需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
基于RPA-CRISPR/Cas12a-SPM和深度学习的DNA病毒检测系统
摘要
我们提出了一种方案,该方案将重组酶聚合酶扩增与CRISPR / Cas12a系统相结合,用于DNA病毒的痕量检测,并构建了具有人工智能辅助分类的便携式智能手机显微镜,用于即时DNA病毒检测。
摘要
我们报告了一种快速、易于实施、高度敏感、序列特异性和即时 (POC) DNA 病毒检测系统,该系统结合了重组酶聚合酶扩增 (RPA) 和 CRISPR/Cas12a 系统,用于 DNA 病毒的痕量检测。靶DNA分别被RPA和CRISPR/Cas12a扩增和识别,触发Cas12a的侧支切割活性,切割荧光团淬灭标记的DNA报告基因并泛化荧光。对于POC检测,便携式智能手机显微镜用于拍摄荧光图像。此外,系统内还部署了深度学习模型,用于正样本或负样本的二元分类,实现了高精度。以青蛙病毒3( FV3,Ranavirus属, 虹膜病毒科)为例,对该DNA病毒POC检测系统进行了检测,40 min内检测限(LoD)可达10 aM。无需熟练的操作人员和笨重的仪器,具有人工智能(AI)辅助分类功能的便携式微型RPA-CRISPR/Cas12a-SPM在POC DNA病毒检测方面显示出巨大的潜力,可以帮助防止此类病毒的传播。
引言
近年来,由不同病毒引起的传染病流行频发,包括2014年1 和2018年2的埃博拉病毒病(EVD)疫情、2015年中东呼吸综合征(MERS)3、2015年寨卡病毒病(4)疫情、2019年由严重急性呼吸系统综合症冠状病毒2(SARS-CoV-2)5引起的冠状病毒病(COVID-19)和 2022年由猴痘病毒(MKPV)引起的持续猴痘6。这些突如其来的流行性传染病的爆发,造成大量死亡,带来巨大的经济损失和社会动荡。迫切需要一个快速、准确的检测系统,以快速诊断感染并防止病毒的进一步传播。
近年来,成簇规则间隔短回文重复序列(CRISPR)和CRISPR相关(Cas)蛋白引起了全世界的关注,并在核酸检测方面显示出有希望的结果7,8,9,10,11,12,13,14,15 .CRISPR/Cas12a 蛋白在 CRISPR RNA (crRNA) 的指导下,与靶 DNA 结合并切割。这种活性导致非特异性单链DNA(ssDNA)的释放,称为反式裂解,可用于增强核酸检测的检测信号。聚合酶链反应 (PCR)、定量实时 PCR (qPCR) 和酶联免疫吸附测定 (ELISA) 等一些传统检测方法对于即时 (POC) 检测来说既复杂又耗时且成本高昂。我们之前的工作成功开发了一种基于CRISPR/Cas12a技术的非洲猪瘟病毒(ASFV)自动化、集成化和具有成本效益的检测系统。在该系统中,我们在 2 小时的时间内实现了 1 pM 的检测限,无需扩增。将CRISPR/Cas12a系统与重组酶聚合酶扩增(RPA)相结合,提高了痕量DNA检测的灵敏度和特异性。与其他等温扩增技术相比,RPA无需复杂的温度控制设备,反应时间更短,操作方便。
对于病原体的 POC 检测,开发了智能手机显微镜 (SPM)、手持式荧光计或侧向层析条等仪器,用于结果读数 16,17,18。SPM通过摄像头捕捉图像,并将其上传到一些移动应用程序以进行快速数据分析。这种显微镜是一种便携式、廉价和小型化的信号采集系统,具有高灵敏度,在检测 H5N1、寨卡病毒和 SARS-CoV-219,20 等病原体方面显示出优势。因此,我们构建了一个便携式SPM来捕获由RPA-CRISPR / Cas12a检测目标DNA病毒触发的荧光信号。当CRISPR/Cas12a识别到目标DNA病毒时,连接荧光团和淬灭基团的ssDNA报告基因探针将被切割,荧光团发出的荧光可以被SPM捕获。
与通常用于从SPM21的荧光图像中获取结果信息的专业软件相比,一些专家在获得荧光图像22后,使用机器学习和深度学习来量化病毒DNA的浓度,这更加耗时。在对医学图像进行分类时,传统神经网络 (CNN) 通常用于以端到端方式从原始像素化图像中学习特征 23,24,25,26。流行的基于 CNN 的深度学习模型,如 AlexNet、DenseNet-121 和 EfficientNet-B7 已成功应用于该领域27,28。然而,在特定领域获取大型数据集可能具有挑战性,需要迁移学习29,30。该方法使用大型数据集预训练深度学习模型,并将预训练模型用作具有小型数据集的新任务的起点。这种技术可以减少对大型数据集的需求,对抗过拟合,并减少训练时间31。在此,我们使用深度学习模型和迁移学习来对阳性和阴性样本的荧光图像进行二元分类。
在该方法中,我们将RPA和CRISPR / Cas12a系统相结合,用于DNA病毒的痕量检测。靶DNA分别被RPA和CRISPR/Cas12a扩增和识别,触发Cas12a的侧支切割活性,切割荧光团淬灭标记的DNA报告基因并泛化荧光。我们构建了一个便携式SPM来获取荧光图像进行POC检测,并开发用于二元分类的深度学习模型。构建的POC检测系统原理图如图 1所示。在没有熟练的操作人员和笨重的仪器的情况下,具有人工智能(AI)辅助分类的RPA-CRISPR/Cas12a-SPM在POC DNA病毒检测方面显示出巨大的潜力。
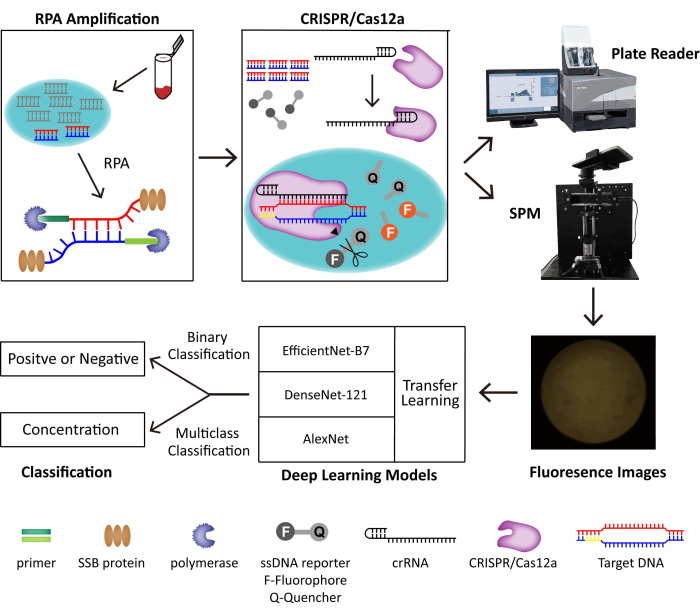
图 1:RPA-CRISPR/Cas12-SPM 检测系统以及用于收集图像的 AI 分类的示意图。 动物源性样本的核酸由PINDBK释放。病毒的靶DNA被RPA-CRISPR/Cas12a系统扩增并特异性识别。CRISPR/Cas12a与crRNA结合,Cas12a-crRNA复合物与靶DNA结合,触发CRISPR/Cas12a在ssDNA报告基因探针上的侧支切割。报告基因上的荧光基团被释放出来,荧光通过商业化的酶标仪或我们制造的 SPM 进行检测。使用三种不同的深度学习模型,包括 AlexNet、DenseNet-121 和具有迁移学习功能的 EfficientNet-B7,对荧光图像进行分类。此图经 Lei et al.35 许可重复使用。 请点击这里查看此图的较大版本.
研究方案
1.样品的处理
- 以青蛙病毒3( FV3,Ranavirus属, 虹膜病毒科)为例,这是一种双链DNA病毒。选择主要衣壳 (mcp) 基因作为 FV3 的检测靶标,因为它高度保守,通常被视为 ranavirus 检测的靶标。选定的目标序列如 表1所示。
注意:青蛙病毒3在此协议中作为示例。 - 对于靶 DNA 片段制备,使用来自 FV3 和传染性脾脏和肾坏死病毒(ISKNV,另一种 Ranavirus)的 mcp 基因的 DNA 片段。
注:在这项研究中,目标DNA片段是从 材料表中列出的一家公司商业获得的。它们被视为后续检测的目标和控制。
| 名字 | 序列 | ||
| FV3 MCP系列 | NTS:5' ...gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg...3' (240 基点) | ||
| TS: 5' ...ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgccgaaagc...3' (240 基点) | |||
| ISKNV MCP(ISKNV MCP) | NTS:5' ...ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg...3' (231 基点) | ||
| TS: 5' ...ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc...3' (231 基点) | |||
表1:此方法中选择的靶序列。
2. RPA反应
- 设计和合成目标序列的 RPA 引物对。RPA引物对的序列如 表2所示。
- 制备5x RPA反应缓冲液(表3)和MgCl2 (100mM)。
- 将四种关键 RPA 酶(UvsX、UvsY、GP32、Bsu 蛋白)混合在 1x RPA 反应缓冲液中,以及预先设计的引物,如 表 4 中所述。
- 彻底涡旋混合物。
- 为每个 RPA 反应添加 1 μL 从步骤 1 获得的靶标,并再次通过涡旋将其充分混合。
- 加入 7 μL MgCl2 (100 mM) 以引发反应。每个 RPA 反应的最终体积为 50 μL。
- 在37°C下进行测定30分钟。
- RPA产品可以在4°C下储存几天。随着RPA产物的降解,应尽快用于进一步检测,以获得更好的诊断结果。
- [可选]进行DNA凝胶电泳。
- 取出 5 μL RPA 产物,加入适当体积的 6x DNA 上样缓冲液,并在 Tris-乙酸 EDTA (TAE) 缓冲液中进行 DNA 凝胶电泳。
- 在 120 V 下电泳约 20 分钟,直到上样缓冲液的条带到达凝胶底部。
- 通过比较样品的条带大小与标记物的条带大小,确定目标序列是否成功从样品中扩增。
| 名字 | 序列 |
| RPA引物F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| RPA引物R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
表 2:该方法中使用的 RPA 引物。
| 元件 | 原始浓度 | 加法 |
| 聚乙二醇 20,000 | - | 114 毫克 |
| ATP酸促成酸酯 | 100毫米 | 125微升 |
| dNTPs的 | 25毫米 | 48微升 |
| Tris-HCl(三盐酸) | 1 米 | 125微升 |
| DTT | 1 米 | 125微升 |
| 磷酸肌酸 | 1 米 | 250微升 |
| 肌酸激酶 | 10微克/微升 | 50微升 |
| ddH2O | - | 277微升 |
| 总体积 | - | 1毫升 |
表3:5x RPA反应缓冲液(pH 7.5)的组成。
| 元件 | 原始浓度 | 加法 |
| 5x RPA反应缓冲液 | - | 10微升 |
| UvsX 蛋白 | 5毫克/毫升 | 2.6微升 |
| UvsY 蛋白 | 5毫克/毫升 | 0.9微升 |
| GP32蛋白 | 5毫克/毫升 | 2.54微升 |
| Bsu 蛋白 | 5毫克/毫升 | 0.88微升 |
| 正向引物 | 100微米 | 0.25微升 |
| 反转引物 | 100微米 | 0.25微升 |
| ddH2O | - | 24.58微升 |
| 目标 | - | 1微升 |
| *氯化镁2 | 100毫米 | 7微升 |
| 总体积 | 50微升 | |
| *最后需要加入 MgCl2 以启动 RPA 反应。 | ||
表4:RPA反应的组成。
3. CRISPR/Cas12a检测,无SPM
- 使用靶序列的 crRNA(材料表)和连接荧光基团和淬灭基团的 ssDNA 报告探针用于 CRISPR/Cas12a。在这里,羧基四甲基罗丹明 (TAMRA) 作为 ssDNA 报告探针 5' 端的荧光团连接,黑洞淬灭剂-2 (BHQ2) 作为 3' 端的淬灭基团。crRNA和ssDNA报告基因的详细序列见 表5。
- 用 10x CRISPR/Cas12a 反应缓冲液制备 Lachnospiraceae 细菌 Cas12a (LbCas12a) 蛋白。
- 将第 2 部分的 1 μL RPA 反应产物溶解在 1x CRISPR/Cas12a 反应缓冲液中,该缓冲液含有 LbCas12a-crRNA 复合物和 500 nM ssDNA 报告探针,反应体积为 100 μL。
- 混合 LbCas12a 和 crRNA 后,让混合物静置至少 5 分钟以形成功能复合物。孵育后,向反应混合物中加入其他组分,并在37°C下进行整个反应。 每个CRISPR / Cas12a反应的最终体积为100μL。 表6描述了每个CRISPR / Cas12a反应中每种组分的详细浓度。
- 在 37 °C 下进行 100 μL CRISPR/Cas12a 检测反应 30 分钟。
- 通过酶标仪在激发波长为535nm和发射波长为595nm,增益为60时不断检查荧光信号。
注:不同波长的激发光和发射光的检测取决于先前设计的 ssDNA 报告探针中荧光团和淬灭基团的选择。 - 对于这些收集的数据,将控制值除以阳性样本的测量值,以对所有数据进行归一化,然后对它们进行积分以进行双样本 t 检验分析。
| 名字 | 序列 | |
| 用于 FV3 的 LbCas12a crRNA | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| ssDNA报告基因 | /5TAMRA/TTATT/3BHQ2 | |
表5:该方法中使用的CRISPR/Cas12a crRNA和ssDNA报告基因的序列。
| 元件 | 原始浓度 | 加法 |
| NEuffer r2.1 | - | 10微升 |
| LBA Cas12a (CPF1) | 10微米 | 0.5微升 |
| crRNA的 | 10微米 | 0.625微升 |
| 等待至少 5 分钟,让 LbCas12a/crRNA 复合物结合。 | ||
| DNA报告基因 | 100微米 | 0.5微升 |
| ddH2O | - | 87.375微升 |
| 目标 | - | 1微升 |
| 总体积 | - | 100微升 |
表 6:CRISPR/Cas12a 反应的组成。
4. SPM 设置
- 对于激发路径,设置激光束穿过中性密度 (ND) 滤光片以衰减激光强度。
- 从非球面透镜(以下简称L1)产生准直光束,并通过二向色镜(DM)将其反射。
- 将光线引导到载玻片上,在载玻片上将样品通过物镜 (20x) 以照亮并激发样品的荧光。样品台可实现精确的焦平面调整,将光束引导至物镜的后焦平面。上述步骤构成了 SPM 仪器的激励路径。
- 对于发射路径,放置一个外部透镜(以下简称L2),以在物镜的另一侧形成一个中间像。物镜同时照亮样品并收集发射信号。
- 使用放置在发射路径末端的智能手机记录来自样品的荧光信号。使用稳定的支架以避免摇晃。
- 在 L1 和智能手机的摄像头之间设置一个带通滤光片,以滤除激发的光,同时只允许样品发射的光到达摄像头,这可以优化检测。
- 将 SPM 设置固定在面包板上,以便进行便携式部署。
注:基于 RPA-CRISPR/Cas12a 反应的荧光检测的 SPM 设备的原理图和物理外观如图 2 所示。CRISPR/Cas12a检测在下一步所述的预处理过的载玻片上进行。
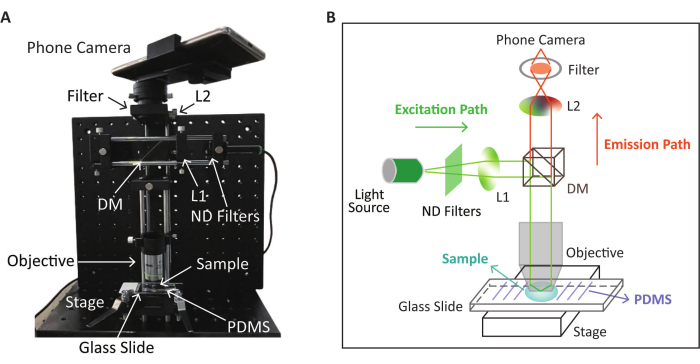
图 2:用于荧光检测的 SPM 设备的原理图和物理外观。 (A) RPA-CRISPR/Cas12a反应后用于荧光图像采集的SPM设备的物理外观。(B)基于RPA-CRISPR/Cas12a反应的SPM荧光检测装置示意图。该图经 Lei 等人许可修改(调整图像位置和颜色)[35。 请点击这里查看此图的较大版本.
5. SPM检测用载玻片的处理
- 通过将碱和固化剂以10:1的比例混合来制备聚二甲基硅氧烷(PDMS),然后在80°C的加热板上烘烤2小时。
- 处理PDMS和载玻片(长度:75 mm;高度:50 毫米)用氧等离子体处理 120 秒,然后将它们压在一起。
- 将玻璃/ PDMS在95°C烘烤2小时;它通过Si-O-Si键永久密封。PDMS透明度高,无自发荧光,有利于SPM检测。载玻片和PMDS的处理按照He等人32进行
6. CRISPR/Cas12a检测与SPM
- 使用靶序列的 crRNA(材料表)和连接荧光基团和淬灭基团的 ssDNA 报告探针用于 CRISPR/Cas12a。在该方法中,羧基四甲基罗丹明(TAMRA)作为ssDNA报告探针的5'端的荧光基团连接,黑洞淬灭剂-2(BHQ2)作为3'端的淬灭基团。crRNA和ssDNA报告基因的详细序列见 表5。
- 用 10x CRISPR/Cas12a 反应缓冲液制备 Lachnospiraceae 细菌 Cas12a (LbCas12a) 蛋白。
- 将第 2 部分的 1 μL RPA 反应产物溶解在 1x CRISPR/Cas12a 反应缓冲液中,该缓冲液含有 LbCas12a-crRNA 复合物和 500 nM ssDNA 报告探针,反应体积为 100 μL。
- 混合 LbCas12a 和 crRNA 后,让混合物静置至少 5 分钟以形成功能复合物。孵育后,向反应混合物中加入其他组分,并在37°C下完成反应。 每个CRISPR / Cas12a反应的最终体积为100μL。 表6描述了每个CRISPR / Cas12a反应中每种组分的详细浓度。
- 在预处理的载玻片上进行100μL CRISPR / Cas12a检测反应,并用盖玻片覆盖。将载玻片与室温反应孵育 10 分钟。
- 通过SPM测量荧光信号。将带有检测反应的载玻片放在SPM的载物台上,保持适当的距离,调整焦距和清晰度,然后寻找反应的视野并聚焦以捕获图像。
注意:应首先获得标准曲线,以便可以将样品的数据缩放到近似浓度范围。使用各种浓度的纯化靶标,包括 10 nM、1 nM、100 pM 和 10 pM,另一种病毒作为阴性对照(RPA 之前)。
7. 数据集和数据增强
- 从第 6 节中的检测测定中收集荧光图像作为数据集。对每个样本至少重复三次平行检测,以确保数据平行性。
- 可以批准一些实现更高并行度的适当方法。例如,在收集每个样品的荧光图像时,手动对焦并寻找相对更亮的场来拍摄图像。同时,对每个样品进行拍照,以获得超过五个不同位置的荧光信号。
- 通过 ImageJ 测量每幅图像的平均灰度值和浓度组中平均灰度值的标准差。
- 设置数据清洗的强度范围[中位数-标准差,中位数+标准差]。
注意:在早期步骤获得的图像中,可能存在差异较大的图像,因此有必要对图像进行筛选。如果图像的强度超出设定的阈值,则应将其视为异常值并排除在外。 - 分别以升序标记浓度增加的纯化目标的图像,浓度为0-6。
- 为了增强系统的鲁棒性并防止过拟合,可以通过在Python中的变换函数实现水平翻转、垂直翻转和随机噪声等图像增强技术。这有助于在数据集中引入变化。
8. 迁移学习
- 作为骨干网络,采用深度学习模型AlexNet33 进行分类。
- 为了满足第 7 节中预训练模型的约束,通过 Python 中的变换函数将输入图像重塑为 224 像素 x 224 像素 x 3 个通道(高度和宽度为 224 像素,红色、绿色和蓝色通道的深度为 3 个通道)。
注意:此步骤是迁移学习中异构数据的常见预处理,包括转换。 - 将预训练的骨干网络与 ImageNet 数据集结合使用来提取特征,同时利用学习到的中间隐藏层的权重。
- 在荧光分类作业上下文中,将神经网络的最后一个全连接层(最初包括 ImageNet 任务的 1000 个神经元)替换为具有 2 个或 7 个神经元的全连接层。
- 使用一系列指标评估设置训练模型的性能,包括混淆矩阵、准确性、精确度、召回率和基于 Lawton 和 Viriri34 的 F1 分数。
结果
该方法侧重于一种快速、易于实施、高度敏感的 DNA 病毒即时 (POC) 检测系统。RPA反应的引物对设计和CRISPR/Cas12a反应的crRNA设计是其中两个重要部分,因为它们会影响RPA-CRISPR/Cas12a反应的效率,并影响后续的检测和分类。
在该方法中,FV3被认为是DNA病毒检测的一个例子。设计了一些用于FV3的RPA引物对,并选择最有效的一种,并确保其显示出良好的扩增效率,?...
讨论
在这种方法中,我们开发了一种快速、易于实现、高灵敏度、序列特异性和 POC DNA 病毒检测系统,并具有 AI 辅助。获得样本后,应用RPA对目标序列进行扩增,然后CRISPR/Cas12a可以识别目标DNA并释放荧光,从而放大检测信号。便携式智能手机显微镜用于拍摄荧光图像,具有迁移学习功能的深度学习模型用于正负样本图像的二元分类。该方法以FV3为阳性样本,从多个角度进行研究,以提高所提系统的?...
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家自然科学基金31970752、深圳市科学技术创新委员会JCYJ20190809180003689、JSGG20200225150707332、JSGG20191129110812708、WDZC20200820173710001的支持;深圳湾实验室开放资助,SZBL2020090501004;中国博士后科学基金 2020M680023;中华人民共和国海关总署 2021HK007。
材料
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
参考文献
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。