Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Система обнаружения ДНК-вирусов на основе RPA-CRISPR/Cas12a-SPM и глубокого обучения
В этой статье
Резюме
Мы представляем протокол, который сочетает амплификацию рекомбиназной полимеразы с системой CRISPR/Cas12a для обнаружения следов ДНК-вирусов и создает портативную микроскопию смартфона с классификацией с помощью искусственного интеллекта для обнаружения ДНК-вирусов в месте оказания медицинской помощи.
Аннотация
Мы сообщаем о быстрой, простой в внедрении, высокочувствительной, последовательно-специфичной и ориентированной на место оказания медицинской помощи (POC) системе обнаружения ДНК-вирусов, которая сочетает в себе амплификацию рекомбиназной полимеразы (RPA) и систему CRISPR/Cas12a для обнаружения следов ДНК-вирусов. ДНК-мишень амплифицируется и распознается RPA и CRISPR/Cas12a по отдельности, что запускает активность коллатерального расщепления Cas12a, которая расщепляет меченный флуорофором репортер ДНК и генерализует флуоресценцию. Для обнаружения POC портативная микроскопия смартфона создана для получения флуоресцентных изображений. Кроме того, в системе развернуты модели глубокого обучения для бинарной классификации положительных или отрицательных образцов, достигающие высокой точности. В качестве примера для этой системы обнаружения POC ДНК-вируса был протестирован вирус лягушки 3 (FV3, роды Ranavirus, семейство Iridoviridae), и пределы обнаружения (LoD) могут достигать 10 aM в течение 40 минут. Без квалифицированных операторов и громоздких инструментов портативный и миниатюрный RPA-CRISPR/Cas12a-SPM с классификацией с помощью искусственного интеллекта (ИИ) демонстрирует большой потенциал для обнаружения ДНК-вирусов POC и может помочь предотвратить распространение таких вирусов.
Введение
В последние годы часто происходили эпидемии инфекционных заболеваний, вызываемых различными вирусами, в том числе эпидемия болезни, вызванной вирусом Эбола (БВВЭ), в 2014 г.1 и 2018 г.2, ближневосточный респираторный синдром (БВРС) в 2015 г.3, эпидемия болезни, вызванной вирусом Зика, в 2015 г.4, коронавирусная болезнь 2019 г. (COVID-19), вызванная коронавирусом тяжелого острого респираторного синдрома 2 (SARS-CoV-2)5 , и продолжающаяся оспа обезьян, вызванная вирусом оспы обезьян (MKPV), в 2022 г.6. Эти внезапные вспышки эпидемических инфекционных заболеваний приводят к большому количеству смертей, огромным экономическим потерям и социальным волнениям. Для быстрой диагностики инфекции и предотвращения дальнейшего распространения вируса срочно требуется система быстрого и точного обнаружения.
В последнее время кластеризованные регулярно чередующиеся короткие палиндромные повторы (CRISPR) и CRISPR-ассоциированные (Cas) белки привлекли внимание всего мира и показали многообещающие результаты в обнаружении нуклеиновых кислот 7,8,9,10,11,12,13,14,15 . Белок CRISPR/Cas12a, управляемый CRISPR RNA (crRNA), связывается с целевой ДНК и расщепляет ее. Эта активность приводит к высвобождению неспецифической одноцепочечной ДНК (ssDNA), известной как транс-расщепление, и может быть использована для усиления сигнала обнаружения нуклеиновых кислот. Некоторые традиционные методы обнаружения, такие как полимеразная цепная реакция (ПЦР), количественная ПЦР в реальном времени (кПЦР) и иммуноферментный анализ (ИФА), являются сложными, трудоемкими и дорогостоящими для обнаружения в месте оказания медицинской помощи (POC). В нашей предыдущей работе была успешно разработана автоматизированная, интегрированная и экономически эффективная система обнаружения вируса африканской чумы свиней (АЧС) на основе технологии CRISPR/Cas12a. В этой системе мы достигли предела обнаружения 1 пМ в течение 2 часов без необходимости усиления. Система CRISPR/Cas12a и амплификация рекомбиназной полимеразы (RPA) объединены для повышения чувствительности и специфичности для обнаружения следов ДНК. По сравнению с другими методами изотермической амплификации, RPA прост в конструкции и удобен в эксплуатации, поскольку имеет более короткое время реакции без сложного оборудования для контроля температуры.
Для обнаружения патогенов с помощью POC разработаны такие приборы, как микроскопия смартфона (SPM), портативный флуориметр или полоски бокового потока для считывания результатов 16,17,18. SPM захватывает изображения через камеру и загружает их в некоторые мобильные приложения для быстрого анализа данных. Такая микроскопия делает портативную, дешевую и миниатюрную систему сбора сигналов с высокой чувствительностью и показала преимущества в обнаружении патогенов, таких как H5N1, вирус Зика и SARS-CoV-219,20. Поэтому мы создаем портативный СЗМ для улавливания сигналов флуоресценции, вызванных обнаружением RPA-CRISPR/Cas12a целевого ДНК-вируса. Репортерный зонд ssDNA, связывающий флуорофор и гаситель, будет расщеплен, когда CRISPR/Cas12a распознает целевой ДНК-вирус, и флуоресценция, испускаемая флуорофором, может быть захвачена СЗМ.
По сравнению с профессиональным программным обеспечением, обычно используемым для получения информации о результатах из флуоресцентных изображений SPM21, некоторые эксперты используют машинное обучение и глубокое обучение для количественной оценки концентраций ДНК вируса после получения флуоресцентных изображений22, что занимает больше времени. Когда дело доходит до классификации медицинских изображений, обычные нейронные сети (СНС) часто используются для изучения особенностей необработанных пиксельных изображений сквозным образом 23,24,25,26. Популярные модели глубокого обучения на основе CNN, такие как AlexNet, DenseNet-121 и EfficientNet-B7, были успешно применены вэтой области. Однако получение больших наборов данных в конкретных областях может быть сложной задачей, требующей трансферного обучения29,30. Этот подход предварительно обучает модель глубокого обучения с большим набором данных, а предварительно обученная модель используется в качестве отправной точки для новой задачи с небольшим набором данных. Этот метод может снизить потребность в больших наборах данных, боевом переобучении и сократить время обучения31. Здесь мы используем модели глубокого обучения с трансферным обучением для бинарной классификации флуоресцентных изображений положительных и отрицательных образцов.
В этом методе мы объединяем RPA и систему CRISPR/Cas12a для обнаружения следов ДНК-вирусов. ДНК-мишень амплифицируется и распознается RPA и CRISPR/Cas12a по отдельности, что запускает активность коллатерального расщепления Cas12a, которая расщепляет меченный флуорофором репортер ДНК и генерализует флуоресценцию. Мы создаем портативный SPM для получения флуоресцентных изображений для обнаружения POC и разрабатываем модели глубокого обучения для двоичной классификации. Схема построенной системы обнаружения POC показана на рисунке 1. Без квалифицированных операторов и громоздких инструментов RPA-CRISPR/Cas12a-SPM с классификацией с помощью искусственного интеллекта (ИИ) демонстрирует большой потенциал для обнаружения ДНК-вирусов POC.
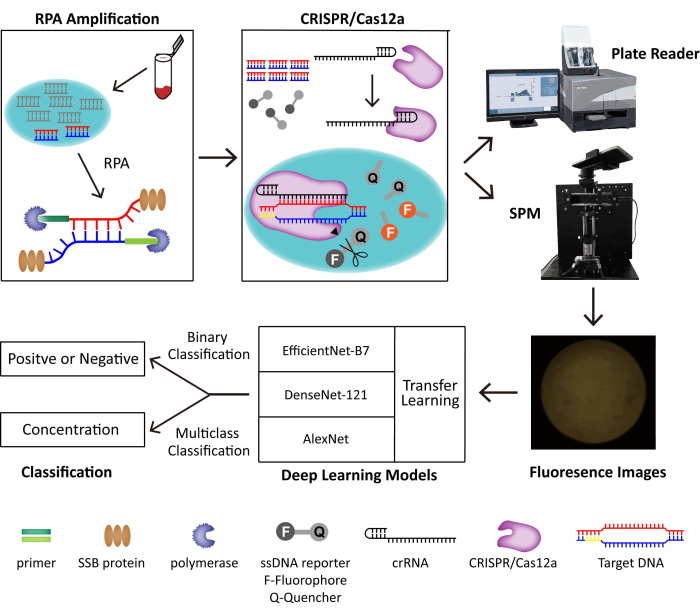
Рисунок 1: Схема системы обнаружения RPA-CRISPR/Cas12-SPM вместе с классификацией ИИ для собранных изображений. Нуклеиновые кислоты образцов животного происхождения высвобождаются ПИНДБК. ДНК-мишень вируса амплифицируется и распознается специфически системой RPA-CRISPR/Cas12a. CRISPR/Cas12a связывается с crRNA, а комплекс Cas12a-crRNA связывается с ДНК-мишенью, что запускает коллатеральное расщепление CRISPR/Cas12a на репортерных зондах ssDNA. Флуорофор на репортере высвобождается, и флуоресценция обнаруживается коммерческим считывателем пластин или SPM, который мы создаем. Для классификации флуоресцентных изображений используются три различные модели глубокого обучения, включая AlexNet, DenseNet-121 и EfficientNet-B7 с трансферным обучением. Этот рисунок повторно используется с разрешения Lei et al.35. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
протокол
1. Обработка образцов
- Возьмем, к примеру, вирус лягушки 3 (FV3, род Ranavirus, семейство Iridoviridae), вирус с двухцепочечной ДНК. Выберите ген главного капсида (MCP) в качестве мишени для обнаружения FV3, поскольку он высоко консервативен и обычно рассматривается как мишень для обнаружения ранавируса. Выбранная целевая последовательность показана в таблице 1.
ПРИМЕЧАНИЕ: В качестве примера в этом протоколе взят Frog Virus 3. - Для получения фрагментов ДНК-мишени используют фрагменты ДНК гена mcp из FV3 и вируса инфекционного некроза селезенки и почек (ISKNV, другой ранавирус).
Примечание: В этом исследовании целевые фрагменты ДНК были коммерчески получены от компании, указанной в Таблице материалов. Они рассматриваются как цель и контроль последующего обнаружения.
| Имя | Последовательность | ||
| ФВ3 МКП | NTS: 5' ... gtaacccggctttcGGGCAGCAGTTTTTCGGTCGGCGTtcccaggtcg... 3' (240.н.) | ||
| ТС: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240.н.) | |||
| МКП ИСКНВ | NTS: 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGACGgtggcgaggg... 3' (231.н.) | ||
| ТС: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231.н.) | |||
Таблица 1: Целевая последовательность, выбранная в этом методе.
2. Реакция RPA
- Проектирование и синтез пар праймеров RPA для целевой последовательности. Последовательности пар праймеров RPA описаны в таблице 2.
- Приготовьте реакционный буфер 5x RPA (табл. 3) и MgCl2 (100 мМ).
- Смешайте четыре ключевых фермента RPA (UvsX, UvsY, GP32, белок Bsu) в реакционном буфере 1x RPA вместе с праймерами, разработанными заранее, как подробно описано в таблице 4.
- Тщательно перемешайте смесь.
- Добавьте 1 мкл мишени, полученной на стадии 1, для каждой реакции RPA и снова тщательно перемешайте ее в вихре.
- Добавьте 7 мкл MgCl2( 100 мМ), чтобы начать реакцию. Конечный объем каждой реакции RPA составляет 50 мкл.
- Проводить анализ при 37 °C в течение 30 мин.
- Продукт RPA можно хранить при температуре 4 °C в течение нескольких дней. По мере разложения продуктов RPA используйте их для дальнейшего обнаружения как можно скорее, чтобы получить лучшие результаты диагностики.
- [ Опционально ] Провести электрофорез ДНК-гелем.
- Извлеките 5 μL продуктов RPA, добавьте соответствующий объем 6-кратного буфера для загрузки ДНК и проведите электрофорез в ДНК-гель-электрофорезе в трисацетатном буфере ЭДТА (TAE).
- Запустите электрофорез при напряжении 120 В в течение примерно 20 минут, пока полосы загрузочного буфера не достигнут дна геля.
- Определить, успешно ли амплифицирована последовательность-мишень из образца, сравнив размер полос образца с полосами маркера.
| Имя | Последовательность |
| Грунтовка RPA F | ATGTCTCTCTGTAACTGGTTCAGGTATCACA |
| Грунтовка RPA R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Таблица 2: Праймеры RPA, используемые в этом методе.
| Компонент | Исходная концентрация | Сложение |
| PEG 20,000 | - | 114 мг |
| СПС | 100 мМ | 125 μл |
| dNTP | 25 мМ | 48 μл |
| Трис-HCl | 1 М | 125 μл |
| ДТТ | 1 М | 125 μл |
| Фосфокреатин | 1 М | 250 μл |
| Креатинкиназа | 10 μг/μл | 50 μл |
| ddH2O | - | 277 μл |
| Общий объем | - | 1 мл |
Таблица 3: Состав реакционного буфера 5x RPA (pH 7,5).
| Компонент | Исходная концентрация | Сложение |
| 5x реакционный буфер RPA | - | 10 μл |
| Белок UvsX | 5 мг/мл | 2,6 мкл |
| Белок UvsY | 5 мг/мл | 0,9 мкл |
| Белок GP32 | 5 мг/мл | 2,54 мкл |
| Белок БГУ | 5 мг/мл | 0,88 мкл |
| Передний праймер | 100 μМ | 0,25 мкл |
| Перевернутый праймер | 100 μМ | 0,25 мкл |
| ddH2O | - | 24,58 μл |
| Цель | - | 1 μл |
| *MgCl2 | 100 мМ | 7 μл |
| Общий объем | 50 μл | |
| *MgCl2 необходимо добавить в последнюю очередь, чтобы инициировать реакцию RPA. | ||
Таблица 4: Состав реакции РПА.
3. Обнаружение CRISPR/Cas12a без СЗМ
- Используйте crRNA (Table of Materials) последовательности-мишени и репортерный зонд ssDNA, связывающий флуорофор и гаситель для CRISPR/Cas12a. Здесь карбокситетраметилродамин (TAMRA) связан как флуорофор на 5'-концах репортерных зондов ssDNA, а гаситель черной дыры-2 (BHQ2) - как гаситель на 3'-концах. Подробная последовательность репортера crRNA и ssDNA описана в таблице 5.
- Приготовьте белок бактерии Lachnospiraceae Cas12a (LbCas12a) с 10-кратным реакционным буфером CRISPR/Cas12a.
- Растворите 1 мкл продукта реакции RPA из секции 2 в 1x реакционном буфере CRISPR/Cas12a с комплексами LbCas12a-crRNA и репортерным зондом 500 нМ ssDNA в реакционном объеме 100 мкл.
- После смешивания LbCas12a и crRNA дайте смеси постоять не менее 5 минут, чтобы образовался функциональный комплекс. После инкубации добавьте в реакционную смесь другие компоненты и проведите всю реакцию при температуре 37 °C. Конечный объем каждой реакции CRISPR/Cas12a составляет 100 мкл. Подробные концентрации каждого компонента в каждой реакции CRISPR/Cas12a описаны в таблице 6.
- Проведите реакцию детектирования CRISPR/Cas12a объемом 100 мкл при 37 °C в течение 30 мин.
- Постоянно исследуйте флуоресцентные сигналы с помощью считывателя микропланшетов на длине волны возбуждения 535 нм и длине волны излучения 595 нм с коэффициентом усиления 60.
ПРИМЕЧАНИЕ: Обнаружение различных длин волн возбуждения и излучения света зависит от выбора флуорофора и гасителя в ранее разработанных репортерных зондах ssDNA. - Для этих собранных данных разделите контрольное значение на измерение положительных образцов, чтобы нормализовать все данные, а затем интегрируйте их для анализа t-критерия с двумя выборками.
| Имя | Последовательность | |
| LbCas12a кРНК для FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| Репортер ssDNA | /5ТАМРА/ТАТТ/3BHQ2 | |
Таблица 5: Последовательности CRISPR/Cas12a crRNA и ssDNA reporter, используемые в этом методе.
| Компонент | Исходная концентрация | Сложение |
| NEBuffer r2.1 | - | 10 μл |
| Lba Cas12a (Cpf1) | 10 μМ | 0,5 мкл |
| крРНК | 10 μМ | 0,625 мкл |
| Подождите не менее 5 минут, чтобы комплекс LbCas12a/crRNA объединился. | ||
| Репортер ДНК | 100 μМ | 0,5 мкл |
| ddH2O | - | 87,375 μл |
| Цель | - | 1 μл |
| Общий объем | - | 100 μл |
Таблица 6: Состав реакции CRISPR/Cas12a.
4. Настройка СЗМ
- Для пути возбуждения установите лазерный луч, проходящий через фильтры нейтральной плотности (ND), чтобы ослабить интенсивность лазера.
- Сформировать коллимированный пучок от асферической линзы (далее L1) и отразить его дихроичным зеркалом (ДМ).
- Направьте свет на предметное стекло, куда помещается образец через объектив (20x), чтобы осветить и возбудить флуоресценцию образца. Предметный столик позволяет точно регулировать фокальную плоскость, направляя луч в заднюю фокальную плоскость объектива. Указанные выше шаги формируют путь возбуждения СЗМ-аппарата.
- Для пути излучения расположите внешнюю линзу (далее L2) так, чтобы сформировать промежуточное изображение с другой стороны объектива. Объектив одновременно освещает образец и собирает сигнал излучения.
- Запишите сигнал флуоресценции от образца с помощью смартфона, расположенного в конце пути излучения. Используйте устойчивый кронштейн, чтобы избежать тряски.
- Установите полосовой фильтр между L1 и камерой смартфона, чтобы отфильтровать возбужденный свет, позволяя только свету, излучаемому образцом, достигать камеры, что может оптимизировать обнаружение.
- Иммобилизуйте установку SPM на макетной плате для переносного развертывания.
ПРИМЕЧАНИЕ: Схема и внешний вид устройства SPM для флуоресцентного детектирования на основе реакции RPA-CRISPR/Cas12a показаны на рисунке 2. Обнаружение CRISPR/Cas12a происходит на предварительно обработанном предметном стекле, описанном на следующем этапе.
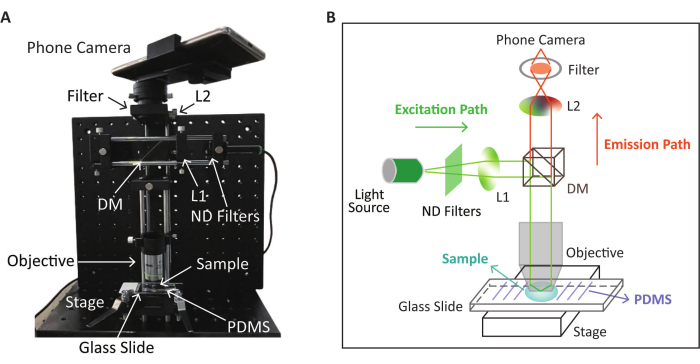
Рисунок 2: Схематический и физический внешний вид устройства SPM, используемого для обнаружения флуоресценции. (A) Внешний вид устройства SPM для сбора флуоресцентных изображений после реакции RPA-CRISPR/Cas12a. (B) Схема устройства SPM для флуоресцентного детектирования на основе реакции RPA-CRISPR/Cas12a. Этот рисунок изменен (скорректировано положение и цвет изображения) с разрешения Lei et al.35. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
5. Обработка предметного стекла для детектирования с помощью СЗМ
- Приготовить полидиметилсилоксан (PDMS) путем смешивания основы и отвердителя в соотношении 10:1 с последующим запеканием на конфорке при 80 °C в течение 2 ч.
- Обработать как PDMS, так и предметное стекло (длина: 75 мм; Высота: 50 мм) с кислородной плазменной обработкой в течение 120 с, затем прижимают их друг к другу.
- Выпекать стекло/PDMS при 95 °C в течение 2 часов; он навсегда герметизирован соединением Si-O-Si. PDMS имеет высокую прозрачность и отсутствие автофлуоресценции, что способствует обнаружению СЗМ. Лечение предметного стекла и PMDS выполняется в соответствии с He et al.32
6. Обнаружение CRISPR/Cas12a с помощью SPM
- Используйте crRNA (Table of Materials) последовательности-мишени и репортерный зонд ssDNA, связывающий флуорофор и гаситель для CRISPR/Cas12a. В этом методе карбокситетраметилродамин (TAMRA) связывают в качестве флуорофора на 5'-концах репортерных зондов ssDNA, а гаситель черных дыр-2 (BHQ2) - в качестве гасителя на 3'-концах. Подробная последовательность репортера crRNA и ssDNA описана в таблице 5.
- Приготовьте белок бактерии Lachnospiraceae Cas12a (LbCas12a) с 10-кратным реакционным буфером CRISPR/Cas12a.
- Растворите 1 мкл продукта реакции RPA из секции 2 в 1x реакционном буфере CRISPR/Cas12a с комплексами LbCas12a-crRNA и репортерным зондом 500 нМ ssDNA в реакционном объеме 100 мкл.
- После смешивания LbCas12a и crRNA дайте смеси постоять не менее 5 минут, чтобы образовался функциональный комплекс. После инкубации добавьте в реакционную смесь другие компоненты и завершите реакцию при 37 °C. Конечный объем каждой реакции CRISPR/Cas12a составляет 100 мкл. Подробные концентрации каждого компонента в каждой реакции CRISPR/Cas12a описаны в таблице 6.
- Выполните реакцию обнаружения CRISPR/Cas12a объемом 100 мкл на предварительно обработанном предметном стекле и накройте его покровным стеклом. Инкубировать предметное стекло с реакцией при RT в течение 10 мин.
- Измерение сигналов флуоресценции с помощью СЗМ. Поместите предметное стекло с реакцией обнаружения на столик СЗМ, соблюдайте соответствующее расстояние, отрегулируйте фокусное расстояние и четкость, затем найдите поле зрения реакции и сфокусируйте его для захвата изображения.
ПРИМЕЧАНИЕ: Сначала следует получить стандартную кривую, чтобы данные проб можно было масштабировать до приблизительного диапазона концентраций. Используются различные концентрации очищенных мишеней, в том числе 10 нМ, 1 нМ, 100 пМ и 10 пМ, другой вирус в качестве отрицательного контроля (до РПА).
7. Расширение наборов данных и данных
- Соберите флуоресцентные изображения из анализа обнаружения в разделе 6 в виде наборов данных. Повторите по крайней мере три параллельных обнаружения для каждого образца, чтобы обеспечить параллелизм данных.
- Могут быть одобрены некоторые подходящие способы достижения более высокого параллелизма. Например, при сборе флуоресцентных изображений из каждого образца вручную сфокусируйтесь и найдите относительно более яркое поле для получения изображений. В то же время сфотографируйте каждый образец, чтобы получить сигнал флуоресценции в более чем пяти разных местах.
- Измерение среднего значения серого каждого изображения и стандартного отклонения среднего значения серого в группе концентраций с помощью ImageJ.
- Установите диапазон интенсивностей [медиана - стандартное отклонение, медиана + стандартное отклонение] для очистки данных.
ПРИМЕЧАНИЕ: На изображениях, полученных на ранних этапах, могут быть изображения с большими различиями, поэтому необходимо экранировать изображения. Если интенсивность изображений выходит за пределы установленного порога, их следует рассматривать как выбросы и исключать. - Пометьте изображения очищенной мишени при возрастающих концентрациях 0-6 в порядке возрастания, соответственно.
- Чтобы повысить надежность системы и предотвратить переобучение, реализуйте методы дополнения изображений, такие как горизонтальное отражение, вертикальное отражение и случайный шум, с помощью функций преобразования в Python. Это помогает вносить изменения в набор данных.
8. Трансферное обучение
- В качестве магистральной сети используйте модель глубокого обучения AlexNet33 для классификации.
- Чтобы удовлетворить ограничение предварительно обученной модели в разделе 7, измените форму входных изображений до 224 x 224 пикселей x 3 канала (высота и ширина 224 пикселя и глубина 3 канала для красного, зеленого и синего цветовых каналов) с помощью функций преобразования в Python.
ПРИМЕЧАНИЕ: Этот шаг является обычной предварительной обработкой для разнородных данных при передаче обучения, включая преобразование. - Используйте предварительно обученную магистральную сеть с набором данных ImageNet для извлечения объектов, используя веса изученных промежуточных скрытых слоев.
- В контексте задания флуоресцентной классификации замените последний полносвязный слой нейронной сети, который изначально включал 1000 нейронов для задачи ImageNet, на полносвязный слой с 2 или 7 нейронами.
- Оцените производительность модели обучения настройки с помощью ряда метрик, включая матрицу несоответствий, точность, прецизионность, полноту и оценку F1 на основе Лоутона и Вирири34.
Результаты
Этот метод основан на быстрой, простой в реализации, высокочувствительной системе обнаружения ДНК-вирусов (POC). Дизайн пар праймеров для реакции RPA и дизайн crRNA для реакции CRISPR/Cas12a являются двумя важными частями, поскольку они влияют на эффективность реакции RPA-CRISPR/Cas12a и влияют на последу?...
Обсуждение
В этом методе мы разрабатываем быструю, простую в реализации, высокочувствительную, последовательно-специфичную и POC-систему обнаружения ДНК-вирусов с помощью искусственного интеллекта. После получения образцов применяется RPA для амплификации последовательности-мишени, а затем CRISPR/Cas...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа поддерживается Национальным фондом естественных наук Китая 31970752, наука, технологии, Комиссия по инновациям муниципалитета Шэньчжэня JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Открытое финансирование лаборатории залива Шэньчжэнь, SZBL2020090501004; Китайский фонд постдокторантуры 2020M680023; и Главное таможенное управление Китайской Народной Республики 2021HK007.
Материалы
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Ссылки
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены