Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Sistema de detección de virus de ADN basado en RPA-CRISPR/Cas12A-SPM y Deep Learning
En este artículo
Resumen
Presentamos un protocolo que combina la amplificación de la polimerasa recombinasa con un sistema CRISPR/Cas12a para la detección de trazas de virus de ADN y construye una microscopía portátil para teléfonos inteligentes con una clasificación asistida por inteligencia artificial para la detección de virus de ADN en el punto de atención.
Resumen
Presentamos un sistema de detección de virus de ADN rápido, fácil de implementar, altamente sensible, específico de secuencia y en el punto de atención (POC), que combina la amplificación de la polimerasa recombinasa (RPA) y el sistema CRISPR/Cas12a para la detección de trazas de virus de ADN. El ADN objetivo es amplificado y reconocido por RPA y CRISPR/Cas12a por separado, lo que desencadena la actividad de escisión colateral de Cas12a que escinde un reportero de ADN marcado con extintores de fluoróforos y generaliza la fluorescencia. Para la detección de POC, la microscopía portátil de los teléfonos inteligentes está diseñada para tomar imágenes fluorescentes. Además, se despliegan dentro del sistema modelos de aprendizaje profundo para la clasificación binaria de muestras positivas o negativas, logrando una alta precisión. El virus de la rana 3 (FV3, géneros Ranavirus, familia Iridoviridae) se probó como ejemplo para este sistema de detección POC de virus de ADN, y los límites de detección (LoD) pueden alcanzar 10 aM en 40 minutos. Sin operadores cualificados ni instrumentos voluminosos, el RPA-CRISPR/Cas12a-SPM portátil y en miniatura con clasificación asistida por inteligencia artificial (IA) muestra un gran potencial para la detección de virus POC DNA y puede ayudar a prevenir la propagación de dichos virus.
Introducción
En los últimos años, se han producido con frecuencia epidemias de enfermedades infecciosas causadas por diferentes virus, como la epidemia de enfermedad por el virus del Ébola (EVE) en 20141 y 20182, el síndrome respiratorio de Oriente Medio (MERS) en 20153, la epidemia de enfermedad por el virus del Zika en 20154, la enfermedad por coronavirus 2019 (COVID-19) causada por el coronavirus 2 del síndrome respiratorio agudo grave (SARS-CoV-2)5 y la continuación de la viruela del mono causada por el virus de la viruela del mono (MKPV) en 20226. Estos brotes repentinos de enfermedades infecciosas epidémicas causan un gran número de muertes y traen enormes pérdidas económicas y disturbios sociales. Se requiere urgentemente un sistema de detección rápido y preciso para diagnosticar rápidamente la infección y evitar una mayor propagación del virus.
Recientemente, las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) y las proteínas asociadas a CRISPR (Cas) han ganado atención mundial y han mostrado resultados prometedores en la detección de ácidos nucleicos 7,8,9,10,11,12,13,14,15 . La proteína CRISPR/Cas12a, guiada por el ARN CRISPR (crRNA), se une y escinde el ADN diana. Esta actividad conduce a la liberación de ADN monocatenario inespecífico (ssDNA), conocido como trans-escisiones, y se puede utilizar para mejorar la señal de detección para la detección de ácidos nucleicos. Algunos métodos de detección tradicionales, como la reacción en cadena de la polimerasa (PCR), la PCR cuantitativa en tiempo real (qPCR) y el ensayo de inmunoabsorción enzimática (ELISA), son complicados, lentos y costosos para la detección en el punto de atención (POC). Nuestro trabajo anterior desarrolló con éxito un sistema de detección automatizado, integrado y rentable para el virus de la peste porcina africana (PPA) basado en la tecnología CRISPR/Cas12a. En este sistema, logramos un límite de detección de 1 pM en un período de tiempo de 2 horas sin necesidad de amplificación. El sistema CRISPR/Cas12a y la amplificación de la polimerasa recombinasa (RPA) se combinan para mejorar la sensibilidad y la especificidad de la detección de trazas de ADN. En comparación con otras técnicas de amplificación isotérmica, RPA tiene un diseño simple y una operación conveniente, ya que tiene un tiempo de reacción más corto sin un equipo sofisticado de control de temperatura.
Para la detección de patógenos POC, se desarrollan instrumentos como la microscopía de teléfono inteligente (SPM), el fluorímetro de mano o las tiras de flujo lateral para las lecturas de resultados 16,17,18. SPM captura imágenes a través de una cámara y las sube a algunas aplicaciones móviles para un análisis rápido de los datos. Dicha microscopía es un sistema de adquisición de señales portátil, barato y en miniatura con alta sensibilidad y ha demostrado ventajas en la detección de patógenos como el H5N1, el virus del Zika y el SARS-CoV-219,20. Por lo tanto, construimos un SPM portátil para captar las señales de fluorescencia desencadenadas por la detección de RPA-CRISPR/Cas12a del virus de ADN objetivo. La sonda reportera de ssDNA que conecta un fluoróforo y un extintor, se escindirá cuando CRISPR/Cas12a reconozca el virus de ADN objetivo, y la fluorescencia emitida por el fluoróforo pueda ser capturada por SPM.
En comparación con el software profesional que se suele utilizar para obtener la información de los resultados de las imágenes de fluorescencia de SPM21, algunos expertos utilizan el aprendizaje automático y el aprendizaje profundo para cuantificar las concentraciones de ADN del virus después de obtener imágenes de fluorescencia22, lo que requiere más tiempo. Cuando se trata de clasificar imágenes médicas, las redes neuronales convencionales (CNN) se utilizan a menudo para aprender características de las imágenes pixeladas en bruto de manera integral 23,24,25,26. Los modelos populares de aprendizaje profundo basados en CNN, como AlexNet, DenseNet-121 y EfficientNet-B7, se han aplicado con éxito en este campo27,28. Sin embargo, la obtención de grandes conjuntos de datos en dominios específicos puede ser un desafío, lo que requiere un aprendizaje por transferencia29,30. Este enfoque entrena previamente un modelo de aprendizaje profundo con un conjunto de datos grande, y el modelo entrenado previamente se usa como punto de partida para una nueva tarea con un conjunto de datos pequeño. Esta técnica puede reducir la necesidad de grandes conjuntos de datos, combatir el sobreajuste y reducir el tiempo de entrenamiento31. En este trabajo, utilizamos modelos de aprendizaje profundo con aprendizaje por transferencia para la clasificación binaria de las imágenes de fluorescencia de las muestras positivas y negativas.
En este método, combinamos RPA y el sistema CRISPR/Cas12a para la detección de trazas de virus de ADN. El ADN objetivo es amplificado y reconocido por RPA y CRISPR/Cas12a por separado, lo que desencadena la actividad de escisión colateral de Cas12a que escinde un reportero de ADN marcado con extintores de fluoróforos y generaliza la fluorescencia. Construimos un SPM portátil para tomar las imágenes fluorescentes para la detección de POC y desarrollamos modelos de aprendizaje profundo para la clasificación binaria. El esquema del sistema de detección POC incorporado se muestra en la Figura 1. Sin operadores cualificados e instrumentos voluminosos, la clasificación asistida por RPA-CRISPR/Cas12a-SPM con inteligencia artificial (IA) muestra un gran potencial para la detección de virus POC ADN.
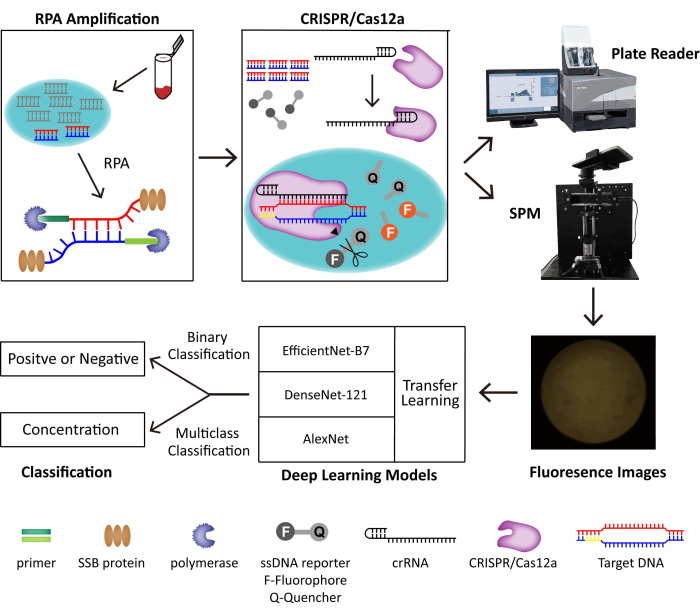
Figura 1: Esquema del sistema de detección RPA-CRISPR/Cas12-SPM junto con la clasificación de IA para las imágenes recopiladas. Los ácidos nucleicos de las muestras de origen animal son liberados por PINDBK. El ADN diana del virus es amplificado y reconocido específicamente por el sistema RPA-CRISPR/Cas12a. CRISPR/Cas12a se une con el crRNA y el complejo Cas12a-crRNA se une con el ADN objetivo, lo que desencadena la escisión colateral de CRISPR/Cas12a en las sondas reporteras de ssDNA. El fluoróforo en el reportero se libera y la fluorescencia es detectada por un lector de placas comercializado o el SPM que construimos. Se utilizan tres modelos diferentes de aprendizaje profundo, incluidos AlexNet, DenseNet-121 y EfficientNet-B7 con aprendizaje por transferencia, para clasificar las imágenes de fluorescencia. Esta figura es reutilizada con permiso de Lei et al.35. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Procesamiento de muestras
- Tomemos como ejemplo el Virus de la Rana 3 (FV3, género Ranavirus, familia Iridoviridae), un virus de ADN bicatenario. Seleccione el gen principal de la cápside (mcp) como objetivo para la detección de FV3, ya que está altamente conservado y generalmente se considera el objetivo para la detección de ranavirus. La secuencia objetivo seleccionada se muestra en la Tabla 1.
NOTA: Frog Virus 3 se toma como ejemplo en este protocolo. - Para la preparación de fragmentos de ADN diana, utilice fragmentos de ADN del gen mcp de FV3 y del virus infeccioso del bazo y la necrosis renal (ISKNV, otro ranavirus).
NOTA: En este estudio, los fragmentos de ADN objetivo se obtuvieron comercialmente de una empresa que figura en la Tabla de Materiales. Se consideran como el objetivo y el control de la detección posterior.
| Nombre | Secuencia | ||
| FV3 MCP | NTS: 5' ... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg... 3' (240 bp) | ||
| TS: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 bp) | |||
| ISKNV MCP | NTS: 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 pb) | ||
| TS: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAAATtggcatggcc... 3' (231 pb) | |||
Tabla 1: Secuencia objetivo seleccionada en este método.
2. Reacción de RPA
- Diseñe y sintetice los pares de cebadores RPA para la secuencia objetivo. Las secuencias de los pares de cebadores de RPA se describen en la Tabla 2.
- Prepare el tampón de reacción RPA 5x (Tabla 3) y MgCl2 (100 mM).
- Mezcle las cuatro enzimas clave de RPA (UvsX, UvsY, GP32, proteína Bsu) en el tampón de reacción RPA 1x, junto con los cebadores diseñados de antemano, como se detalla en la Tabla 4.
- Agita la mezcla a fondo.
- Añadir 1 μL del objetivo obtenido en el paso 1 para cada reacción de RPA, y mezclar bien mediante vórtice de nuevo.
- Añadir 7 μL de MgCl2 (100 mM) para iniciar la reacción. El volumen final de cada reacción de RPA es de 50 μL.
- Realice el ensayo a 37 °C durante 30 min.
- El producto RPA se puede almacenar a 4 °C durante unos días. A medida que los productos de RPA se degradan, utilícelos para una mayor detección lo antes posible para obtener mejores resultados de diagnóstico.
- [Opcional] Realizar electroforesis en gel de ADN.
- Retire 5 μL de productos RPA, agregue un volumen adecuado de tampón de carga de ADN 6x y realice electroforesis en gel de ADN en el tampón de trisacetato EDTA (TAE).
- Ejecute la electroforesis a menos de 120 V durante unos 20 minutos hasta que las bandas de tampón de carga lleguen al fondo del gel.
- Determine si la secuencia objetivo se amplifica con éxito a partir de la muestra comparando el tamaño de las bandas de la muestra con las bandas del marcador.
| Nombre | Secuencia |
| Imprimación RPA F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| Imprimación RPA R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Tabla 2: Imprimaciones RPA utilizadas en este método.
| Componente | Concentración original | Adición |
| PEG 20.000 | - | 114 mg |
| ATP | 100 mM | 125 μL |
| dNTPs | 25 mM | 48 μL |
| Tris-HCl | 1 M | 125 μL |
| TDT | 1 M | 125 μL |
| Fosfocreatina | 1 M | 250 μL |
| Creatina quinasa | 10 μg/μL | 50 μL |
| ddH2O | - | 277 μL |
| Volumen total | - | 1 mL |
Tabla 3: Composición del tampón de reacción RPA 5x (pH 7,5).
| Componente | Concentración original | Adición |
| 5 tampones de reacción RPA | - | 10 μL |
| Proteína UvsX | 5 mg/mL | 2,6 μL |
| Proteína UvsY | 5 mg/mL | 0,9 μL |
| Proteína GP32 | 5 mg/mL | 2,54 μL |
| Proteína Bsu | 5 mg/mL | 0,88 μL |
| Cebador delantero | 100 μM | 0,25 μL |
| imprimación invertida | 100 μM | 0,25 μL |
| ddH2O | - | 24,58 μL |
| Blanco | - | 1 μL |
| *MgCl2 | 100 mM | 7 μL |
| Volumen total | 50 μL | |
| *Por último, es necesario añadir MgCl2 para iniciar la reacción de RPA. | ||
Tabla 4: Composición de la reacción RPA.
3. Detección de CRISPR/Cas12a sin SPM
- Utilice los crRNAs (Tabla de Materiales) de la secuencia diana y la sonda reportera de ssDNA que conecta un fluoróforo y un extintor para CRISPR/Cas12a. Aquí, la carboxitetrametilrodamina (TAMRA) está unida como fluoróforo en los extremos 5' de las sondas reporteras de ssDNA y Black Hole Quencher-2 (BHQ2) como el extinguidor en los extremos 3'. La secuencia detallada del crRNA y el reportero de ssDNA se describe en la Tabla 5.
- Prepare la proteína Cas12a (LbCas12a) de la bacteria Lachnospiraceae con tampón de reacción 10x CRISPR/Cas12a.
- Disuelva 1 μL de producto de reacción RPA de la sección 2 en 1x tampón de reacción CRISPR/Cas12a con complejos LbCas12a-crRNA y sonda reportera de ssDNA de 500 nM en un volumen de reacción de 100 μL.
- Después de mezclar LbCas12a y crRNA, deje reposar la mezcla durante al menos 5 minutos para formar un complejo funcional. Después de la incubación, agregue otros componentes a la mezcla de reacción y realice toda la reacción a 37 °C. El volumen final de cada reacción CRISPR/Cas12a es de 100 μL. Las concentraciones detalladas de cada componente en cada reacción CRISPR/Cas12a se describen en la Tabla 6.
- Realice la reacción de detección CRISPR/Cas12a de 100 μL a 37 °C durante 30 min.
- Examine las señales de fluorescencia constantemente con un lector de microplacas a una longitud de onda de excitación de 535 nm y una longitud de onda de emisión de 595 nm con una ganancia de 60.
NOTA: La detección de diferentes longitudes de onda de excitación y emisión de luz depende de la elección del fluoróforo y el extindor en las sondas reporteras de ssDNA previamente diseñadas. - Para estos datos recopilados, divida el valor de control por la medición de las muestras positivas para normalizar todos los datos, luego intégrelos para un análisis de prueba t de dos muestras.
| Nombre | Secuencia | |
| LbCas12a crRNA para FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| Reportero de ssDNA | /5TAMRA/TTATT/3BHQ2 | |
Tabla 5: Secuencias de crRNA CRISPR/Cas12a y reportero de ssDNA utilizadas en este método.
| Componente | Concentración original | Adición |
| NEBuffer r2.1 | - | 10 μL |
| lba cas12a (cpf1) | 10 μM | 0,5 μL |
| ARNcr | 10 μM | 0,625 μL |
| Espere al menos 5 minutos para que el complejo LbCas12a/crRNA se combine. | ||
| Reportero de ADN | 100 μM | 0,5 μL |
| ddH2O | - | 87.375 μL |
| Blanco | - | 1 μL |
| Volumen total | - | 100 μL |
Tabla 6: Composición de la reacción CRISPR/Cas12a.
4. Configuración de SPM
- Para la trayectoria de excitación, configure un rayo láser para que pase a través de los filtros de densidad neutra (ND) para atenuar la intensidad del láser.
- Genere un haz colimado a partir de una lente asférica (en lo sucesivo denominada L1) y refleje mediante el espejo dicroico (DM).
- Dirija la luz sobre el portaobjetos de vidrio donde se coloca la muestra a través del objetivo (20x) para iluminar y excitar la fluorescencia de la muestra. La etapa de muestreo permite un ajuste preciso del plano focal, dirigiendo el haz al plano focal posterior del objetivo. Los pasos mencionados anteriormente forman la ruta de excitación del instrumento SPM.
- Para la trayectoria de emisión, coloque una lente externa (en lo sucesivo denominada L2) para formar una imagen intermedia en el otro lado del objetivo. El objetivo ilumina simultáneamente la muestra y recoge la señal de emisión.
- Registre la señal de fluorescencia de la muestra utilizando el teléfono inteligente ubicado al final de la ruta de emisión. Use un soporte estable para evitar temblores.
- Establezca un filtro de paso de banda entre L1 y la cámara del teléfono inteligente para filtrar la luz excitada y permitir que solo la luz emitida por la muestra llegue a la cámara, lo que puede optimizar la detección.
- Inmovilice la configuración de SPM en una placa de pruebas para una implementación portátil.
NOTA: En la Figura 2 se muestra el esquema y el aspecto físico del dispositivo SPM para la detección de fluorescencia basado en la reacción RPA-CRISPR/Cas12a. La detección de CRISPR/Cas12a se lleva a cabo en el portaobjetos de vidrio pretratado descrito en el siguiente paso.
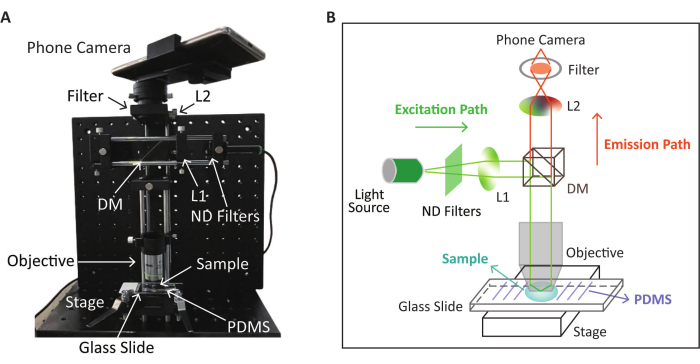
Figura 2: Aspecto esquemático y físico del dispositivo SPM utilizado para la detección de fluorescencia. (A) El aspecto físico del dispositivo SPM para la recogida de imágenes de fluorescencia después de la reacción RPA-CRISPR/Cas12a. (B) Esquema del dispositivo SPM para la detección de fluorescencia basado en la reacción RPA-CRISPR/Cas12a. Esta figura ha sido modificada (se ha ajustado la posición y el color de la imagen) con permiso de Lei et al.35. Haga clic aquí para ver una versión más grande de esta figura.
5. Tratamiento de portaobjetos de vidrio para detección con SPM
- Prepare el polidimetilsiloxano (PDMS) mezclando la base y el agente de curado en una proporción de 10:1, seguido de una cocción en una placa calefactora a 80 °C durante 2 h.
- Trate tanto el PDMS como el portaobjetos de vidrio (Longitud: 75 mm; Altura: 50 mm) con tratamiento con plasma de oxígeno durante 120 s, luego presiónelos juntos.
- Hornee el vaso/PDMS a 95 °C durante 2 h; está sellado permanentemente por el enlace Si-O-Si. PDMS tiene alta transparencia y no tiene autofluorescencia, lo que favorece la detección de SPM. El tratamiento del portaobjetos de vidrio y PMDS se realiza según He et al.32
6. Detección de CRISPR/Cas12a con SPM
- Utilice los crRNAs (Tabla de Materiales) de la secuencia diana y la sonda reportera de ssDNA que conecta un fluoróforo y un extintor para CRISPR/Cas12a. En este método, la carboxitetrametilrodamina (TAMRA) se une como fluoróforo en los extremos 5' de las sondas reporteras de ssDNA y Black Hole Quencher-2 (BHQ2) como el extinguidor en los extremos 3'. La secuencia detallada del crRNA y el reportero de ssDNA se describe en la Tabla 5.
- Prepare la proteína Cas12a (LbCas12a) de la bacteria Lachnospiraceae con 10x tampón de reacción CRISPR/Cas12a.
- Disuelva 1 μL de producto de reacción RPA de la sección 2 en 1x tampón de reacción CRISPR/Cas12a con complejos LbCas12a-crRNA y sonda reportera de ssDNA de 500 nM en un volumen de reacción de 100 μL.
- Después de mezclar LbCas12a y crRNA, deje reposar la mezcla durante al menos 5 minutos para formar un complejo funcional. Después de la incubación, agregue otros componentes a la mezcla de reacción y complete la reacción a 37 °C. El volumen final de cada reacción CRISPR/Cas12a es de 100 μL. Las concentraciones detalladas de cada componente en cada reacción CRISPR/Cas12a se describen en la Tabla 6.
- Realice la reacción de detección CRISPR/Cas12a de 100 μL en el portaobjetos de vidrio pretratado y cúbralo con un cubreobjetos. Incubar el portaobjetos de vidrio con reacción a RT durante 10 min.
- Mida las señales de fluorescencia por el SPM. Coloque el portaobjetos de vidrio con la reacción de detección en la etapa del SPM, mantenga una distancia adecuada, ajuste la distancia focal y la claridad, luego busque el campo de visión de la reacción y enfóquelo para capturar una imagen.
NOTA: Primero se debe obtener una curva estándar para que los datos de las muestras se puedan escalar al rango de concentración aproximado. Se utilizan varias concentraciones de objetivos purificados, incluidos 10 nM, 1 nM, 100 pM y 10 pM, otro virus como control negativo (antes de RPA).
7. Aumento de conjuntos de datos y datos
- Recopile las imágenes de fluorescencia del ensayo de detección de la sección 6 como conjuntos de datos. Repita al menos tres detecciones paralelas para cada muestra a fin de garantizar el paralelismo de los datos.
- Se pueden aprobar algunas formas apropiadas de lograr un mayor paralelismo. Por ejemplo, al recolectar las imágenes de fluorescencia de cada muestra, enfoque manualmente y busque un campo relativamente más brillante para tomar imágenes. Al mismo tiempo, fotografíe cada muestra para obtener la señal de fluorescencia en más de cinco ubicaciones diferentes.
- Mida el valor medio de gris de cada imagen y la desviación estándar del valor medio de gris en un grupo de concentración por ImageJ.
- Establezca un rango de intensidades [mediana - desviación estándar, mediana + desviación estándar] para la limpieza de datos.
NOTA: En las imágenes obtenidas en los primeros pasos, puede haber imágenes con grandes diferencias, por lo que es necesario cribar las imágenes. Si las intensidades de las imágenes están fuera del umbral establecido, deben considerarse valores atípicos y excluirse. - Etiquete las imágenes para el objetivo purificado a concentraciones crecientes con 0-6 en orden ascendente, respectivamente.
- Para mejorar la robustez del sistema y evitar el sobreajuste, implemente técnicas de aumento de imagen como el volteo horizontal, el volteo vertical y el ruido aleatorio a través de funciones de transformación en Python. Esto ayuda a introducir variaciones en el conjunto de datos.
8. Transferir el aprendizaje
- Como red troncal, adopte el modelo de aprendizaje profundo AlexNet33 para la clasificación.
- Para satisfacer la restricción del modelo previamente entrenado en la sección 7, cambie la forma de las imágenes de entrada a 224 píxeles x 224 píxeles x 3 canales (altura y anchura de 224 píxeles y una profundidad de 3 canales para los canales de color rojo, verde y azul) a través de funciones de transformación en Python.
NOTA: Este paso es un preprocesamiento común para datos heterogéneos en el aprendizaje por transferencia, incluida la transformación. - Utilice una red troncal previamente entrenada con el dataset de ImageNet para extraer entidades mientras aprovecha los pesos de las capas ocultas intermedias que se aprendieron.
- En el contexto del trabajo de clasificación de fluorescencia, sustituya la capa final completamente conectada de la red neuronal, que originalmente incluía 1000 neuronas para la tarea ImageNet, por una capa completamente conectada con 2 o 7 neuronas.
- Evalúe el rendimiento del modelo de entrenamiento de configuración mediante una serie de métricas, incluida la matriz de confusión, la exactitud, la precisión, el recuerdo y la puntuación F1 basada en Lawton y Viriri34.
Resultados
Este método se centra en un sistema de detección de virus de ADN rápido, fácil de implementar, altamente sensible y en el punto de atención (POC). El diseño de pares de cebadores para la reacción RPA y el diseño de crRNA para la reacción CRISPR/Cas12a son dos de las partes esenciales, ya que afectarán a la eficiencia de la reacción RPA-CRISPR/Cas12a e influirán en la posterior detección y clasificación.
En este método, FV3 se considera un ejemplo de detecci...
Discusión
En este método, desarrollamos un sistema de detección de virus de ADN POC rápido, fácil de implementar, altamente sensible, específico de secuencia y POC con asistencia de IA. Después de obtener las muestras, se aplica RPA para amplificar la secuencia objetivo, y luego CRISPR/Cas12a puede reconocer el ADN objetivo y liberar fluorescencia, lo que amplía la señal de detección. La microscopía portátil de los teléfonos inteligentes está diseñada para tomar imágenes de fluorescencia, y los modelos de aprendizaj...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo cuenta con el apoyo de la Fundación Nacional de Ciencias Naturales de China 31970752, Ciencia, Tecnología e Innovación de la Comisión de JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Financiación abierta del Laboratorio de la Bahía de Shenzhen, SZBL2020090501004; Fundación de Ciencias Postdoctorales de China 2020M680023; y Administración General de Aduanas de la República Popular China 2021HK007.
Materiales
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Referencias
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados