このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
RPA-CRISPR/Cas12A-SPMとディープラーニングに基づくDNAウイルス検出システム
要約
DNAウイルスの微量検出のための組換えポリメラーゼ増幅とCRISPR/Cas12aシステムを組み合わせたプロトコルを提示し、ポイントオブケアDNAウイルス検出のための人工知能支援分類を備えたポータブルスマートフォン顕微鏡を構築します。
要約
DNAウイルスの微量検出のために、組換えポリメラーゼ増幅(RPA)とCRISPR/Cas12aシステムを組み合わせた、迅速で実装が簡単で、高感度で、配列特異的なポイントオブケア(POC)DNAウイルス検出システムを報告しています。標的DNAはRPAとCRISPR/Cas12aによって別々に増幅および認識され、Cas12aの側副切断活性が引き起こされ、蛍光を消す標識DNAレポーターを切断し、蛍光を一般化します。POC検出のために、ポータブルスマートフォン顕微鏡は蛍光画像を撮影するように構築されています。さらに、高精度を達成した正または負のサンプルの二項分類のための深層学習モデルがシステム内に展開されています。このDNAウイルスPOC検出系の例として、カエルウイルス3(FV3、 ラナウイルス属、 イリドウイルス科)を試験したところ、検出限界(LoD)は40分以内に10aMを達成できます。熟練したオペレーターやかさばる機器がなくても、人工知能(AI)支援分類を備えたポータブルで小型のRPA-CRISPR/Cas12a-SPMは、POC DNAウイルス検出の大きな可能性を示し、そのようなウイルスの拡散を防ぐのに役立ちます。
概要
近年、2014年のエボラウイルス病(EVD)の流行1と20182、2015年の中東呼吸器症候群(MERS)3、2015年のジカウイルス病の流行4、重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)5によるコロナウイルス病2019(COVID-19)5、2022年のサル痘ウイルスによるサル痘の継続(MKPV)6など、さまざまなウイルスによる感染症の流行が頻繁に発生していますで囲まれています。これらの流行感染症の突然の発生は、多数の死者を引き起こし、莫大な経済的損失と社会不安をもたらします。感染を迅速に診断し、ウイルスのさらなる拡散を防ぐためには、迅速かつ正確な検出システムが緊急に必要とされています。
最近、クラスター化された規則的な間隔を空けた短い回文反復(CRISPR)およびCRISPR関連(Cas)タンパク質が世界的な注目を集めており、核酸検出において有望な結果を示しています7,8,9,10,11,12,13,14,15 .CRISPR/Cas12aタンパク質は、CRISPR RNA(crRNA)に導かれて、標的DNAに結合して切断します。この活性は、トランス切断として知られる非特異的一本鎖DNA(ssDNA)の放出につながり、核酸検出の検出シグナルを増強するために利用することができます。ポリメラーゼ連鎖反応(PCR)、定量的リアルタイムPCR(qPCR)、酵素結合免疫吸着アッセイ(ELISA)などの従来の検出法は、ポイントオブケア(POC)検出に複雑で時間とコストがかかります。私たちの以前の研究では、CRISPR/Cas12a技術に基づくアフリカ豚コレラウイルス(ASFV)の自動化、統合、費用対効果の高い検出システムの開発に成功しました。このシステムでは、増幅を必要とせずに、2時間の時間枠で1pMの検出限界を達成しました。CRISPR/Cas12aシステムと組換えポリメラーゼ増幅(RPA)を組み合わせることで、微量DNA検出の感度と特異性が向上します。RPAは、他の等温増幅技術と比較して、設計が簡単で、高度な温度制御装置なしで反応時間が短いため、操作が便利です。
病原体のPOC検出のために、スマートフォン顕微鏡(SPM)、ハンドヘルド蛍光計、またはラテラルフローストリップなどの機器が、結果の読み取り16、17、18用に開発されています。SPMはカメラを通して画像をキャプチャし、高速データ分析のためにいくつかのモバイルアプリケーションにアップロードします。このような顕微鏡法は、高感度でポータブルで安価で小型の信号取得システムを作成し、H5N1、ジカウイルス、SARS-CoV-2などの病原体の検出に利点を示しています19,20。そこで、RPA-CRISPR/Cas12aの標的DNAウイルスの検出によって引き起こされる蛍光シグナルを捕捉するポータブルSPMを構築しました。CRISPR/Cas12aが標的DNAウイルスを認識すると、蛍光色素とクエンチャーをつなぐssDNAレポータープローブが切断され、蛍光色素分子が発する蛍光をSPMで捕捉することができます。
SPM21からの蛍光画像から結果情報を取得するために通常使用される専門的なソフトウェアと比較して、一部の専門家は、蛍光画像22を達成した後、機械学習と深層学習を使用してウイルスDNAの濃度を定量化しますが、これはより時間がかかります。医用画像の分類に関しては、従来のニューラルネットワーク(CNN)が、生のピクセル化された画像からエンドツーエンドの方法で特徴を学習するためによく使用されます23、24、25、26。AlexNet、DenseNet-121、EfficientNet-B7などの一般的なCNNベースのディープラーニングモデルは、この分野にうまく適用されています27,28。しかし、特定のドメインで大規模なデータセットを取得することは困難であり、転移学習が必要です29,30。このアプローチでは、大規模なデータセットを使用してディープ ラーニング モデルを事前トレーニングし、事前トレーニング済みのモデルを小さなデータセットを使用した新しいタスクの開始点として使用します。この手法は、大規模なデータセットの必要性を減らし、過学習に対処し、トレーニング時間を短縮することができます31。ここでは、転移学習による深層学習モデルを用いて、ポジティブサンプルとネガティブサンプルの蛍光画像の二値分類を行います。
この手法では、RPAとCRISPR/Cas12aシステムを組み合わせて、DNAウイルスの微量検出を行います。標的DNAはRPAとCRISPR/Cas12aによって別々に増幅および認識され、Cas12aの側副切断活性が引き起こされ、蛍光を消す標識DNAレポーターを切断し、蛍光を一般化します。POC検出用の蛍光画像を取得するためのポータブルSPMを構築し、バイナリ分類用の深層学習モデルを開発します。構築されたPOC検出システムの概略図を 図1に示します。熟練したオペレーターやかさばる機器がなくても、人工知能(AI)支援分類を備えたRPA-CRISPR/Cas12a-SPMは、POC DNAウイルス検出の大きな可能性を示しています。
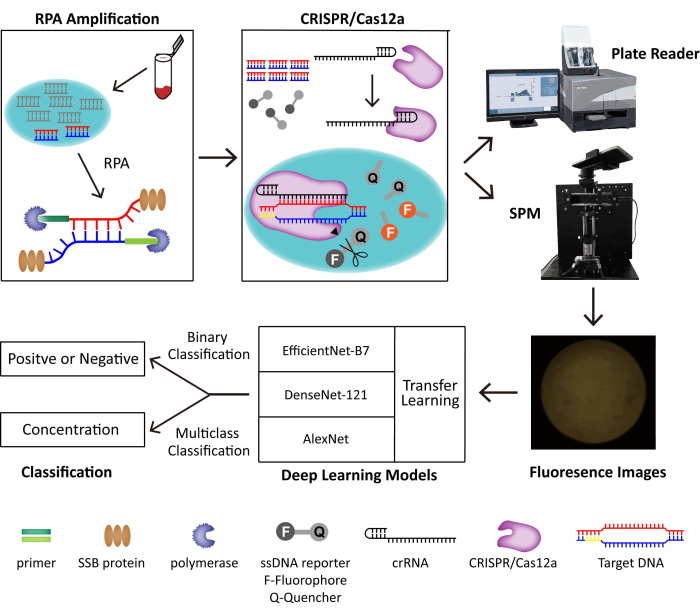
図1:RPA-CRISPR/Cas12-SPM検出システムの概略図と、収集された画像のAI分類。 動物由来サンプルの核酸は、PINDBKによって放出されます。ウイルスの標的DNAは、RPA-CRISPR/Cas12aシステムによって特異的に増幅され、認識されます。CRISPR/Cas12aはcrRNAと結合し、Cas12a-crRNA複合体は標的DNAと結合し、ssDNAレポータープローブ上のCRISPR/Cas12aの側副切断を引き起こします。レポーター上の蛍光色素が放出され、蛍光は市販のプレートリーダーまたは当社が構築したSPMによって検出されます。AlexNet、DenseNet-121、転移学習を備えたEfficientNet-B7を含む3つの異なる深層学習モデルを使用して、蛍光画像を分類します。この図は、Lei et al.35の許可を得て再利用されています。 この図の拡大版を見るには、ここをクリックしてください。
プロトコル
1.サンプルの処理
- 二本鎖DNAウイルスであるカエルウイルス3(FV3、 ラナウイルス属、 イリドウイルス科)を例にとってみましょう。FV3は高度に保存されており、通常はラナウイルス検出の標的と見なされているため、FV3の検出ターゲットとしてメジャーカプシド(mcp)遺伝子を選択します。選択したターゲット配列を 表1に示します。
注:Frog Virus 3は、このプロトコルの例として取り上げられています。 - 標的DNA断片の調製には、FV3および感染性脾臓および腎壊死ウイルス(ISKNV、別のラナウイルス)由来の mcp 遺伝子のDNA断片を使用します。
注:この研究では、標的DNA断片は、 材料の表に記載されている会社から商業的に入手しました。それらは、ターゲットおよびその後の検出の制御と見なされます。
| 名前 | 順序 | ||
| FV3 MCP | NTS:5フィート...gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg...3フィート(240bp) | ||
| TS:5フィート...ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc...3フィート(240bp) | |||
| ISKNV MCPの | NTS:5フィート...ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg...3フィート(231bp) | ||
| TS:5フィート...ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc...3フィート(231bp) | |||
表1:この方法で選択したターゲット配列。
2. RPAの反応
- ターゲット配列のRPAプライマーペアを設計および合成します。RPAプライマーペアの配列を 表2に示します。
- 5x RPA反応バッファー(表3)とMgCl2 (100 mM)を調製します。
- 表4に詳述するように、4つの主要なRPA酵素(UvsX、UvsY、GP32、Bsuタンパク質)を1x RPA反応バッファーに混合し、事前に設計されたプライマーを混合します。
- 混合物を完全にボルテックスします。
- RPA反応1回につき、ステップ1で得られたターゲットを1μL加え、再度ボルテックスで十分に混合します。
- 7 μLのMgCl2 (100 mM)を加えて反応を開始します。各RPA反応の最終容量は50μLです。
- アッセイを37°Cで30分間実行します。
- RPA製品は4°Cで数日間保存できます。RPA製品は劣化しますので、より良い診断結果を得るために、できるだけ早くさらなる検出に使用してください。
- [オプション]DNAゲル電気泳動を行います。
- 5 μLのRPA製品を取り出し、適量の6x DNAローディングバッファーを加え、Tris-acetate EDTA(TAE)バッファーでDNAゲル電気泳動を行います。
- ローディングバッファーのバンドがゲルの底に達するまで、120 V下で約20分間電気泳動を実行します。
- サンプルのバンドのサイズとマーカーのバンドを比較して、ターゲット配列がサンプルから正常に増幅されているかどうかを判断します。
| 名前 | 順序 |
| RPA入門書F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| RPA入門書 R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
表2:この方法で使用されるRPAプライマー。
| コンポーネント | 元の濃度 | 足し算 |
| 20,000ペソ | - | 114ミリグラム |
| ATPの | 100 mM | 125 μL |
| dNTPs | 25 mM | 48 μL |
| トリス-HCl | 1メートル | 125 μL |
| DTTの | 1メートル | 125 μL |
| クレアチンリン酸 | 1メートル | 250μL |
| クレアチンキナーゼ | 10 μg/μL | 50μL |
| ddH2O | - | 277 μL |
| 総ボリューム | - | 1 mL |
表3:5x RPA反応バッファー(pH 7.5)の組成。
| コンポーネント | 元の濃度 | 足し算 |
| 5x RPA反応バッファー | - | 10 μL |
| UvsXタンパク質 | 5 mg/mL | 2.6 μL |
| UvsYタンパク質 | 5 mg/mL | 0.9μL |
| GP32タンパク質 | 5 mg/mL | 2.54μL |
| Bsuタンパク質 | 5 mg/mL | 0.88μL |
| フォワードプライマー | 100 μM | 0.25μL |
| 反転プライマー | 100 μM | 0.25μL |
| ddH2O | - | 24.58μL |
| ターゲット | - | 1 μL |
| *MgCl2 | 100 mM | 7 μL |
| 総ボリューム | 50μL | |
| *RPA反応を開始するには、最後にMgCl2 を添加する必要があります。 | ||
表4:RPA反応の構成。
3. SPMを使用しないCRISPR/Cas12a検出
- 標的配列のcrRNA(Table of Materials)と、CRISPR/Cas12a用の蛍光色素とクエンチャーをつなぐssDNAレポータープローブを使用します。ここでは、カルボキシテトラメチルローダミン(TAMRA)がssDNAレポータープローブの5'末端の蛍光色素として結合し、ブラックホールクエンチャー-2(BHQ2)が3'末端のクエンチャーとして結合しています。crRNAおよびssDNAレポーターの詳細な配列を 表5に記載する。
- Lachnospiraceae細菌Cas12a(LbCas12a)タンパク質を10x CRISPR/Cas12a反応バッファーで調製します。
- セクション2のRPA反応生成物1 μLを、LbCas12a-crRNA複合体および500 nM ssDNAレポータープローブを含む1x CRISPR/Cas12a反応バッファーに溶解し、100 μLの反応容量で溶解します。
- LbCas12aとcrRNAを混合した後、混合物を少なくとも5分間放置して、機能的な複合体を形成します。インキュベーション後、反応混合物に他の成分を加え、37°Cで全反応を行います。 各CRISPR/Cas12a反応の最終容量は100μLです。各CRISPR/Cas12a反応における各成分の詳細な濃度を 表6に記載する。
- 100 μL CRISPR/Cas12a検出反応を37°Cで30分間行ってください。
- マイクロプレートリーダーにより、励起波長535 nm、発光波長595 nm、ゲイン60の蛍光シグナルを常時調べます。
注:異なる波長の励起光と発光光の検出は、以前に設計されたssDNAレポータープローブの蛍光色素とクエンチャーの選択に依存します。 - これらの収集されたデータについて、制御値を陽性サンプルの測定値で割ってすべてのデータを正規化し、それらを積分して2サンプルのt検定分析を行います。
| 名前 | 順序 | |
| FV3用のLbCas12a crRNA | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| ssDNAレポーター | /5タムラ/TTATT/3BHQ2 | |
表5:この方法で使用されるCRISPR/Cas12a crRNAおよびssDNAレポーターの配列。
| コンポーネント | 元の濃度 | 足し算 |
| NEBuffer r2.1 | - | 10 μL |
| LBA CAS12A ( CPF1 ) | 10 μM | 0.5μL |
| crRNA | 10 μM | 0.625μL |
| LbCas12a/crRNA複合体が結合するまで、少なくとも5分間待ちます。 | ||
| DNAレポーター | 100 μM | 0.5μL |
| ddH2O | - | 87.375μL |
| ターゲット | - | 1 μL |
| 総ボリューム | - | 100 μL |
表6:CRISPR/Cas12a反応の組成。
4. SPMのセットアップ
- 励起経路では、レーザービームがND(ND)フィルターを通過するように設定して、レーザー強度を減衰させます。
- 非球面レンズ(以下、L1)からコリメートビームを発生させ、ダイクロイックミラー(DM)で反射します。
- サンプルが置かれているスライドガラスに光を向け、対物レンズ(20x)を通してサンプルの蛍光を照らして励起します。サンプルステージは、正確な焦点面の調整を可能にし、ビームを対物レンズの背面焦点面に向けます。上記のステップは、SPM機器の励起経路を形成します。
- 発光路は、対物レンズの反対側に外部レンズ(以下、L2)を配置して中間像を形成します。対物レンズは、サンプルの照明と発光信号の収集を同時に行います。
- 蛍光経路の最後に設置したスマートフォンを使用して、サンプルからの蛍光信号を記録します。揺れないように安定したブラケットを使用してください。
- L1とスマートフォンのカメラの間にバンドパスフィルターを設定して励起光をフィルタリングし、サンプルから放出された光のみをカメラに届けることで、検出を最適化することができます。
- SPM セットアップをブレッドボードに固定して、ポータブル展開を実現します。
注:RPA-CRISPR/Cas12a反応に基づく蛍光検出用のSPMデバイスの概略図と物理的外観を 図2に示します。CRISPR/Cas12aの検出は、次のステップで説明する前処理されたスライドガラス上で行われます。
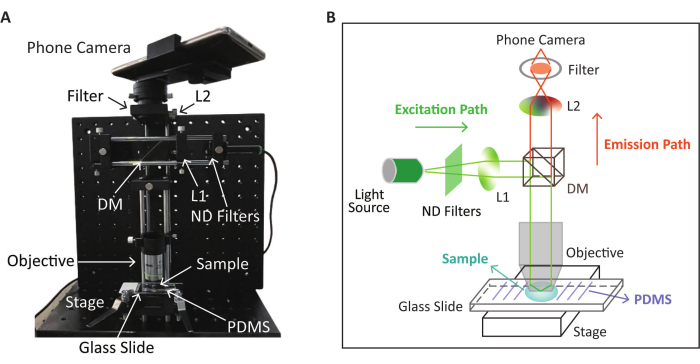
図2:蛍光検出に用いたSPM装置の概略図と外観 (A)RPA-CRISPR/Cas12a反応後の蛍光画像収集用SPM装置の外観。(B)RPA-CRISPR/Cas12a反応に基づく蛍光検出用SPMデバイスの概略図。この図は、Lei et al.35の許可を得て変更(画像の位置と色を調整)したものである。 この図の拡大版を見るには、ここをクリックしてください。
5. SPMによる検出のためのスライドガラスの処理
- ポリジメチルシロキサン(PDMS)は、塩基と硬化剤を10:1の比率で混合し、次いでホットプレート上で80°Cで2時間焼成します。
- PDMSとスライドガラスの両方を処理します(長さ:75 mm;高さ:50mm)に酸素プラズマ処理を120秒間施してから、押し合わせます。
- ガラス/ PDMSを95°Cで2時間焼きます。Si-O-Si結合によって恒久的に密封されています。PDMSは透明性が高く、自家蛍光がないため、SPMの検出に役立ちます。スライドガラスとPMDSの治療は、Heらに従って行われます32。
6. SPMによるCRISPR/Cas12a検出
- 標的配列のcrRNA(Table of Materials)と、CRISPR/Cas12a用の蛍光色素とクエンチャーをつなぐssDNAレポータープローブを使用します。この方法では、カルボキシテトラメチルローダミン(TAMRA)がssDNAレポータープローブの5'末端の蛍光色素として結合され、ブラックホールクエンチャー-2(BHQ2)が3'末端のクエンチャーとして結合されます。crRNAおよびssDNAレポーターの詳細な配列を 表5に記載する。
- Lachnospiraceae細菌Cas12a(LbCas12a)タンパク質を10x CRISPR/Cas12a反応バッファーで調製します。
- セクション2のRPA反応生成物1 μLを、LbCas12a-crRNA複合体および500 nM ssDNAレポータープローブを含む1x CRISPR/Cas12a反応バッファーに溶解し、100 μLの反応容量で溶解します。
- LbCas12aとcrRNAを混合した後、混合物を少なくとも5分間放置して、機能的な複合体を形成します。インキュベーション後、反応混合物に他の成分を加え、37°Cで反応を完了させます。 各CRISPR/Cas12a反応の最終容量は100μLです。各CRISPR/Cas12a反応における各成分の詳細な濃度を 表6に記載する。
- 前処理したスライドガラスに対して100 μL CRISPR/Cas12a検出反応を行い、カバースリップで覆います。スライドガラスを室温で反応させて10分間インキュベートします。
- SPMで蛍光シグナルを測定します。検出反応を起こしたスライドガラスをSPMのステージに置き、適度な距離を保ち、焦点距離と明瞭度を調整してから、反応の視野を探してピントを合わせ、画像を撮影します。
注:サンプルからのデータをおおよその濃度範囲にスケーリングできるように、最初に標準曲線を取得する必要があります。10 nM、1 nM、100 pM、および10 pMを含むさまざまな濃度の精製ターゲットが使用され、ネガティブコントロールとして別のウイルス(RPA前)が使用されます。
7. データセットとデータの拡張
- セクション6の検出アッセイから蛍光画像をデータセットとして収集します。データの並列性を確保するために、サンプルごとに少なくとも3つの並列検出を繰り返します。
- より高い並列処理を実現するためのいくつかの適切な方法が承認されます。たとえば、各サンプルから蛍光画像を収集するときは、手動で焦点を合わせ、比較的明るい視野を探して画像を撮影します。同時に、各サンプルを撮影して、5つ以上の異なる場所で蛍光シグナルを取得します。
- 各画像の平均グレー値と、集中グループの平均グレー値の標準偏差をImageJで測定します。
- データクリーニングの強度範囲(中央値-標準偏差、中央値+標準偏差)を設定します。
注:初期の手順で得られた画像では、大きな違いのある画像がある場合があるため、画像をスクリーニングする必要があります。画像の強度が設定されたしきい値を超えている場合は、外れ値と見なされ、除外されます。 - 精製したターゲットの画像を、それぞれ昇順で0〜6の濃度でラベル付けします。
- システムのロバスト性を高め、過剰適合を防ぐには、Python の変換関数を使用して、水平反転、垂直反転、ランダム ノイズなどの画像拡張手法を実装します。これは、データセットにバリエーションを導入するのに役立ちます。
8.転移学習
- 基幹ネットワークとして、分類に深層学習モデルAlexNet33 を採用する。
- セクション 7 の事前トレーニング済みモデルの制約を満たすには、Python の変換関数を使用して、入力画像を 224 ピクセル x 224 ピクセル x 3 チャネル (高さと幅 224 ピクセル、赤、緑、青のカラー チャネルの深さ 3 チャネル) に形状を変更します。
注: このステップは、変換を含む転移学習における異種データの一般的な前処理です。 - ImageNet データセットで事前学習済みのバックボーン ネットワークを使用して、学習した中間隠れ層の重みを活用しながら特徴を抽出します。
- 蛍光分類ジョブのコンテキストでは、ニューラル ネットワークの最終的な全結合層 (ImageNet タスク用に元々 1000 個のニューロンが含まれていた) を、2 個または 7 個のニューロンを含む全結合層に置き換えます。
- Lawton と Viriri34 に基づく混同行列、精度、精度、再現率、F1 スコアなどの一連のメトリックを使用して、セットアップ トレーニング モデルのパフォーマンスを評価します。
結果
この方法は、DNAウイルスの迅速で実装が簡単で高感度のポイントオブケア(POC)検出システムに焦点を当てています。RPA反応のプライマーペア設計とCRISPR/Cas12a反応のcrRNA設計は、RPA-CRISPR/Cas12a反応の効率に影響を与え、その後の検出と分類に影響を与えるため、重要な部分の2つです。
この方法では、FV3はDNAウイルス検出の一例と見なされます。FV3用のRPAプ?...
ディスカッション
この手法では、AIの支援を受けて、迅速で実装が容易で、高感度で、配列特異的なPOC DNAウイルス検出システムを開発します。サンプルを取得した後、RPAを適用して標的配列を増幅すると、CRISPR / Cas12aが標的DNAを認識して蛍光を放出し、検出信号が拡大します。ポータブルスマートフォン顕微鏡は蛍光画像を撮影するように構築されており、転移学習による深層学習モデルは、ポジティブサン?...
開示事項
著者らは何も開示していない。
謝辞
この作業は、中国国家自然科学基金会31970752、科学、技術、深セン市JCYJ20190809180003689、JSGG20200225150707332、JSGG20191129110812708、WDZC20200820173710001のイノベーション委員会によってサポートされています。深センベイ研究所オープンファンディング、SZBL2020090501004;中国ポスドク科学財団2020M680023;中華人民共和国税関総署2021HK007。
資料
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
参考文献
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved