É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Sistema de detecção de vírus de DNA baseado em RPA-CRISPR/Cas12a-SPM e aprendizado profundo
Neste Artigo
Resumo
Apresentamos um protocolo que combina amplificação da polimerase recombinase com um sistema CRISPR/Cas12a para detecção de traços de vírus de DNA e constrói microscopia portátil para smartphone com uma classificação assistida por inteligência artificial para detecção de vírus de DNA no local de atendimento.
Resumo
Relatamos um sistema de detecção de vírus de DNA rápido, fácil de implementar, altamente sensível, específico de sequência e ponto de atendimento (POC), que combina amplificação da polimerase recombinase (RPA) e sistema CRISPR/Cas12a para detecção de vestígios de vírus de DNA. O DNA alvo é amplificado e reconhecido por RPA e CRISPR/Cas12a separadamente, o que desencadeia a atividade de clivagem colateral de Cas12a que cliva um repórter de DNA rotulado como fluoróforo-supressor e generaliza a fluorescência. Para detecção de POC, a microscopia portátil de smartphone é construída para tirar imagens fluorescentes. Além disso, modelos de deep learning para classificação binária de amostras positivas ou negativas, alcançando alta precisão, são implantados no sistema. O vírus 3 da rã (FV3, gênero Ranavirus, família Iridoviridae) foi testado como exemplo para este sistema de detecção de POC de vírus de DNA, e os limites de detecção (LoD) podem atingir 10 aM em 40 min. Sem operadores qualificados e instrumentos volumosos, a classificação portátil e em miniatura RPA-CRISPR/Cas12a-SPM com inteligência artificial (IA) mostra grande potencial para detecção de vírus de DNA POC e pode ajudar a prevenir a propagação de tais vírus.
Introdução
Nos últimos anos, epidemias de doenças infecciosas causadas por diferentes vírus têm ocorrido com frequência, incluindo a epidemia da doença do vírus Ebola (EVD) em 20141 e 20182, a Síndrome Respiratória do Oriente Médio (MERS) em 20153, a epidemia da doença do vírus Zika em 20154, a doença do Coronavírus 2019 (COVID-19) causada pela síndrome respiratória aguda grave coronavírus 2 (SARS-CoV-2)5 e a continuação da varíola dos macacos causada pelo vírus da varíola dos macacos (MKPV) em 20226. Esses surtos repentinos de doenças infecciosas epidêmicas causam um grande número de mortes e trazem enormes perdas econômicas e agitação social. Um sistema de detecção rápido e preciso é urgentemente necessário para diagnosticar rapidamente a infecção e evitar a propagação do vírus.
Recentemente, repetições palindrômicas curtas agrupadas regularmente interespaçadas (CRISPR) e proteínas associadas a CRISPR (Cas) ganharam atenção mundial e mostraram resultados promissores na detecção de ácidos nucleicos 7,8,9,10,11,12,13,14,15 . A proteína CRISPR/Cas12a, guiada pelo RNA CRISPR (crRNA), liga-se e cliva o DNA alvo. Essa atividade leva à liberação de DNA de fita simples inespecífico (ssDNA), conhecido como transclivagem, e pode ser utilizada para aumentar o sinal de detecção de ácido nucleico. Alguns métodos tradicionais de detecção, como reação em cadeia da polimerase (PCR), PCR quantitativo em tempo real (qPCR) e ensaio de imunoabsorção enzimática (ELISA) são complicados, demorados e caros para detecção no local de atendimento (POC). Nosso trabalho anterior desenvolveu com sucesso um sistema de detecção automatizado, integrado e econômico para o vírus da peste suína africana (ASFV) baseado na tecnologia CRISPR/Cas12a. Neste sistema, atingimos um limite de detecção de 1 pM em um período de 2 horas sem a necessidade de amplificação. O sistema CRISPR/Cas12a e a amplificação da polimerase recombinase (RPA) são combinados para melhorar a sensibilidade e a especificidade para a detecção de traços de DNA. Comparado com outras técnicas de amplificação isotérmica, o RPA é simples em design e conveniente em operação, pois tem um tempo de reação mais curto sem equipamentos sofisticados de controle de temperatura.
Para a detecção de patógenos por POC, instrumentos como microscopia de smartphone (SPM), fluorímetro portátil ou tiras de fluxo lateral são desenvolvidos para as leituras dos resultados 16,17,18. O SPM captura imagens por meio de uma câmera e as carrega em alguns aplicativos móveis para análise rápida de dados. Essa microscopia produz um sistema de aquisição de sinal portátil, barato, miniado e com alta sensibilidade e tem mostrado vantagens na detecção de patógenos como o H5N1, o vírus Zika e o SARS-CoV-2 19,20. Portanto, construímos um SPM portátil para capturar os sinais de fluorescência acionados pela detecção RPA-CRISPR/Cas12a do vírus de DNA alvo. A sonda repórter ssDNA ligando um fluoróforo e um supressor será clivada quando o CRISPR/Cas12a reconhecer o vírus de DNA alvo, e a fluorescência emitida pelo fluoróforo puder ser capturada pelo SPM.
Em comparação com o software profissional geralmente usado para obter as informações de resultados das imagens de fluorescência do SPM21, alguns especialistas usam aprendizado de máquina e aprendizado profundo para quantificar as concentrações de DNA do vírus após obter imagens de fluorescência22, o que é mais demorado. Quando se trata de classificar imagens médicas, as redes neurais convencionais (CNNs) são frequentemente usadas para aprender recursos das imagens pixeladas brutas de maneira ponta a ponta 23,24,25,26. Modelos populares de aprendizado profundo baseados em CNN, como AlexNet, DenseNet-121 e EfficientNet-B7, foram aplicados com sucesso neste campo27,28. No entanto, a obtenção de grandes conjuntos de dados em domínios específicos pode ser desafiadora, necessitando de transferência de aprendizado29,30. Essa abordagem pré-treina um modelo de aprendizado profundo com um grande conjunto de dados, e o modelo pré-treinado é usado como ponto de partida para uma nova tarefa com um pequeno conjunto de dados. Essa técnica pode reduzir a necessidade de grandes conjuntos de dados, combater o overfitting e reduzir o tempo de treinamento31. Aqui, usamos modelos de aprendizado profundo com aprendizado de transferência para a classificação binária das imagens de fluorescência das amostras positivas e negativas.
Neste método, combinamos RPA e o sistema CRISPR/Cas12a para a detecção de vestígios de vírus de DNA. O DNA alvo é amplificado e reconhecido por RPA e CRISPR/Cas12a separadamente, o que desencadeia a atividade de clivagem colateral de Cas12a que cliva um repórter de DNA rotulado como fluoróforo-supressor e generaliza a fluorescência. Construímos um SPM portátil para obter as imagens fluorescentes para detecção de POC e desenvolvemos modelos de aprendizado profundo para classificação binária. O esquema do sistema de detecção de POC construído é mostrado na Figura 1. Sem operadores qualificados e instrumentos volumosos, a classificação assistida por RPA-CRISPR/Cas12a-SPM com inteligência artificial (IA) mostra grande potencial para detecção de vírus de DNA POC.
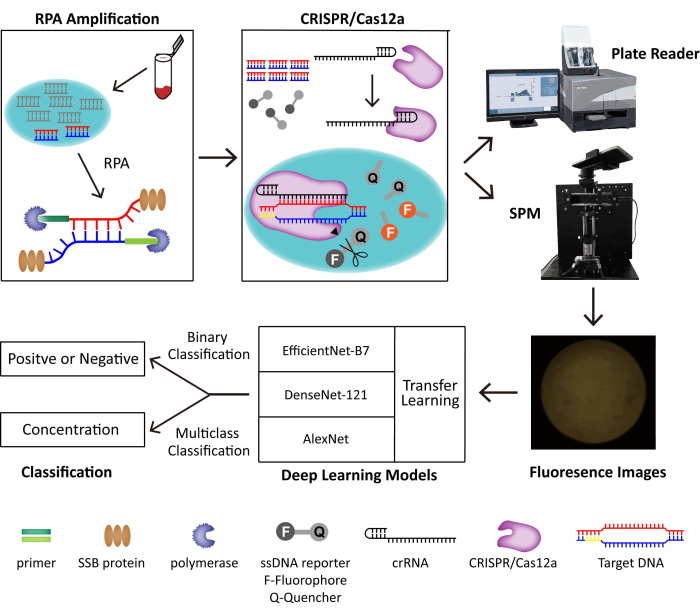
Figura 1: O esquema do sistema de detecção RPA-CRISPR/Cas12-SPM junto com a classificação de IA para imagens coletadas. Os ácidos nucléicos de amostras derivadas de animais são liberados por PINDBK. O DNA alvo do vírus é amplificado e reconhecido especificamente pelo sistema RPA-CRISPR/Cas12a. O CRISPR/Cas12a se liga ao crRNA e o complexo Cas12a-crRNA se liga ao DNA alvo, o que desencadeia a clivagem colateral do CRISPR/Cas12a nas sondas repórter do ssDNA. O fluoróforo no repórter é liberado e a fluorescência é detectada por um leitor de placas comercializado ou pelo SPM que construímos. Três modelos diferentes de aprendizado profundo, incluindo AlexNet, DenseNet-121 e EfficientNet-B7 com aprendizado de transferência, são usados para classificar as imagens de fluorescência. Essa figura é reutilizada com permissão de Lei et al.35. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Processamento de amostras
- Veja o Frog Virus 3 (FV3, gênero Ranavirus, família Iridoviridae), um vírus de DNA de fita dupla. Selecione o gene do capsídeo principal (mcp) como alvo para a detecção de FV3, pois é altamente conservado e geralmente considerado como alvo para detecção de ranavírus. A sequência alvo selecionada é mostrada na Tabela 1.
NOTA: O Frog Virus 3 é tomado como exemplo neste protocolo. - Para a preparação de fragmentos de DNA alvo, use fragmentos de DNA do gene mcp de FV3 e Vírus de Necrose Infecciosa do Baço e Rim (ISKNV, outro Ranavirus).
NOTA: Neste estudo, os fragmentos de DNA alvo foram obtidos comercialmente de uma empresa listada na Tabela de Materiais. Eles são considerados o alvo e o controle da detecção subsequente.
| Nome | Seqüenciar | ||
| FV3 MCP | NTS: 5' ... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcgg... 3' (240 bp) | ||
| TS: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 bp) | |||
| ISKNV MCP | NTS: 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 bp) | ||
| TS: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 bp) | |||
Tabela 1: Sequência alvo selecionada neste método.
2. Reação de RPA
- Projete e sintetize os pares de primers RPA para a sequência alvo. As sequências de pares de primers RPA são descritas na Tabela 2.
- Prepare o tampão de reação 5x RPA (Tabela 3) e MgCl2 (100 mM).
- Misture as quatro principais enzimas RPA (UvsX, UvsY, GP32, proteína Bsu) no tampão de reação 1x RPA, juntamente com os primers projetados com antecedência, conforme detalhado na Tabela 4.
- Vortex a mistura completamente.
- Adicione 1 μL do alvo obtido na etapa 1 para cada reação de RPA e misture bem por vórtice novamente.
- Adicione 7 μL de MgCl2 (100 mM) para iniciar a reação. O volume final de cada reação RPA é de 50 μL.
- Efectuar o ensaio a 37 °C durante 30 min.
- O produto RPA pode ser armazenado a 4 °C por alguns dias. À medida que os produtos RPA se degradam, use para detecção adicional o mais rápido possível para obter melhores resultados de diagnóstico.
- [Opcional] Realize eletroforese em gel de DNA.
- Retire 5 μL de produtos RPA, adicione um volume apropriado de tampão de carga de DNA 6x e conduza a eletroforese em gel de DNA no tampão EDTA de trisacetato (TAE).
- Execute a eletroforese abaixo de 120 V por cerca de 20 min até que as bandas do tampão de carga atinjam o fundo do gel.
- Determine se a sequência alvo foi amplificada com sucesso a partir da amostra, comparando o tamanho das bandas da amostra com as bandas do marcador.
| Nome | Seqüenciar |
| RPA primer F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| RPA primer R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Tabela 2: Primers RPA usados neste método.
| Componente | Concentração original | Adição |
| PEG 20.000 | - | 114 mg |
| ATP | 100 mM | 125 μL |
| dNTPs | 25 mM | 48 μL |
| Tris-HCl | 1 milhão | 125 μL |
| TDT | 1 milhão | 125 μL |
| Fosfocreatina | 1 milhão | 250 μL |
| Creatina quinase | 10 μg/μL | 50 μL |
| ddH2O | - | 277 μL |
| Total Volume | - | 1 mL |
Tabela 3: A composição do tampão de reação 5x RPA (pH 7,5).
| Componente | Concentração original | Adição |
| 5x tampão de reação RPA | - | 10 μL |
| Proteína UvsX | 5 mg/ml | 2,6 μL |
| Proteína UvsY | 5 mg/ml | 0,9 μL |
| Proteína GP32 | 5 mg/ml | 2,54 μL |
| Proteína Bsu | 5 mg/ml | 0,88 μL |
| primer para frente | 100 μM | 0,25 μL |
| primer invertido | 100 μM | 0,25 μL |
| ddH2O | - | 24,58 μL |
| Alvo | - | 1 μL |
| *MgCl2 | 100 mM | 7 μL |
| Total Volume | 50 μL | |
| *MgCl2 precisa ser adicionado por último para iniciar a reação de RPA. | ||
Tabela 4: A composição da reação RPA.
3. Detecção de CRISPR/Cas12a sem SPM
- Use os crRNAs (Tabela de Materiais) da sequência alvo e a sonda repórter ssDNA ligando um fluoróforo e um supressor para CRISPR/Cas12a. Aqui, a carboxitetrametilrodamina (TAMRA) está ligada como o fluoróforo nas extremidades 5 'das sondas repórter ssDNA e o Black Hole Quencher-2 (BHQ2) como o supressor nas extremidades 3 '. A sequência detalhada do repórter crRNA e ssDNA é descrita na Tabela 5.
- Prepare a bactéria Lachnospiraceae Cas12a (LbCas12a)
- Dissolva 1 μL do produto da reação RPA da seção 2 em 1x tampão de reação CRISPR/Cas12a com complexos LbCas12a-crRNA e sonda repórter ssDNA de 500 nM em um volume de reação de 100 μL.
- Depois de misturar LbCas12a e crRNA, deixe a mistura descansar por pelo menos 5 min para formar um complexo funcional. Após a incubação, adicionar outros componentes à mistura de reação e realizar toda a reação a 37 °C. O volume final de cada reação CRISPR/Cas12a é de 100 μL. As concentrações detalhadas de cada componente em cada reação CRISPR/Cas12a estão descritas na Tabela 6.
- Execute a reação de detecção CRISPR/Cas12a de 100 μL a 37 °C por 30 min.
- Examinar constantemente os sinais de fluorescência por um leitor de microplacas com um comprimento de onda de excitação de 535 nm e um comprimento de onda de emissão de 595 nm com um ganho de 60.
NOTA: A detecção de diferentes comprimentos de onda de excitação e emissão de luz depende da escolha do fluoróforo e do atenuador nas sondas repórter ssDNA projetadas anteriormente. - Para esses dados coletados, divida o valor de controle pela medição das amostras positivas para normalizar todos os dados e, em seguida, integre-os para uma análise de teste t de duas amostras.
| Nome | Seqüenciar | |
| LbCas12a crRNA para FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| repórter ssDNA | /5TAMRA/TTATT/3BHQ2 | |
Tabela 5: Sequências de crRNA CRISPR/Cas12a e repórter ssDNA usadas neste método.
| Componente | Concentração original | Adição |
| NEBuffer r2.1 | - | 10 μL |
| Lba Cas12a (Cpf1) | 10 μM | 0,5 μL |
| crRNA | 10 μM | 0,625 μL |
| Aguarde pelo menos 5 minutos para permitir que o complexo LbCas12a/crRNA se combine. | ||
| Repórter de DNA | 100 μM | 0,5 μL |
| ddH2O | - | 87,375 μL |
| Alvo | - | 1 μL |
| Total Volume | - | 100 μL |
Tabela 6: A composição da reação CRISPR/Cas12a.
4. Configuração do SPM
- Para o caminho de excitação, defina um feixe de laser para passar pelos filtros de densidade neutra (ND) para atenuar a intensidade do laser.
- Gerar um feixe colimado a partir de uma lente asférica (a seguir designada por L1) e refleti-lo através do espelho dicróico (DM).
- Direcione a luz para a lâmina de vidro onde a amostra é colocada através da objetiva (20x) para iluminar e excitar a fluorescência da amostra. A platina da amostra permite o ajuste preciso do plano focal, direcionando o feixe para o plano focal traseiro da objetiva. As etapas mencionadas acima formam o caminho de excitação do instrumento SPM.
- Para o caminho de emissão, posicione uma lente externa (doravante denominada L2) para formar uma imagem intermediária do outro lado da objetiva. A objetiva ilumina simultaneamente a amostra e coleta o sinal de emissão.
- Registre o sinal de fluorescência da amostra usando o smartphone colocado no final do caminho de emissão. Use um suporte estável para evitar tremores.
- Defina um filtro passa-banda entre L1 e a câmera do smartphone para filtrar a luz excitada, permitindo que apenas a luz emitida da amostra chegue à câmera, o que pode otimizar a detecção.
- Imobilize a configuração do SPM em uma placa de ensaio para implantação portátil.
NOTA: O esquema e a aparência física do dispositivo SPM para detecção de fluorescência com base na reação RPA-CRISPR/Cas12a são mostrados na Figura 2. A detecção de CRISPR/Cas12a ocorre na lâmina de vidro pré-tratada descrita na próxima etapa.
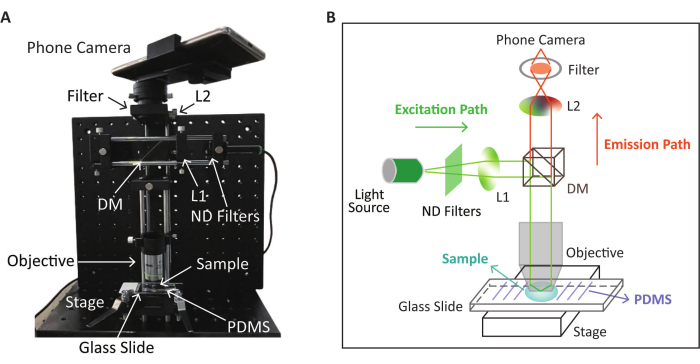
Figura 2: Aparência esquemática e física do dispositivo SPM usado para detecção de fluorescência. (A) A aparência física do dispositivo SPM para coleta de imagens de fluorescência após a reação RPA-CRISPR/Cas12a. (B) Esquema do dispositivo SPM para detecção de fluorescência com base na reação RPA-CRISPR/Cas12a. Essa figura é modificada (posição e cor da imagem ajustadas) com permissão de Lei et al.35. Clique aqui para ver uma versão maior desta figura.
5. Tratamento de lâmina de vidro para detecção com SPM
- Preparar o polidimetilsiloxano (PDMS) misturando a base e o agente de cura na proporção de 10:1, seguido de cozedura numa placa de aquecimento a 80 °C durante 2 h.
- Trate o PDMS e a lâmina de vidro (Comprimento: 75 mm; Altura: 50 mm) com tratamento de plasma de oxigênio por 120 s, depois pressione-os juntos.
- Asse o vidro/PDMS a 95 °C por 2 h; é selado permanentemente pela ligação Si-O-Si. O PDMS tem alta transparência e sem autofluorescência, o que é propício para a detecção de SPM. O tratamento da lâmina de vidro e da PMDS é realizado de acordo com He et al.32
6. Detecção CRISPR/Cas12a com SPM
- Use os crRNAs (Tabela de Materiais) da sequência alvo e a sonda repórter ssDNA ligando um fluoróforo e um supressor para CRISPR/Cas12a. Neste método, a carboxitetrametilrodamina (TAMRA) está ligada como o fluoróforo nas extremidades 5' das sondas repórter ssDNA e o Black Hole Quencher-2 (BHQ2) como o supressor nas extremidades 3'. A sequência detalhada do repórter crRNA e ssDNA é descrita na Tabela 5.
- Prepare a bactéria Lachnospiraceae Cas12a (LbCas12a)
- Dissolva 1 μL do produto da reação RPA da seção 2 em 1x tampão de reação CRISPR/Cas12a com complexos LbCas12a-crRNA e sonda repórter ssDNA de 500 nM em um volume de reação de 100 μL.
- Depois de misturar LbCas12a e crRNA, deixe a mistura descansar por pelo menos 5 min para formar um complexo funcional. Após a incubação, adicionar outros componentes à mistura de reação e completar a reação a 37 °C. O volume final de cada reação CRISPR/Cas12a é de 100 μL. As concentrações detalhadas de cada componente em cada reação CRISPR/Cas12a estão descritas na Tabela 6.
- Execute a reação de detecção CRISPR/Cas12a de 100 μL na lâmina de vidro pré-tratada e cubra-a com uma lamínula. Incubar a lâmina de vidro com reação em RT durante 10 min.
- Meça os sinais de fluorescência pelo SPM. Coloque a lâmina de vidro com a reação de detecção no palco do SPM, mantenha uma distância adequada, ajuste a distância focal e a clareza, procure o campo de visão da reação e foque-o para capturar uma imagem.
NOTA: Uma curva padrão deve ser obtida primeiro para que os dados das amostras possam ser dimensionados para a faixa de concentração aproximada. Várias concentrações de alvos purificados são usadas, incluindo 10 nM, 1 nM, 100 pM e 10 pM, outro vírus como controle negativo (antes do RPA).
7. Aumento de dados e dados
- Colete as imagens de fluorescência do ensaio de detecção na seção 6 como conjuntos de dados. Repita pelo menos três detecções paralelas para cada exemplo para garantir o paralelismo de dados.
- Algumas maneiras apropriadas de obter maior paralelismo podem ser aprovadas. Por exemplo, ao coletar as imagens de fluorescência de cada amostra, foque manualmente e procure um campo relativamente mais claro para tirar imagens. Ao mesmo tempo, fotografe cada amostra para obter o sinal de fluorescência em mais de cinco locais diferentes.
- Meça o valor médio de cinza de cada imagem e o desvio padrão do valor médio de cinza em um grupo de concentração por ImageJ.
- Defina um intervalo de intensidades [mediana - desvio padrão, mediana + desvio padrão] para limpeza de dados.
NOTA: Nas imagens obtidas nos primeiros passos, pode haver imagens com grandes diferenças, por isso é necessário rastrear as imagens. Se as intensidades das imagens estiverem fora do limite definido, elas devem ser consideradas discrepantes e excluídas. - Rotule as imagens para o alvo purificado em concentrações crescentes com 0-6 em ordem crescente, respectivamente.
- Para aumentar a robustez do sistema e evitar o ajuste excessivo, implemente técnicas de aumento de imagem, como inversão horizontal, inversão vertical e ruído aleatório por meio de funções de transformação em Python. Isso ajuda a introduzir variações no conjunto de dados.
8. Transferir aprendizado
- Como rede de backbone, adote o modelo de aprendizado profundo AlexNet33 para classificação.
- Para atender à restrição do modelo pré-treinado na seção 7, remodele as imagens de entrada para 224 pixels x 224 pixels x 3 canais (altura e largura de 224 pixels e uma profundidade de 3 canais para os canais de cores vermelho, verde e azul) por meio de funções de transformação em Python.
NOTA: Esta etapa é um pré-processamento comum para dados heterogêneos no aprendizado de transferência, incluindo transformação. - Use uma rede de backbone pré-treinada com o conjunto de dados ImageNet para extrair recursos enquanto aproveita os pesos das camadas ocultas intermediárias que foram aprendidas.
- No contexto do trabalho de classificação de fluorescência, substitua a camada final totalmente conectada da rede neural, que originalmente incluía 1000 neurônios para a tarefa ImageNet, por uma camada totalmente conectada com 2 ou 7 neurônios.
- Avalie o desempenho do modelo de treinamento de configuração usando uma série de métricas, incluindo matriz de confusão, exatidão, precisão, recall e pontuação F1 com base em Lawton e Viriri34.
Resultados
Este método se concentra em um sistema de detecção rápido, fácil de implementar, altamente sensível e no local de atendimento (POC) para vírus de DNA. O design dos pares de primers para a reação RPA e o design do crRNA para a reação CRISPR/Cas12a são duas das partes essenciais, pois afetarão a eficiência da reação RPA-CRISPR/Cas12a e influenciarão a detecção e classificação subsequentes.
Neste método, o FV3 é considerado um exemplo de detecção de ...
Discussão
Nesse método, desenvolvemos um sistema de detecção de vírus de DNA POC rápido, fácil de implementar, altamente sensível, específico de sequência e POC com assistência de IA. Após a obtenção das amostras, o RPA é aplicado para amplificar a sequência alvo e, em seguida, o CRISPR/Cas12a pode reconhecer o DNA alvo e liberar fluorescência, o que amplia o sinal de detecção. A microscopia portátil de smartphone é construída para obter imagens de fluorescência, e modelos de aprendizado profundo com aprendiz...
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho é apoiado pela Fundação Nacional de Ciências Naturais da China 31970752, Ciência, Tecnologia e Comissão de Inovação do Município de Shenzhen JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Financiamento Aberto do Laboratório da Baía de Shenzhen, SZBL2020090501004; Fundação de Ciência de Pós-Doutorado da China 2020M680023; e Administração Geral das Alfândegas da República Popular da China 2021HK007.
Materiais
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Referências
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados