A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מערכת זיהוי וירוסי DNA מבוססת על RPA-CRISPR/Cas12A-SPM ולמידה עמוקה
In This Article
Summary
אנו מציגים פרוטוקול המשלב הגברה של רקומבינאז פולימראז עם מערכת CRISPR/Cas12a לאיתור עקבות של נגיפי DNA ובונים מיקרוסקופ סמארטפון נייד עם סיווג בעזרת בינה מלאכותית לזיהוי נגיפי DNA נקודתיים.
Abstract
אנו מדווחים על מערכת זיהוי נגיפי DNA מהירה, קלה ליישום, רגישה מאוד, ספציפית לרצף ונקודת טיפול (POC), המשלבת הגברה של רקומבינאז פולימראז (RPA) ומערכת CRISPR/Cas12a לאיתור עקבות של נגיפי DNA. דנ"א המטרה מוגבר ומזוהה על ידי RPA ו-CRISPR/Cas12a בנפרד, מה שמפעיל את פעילות המחשוף ההיקפי של Cas12a שחותכת כתב DNA עם תווית פלואורופורית ומכלילה פלואורסצנטיות. לזיהוי POC, מיקרוסקופ סמארטפון נייד בנוי לצלם תמונות פלואורסצנטיות. חוץ מזה, מודלים של למידה עמוקה לסיווג בינארי של דגימות חיוביות או שליליות, להשגת דיוק גבוה, נפרסים בתוך המערכת. וירוס צפרדע 3 (FV3, סוג Ranavirus, משפחה Iridoviridae) נבדק כדוגמה למערכת זיהוי POC זו של נגיף DNA, וגבולות הזיהוי (LoD) יכולים להשיג 10 aM תוך 40 דקות. ללא מפעילים מיומנים ומכשירים מגושמים, הסיווג הנייד והזעיר בסיוע RPA-CRISPR/Cas12a-SPM עם בינה מלאכותית (AI) מראה פוטנציאל גדול לזיהוי וירוסי POC DNA ויכול לסייע במניעת התפשטות וירוסים כאלה.
Introduction
בשנים האחרונות, מגיפות של מחלות זיהומיות הנגרמות על ידי וירוסים שונים התרחשו לעתים קרובות, כולל מגיפת נגיף האבולה (EVD) בשנת 20141 ו 20182, תסמונת הנשימה במזרח התיכון (MERS) בשנת 20153, מגיפת מחלת נגיף זיקה בשנת 20154, מחלת נגיף הקורונה 2019 (COVID-19) הנגרמת על ידי תסמונת נשימה חריפה חמורה קורונה 2 (SARS-CoV-2)5 ואבעבועות הקוף המתמשכת הנגרמת על ידי נגיף אבעבועות הקוף (MKPV) בשנת 20226. התפרצויות פתאומיות אלה של מחלות זיהומיות מגיפות גורמות למספר רב של מקרי מוות ומביאות להפסדים כלכליים עצומים ותסיסה חברתית. נדרשת בדחיפות מערכת זיהוי מהירה ומדויקת כדי לאבחן במהירות את הזיהום ולמנוע את המשך התפשטות הנגיף.
לאחרונה, חלבונים פלינדרומיים קצרים מקובצים במרווחים קבועים (CRISPR) וחלבונים הקשורים לקריספר (Cas) זכו לתשומת לב עולמית והראו תוצאות מבטיחות בזיהוי חומצות גרעין 7,8,9,10,11,12,13,14,15 . חלבון CRISPR/Cas12a, מונחה על ידי CRISPR RNA (crRNA), נקשר לדנ"א המטרה וקורע אותו. פעילות זו מובילה לשחרור DNA חד-גדילי לא ספציפי (ssDNA), המכונה trans-cleavage, וניתן להשתמש בה כדי לשפר את אות הזיהוי לזיהוי חומצות גרעין. כמה שיטות זיהוי מסורתיות כמו תגובת שרשרת פולימראז (PCR), PCR כמותי בזמן אמת (qPCR) ובדיקת אימונוסורבנט מקושרת אנזים (ELISA) הן מסובכות, גוזלות זמן ויקרות לזיהוי נקודת טיפול (POC). בעבודתנו הקודמת פיתחנו בהצלחה מערכת זיהוי אוטומטית, משולבת וחסכונית לנגיף קדחת החזירים האפריקנית (ASFV) המבוססת על טכנולוגיית CRISPR/Cas12a. במערכת זו השגנו מגבלת זיהוי של 1 pM בתוך מסגרת זמן של שעתיים ללא צורך בהגברה. מערכת CRISPR/Cas12a והגברה של רקומבינאז פולימראז (RPA) משולבות כדי לשפר את הרגישות והספציפיות לזיהוי עקבות DNA. בהשוואה לטכניקות הגברה איזותרמיות אחרות, RPA הוא פשוט בעיצוב ונוח לתפעול מכיוון שיש לו זמן תגובה קצר יותר ללא ציוד בקרת טמפרטורה מתוחכם.
לזיהוי POC של פתוגנים, מכשירים כגון מיקרוסקופ סמארטפון (SPM), פלואורימטר כף יד או פסי זרימה רוחביים מפותחים עבור קריאות התוצאות 16,17,18. SPM לוכד תמונות באמצעות מצלמה ומעלה אותן ליישומים ניידים מסוימים לצורך ניתוח נתונים מהיר. מיקרוסקופיה כזו מייצרת מערכת קליטת אותות ניידת, זולה וממוזערת עם רגישות גבוהה והראתה יתרונות באיתור פתוגנים כגון H5N1, נגיף זיקה ו- SARS-CoV-219,20. לכן, אנו בונים SPM נייד כדי לתפוס את אותות הפלואורסצנטיות המופעלים על ידי זיהוי RPA-CRISPR/Cas12a של נגיף ה- DNA המטרה. גשושית כתב ssDNA המקשרת בין פלואורופור ומרווה תיבקע כאשר CRISPR/Cas12a יזהה את נגיף ה- DNA המטרה, והפלואורסצנטיות הנפלטת מהפלואורופור יכולה להילכד על ידי SPM.
בהשוואה לתוכנה המקצועית המשמשת בדרך כלל להשגת מידע התוצאות מתמונות הפלואורסצנטיות מ- SPM21, חלק מהמומחים משתמשים בלמידת מכונה ובלמידה עמוקה כדי לכמת את ריכוזי ה- DNA של הנגיף לאחר השגת תמונות פלואורסצנטיות22, דבר הגוזל זמן רב יותר. כשמדובר בסיווג תמונות רפואיות, רשתות עצביות קונבנציונליות (CNN) משמשות לעתים קרובות כדי ללמוד תכונות מהתמונות המפוקסלות הגולמיות באופן מקצה לקצה 23,24,25,26. מודלים פופולריים מבוססי למידה עמוקה מבוססי CNN כמו AlexNet, DenseNet-121 ו- EfficientNet-B7 יושמו בהצלחה בתחום זה27,28. עם זאת, השגת מערכי נתונים גדולים בתחומים ספציפיים יכולה להיות מאתגרת, ומחייבת למידה העברה29,30. גישה זו מאמנת מראש מודל למידה עמוקה עם מערך נתונים גדול, והמודל שהוכשר מראש משמש כנקודת התחלה למשימה חדשה עם מערך נתונים קטן. טכניקה זו יכולה להפחית את הצורך במערכי נתונים גדולים, להילחם בהתאמת יתר ולהפחית את זמן האימון31. כאן, אנו משתמשים במודלים של למידה עמוקה עם למידת העברה לסיווג בינארי של תמונות פלואורסצנטיות של הדגימות החיוביות והשליליות.
בשיטה זו אנו משלבים RPA ומערכת CRISPR/Cas12a לאיתור עקבות של נגיפי DNA. דנ"א המטרה מוגבר ומזוהה על ידי RPA ו-CRISPR/Cas12a בנפרד, מה שמפעיל את פעילות המחשוף ההיקפי של Cas12a שחותכת כתב DNA עם תווית פלואורופורית ומכלילה פלואורסצנטיות. אנו בונים SPM נייד כדי לצלם את התמונות הפלואורסצנטיות לזיהוי POC ולפתח מודלים של למידה עמוקה לסיווג בינארי. הסכימה של מערכת זיהוי POC הבנויה מוצגת באיור 1. ללא מפעילים מיומנים ומכשירים מגושמים, הסיווג בסיוע RPA-CRISPR/Cas12a-SPM עם בינה מלאכותית (AI) מראה פוטנציאל גדול לזיהוי וירוסי DNA POC.
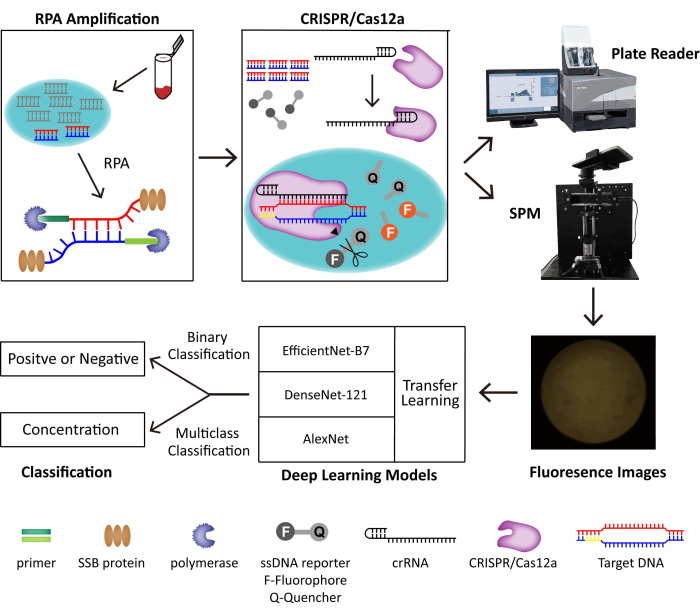
איור 1: הסכימה של מערכת זיהוי RPA-CRISPR/Cas12-SPM יחד עם סיווג AI עבור תמונות שנאספו. חומצות הגרעין של דגימות שמקורן בבעלי חיים משוחררות על ידי PINDBK. דנ"א המטרה של הנגיף מוגבר ומזוהה באופן ספציפי על ידי מערכת RPA-CRISPR/Cas12a. קשרי CRISPR/Cas12a עם crRNA וקשרים מורכבים Cas12a-crRNA עם DNA מטרה, מה שמפעיל את הפיצול הבטחוני של CRISPR/Cas12a על גשושיות כתב ssDNA. הפלואורופור על הכתב משתחרר, והפלואורסצנטיות מזוהה על ידי קורא לוחות ממוסחר או SPM שאנו בונים. שלושה מודלים שונים של למידה עמוקה, כולל AlexNet, DenseNet-121 ו- EfficientNet-B7 עם למידת העברה, משמשים לסיווג תמונות הפלואורסצנטיות. נעשה שימוש חוזר בנתון זה באישור Lei et al.35. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
1. עיבוד דגימות
- קח וירוס צפרדע 3 (FV3, סוג Ranavirus, משפחה Iridoviridae), וירוס DNA דו גדילי. בחר את הגן הראשי capsid (mcp) כיעד לזיהוי FV3 מכיוון שהוא שמור מאוד ונחשב בדרך כלל כיעד לזיהוי ranavirus. רצף היעד שנבחר מוצג בטבלה 1.
הערה: וירוס צפרדע 3 נלקח כדוגמה בפרוטוקול זה. - להכנת מקטעי DNA ממוקדים, השתמש במקטעי DNA של הגן mcp מ- FV3 וב- Infectious Spleen and Kidney Necrosis Virus (ISKNV, Ranavirus אחר).
הערה: במחקר זה, מקטעי דנ"א המטרה התקבלו באופן מסחרי מחברה הרשומה בטבלת החומרים. הם נחשבים המטרה ואת השליטה של גילוי לאחר מכן.
| שם | רצף | ||
| מק"פ FV3 | NTS: 5 '... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg... 3' (240 bp) | ||
| TS: 5 '... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 bp) | |||
| ISKNV MCP | NTS: 5 '... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 כ"ס) | ||
| TS: 5 '... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 כ"ס) | |||
טבלה 1: רצף היעד שנבחר בשיטה זו.
2. תגובת RPA
- עצב וסנתז את זוגות פריימר RPA עבור רצף היעד. הרצפים של זוגות פריימר RPA מתוארים בטבלה 2.
- הכן את מאגר התגובה 5x RPA (טבלה 3) ואת MgCl2 (100 מילימול).
- ערבבו את ארבעת אנזימי ה-RPA העיקריים (UvsX, UvsY, GP32, חלבון Bsu) במאגר התגובה 1x RPA, יחד עם הפריימרים שתוכננו מראש, כמפורט בטבלה 4.
- מערבבים היטב את התערובת.
- הוסף 1 μL של המטרה המתקבלת משלב 1 עבור כל תגובת RPA, וערבב אותו היטב על ידי מערבולת שוב.
- הוסף 7 μL של MgCl2 (100 mM) כדי להתחיל את התגובה. הנפח הסופי של כל תגובת RPA הוא 50 μL.
- בצע את הבדיקה ב 37 ° C במשך 30 דקות.
- מוצר RPA ניתן לאחסן ב 4 ° C במשך כמה ימים. כאשר מוצרי RPA מתפרקים, השתמש לזיהוי נוסף בהקדם האפשרי כדי להשיג תוצאות אבחון טובות יותר.
- [אופציונלי] לבצע אלקטרופורזה ג'ל DNA.
- הוציאו 5 מיקרוליטר של מוצרי RPA, הוסיפו נפח מתאים של מאגר טעינת DNA פי 6 ובצעו אלקטרופורזה של ג'ל DNA במאגר Tris-acetate EDTA (TAE).
- הפעל את האלקטרופורזה תחת 120 וולט במשך כ -20 דקות עד שרצועות חיץ ההעמסה מגיעות לתחתית הג'ל.
- קבע אם רצף היעד מוגבר בהצלחה מהדגימה על-ידי השוואת גודל הפסים של הדגימה עם הפסים של הסמן.
| שם | רצף |
| פריימר RPA F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| פריימר RPA R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
טבלה 2: פריימרים של RPA המשמשים בשיטה זו.
| רכיב | ריכוז מקורי | תוספת |
| PEG 20,000 | - | 114 מ"ג |
| ATP | 100 מ"מ | 125 מיקרוליטר |
| dNTPs | 25 מ"מ | 48 μL |
| Tris-HCl | 1 מטר | 125 מיקרוליטר |
| DTT | 1 מטר | 125 מיקרוליטר |
| פוספוקריאטין | 1 מטר | 250 מיקרוליטר |
| קריאטין קינאז | 10 מיקרוגרם/μL | 50 מיקרוליטר |
| ddH2O | - | 277 מיקרוליטר |
| נפח כולל | - | 1 מ"ל |
טבלה 3: הרכב מאגר תגובת RPA 5x (pH 7.5).
| רכיב | ריכוז מקורי | תוספת |
| 5x מאגר תגובת RPA | - | 10 מיקרוליטר |
| חלבון UvsX | 5 מ"ג/מ"ל | 2.6 מיקרוליטר |
| חלבון UvsY | 5 מ"ג/מ"ל | 0.9 מיקרוליטר |
| חלבון GP32 | 5 מ"ג/מ"ל | 2.54 מיקרוליטר |
| חלבון Bsu | 5 מ"ג/מ"ל | 0.88 מיקרוליטר |
| פריימר קדימה | 100 מיקרומטר | 0.25 מיקרוליטר |
| פריימר הפוך | 100 מיקרומטר | 0.25 מיקרוליטר |
| ddH2O | - | 24.58 מיקרוליטר |
| יעד | - | 1 μL |
| *MgCl2 | 100 מ"מ | 7 מיקרוליטר |
| נפח כולל | 50 מיקרוליטר | |
| *יש להוסיף MgCl2 באופן אחרון כדי להתחיל את תגובת ה-RPA. | ||
טבלה 4: הרכב תגובת ה-RPA.
3. זיהוי CRISPR/Cas12a ללא SPM
- השתמש ב- crRNA (טבלת חומרים) של רצף המטרה ובבדיקה של כתב ssDNA המקשרת בין פלואורופור ומרווה עבור CRISPR/Cas12a. כאן, קרבוקסי טטרמתילרודאמין (TAMRA) מקושר כפלואורופור בקצוות 5' של בדיקות כתב ssDNA וחור שחור Quencher-2 (BHQ2) כמו מרווה בקצוות 3'. הרצף המפורט של crRNA וכתב ssDNA מתואר בטבלה 5.
- הכינו את חלבון החיידק Lachnospiraceae Cas12a (LbCas12a) עם חיץ תגובה 10x CRISPR/Cas12a.
- המסת 1 μL של תוצר תגובת RPA מסעיף 2 במאגר תגובה 1x CRISPR/Cas12a עם קומפלקסים LbCas12a-crRNA ובדיקת כתב ssDNA של 500 ננומטר בנפח תגובה של 100 μL.
- לאחר ערבוב LbCas12a ו-crRNA, הניחו לתערובת לשבת לפחות 5 דקות כדי ליצור קומפלקס פונקציונלי. לאחר הדגירה, הוסף רכיבים אחרים לתערובת התגובה ובצע את התגובה כולה ב 37 מעלות צלזיוס. הנפח הסופי של כל תגובת CRISPR/Cas12a הוא 100 μL. הריכוזים המפורטים של כל רכיב בכל תגובת CRISPR/Cas12a מתוארים בטבלה 6.
- בצע את תגובת הזיהוי CRISPR/Cas12a של 100 μL ב-37°C למשך 30 דקות.
- בחן את אותות הפלואורסצנטיות כל הזמן על ידי קורא מיקרו-לוחות באורך גל עירור של 535 ננומטר ובאורך גל פליטה של 595 ננומטר עם רווח של 60.
הערה: הזיהוי של אורכי גל שונים של אור עירור ופליטה תלוי בבחירת הפלואורופור והמרווה בבדיקות כתב ssDNA שתוכננו בעבר. - עבור נתונים אלה שנאספו, חלק את ערך הבקרה במדידת הדגימות החיוביות כדי לנרמל את כל הנתונים, ולאחר מכן שלב אותם לניתוח מבחן t של שתי דגימות.
| שם | רצף | |
| LbCas12a crRNA עבור FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| כתב ssDNA | /5טמרה/TTATT/3BHQ2 | |
טבלה 5: רצפים של CRISPR/Cas12a crRNA וכתב ssDNA המשמשים בשיטה זו.
| רכיב | ריכוז מקורי | תוספת |
| NEBuffer r2.1 | - | 10 מיקרוליטר |
| Lba Cas12a (Cpf1) | 10 מיקרומטר | 0.5 מיקרוליטר |
| crRNA | 10 מיקרומטר | 0.625 מיקרוליטר |
| המתן לפחות 5 דקות כדי לאפשר לקומפלקס LbCas12a/crRNA להשתלב. | ||
| כתב DNA | 100 מיקרומטר | 0.5 מיקרוליטר |
| ddH2O | - | 87.375 מיקרוליטר |
| יעד | - | 1 μL |
| נפח כולל | - | 100 מיקרוליטר |
טבלה 6: הרכב תגובת CRISPR/Cas12a.
4. הגדרת SPM
- עבור נתיב העירור, הגדר קרן לייזר שתעבור דרך מסנני הצפיפות הנייטרלית (ND) כדי להחליש את עוצמת הלייזר.
- ליצור קרן התנגשות מעדשה אספרית (להלן L1) ולשקף אותה על ידי המראה הדיכרואית (DM).
- כוונו את האור אל שקופית הזכוכית שבה הדגימה ממוקמת דרך המטרה (20x) כדי להאיר ולעורר את הפלואורסצנטיות של הדגימה. שלב הדגימה מאפשר התאמה מדויקת של מישור המוקד, המכוון את הקרן למישור המוקד האחורי של המטרה. השלבים שהוזכרו לעיל יוצרים את נתיב העירור של מכשיר SPM.
- עבור נתיב הפליטה, מקם עדשה חיצונית (להלן L2) ליצירת תמונת ביניים בצד השני של המטרה. המטרה מאירה בו זמנית את הדגימה ואוספת את אות הפליטה.
- הקלט את אות הפלואורסצנטיות מהדגימה באמצעות הטלפון החכם הממוקם בסוף נתיב הפליטה. השתמש בתושבת יציבה כדי למנוע רעידות.
- הגדר מסנן פס בין L1 למצלמת הטלפון החכם כדי לסנן את האור הנרגש תוך מתן אפשרות רק לאור הנפלט מהדגימה להגיע למצלמה, מה שיכול לייעל את הזיהוי.
- שתק את הגדרת SPM על קרש לחם לצורך פריסה ניידת.
הערה: המראה הסכמטי והפיזי של התקן SPM לזיהוי פלואורסצנטיות בהתבסס על תגובת RPA-CRISPR/Cas12a מוצגים באיור 2. זיהוי CRISPR/Cas12a מתבצע בשקופית הזכוכית שטופלה מראש המתוארת בשלב הבא.
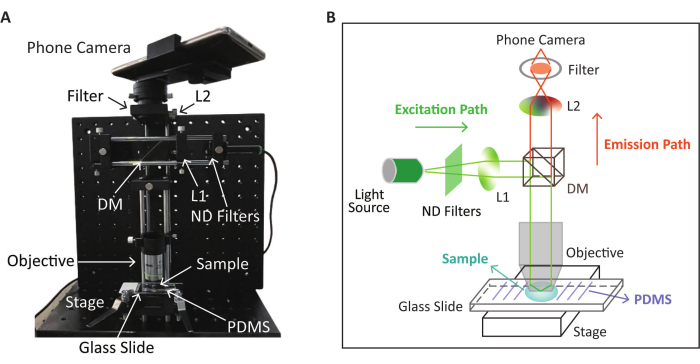
איור 2: המראה הסכמטי והפיזי של התקן SPM המשמש לזיהוי פלואורסצנטי. (A) המראה הפיזי של התקן SPM עבור אוסף תמונות פלואורסצנטיות לאחר תגובת RPA-CRISPR/Cas12a. (B) סכימה של התקן SPM לזיהוי פלואורסצנטיות בהתבסס על תגובת RPA-CRISPR/Cas12a. איור זה שונה (מיקום תמונה וצבע מותאמים) באישור Lei et al.35. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
5. טיפול במגלשת זכוכית לגילוי עם SPM
- הכינו פולידימתילסילוקסאן (PDMS) על ידי ערבוב הבסיס וחומר הריפוי ביחס של 10:1, ולאחר מכן אפו על פלטה חמה בטמפרטורה של 80°C למשך שעתיים.
- יש לטפל הן ב-PDMS והן במגלשת הזכוכית (אורך: 75 מ"מ; גובה: 50 מ"מ) עם טיפול פלזמה חמצן במשך 120 שניות, ולאחר מכן לחץ אותם יחד.
- אופים את הכוס/PDMS בטמפרטורה של 95°C למשך שעתיים; הוא חתום לצמיתות על ידי קשר Si-O-Si. PDMS הוא בעל שקיפות גבוהה וללא פלואורסצנטיות אוטומטית, מה שתורם לזיהוי SPM. הטיפול במגלשת הזכוכית וב-PMDS מתבצע לפי He et al.32
6. זיהוי CRISPR/Cas12a עם SPM
- השתמש ב- crRNA (טבלת חומרים) של רצף המטרה ובבדיקה של כתב ssDNA המקשרת בין פלואורופור ומרווה עבור CRISPR/Cas12a. בשיטה זו, קרבוקסי טטרמתילרודאמין (TAMRA) מקושר כפלואורופור בקצוות 5' של בדיקות כתב ssDNA וחור שחור קוונצ'ר-2 (BHQ2) כמרווה בקצוות 3'. הרצף המפורט של crRNA וכתב ssDNA מתואר בטבלה 5.
- הכינו את החלבון Lachnospiraceae bacterium Cas12a (LbCas12a) עם 10x CRISPR/Cas12a Reaction Buffer.
- המסת 1 μL של תוצר תגובת RPA מסעיף 2 במאגר תגובה 1x CRISPR/Cas12a עם קומפלקסים LbCas12a-crRNA ובדיקת כתב ssDNA של 500 ננומטר בנפח תגובה של 100 μL.
- לאחר ערבוב LbCas12a ו-crRNA, הניחו לתערובת לשבת לפחות 5 דקות כדי ליצור קומפלקס פונקציונלי. לאחר הדגירה, הוסף רכיבים אחרים לתערובת התגובה והשלם את התגובה ב 37 ° C. הנפח הסופי של כל תגובת CRISPR/Cas12a הוא 100 μL. הריכוזים המפורטים של כל רכיב בכל תגובת CRISPR/Cas12a מתוארים בטבלה 6.
- בצע את תגובת הזיהוי של 100 μL CRISPR/Cas12a על מגלשת הזכוכית שטופלה מראש וכסה אותה בכיסוי. דגרו על מגלשת הזכוכית בתגובה ב-RT למשך 10 דקות.
- מדוד את אותות הפלואורסצנטיות על-ידי SPM. שים את שקופית הזכוכית עם תגובת הזיהוי על הבמה של SPM, לשמור על מרחק מתאים, להתאים את אורך מוקד ובהירות, ולאחר מכן לחפש את שדה הראייה של התגובה ולמקד אותו כדי ללכוד תמונה.
הערה: תחילה יש לקבל עקומה סטנדרטית כדי שניתן יהיה לשנות את קנה המידה של הנתונים מהדגימות לטווח הריכוז המשוער. נעשה שימוש בריכוזים שונים של מטרות מטוהרות, כולל 10 ננומטר, 1 ננומטר, 100 pM ו-10 pM, וירוס נוסף כבקרה שלילית (לפני RPA).
7. מערך נתונים והגדלת נתונים
- אסוף את התמונות הפלואורסצנטיות מבדיקת הזיהוי בסעיף 6 כערכות הנתונים. חזור על לפחות שלושה זיהויים מקבילים עבור כל דגימה כדי להבטיח הקבלה בין הנתונים.
- ניתן לאשר כמה דרכים מתאימות להשגת הקבלה גבוהה יותר. לדוגמה, בעת איסוף תמונות פלואורסצנטיות מכל דגימה, התמקד ידנית וחפש שדה בהיר יחסית לצילום תמונות. במקביל, צלמו כל דגימה כדי להשיג את האות הפלואורסצנטי ביותר מחמישה מקומות שונים.
- מדוד את הערך האפור הממוצע של כל תמונה ואת סטיית התקן של הערך האפור הממוצע בקבוצת ריכוז לפי ImageJ.
- הגדר טווח עוצמות [חציון - סטיית תקן, חציון + סטיית תקן] לניקוי נתונים.
הערה: בתמונות המתקבלות בשלבים המוקדמים, ייתכנו תמונות עם הבדלים גדולים, ולכן יש צורך לסנן את התמונות. אם העוצמות של התמונות חורגות מהסף שנקבע, יש לראות בהן חריגות ולא נכללות. - תייגו את התמונות עבור המטרה המטוהרת בריכוזים הולכים וגדלים עם 0-6 בסדר עולה, בהתאמה.
- כדי לשפר את עמידות המערכת ולמנוע התאמת יתר, הטמיעו טכניקות הגדלת תמונה כגון היפוך אופקי, היפוך אנכי ורעש אקראי באמצעות פונקציות טרנספורמציה בפייתון. פעולה זו מסייעת להציג וריאציות בערכת הנתונים.
8. העברת למידה
- כרשת עמוד השדרה, אמצו את מודל הלמידה העמוקה AlexNet33 לסיווג.
- כדי לספק את האילוץ של המודל שהוכשר מראש בסעיף 7, עצב מחדש את תמונות הקלט ל- 224 פיקסלים x 224 פיקסלים x 3 ערוצים (גובה ורוחב של 224 פיקסלים ועומק של 3 ערוצים לערוצי הצבע אדום, ירוק וכחול) באמצעות פונקציות שינוי צורה ב- Python.
הערה: שלב זה הוא עיבוד מקדים נפוץ עבור נתונים הטרוגניים בלמידת העברה, כולל המרה. - השתמש ברשת עמוד שדרה מאומנת מראש עם ערכת הנתונים של ImageNet כדי לחלץ תכונות תוך מינוף המשקולות של שכבות ביניים נסתרות שנלמדו.
- בהקשר של משימת סיווג פלואורסצנטי, החלף את השכבה הסופית המחוברת במלואה של הרשת העצבית, שכללה במקור 1000 נוירונים עבור משימת ImageNet, בשכבה מחוברת במלואה עם 2 או 7 נוירונים.
- הערך את הביצועים של מודל אימון ההתקנה באמצעות סדרה של מדדים, כולל מטריצת בלבול, דיוק, דיוק, היזכרות וציון F1 בהתבסס על Lawton ו- Viriri34.
תוצאות
שיטה זו מתמקדת במערכת זיהוי מהירה, קלה ליישום, רגישה ביותר ונקודת טיפול (POC) עבור נגיפי DNA. תכנון זוגות פריימרים עבור תגובת RPA ועיצוב crRNA עבור תגובת CRISPR/Cas12a הם שניים מהחלקים החיוניים מכיוון שהם ישפיעו על יעילות תגובת RPA-CRISPR/Cas12a וישפיעו על הזיהוי והסיווג הבאים.
בשיטה ...
Discussion
בשיטה זו, אנו מפתחים מערכת מהירה, קלה ליישום, רגישה מאוד, ספציפית לרצף וזיהוי וירוסי DNA POC בסיוע AI. לאחר קבלת דגימות, RPA מוחל כדי להגביר את רצף המטרה, ולאחר מכן CRISPR/Cas12a יכול לזהות את DNA המטרה ולשחרר פלואורסצנטיות, אשר מגדילה את אות האיתור. מיקרוסקופ סמארטפון נייד בנוי לצלם תמונות פלואורסצנטיות,...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכת על ידי הקרן הלאומית למדעי הטבע של סין 31970752, מדע, טכנולוגיה, חדשנות הוועדה של עיריית שנזן JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; מימון פתוח של מעבדת מפרץ שנזן, SZBL2020090501004; הקרן למדע פוסט-דוקטורט בסין 2020M680023; והמנהל הכללי של המכס של הרפובליקה העממית של סין 2021HK007.
Materials
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
References
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved