Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
DNA-Virusnachweissystem basierend auf RPA-CRISPR/Cas12a-SPM und Deep Learning
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir stellen ein Protokoll vor, das die Rekombinase-Polymerase-Amplifikation mit einem CRISPR/Cas12a-System für den Spurennachweis von DNA-Viren kombiniert und eine tragbare Smartphone-Mikroskopie mit einer durch künstliche Intelligenz unterstützten Klassifizierung für den Point-of-Care-Nachweis von DNA-Viren entwickelt.
Zusammenfassung
Wir berichten über ein schnelles, einfach zu implementierendes, hochsensitives, sequenzspezifisches und Point-of-Care (POC) DNA-Virus-Nachweissystem, das die Rekombinase-Polymerase-Amplifikation (RPA) und das CRISPR/Cas12a-System für den Spurennachweis von DNA-Viren kombiniert. Die Ziel-DNA wird durch RPA und CRISPR/Cas12a separat amplifiziert und erkannt, was die kollaterale Spaltungsaktivität von Cas12a auslöst, die einen mit Fluorophor-Quenchern markierten DNA-Reporter spaltet und die Fluoreszenz verallgemeinert. Für die POC-Erkennung ist die tragbare Smartphone-Mikroskopie für die Aufnahme von Fluoreszenzbildern ausgelegt. Darüber hinaus werden Deep-Learning-Modelle für die binäre Klassifizierung positiver oder negativer Proben eingesetzt, die eine hohe Genauigkeit erreichen. Das Froschvirus 3 (FV3, Gattung Ranavirus, Familie Iridoviridae) wurde als Beispiel für dieses DNA-Virus-POC-Nachweissystem getestet, und die Nachweisgrenzen (LoD) können innerhalb von 40 Minuten 10 aM erreichen. Ohne geschulte Bediener und sperrige Instrumente zeigt das tragbare und miniaturisierte RPA-CRISPR/Cas12a-SPM mit künstlicher Intelligenz (KI) unterstützte Klassifizierung ein großes Potenzial für den Nachweis von POC-DNA-Viren und kann dazu beitragen, die Ausbreitung solcher Viren zu verhindern.
Einleitung
In den letzten Jahren kam es häufig zu Epidemien von Infektionskrankheiten, die durch verschiedene Viren verursacht wurden, darunter die Ebola-Virusvirus-Epidemie (EVD) in den Jahren 20141 und 20182, das Middle East Respiratory Syndrome (MERS) im Jahr 20153, die Zika-Virus-Epidemie im Jahr 20154, die Coronavirus-Krankheit 2019 (COVID-19), die durch das schwere akute respiratorische Syndrom Coronavirus 2 (SARS-CoV-2)5 verursacht wurde, und die anhaltenden Affenpocken, die durch das Affenpockenvirus (MKPV) verursacht wurden, im Jahr 20226. Diese plötzlichen Ausbrüche epidemischer Infektionskrankheiten verursachen eine große Zahl von Todesfällen und bringen enorme wirtschaftliche Verluste und soziale Unruhen mit sich. Ein schnelles und genaues Nachweissystem ist dringend erforderlich, um die Infektion schnell zu diagnostizieren und eine weitere Ausbreitung des Virus zu verhindern.
In jüngster Zeit haben geclusterte CRISPR-Proteine (Regularly Interspaced Short Palindromic Repeats) und CRISPR-assoziierte (Cas) Proteine weltweite Aufmerksamkeit erregt und vielversprechende Ergebnisse beim Nachweis von Nukleinsäuren gezeigt 7,8,9,10,11,12,13,14,15 . Das CRISPR/Cas12a-Protein, das von CRISPR-RNA (crRNA) gesteuert wird, bindet an die Ziel-DNA und spaltet sie. Diese Aktivität führt zur Freisetzung von unspezifischer einzelsträngiger DNA (ssDNA), die als Trans-Spaltung bekannt ist, und kann verwendet werden, um das Nachweissignal für den Nukleinsäurenachweis zu verbessern. Einige traditionelle Nachweismethoden wie die Polymerase-Kettenreaktion (PCR), die quantitative Echtzeit-PCR (qPCR) und der Enzyme-Linked Immunosorbent Assay (ELISA) sind kompliziert, zeitaufwändig und kostspielig für den Point-of-Care-Nachweis (POC). In unserer bisherigen Arbeit haben wir erfolgreich ein automatisiertes, integriertes und kostengünstiges Nachweissystem für das Afrikanische Schweinepestvirus (ASFV) entwickelt, das auf der CRISPR/Cas12a-Technologie basiert. In diesem System erreichten wir eine Nachweisgrenze von 1 pM innerhalb eines Zeitrahmens von 2 Stunden, ohne dass eine Amplifikation erforderlich war. Das CRISPR/Cas12a-System und die Rekombinase-Polymerase-Amplifikation (RPA) werden kombiniert, um die Sensitivität und Spezifität für den Nachweis von DNA-Spuren zu verbessern. Im Vergleich zu anderen isothermen Amplifikationstechniken ist RPA einfach aufgebaut und bequem zu bedienen, da es eine kürzere Reaktionszeit ohne ausgeklügelte Temperaturkontrollgeräte hat.
Für den POC-Nachweis von Krankheitserregern werden Instrumente wie Smartphone-Mikroskopie (SPM), Handheld-Fluorimeter oder Lateral-Flow-Strips für die Ergebnisauslesungen 16,17,18 entwickelt. SPM erfasst Bilder über eine Kamera und lädt sie zur schnellen Datenanalyse in einige mobile Anwendungen hoch. Eine solche Mikroskopie stellt ein tragbares, kostengünstiges und miniaturisiertes Signalerfassungssystem mit hoher Empfindlichkeit her und hat Vorteile beim Nachweis von Krankheitserregern wie H5N1, dem Zika-Virus und SARS-CoV-2 gezeigt19,20. Daher bauen wir ein tragbares SPM, um die Fluoreszenzsignale abzufangen, die durch die RPA-CRISPR/Cas12a-Detektion des Ziel-DNA-Virus ausgelöst werden. Die ssDNA-Reportersonde, die einen Fluorophor und einen Quencher verbindet, wird gespalten, wenn CRISPR/Cas12a das Ziel-DNA-Virus erkennt, und die vom Fluorophor emittierte Fluoreszenz kann von SPM erfasst werden.
Im Vergleich zu der professionellen Software, die üblicherweise verwendet wird, um die Ergebnisinformationen aus den Fluoreszenzbildern von SPM21 zu erhalten, verwenden einige Experten maschinelles Lernen und Deep Learning, um die Konzentrationen der Virus-DNA zu quantifizieren, nachdem sie Fluoreszenzbildererhalten haben 22, was zeitaufwändiger ist. Wenn es um die Klassifizierung medizinischer Bilder geht, werden häufig herkömmliche neuronale Netze (CNNs) verwendet, um Merkmale aus den verpixelten Rohbildern auf End-to-End-Weise zu lernen 23,24,25,26. Beliebte CNN-basierte Deep-Learning-Modelle wie AlexNet, DenseNet-121 und EfficientNet-B7 wurden in diesem Bereich erfolgreich eingesetzt27,28. Die Beschaffung großer Datensätze in bestimmten Bereichen kann jedoch eine Herausforderung darstellen und erfordert Transferlernen29,30. Bei diesem Ansatz wird ein Deep-Learning-Modell mit einem großen Datensatz vortrainiert, und das vortrainierte Modell wird als Ausgangspunkt für eine neue Aufgabe mit einem kleinen Datensatz verwendet. Diese Technik kann den Bedarf an großen Datensätzen reduzieren, Überanpassung bekämpfen und die Trainingszeit verkürzen31. Hier verwenden wir Deep-Learning-Modelle mit Transfer-Learning für die binäre Klassifizierung der Fluoreszenzbilder der positiven und negativen Proben.
Bei dieser Methode kombinieren wir RPA und das CRISPR/Cas12a-System zur Spurendetektion von DNA-Viren. Die Ziel-DNA wird durch RPA und CRISPR/Cas12a separat amplifiziert und erkannt, was die kollaterale Spaltungsaktivität von Cas12a auslöst, die einen mit Fluorophor-Quenchern markierten DNA-Reporter spaltet und die Fluoreszenz verallgemeinert. Wir bauen ein tragbares SPM, um die Fluoreszenzbilder für die POC-Detektion aufzunehmen und entwickeln Deep-Learning-Modelle für die binäre Klassifizierung. Das Schema des gebauten POC-Erkennungssystems ist in Abbildung 1 dargestellt. Ohne geschickte Bediener und sperrige Instrumente zeigt das RPA-CRISPR/Cas12a-SPM mit künstlicher Intelligenz (KI) unterstützter Klassifizierung ein großes Potenzial für den Nachweis von POC-DNA-Viren.
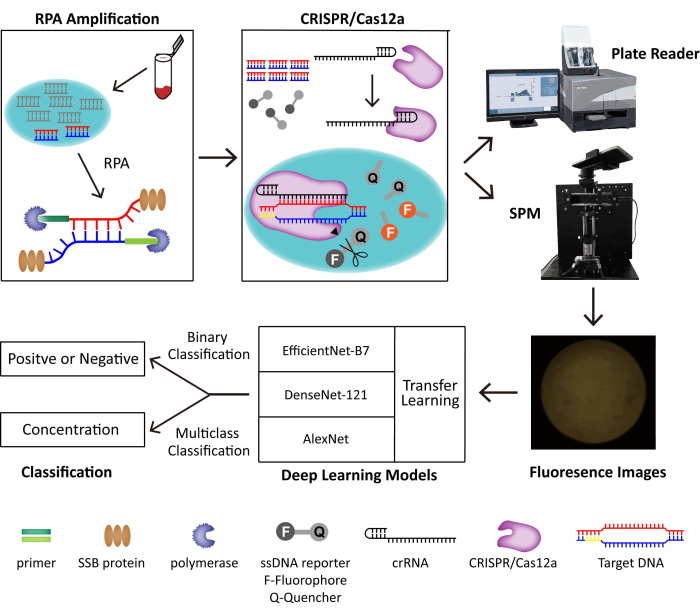
Abbildung 1: Das Schema des RPA-CRISPR/Cas12-SPM-Detektionssystems zusammen mit der KI-Klassifizierung für gesammelte Bilder. Die Nukleinsäuren tierischer Proben werden von PINDBK freigesetzt. Die Ziel-DNA des Virus wird durch das RPA-CRISPR/Cas12a-System amplifiziert und spezifisch erkannt. CRISPR/Cas12a bindet an crRNA und der Cas12a-crRNA-Komplex an die Ziel-DNA, was die kollaterale Spaltung von CRISPR/Cas12a auf den ssDNA-Reportersonden auslöst. Das Fluorophor auf dem Reporter wird freigesetzt und die Fluoreszenz wird von einem kommerzialisierten Platten-Reader oder dem von uns entwickelten SPM detektiert. Drei verschiedene Deep-Learning-Modelle, darunter AlexNet, DenseNet-121 und EfficientNet-B7 mit Transfer-Learning, werden zur Klassifizierung der Fluoreszenzbilder verwendet. Diese Zahl wird mit Genehmigung von Lei et al.35 wiederverwendet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
1. Verarbeitung der Proben
- Nehmen wir das Froschvirus 3 (FV3, Gattung Ranavirus, Familie Iridoviridae), ein doppelsträngiges DNA-Virus. Wählen Sie das Major Capsid (mcp)-Gen als Ziel für den Nachweis von FV3, da es hochkonserviert ist und normalerweise als Ziel für den Ranavirus-Nachweis angesehen wird. Die ausgewählte Zielsequenz ist in Tabelle 1 dargestellt.
HINWEIS: Frog Virus 3 wird in diesem Protokoll als Beispiel genommen. - Verwenden Sie für die Vorbereitung der Ziel-DNA-Fragmente DNA-Fragmente des mcp-Gens von FV3 und dem Infectious Spleen and Kidney Necrosis Virus (ISKNV, ein weiteres Ranavirus).
HINWEIS: In dieser Studie wurden die Ziel-DNA-Fragmente kommerziell von einem Unternehmen bezogen, das in der Materialtabelle aufgeführt ist. Sie gelten als Ziel und Kontrolle der anschließenden Detektion.
| Name | Reihenfolge | ||
| FV3 MCP | NTS: 5' ... gtaacccggctttcGGGCAGCAGTTTCGGGGGGCGTtcccaggtcg... 3' (240 bps) | ||
| TS: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgccgaaagc... 3' (240 bps) | |||
| ISKNV MCP | NTS: 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 bps) | ||
| TS: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 bps) | |||
Tabelle 1: In dieser Methode ausgewählte Zielsequenz.
2. RPA-Reaktion
- Entwerfen und synthetisieren Sie die RPA-Primer-Paare für die Zielsequenz. Die Sequenzen von RPA-Primer-Paaren sind in Tabelle 2 beschrieben.
- Bereiten Sie den 5x RPA-Reaktionspuffer (Tabelle 3) und MgCl2 (100 mM) vor.
- Mischen Sie die vier wichtigsten RPA-Enzyme (UvsX, UvsY, GP32, Bsu-Protein) im 1x RPA-Reaktionspuffer zusammen mit den im Voraus entwickelten Primern, wie in Tabelle 4 beschrieben.
- Die Mischung gründlich einreiben.
- Fügen Sie 1 μl des in Schritt 1 erhaltenen Ziels für jede RPA-Reaktion hinzu und mischen Sie es erneut gründlich durch Vortex.
- Fügen Sie 7 μl MgCl2 (100 mM) hinzu, um die Reaktion zu starten. Das Endvolumen jeder RPA-Reaktion beträgt 50 μl.
- Führen Sie den Assay 30 Minuten lang bei 37 °C durch.
- Das RPA-Produkt kann einige Tage bei 4 °C gelagert werden. Wenn sich RPA-Produkte verschlechtern, sollten Sie sie so schnell wie möglich zur weiteren Erkennung verwenden, um bessere Diagnoseergebnisse zu erhalten.
- [Fakultativ] Führen Sie eine DNA-Gelelektrophorese durch.
- Nehmen Sie 5 μl RPA-Produkte heraus, fügen Sie ein entsprechendes Volumen von 6x DNA-Ladepuffer hinzu und führen Sie die DNA-Gelelektrophorese im Trisacetat-EDTA (TAE)-Puffer durch.
- Lassen Sie die Elektrophorese etwa 20 Minuten lang unter 120 V laufen, bis die Bänder des Ladepuffers den Boden des Gels erreichen.
- Bestimmen Sie, ob die Zielsequenz erfolgreich aus der Probe amplifiziert wurde, indem Sie die Größe der Banden der Probe mit den Banden des Markers vergleichen.
| Name | Reihenfolge |
| RPA-Einführung F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| RPA-Einführung R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Tabelle 2: RPA-Primer, die bei dieser Methode verwendet werden.
| Bestandteil | Ursprüngliche Konzentration | Addition |
| PEG 20.000 | - | 114 mg |
| ATP | ca. 100 mM | 125 μL |
| dNTPs | 25 mM | 48 μL |
| Tris-HCl | 1 Mio. | 125 μL |
| DVB-T | 1 Mio. | 125 μL |
| Phosphokreatin | 1 Mio. | 250 μL |
| Kreatinkinase | 10 μg/μL | 50 μL |
| ddH2Ω | - | 277 μL |
| Gesamtvolumen | - | 1 ml |
Tabelle 3: Die Zusammensetzung des 5x RPA-Reaktionspuffers (pH 7,5).
| Bestandteil | Ursprüngliche Konzentration | Addition |
| 5x RPA-Reaktionspuffer | - | 10 μL |
| UvsX-Eiweiß | 5 mg/ml | 2,6 μl |
| UvsY-Protein | 5 mg/ml | 0,9 μl |
| GP32-Eiweiß | 5 mg/ml | 2,54 μl |
| Bsu-Protein | 5 mg/ml | 0,88 μl |
| Vorwärts-Grundierung | 100 μM | 0,25 μl |
| Umgekehrte Grundierung | 100 μM | 0,25 μl |
| ddH2Ω | - | 24,58 μl |
| Ziel | - | 1 μL |
| *MgCl2 | ca. 100 mM | 7 μL |
| Gesamtvolumen | 50 μL | |
| *MgCl2 muss zuletzt hinzugefügt werden, um die RPA-Reaktion zu initiieren. | ||
Tabelle 4: Die Zusammensetzung der RPA-Reaktion.
3. CRISPR/Cas12a-Nachweis ohne SPM
- Verwenden Sie die crRNAs (Table of Materials) der Zielsequenz und die ssDNA-Reportersonde, die ein Fluorophor und einen Quencher für CRISPR/Cas12a verbindet. Hier ist Carboxytetramethylrhodamin (TAMRA) als Fluorophor an den 5'-Enden der ssDNA-Reportersonden und Black Hole Quencher-2 (BHQ2) als Quencher an den 3'-Enden verknüpft. Die detaillierte Sequenz von crRNA und ssDNA-Reporter ist in Tabelle 5 beschrieben.
- Bereiten Sie das Protein des Bakteriums Lachnospiraceae Cas12a (LbCas12a) mit 10x CRISPR/Cas12a-Reaktionspuffer vor.
- 1 μl RPA-Reaktionsprodukt aus Abschnitt 2 in 1x CRISPR/Cas12a-Reaktionspuffer mit LbCas12a-crRNA-Komplexen und 500 nM ssDNA-Reportersonde in einem 100 μl Reaktionsvolumen lösen.
- Lassen Sie die Mischung nach dem Mischen von LbCas12a und crRNA mindestens 5 Minuten ruhen, um einen funktionellen Komplex zu bilden. Nach der Inkubation werden dem Reaktionsgemisch weitere Komponenten zugesetzt und die gesamte Reaktion bei 37 °C durchgeführt. Das Endvolumen jeder CRISPR/Cas12a-Reaktion beträgt 100 μl. Die detaillierten Konzentrationen der einzelnen Komponenten in jeder CRISPR/Cas12a-Reaktion sind in Tabelle 6 beschrieben.
- Die 100 μl CRISPR/Cas12a-Nachweisreaktion bei 37 °C für 30 min durchführen.
- Untersuchen Sie die Fluoreszenzsignale konstant mit einem Mikroplatten-Reader bei einer Anregungswellenlänge von 535 nm und einer Emissionswellenlänge von 595 nm mit einer Verstärkung von 60.
HINWEIS: Die Detektion verschiedener Wellenlängen von Anregungs- und Emissionslicht hängt von der Wahl des Fluorophors und des Quenchers in den zuvor entwickelten ssDNA-Reportersonden ab. - Teilen Sie für diese gesammelten Daten den Kontrollwert durch die Messung der positiven Proben, um alle Daten zu normalisieren, und integrieren Sie sie dann für eine t-Test-Analyse mit zwei Stichproben.
| Name | Reihenfolge | |
| LbCas12a crRNA für FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGCGT | |
| ssDNA-Reporter | /5TAMRA/TTATT/3BHQ2 | |
Tabelle 5: Sequenzen von CRISPR/Cas12a crRNA und ssDNA-Reportern, die bei dieser Methode verwendet werden.
| Bestandteil | Ursprüngliche Konzentration | Addition |
| NEBuffer r2.1 | - | 10 μL |
| Lba Cas12a (Cpf1) | 10 μM | 0,5 μl |
| crRNA | 10 μM | 0,625 μl |
| Warten Sie mindestens 5 Minuten, bis sich der LbCas12a/crRNA-Komplex verbindet. | ||
| DNA-Reporter | 100 μM | 0,5 μl |
| ddH2Ω | - | 87,375 μL |
| Ziel | - | 1 μL |
| Gesamtvolumen | - | 100 μL |
Tabelle 6: Die Zusammensetzung der CRISPR/Cas12a-Reaktion.
4. SPM-Einrichtung
- Stellen Sie für den Anregungspfad einen Laserstrahl so ein, dass er durch die ND-Filter (Neutral Density) geht, um die Laserintensität zu dämpfen.
- Erzeugen Sie einen kollimierten Strahl aus einer asphärischen Linse (im Folgenden als L1 bezeichnet) und reflektieren Sie ihn durch den dichroitischen Spiegel (DM).
- Richten Sie das Licht durch das Objektiv (20x) auf den Objektträger, wo die Probe platziert wird, um die Fluoreszenz der Probe zu beleuchten und anzuregen. Der Probentisch ermöglicht eine präzise Einstellung der Brennebene, indem der Strahl auf die hintere Brennebene des Objektivs gerichtet wird. Die oben genannten Schritte bilden den Anregungspfad des SPM-Instruments.
- Für den Emissionspfad ist eine externe Linse (im Folgenden als L2 bezeichnet) so zu positionieren, dass auf der anderen Seite des Objektivs ein Zwischenbild entsteht. Das Objektiv beleuchtet gleichzeitig die Probe und sammelt das Emissionssignal.
- Zeichnen Sie das Fluoreszenzsignal der Probe mit dem Smartphone auf, das am Ende des Emissionspfads platziert ist. Verwenden Sie eine stabile Halterung, um ein Schütteln zu vermeiden.
- Stellen Sie einen Bandpassfilter zwischen L1 und der Kamera des Smartphones ein, um das angeregte Licht herauszufiltern, während nur das von der Probe emittierte Licht die Kamera erreicht, wodurch die Detektion optimiert werden kann.
- Immobilisieren Sie das SPM-Setup auf einem Steckbrett für den portablen Einsatz.
HINWEIS: Das Schema und das physikalische Erscheinungsbild des SPM-Geräts für die Fluoreszenzdetektion auf der Grundlage der RPA-CRISPR/Cas12a-Reaktion sind in Abbildung 2 dargestellt. Die CRISPR/Cas12a-Detektion erfolgt auf dem im nächsten Schritt beschriebenen vorbehandelten Objektträger.
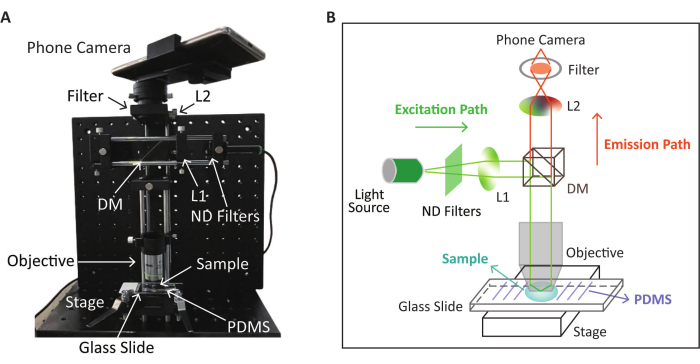
Abbildung 2: Schematisches und physikalisches Erscheinungsbild des SPM-Geräts, das für die Fluoreszenzdetektion verwendet wird. (A) Das physikalische Erscheinungsbild des SPM-Geräts für die Fluoreszenzbilderfassung nach der RPA-CRISPR/Cas12a-Reaktion. (B) Schematische Darstellung des SPM-Geräts zur Fluoreszenzdetektion auf der Grundlage der RPA-CRISPR/Cas12a-Reaktion. Diese Abbildung wurde mit Genehmigung von Lei et al.35 geändert (Bildposition und -farbe angepasst). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Behandlung des Objektträgers zur Detektion mit SPM
- Polydimethylsiloxan (PDMS) wird durch Mischen von Base und Härter im Verhältnis 10:1 hergestellt und anschließend auf einer Herdplatte bei 80 °C für 2 h gebacken.
- Behandeln Sie sowohl PDMS als auch den Objektträger (Länge: 75 mm; Höhe: 50 mm) 120 s lang mit Sauerstoffplasma behandeln, dann zusammendrücken.
- Das Glas/PDMS bei 95 °C 2 h backen; Es wird durch die Si-O-Si-Bindung dauerhaft abgedichtet. PDMS hat eine hohe Transparenz und keine Autofluoreszenz, was der SPM-Detektion förderlich ist. Die Behandlung des Objektträgers und des PMDS erfolgt nach He et al.32
6. CRISPR/Cas12a-Nachweis mit SPM
- Verwenden Sie die crRNAs (Table of Materials) der Zielsequenz und die ssDNA-Reportersonde, die ein Fluorophor und einen Quencher für CRISPR/Cas12a verbindet. Bei dieser Methode wird Carboxytetramethylrhodamin (TAMRA) als Fluorophor an den 5'-Enden der ssDNA-Reportersonden und Black Hole Quencher-2 (BHQ2) als Quencher an den 3'-Enden verknüpft. Die detaillierte Sequenz von crRNA und ssDNA-Reporter ist in Tabelle 5 beschrieben.
- Bereiten Sie das Protein des Bakteriums Lachnospiraceae Cas12a (LbCas12a) mit 10x CRISPR/Cas12a-Reaktionspuffer vor.
- 1 μl RPA-Reaktionsprodukt aus Abschnitt 2 in 1x CRISPR/Cas12a-Reaktionspuffer mit LbCas12a-crRNA-Komplexen und 500 nM ssDNA-Reportersonde in einem 100 μl Reaktionsvolumen lösen.
- Lassen Sie die Mischung nach dem Mischen von LbCas12a und crRNA mindestens 5 Minuten ruhen, um einen funktionellen Komplex zu bilden. Nach der Inkubation werden dem Reaktionsgemisch weitere Komponenten zugesetzt und die Reaktion bei 37 °C abgeschlossen. Das Endvolumen jeder CRISPR/Cas12a-Reaktion beträgt 100 μl. Die detaillierten Konzentrationen der einzelnen Komponenten in jeder CRISPR/Cas12a-Reaktion sind in Tabelle 6 beschrieben.
- Führen Sie die 100 μl CRISPR/Cas12a-Nachweisreaktion auf dem vorbehandelten Objektträger durch und decken Sie ihn mit einem Deckglas ab. Inkubieren Sie den Objektträger mit Reaktion bei RT für 10 min.
- Messen Sie die Fluoreszenzsignale mit dem SPM. Legen Sie den Objektträger mit der Detektionsreaktion auf den Tisch des SPM, halten Sie einen angemessenen Abstand ein, stellen Sie die Brennweite und Klarheit ein, suchen Sie dann nach dem Sichtfeld der Reaktion und fokussieren Sie es, um ein Bild aufzunehmen.
HINWEIS: Zuerst sollte eine Standardkurve erstellt werden, damit die Daten aus den Proben auf den ungefähren Konzentrationsbereich skaliert werden können. Es werden verschiedene Konzentrationen von gereinigten Zielmolekülen verwendet, darunter 10 nM, 1 nM, 100 pM und 10 pM, ein weiteres Virus als Negativkontrolle (vor RPA).
7. Datensatz- und Datenerweiterung
- Sammeln Sie die Fluoreszenzbilder aus dem Detektionsassay in Abschnitt 6 als Datensätze. Wiederholen Sie mindestens drei parallele Erkennungen für jede Stichprobe, um die Datenparallelität sicherzustellen.
- Einige geeignete Wege zur Erreichung einer höheren Parallelität können genehmigt werden. Wenn Sie beispielsweise die Fluoreszenzbilder von jeder Probe sammeln, fokussieren Sie manuell und suchen Sie nach einem relativ helleren Feld, um Bilder aufzunehmen. Fotografieren Sie gleichzeitig jede Probe, um das Fluoreszenzsignal an mehr als fünf verschiedenen Stellen zu erhalten.
- Messen Sie den mittleren Grauwert jedes Bildes und die Standardabweichung des mittleren Grauwerts in einer Konzentrationsgruppe mit ImageJ.
- Legen Sie einen Intensitätsbereich [Median - Standardabweichung, Median + Standardabweichung] für die Datenbereinigung fest.
HINWEIS: In den Bildern, die in den ersten Schritten erhalten wurden, kann es Bilder mit großen Unterschieden geben, so dass es notwendig ist, die Bilder zu überprüfen. Wenn die Intensitäten der Bilder außerhalb des festgelegten Schwellenwerts liegen, sollten sie als Ausreißer betrachtet und ausgeschlossen werden. - Beschriften Sie die Bilder für das gereinigte Ziel bei steigenden Konzentrationen mit 0-6 in aufsteigender Reihenfolge.
- Um die Robustheit des Systems zu verbessern und eine Überanpassung zu verhindern, implementieren Sie Bilderweiterungstechniken wie horizontales Drehen, vertikales Spiegeln und zufälliges Rauschen durch Transformationsfunktionen in Python. Dies hilft, Variationen im Datensatz einzuführen.
8. Lernen übertragen
- Verwenden Sie als Backbone-Netzwerk das Deep-Learning-Modell AlexNet33 für die Klassifizierung.
- Um die Einschränkung des vortrainierten Modells in Abschnitt 7 zu erfüllen, formen Sie die Eingabebilder mithilfe von Transformationsfunktionen in Python auf 224 Pixel x 224 Pixel x 3 Kanäle (Höhe und Breite von 224 Pixeln und eine Tiefe von 3 Kanälen für die Farbkanäle Rot, Grün und Blau) um.
HINWEIS: Dieser Schritt ist eine gängige Vorverarbeitung für heterogene Daten im Transferlernen, einschließlich Transformieren. - Verwenden Sie ein vortrainiertes Backbone-Netzwerk mit dem ImageNet-Dataset, um Features zu extrahieren und gleichzeitig die Gewichtungen der zwischengeschalteten verborgenen Schichten zu nutzen, die Sie gelernt haben.
- Ersetzen Sie im Auftragskontext für die Fluoreszenzklassifizierung die letzte vollständig verbundene Schicht des neuronalen Netzwerks, die ursprünglich 1000 Neuronen für die ImageNet-Aufgabe umfasste, durch eine vollständig verbundene Schicht mit 2 oder 7 Neuronen.
- Bewerten Sie die Leistung des Setup-Trainingsmodells mithilfe einer Reihe von Metriken, darunter Konfusionsmatrix, Genauigkeit, Präzision, Abruf und F1-Punktzahl basierend auf Lawton und Viriri34.
Ergebnisse
Diese Methode konzentriert sich auf ein schnelles, einfach zu implementierendes, hochsensitives und Point-of-Care (POC) Nachweissystem für DNA-Viren. Das Design der Primerpaare für die RPA-Reaktion und das crRNA-Design für die CRISPR/Cas12a-Reaktion sind zwei der wesentlichen Teile, da sie die Effizienz der RPA-CRISPR/Cas12a-Reaktion beeinflussen und den anschließenden Nachweis und die Klassifizierung beeinflussen.
Bei dieser Methode gilt FV3 als Beispiel für den Nac...
Diskussion
Bei dieser Methode entwickeln wir mit KI-Unterstützung ein schnelles, einfach zu implementierendes, hochsensitives, sequenzspezifisches und POC-DNA-Virusnachweissystem. Nach der Entnahme von Proben wird RPA angewendet, um die Zielsequenz zu amplifizieren, und dann kann CRISPR/Cas12a die Ziel-DNA erkennen und Fluoreszenz freisetzen, die das Detektionssignal vergrößert. Die tragbare Smartphone-Mikroskopie ist für die Aufnahme von Fluoreszenzbildern ausgelegt, und Deep-Learning-Modelle mit Transferlernen werden für die...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wird unterstützt von der National Natural Science Foundation of China 31970752, Science, Technology, Innovation Commission of Shenzhen Municipality JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Offene Finanzierung des Shenzhen Bay Laboratory, SZBL2020090501004; Stiftung für Postdoktorandenwissenschaften in China 2020M680023; und Allgemeine Zollverwaltung der Volksrepublik China 2021HK007.
Materialien
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Referenzen
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten