È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sistema di rilevamento del virus del DNA basato su RPA-CRISPR/Cas12a-SPM e Deep Learning
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentiamo un protocollo che combina l'amplificazione della polimerasi ricombinasi con un sistema CRISPR/Cas12a per il rilevamento di tracce di virus a DNA e costruisce una microscopia portatile per smartphone con una classificazione assistita dall'intelligenza artificiale per il rilevamento di virus a DNA point-of-care.
Abstract
Segnaliamo un sistema di rilevamento del DNA virus veloce, facile da implementare, altamente sensibile, specifico per la sequenza e point-of-care (POC), che combina l'amplificazione della ricombinasi polimerasi (RPA) e il sistema CRISPR/Cas12a per il rilevamento di tracce di virus a DNA. Il DNA bersaglio viene amplificato e riconosciuto separatamente da RPA e CRISPR/Cas12a, che innesca l'attività di scissione collaterale di Cas12a che slega un reporter di DNA marcato con fluoroforo e generalizza la fluorescenza. Per il rilevamento POC, la microscopia portatile per smartphone è progettata per acquisire immagini fluorescenti. Inoltre, all'interno del sistema vengono implementati modelli di deep learning per la classificazione binaria di campioni positivi o negativi, ottenendo un'elevata precisione. Il virus della rana 3 (FV3, generi Ranavirus, famiglia Iridoviridae) è stato testato come esempio per questo sistema di rilevamento POC del virus a DNA e i limiti di rilevamento (LoD) possono raggiungere 10 aM entro 40 minuti. Senza operatori qualificati e strumenti ingombranti, l'RPA-CRISPR/Cas12a-SPM portatile e miniaturizzato con classificazione assistita dall'intelligenza artificiale (AI) mostra un grande potenziale per il rilevamento del virus POC DNA e può aiutare a prevenire la diffusione di tali virus.
Introduzione
Negli ultimi anni si sono verificate frequentemente epidemie di malattie infettive causate da diversi virus, tra cui l'epidemia di malattia da virus Ebola (EVD) nel 20141 e nel 20182, la sindrome respiratoria del Medio Oriente (MERS) nel 20153, l'epidemia di malattia da virus Zika nel 20154, la malattia da coronavirus 2019 (COVID-19) causata dalla sindrome respiratoria acuta grave coronavirus 2 (SARS-CoV-2)5 e il continuo vaiolo delle scimmie causato dal virus del vaiolo delle scimmie (MKPV) nel 20226. Questi improvvisi focolai di malattie infettive epidemiche causano un gran numero di morti e portano enormi perdite economiche e disordini sociali. È urgentemente necessario un sistema di rilevamento rapido e accurato per diagnosticare rapidamente l'infezione e prevenire l'ulteriore diffusione del virus.
Recentemente, le brevi ripetizioni palindromiche (CRISPR) e le proteine associate a CRISPR (Cas) hanno guadagnato l'attenzione di tutto il mondo e hanno mostrato risultati promettenti nella rilevazione degli acidi nucleici 7,8,9,10,11,12,13,14,15 . La proteina CRISPR/Cas12a, guidata dall'RNA CRISPR (crRNA), si lega e scinde il DNA bersaglio. Questa attività porta al rilascio di DNA a filamento singolo (ssDNA) aspecifico, noto come trans-scissione, e può essere utilizzata per migliorare il segnale di rilevamento per il rilevamento dell'acido nucleico. Alcuni metodi di rilevamento tradizionali come la reazione a catena della polimerasi (PCR), la PCR quantitativa in tempo reale (qPCR) e il saggio di immunoassorbimento enzimatico (ELISA) sono complicati, dispendiosi in termini di tempo e costosi per il rilevamento point-of-care (POC). Il nostro lavoro precedente ha sviluppato con successo un sistema di rilevamento automatizzato, integrato ed economico per il virus della peste suina africana (ASFV) basato sulla tecnologia CRISPR/Cas12a. In questo sistema, abbiamo raggiunto un limite di rilevamento di 1 pM in un intervallo di tempo di 2 ore senza la necessità di amplificazione. Il sistema CRISPR/Cas12a e l'amplificazione della polimerasi ricombinasi (RPA) sono combinati per migliorare la sensibilità e la specificità per il rilevamento di tracce di DNA. Rispetto ad altre tecniche di amplificazione isotermica, l'RPA è semplice nel design e conveniente nel funzionamento poiché ha un tempo di reazione più breve senza sofisticate apparecchiature di controllo della temperatura.
Per il rilevamento POC di agenti patogeni, vengono sviluppati strumenti come la microscopia per smartphone (SPM), il fluorimetro portatile o le strisce a flusso laterale per le letture dei risultati 16,17,18. SPM cattura le immagini attraverso una fotocamera e le carica su alcune applicazioni mobili per una rapida analisi dei dati. Tale microscopia rende un sistema di acquisizione del segnale portatile, economico e miniaturizzato con un'elevata sensibilità e ha mostrato vantaggi nel rilevare agenti patogeni come H5N1, il virus Zika e SARS-CoV-219,20. Pertanto, costruiamo un SPM portatile per catturare i segnali di fluorescenza innescati dal rilevamento RPA-CRISPR/Cas12a del virus a DNA bersaglio. La sonda reporter ssDNA che collega un fluoroforo e un quencher verrà tagliata quando CRISPR/Cas12a riconosce il virus a DNA bersaglio e la fluorescenza emessa dal fluoroforo può essere catturata da SPM.
Rispetto al software professionale solitamente utilizzato per ottenere le informazioni sui risultati dalle immagini a fluorescenza di SPM21, alcuni esperti utilizzano l'apprendimento automatico e l'apprendimento profondo per quantificare le concentrazioni di DNA virale dopo aver ottenuto immagini a fluorescenza22, il che richiede più tempo. Quando si tratta di classificare le immagini mediche, le reti neurali convenzionali (CNN) vengono spesso utilizzate per apprendere le caratteristiche dalle immagini pixelate grezze in modo end-to-end 23,24,25,26. I popolari modelli di deep learning basati su CNN come AlexNet, DenseNet-121 ed EfficientNet-B7 sono stati applicati con successo in questo campo27,28. Tuttavia, ottenere grandi set di dati in domini specifici può essere impegnativo, richiedendo l'apprendimento del trasferimento29,30. Questo approccio esegue il pre-training di un modello di deep learning con un set di dati di grandi dimensioni e il modello pre-addestrato viene utilizzato come punto di partenza per una nuova attività con un set di dati di piccole dimensioni. Questa tecnica può ridurre la necessità di grandi set di dati, combattere l'overfitting e ridurre i tempi di addestramento31. In questo caso, utilizziamo modelli di deep learning con transfer learning per la classificazione binaria delle immagini di fluorescenza dei campioni positivi e negativi.
In questo metodo, combiniamo RPA e il sistema CRISPR/Cas12a per il rilevamento di tracce di virus a DNA. Il DNA bersaglio viene amplificato e riconosciuto separatamente da RPA e CRISPR/Cas12a, che innesca l'attività di scissione collaterale di Cas12a che slega un reporter di DNA marcato con fluoroforo e generalizza la fluorescenza. Costruiamo un SPM portatile per acquisire le immagini fluorescenti per il rilevamento POC e sviluppiamo modelli di deep learning per la classificazione binaria. Lo schema del sistema di rilevamento POC integrato è mostrato nella Figura 1. Senza operatori qualificati e strumenti ingombranti, l'RPA-CRISPR/Cas12a-SPM con classificazione assistita dall'intelligenza artificiale (AI) mostra un grande potenziale per il rilevamento del virus POC DNA.
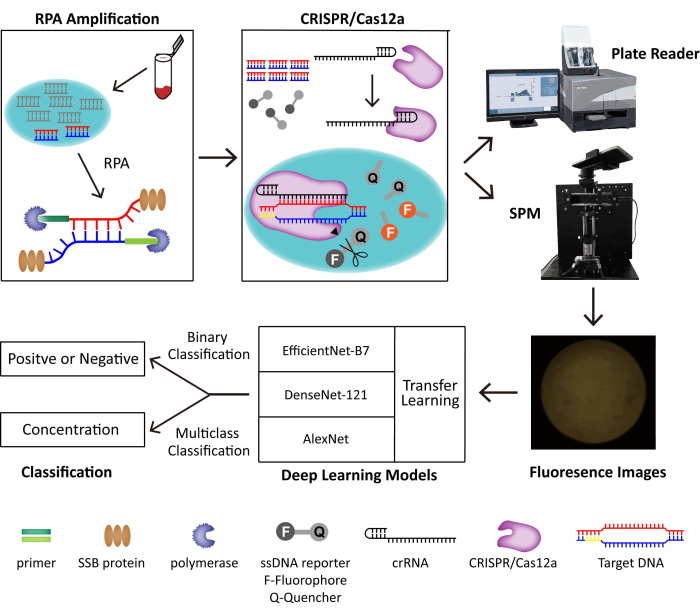
Figura 1: Lo schema del sistema di rilevamento RPA-CRISPR/Cas12-SPM insieme alla classificazione AI per le immagini raccolte. Gli acidi nucleici dei campioni di origine animale vengono rilasciati da PINDBK. Il DNA bersaglio del virus viene amplificato e riconosciuto in modo specifico dal sistema RPA-CRISPR/Cas12a. CRISPR/Cas12a si lega al crRNA e il complesso Cas12a-crRNA si lega al DNA bersaglio, che innesca la scissione collaterale di CRISPR/Cas12a sulle sonde reporter ssDNA. Il fluoroforo sul reporter viene rilasciato e la fluorescenza viene rilevata da un lettore di piastre commercializzato o dall'SPM che costruiamo. Per classificare le immagini a fluorescenza vengono utilizzati tre diversi modelli di deep learning, tra cui AlexNet, DenseNet-121 ed EfficientNet-B7 con transfer learning. Questa figura è riutilizzata con il permesso di Lei et al.35. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Trattamento dei campioni
- Prendiamo il Frog Virus 3 (FV3, generi Ranavirus, famiglia Iridoviridae), un virus a DNA a doppio filamento. Selezionare il gene del capside maggiore (mcp) come bersaglio per la rilevazione di FV3 poiché è altamente conservato e solitamente considerato come bersaglio per la rilevazione del ranavirus. La sequenza di destinazione selezionata è mostrata nella Tabella 1.
NOTA: Frog Virus 3 è preso come esempio in questo protocollo. - Per la preparazione dei frammenti di DNA bersaglio, utilizzare frammenti di DNA del gene mcp di FV3 e del virus della necrosi renale e della milza infettiva (ISKNV, un altro Ranavirus).
NOTA: In questo studio, i frammenti di DNA bersaglio sono stati ottenuti commercialmente da una società elencata nella Tabella dei materiali. Sono considerati l'obiettivo e il controllo del rilevamento successivo.
| Nome | Sequenza | ||
| Centralina multifunzione FV3 | NTS: 5' ... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcgg... 3' (240 pb) | ||
| TS: 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcccgaaagc... 3' (240 pb) | |||
| ISKNV MCP | NTS: 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3' (231 pb) | ||
| TS: 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3' (231 pb) | |||
Tabella 1: sequenza target selezionata in questo metodo.
2. Reazione RPA
- Progettare e sintetizzare le coppie di primer RPA per la sequenza target. Le sequenze di coppie di primer RPA sono descritte nella Tabella 2.
- Preparare il tampone di reazione RPA 5x (Tabella 3) e MgCl2 (100 mM).
- Mescolare i quattro enzimi RPA chiave (UvsX, UvsY, GP32, proteina Bsu) nel tampone di reazione RPA 1x, insieme ai primer progettati in anticipo, come dettagliato nella Tabella 4.
- Agitare accuratamente la miscela.
- Aggiungere 1 μL del bersaglio ottenuto dalla fase 1 per ogni reazione RPA e mescolarlo accuratamente a vortice di nuovo.
- Aggiungere 7 μL di MgCl2 (100 mM) per avviare la reazione. Il volume finale di ciascuna reazione RPA è di 50 μL.
- Eseguire il test a 37 °C per 30 min.
- Il prodotto RPA può essere conservato a 4 °C per alcuni giorni. Man mano che i prodotti RPA si degradano, utilizzarli per ulteriori rilevamenti il prima possibile per ottenere risultati di diagnosi migliori.
- [Facoltativo] Condurre l'elettroforesi su gel di DNA.
- Estrarre 5 μL di prodotti RPA, aggiungere un volume appropriato di tampone di caricamento del DNA 6x ed eseguire l'elettroforesi su gel di DNA nel tampone EDTA (TAE) di acetato di tris.
- Eseguire l'elettroforesi a 120 V per circa 20 minuti fino a quando le bande del tampone di caricamento raggiungono il fondo del gel.
- Determinare se la sequenza target viene amplificata con successo dal campione confrontando la dimensione delle bande del campione con le bande del marcatore.
| Nome | Sequenza |
| RPA primer F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| RPA primer R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Tabella 2: Primer RPA utilizzati in questo metodo.
| Componente | Concentrazione originale | Addizione |
| PESO 20.000 | - | 114 mg |
| ATP | 100 mM | 125 μL |
| dNTP | 25 mM | 48 μl |
| Tris-HCl | 1 M | 125 μL |
| DTT | 1 M | 125 μL |
| Creatinfosfato | 1 M | 250 μl |
| Creatinchinasi | 10 μg/μL | 50 μl |
| ddH2O | - | 277 μl |
| Volume totale | - | 1 ml |
Tabella 3: La composizione del tampone di reazione RPA 5x (pH 7,5).
| Componente | Concentrazione originale | Addizione |
| 5x tampone di reazione RPA | - | 10 μl |
| Proteina UvsX | 5 mg/ml | 2,6 μl |
| Proteina UvsY | 5 mg/ml | 0,9 μl |
| Proteina GP32 | 5 mg/ml | 2,54 μL |
| Proteina Bsu | 5 mg/ml | 0,88 μl |
| Primer in avanti | 100 μM | 0,25 μl |
| primer invertito | 100 μM | 0,25 μl |
| ddH2O | - | 24,58 μL |
| Bersaglio | - | 1 μl |
| *MgCl2 | 100 mM | 7 μl |
| Volume totale | 50 μl | |
| *MgCl2 deve essere aggiunto per ultimo per avviare la reazione RPA. | ||
Tabella 4: La composizione della reazione RPA.
3. Rilevamento CRISPR/Cas12a senza SPM
- Utilizzare i crRNA (Table of Materials) della sequenza target e la sonda reporter ssDNA che collega un fluoroforo e un quencher per CRISPR/Cas12a. Qui, la carbossi-tetrametilrodamina (TAMRA) è legata come fluoroforo alle estremità 5' delle sonde reporter ssDNA e Black Hole Quencher-2 (BHQ2) come quencher alle estremità 3'. La sequenza dettagliata del crRNA e del reporter ssDNA è descritta nella Tabella 5.
- Preparare la proteina del batterio delle Lachnospiraceae Cas12a (LbCas12a) con 10 tamponi di reazione CRISPR/Cas12a.
- Sciogliere 1 μL di prodotto di reazione RPA dalla sezione 2 in 1 tampone di reazione CRISPR/Cas12a con complessi LbCas12a-crRNA e sonda reporter ssDNA da 500 nM in un volume di reazione di 100 μL.
- Dopo aver mescolato LbCas12a e crRNA, lasciare riposare la miscela per almeno 5 minuti per formare un complesso funzionale. Dopo l'incubazione, aggiungere altri componenti alla miscela di reazione ed eseguire l'intera reazione a 37 °C. Il volume finale di ciascuna reazione CRISPR/Cas12a è di 100 μl. Le concentrazioni dettagliate di ciascun componente in ciascuna reazione CRISPR/Cas12a sono descritte nella Tabella 6.
- Eseguire la reazione di rilevamento CRISPR/Cas12a da 100 μl a 37 °C per 30 min.
- Esaminare costantemente i segnali di fluorescenza da un lettore di micropiastre a una lunghezza d'onda di eccitazione di 535 nm e una lunghezza d'onda di emissione di 595 nm con un guadagno di 60.
NOTA: Il rilevamento di diverse lunghezze d'onda della luce di eccitazione ed emissione dipende dalla scelta del fluoroforo e del quencher nelle sonde reporter ssDNA precedentemente progettate. - Per questi dati raccolti, dividere il valore di controllo per la misurazione dei campioni positivi per normalizzare tutti i dati, quindi integrarli per un'analisi t-test a due campioni.
| Nome | Sequenza | |
| LbCas12a crRNA per FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTCGGTCGGGGCGT | |
| Reporter ssDNA | /5TAMRA/TTATT/3BHQ2 | |
Tabella 5: Sequenze di crRNA CRISPR/Cas12a e reporter ssDNA utilizzate in questo metodo.
| Componente | Concentrazione originale | Addizione |
| NEBuffer r2.1 | - | 10 μl |
| Lba Cas12a (CPF1) | 10 μM | 0,5 μl |
| crRNA | 10 μM | 0,625 μl |
| Attendere almeno 5 minuti per consentire al complesso LbCas12a/crRNA di combinarsi. | ||
| Reporter del DNA | 100 μM | 0,5 μl |
| ddH2O | - | 87,375 μL |
| Bersaglio | - | 1 μl |
| Volume totale | - | 100 μl |
Tabella 6: La composizione della reazione CRISPR/Cas12a.
4. Configurazione SPM
- Per il percorso di eccitazione, impostare un raggio laser in modo che passi attraverso i filtri a densità neutra (ND) per attenuare l'intensità del laser.
- Generare un fascio collimato da una lente asferica (di seguito denominata L1) e rifletterlo dallo specchio dicroico (DM).
- Dirigere la luce sul vetrino dove è posizionato il campione attraverso l'obiettivo (20x) per illuminare ed eccitare la fluorescenza del campione. Il tavolino del campione consente una regolazione precisa del piano focale, dirigendo il fascio verso il piano focale posteriore dell'obiettivo. I passaggi sopra menzionati formano il percorso di eccitazione dello strumento SPM.
- Per il percorso di emissione, posizionare una lente esterna (di seguito denominata L2) per formare un'immagine intermedia sull'altro lato dell'obiettivo. L'obiettivo illumina contemporaneamente il campione e raccoglie il segnale di emissione.
- Registrare il segnale di fluorescenza dal campione utilizzando lo smartphone posto alla fine del percorso di emissione. Utilizzare una staffa stabile per evitare scosse.
- Imposta un filtro passa-banda tra L1 e la fotocamera dello smartphone per filtrare la luce eccitata consentendo solo alla luce emessa dal campione di raggiungere la fotocamera, il che può ottimizzare il rilevamento.
- Immobilizzare la configurazione SPM su una breadboard per la distribuzione portatile.
NOTA: Lo schema e l'aspetto fisico del dispositivo SPM per il rilevamento della fluorescenza basato sulla reazione RPA-CRISPR/Cas12a sono mostrati nella Figura 2. Il rilevamento CRISPR/Cas12a avviene sul vetrino pretrattato descritto nella fase successiva.
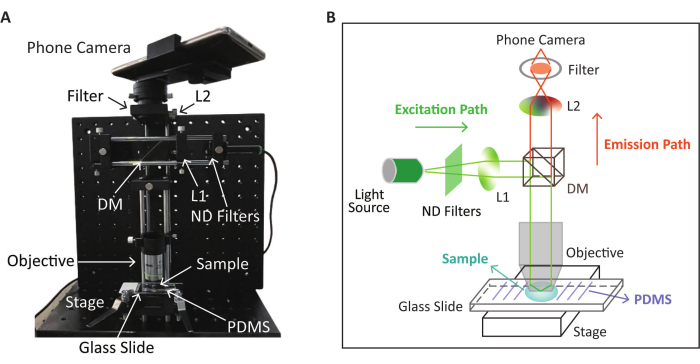
Figura 2: Schema e aspetto fisico del dispositivo SPM utilizzato per la rilevazione della fluorescenza. (A) L'aspetto fisico del dispositivo SPM per la raccolta di immagini a fluorescenza dopo la reazione RPA-CRISPR/Cas12a. (B) Schema del dispositivo SPM per la rilevazione della fluorescenza basato sulla reazione RPA-CRISPR/Cas12a. Questa figura è stata modificata (posizione e colore dell'immagine regolati) con il permesso di Lei et al.35. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Trattamento del vetrino per il rilevamento con SPM
- Preparare il polidimetilsilossano (PDMS) mescolando la base e l'agente indurente in rapporto 10:1, quindi cuocere su una piastra a 80 °C per 2 ore.
- Trattare sia il PDMS che il vetrino (Lunghezza: 75 mm; Altezza: 50 mm) con trattamento al plasma ad ossigeno per 120 s, quindi premerli insieme.
- Cuocere il bicchiere/PDMS a 95 °C per 2 h; è sigillato in modo permanente dal legame Si-O-Si. Il PDMS ha un'elevata trasparenza e nessuna autofluorescenza, il che favorisce il rilevamento SPM. Il trattamento del vetrino e del PMDS viene eseguito secondo He et al.32
6. Rilevamento CRISPR/Cas12a con SPM
- Utilizzare i crRNA (Table of Materials) della sequenza target e la sonda reporter ssDNA che collega un fluoroforo e un quencher per CRISPR/Cas12a. In questo metodo, la carbossi-tetrametilrodamina (TAMRA) è legata come fluoroforo alle estremità 5' delle sonde reporter ssDNA e Black Hole Quencher-2 (BHQ2) come quencher alle estremità 3'. La sequenza dettagliata del crRNA e del reporter ssDNA è descritta nella Tabella 5.
- Preparare la proteina del batterio delle Lachnospiraceae Cas12a (LbCas12a) con 10x tampone di reazione CRISPR/Cas12a.
- Sciogliere 1 μL di prodotto di reazione RPA dalla sezione 2 in 1 tampone di reazione CRISPR/Cas12a con complessi LbCas12a-crRNA e sonda reporter ssDNA da 500 nM in un volume di reazione di 100 μL.
- Dopo aver mescolato LbCas12a e crRNA, lasciare riposare la miscela per almeno 5 minuti per formare un complesso funzionale. Dopo l'incubazione, aggiungere altri componenti alla miscela di reazione e completare la reazione a 37 °C. Il volume finale di ciascuna reazione CRISPR/Cas12a è di 100 μl. Le concentrazioni dettagliate di ciascun componente in ciascuna reazione CRISPR/Cas12a sono descritte nella Tabella 6.
- Eseguire la reazione di rilevamento CRISPR/Cas12a da 100 μl sul vetrino pretrattato e coprirlo con un vetrino coprioggetti. Incubare il vetrino con reazione a RT per 10 min.
- Misurare i segnali di fluorescenza tramite l'SPM. Posizionare il vetrino con la reazione di rilevamento sul tavolino dell'SPM, mantenere una distanza adeguata, regolare la lunghezza focale e la chiarezza, quindi cercare il campo visivo della reazione e metterlo a fuoco per catturare un'immagine.
NOTA: È necessario ottenere prima una curva standard in modo che i dati dei campioni possano essere scalati all'intervallo di concentrazione approssimativo. Vengono utilizzate varie concentrazioni di bersagli purificati, tra cui 10 nM, 1 nM, 100 pM e 10 pM, un altro virus come controllo negativo (prima dell'RPA).
7. Aumento di set di dati e dati
- Raccogliere le immagini di fluorescenza dal saggio di rilevamento nella sezione 6 come set di dati. Ripetere almeno tre rilevamenti paralleli per ogni campione per garantire il parallelismo dei dati.
- È possibile approvare alcuni modi appropriati per ottenere un parallelismo più elevato. Ad esempio, quando raccogli le immagini a fluorescenza da ciascun campione, metti a fuoco manualmente e cerca un campo relativamente più luminoso per scattare le immagini. Allo stesso tempo, fotografa ogni campione per ottenere il segnale di fluorescenza in più di cinque posizioni diverse.
- Misurare il valore medio di grigio di ciascuna immagine e la deviazione standard del valore medio di grigio in un gruppo di concentrazione mediante ImageJ.
- Impostare un intervallo di intensità [mediana - deviazione standard, mediana + deviazione standard] per la pulizia dei dati.
NOTA: Nelle immagini ottenute nei primi passaggi, potrebbero esserci immagini con grandi differenze, quindi è necessario schermare le immagini. Se le intensità delle immagini sono al di fuori della soglia impostata, devono essere considerate anomale ed escluse. - Etichettare le immagini per il bersaglio purificato a concentrazioni crescenti con 0-6 in ordine crescente, rispettivamente.
- Per migliorare la robustezza del sistema e prevenire l'overfitting, implementa tecniche di aumento delle immagini come il capovolgimento orizzontale, il capovolgimento verticale e il rumore casuale attraverso le funzioni di trasformazione in Python. Ciò consente di introdurre variazioni nel set di dati.
8. Trasferisci l'apprendimento
- Come rete backbone, adotta il modello di deep learning AlexNet33 per la classificazione.
- Per soddisfare il vincolo del modello pre-addestrato nella sezione 7, rimodellare le immagini di input a 224 pixel x 224 pixel x 3 canali (altezza e larghezza di 224 pixel e una profondità di 3 canali per i canali di colore rosso, verde e blu) tramite le funzioni di trasformazione in Python.
NOTA: questo passaggio è una pre-elaborazione comune per dati eterogenei nell'apprendimento del trasferimento, inclusa la trasformazione. - Utilizza una rete backbone pre-addestrata con il set di dati ImageNet per estrarre le feature sfruttando i pesi dei livelli nascosti intermedi che sono stati appresi.
- Nel contesto del lavoro di classificazione della fluorescenza, sostituire lo strato finale completamente connesso della rete neurale, che originariamente includeva 1000 neuroni per l'attività ImageNet, con uno strato completamente connesso con 2 o 7 neuroni.
- Valutare le prestazioni del modello di training di configurazione usando una serie di metriche, tra cui matrice di confusione, accuratezza, precisione, richiamo e punteggio F1 basato su Lawton e Viriri34.
Risultati
Questo metodo si concentra su un sistema di rilevamento veloce, facile da implementare, altamente sensibile e point-of-care (POC) per i virus a DNA. La progettazione di coppie di primer per la reazione RPA e la progettazione di crRNA per la reazione CRISPR/Cas12a sono due delle parti essenziali poiché influenzeranno l'efficienza della reazione RPA-CRISPR/Cas12a e influenzeranno la successiva rilevazione e classificazione.
In questo metodo, FV3 è considerato un esempio d...
Discussione
Con questo metodo, sviluppiamo un sistema di rilevamento del virus a DNA veloce, facile da implementare, altamente sensibile, specifico per la sequenza e POC con l'assistenza dell'intelligenza artificiale. Dopo aver ottenuto i campioni, viene applicata RPA per amplificare la sequenza target, quindi CRISPR/Cas12a può riconoscere il DNA target e rilasciare fluorescenza, che amplia il segnale di rilevamento. La microscopia portatile per smartphone è costruita per acquisire immagini a fluorescenza e i modelli di deep learn...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è supportato dalla National Natural Science Foundation of China 31970752, Science, Technology, Innovation Commission of Shenzhen Municipality JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Finanziamento aperto del laboratorio della baia di Shenzhen, SZBL2020090501004; Fondazione scientifica post-dottorato cinese 2020M680023; e Amministrazione generale delle dogane della Repubblica popolare cinese 2021HK007.
Materiali
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Riferimenti
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon