Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Système de détection de virus à ADN basé sur RPA-CRISPR/Cas12a-SPM et Deep Learning
Dans cet article
Résumé
Nous présentons un protocole qui combine l’amplification de la polymérase recombinase avec un système CRISPR/Cas12a pour la détection de traces de virus à ADN et construisons une microscopie portable pour smartphone avec une classification assistée par l’intelligence artificielle pour la détection des virus à ADN au point de service.
Résumé
Nous présentons un système de détection de virus à ADN rapide, facile à mettre en œuvre, très sensible, spécifique à une séquence et au point de service (POC), qui combine l’amplification par polymérase recombinase (RPA) et le système CRISPR/Cas12a pour la détection de traces de virus à ADN. L’ADN cible est amplifié et reconnu séparément par RPA et CRISPR/Cas12a, ce qui déclenche l’activité de clivage collatéral de Cas12a qui clive un rapporteur d’ADN marqué par fluorophore et généralise la fluorescence. Pour la détection POC, la microscopie portable pour smartphone est conçue pour prendre des images fluorescentes. En outre, des modèles d’apprentissage profond pour la classification binaire d’échantillons positifs ou négatifs, atteignant une grande précision, sont déployés au sein du système. Le virus de la grenouille 3 (FV3, genres Ranavirus, famille des Iridoviridae) a été testé à titre d’exemple pour ce système de détection POC de virus à ADN, et les limites de détection (LoD) peuvent atteindre 10 aM en 40 min. En l’absence d’opérateurs qualifiés et d’instruments encombrants, le RPA-CRISPR/Cas12a-SPM portable et miniature avec classification assistée par l’intelligence artificielle (IA) présente un grand potentiel pour la détection des virus ADN POC et peut aider à prévenir la propagation de ces virus.
Introduction
Ces dernières années, des épidémies de maladies infectieuses causées par différents virus se sont produites fréquemment, notamment l’épidémie de maladie à virus Ebola (MVE) en 20141 et 20182, le syndrome respiratoire du Moyen-Orient (MERS) en 20153, l’épidémie de maladie à virus Zika en 20154, la maladie à coronavirus 2019 (COVID-19) causée par le coronavirus du syndrome respiratoire aigu sévère 2 (SRAS-CoV-2)5 et la variole du singe persistante causée par le virus de la variole du singe (MKPV) en 20226. Ces épidémies soudaines de maladies infectieuses épidémiques causent un grand nombre de décès et entraînent d’énormes pertes économiques et des troubles sociaux. Il est urgent de disposer d’un système de détection rapide et précis pour diagnostiquer rapidement l’infection et empêcher la propagation du virus.
Récemment, les répétitions palindromiques courtes (CRISPR) et les protéines associées à CRISPR (Cas) ont attiré l’attention du monde entier et ont montré des résultats prometteurs dans la détection des acides nucléiques 7,8,9,10,11,12,13,14,15 . La protéine CRISPR/Cas12a, guidée par l’ARN CRISPR (ARNcr), se lie à l’ADN cible et le clive. Cette activité conduit à la libération d’ADN simple brin non spécifique (ADNsb), connu sous le nom de trans-clivage, et peut être utilisée pour améliorer le signal de détection pour la détection des acides nucléiques. Certaines méthodes de détection traditionnelles, telles que la réaction en chaîne par polymérase (PCR), la PCR quantitative en temps réel (qPCR) et le test immuno-enzymatique (ELISA) sont compliquées, longues et coûteuses pour la détection au point de service (POC). Nos travaux précédents ont permis de développer un système de détection automatisé, intégré et rentable du virus de la peste porcine africaine (PPA) basé sur la technologie CRISPR/Cas12a. Dans ce système, nous avons atteint une limite de détection de 1 pM dans un laps de temps de 2 h sans avoir besoin d’amplification. Le système CRISPR/Cas12a et l’amplification par polymérase recombinase (RPA) sont combinés pour améliorer la sensibilité et la spécificité de la détection de l’ADN trace. Comparé à d’autres techniques d’amplification isotherme, le RPA est simple dans sa conception et pratique dans son fonctionnement car il a un temps de réaction plus court sans équipement sophistiqué de contrôle de la température.
Pour la détection des agents pathogènes au point de service, des instruments tels que la microscopie sur smartphone (SPM), le fluorimètre portable ou les bandelettes à flux latéral ont été développés pour la lecture des résultats 16,17,18. SPM capture des images via une caméra et les télécharge sur certaines applications mobiles pour une analyse rapide des données. Une telle microscopie constitue un système d’acquisition de signaux portable, bon marché et miniature avec une sensibilité élevée et a montré des avantages dans la détection d’agents pathogènes tels que H5N1, le virus Zika et le SRAS-CoV-219,20. Par conséquent, nous construisons un SPM portable pour capter les signaux de fluorescence déclenchés par la détection RPA-CRISPR/Cas12a du virus à ADN cible. La sonde rapporteure d’ADNsb reliant un fluorophore et un quencher sera clivée lorsque CRISPR/Cas12a reconnaîtra le virus à ADN cible, et la fluorescence émise par le fluorophore pourra être capturée par SPM.
Par rapport aux logiciels professionnels habituellement utilisés pour obtenir les informations sur les résultats des images de fluorescence de SPM21, certains experts utilisent l’apprentissage automatique et l’apprentissage profond pour quantifier les concentrations d’ADN viral après avoir obtenu des images de fluorescence22, ce qui prend plus de temps. Lorsqu’il s’agit de classifier des images médicales, les réseaux neuronaux conventionnels (CNN) sont souvent utilisés pour apprendre des caractéristiques à partir d’images pixélisées brutes de bout en bout 23,24,25,26. Des modèles populaires d’apprentissage profond basés sur CNN tels que AlexNet, DenseNet-121 et EfficientNet-B7 ont été appliqués avec succès dans ce domaine27,28. Cependant, l’obtention de grands ensembles de données dans des domaines spécifiques peut être difficile, nécessitant un apprentissage par transfert 29,30. Cette approche pré-entraîne un modèle de Deep Learning avec un grand ensemble de données, et le modèle pré-entraîné est utilisé comme point de départ pour une nouvelle tâche avec un petit jeu de données. Cette technique peut réduire le besoin de grands ensembles de données, lutter contre le surajustement et réduire le temps d’entraînement31. Ici, nous utilisons des modèles d’apprentissage profond avec apprentissage par transfert pour la classification binaire des images de fluorescence des échantillons positifs et négatifs.
Dans cette méthode, nous combinons la RPA et le système CRISPR/Cas12a pour la détection de traces de virus à ADN. L’ADN cible est amplifié et reconnu séparément par RPA et CRISPR/Cas12a, ce qui déclenche l’activité de clivage collatéral de Cas12a qui clive un rapporteur d’ADN marqué par fluorophore et généralise la fluorescence. Nous construisons un SPM portable pour prendre les images fluorescentes pour la détection POC et développons des modèles d’apprentissage profond pour la classification binaire. Le schéma du système de détection POC construit est illustré à la figure 1. En l’absence d’opérateurs qualifiés et d’instruments encombrants, le RPA-CRISPR/Cas12a-SPM avec classification assistée par l’intelligence artificielle (IA) présente un grand potentiel pour la détection des virus ADN POC.
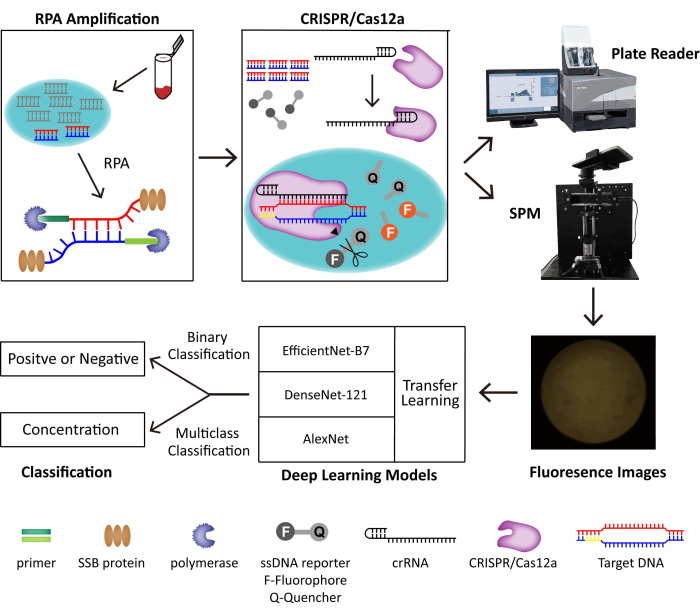
Figure 1 : Le schéma du système de détection RPA-CRISPR/Cas12-SPM ainsi que la classification par IA des images collectées. Les acides nucléiques des échantillons d’origine animale sont libérés par le PINDBK. L’ADN cible du virus est amplifié et reconnu spécifiquement par le système RPA-CRISPR/Cas12a. Les liaisons CRISPR/Cas12a avec l’ARNcr et le complexe Cas12a-ARNcr se lie à l’ADN cible, ce qui déclenche le clivage collatéral de CRISPR/Cas12a sur les sondes rapporteures de l’ADNss. Le fluorophore sur le rapporteur est libéré et la fluorescence est détectée par un lecteur de plaques commercialisé ou le SPM que nous construisons. Trois modèles d’apprentissage profond différents, dont AlexNet, DenseNet-121 et EfficientNet-B7 avec apprentissage par transfert, sont utilisés pour classer les images de fluorescence. Cette figure est réutilisée avec la permission de Lei et al.35. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Traitement des échantillons
- Prenez le virus de la grenouille 3 (FV3, genres Ranavirus, famille des Iridoviridae), un virus à ADN double brin. Sélectionnez le gène de la capside majeure (mcp) comme cible pour la détection du FV3, car il est hautement conservé et généralement considéré comme la cible de la détection du ranavirus. La séquence cible sélectionnée est indiquée dans le Tableau 1.
REMARQUE : Frog Virus 3 est pris comme exemple dans ce protocole. - Pour la préparation des fragments d’ADN cibles, utilisez des fragments d’ADN du gène mcp du FV3 et du virus de la nécrose infectieuse de la rate et du rein (ISKNV, un autre ranavirus).
REMARQUE : Dans cette étude, les fragments d’ADN cibles ont été obtenus commercialement auprès d’une entreprise figurant dans la Table des matières. Ils sont considérés comme la cible et le contrôle de la détection ultérieure.
| Nom | Séquence | ||
| FV3 MCP | SNRC : 5' ... gtaacccggctttcGGGCAGCAGTTTCGGTCGGCGTtcccaggtcg... 3 pi (240 pb) | ||
| TS : 5' ... ccgacctgggaACGCCGACCGAAACTGCTGCCCtgctgcgcgaaagc... 3 pi (240 pb) | |||
| ISKNV MCP | SNRC : 5' ... ggccatgccaatttTGGGCAGGAGTTTAGTGTGACGgtggcgaggg... 3 pi (231 pb) | ||
| TS : 5' ... ccctcgccaccgtcACACTAAACTCCTGCCCAAAATtggcatggcc... 3 pi (231 pb) | |||
Tableau 1 : Séquence cible sélectionnée dans cette méthode.
2. Réaction RPA
- Concevez et synthétisez les paires d’amorces RPA pour la séquence cible. Les séquences des paires d’amorces RPA sont décrites dans le tableau 2.
- Préparez le tampon de réaction RPA 5x (Tableau 3) et le MgCl2 (100 mM).
- Mélangez les quatre enzymes clés de la RPA (UvsX, UvsY, GP32, protéine Bsu) dans le tampon de réaction 1x RPA, ainsi que les amorces conçues à l’avance, comme détaillé dans le tableau 4.
- Mélangez soigneusement le mélange.
- Ajoutez 1 μL de la cible obtenue à l’étape 1 pour chaque réaction RPA, et mélangez-le soigneusement par vortex à nouveau.
- Ajouter 7 μL de MgCl2 (100 mM) pour amorcer la réaction. Le volume final de chaque réaction RPA est de 50 μL.
- Effectuez le test à 37 °C pendant 30 min.
- Le produit RPA peut être conservé à 4 °C pendant quelques jours. Au fur et à mesure que les produits RPA se dégradent, utilisez-les pour une détection plus poussée dès que possible afin d’obtenir de meilleurs résultats de diagnostic.
- [Facultatif] Effectuer une électrophorèse sur gel d’ADN.
- Prélever 5 μL de produits RPA, ajouter un volume approprié de tampon de charge d’ADN 6x et effectuer une électrophorèse sur gel d’ADN dans le tampon Tris-acétate EDTA (TAE).
- Faites fonctionner l’électrophorèse sous 120 V pendant environ 20 minutes jusqu’à ce que les bandes de tampon de charge atteignent le fond du gel.
- Déterminez si la séquence cible est amplifiée avec succès à partir de l’échantillon en comparant la taille des bandes de l’échantillon avec les bandes du marqueur.
| Nom | Séquence |
| Apprêt RPA F | ATGTCTTCTGTAACTGGTTCAGGTATCACA |
| Apprêt RPA R | GGCGTTGAGGATGTAATCCCCCGACCTGGG |
Tableau 2 : Amorces RPA utilisées dans cette méthode.
| Composant | Concentration d’origine | Addition |
| PEG 20 000 | - | 114 mg |
| ATP | 100 millions de mètres | 125 μL |
| dNTP | 25 millions d’euros | 48 μL |
| Tris-HCl | 1 M | 125 μL |
| La TNT | 1 M | 125 μL |
| Phosphocréatine | 1 M | 250 μL |
| Créatine kinase | 10 μg/μL | 50 μL |
| jjH2O | - | 277 μL |
| Total Volume | - | 1 mL |
Tableau 3 : La composition du tampon de réaction 5x RPA (pH 7,5).
| Composant | Concentration d’origine | Addition |
| 5x tampon de réaction RPA | - | 10 μL |
| Protéine UvsX | 5 mg/mL | 2,6 μL |
| Protéine UvsY | 5 mg/mL | 0,9 μL |
| Protéine GP32 | 5 mg/mL | 2,54 μL |
| Protéine Bsu | 5 mg/mL | 0,88 μL |
| apprêt avant | 100 μM | 0,25 μL |
| apprêt inversé | 100 μM | 0,25 μL |
| jjH2O | - | 24,58 μL |
| Cible | - | 1 μL |
| *MgCl2 | 100 millions de mètres | 7 μL |
| Total Volume | 50 μL | |
| *MgCl2 doit être ajouté en dernier lieu pour initier la réaction RPA. | ||
Tableau 4 : Composition de la réaction RPA.
3. Détection CRISPR/Cas12a sans SPM
- Utilisez les ARNc (Table des Matériaux) de la séquence cible et la sonde rapporteure d’ADNsb reliant un fluorophore et un quencher pour CRISPR/Cas12a. Ici, la carboxy tétraméthylrhodamine (TAMRA) est liée en tant que fluorophore aux extrémités 5' des sondes rapporteures d’ADNss et le trou noir Quencher-2 (BHQ2) en tant que quencher aux extrémités 3'. La séquence détaillée de l’ARNcr et du rapporteur d’ADNsb est décrite dans le tableau 5.
- Préparez la protéine Cas12a (LbCas12a) de la bactérie Lachnospiraceae avec 10 tampons de réaction CRISPR/Cas12a.
- Dissoudre 1 μL de produit de réaction RPA de la section 2 dans 1 tampon de réaction CRISPR/Cas12a avec des complexes LbCas12a-crRNA et une sonde rapporteure d’ADNsb de 500 nM dans un volume de réaction de 100 μL.
- Après avoir mélangé LbCas12a et l’ARNcr, laissez reposer le mélange pendant au moins 5 minutes pour former un complexe fonctionnel. Après l’incubation, ajoutez d’autres composants au mélange réactionnel et effectuez l’ensemble de la réaction à 37 °C. Le volume final de chaque réaction CRISPR/Cas12a est de 100 μL. Les concentrations détaillées de chaque composant dans chaque réaction CRISPR/Cas12a sont décrites dans le tableau 6.
- Effectuez la réaction de détection CRISPR/Cas12a de 100 μL à 37 °C pendant 30 min.
- Examiner en permanence les signaux de fluorescence par un lecteur de microplaques à une longueur d’onde d’excitation de 535 nm et une longueur d’onde d’émission de 595 nm avec un gain de 60.
REMARQUE : La détection de différentes longueurs d’onde d’excitation et d’émission de lumière dépend du choix du fluorophore et de l’extincteur dans les sondes rapporteures d’ADNsb précédemment conçues. - Pour ces données collectées, divisez la valeur de contrôle par la mesure des échantillons positifs pour normaliser toutes les données, puis intégrez-les pour une analyse de test t à deux échantillons.
| Nom | Séquence | |
| ARNc LbCas12a pour FV3 | uaauuucuacuaaguguagauGGGCAGCAGTTTTTTCGGTCGGCGT | |
| Rapporteur d’ADNss | /5TAMRA/TTATT/3BHQ2 | |
Tableau 5 : Séquences de l’ARNcr CRISPR/Cas12a et du rapporteur d’ADNsb utilisés dans cette méthode.
| Composant | Concentration d’origine | Addition |
| NEBuffer r2.1 | - | 10 μL |
| LBA Cas12a (Cpf1) | 10 μM | 0,5 μL |
| ARNcr | 10 μM | 0,625 μL |
| Attendez au moins 5 min pour laisser le complexe LbCas12a/ARNcr se combiner. | ||
| Rapporteure ADN | 100 μM | 0,5 μL |
| jjH2O | - | 87,375 μL |
| Cible | - | 1 μL |
| Total Volume | - | 100 μL |
Tableau 6 : La composition de la réaction CRISPR/Cas12a.
4. Configuration SPM
- Pour le chemin d’excitation, réglez un faisceau laser pour qu’il passe à travers les filtres de densité neutre (ND) afin d’atténuer l’intensité du laser.
- Générer un faisceau collimaté à partir d’une lentille asphérique (ci-après dénommée L1) et le réfléchir par le miroir dichroïque (DM).
- Dirigez la lumière sur la lame de verre où l’échantillon est placé à travers l’objectif (20x) pour éclairer et exciter la fluorescence de l’échantillon. La platine d’échantillonnage permet un réglage précis du plan focal, en dirigeant le faisceau vers le plan focal arrière de l’objectif. Les étapes mentionnées ci-dessus forment le chemin d’excitation de l’instrument SPM.
- Pour le trajet d’émission, positionnez une lentille externe (ci-après dénommée L2) de manière à former une image intermédiaire de l’autre côté de l’objectif. L’objectif éclaire simultanément l’échantillon et recueille le signal d’émission.
- Enregistrez le signal de fluorescence de l’échantillon à l’aide du smartphone placé à la fin du trajet d’émission. Utilisez un support stable pour éviter les secousses.
- Réglez un filtre passe-bande entre L1 et l’appareil photo du smartphone pour filtrer la lumière excitée tout en ne laissant que la lumière émise de l’échantillon atteindre l’appareil photo, ce qui peut optimiser la détection.
- Immobilisez la configuration SPM sur une carte d’essai pour un déploiement portable.
REMARQUE : Le schéma et l’aspect physique du dispositif SPM pour la détection de fluorescence basé sur la réaction RPA-CRISPR/Cas12a sont illustrés à la figure 2. La détection CRISPR/Cas12a a lieu sur la lame de verre prétraitée décrite à l’étape suivante.
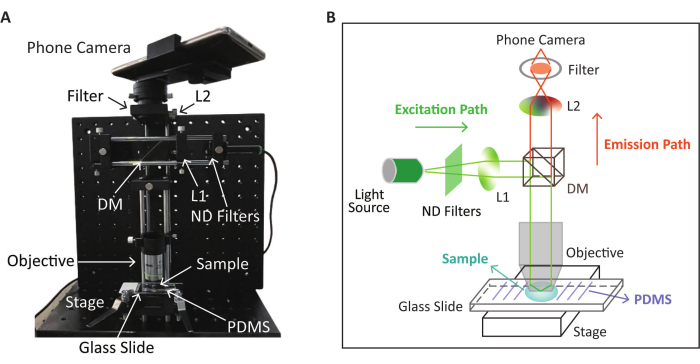
Figure 2 : Schéma et aspect physique du dispositif SPM utilisé pour la détection de fluorescence. (A) Aspect physique du dispositif SPM pour la collecte d’images de fluorescence après la réaction RPA-CRISPR/Cas12a. (B) Schéma du dispositif SPM pour la détection de fluorescence basé sur la réaction RPA-CRISPR/Cas12a. Cette figure est modifiée (position et couleur de l’image ajustées) avec la permission de Lei et al.35. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Traitement de la lame de verre pour la détection avec SPM
- Préparez le polydiméthylsiloxane (PDMS) en mélangeant la base et l’agent de durcissement dans un rapport de 10:1, puis faites cuire sur une plaque chauffante à 80 °C pendant 2 h.
- Traitez à la fois le PDMS et la lame de verre (Longueur : 75 mm ; Hauteur : 50 mm) avec un traitement au plasma d’oxygène pendant 120 s, puis pressez-les ensemble.
- Cuire le verre/PDMS à 95 °C pendant 2 h ; il est scellé de manière permanente par la liaison Si-O-Si. Le PDMS a une transparence élevée et aucune auto-fluorescence, ce qui est propice à la détection SPM. Le traitement de la lame de verre et du PMDS est effectué conformément à He et al.32
6. Détection CRISPR/Cas12a avec SPM
- Utilisez les ARNc (Table des Matériaux) de la séquence cible et la sonde rapporteure d’ADNsb reliant un fluorophore et un quencher pour CRISPR/Cas12a. Dans cette méthode, la carboxy tétraméthylrhodamine (TAMRA) est liée en tant que fluorophore aux extrémités 5' des sondes rapporteures d’ADNss et le trou noir Quencher-2 (BHQ2) en tant que quencher aux extrémités 3'. La séquence détaillée de l’ARNcr et du rapporteur d’ADNsb est décrite dans le tableau 5.
- Préparez la protéine Cas12a (LbCas12a) de la bactérie Lachnospiraceae avec 10 tampons de réaction CRISPR/Cas12a.
- Dissoudre 1 μL de produit de réaction RPA de la section 2 dans 1 tampon de réaction CRISPR/Cas12a avec des complexes LbCas12a-crRNA et une sonde rapporteure d’ADNsb de 500 nM dans un volume de réaction de 100 μL.
- Après avoir mélangé LbCas12a et l’ARNcr, laissez reposer le mélange pendant au moins 5 minutes pour former un complexe fonctionnel. Après l’incubation, ajoutez d’autres composants au mélange réactionnel et terminez la réaction à 37 °C. Le volume final de chaque réaction CRISPR/Cas12a est de 100 μL. Les concentrations détaillées de chaque composant dans chaque réaction CRISPR/Cas12a sont décrites dans le tableau 6.
- Effectuez la réaction de détection CRISPR/Cas12a de 100 μL sur la lame de verre prétraitée et couvrez-la d’une lamelle. Incuber la lame de verre avec réaction à RT pendant 10 min.
- Mesurez les signaux de fluorescence à l’aide du SPM. Placez la lame de verre avec la réaction de détection sur la platine du SPM, maintenez une distance appropriée, ajustez la distance focale et la clarté, puis recherchez le champ de vision de la réaction et faites-le correspondre pour capturer une image.
REMARQUE : Il faut d’abord obtenir une courbe standard afin que les données des échantillons puissent être mises à l’échelle à la plage de concentration approximative. Diverses concentrations de cibles purifiées sont utilisées, notamment 10 nM, 1 nM, 100 pM et 10 pM, un autre virus comme contrôle négatif (avant RPA).
7. Ensemble de données et augmentation des données
- Recueillir les images de fluorescence du test de détection de la section 6 en tant qu’ensembles de données. Répétez au moins trois détections parallèles pour chaque échantillon afin de garantir le parallélisme des données.
- Certaines méthodes appropriées pour obtenir un parallélisme plus élevé peuvent être approuvées. Par exemple, lorsque vous collectez les images de fluorescence de chaque échantillon, faites la mise au point manuellement et recherchez un champ relativement plus clair pour prendre des images. En même temps, photographiez chaque échantillon pour obtenir le signal de fluorescence dans plus de cinq endroits différents.
- Mesurez la valeur moyenne de gris de chaque image et l’écart-type de la valeur moyenne de gris dans un groupe de concentration à l’aide d’ImageJ.
- Définissez une plage d’intensités [médiane - écart-type, médiane + écart-type] pour le nettoyage des données.
REMARQUE : Dans les images obtenues dans les premières étapes, il peut y avoir des images avec de grandes différences, il est donc nécessaire de filtrer les images. Si l’intensité des images est en dehors du seuil défini, elles doivent être considérées comme des valeurs aberrantes et exclues. - Étiquetez les images de la cible purifiée à des concentrations croissantes de 0 à 6 dans l’ordre croissant, respectivement.
- Pour améliorer la robustesse du système et éviter le sur-ajustement, implémentez des techniques d’augmentation d’image telles que le retournement horizontal, le retournement vertical et le bruit aléatoire via des fonctions de transformation en Python. Cela permet d’introduire des variations dans l’ensemble de données.
8. Apprentissage par transfert
- En tant que réseau de base, adoptez le modèle d’apprentissage profond AlexNet33 pour la classification.
- Pour satisfaire la contrainte du modèle pré-entraîné de la section 7, remodelez les images d’entrée à 224 pixels x 224 pixels x 3 canaux (hauteur et largeur de 224 pixels et profondeur de 3 canaux pour les canaux de couleur rouge, vert et bleu) à l’aide de fonctions de transformation dans Python.
REMARQUE : Cette étape est un prétraitement courant pour les données hétérogènes dans l’apprentissage par transfert, y compris la transformation. - Utilisez un réseau dorsal pré-entraîné avec l’ensemble de données ImageNet pour extraire des entités tout en exploitant les poids des couches cachées intermédiaires qui ont été apprises.
- Dans le contexte de la classification de fluorescence, remplacez la couche finale entièrement connectée du réseau neuronal, qui comprenait à l’origine 1000 neurones pour la tâche ImageNet, par une couche entièrement connectée avec 2 ou 7 neurones.
- Évaluez les performances du modèle d’entraînement de configuration à l’aide d’une série de mesures, notamment la matrice de confusion, l’exactitude, la précision, le rappel et le score F1 basé sur Lawton et Viriri34.
Résultats
Cette méthode se concentre sur un système de détection rapide, facile à mettre en œuvre, très sensible et au point de service (POC) pour les virus à ADN. La conception des paires d’amorces pour la réaction RPA et la conception de l’ARNcr pour la réaction CRISPR/Cas12a sont deux des éléments essentiels car elles affecteront l’efficacité de la réaction RPA-CRISPR/Cas12a et influenceront la détection et la classification ultérieures.
Dans cette méthode,...
Discussion
Dans cette méthode, nous développons un système de détection de virus à ADN rapide, facile à mettre en œuvre, très sensible, spécifique à une séquence et POC avec l’aide de l’IA. Après avoir obtenu des échantillons, la RPA est appliquée pour amplifier la séquence cible, puis CRISPR/Cas12a peut reconnaître l’ADN cible et libérer de la fluorescence, ce qui élargit le signal de détection. La microscopie portable pour smartphone est conçue pour prendre des images de fluorescence, et les modèles d?...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail est soutenu par la Fondation nationale des sciences naturelles de Chine 31970752, la Commission de la science, de la technologie et de l’innovation de la municipalité de Shenzhen JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708 WDZC20200820173710001 ; Laboratoire de la baie de Shenzhen, SZBL2020090501004 ; Fondation chinoise des sciences postdoctorales 2020M680023 ; et Administration générale des douanes de la République populaire de Chine 2021HK007.
matériels
| Name | Company | Catalog Number | Comments |
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25x36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Références
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak - nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon