Method Article
一种可验证的液滴数字聚合酶链反应测定法,用于检测泪液生物脱落研究中的腺相关病毒载体
摘要
在这里,我们提出了一种协议,用于开发和验证通过液滴数字聚合酶链反应合规检测人泪液中腺相关病毒载体的良好实验室规范,以支持基因治疗载体的临床开发。
摘要
近年来,使用病毒载体治疗遗传疾病的使用大幅增加,迄今为止已注册了2,000多项研究。腺相关病毒(AAV)载体在治疗眼相关疾病方面特别成功,例如voreticigene neparvovec-rzyl的批准。为了将新疗法推向市场,监管机构通常会要求进行合格或经过验证的生物脱落研究,以评估载体向环境中的释放。然而,美国食品和药物管理局尚未发布开发基于分子的测定以支持此类脱落研究的官方指南,让开发人员自己确定最佳实践。该协议的目的是提供一种可验证的方案,用于通过液滴数字聚合酶链反应(ddPCR)检测人泪液中的AAV载体,以支持临床生物脱落研究。本手稿讨论了当前行业分子检测验证的方法,并证明该方法超出了白皮书中目前提出的靶标检测验收标准。最后,讨论了对任何ddPCR检测性能至关重要的步骤,无论应用如何。
引言
基因治疗的定义各不相同,但通常会诱导细胞基因组的特定DNA序列的有意且通常预期的永久交替,以修改或操纵基因的表达或改变活细胞的生物学特性以用于临床目的1,2。由于其转导效率,病毒载体越来越多地被用作基因治疗的载体,一份报告表明,目前超过70%的基因治疗临床试验使用病毒载体3。人们对用于基因治疗的病毒载体的兴趣一直在稳步增长。美国基因和细胞治疗学会发布的 2022 年第四季度基因、细胞和 RNA 治疗领域季度数据报告报告称,2022 年,从临床前到预注册的基因、细胞和 RNA 治疗管道增长了 7%,使在研疗法总数达到 3,726 种,其中 2,053 种 (55%) 是基因疗法4.美国食品和药物管理局(US FDA)目前已批准27种细胞和基因疗法用于人类临床,其中5种专门利用病毒载体5。
腺相关病毒(AAV)作为基因治疗的载体引起了人们的特别兴趣。最近的一项荟萃分析显示,在过去二十年中,大约有136项临床试验调查了AAV的使用6。此外,美国FDA批准的五种基因疗法中有三种是基于AAV的。这是由于其高度可编辑的性质,广泛的宿主范围,可以根据使用特定的天然存在或人工工程载体进行调整,对人类的低致病性和毒性,以及通常低免疫原性7,8。AAV也已成功用于在批准的临床环境中治疗眼部疾病。Voretigene neparvovec-rzyl 是一种基于 AAV2 的疗法,于 2017 年获得美国 FDA 批准,并于 2018 年获得欧洲药品管理局 (EMA) 的批准,用于治疗双等位基因 RPE65 突变相关的视网膜营养不良症9。
随着人们对开发基于AAV的疗法的兴趣日益浓厚,需要对检测进行监管指导。任何病毒载体的准确检测和定量是产品开发的发现、制造和临床前/临床测试阶段不可或缺的一部分。美国FDA已经开始发布一些基因治疗指南,包括人类基因治疗研究性新药应用的化学、制造和控制10,基因治疗给药后的长期随访11,复制能力逆转录病毒检测12,以及基因治疗中使用的微生物载体的建议13.EMA还发布了一系列关于基因治疗产品开发的指南,这些指南通常与FDA的建议保持一致,尽管确实存在一些差异14。值得注意的是,虽然这些指南没有规定法律上可执行的责任,但除非参考了具体法规,但它们明确了监管机构对该主题的当前想法以及他们对药物备案和监管批准所需检测的期望。
FDA 特别建议开展研究以评估载体从给药部位到靶向眼和非眼组织、眼内液和血液的分布、持久性和清除性15。这些采取生物分布和脱落研究的形式。生物分布研究通过调查产品如何从给药部位传播到患者全身来评估暴露。脱落专门评估产品从患者释放到环境中的情况,并提高载体传播给未经治疗的个体的可能性16。FDA就样本采集频率、样本采集持续时间、采集样本类型和储存条件,对生物分布和脱落研究的设计提出了建议。
此外,FDA建议使用定量聚合酶链反应(qPCR或实时PCR)进行载体基因组的定量检测,因为它易于操作,高通量形式,快速周转时间和检测灵敏度。然而,与现有的小分子和大分子相比,相对缺乏分子方法的设计和性能评估建议。由于产品和检测本身的独特而复杂的设计,许多此类研究的指南难以应用于分子方法,这引发了有关推荐评估的可用平台和适当的检测验证方法的适当性的问题。迄今为止,FDA尚未要求对基于PCR的检测进行正式验证,尽管EMA已施加了这一要求17。鉴于这一空白,不同的团体和车间发布了白皮书和建议,制造商和合同研究组织试图遵循18,19,20,21,22,23,24,25。这些建议中的大多数都是专门针对qPCR检测编写的,并针对新兴平台(例如微滴数字PCR(ddPCR))提出建议或更改,仅在被认为相关的情况下包括在内。最近的建议集中在ddPCR测定的考虑因素上,但主要集中在它们在制造环境中的载体基因组定量中的应用,而不是在生物脱落研究中遇到的复杂生物基质中的应用。
根据临床应用和目标,由于与qPCR相比,ddPCR具有更高的灵敏度和处理基质干扰的能力,因此在支持生物分布和脱落研究方面,ddPCR可能优于qPCR。此外,由于将样品分成大约20,000个液滴,因此无需使用泊松统计的标准曲线即可实现拷贝数的准确定量,从而简化了方法开发和验证。该协议的目标是描述一种标准化方法,用于开发和验证基于ddPCR的方法,用于检测从眼表收集的泪液中的AAV载体,以支持临床生物脱落研究。
研究方案
1. 合成DNA片段的制备
- 设计并订购含有靶标扩增区域的合成DNA片段,用作质控品。
- 确保序列包含从靶基因的正向引物到反向引物的整个扩增子序列,在每个引物结合序列的5'末端延伸四到六个碱基对序列。
- 避免使用大于12个碱基对的腺嘌呤和胸腺嘧啶的均聚物或大于8个碱基对的鸟嘌呤和胞嘧啶碱基对,因为只要均聚物可能会干扰基因片段的合成。
注意:如果扩增子包含此类序列,只要保持引物和探针的退火位点,就可以进行碱基替换。 - 或者,使用典型的克隆策略制备含有扩增子的线性化质粒。
- 在微量离心机中离心含有合成DNA片段的试管~10秒,以确保材料收集在管底部。
- 使用tris-EDTA(TE)缓冲液将合成DNA片段重悬至1.0×1010 拷贝/μL的浓度,或根据目标测定范围酌情重悬。
- 短暂涡旋,然后在50°C孵育20±5分钟。在冰上冷却。
- 准备多个,理想情况下一次性等分试样,并储存在-70至-90°C直至使用。
注意:以这种方式制备的合成DNA片段通常自重悬之日起至少24个月内稳定。 - 如果需要,在用作质量控制之前确定制备的合成DNA储备液的确切浓度,或根据所使用的重悬估计标称浓度。
2. 引物和探针的制备
- 使用典型设计策略26,27设计和订购引物和水解探针,以靶向所需的扩增区域。
- 使用与 ddPCR 系统兼容的 5' 荧光报告染料(例如 FAM)和 3' 淬灭剂(例如爱荷华黑深色淬灭剂)。
注意:存在许多PCR测定设计软件包,可以使用任何软件包。例如,美国国家生物技术信息中心28 的引物-BLAST被广泛使用,因为它具有强大的检测设计选项,并且易于进行生物信息学特异性评估以识别可能的脱靶效应。应该注意的是,引物和探针的制备可能与此处列出的步骤不同,具体取决于它们的供应形式。
- 使用与 ddPCR 系统兼容的 5' 荧光报告染料(例如 FAM)和 3' 淬灭剂(例如爱荷华黑深色淬灭剂)。
- 将含有正向引物、反向引物和探针的试管在微量离心机中离心~10秒,以将材料沉淀到管底部。
- 使用TE缓冲液将引物重悬至20μM。短暂漩涡。
- 使用 TE 缓冲液将探针重悬至 10 μM。短暂漩涡。
- 准备多个,理想情况下一次性等分试样,并在至少-20°C下储存直至使用。
注意:以这种方式制备的引物和探针通常自重悬之日起至少24个月内稳定。
3. 样品稀释缓冲液的制备
- 在室温下解冻PCR缓冲液和剪切的鲑鱼精子DNA。彻底涡旋混合。
- 按照 表1制备样品稀释缓冲液。
- 彻底涡旋。制备后在2-8°C下储存长达1个月。
表1:样品稀释缓冲液的制备。请按此下载此表格。
4. 预混液的制备
- 在室温下解冻用于探针、正向引物、反向引物和探针的ddPCR预混液,并在解冻后加热至少10分钟后使用。在室温下储存直至使用。
注意:这些试剂必须完全置于室温下,以确保有效形成液滴。制备过程中不要将试剂放在冰上。- 使用前在小型离心机中彻底、短暂地涡旋离心。
注意:限制性内切酶通常以甘油形式提供,应在使用前立即从储存中取出。轻轻混合。不要涡旋。
- 使用前在小型离心机中彻底、短暂地涡旋离心。
- 为每个扩增靶标制备PCR预混液。有关建议的PCR预混液组成,请参见 表2 ,并根据需要修改引物和探针的浓度。
- 在加入限制性内切酶之前彻底涡旋并短暂离心。加入限制性内切酶并倒置混合。
注意:在此步骤中,需要 22 μL PCR 反应才能在液滴形成后获得 40 μL PCR 反应的最终体积(由 15 μL PCR 预混液、5.0 μL 模板和 20 μL 液滴生成油组成)。
- 在加入限制性内切酶之前彻底涡旋并短暂离心。加入限制性内切酶并倒置混合。
- 根据板图向每个孔中加入 16.5 μL 预混液。有关验证准确度和精密度运行的示例板图,请参见 图 1 。
- 确保板包含三种独立的质控品(QC)系列制剂、三种独立测试的内源性撕裂等分试样,加标至高低水平且未加标,以及三种独立的无模板对照(NTC)。
- 改变这些孔在板上的布局,其中组1按浓度降低的顺序上样,组2按浓度增加的顺序上样,组3以随机顺序上样,以评估是否存在任何板位置特定的效应。
- 排列样品以填充尽可能多的色谱柱,并用对照缓冲液填充色谱柱内未使用的孔。如果需要,在剩余的孔中包括多个内源性对照批次(例如,更多的眼泪池或从个体收集的眼泪)。
- 用透明胶膜密封板。在模板制备过程中将板保持在室温下。或者,在2-8°C下保持板长达4小时,但在加入模板之前将其恢复到室温至少10分钟。
表2:PCR预混液制备示例。请按此下载此表格。
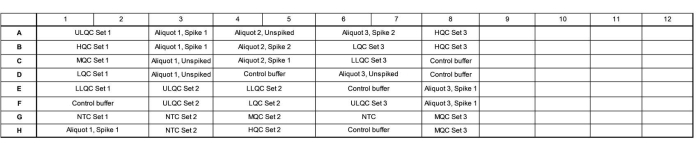
图 1:验证准确性和精度运行的示例板图。缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。请点击此处查看此图的大图。
5. 准备质量控制
- 在室温下解冻合成DNA片段或线性化质粒,并在解冻后加热至少10分钟,然后再使用。将模板置于室温,以确保有效形成液滴。
- 在室温下储存直至使用。使用前在小型离心机中彻底、短暂地涡旋离心。
- 使用样品稀释缓冲液作为稀释剂制备QC稀释液。 表3列出了为验证准确度和精密度运行做准备的推荐浓度示例。
注意:成功完成准确度和精密度运行后,只需在每个板上运行高质量控制 (HQC)、中等质量控制 (MQC) 和低质量控制 (LQC)。对于准确度和精密度运行,至少包括三次独立的QC稀释液,以评估测定内的准确性和精密度。在准确度和精密度运行之后,只需要包括一个稀释系列。 - 制备后,将稀释液储存在室温下,直到添加到板中。
- 如果需要,将稀释液储存在冰上或2-8°C。在随后使用之前,在使用前让稀释液加热至室温至少 10 分钟。在一天结束时丢弃QC。
表3:使用合成双链DNA片段的质量控制(QC)制备示例。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
6. 样品制备
- 从临床试验中收集的泪液样品在室温下解冻直至解冻,并在解冻后加热至少 10 分钟后使用前。
- 在室温下储存直至使用。使用前在微型离心机中彻底短暂地涡旋。
- 使用样品稀释缓冲液作为稀释剂,将泪液以 1:10(或更高)的比例稀释到 0.2 mL PCR 管或 8 孔 PCR 试管中。密封试管。
注意:根据眼泪中目标的预期浓度,可能需要进一步稀释样品或测试每个样品的多次稀释。 - 在95°C的热循环仪中加热样品10分钟,然后在4°C下保持至少5分钟以冷却。使用3°C/s的升温速率。
注意:样品可以停留在4°C的热循环仪中,直到在同一天使用,或者可以在-70至-90°C下冷冻以延长储存时间。此步骤用于使载体衣壳变性,释放基因组。由于QC合成DNA片段或线性化质粒是双链的,因此不应进行此加热步骤。 - 冷却后将样品放回室温(或如果冷冻,则在室温下解冻)并加热至少10分钟。
注意:样品必须完全达到室温,以确保有效形成液滴。
7. 模板添加
- 取回含有预混液的ddPCR板。彻底涡旋每个样品或QC稀释管,并短暂离心以收集材料。
- 根据板图,去除粘合膜并将 5.5 μL QC 或样品添加到 96 孔板的适当孔中。
注意:有关所需卷的说明,请参阅步骤 4.2.1 - 向NTC孔中加入5.5 μL样品稀释缓冲液。
- 液滴生成要求色谱柱的所有孔都具有反应或缓冲液对照。如果色谱柱的任何孔不含样品反应,则使用不含核酸酶的水以1:2的比例稀释2x ddPCR缓冲液对照。向色谱柱的任何空孔中加入 22 μL 1x ddPCR 缓冲液对照品。
注意:如果未使用整个色谱柱,则无需向这些孔添加缓冲液控制。 - 在板上添加可刺穿的箔密封。将板放入板封口机中,并在180°C下密封5秒。
- 或者,根据 ddPCR 系统制造商的建议密封板。
- 以最大速度涡旋板至少30秒(使用连续涡旋设置;不要使用触摸涡旋),并在平板旋转器中短暂离心。
注意:在此步骤中彻底彻底混合板对于将PCR反应正确分配为液滴至关重要。确保孔中没有可见的气泡。如有必要,板可以在液滴产生之前保持在2-8°C最多4小时。如果保持,在液滴产生之前让板达到室温至少 10 分钟。
8. 自动液滴生成、热循环和液滴读取
- 在自动液滴发生器中生成液滴,如下所示。
- 在触摸屏上,选择包含样品的板图上的列。仪器的甲板将亮起,指示需要哪些耗材(DG32 墨盒、吸头、废液容器、液滴发生油)。黄灯表示需要添加耗材,绿灯表示有足够的耗材可用。
- 从后到前加载液滴发生器。
- 对于水解探头,请确保安装了用于探头的液滴发生油,并且有足够的油用于孔数。如果使用替代PCR填料,请确保安装兼容的液滴发生油。
- 将冷块放入液滴板支架中。确保块是完全蓝色的,并且没有粉红色可见。将新的 96 孔 ddPCR 板放入冷块中。
- 将准备好的PCR板放入样品板架中。合上机器盖。按 开始 以产生液滴。
- 液滴形成后,每个反应总共 40 μL 自动转移到新的 PCR 板中。
- 在液滴生成完成后的30分钟内,从冷块中取出含有液滴的板。轻轻工作,因为液滴在这个阶段最脆弱。
- 在板上添加可刺穿的箔密封。将板放入板封口机中,并在180°C下密封5秒。
- 或者,根据 ddPCR 系统制造商的建议密封板。
- 将板放入兼容的热循环仪中。输入循环条件(见 表4)。
- 热循环结束后,将板保持在热循环仪中,转移到2-8°C,或立即读取。
注意:在4-12°C下保持板12小时可能会改善液滴计数,但这不是必需的。应在没有保持的情况下获得足够的液滴。 - 将板装入液滴读取器,确保有足够的读数器油残留,并且废物容器有足够的空间。阅读液滴。在热循环开始后24小时内进行液滴读数。
表 4:典型的热循环条件。请按此下载此表格。
9. 数据分析
注意:每孔至少需要10,000个液滴才能使用泊松统计正确计算浓度。不要尝试对任何液滴少于10,000的孔进行分析。
- 需要阈值才能将液滴定义为阳性或阴性。ddPCR 分析软件会自动应用可能因孔而异的阈值。但是,手动将板的所有孔的阈值设置为略高于NTC孔的荧光强度,以获得更一致,准确和精确的结果。
注意:阈值的正确放置可能需要优化,具体取决于正负液滴的分离以及存在的液滴雨量(见 图2)。在本例中,液滴振幅图显示了每个QC水平和NTC的示例孔。紫线表示阈值为 1,000,设置略高于负液滴群体。 - 泊松统计模型需要至少三个正液滴才能以 95% 的置信度计算浓度。考虑所有含有零、一或两个正液滴的孔均为负数,浓度设置为零27。
- 反向计算每个泪液样本中的拷贝数。
- 浓度(以拷贝/μL为单位)在数据报告中提供。使用此值可确定原始样品(即泪液样品)的浓度(以拷贝/μL为单位)。
- 要计算ddPCR反应稀释度,请将液滴形成前的初始PCR反应体积除以添加的模板体积。当使用此方法中提供的卷时,这将产生值 4。
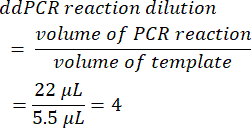
- 确定原始样品的连续稀释因子(步骤6.2)。
- 要确定样品中的拷贝数/μL,请将拷贝数/μL乘以ddPCR反应稀释液,然后乘以连续稀释因子。例如,数据报告中生成的拷贝/μL浓度为966;每 22 μL 反应加入 5.5 μL 模板。使用样品的1:50,000连续稀释。
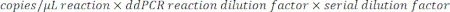
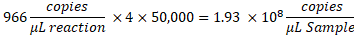
- 如果测试同一样品的多次稀释,请分析所有有效的范围内稀释液并计算平均值。
- 对于每个QC,通过将给定QC稀释液的浓度(以拷贝/μL为单位)除以ddPCR反应体积(20μL)来计算预期的拷贝数/ μL PCR反应。这允许将该标称值与数据报告中提供的拷贝/μL值直接进行比较,而无需进一步计算。
注意:这种方法还用于分析代表性结果中使用的加标泪液样品。 - 使用重复孔确定样品标称浓度(%RE)的平均值、标准偏差、变异系数(%CV)和相对误差百分比(%RE)或QC值(如果适用,包括多次稀释)。
- 为了评估孔间精度,请确定每个重复孔(如果包括)的精度。
- 为了评估测定内的准确性和精密度,请针对批次中使用的每个稀释系列或等分试样确定这一点。
- 为了评估测定间的准确性和精密度,请使用每个包含批次的测定内方法确定这一点。
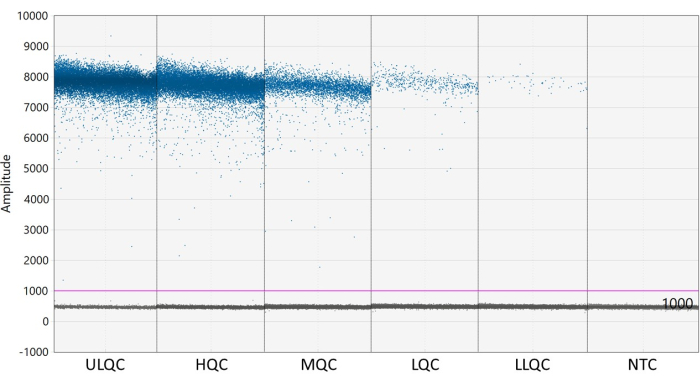
图 2:设置阈值的示例。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请点击此处查看此图的大图。
10. 检测验收标准
- 对每个批次的计算数据使用以下规范来确定该批次是否可接受。如果不满足这些条件,请使批处理失效并重复。
注意:这些标准是根据已发表的基于PCR的检测验证白皮书18,19,20,21,22,23,24,25确定的共识。可能需要根据临床应用修改目标标准。 - 无模板控件 (NTC)
- 确保每个NTC孔至少有10,000个液滴。
- 确保每个NTC孔的正液滴少于3个。
- 质量控制和检测范围
- 确保每个QC孔至少有10,000个液滴。
- 确保QC浓度的重复孔的精度为CV≤25.0%,但定量上限和下限除外,其中≤30.0%是可接受的。针对每个QC组和浓度水平独立评估。
- 确保每个平均QC水平下反向计算浓度的相对误差在标称浓度(拷贝/PCR反应)的±25.0%RE范围内,但在定量上限和下限除外,其中±30.0%RE是可接受的。针对每个QC组和浓度水平独立评估。
- 确保至少 2/3 的 QC 样品(例如,六个结果中的四个)和每个级别(低、中、高)的 50% 的 QC 样品符合这些准则。
- 样品
- 确保要分析的样品孔至少有10,000个液滴。
- 确保要分析的样品稀释液的重复孔的精度为≤25.0% CV。
- 确保给定样品的至少一种包含稀释液在测定的定义定量范围内,如上文根据上限和下限QC定义的那样。
- 如果所有稀释液的产量结果都大于定义的定量上限,并且如果样品体积足够,则使用更高的样品稀释度重复测定。
- 如果包含的所有稀释液产生的结果都低于定量下限,并且如果样品体积足够,则使用较低的样品稀释度重复测定。
注意:含有三个以上阳性液滴但浓度低于定量下限的样品可以描述为可检测,但不可定量。
结果
为了演示目的,开发了一种旨在检测市售的增强型绿色荧光蛋白(eGFP)表达AAV2载体的测定方法,其中含有eGFP的合成双链DNA片段作为质量控制。目前,关于载体本身或合成DNA片段或线性化质粒是否最适合用作QC的争论仍在继续。通常,如果在方法开发中证明与载体等效,则可以使用合成DNA片段或线性化质粒(数据未显示)。设计和优化引物和探针以检测eGFP转基因。有关本文中使用的序列,请参阅 补充表S1 。使用ddPCR经验测定QC片段原液的浓度。所有测定均使用协议部分中作为示例给出的浓度和PCR条件进行。
对于qPCR检测,建议评估标准曲线的线性、灵敏度、动态范围、准确度和精密度。由于ddPCR不依赖于标准曲线进行靶标定量,因此必须修改这些建议。取而代之的是,利用由合成双链DNA片段组成的QC,稀释到各种浓度,以跨越基于泊松统计模型的ddPCR反应的预期可定量范围29,30,31,32来定义动态范围和灵敏度,并评估准确性和精密度。QC浓度的选择主要基于给定浓度下孔内正液滴与总液滴的预期比率。从数学上讲,当大约80%的分区正扩增时,ddPCR理论上最准确。当正液滴与总液滴之比增加到0.8以上时,由于分区饱和,精度会降低,一旦100%的液滴为正,就无法定量。在低端,理论上,只需检测和定量一个阳性液滴,尽管准确性较差,并且检测受到低水平假阳性的影响。通常,必须至少有三个液滴为阳性才能以 95% 的置信度计算结果,这是我们在这里使用的计算浓度的阈值。
制备了一系列五种不同的QC浓度,计算出的目标拷贝数/μL PCR反应体积有望产生跨越ddPCR可量化范围的正液滴与总液滴比,如 表5所示。这些用于评估测定的准确性和精密度。在此评估中,定量的上限和下限没有被推到ddPCR中可能的理论最大值。在比此处所示的更高和更低的水平下,可以进行准确的定量。应根据该方法的下游应用开发该范围。
在每批样品稀释缓冲液中总共制备了三个独立制备的QC稀释系列,以评估测定内的准确性和精密度。包括每个QC稀释液的重复孔。为了模拟实际的验证方案,多名分析人员在多天内总共执行了六个准确度和精密度批次。分析这六个批次的结果,以确定方法的测定内和测定间准确性和精密度,并确定测定的动态范围。
在每个QC水平上评估每个批次的测定内性能。我们预计所有QC和NTC孔至少具有10,000个液滴。在所有六个批次测试的216个孔中,有216个井达到了这一要求,平均液滴计数为19,748个液滴/孔(表6)。其次,每个QC的每组重复孔的孔间%CV预计为≤25.0%,但上限和下限QC预计为≤30.0%。在所有六个批次的QC测试的90个孔中,有90个孔一组满足了这一要求,所有QC水平的平均孔间%CV为3.9%(表7)。所有QC均在上述预期范围内产生平均正液滴与总液滴比(表6)。
在每个批次中,计算每个独立制备的稀释系列点的测定内平均值和标准偏差,并用于计算每个测定中每个浓度的测定内平均值。这用于评估测定的准确性和精密度(表8)。精密度是指在正常测定条件下同一均质样品重复的数据变异性,并通过计算多个包含的等分试样的%CV来评估。我们预计每批中测试的三个等分试样将产生测定内%CV≤25.0%,但上限和下限QC除外,预计≤30.0%。60个批次中每个批次的所有五个QC级别都达到了这一要求(30个总性能中的30个)。通常,可以实现比目标标准更高的测定内精密度,所有QC水平的平均测定内%CV为7.7%。准确度是指实验确定的值与标称值之间的一致性。这是通过计算每个QC的计算浓度与其理论上预期的标称浓度之间的相对误差百分比(%RE或%Bias)来评估的。预计三种等分试样的测定内平均值将为标称浓度的±25.0%RE,但上限和下限QC除外,其中预计为±30.0%。60个批次中每个批次的所有五个QC级别都达到了这一要求(30个总性能中的30个)。一般来说,可以实现比我们的目标更高的检测内准确度,所有QC水平的平均绝对检测内%RE为4.2%。在NTC的所有性能(总共30个)中,没有检测到阳性液滴。
还使用每批中每个QC水平的测定内平均值计算测定间准确度和精密度。测定间精密度预计为≤25.0% CV,但上限和下限QC预计为≤30.0%。同样,对于测定间的准确性,预计RE为±25.0%,但上限和下限QC预计为±30.0%。观察到的测定间准确度和精密度明显高于这些靶标(表9),检测间精密度范围为4.0%至8.5%,测定间绝对准确度范围为1.0%至3.2%。总的来说,这些结果表明,该方法可以在当前的行业目标范围内实现足够的测定内和测定间准确度和精密度。根据这些结果,可以定义该测定的动态范围为每μL PCR反应2,500-2.5拷贝,总体测定灵敏度为每μL PCR反应2.5拷贝。如前所述,可以验证更宽的动态范围。
接下来,有必要评估目标基质内的测定准确性和精密度 - 在这种情况下,眼泪。通常,在临床研究开始之前对检测进行验证,这意味着从载体治疗的患者收集的眼泪不太可能用于验证目的。这可以通过将目标AAV载体加标到从志愿者捐赠者那里收集的眼泪中以创建基质加标QC来人工创建。 收集的人类眼泪由第三方(BioIVT)收集。为了验证原理,使用了从商业来源获得的表达eGFP的AAV2载体。使用ddPCR经验确定AAV2载体储备液的浓度,无需使用DNA分离步骤,如本协议所述。在每次运行中,AAV2 以高(预期 1.41 x 103 拷贝/μL PCR 反应)和低(28.2 拷贝/μL PCR 反应)水平独立加标到三个撕裂等分试样中。包括未加标的等分试样作为对照,以证明该方法的特异性。
在每个加标水平下评估每个批次的测定内性能。预计所有泪液样本至少会有10,000个液滴。在所有六个批次测试的108个孔中,有108个孔达到了这一要求,平均总液滴数为20,208个液滴/孔(表10)。接下来,对于高和低尖峰水平,每个QC的每组重复孔的孔间%CV预计为≤25.0%。在所有六个批次的QC测试的36组孔中,有36组达到了这一要求,平均井间%CV为3.2%(表11)。
在每个批次中,计算每个独立制备的泪液尖峰的测定内平均值和标准偏差,并使用这些平均值计算每个测定中每个浓度的测定内平均值。这用于评估基质中测定的准确性和精密度(表12)。我们预计测定内%CV为≤25.0%,峰值水平高低。每个级别的六批中有六批达到了这一要求。通常,可以实现比目标更高的基质测定内精度,高水平的平均测定内%CV为3.7%,低水平为12.2%(总体8.0%)。还预计在两个加标水平下测定内%RE将为±25.0%。每个级别的六批中有六批达到了这一要求。同样,通常发现基质的测定内准确度高于目标,低水平的平均测定内绝对 %RE 为 8.1%,高水平为 11.3%(总体为 9.7%)。对于未加标的对照,在任何等分试样中均未检测到eGFP信号(表12),证明了该方法在人泪液基质中的特异性。
还使用每批中每个加标水平的测定内平均值计算了泪液基质中的测定间准确度和精密度。预计检测间精密度为≤25.0% CV,对于检测间准确度,我们预计RE为±25.0%。观察到的测定间准确度和精密度明显高于这些靶标(表13),高水平检测间精密度为5.5%,低水平检测间精密度为7.1%,高水平绝对测定间准确度为11.3%,低水平为8.1%。总的来说,这些结果证明了该方法在泪液基质中的准确性、精密度和特异性。
表5:用于定义测定动态范围的质控品。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
表6:合成双链DNA质量控制和NTC的总液滴计数和正液滴与总液滴比。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
表7:QC孔间统计(拷贝靶标/μL PCR反应)。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
表8:QC的测定内准确度和精密度(拷贝靶标/μL PCR反应)。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
表9:QC的测定间准确度和精密度(拷贝靶标/μL PCR反应)。 缩写:ULQC = 质量控制上限;HQC = 高质量控制;MQC = 中等质量控制;LQC = 低质量控制;LLQC = 下限质量控制;NTC = 无模板控件。 请按此下载此表格。
表10:泪液样品的总液滴计数。请按此下载此表格。
表11:撕裂样品孔间统计(拷贝靶标/μL PCR反应)。请按此下载此表格。
表12:泪液样品的测定内准确度和精密度(拷贝靶标/μL PCR反应)。请按此下载此表格。
表13:泪液样品的测定间准确度和精密度(拷贝靶标/μL PCR反应)。请按此下载此表格。
补充表S1:本研究中使用的引物,探针和合成双链DNA质量控制序列。请按此下载此表格。
讨论
ddPCR方案的几个步骤对于测定的正确性能至关重要。第一个关键步骤是引物和探针的设计和优化。一般来说,建议在临床前或临床环境中使用基于水解探针的化学而不是基于染料的化学(例如,SYBR Green),因为它们具有优越的特异性。此外,扩增靶标的选择也很关键。通常,靶向载体感兴趣的转基因。然而,在早期临床前阶段或可能无法区分载体转基因与基因组DNA的载体中,使用标准化的载体靶标可能是合适的。例如,可以针对倒置末端重复区域、启动子、poly-A 尾或这些载体分量之间的段间连接。目标的选择将根据矢量设计而有所不同。传统的qPCR引物和探针设计策略和软件通常适用于ddPCR。应选择预期产生一致退火温度(例如 60°C)的设计参数,以减少所需的优化量。还建议为每个目标设计、订购和评估至少三套不同的套装。然后,应选择具有最大特异性(阴性对照孔或相关靶DNA基质中无扩增)和灵敏度(即检测限)的集合20。
如果能够在qPCR和ddPCR之间转换测定是有利的,建议首先使用qPCR优化测定条件,并确定所选集合的条件,使扩增效率为90%-110%,R2≥0.98。然而,由于扩增效率的差异,ddPCR 作为一种终点方法的灵敏度通常低于 qPCR。至少,建议在退火/延伸步骤中运行热温度梯度,以覆盖高于和低于预期退火温度的温度,并评估负液滴簇和正液滴簇之间的雨水和荧光幅度分离作为温度的函数。如果工作区允许,建议使用单独的专用工作站进行预混液制备、模板添加和扩增。在可能的情况下,这些应通过具有内置工程控制的单向工作流程(例如受控访问和压差)进行物理隔离,以降低交叉污染和误报的风险。如果无法做到这一点,则必须格外小心,以防止交叉污染。
该协议中有两个步骤对于那些更习惯于qPCR检测开发的人来说可能看起来不寻常。首先是在PCR预混液中加入限制性内切酶。在ddPCR扩增过程中,每个液滴都被热循环到终点。在适当优化的测定中,这导致两个液滴群,一组显示始终高水平的荧光信号 - 阳性 - 另一组始终显示低水平的荧光信号 - 阴性。如果发生PCR干扰,可能会使PCR扩增的启动不同步,导致液滴无法达到扩增平台,从而产生不一致的荧光终点。在这种情况下,液滴将分布在负极和正极之间,从而产生一种称为ddPCR雨的现象。这可能导致目标量化不准确,阈值应用不一致和主观。我们建议将阈值设置为略高于NTC信号的水平,这应该最大限度地减少最终定量中雨水的影响,因为即使没有完全循环到终点,所有液滴仍然被认为是阳性的。AAV具有高度复杂的二级结构,根据扩增靶标的不同,可能会降低引物和探针的可及性,从而导致PCR干扰,从而下雨。在预混液中加入限制性内切酶可切割该二级结构,以增加引物和探针的可及性,从而减少雨水,从而提高测定的准确性。在ddPCR反应中加入限制性内切酶的作用已在前面描述过25,32。可以使用任何限制性内切酶,只要确认其在靶标扩增区域内不切割即可。无需预酶切步骤或替代缓冲液组合物。
第二个不寻常的步骤是制备含有AAV的泪液样品。在该协议中,使用1:10(或更高)的眼泪比例,随后加热样品。通常,当通过毛细管收集眼泪(这是一种广泛使用的收集方法)时,平均可以收集约10.0μL33。稀释有助于解决样品体积有限的问题,并为重复的孔测试提供足够的材料。虽然这确实降低了检测的理论限,但ddPCR的强大灵敏度仍应能够检测除极少数载体颗粒之外的所有载体颗粒。这种方法还可以在意外失败时创建"备份"。在这种情况下,或者在样品体积不足以运行两个孔的情况下,泊松误差可用于评估精度。此外,在浓度低于检测限的情况下,它创造了合并孔数据以确定浓度的机会。有必要从病毒衣壳中释放AAV载体以进行ddPCR检测。一些用于定量AAV的方法包括蛋白酶K消化步骤以去除病毒衣壳34,35,36。所有天然存在的AAV血清型的熔化温度都在或低于约90°C,大多数低于80°C;因此,这似乎是不必要的包含37.仅加热似乎就足以释放载体DNA。
此外,ddPCR 通常不太容易受到可能影响 qPCR 测定的样品中可能存在的 PCR 抑制剂的影响。如果包括特定的DNA分离步骤,这也需要特定的验证,这在该协议中是避免的。由于载体基因组在液体中的扩散动力学,样品在加热前被稀释。在加热和随后的冷却过程中,如果浓度足够高,单链DNA基因组的正义和负义链可以一起退火以产生双链中间体。加热前的稀释会降低浓度,并且在数学上不太可能形成足够的双链中间体对定量准确性产生不利影响。应该注意的是,用作质量控制的合成DNA片段或线性化质粒不得经过此加热步骤。由于它们是双链的,加热会导致转化为单链中间体。将这些单链QC独立分配成液滴后,预计QC浓度相对于标称浓度增加两倍。或者,如果要加热QC以标准化方法,则必须将其考虑在标称浓度的重构和分配中。
最后,关于样品制备,许多方案还建议包含DNase处理步骤以去除任何未包封的载体DNA。在不需要定量与载体制备相关的游离DNA的情况下(例如在定量剂量期间),此步骤至关重要。然而,在生物分布和生物脱落研究的背景下,人们通常希望知道任何载体DNA的传播位置,无论它是否被包衣。因此,建议在此类研究中通常不执行DNase处理步骤。如果确定有必要包括DNase步骤,此步骤应在稀释和加热之前进行。
在本文中,介绍了在符合目的的良好实验室规范验证背景下评估方法动态范围、灵敏度、准确度和精密度的数据。目前缺乏关于该主题的指导,使得验证实验室无法自行确定靶标检测标准,这与当前的行业思维一致。不同的组提出了比本研究中使用的更高和更低的目标标准19,20,21,22,23,24,25。在更严格定义之前,应根据方法的预期临床应用在验证之前选择目标测定标准。根据根据数据做出的下游决策,可能需要更高水平的准确度和精密度。相反,简单的阳性与阴性结果可能就足够了。
该方法还涉及评估特异性和基质效应的建议。从未经治疗的个体收集的泪液池在该测定中未能产生阳性结果,而当载体在推荐的回收率内以高浓度和低浓度加标到眼泪中时,可以检测到靶标。理想情况下,含有内源性载体的基质(例如,用病毒载体处理后收集的)也将包含在这些评估中。但是,此类样本不太可能可用于验证。为了提高验证的稳健性,可以评估多个眼泪池或从各种个体收集的眼泪,以确定是否发生患者特异性基质效应。最后,建议评估稳定性。在从生物基质中提取DNA的工作流程中,可能需要评估样品和提取的DNA的稳定性。在该工作流程中,样品直接在检测中进行测试,无需提取DNA。因此,考虑到该方法稳定性的评估,必须评估泪液样品的稳定性。通常,建议进行台式、冰箱、冻融和长期稳定性评估。这些不是作为本研究的一部分进行的,但这里开发的方法可用于本次评估,在对输入样本进行操作之后。
总体而言,该方法已被证明是一种稳定、可重复且可验证的检测方法,可用于检测泪液样品中基于 AAV 的载体。它可以作为一个平台,适应特定载体以支持临床试验,并为验证符合良好实验室规范的测定提供基础。
披露声明
所有作者都是KCAS生物分析和生物标志物服务的员工,KCAS是一家合同研究组织,提供全面的良好实验室规范/良好临床规范合规开发服务,从早期发现支持到产品注册,包括支持基于AAV的测定,如本协议所述。KCAS资助了该项目并出版了该协议。虽然目前没有FDA指南来设计和验证本文中提出的实验,但在KCAS进行此类研究的专家和思想领袖已采用这种方法作为基线方法。其他标准和参数在逐个项目的基础上进行讨论,并可根据预期用途包括在内。
致谢
我们要感谢Bio-Rad的Nick Russell和Brandon McKethan在这种方法的开发过程中进行的有益讨论。
材料
| Name | Company | Catalog Number | Comments |
| AAV-eGFP Vector | Charles River Laboratories | RS-AAV2-FL | Lot AAV2-0720-FL, used as a proof of principle vector |
| AutoDG Droplet Digital PCR system | Bio-Rad | QX200 | Alternative ddPCR system may be used following manufacturer’s protocol. |
| AutoDG Oil for Probes | Bio-Rad | 1864110 | Or use material compatible with ddPCR system. |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | Or use material compatible with ddPCR system and PCR chemistry. |
| ddPCR Droplet Reader Oil | Bio-Rad | 1863004 | Or use material compatible with ddPCR system. |
| ddPCR Piercable Foil Seals | Bio-Rad | 1814040 | Or use material compatible with ddPCR system. |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad | 12001925 | Or use material compatible with ddPCR system. |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023, 1863024, or 1863025 | Use master mix compatible with primers/probes and ddPCR system. |
| DG32 AutoDG Cartidges | Bio-Rad | 1864108 | Or use material compatible with ddPCR system. |
| Droplet Reader | Bio-Rad | QX200 | Alternative ddPCR system may be used following manufacturer’s protocol. |
| GeneAmp PCR Buffer | Applied Biosystems | N8080129 | N/A |
| Nuclease-Free Water | Ambion | AM9906 | N/A |
| PCR Plate Sealer | Bio-Rad | PX1 | Or use material compatible with ddPCR system. |
| Pipet Tips for AutoDG | Bio-Rad | 1864120 | Or use material compatible with ddPCR system. |
| Pluronic F-68 Non-ionic Surfactant | Gibco | 24040 | N/A |
| Primer and Hydrolysis Probes | Various | Various | Design based on target sequence using general approaches for primer/probe design. Select fluorphores and quenchers compatible with ddPCR system. |
| Restriction Enzyme | Various | Various | Varies with target amplification sequence. Use restriction enzyme that does not cut in the amplified sequence |
| Sheared salmon sperm DNA | ThermoFisher | AM9680 | N/A |
| Synthetic DNA gene fragment or linearized plasmid | Various | Various | Design a synthetic DNA fragment containing the target amplification region for use as a quality control |
| TE Buffer | Teknova | T0224 | Ensure prepared or purchases nuclease free. 10 mM Tris-HCl, 1.0 mM EDTA, pH=8.0 |
| Touch Thermal Cycler | Bio-Rad | C1000 | Or use material compatible with ddPCR system. |
参考文献
- Sherkow, J. S., Zettler, P. J., Greeley, H. T. Is it 'gene therapy. Journal of Law and the Biosciences. 5 (3), 786-793 (2018).
- Ginn, S. L., Amaya, A. K., Alexander, I. E., Edelstein, M., Abedi, M. R. Gene therapy clinical trials worldwide to 2017: an update. The Journal of Gene Medicine. 20 (5), 3015 (2018).
- Ghosh, S., Brown, A. M., Jenkins, C., Campbell, K. Viral vector systems for gene therapy: a comprehensive literature review of progress and biosafety challenges. Applied Biosafety. 25 (1), 7-18 (2020).
- Gene, Cell, & RNA therapy landscape, Q4 2022 quarterly data report. American Society for Gene & Cell Therapy Available from: https://asgct.org/global/documents/asgct_citeline-q4-2022-report_final.aspx (2022)
- Approved Cellular and Gene Therapy Products. United States Food and Drug Administration Available from: https://www.fda.gov/vaccines-blood-biologics/cellular-gene-therapy-products/approved-cellular-and-gene-therapy-products (2022)
- Au, H. K. E., Isalan, M., Meilcarek, M. Gene therapy advances: a meta-analysis of AAV usage in clinical settings. Frontiers in Medicine. 8, 809118 (2022).
- Lundstrom, K. Viral vectors in gene therapy. Diseases. 6 (2), 42 (2018).
- Naso, M. F., Tomkowicz, B., Perry, W. L., Strohl, W. R. Adeno-associated virus (AAV) as a vector for gene therapy. BioDrugs. 31, 317-334 (2017).
- Russell, S., et al. Efficacy and safety of voretigene neparvovec (AAV2-hRPE65v2) in patients with RPE65-mediated inherited retinal dystrophy: a randomized, controlled, open-label, phase 3 trial. Lancet. 390 (10097), 849-860 (2017).
- Chemistry, manufacturing, and control (CMC) information for human gene therapy investigational new drugs (INDs). United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/chemistry-manufacturing-and-control-cmc-information-human-gene-therapy-investigationsal-new-drug (2020)
- Long term follow-up after administration of human gene therapy products. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/long-term-follow-after-adminstration-human-gene-therapy-products (2020)
- Testing of retroviral vector-based human gene therapy products for replication competent retrovirus during product manufacture and patient follow-up. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/testing-retroviral-vector-based-human-gene-therapy-products-replication-competent-retrovirus-during (2020)
- Recommendations for microbial vectors used in gene therapy. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/recommendations-microbial-vectors-used-gene-therapy (2016)
- Multidisciplinary: gene therapy. European Medicines Agency Available from: https://www.europa.eu/en/human-regulatory-development/scientific-guidelines/multidisciplinary/multidisciplinary-gene-therapy (2023)
- Human gene therapy for retinal disorders. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/human-gene-therapy-retinal-disorders (2020)
- Design and analysis of shedding studies for virus or bacterial-based gene therapy and oncolytic products. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/design-and-analysis-shedding-studies-virus-or-bacteria-based-gene-therapy-and-oncolytic-products (2015)
- Guideline on the quality, non-clinical and clinical aspects of gene therapy medicinal products. European Medicines Agency Available from: https://www.ema.europa.eu/en/documents/scientific-guideline/guideline-quality-non-clinical-clinical-aspects-gene-therapy-medicinal-products_en.pdf (2018)
- Kaur, S. white paper on recent issues in bioanalysis: mass spec of proteins, extracellular vesicles, CRISPR, chiral assays, oligos; nanomedicines bioanalysis; ICH M10 section 7.1; non-liquid & rare matrices; regulatory inputs (part 1A - recommendations on endogenous compounds, small molecules, complex methods, regulated mass spec of large molecules, small molecule, PoC & part 1B - regulatory agencies' inputs on bioanalysis, biomarkers, immunogenicity, gene & cell therapy and vaccine). Bioanalysis. 14 (9), 505-580 (2022).
- Hays, A., Islam, R., Matys, K., Williams, D. Best practices in qPCR and qPCR validation in regulated bio analytical laboratories. The AAPS Journal. 24 (2), 36 (2022).
- Ma, H., Bell, K. N., Loker, R. N. qPCR and qRT-PCR analysis: Regulatory points to consider when conducting biodistribution and vector shedding studies. Molecular Therapy. Methods & Clinical Development. 17 (20), 152-168 (2020).
- Wissel, M. Recommendations on qPCR/ddPCR assay validation by GCC. Bioanalysis. 14 (12), 853-863 (2022).
- Expectations for biodistribution (BD) assessments for gene therapy (GT) products. International Pharmaceutical Regulators Programme Available from: https://admin.iprp.global/sites/default/files/2018-09/IPRP_GTWG_ReflectionPaper_BD_Final_2018_0713.pdf (2018)
- Pinheiro, L., Emslie, K. R. Basic concepts and validation of digital PCR measurements. Methods in Molecular Biology. 1768, 11-24 (2018).
- Tzonev, S. Fundamentals of counting statistics in digital PCR: I measured two target copies-what does it mean. Methods in Molecular Biology. 1768, 25-43 (2018).
- Prantner, A., Marr, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18, 0280242 (2023).
- Primer-BLAST. National Library of Medicine, National Center for Biotechnology Information Available from: https://www.ncbi.nim.nih.gov/tools/primer-blast/ (2023)
- Koressaar, T., Remm, M. Enhancements and modifications of primer design program Primer 3. Bioinformatics. 23 (10), 1289-1291 (2007).
- Ye, J. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134 (2012).
- Droplet Digital PCR Application Guide. Bio-Rad Available from: https://www.bio-rad.com/webroot/we/pdf/lsr/literature/Bulletin_6407.pdf (2023)
- Qian, P. L., Sauzade, M., Brouzes, E. dPCR: A technology review. Sensors. 18 (4), 1271 (2018).
- Basu, A. Digital assays part I: portioning statistics and digital PCR. SLAS Technology. 22 (4), 369-386 (2017).
- Sanmiguel, J., Gao, G., Vandeberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods in Molecular Biology. 1950, 51-83 (2019).
- Bachhuber, F., Huss, A., Senel, M., Tumani, H. Diagnostic biomarkers in tear fluid: from sampling to preanalytical processing. Scientific Reports. 11, 10064 (2021).
- Martinez-Fernandez de la Camara, C., McClements, M. E., MacLaren, R. E. Accurate quantification of AAV vector genomes by quantitative PCR. Genes. 12 (4), 601 (2021).
- Ai, J., Ibraheim, R., Tai, P. W. L., Gao, G. A scalable and accurate method for quantifying vector genomes of recombinant adeno-associated viruses in crude lysate. Human Gene TherapyMethods. 28 (3), 139-147 (2017).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Frontiers in Microbiology. 10, 1570 (2019).
- Bennett, A., et al. Thermal stability as a determinant of AAV serotype identity. Molecular Therapy Methods and Clinical Development. 6, 171-182 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。