Method Article
涙液のバイオシェディング研究におけるアデノ随伴ウイルスベクターの検出のための検証可能な液滴デジタルポリメラーゼ連鎖反応アッセイ
要約
ここでは、遺伝子治療ベクターの臨床開発を支援するために、液滴デジタルポリメラーゼ連鎖反応によるヒト涙液中のアデノ随伴ウイルスベクターのコンプライアント検出における優れた実験室慣行の開発と検証のためのプロトコルを提示します。
要約
遺伝病を治療するためのウイルスベクターの使用は近年大幅に増加しており、これまでに2,000を超える研究が登録されています。アデノ随伴ウイルス(AAV)ベクターは、ボレチゲンネパルボヴェック−rzylの承認によって例示されるように、眼関連疾患の治療において特に成功を見出した。新しい治療法を市場に投入するために、規制当局は通常、環境へのベクターの放出を評価するために、適格または検証済みのバイオシェディング研究を要求します。ただし、このような脱落研究をサポートするための分子ベースのアッセイの開発に関する公式のガイドラインは、米国食品医薬品局によってリリースされておらず、開発者は自分でベストプラクティスを決定する必要があります。このプロトコルの目的は、臨床バイオシェディング研究をサポートするために、液滴デジタルポリメラーゼ連鎖反応(ddPCR)によるヒト涙液中のAAVベクターを検出するための検証可能なプロトコルを提示することです。この原稿では、分子アッセイバリデーションに対する現在の業界のアプローチについて説明し、この方法がホワイトペーパーで現在提案されているターゲットアッセイの受け入れ基準を超えていることを示しています。最後に、アプリケーションに関係なく、ddPCRアッセイの性能に重要なステップについて説明します。
概要
遺伝子治療の定義はさまざまですが、一般に、遺伝子の発現を変更または操作したり、臨床目的で生細胞の生物学的特性を変更したりするために、細胞ゲノムの特定のDNA配列の意図的でしばしば予想される永続的な交替を誘発します1,2。ウイルスベクターは、形質導入の効率から遺伝子治療のビヒクルとしてますます使用されており、現在の遺伝子治療臨床試験の70%以上がウイルスベクターを利用していることを示唆する報告もあります3。遺伝子治療用のウイルスベクターへの関心は着実に高まっています。米国遺伝子細胞治療学会の遺伝子、細胞、RNA療法の状況に関する2022年第4四半期データレポートによると、2022年には、前臨床から事前登録までの遺伝子、細胞、RNA治療のパイプラインが7%増加し、開発中の治療法の総数は3,726になり、そのうち2,053(55%)が遺伝子治療でした4.米国食品医薬品局(USA FDA)は現在、ヒトでの臨床使用のために27の細胞および遺伝子治療を承認しており、そのうち5つはウイルスベクターを特異的に利用しています5。
アデノ随伴ウイルス(AAV)は、遺伝子治療の媒体として特に関心を集めています。最近のメタアナリシスでは、過去136年間にAAVの使用を調査した約6の臨床試験があったことが明らかになりました。さらに、米国FDAが承認した5つの遺伝子治療のうち3つはAAVベースです。これは、それらの高度に編集可能な性質、特定の天然または人工的に設計されたベクターの使用に基づいて調整できる幅広い宿主範囲、ヒトにおける低い病原性と毒性、および一般的に低い免疫原性によるものです7,8。AAVは、承認された臨床現場での眼疾患の治療にも成功裏に使用されています。ボレチゲンネパルボヴェック-rzylは、2017年に米国FDAによって、2018年に欧州医薬品庁(EMA)によって、二対立遺伝子RPE65変異関連網膜ジストロフィーを治療するために承認されたAAV2ベースの治療法です9。
AAVベースの治療法の開発への関心が高まるにつれ、アッセイに関する規制ガイダンスの必要性が高まっています。ウイルスベクターの正確な検出と定量は、製品開発の発見、製造、前臨床/臨床試験の段階の不可欠な部分です。米国FDAは、ヒト遺伝子治療治験薬申請10、遺伝子治療投与後の長期フォローアップ11、複製能力のあるレトロウイルス検査12、遺伝子治療に使用される微生物ベクターの推奨13など、遺伝子治療に関するガイダンスの発行を開始しました.EMAはまた、FDAの推奨事項に一般的に一致する遺伝子治療製品の開発に関する一連のガイドラインをリリースしましたが、いくつかの違いがあります14。これらのガイダンスは、特定の規制が参照されている場合を除き、法的強制力のある責任を確立するものではありませんが、このトピックに関する規制当局の現在の考え方と、医薬品の申請と規制当局の承認に必要なアッセイに対する期待を明確にしていることに注意することが重要です。
FDAは、眼および非眼組織、眼内液、および血液を標的とする投与部位からのベクターの分布、持続性、およびクリアランスを評価するための研究を実施する必要があることを特に推奨しています15。これらは、生体内分布および脱落研究の形をとります。生体内分布研究では、製品が投与部位から患者の体全体にどのように広がっているかを調査することにより、曝露を評価します。脱落は、患者から環境への生成物の放出を具体的に評価し、未治療の個体へのベクターの伝播の可能性を高める16。FDAは、サンプル収集の頻度、サンプル収集の期間、収集されたサンプルの種類、および保管条件に関して、生体内分布および脱落研究の設計に関する推奨事項を作成します。
さらに、FDAは、性能の容易さ、ハイスループットフォーマット、迅速なターンアラウンドタイム、およびアッセイ感度により、ベクターゲノムの定量的検出に定量的ポリメラーゼ連鎖反応(qPCR、またはリアルタイムPCR)の使用を推奨しています。ただし、分子法の設計と性能評価に関する推奨事項は、低分子および高分子に存在するものと比較して相対的に不足しています。このような研究のガイドラインの多くは、製品とアッセイ自体の両方の独特で複雑な設計のために分子法に適用するのが困難であり、推奨される評価に利用可能なプラットフォームの適切性とアッセイ検証のための適切な方法に関して疑問を投げかけています。現在まで、FDAはPCRベースのアッセイの正式な検証を要求していませんが、EMAはこの要件を課しています17。この空白に照らして、さまざまなグループやワークショップが、製造業者や受託研究機関が従うことを求めているホワイトペーパーと推奨事項を発行しました18,19,20,21,22,23,24,25。これらの推奨事項のほとんどは、特にqPCRアッセイを念頭に置いて書かれており、ドロップレットデジタルPCR(ddPCR)などの新しいプラットフォームの提案または変更は、関連性があると見なされる場合にのみ含まれています。最近の推奨事項では、ddPCRアッセイの考慮事項に焦点を当てていますが、バイオシェディング研究で遭遇する複雑な生物学的マトリックスではなく、製造環境でのベクターゲノム定量への応用に主に焦点を当てています。
臨床アプリケーションと目標によっては、qPCRと比較してddPCRの感度とマトリックス干渉を処理する能力が向上しているため、生体内分布および脱落研究をサポートするために、ddPCRがqPCRよりも好まれる可能性があります。さらに、サンプルを約20,000個の液滴に分割するため、ポアソン統計量を用いた検量線を使用せずにコピー数の正確な定量化が可能となり、メソッドの開発と検証が簡素化されます。このプロトコルの目的は、臨床バイオシェディング研究をサポートするために、眼表面から収集された涙液中のAAVベクターを検出するためのddPCRベースの方法の開発と検証のための標準化されたアプローチを説明することです。
プロトコル
1. 合成DNA断片の調製
- 品質管理として使用するために、標的増幅領域を含む合成DNA断片を設計して注文します。
- 配列に、目的の標的遺伝子のフォワードプライマーからリバースプライマーまでのアンプリコン配列全体が含まれ、各プライマー結合配列の5'末端に4〜6塩基対の配列が延長されていることを確認します。
- 12塩基対を超えるアデニンおよびチミンのホモポリマー、または8塩基対を超えるグアニンおよびシトシン塩基対のホモポリマーは、ホモポリマーが遺伝子断片の合成を妨げる可能性があるため避けてください。
注:アンプリコンにそのような配列が含まれている場合、プライマーおよびプローブのアニーリング部位が維持されている限り、塩基置換を行うことができます。 - あるいは、典型的なクローニング戦略を用いてアンプリコンを含む直鎖化プラスミドを調製します。
- 合成DNA断片を含むチューブをマイクロ遠心分離機で~10秒間遠心分離し、材料がチューブの底に集められるようにします。
- tris-EDTA(TE)バッファーを使用して合成DNA断片を1.0 ×10 10 コピー/μLの濃度に、またはターゲットアッセイ範囲に基づいて適切に再懸濁します。
- 短時間ボルテックスした後、50°Cで20±5分間インキュベートします。氷の上で冷やす。
- 複数の、理想的には使い捨てのアリコートを準備し、使用するまで-70〜-90°Cで保存します。
注:この方法で調製された合成DNA断片は、通常、再懸濁日から少なくとも24ヶ月間安定です。 - 必要に応じて、品質管理として使用する前に調製した合成DNAストックの正確な濃度を決定するか、使用する再懸濁液に基づいて公称濃度を推定します。
2. プライマーとプローブの調製
- 典型的な設計戦略26、27を使用して所望の増幅領域を標的とするプライマーおよび加水分解プローブを設計および注文する。
- ddPCRシステムと互換性のある5'蛍光レポーター色素(FAMなど)と3'クエンチャー(アイオワブラックダーククエンチャーなど)を利用します。
注:多数のPCRアッセイデザインソフトウェアパッケージが存在し、どれでも利用できます。例えば、国立バイオテクノロジー情報センター28 によるPrimer-BLASTは、アッセイ設計のための堅牢なオプションと、可能性のあるオフターゲット効果を特定するために特異性をバイオインフォマティックに評価することが容易であるため、広く使用されています。プライマーおよびプローブの調製は、それらが供給される形式に応じて、ここに列挙されたステップとは異なる場合があることに留意されたい。
- ddPCRシステムと互換性のある5'蛍光レポーター色素(FAMなど)と3'クエンチャー(アイオワブラックダーククエンチャーなど)を利用します。
- フォワードプライマー、リバースプライマー、プローブを含むチューブをマイクロ遠心分離機で~10秒間遠心分離し、材料をチューブの底までペレット化します。
- TE バッファーを使用してプライマーを 20 μM に再懸濁します。簡単に渦。
- TE バッファーを使用してプローブを 10 μM に再懸濁します。簡単に渦。
- 複数の理想的には使い捨てのアリコートを準備し、使用するまで最低-20°Cで保管してください。
注:この方法で調製されたプライマーおよびプローブは、通常、再懸濁日から少なくとも24ヶ月間安定です。
3. サンプル希釈バッファーの調製
- PCRバッファーと剪断したサケ精子DNAを室温で解凍する。徹底的に渦をかけて混ぜます。
- 表1に従ってサンプル希釈バッファーを調製します。
- 徹底的に渦。調製後2〜8°Cで最大1か月間保管してください。
表1:サンプル希釈バッファーの調製。この表をダウンロードするには、ここをクリックしてください。
4.マスターミックスの調製
- プローブ、フォワードプライマー、リバースプライマー、およびプローブ用のddPCRマスターミックスを室温で解凍し、使用前に解凍後少なくとも10分間温めます。使用するまで室温で保管してください。
注:これらの試薬は、効率的な液滴形成を確実にするために、完全に室温にする必要があります。調製中は試薬を氷の上に置かないでください。- ボルテックスは、使用前にミニ遠心分離機で徹底的にそして短時間遠心分離する。
注意: 制限酵素は通常グリセロールで供給され、使用直前に保管場所から取り除く必要があります。穏やかに混ぜる。渦巻かないでください。
- ボルテックスは、使用前にミニ遠心分離機で徹底的にそして短時間遠心分離する。
- 各増幅ターゲットのPCRマスターミックスを調製します。推奨されるPCRマスターミックス組成については 表2 を参照し、必要に応じてプライマーとプローブの濃度を変更します。
- 制限酵素を添加する前に十分にボルテックスおよび短時間遠心分離する。制限酵素を加え、反転させて混ぜます。
注:このステップでは、液滴形成後の最終容量40 μLのPCR反応(15 μLのPCRマスターミックス、5.0 μLのテンプレート、および20 μLの液滴生成オイルで構成される)を得るために、22 μLのPCR反応が必要です。
- 制限酵素を添加する前に十分にボルテックスおよび短時間遠心分離する。制限酵素を加え、反転させて混ぜます。
- プレートマップに従って、16.5 μLのマスターミックスを各ウェルに追加します。検証精度と精度実行のプレートマップの例については、 図1 を参照してください。
- プレートに、品質管理(QC)シリーズの3つの独立した調製物、テストされた3つの独立してテストされた内因性涙液アリコート、高レベルと低レベルにスパイクされ、スパイクされていない、および3つの独立したテンプレートなしコントロール(NTC)が含まれていることを確認します。
- プレート全体でこれらのウェルのレイアウトを変更し、セット1を濃度の高い順にロードし、セット2を濃度の高い順にロードし、セット3をランダムな順序でロードして、プレートの位置固有の効果があるかどうかを評価します。
- サンプルを配列して、できるだけ多くのカラムを満たし、カラム内の未使用のウェルをコントロールバッファーで満たします。必要に応じて、複数の内因性コントロールロット(例えば、より多くの涙のプールまたは個人から収集された涙)を残りのウェルに含めます。
- 透明な粘着フィルムでプレートをシールします。テンプレートの準備中は、プレートを室温で保持します。または、プレートを4〜2°Cで最大8時間保持しますが、テンプレートを追加する前に少なくとも10分間室温に戻します。
表2:PCRマスターミックス調製例。この表をダウンロードするには、ここをクリックしてください。
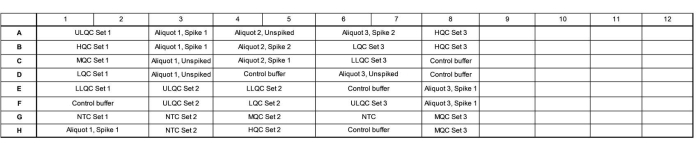
図1:検証精度と精密実行のプレートマップの例。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この図の拡大版を表示するには、ここをクリックしてください。
5. 品質管理の準備
- 合成DNA断片または直鎖状プラスミドを室温で解凍し、使用前に解凍後少なくとも10分間温めます。テンプレートを室温にして、効率的な液滴形成を確保します。
- 使用するまで室温で保管してください。ボルテックスは、使用前にミニ遠心分離機で徹底的にそして短時間遠心分離する。
- サンプル希釈バッファーを希釈液として使用してQC希釈液を調製します。検証精度と精度の実行を準備するための推奨濃度の例を 表3に示します。
注意: 精度と精度の実行が正常に完了した後、各プレートで高品質制御(HQC)、中品質制御(MQC)、および低品質管理(LQC)のみを実行する必要があります。精度と精度のために、アッセイ内の精度と精度を評価するために、QCの少なくとも3つの独立した希釈が含まれています。精度と精度の実行に続いて、1つの希釈シリーズのみを含める必要があります。 - 調製後、希釈液をプレートに添加するまで室温で保存する。
- 希釈液を氷上または必要に応じて2〜8°Cで保管します。その後の使用の前に、希釈液を使用前に少なくとも10分間室温に温めてください。.一日の終わりにQCを破棄します。
表3:合成二本鎖DNA断片を用いた品質管理(QC)調製例。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
6. サンプルの調製
- 臨床試験から収集した涙液サンプルを室温で解凍するまで解凍し、使用前に解凍後少なくとも10分間温めます。
- 使用するまで室温で保管してください。ボルテックスは、使用前にマイクロ遠心分離機で徹底的かつ短時間遠心分離します。
- サンプル希釈バッファーを希釈液として使用して、涙液サンプルを 1:10 (またはそれ以上) に希釈して、0.2 mL の PCR チューブまたは 8 ウェル PCR ストリップにします。チューブを密封します。
注:涙液中のターゲットの予想される濃度によっては、サンプルをさらに希釈するか、各サンプルの複数の希釈をテストする必要がある場合があります。 - サンプルをサーマルサイクラーで95°Cで10分間加熱した後、4°Cで5分以上保持して冷却します。3°C/sのランプレートを使用してください。
注意: サンプルは、同じ日に使用するまで4°Cでサーマルサイクラーに留まるか、-70〜-90°Cで凍結して長期間保管することができます。このステップは、ベクターキャプシドを変性させ、ゲノムを放出する役割を果たす。QC合成DNAフラグメントまたは直鎖化プラスミドは二本鎖であるため、この加熱ステップを経ないでください。 - 冷却後のサンプルを室温に戻し(または凍結した場合は室温で解凍)、少なくとも10分間温めます。
注:効率的な液滴形成を確実にするために、サンプルを完全に室温にする必要があります。
7. テンプレートの追加
- マスターミックスを含むddPCRプレートを取り出します。各サンプルまたはQC希釈管を徹底的に短時間遠心分離してボルテックスし、材料を回収します。
- 接着フィルムをはがし、プレートマップに従って、5.5 μLのQCまたはサンプルを96ウェルプレートの適切なウェルに加えます。
メモ: 必要なボリュームの説明については、手順 4.2.1 を参照してください - 5.5 μLのサンプル希釈バッファーをNTCウェルに追加します。
- 液滴生成には、カラムのすべてのウェルに反応制御または緩衝制御が必要です。カラムのウェルにサンプル反応が含まれていない場合は、ヌクレアーゼフリー水を使用して 2x ddPCR バッファーコントロールを 1:2 に希釈します。22 μL の 1x ddPCR バッファーコントロールをカラムの空のウェルに追加します。
注: カラム全体を使用しない場合は、これらのウェルにバッファー制御を追加する必要はありません。 - プレートに穴を開け可能なホイルシールを追加します。プレートをプレートシーラーに入れ、180°Cで5秒間シールします。
- または、ddPCRシステムメーカーの推奨に従ってプレートをシールします。
- プレートを最高速度で少なくとも30秒間渦流し(連続渦渦設定を使用、タッチボルテックスは使用しないでください)、プレートスピナーで短時間遠心分離します。
注:このステップでのプレートの完全かつ完全な混合は、PCR反応を液滴に適切に分割するために重要です。ウェル内に気泡が見えないことを確認してください。必要に応じて、液滴生成前にプレートを2〜8°Cで最大4時間保持することができます。保持されている場合は、液滴を生成する前に、プレートを最低10分間室温に戻します。
8.自動液滴生成、熱サイクル、および液滴読み取り
- 次のように自動液滴発生器で液滴を生成します。
- タッチスクリーンで、サンプルを含むプレートマップ上の列を選択します。機器のデッキが点灯し、必要な消耗品(DG32カートリッジ、チップ、廃棄物容器、液滴生成オイル)を示します。黄色のライトは消耗品を追加する必要があることを示し、緑色のライトは十分な消耗品があることを示します。
- 液滴発生器を後ろから前にロードします。
- 加水分解プローブの場合は、プローブ用の液滴生成オイルが取り付けられ、ウェル数に対して十分なオイルが残っていることを確認してください。代替PCR化学物質を使用する場合は、互換性のある液滴生成オイルが取り付けられていることを確認してください。
- 液滴プレートホルダーにコールドブロックを置きます。ブロックが完全に青色で、ピンクが見えないことを確認します。新しい 96 ウェル ddPCR プレートをコールドブロックに入れます。
- 準備したPCRプレートをサンプルプレートホルダーに入れます。機械の蓋を閉めます。液滴生成のために 開始 を押します。
- 液滴形成後、反応あたり合計40 μLが新しいPCRプレートに自動的に移されます。
- 液滴生成が完了してから30分以内に、液滴を含むプレートをコールドブロックから取り出します。この段階で液滴が最も壊れやすいので、穏やかに作業してください。
- プレートに穴を開け可能なホイルシールを追加します。プレートをプレートシーラーに入れ、180°Cで5秒間シールします。
- または、ddPCRシステムメーカーの推奨に従ってプレートをシールします。
- プレートを互換性のあるサーマルサイクラーに置きます。サイクリング条件を入力します( 表4を参照)。
- サーマルサイクルの終了後、プレートをサーマルサイクラーに保持し、2〜8°Cに移すか、すぐに読み取ります。
注意: プレートを12〜4°Cで12時間保持すると、液滴数が改善される可能性がありますが、これは必須ではありません。ホールドなしで十分な液滴が得られるはずです。 - プレートを液滴リーダーにロードし、十分なリーダーオイルが残り、廃棄物容器に十分なスペースがあることを確認します。液滴を読み取ります。熱サイクル開始から24時間以内に液滴の読み取りを実行します。
表4:典型的な熱サイクル条件。この表をダウンロードするには、ここをクリックしてください。
9.データ分析
注:ポアソン統計量を使用して濃度を適切に計算するには、ウェルあたり最低10,000液滴が必要です。液滴が10,000個未満のウェルでは分析を試みないでください。
- 液滴を正または負として定義するには、しきい値が必要です。ddPCR分析ソフトウェアは、ウェル間で異なる可能性のある閾値を自動的に適用します。ただし、プレートのすべてのウェルのしきい値をNTCウェルの蛍光強度よりわずかに高く設定すると、より一貫性があり、正確で、正確な結果が得られます。
注:しきい値を適切に配置するには、正と負の液滴の分離と液滴雨の存在量に応じて最適化が必要になる場合があります( 図2を参照)。この例では、液滴振幅グラフは、各QCレベルおよびNTCにおけるウェルの例を示している。紫色の線は、負の液滴集団よりわずかに上に設定された1,000のしきい値を示します。 - ポアソン統計モデリングでは、95%の信頼度で濃度を計算するために、少なくとも3つの正の液滴が必要です。ゼロ、1、または2つの正の液滴を含むすべてのウェルを陰性と見なし、ゼロ27の濃度に設定します。
- 各涙サンプルのコピー数を逆算します。
- 濃度(コピー/μL)は、データレポートに記載されています。この値を使用して、元のサンプル(つまり、涙液サンプル)のコピー/ μLで濃度を決定します。
- ddPCR反応希釈率を計算するには、液滴形成前の初期PCR反応量を添加したテンプレートの体積で割ります。このメソッドで示されたボリュームを使用すると、値は 4 になります。
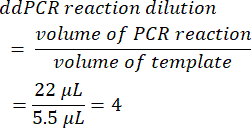
- 元のサンプルから段階希釈係数を決定します(ステップ6.2)。
- サンプル中のコピー/μLを決定するには、コピー/μLにddPCR反応希釈を掛け、次に段階希釈係数を掛けます。たとえば、データレポートで生成されたコピー/μLの濃度は966でした。反応液22 μLあたり5.5 μLのテンプレートを添加しました。サンプルの1:50,000段階希釈を利用しました。
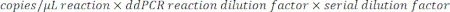
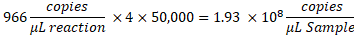
- 同じサンプルの複数の希釈がテストされた場合は、すべての有効な範囲内の希釈を分析し、平均を計算します。
- 各QCについて、所定のQC希釈液の濃度(コピー/μL)をddPCR反応量(20 μL)で割ることにより、予想されるコピー/μL PCR反応を計算します。これにより、この公称値を、追加の計算なしでデータレポートで提供されるコピー/μL値と直接比較できます。
注:このアプローチは、代表的な結果で利用されたスパイク涙液サンプルの分析にも使用されました。 - レプリケートウェルを使用して、平均値、標準偏差、変動係数(%CV)、およびサンプルの公称濃度(%RE)に対する相対誤差の割合(%RE)またはQC値を決定します(該当する場合は複数の希釈液を含めます)。
- ウェル間精度の評価では、ウェルの重複(含まれている場合)ごとにこれを決定します。
- アッセイ内の精度と精度を評価するには、バッチ内で使用する希釈シリーズまたはアリコートごとにこれを決定します。
- アッセイ間の精度と精度を評価するには、含まれている各バッチのアッセイ内平均を使用してこれを決定します。
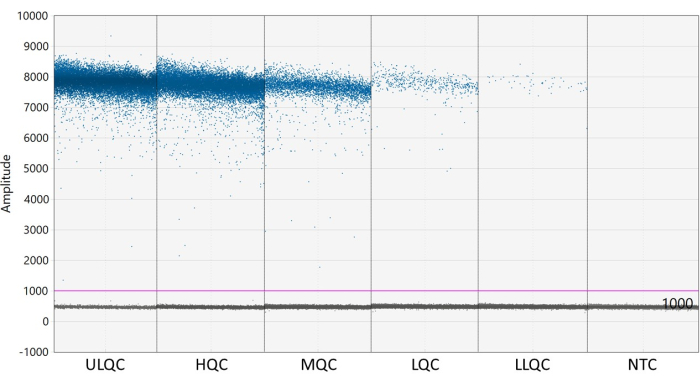
図2:しきい値の設定例 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この図の拡大版を表示するには、ここをクリックしてください。
10.アッセイの合格基準
- 各バッチの計算データに次の仕様を使用して、バッチが許容可能かどうかを判断します。これらの条件が満たされない場合は、バッチを無効にして繰り返します。
注:これらの基準は、PCRベースのアッセイバリデーション18、19、20、21、22、23、24、25に関する公開されたホワイトペーパーからのコンセンサスとして決定されました。臨床応用に応じてターゲット基準を変更する必要があるかもしれません。 - テンプレート コントロールなし (NTC)
- 各NTCウェルに少なくとも10,000個の液滴があることを確認します。
- 各NTCウェルの正の液滴が3つ未満であることを確認します。
- QC およびアッセイ範囲
- 各QCウェルに少なくとも10,000個の液滴があることを確認します。
- QC濃度の複製ウェルの精度が≤25.0%CVであることを確認してください(定量の上限と下限を除いて、≤30.0%が許容範囲です)。これをQCセットと濃度レベルごとに個別に評価します。
- 各平均QCレベルでの逆算濃度の相対誤差が、±30.0%REが許容される定量の上限と下限を除いて、公称濃度(コピー/PCR反応)の±25.0%RE以内であることを確認してください。これをQCセットと濃度レベルごとに個別に評価します。
- QCサンプルの少なくとも2/3(たとえば、6つの結果のうち4つ)および各レベル(低、中、高)のQCサンプルの50%がこれらのガイドラインを満たしていることを確認してください。
- サンプル
- 分析するサンプルウェルに少なくとも10,000個の液滴があることを確認してください。
- 分析するサンプル希釈液の複製ウェルの精度が≤25.0%CVであることを確認してください。
- 所定のサンプルの少なくとも1つの含まれる希釈液が、上限および下限QCに基づいて上記で定義されたアッセイの定義された定量範囲内にあることを確認してください。
- 含まれるすべての希釈液で定義された定量上限を超える結果が得られ、十分なサンプル量が残っている場合は、サンプルのより高い希釈率を使用してアッセイを繰り返します。
- 含まれるすべての希釈液で定量下限よりも低い結果が得られ、十分なサンプル量が残っている場合は、サンプルの下限希釈率を使用してアッセイを繰り返します。
注:3つ以上の陽性液滴を含むが、濃度が定量下限を下回るサンプルは、検出可能と説明できますが、定量化できません。
結果
実証目的のために、品質管理としてeGFPを含む合成二本鎖DNA断片を用いて、市販の増強緑色蛍光タンパク質(eGFP)発現AAV2ベクターを検出するように設計されたアッセイが開発されました。現在、ベクター自体、合成DNA断片、直鎖化プラスミドのどちらがQCとしての使用に最も適しているかについて議論が続いています。一般に、合成DNA断片または直鎖化プラスミドは、ベクターとの同等性がメソッド開発において実証される場合に使用され得る(データは示されていない)。プライマーおよびプローブは、eGFP導入遺伝子を検出するように設計および最適化した。本作業で用いた配列については別 表S1 を参照されたい。QCフラグメントストックの濃度は、ddPCRを用いて経験的に決定した。全てのアッセイは、プロトコールの項に例として挙げた濃度及びPCR条件を用いて実施した。
qPCRアッセイでは、検量線の直線性、感度、ダイナミックレンジ、精度、精度を評価することをお勧めします。ddPCRはターゲット定量に標準曲線に依存しないため、これらの推奨事項を変更する必要があります。代わりに、ポアソン統計モデリングに基づくddPCR反応の予想される定量可能範囲をカバーするようにさまざまな濃度に希釈された合成二本鎖DNA断片からなるQCが、ダイナミックレンジと感度を定義し、精度と精度を評価するために利用されました29、30、31、32。QC濃度の選択は、主に、所与の濃度におけるウェル内の全液滴に対する陽性の予想される比率に基づいていた。数学的には、ddPCRは、パーティションの約80%が正に増幅するときに理論的に最も正確です。総液滴に対する正の比率が0.8を超えると、分配の飽和により精度が低下し、液滴の100%が正になると定量化できなくなります。ローエンドでは、理論的には、わずか1つの陽性液滴を検出および定量できますが、精度は低く、アッセイは低レベルの偽陽性の影響を受けます。通常、結果が95%の信頼度で計算されるためには、少なくとも3つの液滴が正でなければなりません(これは、ここで使用した濃度を計算するためのしきい値です)。
一連の5つの異なるQC濃度を調製し、目標コピー数/μLのPCR反応量を計算して、 表5に示すように、ddPCRの定量可能な範囲にまたがる総液滴比に対して陽性が得られると予想しました。これらを用いて、アッセイの精度および精度を評価した。ここでの評価では、定量の上限と下限は、ddPCRで可能な理論上の最大値に押し上げられませんでした。正確な定量は、ここで示すよりも高いレベルと低いレベルで可能かもしれません。範囲は、この方法の下流のアプリケーションに合わせて開発する必要があります。
これらのQCの合計3つの独立して調製された希釈シリーズを、アッセイ内精度および精度を評価するために、各バッチのサンプル希釈バッファー中で調製した。各QC希釈液の重複ウェルを含めた。実際の検証プロトコルをシミュレートするために、複数のアナリストが数日間にわたって合計6つの精度と精度のバッチを実行しました。これらの6つのバッチの結果を分析して、アッセイ内およびアッセイ間の精度と精度を定義し、アッセイのダイナミックレンジを定義しました。
アッセイ内性能は、各QCレベルで各バッチについて評価した。すべてのQCおよびNTCウェルには少なくとも10,000個の液滴があると予想していました。これは、6つのバッチすべてでテストされた216ウェルのうち216ウェルで満たされ、平均液滴数は19,748液滴/ウェルでした(表6)。次に、各QCの重複ウェルの各セットのウェル間%CVは、≤30.0%が予想された上限および下限QCを除いて、≤25.0%であると予想された。これは、QCの6つのバッチすべてでテストされた90ウェルのうち90セットで満たされ、すべてのQCレベルで平均ウェル間%CVが3.9%でした(表7)。すべてのQCは、上記で概説した予想される範囲内で、総液滴比に対して平均正を示しました(表6)。
各バッチ内で、独立して調製された希釈系列ポイントのそれぞれについてアッセイ内平均および標準偏差を計算し、これらを使用して各アッセイにおける各濃度のアッセイ内平均を計算した。これを、アッセイの精度および精度を評価するために使用した(表8)。精度とは、通常のアッセイ条件下での同じ均質サンプルの反復からのデータの変動性を指し、複数の含まれるアリコートの%CVを計算することによって評価されます。各バッチ内でテストされた3つのアリコートは、≤30.0%が予想される上限および下限QCを除いて、アッセイ内%CV ≤25.0%をもたらすと予想しました。これは、60バッチのそれぞれ(合計30のパフォーマンスのうち30)の5つのQCレベルすべてで満たされました。一般に、目標基準よりも高いアッセイ内精度を達成でき、すべてのQCレベルで平均アッセイ内%CVは7.7%でした。精度とは、実験的に決定された値と公称値との間の一致の近さを指します。これは、各QCの計算された濃度と理論的に予想される公称濃度との間の相対誤差(%RE、または%Bias)を計算することによって評価されます。3つのアリコートのアッセイ内平均は、±30.0%が予想される上限および下限QCを除いて、公称濃度の±25.0%REになると予想されました。これは、60バッチのそれぞれ(合計30のパフォーマンスのうち30)の5つのQCレベルすべてで満たされました。一般的に、目標よりも高いアッセイ内精度を達成でき、すべてのQCレベルで平均絶対アッセイ内%REは4.2%でした。NTCのすべてのパフォーマンス(合計30)で、陽性の飛沫は検出されませんでした。
アッセイ間の精度および精度も、各バッチ内の各QCレベルのアッセイ内平均を用いて計算した。アッセイ間の精度は≤25.0%CVと予想されたが、上限QCと下限QCは≤30.0%と予想された。同様に、アッセイ間の精度については、±±25.0%のREが予想されましたが、30.0%が期待された上限および下限QCは例外でした。これらのターゲットよりも有意に高いアッセイ間精度および精度が観察され(表9)、アッセイ間精度は4.0%から8.5%の範囲、アッセイ間絶対精度は1.0%から3.2%の範囲でした。まとめると、これらの結果は、この方法が現在の業界目標の範囲内で十分なアッセイ内およびアッセイ間の精度と精度を達成できることを示しています。PCR反応1μLあたり2,500〜2.5コピーのこのアッセイのダイナミックレンジは、これらの結果に基づいて定義でき、全体的なアッセイ感度はPCR反応1μLあたり2.5コピーです。前述のように、より広いダイナミックレンジを検証できる場合があります。
次に、アッセイの精度とターゲットマトリックス内の精度(この場合は涙)を評価する必要がありました。通常、アッセイは臨床試験の開始前に検証されるため、ベクター治療を受けた患者から採取した涙液が検証目的で利用される可能性は低いです。これは、ターゲットAAVベクターをボランティアドナーから収集した涙にスパイクしてマトリックススパイクQCを作成することによって人工的に作成できます。 プールされた人間の涙は、第三者(BioIVT)によって収集されました。原理の証明のために、市販のソースから取得したeGFP発現AAV2ベクターを利用しました。AAV2ベクターストックの濃度は、このプロトコルに記載されているように、DNA単離ステップを使用せずに、ddPCRを使用して経験的に決定しました。各ランで、AAV2は、高レベル(予想される1.41 x 10 3コピー/μLPCR反応)および低レベル(28.2コピー/μLPCR反応)で3 つの涙液アリコートに独立して添加されました。非スパイクアリコートを、方法の特異性を実証するための対照として含めた。
アッセイ内性能は、各スパイクレベルで各バッチについて評価した。すべての涙液サンプルには少なくとも10,000個の液滴があると予想されていました。これは、6つのバッチすべてでテストされた108ウェルのうち108ウェルで満たされ、平均総液滴数は20,208液滴/ウェルでした(表10)。次に、各QCの重複ウェルの各セットのウェル間%CVは、高スパイクレベルと低スパイクレベルで≤25.0%であると予想されました。これは、QCの6つのバッチすべてでテストされた36セットのウェルのうち36セットで満たされ、平均ウェル間%CVは3.2%でした(表11)。
各バッチ内で、独立して調製された涙液スパイクのそれぞれについてアッセイ内平均および標準偏差を計算し、これらを使用して、各アッセイにおける各濃度のアッセイ内平均を計算した。これを、マトリックス中のアッセイの精度および精度を評価するために使用した(表12)。アッセイ内%CVは≤25.0%で、スパイクレベルが高いと低いと予想した。これは、各レベルの6つのバッチのうち6つで満たされました。一般に、マトリックス中のアッセイ内精度は目標よりも高く、平均アッセイ内%CVは高レベルで3.7%、低レベルで12.2%(全体で8.0%)でした。また、アッセイ内%REは両方のスパイクレベルで±25.0%になると予想されました。これは、各レベルの6つのバッチのうち6つで満たされました。同様に、マトリックスのアッセイ内精度は目標よりも高く、アッセイ内平均絶対%REは低レベルで8.1%、高レベルで11.3%(全体で9.7%)であることが一般的にわかりました。非スパイク対照については、いずれのアリコートにおいてもeGFPシグナルは検出されず(表12)、ヒト涙液マトリックスにおける本方法の特異性を実証した。
アッセイ間精度および涙液マトリックスの精度も、各バッチ内の各スパイクレベルのアッセイ内平均を使用して計算した。アッセイ間精度は≤25.0%CVと予想され、アッセイ間精度は±25.0%REと予想した。これらのターゲットよりも有意に高いアッセイ間精度および精度が観察され(表13)、アッセイ間精度は高レベルで5.5%、低レベルで7.1%、絶対アッセイ間精度は高レベルで11.3%、低レベルで8.1%でした。まとめると、これらの結果は、涙液マトリックスにおける方法の精度、精度、および特異性を示しています。
表5:アッセイのダイナミックレンジを定義するために使用される品質管理。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
表6:合成二本鎖DNA品質管理およびNTCの総液滴数および総液滴に対する正の比率。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
表7:QCウェル間統計(コピーターゲット/μL PCR反応)。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
表8:アッセイ内の精度とQCの精度(コピーターゲット/μL PCR反応)。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
表9:アッセイ間の精度とQC(コピーターゲット/μL PCR反応)の精度。 略語:ULQC =上限品質管理;HQC = 高品質管理;MQC = 中程度の品質管理;LQC = 低品質管理;LLQC = 品質管理の下限;NTC = テンプレート コントロールなし。 この表をダウンロードするには、ここをクリックしてください。
表10:涙液サンプルの総液滴数。この表をダウンロードするには、ここをクリックしてください。
表11:涙液サンプルウェル間統計(コピーターゲット/μL PCR反応)。この表をダウンロードするには、ここをクリックしてください。
表12:涙液サンプルのアッセイ内精度および精度(コピーターゲット/μL PCR反応)。この表をダウンロードするには、ここをクリックしてください。
表13:アッセイ間の精度と涙液サンプルの精度(コピーターゲット/μL PCR反応)。この表をダウンロードするには、ここをクリックしてください。
補足表S1:この研究で利用されたプライマー、プローブ、および合成二本鎖DNA品質管理の配列。この表をダウンロードするには、ここをクリックしてください。
ディスカッション
ddPCRプロトコルには、アッセイの適切な性能に不可欠ないくつかのステップがあります。最初の重要なステップは、プライマーとプローブの設計と最適化です。一般に、前臨床または臨床設定では、色素ベースの化学(SYBR Greenなど)よりも加水分解プローブベースの化学を使用することが、その優れた特異性のために推奨されます。さらに、増幅ターゲットの選択も重要です。典型的には、ベクターの目的の導入遺伝子が標的化される。ただし、ベクター導入遺伝子とゲノムDNAを区別できない可能性のある初期の前臨床段階またはベクターでは、標準化されたベクターターゲットを使用することが適切な場合があります。例えば、逆末端リピート領域、プロモーター、poly-Aテール、またはこれらのベクター成分間のセグメント間接合部を標的とすることができる。ターゲットの選択は、ベクターの設計によって異なります。従来の qPCR プライマーおよびプローブ設計戦略とソフトウェアは、通常、ddPCR に適しています。必要な最適化の量を減らすために、一貫したアニーリング温度(たとえば、60°C)が得られると予想される設計パラメータを選択する必要があります。また、ターゲットごとに少なくとも3つの異なるセットを設計、注文、および評価することも推奨されています。次に、最大の特異性(ネガティブコントロールウェルまたは関連する標的DNAのマトリックスで増幅されない)と感度(つまり、検出限界)を示すセットを選択する必要があります20。
qPCRとddPCRの間でアッセイを移行できることが有利な場合は、最初にqPCRを使用してアッセイ条件を最適化し、R2≥0.98で90%〜110%の増幅効率をもたらす選択されたセットの条件を特定することが推奨されます。ただし、エンドポイントメソッドとしてのddPCRは、増幅効率にばらつきがあるため、通常、qPCRよりも感度が低くなります。少なくとも、アニーリング/伸長ステップで熱温度勾配を実行して、予想されるアニーリング温度の上下の温度をカバーし、負と正の液滴クラスター間の雨と蛍光の振幅分離を温度の関数として評価することをお勧めします。ワークスペースで許可されている場合は、マスターミックスの準備、テンプレートの追加、および増幅のために個別の専用ワークステーションを用意することをお勧めします。可能であれば、これらは、制御されたアクセスや差圧などのエンジニアリング制御が組み込まれた一方向ワークフローによって物理的に分離され、クロスコンタミネーションや誤検知のリスクを軽減する必要があります。これが不可能な場合は、クロスコンタミネーションを防ぐために細心の注意を払う必要があります。
このプロトコルには、qPCRアッセイの開発に慣れている人には珍しいように見える2つのステップがあります。1つ目は、PCRマスターミックスに制限酵素を含めることです。ddPCR増幅中、各液滴はエンドポイントまでサーモサイクルされます。適切に最適化されたアッセイでは、これにより2つの液滴集団が得られ、1つのセットは一貫して高レベルの蛍光シグナル(ポジティブ)を示し、もう1つのセットは一貫して低レベルの蛍光シグナル(ネガティブ)を示します。PCR干渉が発生すると、PCR増幅の開始が非同期化し、液滴が増幅プラトーに到達せず、蛍光エンドポイントに一貫性がなくなる可能性があります。この場合、液滴はネガティブとポジティブの間に分布し、ddPCRレインと呼ばれる現象が発生します。これにより、ターゲットの定量化が不正確になり、一貫性のない主観的なしきい値が適用される可能性があります。閾値をNTCの信号よりわずかに高く設定することを推奨し、エンドポイントまで完全に循環していなくても、すべての液滴が陽性と見なされるため、最終的な定量化における雨の影響を最小限に抑える必要があります。AAVは非常に複雑な二次構造を有しており、増幅ターゲットによっては、プライマーやプローブへのアクセス性が低下し、PCR干渉や雨が発生する可能性があります。マスターミックスに制限酵素を含めると、この二次構造が切断され、プライマーとプローブによるアクセスが増加し、雨が減少し、アッセイの精度が向上する可能性があります。ddPCR反応に制限酵素を含めることの効果は、以前に25、32に記載されている。標的増幅領域内で切断しないことが確認される限り、任意の制限酵素を使用することができる。前消化ステップや代替バッファー組成物は必要ありません。
2番目の珍しいステップは、AAVを含む涙液サンプルの調製です。このプロトコルでは、1:10(またはそれ以上)の涙の比率を利用し、続いてサンプルを加熱した。通常、広く利用されている収集方法であるキャピラリーチューブを介して涙を採取する場合、平均して約10.0μLを採取できます33。希釈は、限られたサンプル量に対処し、重複した井戸試験に十分な材料を提供するのに役立ちます。これにより、検出の理論上の限界は減少しますが、ddPCRの堅牢な感度により、非常に少数のベクター粒子を除くすべてのベクトル粒子を検出できるはずです。このアプローチでは、予期せず障害が発生した場合に「バックアップ」ウェルを追加で作成します。この場合、または2つのウェルを実行するのに十分なサンプル量がない場合は、ポアソン誤差を使用して精度を評価できます。さらに、濃度が検出限界を下回っている場合は、井戸データをマージして濃度を決定する機会が生まれます。ddPCR検出のためには、ウイルスカプシドからAAVベクターを遊離させる必要があります。AAVの定量のためのいくつかの方法は、ウイルスキャプシド34、35、36を除去するためのプロテイナーゼK消化ステップを含んでいる。すべての天然に存在するAAV血清型の融解温度は約90°C以下で、ほとんどが80°Cを下回ります。したがって、これは不必要な包含37であると思われる。加熱だけでベクターDNAを放出するのに十分であるように思われる。
さらに、ddPCRは一般に、qPCRアッセイに影響を与える可能性のあるサンプル中に存在する可能性のあるPCR阻害剤の影響を受けにくい。特定のDNA単離ステップが含まれる場合、これには特定の検証も必要になりますが、このプロトコルでは回避されます。サンプルは、液体中でのベクターゲノムの拡散の速度論のために加熱前に希釈される。加熱およびその後の冷却プロセス中に、一本鎖DNAゲノムの正および負のセンス鎖は、濃度が十分に高い場合、一緒にアニーリングして二本鎖中間体を生成することができる。加熱前に希釈すると濃度が低下し、定量の精度に悪影響を与えるのに十分な二本鎖中間体が形成されることが数学的に低くなります。品質管理として使用される合成DNA断片または直鎖化プラスミドは、この加熱ステップを経てはならないことに留意すべきである。これらは二本鎖であるため、加熱すると一本鎖中間体に変換されます。これらの一本鎖QCを液滴に独立して分割した後、これは公称濃度と比較してQC濃度の2倍の増加をもたらすと予想されます。あるいは、QCを加熱して方法を標準化する場合は、これを再構成と公称濃度の割り当てに考慮する必要があります。
最後に、サンプル調製に関して、多くのプロトコルでは、カプセル化されていないベクターDNAを除去するためのDNase処理ステップを含めることも推奨されています。このステップは、ベクター調製に関連する遊離DNAを定量することが望ましくない場合(投薬目的の定量中など)に重要です。しかし、生体内分布およびバイオシェディング研究の文脈では、通常、ベクターDNAがカプセル化されているかどうかに関係なく、ベクターDNAがどこに移動したかを知りたいと思う。したがって、通常、このような研究中にDNase処理ステップを実行しないことが示唆されます。DNaseステップを含める必要があると判断された場合、このステップは希釈および加熱の前に行う必要があります。
この論文では、目的に合った優れたラボプラクティスに準拠した検証のコンテキスト内でメソッドのダイナミックレンジ、感度、精度、および精度を評価するためのアプローチを代表するデータを紹介します。現在、このトピックに関するガイダンスが不足しているため、検証ラボは、現在の業界の考え方に沿って、ターゲットアッセイ基準を独自に決定する必要があります。異なるグループは、この研究で使用されたよりも高いターゲット基準と低いターゲット基準の両方を提起しました19、20、21、22、23、24、25。標的アッセイ基準は、より厳密に定義されるまで、本方法の意図された臨床用途に基づいてバリデーションの前に選択されるべきである。データに基づいて下される下流の決定によっては、より高いレベルの精度と精度が必要になる場合があります。逆に、単純な正と負の結果で十分な場合があります。
このアプローチでは、特異性とマトリックス効果の評価に関する推奨事項にも対処しました。未治療の個人から収集された涙のプールは、このアッセイで肯定的な結果をもたらさなかったが、ベクターが推奨される回収率の範囲内で高濃度および低濃度で涙液にスパイクされたときに標的を検出できた。理想的には、内因性ベクターを含むマトリックス(例えば、ウイルスベクターによる処理後に収集)もこれらの評価に含まれるであろう。ただし、このようなサンプルを検証に使用できる可能性はほとんどありません。検証の頑健性を高めるために、複数の涙液のプール、またはさまざまな個人から収集された涙を評価して、患者固有のマトリックス効果が発生するかどうかを判断できます。最後に、安定性を評価することをお勧めします。生物学的マトリックスからDNAを抽出するワークフローでは、サンプルと抽出されたDNAの両方の安定性を評価する必要がある場合があります。このワークフローでは、サンプルはDNA抽出を必要とせずにアッセイで直接テストされます。したがって、この方法に対する安定性の評価を考慮して、涙液サンプルの安定性を評価しなければならない。通常、ベンチトップ、冷蔵庫、凍結/解凍、および長期安定性評価が推奨されます。これらはこの研究の一部として実行されていませんが、ここで開発された方法は、入力サンプルの操作に続いて、この評価で使用できます。
全体として、この方法は、涙液サンプル中のAAVベースのベクターを検出するための堅牢で再現性があり、検証可能なアッセイであることが実証されています。これは、臨床試験をサポートするために特定のベクターに適応するためのプラットフォームとして機能し、優れたラボプラクティスと一致するアッセイの検証の基礎を提供します。
開示事項
すべての著者は、このプロトコルに記載されているように、AAVベースのアッセイのサポートを含む、早期発見サポートから製品登録までの包括的なグッドラボラトリープラクティス/グッドクリニカルプラクティスに準拠した開発サービスを提供する受託研究機関であるKCASバイオアナリシスおよびバイオマーカーサービスの従業員です。KCASはこのプロジェクトとこのプロトコルの出版に資金を提供しました。この論文で提示された実験の設計と検証に関する現在のFDAガイドラインはありませんが、KCASでそのような研究を開発している専門家とソートリーダーは、ベースラインアプローチとしてこのアプローチを採用しています。追加の基準とパラメーターは、プロジェクトごとに説明され、使用目的に基づいて含まれる場合があります。
謝辞
バイオ・ラッドのニック・ラッセル氏とブランドン・マッケサン氏には、この方法の開発中に有益な議論をしていただき、ありがとうございました。
資料
| Name | Company | Catalog Number | Comments |
| AAV-eGFP Vector | Charles River Laboratories | RS-AAV2-FL | Lot AAV2-0720-FL, used as a proof of principle vector |
| AutoDG Droplet Digital PCR system | Bio-Rad | QX200 | Alternative ddPCR system may be used following manufacturer’s protocol. |
| AutoDG Oil for Probes | Bio-Rad | 1864110 | Or use material compatible with ddPCR system. |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | Or use material compatible with ddPCR system and PCR chemistry. |
| ddPCR Droplet Reader Oil | Bio-Rad | 1863004 | Or use material compatible with ddPCR system. |
| ddPCR Piercable Foil Seals | Bio-Rad | 1814040 | Or use material compatible with ddPCR system. |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad | 12001925 | Or use material compatible with ddPCR system. |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023, 1863024, or 1863025 | Use master mix compatible with primers/probes and ddPCR system. |
| DG32 AutoDG Cartidges | Bio-Rad | 1864108 | Or use material compatible with ddPCR system. |
| Droplet Reader | Bio-Rad | QX200 | Alternative ddPCR system may be used following manufacturer’s protocol. |
| GeneAmp PCR Buffer | Applied Biosystems | N8080129 | N/A |
| Nuclease-Free Water | Ambion | AM9906 | N/A |
| PCR Plate Sealer | Bio-Rad | PX1 | Or use material compatible with ddPCR system. |
| Pipet Tips for AutoDG | Bio-Rad | 1864120 | Or use material compatible with ddPCR system. |
| Pluronic F-68 Non-ionic Surfactant | Gibco | 24040 | N/A |
| Primer and Hydrolysis Probes | Various | Various | Design based on target sequence using general approaches for primer/probe design. Select fluorphores and quenchers compatible with ddPCR system. |
| Restriction Enzyme | Various | Various | Varies with target amplification sequence. Use restriction enzyme that does not cut in the amplified sequence |
| Sheared salmon sperm DNA | ThermoFisher | AM9680 | N/A |
| Synthetic DNA gene fragment or linearized plasmid | Various | Various | Design a synthetic DNA fragment containing the target amplification region for use as a quality control |
| TE Buffer | Teknova | T0224 | Ensure prepared or purchases nuclease free. 10 mM Tris-HCl, 1.0 mM EDTA, pH=8.0 |
| Touch Thermal Cycler | Bio-Rad | C1000 | Or use material compatible with ddPCR system. |
参考文献
- Sherkow, J. S., Zettler, P. J., Greeley, H. T. Is it 'gene therapy. Journal of Law and the Biosciences. 5 (3), 786-793 (2018).
- Ginn, S. L., Amaya, A. K., Alexander, I. E., Edelstein, M., Abedi, M. R. Gene therapy clinical trials worldwide to 2017: an update. The Journal of Gene Medicine. 20 (5), 3015 (2018).
- Ghosh, S., Brown, A. M., Jenkins, C., Campbell, K. Viral vector systems for gene therapy: a comprehensive literature review of progress and biosafety challenges. Applied Biosafety. 25 (1), 7-18 (2020).
- Gene, Cell, & RNA therapy landscape, Q4 2022 quarterly data report. American Society for Gene & Cell Therapy Available from: https://asgct.org/global/documents/asgct_citeline-q4-2022-report_final.aspx (2022)
- Approved Cellular and Gene Therapy Products. United States Food and Drug Administration Available from: https://www.fda.gov/vaccines-blood-biologics/cellular-gene-therapy-products/approved-cellular-and-gene-therapy-products (2022)
- Au, H. K. E., Isalan, M., Meilcarek, M. Gene therapy advances: a meta-analysis of AAV usage in clinical settings. Frontiers in Medicine. 8, 809118 (2022).
- Lundstrom, K. Viral vectors in gene therapy. Diseases. 6 (2), 42 (2018).
- Naso, M. F., Tomkowicz, B., Perry, W. L., Strohl, W. R. Adeno-associated virus (AAV) as a vector for gene therapy. BioDrugs. 31, 317-334 (2017).
- Russell, S., et al. Efficacy and safety of voretigene neparvovec (AAV2-hRPE65v2) in patients with RPE65-mediated inherited retinal dystrophy: a randomized, controlled, open-label, phase 3 trial. Lancet. 390 (10097), 849-860 (2017).
- Chemistry, manufacturing, and control (CMC) information for human gene therapy investigational new drugs (INDs). United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/chemistry-manufacturing-and-control-cmc-information-human-gene-therapy-investigationsal-new-drug (2020)
- Long term follow-up after administration of human gene therapy products. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/long-term-follow-after-adminstration-human-gene-therapy-products (2020)
- Testing of retroviral vector-based human gene therapy products for replication competent retrovirus during product manufacture and patient follow-up. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/testing-retroviral-vector-based-human-gene-therapy-products-replication-competent-retrovirus-during (2020)
- Recommendations for microbial vectors used in gene therapy. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/recommendations-microbial-vectors-used-gene-therapy (2016)
- Multidisciplinary: gene therapy. European Medicines Agency Available from: https://www.europa.eu/en/human-regulatory-development/scientific-guidelines/multidisciplinary/multidisciplinary-gene-therapy (2023)
- Human gene therapy for retinal disorders. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/human-gene-therapy-retinal-disorders (2020)
- Design and analysis of shedding studies for virus or bacterial-based gene therapy and oncolytic products. United States Food and Drug Administration Available from: https://www.fda.gov/regulatory-information/search-fda-guidance-documents/design-and-analysis-shedding-studies-virus-or-bacteria-based-gene-therapy-and-oncolytic-products (2015)
- Guideline on the quality, non-clinical and clinical aspects of gene therapy medicinal products. European Medicines Agency Available from: https://www.ema.europa.eu/en/documents/scientific-guideline/guideline-quality-non-clinical-clinical-aspects-gene-therapy-medicinal-products_en.pdf (2018)
- Kaur, S. white paper on recent issues in bioanalysis: mass spec of proteins, extracellular vesicles, CRISPR, chiral assays, oligos; nanomedicines bioanalysis; ICH M10 section 7.1; non-liquid & rare matrices; regulatory inputs (part 1A - recommendations on endogenous compounds, small molecules, complex methods, regulated mass spec of large molecules, small molecule, PoC & part 1B - regulatory agencies' inputs on bioanalysis, biomarkers, immunogenicity, gene & cell therapy and vaccine). Bioanalysis. 14 (9), 505-580 (2022).
- Hays, A., Islam, R., Matys, K., Williams, D. Best practices in qPCR and qPCR validation in regulated bio analytical laboratories. The AAPS Journal. 24 (2), 36 (2022).
- Ma, H., Bell, K. N., Loker, R. N. qPCR and qRT-PCR analysis: Regulatory points to consider when conducting biodistribution and vector shedding studies. Molecular Therapy. Methods & Clinical Development. 17 (20), 152-168 (2020).
- Wissel, M. Recommendations on qPCR/ddPCR assay validation by GCC. Bioanalysis. 14 (12), 853-863 (2022).
- Expectations for biodistribution (BD) assessments for gene therapy (GT) products. International Pharmaceutical Regulators Programme Available from: https://admin.iprp.global/sites/default/files/2018-09/IPRP_GTWG_ReflectionPaper_BD_Final_2018_0713.pdf (2018)
- Pinheiro, L., Emslie, K. R. Basic concepts and validation of digital PCR measurements. Methods in Molecular Biology. 1768, 11-24 (2018).
- Tzonev, S. Fundamentals of counting statistics in digital PCR: I measured two target copies-what does it mean. Methods in Molecular Biology. 1768, 25-43 (2018).
- Prantner, A., Marr, D. Genome concentration, characterization, and integrity analysis of recombinant adeno-associated viral vectors using droplet digital PCR. PLoS One. 18, 0280242 (2023).
- Primer-BLAST. National Library of Medicine, National Center for Biotechnology Information Available from: https://www.ncbi.nim.nih.gov/tools/primer-blast/ (2023)
- Koressaar, T., Remm, M. Enhancements and modifications of primer design program Primer 3. Bioinformatics. 23 (10), 1289-1291 (2007).
- Ye, J. Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 13, 134 (2012).
- Droplet Digital PCR Application Guide. Bio-Rad Available from: https://www.bio-rad.com/webroot/we/pdf/lsr/literature/Bulletin_6407.pdf (2023)
- Qian, P. L., Sauzade, M., Brouzes, E. dPCR: A technology review. Sensors. 18 (4), 1271 (2018).
- Basu, A. Digital assays part I: portioning statistics and digital PCR. SLAS Technology. 22 (4), 369-386 (2017).
- Sanmiguel, J., Gao, G., Vandeberghe, L. H. Quantitative and digital droplet-based AAV genome titration. Methods in Molecular Biology. 1950, 51-83 (2019).
- Bachhuber, F., Huss, A., Senel, M., Tumani, H. Diagnostic biomarkers in tear fluid: from sampling to preanalytical processing. Scientific Reports. 11, 10064 (2021).
- Martinez-Fernandez de la Camara, C., McClements, M. E., MacLaren, R. E. Accurate quantification of AAV vector genomes by quantitative PCR. Genes. 12 (4), 601 (2021).
- Ai, J., Ibraheim, R., Tai, P. W. L., Gao, G. A scalable and accurate method for quantifying vector genomes of recombinant adeno-associated viruses in crude lysate. Human Gene TherapyMethods. 28 (3), 139-147 (2017).
- Dobnik, D., et al. Accurate quantification and characterization of adeno-associated viral vectors. Frontiers in Microbiology. 10, 1570 (2019).
- Bennett, A., et al. Thermal stability as a determinant of AAV serotype identity. Molecular Therapy Methods and Clinical Development. 6, 171-182 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved