需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
BrainBeats 作为开源 EEGLAB 插件,用于联合分析 EEG 和心血管信号
摘要
BrainBeats 工具箱是一个开源的 EEGLAB 插件,旨在联合分析 EEG 和心血管 (ECG/PPG) 信号。它包括心跳诱发电位 (HEP) 评估、基于特征的分析以及从 EEG 信号中提取心脏伪影。该协议将有助于通过两个镜头(HEP 和特征)研究脑心相互作用,从而提高可重复性和可及性。
摘要
大脑和心血管系统之间的相互作用因其在促进我们对人类生理学的理解和改善健康状况方面的潜力而受到越来越多的关注。然而,由于缺乏指南、标准化信号处理和统计工具、图形用户界面 (GUI) 以及用于处理大型数据集或提高可重复性的自动化,因此对这些信号进行多模态分析具有挑战性。标准化的 EEG 和心率变异性 (HRV) 特征提取方法还存在进一步的空白,破坏了临床诊断或机器学习 (ML) 模型的稳健性。为了应对这些限制,我们引入了 BrainBeats 工具箱。BrainBeats 作为开源 EEGLAB 插件实现,集成了三个主要协议:1) 心跳诱发电位 (HEP) 和振荡 (HEO),用于以毫秒级精度评估时间锁定的脑心相互作用;2) EEG 和 HRV 特征提取,用于检查各种大脑和心脏指标之间的关联/差异或构建强大的基于特征的 ML 模型;3) 在进行 EEG 分析时,从 EEG 信号中自动提取心脏伪影,以去除任何潜在的心血管污染。我们提供了一个分步教程,将这三种方法应用于包含同步 64 通道 EEG、ECG 和 PPG 信号的开源数据集。用户可以使用图形用户界面 (GUI) 或命令行轻松微调参数,以定制他们独特的研究需求。BrainBeats 应该使脑心相互作用研究更容易获得和可重复性。
引言
长期以来,还原论方法一直主导着人类生理学和认知学的科学研究。这种方法涉及将复杂的身体和心理过程分解成更小、更易于管理的组件,使研究人员能够孤立地关注单个系统。这种策略的出现是由于研究人类身心错综复杂和相互关联的本质所面临的挑战1。还原论有助于孤立地理解各个子系统,例如阐明离子通道和动作电位在神经2 或心脏3 通信中的作用。然而,我们对这些孤立的系统如何在更大的空间和时间尺度上相互作用的理解仍然存在重大差距。多模态(综合或生态)框架认为人体是一个复杂的多维系统,其中思想不被视为大脑的产物,而是生物的一种活动,一种将大脑整合到人体日常功能中的活动4。多模态和还原论方法并不排他性,就像我们不能在没有整个大脑的情况下研究一个神经元,或者在不了解单个神经元特性的情况下就无法研究整个大脑一样。它们共同为对人类健康、病理学、认知、心理学和意识的更全面、协同的理解铺平了道路。该方法旨在通过提供脑电图 (EEG) 和心血管信号的联合分析,即心电图 (ECG) 和光电容积脉搏波 (PPG),来简化大脑和心脏之间相互作用的多模态研究。该工具箱在 MATLAB 中作为 EEGLAB 插件实现,解决了现有的方法限制,并成为开源的,以促进科学领域的可访问性和可重复性。它将最新的准则和建议实施到其设计和默认参数中,以鼓励用户遵循已知的最佳实践。对于感兴趣的研究人员和临床医生来说,所提出的工具箱应该是宝贵的资源 1) 研究心跳诱发电位,2) 从 EEG 和 ECG/PPG 信号中提取特征,或 3) 从 EEG 信号中去除心脏伪影。
心脑研究
心脏和大脑之间的关系历来通过神经影像学方法进行研究,例如功能磁共振成像 (fMRI) 和正电子发射断层扫描 (PET)。使用这些工具,研究人员突出显示了一些与心血管控制相关的大脑区域(例如,心率和血压的操纵5),显示了心率对 BOLD 信号的影响6,或确定了导致冠心病的潜在脑身通路(即压力诱发的血压7).虽然这些研究极大地促进了我们对中枢神经系统 (CNS) 和心血管功能之间复杂相互作用的理解,但这些神经影像学技术价格昂贵、可用性有限,并且仅限于受控的实验室环境,这限制了它们在现实世界和大规模应用中的实用性。
相比之下,EEG 和 ECG/PPG 是更实惠和便携的工具,为在更多样化的环境和人群中或更长时间内研究脑心相互作用提供了潜力,提供了新的机会。ECG 通过放置在皮肤(通常位于胸部或手臂)上的电极测量心脏收缩和放松时每次心跳产生的电信号8。PPG 使用光源(例如 LED)和光电探测器(通常放置在指尖、手腕或前额)测量微血管组织中的血容量变化(即血流和脉搏率),具体取决于血液如何比周围组织吸收更多的光9。这两种方法都提供了有关心血管功能的宝贵信息,但用途不同,并提供不同的数据类型。与心电图一样,脑电图记录数千个皮层神经元同步活动产生的电场,这些神经元通过细胞外基质、组织、颅骨和头皮传播,直到到达放置在头皮表面的电极10。因此,脑电图和心电图/PPG 的使用对于促进我们对脑心相互作用背后的生理、认知和情感过程及其对人类健康和福祉的影响的理解具有巨大的前景。因此,使用 BrainBeats 工具箱从 EEG、ECG/PPG 信号捕获心脑相互作用可能对以下科学领域特别有用:临床诊断和预测、大数据机器学习 (ML)、真实世界自我监测11 和移动脑/身体成像 (MoBI)12,13。
联合分析 EEG 和 ECG 信号的两种方法
研究脑电图和心血管信号之间相互作用有两种主要方法:
时域中的心跳诱发电位 (HEP):事件相关电位 (ERP),时频域中的心跳诱发电位 (HEO):事件相关频谱扰动 (ERSP) 和试验间相干性 (ITC)。这种方法检查大脑如何处理每个心跳。以毫秒 (ms) 的精度,这种方法要求两个时间序列完全同步,并且心跳要在 EEG 信号中标记。这种方法近年来引起了人们的兴趣 14,15,16,17,18,19。
基于特征的方法:这种方法从连续信号中提取 EEG 和心率变异性 (HRV) 特征,并检查它们之间的关联。这是独立进行的 EEG(通常称为定量 EEG 或 qEEG20)、ECG21、22、23 和 PPG24、25、26。这种方法通过捕获与状态和特征相关的变量来提供有前途的应用。请注意,对于 EEG 和心血管信号,记录时间越长,性状变量 27,28,29 就越显性。因此,应用程序取决于记录参数。基于特征的分析越来越受到关注,为预测精神和神经系统疾病的发展、治疗反应或复发提供了新的定量指标 30,31,32,33,34,35。这种方法对于大型和真实世界的数据集(例如,诊所、远程监控)尤其引人注目,这要归功于可穿戴神经技术的最新创新11。一个较少探索的应用是识别特定大脑和心脏特征之间的关联,突出潜在的潜在中枢神经系统动力学。心率变异性 (HRV) 可以根据 ECG 和 PPG 信号计算。它通过测量心跳之间时间间隔的变化(即正常到正常的间隔)来提供有关自主神经系统 (ANS) 的信息27。交感神经 (SNS) 活动增加(例如,在压力或运动期间)通常会降低 HRV,而副交感神经 (PNS) 活动(例如,在放松期间)会增加 HRV。由于 PNS 活动增强,较慢的呼吸频率通常会增加 HRV,尤其是对于短记录(<10 分钟)27。较高的 HRV 分数通常表明 ANS 更具弹性和适应性,而较低的 HRV 可能表明压力、疲劳或潜在的健康问题。长时间的 HRV 记录(即至少 24 小时)为各种健康状况提供预测预后,包括心血管疾病、压力、焦虑和一些神经系统疾病27。血压、心率或胆固醇水平等指标提供有关心血管系统状态的信息。相比之下,HRV 增加了一个动态方面,显示心脏如何响应压力并从压力中恢复。
BrainBeats 相对于现有方法的优势
虽然存在工具,如下所述,可以彼此独立地处理心血管和 EEG 信号,但它们不能联合分析。此外,大多数处理心血管信号的可用方法都涉及昂贵的许可,不允许自动处理(尤其有利于大型数据集),具有阻止透明度和可重复性的专有算法,或者由于不提供图形用户界面 (GUI) 而需要高级编程技能36。据我们所知,四个开源 MATLAB 工具箱支持使用 GUI 进行 HEP/HEO 分析:ecg-kit 工具箱37、BeMoBIL 管道38、HEPLAB EEGLAB 插件39 和 CARE-rCortex 工具箱40。虽然 HEPLAB、BeMoBIL 和 ecg-kit 通过检测心跳并在 EEG 信号中标记它们来促进 HEP 分析,但它们不提供统计分析或仅限于时域(即 HEP)。CARE-rCortex 插件通过支持 ECG 和呼吸信号、时频域分析、统计以及适用于 HEP/HEO 分析的高级基线归一化和校正方法来解决这些问题。然而,它使用 Bonferroni 方法对 1 型错误(即假阳性)进行统计校正,这对于 EEG 应用来说过于保守且生理上不合理,导致 II 型错误(即假阴性)增加41。此外,该工具箱不提供用于自动化的命令行访问。最后,最近的研究建议不要使用基线校正方法 42,43,44,因为它们会降低信噪比 (SNR),并且在统计上是不必要的和不可取的。
为了解决这些限制,我们引入了 BrainBeats 工具箱,目前在 MATLAB 环境中作为开源 EEGLAB 插件实现。与以前的方法相比,它具有以下优点:
1) 易于使用的 GUI 和命令行功能(适用于旨在执行自动化处理的程序员)。2) 经过验证的算法、参数和处理心血管信号的指南,例如检测 R 峰、插值 RR 伪影和计算 HRV 指标(例如,用于窗口化、重采样、归一化等的植入指南27,45,46)。这很重要,因为 Vest 等人证明了这些处理步骤中的适度差异如何导致不同的结果,从而导致 HRV 指标缺乏可重复性和临床适用性46。3) 经过验证的算法、默认参数和处理脑电信号的指南,包括过滤和窗口化44,47,重新引用48,49,去除异常通道和伪影 50,51,52,优化的 ICA 分解和独立组件的分类 53,54,55,56.在使用工具箱满足他们的需求之前,用户可以微调所有预处理参数,甚至使用他们喜欢的方法预处理他们的 EEG 数据(例如,使用 EEGLAB clean_rawdata 插件50,52、BeMoBIL 管道38、PREP 管道57 等)。4) 心电图信号的心跳诱发电位(HEP,即时域)和振荡(HEO;使用小波或 FFT 方法的事件相关频谱扰动,以及试验间相干性可通过标准 EEGLAB 软件获得)。通过 EEGLAB 的标准软件可获得带有 1 类误差校正的参数和非参数统计。非参数统计包括排列统计和多重比较的时空校正(例如,时空聚类或无阈值聚类增强)58,59。用户可以使用 LIMO-EEG 插件来实现分层线性建模,该模型很好地考虑了受试者内和受试者间的方差,并实施了无假设的质量单变量方法,对 I 型和 II 型误差进行了稳健控制60,61。HEP/HEO 数据统计分析可以在通道域和独立组件域中进行。5) 来自 PPG 信号的 HEP/HEO 和 HRV 分析(首次用于 HEP/HEO)。6) 首次支持脑电和 HRV 特征的联合提取。7) 该工具箱提供各种数据可视化,以检查各种必要处理步骤中的信号,并在主题级别进行输出。
| 方法 | 从 ECG 中检测 R 峰 | 检测 PPG 的 R 波 | HEP/HEO 系列 | EEG & HRV 特性 | 从 EEG 中去除心脏伪影 | 图形用户界面 | 命令行 |
| 心电图套件 | X | X | X | X | |||
| BeMoBIL 贝莫比尔 | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CARE-rCortex 皮层 | X | X | X | X | |||
| 脑跳 | X | X | X | X | X | X | X |
表 1:BrainBeats 相对于先前存在的类似方法带来的新颖性。
帮助读者确定该方法是否适合他们的信息
该工具箱适用于任何拥有 EEG 和 ECG/PPG 数据的研究人员或临床医生。该插件尚不支持从单独的文件导入 EEG 和 ECG/PPG 信号(尽管此功能将很快推出)。该工具箱适用于任何旨在执行 HEP/HEO 分析、使用标准化方法提取 EEG 和/或 HRV 特征或简单地从 EEG 信号中去除心脏伪影的任何人。图 1 中总结了 BrainBeats 的整体流程和方法的框图。
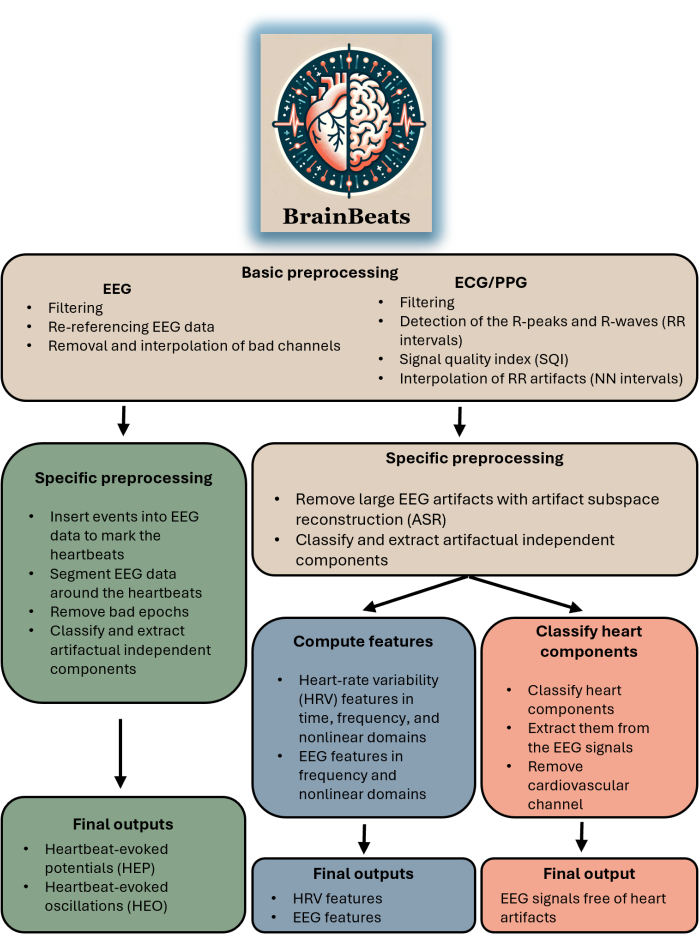
图 1.总结 BrainBeats 整体架构和流程的框图。这三种方法中常见的运算是棕色的。特定于心跳诱发电位 (HEP) 和振荡 (HEO) 的操作为绿色。特定于提取 EEG 和 HRV 特征的操作为蓝色。专门用于从 EEG 信号中去除心脏伪影的操作为红色。 请单击此处查看此图的较大版本。
研究方案
获得每个参与者的知情同意,乌拉尔联邦大学伦理委员会批准了实验方案。
1. BrainBeats 要求
- 在计算机上安装 MATLAB 和 EEGLAB。EEGLAB 可以在 https://github.com/sccn/eeglab 下载并解压缩(或为 Git 用户克隆)计算机上的任何位置。有关安装的更多详细信息,请参阅 GitHub 页面。
- 通过单击 Set Path 按钮,将路径添加到 MATLAB 主面板中的 EEGLAB 文件夹中。选择包含解压缩文件的 eeglab 文件夹,然后单击 Save > Close。
- 通过在 MATLAB 的命令窗口中键入 eeglab 来启动 EEGLAB。
- 通过单击安装 BrainBeats 插件 文件 > 管理 EEGLAB 扩展.在搜索栏中键入 brainbeats ,在列表中选择 BrainBeats 插件,然后单击 安装/更新。
- 将示例数据集加载到 EEGLAB 中。单击 File > Load existing data。导航到 EEGLAB 文件夹,转到 plugins 文件夹,转到 BrainBeats 文件夹并打开 sample_data 文件夹。选择文件 dataset.set。
注意:该数据集对应于开源多主题数据集62,63 中的 sub-032(睁开眼睛的静息状态)。之所以选择它,是因为它以 1000 Hz 的采样率同时包含脑电图(64 个通道)、心电图(一个通道)和 PPG(一个通道)。使用 actiCHamp 系统的辅助输入收集 ECG 和 PPG 信号。脑电数据是用根据 10-20 系统放置的有源电极记录的,以 FCz 作为在线参考,Fpz 作为接地电极,阻抗保持在 25 kOm 以下。使用放置在右手腕上的一个有源电极、左手腕上的参比电极和距肘部 3 cm 处左前臂内侧的地面记录心电图。PPG 是从左手食指记录的。EEG、ECG 和 PPG 数据是时间同步的,因为它们是用同一个放大器同时记录的。有关更多详细信息,请参阅参考文献62,63。 - 选中 Save Outputs 复选框,将相应 .set 文件中的所有内容保存在与加载到 EEGLAB 中的原始文件相同的位置。
注意:在本教程中,我们将 EEG、ECG 和 PPG 数据合并到一个 EEGLAB 数据集中,加载 3D 边界元法 (BEM) 电极坐标,并将信号下采样至 250 Hz,以减少文件重量(供用户下载)并加快计算时间。由于该数据集没有异常的脑电通道,我们人为修改了通道 TP9 来说明 BrainBeats 的坏通道检测和去除算法。同样,我们在文件开头人为模拟了一个大的脑电图伪影,并在 3-6 秒的颞通道中模拟了一个高频肌肉状伪影,以说明 BrainBeats 的伪影去除功能。
2. 心跳诱发电位 (HEP) 和振荡 (HEO)
- 打开 BrainBeats 的第一个通用用户界面 (GUI) 以选择主要参数。在 EEGLAB 中,单击 Tools > BrainBeats > 1st level (subject level)”。选择 Heartbeat-evoked potentials HEP 作为要运行的分析,选择 ECG 作为心脏数据类型,然后单击按钮以显示通道列表,以选择标记为 ECG 的 ECG 通道(或直接在文本框中键入)。保持选项 可视化输出(推荐) 和 保存输出 选中,然后单击 确定。
- 根据之前的选择(即,使用 ECG 数据进行 HEP 分析)弹出第二个 GUI 窗口。选择 EEG 和 ECG 信号的预处理参数。通过取消选中 Preprocess ECG (预处理 ECG) 和 Preprocess EEG (预处理脑电图) 复选框来关闭预处理,以查看在启动 BrainBeats 之前是否已经对数据进行了预处理。在预处理 EEG 部分将电源线噪声更改为 50 Hz,因为这些 EEG 数据是在俄罗斯记录的。单击 OK 启动。
- 将出现一条警告消息,要求确认删除已检测到的 PPG 通道。这是因为该工具箱并非旨在同时分析 ECG 和 PPG(或其他辅助通道),将其保留在数据集中会导致严重错误(例如,伪影去除、ICA 分解不良等)。单击 Yes (是)。BrainBeats 开始执行一些检查,设置一些默认参数,并将 ECG 与 EEG 数据分离,以预处理 ECG 信号并计算 RR 间期。
- ECG 和 RR 时间序列使用 Physionet 心血管信号工具箱46,64 中经过验证的算法进行预处理,以适应 BrainBeats 的数据格式、提高清晰度、参数调整和计算时间(参见参考文献了解算法的验证)。该插件输出 RR 间期、时间戳、过滤后的 ECG 信号、R 峰值指数和心率 (HR)。通过 GUI 或命令行调整这些参数。
注意:ECG 信号使用定制的、经过验证的滤波器 (1-30 Hz) 进行带通过滤,并使用 Pan-Tompkins (P&T) 方法65 扫描信号以识别 QRS 波群和 R 峰,实施一些信号处理操作,包括微分、平方、积分和平滑以获得最佳性能。P&T 能量阈值是根据采样率和平滑的 ECG 值估计的,以避免大颠簸造成的中断。如果 RR 间期变异性超过中位数的 1.5 倍,则搜索错过的峰值。计算 30 s 段的平均 R 峰征,并通过不应期检查来细化峰点,管理平坦线条件并确保检测一致性。 - 接下来,BrainBeats 使用向前-向后搜索和生理阈值识别异常的 RR 间期或 RR 间期内的峰值。信号质量指数 (SQI) 计算为46;如果系统显示警告,其中超过 20% 的 RR 时间序列包含 RR 伪影(超出生理限制或 SQI 低于 0.9),请选中此选项。该图显示过滤后的 ECG 信号、识别的 R 峰、NN 间期和插值伪影(参见 图 2)。
注意:默认情况下,RR 伪影使用形状不变的分段三次方法进行插值,以获得法向到法向 (NN) 间隔,但会将其删除(不推荐)或根据需要使用其他插值方法(线性、立方、最近邻、上/下邻、样条、三次卷积或修改后的 Akima 立方)。当存在多个 ECG 通道时,估计每个通道的 RR 间期,并为以下步骤选择 RR 伪像数量最少的通道。 - 按 右/左箭头滚动 R 峰的放大 30 秒窗口以进行更仔细的检查。如果数据包含多个 ECG/PPG 通道,则使用 RR 间期信号质量指数最好的通道。BrainBeats 目前不支持同时支持 ECG 和 PPG 信号。出于教程目的,我们选择了一个包含这两种数据类型的示例数据集。
- 完成 ECG 信号后,默认情况下,BrainBeats 带通使用非线性因果最小相位 FIR 滤波器以 1-40 Hz 过滤 EEG 数据,以减少心跳前后周期之间的涂抹活动,保留因果关系,并避免不希望的群体延迟44。这对于检查检测信号前周期的用户尤其重要。如果低通滤波器设置为高于电源线频率的值(例如,80 Hz 低通,电源线频率为 50 Hz),请使用尖锐的陷波滤波器来消除线路噪声伪影。然后使用 REST 算法(最适合 HEP 分析49)将 EEG 数据重新引用到无穷大,除非检测到少于 30 个通道(在这种情况下,它们无法可靠地重新引用,并且会生成警告以告知用户)。
- 然后,BrainBeats 检测、删除和插值异常的 EEG 通道(图 3)。检查默认参数是否为大于 5 s 的平坦线(clean_flatlines 算法)、最大高频噪声标准差 10、窗口长度 5 s(以更好地捕获慢频伪影52)、相邻通道之间的最小相关性 .65 和最大容忍部分 33%(clean_channels 算法)。
注意:默认情况下,RANSAC 样本数设置为 500,以提高不良通道抑制的收敛性和可复制性(尽管它会增加计算时间)。 - 接下来,将 R 峰值作为事件标记插入 EEG 数据中,以标记每个心跳并围绕这些标记分割数据,而无需删除基线(根据指南43,66;图 4)。由于 NN 间期具有不同的长度,并且 EEG 必须以恒定长度分割,因此使用心跳间期 (IBI) 数据的第 5 个百分位数(即,低于 5% 的最短 IBI 的值,在直方图上显示为红色虚线;见图 5)估计 R 峰值事件后的最小时期大小截止值。
注意:这个第 5 个百分位值是一个很好的折衷方案,可以保留尽可能多的纪元,同时确保它们不会太短,因为 HEP/HEO 分析的兴趣期是心跳后 200-600 毫秒49,67。 - 从 R 峰值之前的 -300 毫秒到 R 峰值后的第 5 个百分位值,将 EEG 数据分段,R 峰值在时间 0。根据指南49,67,拒绝短于 550 ms 或包含多个 R 峰(这将使 ERP/ERSP 偏倚)的时期。使用均方根 (RMS) 和信噪比 (SNR) 指标以及 MATLAB 的 isoutlier 函数检测包含大量 EEG 伪影的时期(图 6)。人为的 epoch 将被删除。
- 使用默认的 Infomax 算法执行盲源分离,考虑数据排名以获得最佳性能53。如果需要,通过选择选项 Picard (快速) 来选择预条件独立成分分析 (PICARD) 算法来快速计算54,54 ,或者通过为字段 ICA 方法选择选项 Modified Infomax (long but replicaable) 来选择修改后的 Infomax 算法(尽管这涉及更长的计算时间)。使用 ICLabel 插件56 自动对 IC 进行分类以提取非大脑伪影(以 90% 的置信度去除眼部成分,而以 99% 的置信度去除肌肉、线噪声和通道噪声;图 7)。
- 在第一个 GUI 窗口中保持选中 Visualize outputs 选项,以可视化最终的 EEG 时间序列、大平均 HEP(图 8 顶部)、每个心跳的 HEP(图 8 底部)和大平均心跳诱发振荡(HEO; 图 9)。根据事件相关频谱扰动(ERSP,即心跳中脑电图功率的变化; 图 9 顶部)和试验间相位相干性(ITC,即跨心跳相位角的一致性; 图 9 底部)。
注意:ERSP 是使用默认的 3 周期小波(应用 Hanning 锥形窗口,焊盘比为 2)计算的,并且用于较高频率的小波周期数缓慢扩展至等效 FFT 窗口中周期数的 20%在其最高频率(1 减去 0.8)。这将控制函数测量的各个窗口的形状以及它们在生成的 time/frequency 窗格中的形状。为了说明目的,删除了任意基线,并计算了 7-25 Hz 频率的 ERSP 以捕获文献中描述的典型 HEO 效应,即 300-450 ms 心跳后 α 波段 (8-13 Hz) 在前中央电极17,67。由于节拍间隔定义的 epoch 大小较短,因此无法估计较低的频率。应用非参数(排列)统计量来可视化 p 值为 0.05 的 HEO,并针对错误发现率(FDR,即 1 型错误或系列错误)进行校正。这些图是为教程目的或单试验分析而生成的。 - 生成预处理图以可视化不同的步骤。要将其关闭,请取消选中 Visualize preprocessing 复选框。最终的 EEG 数据(在 R 峰值周围清理和分割)不包括 ECG 数据,因为它会使 ERP/ERSP 分析产生偏差。要在最终输出中保留心形通道,请选中 Keep heart channel 复选框。
注意:如果需要,请在处理下一个文件(下一个条件或参与者)之前在此处暂停。 - BrainBeats 支持 EEGLAB 的历史功能。在所有操作结束时,在 MATLAB 的命令窗口中键入 eegh 以打印命令行,以通过单个命令行重复上述所有步骤,并使用在 GUI 中手动选择的参数,从而轻松实现自动化。在 EEGLAB 结构中找到预处理输出(例如,心血管时间序列的信号质量指数、NN 间期、去除的 EEG 通道、段和组件等):EEG.brainbeats.preprocessings。所有参数也都导出在 EEG.brainbeats.parameters 中。
- 对于高级用户,请使用以下命令行使用默认参数执行上述所有步骤(有关更多选项,请参阅 BrainBeats 存储库中的教程脚本):
EEGLAB 的;关闭;在没有 GUI 的情况下启动 EEGLAB 的百分比
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
脑电图 = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data'));%加载示例数据集
脑电图 = brainbeats_process(脑电图,'分析',
'hep'、'heart_signal'、'ECG'、'heart_channels'、
{'心电图'},'clean_eeg',true);% 启动 BrainBeats1st 级别,以使用默认参数处理文件以进行 HEP 分析 - 上述步骤从 ECG 信号执行 HEP/HEO。对 PPG 信号使用以下步骤。
- 在以下步骤中,执行相同的操作,但使用 PPG 信号。由于前面的操作覆盖了相同的数据集(请参阅步骤 1.5),然后再次打开 BrainBeats 的第一个 GUI 以选择主要参数。单击 BrainBeats > BrainBeats >1st level (subject level) (工具)。选择 Heartbeat-evoked potentials (HEP) 作为要运行的分析,选择 PPG 作为心脏数据类型,然后单击按钮以显示通道列表以选择 PPG 通道。保持选项 可视化输出(推荐) 和 保存输出 选中 状态,然后单击 确定。
- 第二个 GUI 窗口以类似于步骤 2.2 的方式弹出。唯一的区别是,我们现在可以微调参数来处理 PPG 信号,而不是 ECG 信号。单击 OK 以使用默认参数运行。
- 此时将显示一条警告消息,要求确认删除已检测到的额外 ECG 通道。同样,这是意料之中的。单击 Yes (是)。默认情况下,该工具箱将预处理 PPG 信号,检测脉冲波以获得 RR 间隔,识别 RR 伪影(如果有)并对其进行插值(图 10)。步骤 2.7.更改为 2.12。,并生成相同的绘图和输出,但基于从 PPG 信号检测到的 R 波(参见 图 11 和 图 12)。
注意:R 波是使用指定窗口内信号的斜率来检测的。当斜率超过动态阈值时,会标记潜在脉冲,该阈值会根据检测历史记录和信号特性进行调整。然后,该算法在闭眼周期内进行搜索以查明最大斜率,随后,通过阈值确定脉搏波的开始。R 波峰值被识别为起点附近的谷值,并记录它们的位置。该算法遍历整个信号,不断调整检测阈值并识别 R 波峰值,然后将其用于计算 RR 间隔。 - 对于高级用户,请使用以下命令行使用默认参数执行上述所有步骤(有关更多选项,请参阅 BrainBeats 存储库中的教程脚本):
EEGLAB 的;关闭;在没有 GUI 的情况下启动 EEGLAB 的百分比
main_path = fileparts(which('eegplugin_
BrainBeats.m'));cd(main_path);
脑电图 = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data'));%加载示例数据集
脑电图 = brainbeats_process(脑电图,'分析',
'hep'、'heart_signal'、'PPG'、'heart_channels'、
{'PPG'},'clean_eeg',true);% 启动 BrainBeats1st 级别,以使用默认参数处理文件以进行 HEP 分析
3. 提取 EEG 和 HRV 特征
- 再次加载相同的数据集(请参阅步骤 1.5;单击文件>加载现有数据集>选择 dataset.set),因为它已被前面的操作覆盖,然后再次打开主 GUI 以选择主要参数(步骤 2.1;点击 工具 > BrainBeats > 1级)。选择提取EEG和HRV特征作为分析类型,选择ECG作为心脏信号类型,并在电极标签列表中选择ECG。单击 OK。
- 第二个 GUI 窗口与步骤 2.2 一样弹出,但具有不同的 EEG 预处理和提取 HRV 和 EEG 特征的参数。如下所述设置这些参数。单击 OK 以使用默认参数启动。
- 在 HRV 部分,单击 按钮频率选项 以选择计算 HRV 功率的方法(默认设置为归一化 Lomb-Scargle 周期图)、窗口重叠(默认设置为 25%)以及执行二级归一化的方法(默认未设置;有关详细信息,请参阅下面的注释)。
- 在 EEG 功能部分,单击 频率选项 按钮以微调某些参数,例如计算功率谱密度(PSD;默认 = 1-40 Hz)、单位(分贝、μV2/Hz 或按总功率归一化)、窗口类型(默认 = 汉明)、窗口重叠(默认 = 50%)、窗口长度(默认 = 2 s)、 以及每个频段的频率边界类型。
注意:默认情况下,HRV 功率是使用归一化的 Lomb-Scargle 周期图计算的,它不需要重新采样(因此更好地保留了原始信息),并且最好处理非均匀采样数据、缺失数据和噪声(典型值为 NN 间隔)68。归一化版本通过信号的方差来缩放功率,提供对不同噪声水平不太敏感的结果,更侧重于周期性分量的相对强度,并且在不同的录音或主题之间更具可比性。其他可用的方法包括非归一化 Lomb-Scargle 周期图、Welch 方法和快速傅里叶变换 (FFT)。将对 Welch 和 FFT 方法自动执行重采样,以创建必要的定期采样时间序列。通过将每个频带的功率除以总功率,可以应用二级归一化,从而更直观地测量每个频率分量对总功率的相对贡献。默认情况下,它是禁用的,因为它仅在所有四个波段都可用时才有意义,需要至少 24 小时的信号。这些算法改编自 Physionet 心血管信号处理工具箱46。
- 出现相同的警告消息,要求确认删除已检测到的额外 PPG 通道。同样,这是意料之中的。单击 Yes (是)。BrainBeats 将开始预处理 ECG 数据并以相同的方式提取 NN 间期,如步骤 2.4 所示。然后,它从时间 (SDNN、RMSSD、pNN50)、频率 (ULF、VLF、LF、HF、LF:HF 比率、总功率) 和非线性 (Poincare、相位校正信号平均、模糊熵和分形维数) 域的 NN 间隔中提取心率变异性 (HRV) 特征。
注意:BrainBeats 会自动检查文件长度以确保满足最低要求(例如,ULF-HRV 功率需要 24 小时的数据),否则发送警告消息,并且不会导出这些功能以防止不可靠的估计。BrainBeats 遵循估计 HRV 指标27,45 的指南和建议。 - BrainBeats 像步骤 2.7 一样预处理 EEG 数据。(参见图 3)。使用伪影子空间重建 (ASR) 算法50,52(默认 SD 标准设置为 30,并使用 80% 的可用 RAM 来提高速度),在连续数据中自动检测大型伪影。这些大的伪影从 EEG 数据中删除(见 图13 )。要在 GUI 中调整这些参数,请选择字段 Threshold to reject bad segments with ASR 和 Available RAM to use for ASR。
注意:对于特征模式,脑电图和心血管时间序列不需要时间同步,因为特征是分别估计每个信号的。因此,可以直接从 EEG 数据中删除 EEG 伪影(红色, 图 13),这与 HEP 模式不同,在 HEP 模式下,包含伪影的 epoch 在两个时间序列中都被拒绝,因为具有毫秒精度的时间同步对于该方法至关重要。 - 使用与 HEP 相同的算法和参数执行 ICA(参见步骤 2.11.),除了这次如果以 99% 的置信度检测到,则去除心脏成分(它们被保留用于 HEP/HEO,因为我们不想去除相关的心脏相关信号)。
- 在步骤 3.2 中选中了 频域 框,因此 BrainBeats 提取了以下频域特征:delta (1-3 Hz)、theta (3-7 Hz)、alpha (8-13 Hz)、beta (13-30 Hz) 和 gamma (30+ Hz) 频段的平均功率谱密度 (PSD)、单个 alpha 频率 (IAF) 和所有可用(对称)电极对的 alpha 不对称性。检查这些功能。
注意:PSD 转换为分贝 (dB) 有助于比较不同录制内容和主题的结果。频率边界可以设置为常规边界(例如,为 alpha 波段预定义的 8-13 Hz)或个性化边界,这些边界是从功率谱密度的分布中检测到的,以解释个体间差异69 (例如,alpha 波段为 7.3-12.6 Hz)。该算法专为 Alpha 频段设计,对于其他频段的性能不佳,尤其是当功率谱分布中不存在峰值时。使用 alpha 重心估计单个 alpha 频率 (IAF),以更好地处理分裂峰或模糊峰69。Alpha 不对称性按照准则计算(2 秒汉明窗,重叠 50%,左通道的 α 功率的对数减去右通道的 α 功率的对数)47。因此,正值表示左大于右 alpha 功效,反之亦然。α 不对称性可以通过将来自每个电极的 α 功率除以所有电极的 α 功率之和47 来标准化。对称对是使用 θ 距离获得的,要求 EEG 数据包含电极坐标。 - 在步骤 3.2 中选中 了非线性域 框,BrainBeats 提取每个 EEG 通道的模糊熵和分形维数。
注意:非线性域特征被认为可以捕获光谱测量遗漏的大脑的非线性、复杂动力学,并在研究各种身体系统之间的相互作用方面显示出特别的前景 70,71,72。模糊熵比其替代方案(样本和近似熵)更可靠、更稳健,但需要更长的计算时间(尤其是对于具有高采样率的长 EEG 时间序列)。为了解决这个问题,当 EEG 信号时长超过 2 分钟且采样率大于 100 Hz 时,它们会自动下采样(或在因子不是整数时抽取)到 90 Hz(即对应于 45 Hz 的奈奎斯特频率,以匹配默认的低通滤波器并尽可能避免线路噪声伪影)。此外,在估计 EEG 特征时,并行计算默认处于激活状态,这减少了计算时间,尤其是在有许多 EEG 通道可用时。 - 在第一个 GUI 中选择 Visualize outputs (参见步骤 3.1.),以生成一个显示 HRV 和 EEG 数据的功率谱密度 (PSD)(图 14)以及显示一些 EEG 特征的头皮地形图(图 15)。
注意:您还可以在 EEG.brainbeats.preprocessing 中找到一些预处理输出,并在 EEG.brainbeats.parameters 中找到所有使用的参数。我们鼓励用户在科学出版物中报告这些输出,以提高结果的可复制性。 - 在第一个 GUI 窗口中保持选中 Save outputs 框,以将所有导出到 EEG.brainbeats.features 中 EEGLAB .set 文件的特征保存在 EEG.brainbeats.features 中,并将它们保存在加载数据集的同一文件夹中的 .mat 文件中。
- BrainBeats 支持 EEGLAB 的历史功能。在所有操作结束时,在 MATLAB 的命令窗口中键入 eegh 以打印命令行,这将允许您通过单个命令行重复上述所有步骤,并使用在 GUI 中手动选择的参数,从而轻松实现操作的自动化和复制。
- 对于高级用户,请使用以下命令执行上述所有步骤:
EEGLAB 的;关闭;在没有 GUI 的情况下启动 EEGLAB 的百分比
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
脑电图 = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data'));% 加载示例数据集
脑电图 = brainbeats_process(EEG,' 分析',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - 前面的步骤从 ECG 信号中提取了 HRV 特征。使用以下步骤从 PPG 信号中提取 HRV 特征(EEG 特征相同)。
- 再次加载相同的数据集(步骤 1.5.),因为它已作覆盖,然后再次打开主 GUI(步骤 2.5)。选择提取EEG和HRV特征进行分析,并选择PPG作为心脏信号类型,PPG作为通道名称。单击 OK。
- 第二个 GUI 窗口现在显示用于预处理 PPG 和提取 HRV 和 EEG 特征的参数。单击 OK 以使用默认参数运行。参数在步骤 2.17 中描述。
- 将出现一条警告消息,要求确认删除检测到的 ECG 通道。这是意料之中的,因为该工具箱并非旨在同时分析 ECG 和 PPG(或其他辅助通道),将其保留在数据集中会导致严重错误(例如,伪影去除、ICA 分解不良等)。单击 Yes (是)。
- BrainBeats 预处理 PPG 信号并估计 NN 间隔,如步骤 2.5 所示。从 NN 区间检查提取的 HRV 特征,与步骤 3.2 中相同。不同之处在于 NN 间隔现在是从 PPG 信号获得的。EEG 信号按照步骤 3.2 进行预处理。BrainBeats 绘制 PSD (图 16)。这里唯一的区别是从 PPG 获得的 NN 间期而不是 ECG 估计的 PSD。
- 对于高级用户,请使用以下命令执行上述所有步骤:
EEGLAB 的;关闭;在没有 GUI 的情况下启动 EEGLAB 的百分比
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
脑电图 = pop_loadset('filename','dataset.set','filepath',
fullfile(main_path, 'sample_data'));%加载示例数据集
脑电图 = brainbeats_process(脑电图,'分析',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. 从脑电信号中提取心脏伪影。
- 加载示例数据集(请参阅步骤 1.5)。
- 单击工具打开 GUI 主窗口> BrainBeats > 1级(主题级别),然后选择 Extract heart artifacts from EEG signals(从 EEG 信号中提取心脏伪影)作为分析类型,选择 ECG 作为心脏信号类型,然后在电极标签列表中选择 ECG。单击 OK。
- 第 2 个 GUI 窗口显示预处理参数。将电源线噪声设置为 50 Hz(欧洲),根据需要编辑置信度,选中 Boost mode (beta) 复选框,然后单击 OK(确定)以使用默认参数运行,因为来自样本数据集的 EEG 信号未经过预处理。
注意:默认情况下,检测心脏成分的置信度设置为 80%,对于某些数据集来说,这可能太低或太高。增加此值将增加检测到心脏成分的机会,但会降低该检测的可靠性。Boost 模式(测试版)是可选的,旨在通过将心脏信号涂抹到 EEG 信号中来提高分类性能。 - 将出现一条警告消息,要求确认删除已检测到的额外 PPG 通道。这是意料之中的,因为该工具箱并非旨在同时分析 ECG 和 PPG(或其他辅助通道),将其保留在数据集中会导致严重错误(例如,伪影去除、ICA 分解不良等)。单击 Yes (是)。
- ECG 信号经过带通滤波,可消除低于 1 Hz 的慢频漂移和高于 20 Hz 的高频噪声(使用非因果零相位 FIR 滤波器)。如步骤 3.4 所示,对 EEG 信号进行预处理。
- 使用预条件 ICA for real data 算法 (PICARD) 执行独立成分分析 (ICA)。要更改此选项,请在步骤 4.3 中从 GUI 中选择用于复制的标准 Infomax 算法或修改的 Infomax 算法。然后,使用 ICLabel 自动对独立组件进行分类。如果某个组件被归类为心脏组件,置信度为 80%,则默认情况下,它会从 EEG 数据中自动检测和提取。
- 在第一个主 GUI(步骤 4.2)中保留框 Visualize outputs(可视化输出),以可视化去除组件的头皮地形(图 17 左)和最终的 EEG 时间序列(蓝色, 图 17 右)提取心脏组件后(红色, 图 17 右)。
注意:保留 ECG 通道以进行可视化以确认 ECG 相关成分的提取,但在此步骤后将其删除,因为它不再包含任何相关信息。 - 对于高级用户,请使用以下命令行执行以下步骤:
EEGLAB 的;关闭;在没有 GUI 的情况下启动 EEGLAB 的百分比
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
脑电图 = pop_loadset('filename','dataset.set','filepath',
fullfile(main_path, 'sample_data'));
脑电图 = brainbeats_process(脑电图,'分析','rm_heart',
'heart_signal','心电图',...
'heart_channels',{'心电图'},'clean_eeg',真,'vis_cleaning',假,...
'conf_thresh',.8,'boost',true);
结果
首先,使用 BrainBeats 插件对 EEG 和 ECG 数据进行预处理,识别和去除伪影,并分析心跳诱发电位 (HEP) 和振荡 (HEO)。BrainBeats 成功地从 ECG 信号和一些 RR 伪影中检测到 RR 间期(图 2)。BrainBeats 还在命令窗口中报告说,11/305 (3.61%) 的检测信号被标记为伪影并进行内插。RR 间隔的平均信号质量指数 (SQI) (插值前) 的值为 1,这是最高值。低信号质量对应于 20% 或更多的...
讨论
协议中的关键步骤
关键步骤在步骤 1.1-1.4 中描述。警告和错误消息在工具箱中的不同位置实现,以帮助用户了解他们可能遇到问题的原因(例如,EEG 数据中未加载电极位置、文件长度太短而无法计算超低频 HRV 的可靠测量、信号质量太低而无法进行任何可靠的分析等)。每个功能都为高级用户记录了数据,并且可以轻松微调参数(推荐参数和典型范围记录在本手稿和代码中)。用?...
披露声明
作者没有什么可披露的。
致谢
认知科学研究所支持这项研究。我们感谢原始开源算法的开发人员,这些算法适用于开发 BrainBeats 的一些算法。
材料
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
参考文献
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。