Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
BrainBeats als Open-Source-EEGLAB-Plugin zur gemeinsamen Analyse von EEG- und kardiovaskulären Signalen
In diesem Artikel
Zusammenfassung
Die BrainBeats-Toolbox ist ein Open-Source-EEGLAB-Plugin, das entwickelt wurde, um EEG- und kardiovaskuläre (EKG/PPG) Signale gemeinsam zu analysieren. Es umfasst die Bewertung von herzschlagevozierten Potentialen (HEP), merkmalsbasierte Analysen und die Extraktion von Herzartefakten aus EEG-Signalen. Das Protokoll wird dazu beitragen, das Zusammenspiel von Gehirn und Herz durch zwei Linsen (HEP und Merkmale) zu untersuchen und so die Reproduzierbarkeit und Zugänglichkeit zu verbessern.
Zusammenfassung
Das Zusammenspiel zwischen dem Gehirn und dem Herz-Kreislauf-System erregt zunehmend Aufmerksamkeit, da es unser Verständnis der menschlichen Physiologie verbessern und die Gesundheitsergebnisse verbessern kann. Die multimodale Analyse dieser Signale stellt jedoch eine Herausforderung dar, da es an Richtlinien, standardisierten Signalverarbeitungs- und Statistikwerkzeugen, grafischen Benutzeroberflächen (GUIs) und Automatisierung für die Verarbeitung großer Datensätze oder an der Erhöhung der Reproduzierbarkeit mangelt. Eine weitere Lücke besteht bei standardisierten EEG- und Herzfrequenzvariabilitäts-Extraktionsmethoden (HRV), die die klinische Diagnostik oder die Robustheit von Modellen des maschinellen Lernens (ML) untergraben. Als Reaktion auf diese Einschränkungen stellen wir die BrainBeats-Toolbox vor. BrainBeats wurde als Open-Source-EEGLAB-Plugin implementiert und integriert drei Hauptprotokolle: 1) Herzschlag-evozierte Potentiale (HEP) und Oszillationen (HEO) zur Beurteilung des zeitgesteuerten Gehirn-Herz-Zusammenspiels mit einer Genauigkeit von Millisekunden; 2) EEG- und HRV-Merkmalsextraktion zur Untersuchung von Assoziationen/Unterschieden zwischen verschiedenen Gehirn- und Herzmetriken oder zur Erstellung robuster merkmalsbasierter ML-Modelle; 3) Automatisierte Extraktion von Herzartefakten aus EEG-Signalen, um mögliche kardiovaskuläre Kontaminationen bei der EEG-Analyse zu entfernen. Wir bieten eine Schritt-für-Schritt-Anleitung für die Anwendung dieser drei Methoden auf einen Open-Source-Datensatz, der simultane 64-Kanal-EEG-, EKG- und PPG-Signale enthält. Benutzer können die Parameter einfach fein abstimmen, um ihre individuellen Forschungsanforderungen über die grafische Benutzeroberfläche (GUI) oder die Befehlszeile anzupassen. BrainBeats soll die Hirn-Herz-Zusammenspiel-Forschung zugänglicher und reproduzierbarer machen.
Einleitung
Lange Zeit dominierte der reduktionistische Ansatz die wissenschaftliche Erforschung der menschlichen Physiologie und Kognition. Bei diesem Ansatz wurden komplexe körperliche und mentale Prozesse in kleinere, besser handhabbare Komponenten zerlegt, so dass sich die Forscher isoliert auf einzelne Systeme konzentrieren konnten. Diese Strategie entstand aufgrund der Herausforderungen bei der Erforschung der komplizierten und miteinander verbundenen Natur des menschlichen Körpers und Geistes1. Der Reduktionismus hat maßgeblich dazu beigetragen, einzelne Subsysteme isoliert zu verstehen, wie z.B. die Aufklärung der Rolle von Ionenkanälen und Aktionspotentialen für die neuronale2 oder kardiale3 Kommunikation. Es gibt jedoch noch eine erhebliche Lücke in unserem Verständnis davon, wie diese isolierten Systeme auf einer größeren räumlichen und zeitlichen Skala interagieren. Der multimodale (integrative oder ökologische) Rahmen betrachtet den menschlichen Körper als ein komplexes multidimensionales System, in dem der Geist nicht als Produkt des Gehirns, sondern als Aktivität des Lebewesens angesehen wird, eine Aktivität, die das Gehirn in die alltäglichen Funktionen des menschlichen Körpers integriert4. Die multimodalen und reduktionistischen Ansätze schließen sich nicht aus, so wie wir nicht ein Neuron ohne das gesamte Gehirn oder das gesamte Gehirn untersuchen können, ohne die Eigenschaften einzelner Neuronen zu verstehen. Zusammen ebnen sie den Weg für ein umfassenderes, synergetisches Verständnis der menschlichen Gesundheit, Pathologie, Kognition, Psychologie und Bewusstsein. Die vorliegende Methode zielt darauf ab, die multimodale Untersuchung des Zusammenspiels zwischen Gehirn und Herz zu erleichtern, indem sie die gemeinsame Analyse von Elektroenzephalographie (EEG) und kardiovaskulären Signalen, nämlich Elektrokardiographie (EKG) und Photoplethysmographie (PPG), ermöglicht. Diese Toolbox, die als EEGLAB-Plugin in MATLAB implementiert ist, adressiert bestehende methodische Einschränkungen und ist Open Source, um die Zugänglichkeit und Reproduzierbarkeit im wissenschaftlichen Bereich zu erleichtern. Es implementiert die neuesten Richtlinien und Empfehlungen in seine Design- und Standardparameter, um Benutzer zu ermutigen, bekannte Best Practices zu befolgen. Die vorgeschlagene Toolbox sollte eine wertvolle Ressource für Forscher und Kliniker sein, die daran interessiert sind, 1) herzschlagevozierte Potentiale zu untersuchen, 2) Merkmale aus EEG- und EKG/PPG-Signalen zu extrahieren oder 3) Herzartefakte aus EEG-Signalen zu entfernen.
Herz-Hirn-Forschung
Die Beziehung zwischen Herz und Gehirn wurde in der Vergangenheit mit Hilfe von bildgebenden Verfahren wie der funktionellen Magnetresonanztomographie (fMRT) und der Positronen-Emissions-Tomographie (PET) untersucht. Mit diesen Instrumenten hoben die Forscher einige Gehirnregionen hervor, die mit der kardiovaskulären Kontrolle verbunden sind (z. B. Manipulation der Herzfrequenz und des Blutdrucks5), zeigten den Einfluss der Herzfrequenz auf das BOLD-Signal6 oder identifizierten potenzielle Gehirn-Körper-Bahnen, die zur koronaren Herzkrankheit beitragen (z. B. stressevozierter Blutdruck7). Während diese Studien unser Verständnis des komplexen Zusammenspiels zwischen dem Zentralnervensystem (ZNS) und der Herz-Kreislauf-Funktion erheblich verbessert haben, sind diese Neuroimaging-Verfahren teuer, nur begrenzt verfügbar und auf kontrollierte Laborumgebungen beschränkt, was ihre Praktikabilität für reale und großtechnische Anwendungen einschränkt.
Im Gegensatz dazu sind EEG und EKG/PPG erschwinglichere und tragbarere Werkzeuge, die das Potenzial bieten, Gehirn-Herz-Interaktionen in vielfältigeren Umgebungen und Populationen oder über längere Zeiträume zu untersuchen, was neue Möglichkeiten eröffnet. Das EKG misst die elektrischen Signale, die bei jedem Herzschlag erzeugt werden, wenn sich das Herz zusammenzieht und entspannt, und zwar über Elektroden, die auf der Haut platziert werden (normalerweise an der Brust oder an den Armen)8. PPG misst Veränderungen des Blutvolumens in den mikrovaskulären Geweben (d. h. Blutfluss und Pulsfrequenz) mit einer Lichtquelle (z. B. LED) und einem Photodetektor (der üblicherweise an einer Fingerspitze, einem Handgelenk oder einer Stirn angebracht wird), wobei es sich darauf verlässt, wie das Blut mehr Licht absorbiert als das umgebende Gewebe9. Beide Methoden liefern wertvolle Informationen über die kardiovaskuläre Funktion, dienen jedoch unterschiedlichen Zwecken und bieten unterschiedliche Datentypen. Wie das EKG zeichnet das EEG die elektrischen Felder auf, die durch die synchronisierte Aktivität von Tausenden von kortikalen Neuronen erzeugt werden, die sich durch die extrazelluläre Matrix, das Gewebe, den Schädel und die Kopfhaut ausbreiten, bis sie die Elektroden erreichen, die auf der Oberfläche der Kopfhaut platziert sind10. Daher ist die Verwendung von EEG und EKG/PPG vielversprechend, um unser Verständnis der physiologischen, kognitiven und emotionalen Prozesse, die den Gehirn-Herz-Interaktionen zugrunde liegen, und ihrer Auswirkungen auf die menschliche Gesundheit und das Wohlbefinden zu verbessern. Daher kann die Erfassung des Herz-Hirn-Zusammenspiels von EEG- und EKG-/PPG-Signalen mit der BrainBeats-Toolbox besonders nützlich für die folgenden wissenschaftlichen Bereiche sein: klinische Diagnostik und Prognose, maschinelles Lernen (ML) in großen Datenmengen, Selbstüberwachung in der realen Welt11 und mobile Gehirn-/Körperbildgebung (MoBI)12,13.
Zwei Ansätze zur gemeinsamen Analyse von EEG- und EKG-Signalen
Es gibt zwei Hauptansätze, um die Wechselwirkungen zwischen EEG und kardiovaskulären Signalen zu untersuchen:
Die herzschlagevozierten Potentiale (HEP) im Zeitbereich: ereigniskorrelierte Potentiale (ERP) und die herzschlagevozierten Oszillationen (HEO) im Zeit-Frequenz-Bereich: ereigniskorrelierte spektrale Störungen (ERSP) und Inter-Trial-Kohärenz (ITC). Dieser Ansatz untersucht, wie das Gehirn jeden Herzschlag verarbeitet. Mit einer Genauigkeit von Millisekunden (ms) erfordert diese Methode, dass beide Zeitreihen perfekt synchronisiert sind und die Herzschläge in den EEG-Signalen markiert werden. Dieser Ansatz hat in den letzten Jahren an Interesse gewonnen 14,15,16,17,18,19.
Merkmalsbasierter Ansatz: Dieser Ansatz extrahiert EEG- und Herzfrequenzvariabilitätsmerkmale (HRV) aus kontinuierlichen Signalen und untersucht Assoziationen zwischen ihnen. Dies wurde unabhängig voneinander für EEG (oft als quantitatives EEG oder qEEG20 bezeichnet), EKG 21,22,23 und PPG 24,25,26 durchgeführt. Dieser Ansatz bietet vielversprechende Anwendungen, indem er sowohl zustands- als auch merkmalsbezogene Variablen erfasst. Es ist zu beachten, dass sowohl für EEG- als auch für kardiovaskuläre Signale die Merkmalsvariable umso dominanter ist, je länger die Aufzeichnung dauert 27,28,29. Die Anwendungen hängen also von den Aufnahmeparametern ab. Merkmalsbasierte Analysen gewinnen zunehmend an Interesse und liefern neue quantitative Metriken für die Vorhersage der Entwicklung psychischer und neurologischer Störungen, des Ansprechens auf die Behandlung oder des Rückfalls 30,31,32,33,34,35. Dieser Ansatz ist besonders überzeugend bei großen und realen Datensätzen (z. B. Klinik, Fernüberwachung), die dank der jüngsten Innovationen in der tragbaren Neurotechnologie11 leichter zu erhalten sind. Eine weniger erforschte Anwendung ist die Identifizierung von Assoziationen zwischen spezifischen Gehirn- und Herzmerkmalen, wobei möglicherweise die zugrunde liegende Dynamik des Zentralnervensystems hervorgehoben wird. Die Herzfrequenzvariabilität (HRV) kann sowohl aus EKG- als auch aus PPG-Signalen berechnet werden. Es liefert Informationen über das autonome Nervensystem (ANS), indem es die Schwankungen in den Zeitintervallen zwischen den Herzschlägen (d. h. den normalen bis normalen Intervallen) misst27. Eine erhöhte sympathische (SNS) Aktivität (z. B. bei Stress oder körperlicher Belastung) reduziert in der Regel die HRV, während die parasympathische (PNS) Aktivität (z. B. während der Entspannung) sie erhöht. Eine langsamere Atemfrequenz erhöht im Allgemeinen die HRV aufgrund der erhöhten PNS-Aktivität, insbesondere bei kurzen Aufzeichnungen (<10 min)27. Höhere HRV-Werte deuten im Allgemeinen auf ein widerstandsfähigeres und anpassungsfähigeres ANS hin, während eine niedrigere HRV auf Stress, Müdigkeit oder zugrunde liegende Gesundheitsprobleme hinweisen kann. Lange HRV-Aufzeichnungen (d. h. mindestens 24 Stunden) bieten eine prädiktive Prognose für verschiedene Gesundheitszustände, einschließlich Herz-Kreislauf-Erkrankungen, Stress, Angstzustände und einige neurologische Erkrankungen27. Messungen wie Blutdruck, Herzfrequenz oder Cholesterinspiegel geben Aufschluss über den Zustand des Herz-Kreislauf-Systems. Im Gegensatz dazu fügt die HRV einen dynamischen Aspekt hinzu und zeigt, wie das Herz auf Stress reagiert und sich davon erholt.
Die Vorteile von BrainBeats gegenüber bestehenden Methoden
Es gibt zwar Instrumente, um kardiovaskuläre und EEG-Signale unabhängig voneinander zu verarbeiten, aber sie können nicht gemeinsam analysiert werden. Darüber hinaus sind die meisten verfügbaren Mittel zur Verarbeitung kardiovaskulärer Signale mit kostspieligen Lizenzierungen verbunden, erlauben keine automatisierte Verarbeitung (besonders vorteilhaft für große Datensätze), verfügen über proprietäre Algorithmen, die Transparenz und Reproduzierbarkeit verhindern, oder erfordern fortgeschrittene Programmierkenntnisse, da sie keine grafische Benutzeroberfläche (GUI) bereitstellen36. Unseres Wissens unterstützen vier Open-Source-MATLAB-Toolboxen die HEP/HEO-Analyse mit einer GUI: die EKG-Kit-Toolbox37, die BeMoBIL-Pipeline38, das HEPLAB EEGLAB-Plugin39 und die CARE-rCortex-Toolbox40. HEPLAB, BeMoBIL und EKG-Kit erleichtern zwar die HEP-Analyse, indem sie Herzschläge erkennen und in den EEG-Signalen markieren, bieten jedoch keine statistische Analyse oder sind auf den Zeitbereich (d. h. HEP) beschränkt. Das CARE-rCortex-Plugin löste diese Probleme, indem es EKG- und Atemsignale, Zeit-Frequenz-Bereichsanalysen, Statistiken und fortschrittliche Baseline-Normalisierungs- und Korrekturmethoden unterstützte, die an die HEP/HEO-Analyse angepasst sind. Es wird jedoch die Bonferroni-Methode zur statistischen Korrektur des Typ-1-Fehlers (d. h. falsch positiver Ergebnisse) verwendet, die für EEG-Anwendungen zu konservativ und physiologisch nicht solide ist, was zu einer Zunahme von Typ-II-Fehlern (d. h. falsch negativen Ergebnissen) führt41. Darüber hinaus bietet die Toolbox keinen Befehlszeilenzugriff für die Automatisierung. Schließlich raten neuere Studien von Baseline-Korrekturmethoden ab 42,43,44, da sie das Signal-Rausch-Verhältnis (SNR) reduzieren und statistisch unnötig und unerwünscht sind.
Um diese Einschränkungen zu beheben, stellen wir die BrainBeats-Toolbox vor, die derzeit als Open-Source-EEGLAB-Plugin in der MATLAB-Umgebung implementiert ist. Es bietet folgende Vorteile gegenüber bisherigen Methoden:
1) Eine einfach zu bedienende GUI und Befehlszeilenfunktionen (für Programmierer, die eine automatisierte Verarbeitung durchführen möchten). 2) Validierte Algorithmen, Parameter und Richtlinien für die Verarbeitung kardiovaskulärer Signale, wie z. B. die Erkennung von R-Peaks, die Interpolation von RR-Artefakten und die Berechnung von HRV-Metriken (z. B. Implantation von Richtlinien für Windowing, Resampling, Normalisierung usw.27,45,46). Dies ist wichtig, da Vest et al. gezeigt haben, wie geringfügige Unterschiede in diesen Verarbeitungsschritten zu unterschiedlichen Ergebnissen führen können, was zur mangelnden Reproduzierbarkeit und klinischen Anwendbarkeit von HRV-Metriken beiträgt46. 3) Validierte Algorithmen, Standardparameter und Richtlinien für die Verarbeitung von EEG-Signalen, einschließlich Filterung und Fensterbildung44,47, Neureferenzierung48,49, Entfernung abnormaler Kanäle und Artefakte 50,51,52, optimierte ICA-Zerlegung und Klassifizierung unabhängiger Komponenten 53,54,55,56. Die Benutzer können alle Vorverarbeitungsparameter feinabstimmen oder sogar ihre EEG-Daten mit ihrer bevorzugten Methode vorverarbeiten, bevor sie die Toolbox nach ihren Bedürfnissen verwenden (z. B. mit EEGLAB clean_rawdata Plugin50,52, der BeMoBIL-Pipeline38, der PREP-Pipeline57 usw.). 4) Herzschlagevozierte Potentiale (HEP, d.h. Zeitbereich) und Oszillationen (HEO; ereigniskorrelierte spektrale Störungen mit Wavelet- oder FFT-Methoden sowie Kohärenz zwischen den Versuchen sind über die Standard-EEGLAB-Software verfügbar) von EKG-Signalen. Parametrische und nichtparametrische Statistiken mit Korrekturen für Fehler vom Typ 1 sind über die Standardsoftware von EEGLAB verfügbar. Zu den nichtparametrischen Statistiken gehören Permutationsstatistiken und raumzeitliche Korrekturen für Mehrfachvergleiche (z. B. raumzeitliches Clustering oder schwellenwertfreie Clusterverstärkung)58,59. Benutzer können das LIMO-EEG-Plugin verwenden, um eine hierarchische lineare Modellierung zu implementieren, die die Varianz innerhalb und zwischen den Probanden gut berücksichtigt und einen annahmefreien massenunivariaten Ansatz mit robuster Kontrolle für Fehler vom Typ I und II implementiert60,61. Die statistischen Analysen der HEP/HEO-Daten können im Kanal- und unabhängigen Komponentenbereich durchgeführt werden. 5) HEP/HEO- und HRV-Analyse von PPG-Signalen (zum ersten Mal für HEP/HEO). 6) Unterstützt zum ersten Mal die gemeinsame Extraktion von EEG- und HRV-Merkmalen. 7) Die Toolbox bietet verschiedene Datenvisualisierungen zur Inspektion von Signalen bei verschiedenen notwendigen Verarbeitungsschritten und Ausgaben auf Subjektebene.
| Methode | Erkennen von R-Peaks aus dem EKG | Erkennen Sie R-Wellen von PPG | HEP/HEO | EEG- und HRV-Merkmale | Entfernen von Herzartefakten aus dem EEG | GUI | Befehlszeile |
| EKG-Kit | X | X | X | X | |||
| BeMoBIL | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| PFLEGE-rKortex | X | X | X | X | |||
| Gehirn-Beats | X | X | X | X | X | X | X |
TABELLE 1: Neuheiten, die BrainBeats im Vergleich zu bereits existierenden, ähnlichen Methoden mit sich bringt.
Informationen, die den Lesern bei der Entscheidung helfen, ob die Methode für sie geeignet ist
Diese Toolbox ist für jeden Forscher oder Kliniker geeignet, der über EEG- und EKG/PPG-Daten verfügt. Das Plugin unterstützt noch nicht den Import von EEG- und EKG/PPG-Signalen aus separaten Dateien (obwohl diese Funktion in Kürze verfügbar sein wird). Die Toolbox eignet sich für alle, die HEP/HEO-Analysen durchführen, EEG- und/oder HRV-Merkmale mit standardisierten Methoden extrahieren oder einfach Herzartefakte aus EEG-Signalen entfernen möchten. In Abbildung 1 finden Sie ein Blockdiagramm, das den Gesamtablauf und die Methoden von BrainBeats zusammenfasst.
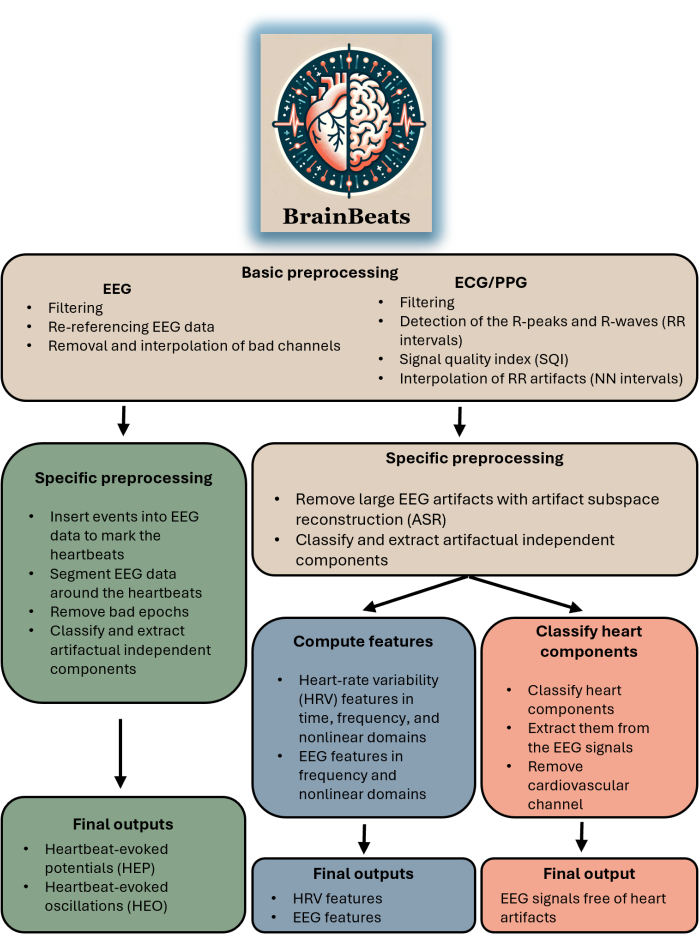
ABBILDUNG 1. Blockdiagramm, das die Gesamtarchitektur und den Ablauf von BrainBeats zusammenfasst. Die Vorgänge, die in den drei Methoden gemeinsam sind, sind braun. Operationen, die für herzschlagevozierte Potentiale (HEP) und Oszillationen (HEO) spezifisch sind, sind grün. Operationen, die für die Extraktion von EEG- und HRV-Merkmalen spezifisch sind, sind blau. Operationen, die spezifisch für die Entfernung von Herzartefakten aus den EEG-Signalen sind, sind rot. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Von jedem Teilnehmer wurde eine Einverständniserklärung eingeholt, und die Ethikkommission der Uralischen Föderalen Universität genehmigte das Versuchsprotokoll.
1. BrainBeats-Anforderungen
- Installieren Sie MATLAB und EEGLAB auf dem Computer. EEGLAB kann unter https://github.com/sccn/eeglab heruntergeladen und für Git-Benutzer überall auf dem Computer entpackt (oder geklont) werden. Weitere Informationen zur Installation finden Sie auf der GitHub-Seite.
- Fügen Sie den Pfad zum EEGLAB-Ordner im Startbereich von MATLAB hinzu, indem Sie auf die Schaltfläche Pfad festlegen klicken. Wählen Sie den eeglab-Ordner mit der entpackten Datei aus und klicken Sie auf Speichern > Schließen.
- Starten Sie EEGLAB, indem Sie eeglab in das Befehlsfenster von MATLAB eingeben.
- Installieren Sie das BrainBeats-Plugin, indem Sie auf Datei > EEGLAB-Erweiterungen verwalten klicken. Geben Sie brainbeats in die Suchleiste ein, wählen Sie das BrainBeats-Plugin in der Liste aus und klicken Sie auf Installieren/Aktualisieren.
- Laden Sie den Beispieldatensatz in EEGLAB. Klicken Sie auf Datei > Vorhandene Daten laden. Navigieren Sie zum EEGLAB-Ordner, gehen Sie zum Plugins-Ordner, gehen Sie zum BrainBeats-Ordner und öffnen Sie den sample_data Ordner. Wählen Sie die Datei dataset.set aus.
HINWEIS: Dieser Datensatz entspricht Sub-032 (Ruhezustand mit geöffneten Augen) aus einem Open-Source-Datensatz mit mehreren Themen62,63. Diese wurde ausgewählt, weil sie gleichzeitiges EEG (64 Kanäle), EKG (ein Kanal) und PPG (ein Kanal) mit einer Abtastrate von 1000 Hz enthält. EKG- und PPG-Signale wurden über die Hilfseingänge des actiCHamp-Systems erfasst. Die EEG-Daten wurden mit aktiven Elektroden aufgezeichnet, die gemäß dem 10-20-System platziert waren, mit FCz als Online-Referenz und Fpz als Masseelektrode, und mit einer Impedanz von unter 25 kOm. Das EKG wurde mit einer aktiven Elektrode aufgezeichnet, die am rechten Handgelenk, die Referenzelektrode am linken Handgelenk und der Boden am linken inneren Unterarm in einem Abstand von 3 cm vom Ellbogen. PPG wurde vom linken Zeigefinger aufgezeichnet. EEG-, EKG- und PPG-Daten sind zeitsynchronisiert, da sie gleichzeitig mit demselben Verstärker aufgezeichnet wurden. Weitere Informationen finden Sie in den Referenzen62,63. - Aktivieren Sie das Kontrollkästchen Ausgaben speichern , um alles in der entsprechenden .set-Datei am selben Ort zu speichern, an dem die Originaldatei in EEGLAB geladen wurde.
HINWEIS: Für dieses Tutorial haben wir die EEG-, EKG- und PPG-Daten in einem EEGLAB-Datensatz zusammengeführt, die Elektrodenkoordinaten der 3D-Randelementmethode (BEM) geladen und das Signal auf 250 Hz heruntergerechnet, um das Dateigewicht zu reduzieren (für den Benutzer-Download) und die Rechenzeit zu beschleunigen. Da dieser Datensatz keinen abnormalen EEG-Kanal aufwies, haben wir den Kanal TP9 künstlich modifiziert, um den Algorithmus von BrainBeats zur Erkennung und Entfernung fehlerhafter Kanäle zu veranschaulichen. In ähnlicher Weise simulieren wir künstlich ein großes EEG-Artefakt am Anfang der Datei und ein hochfrequentes muskelähnliches Artefakt in den temporalen Kanälen von 3-6 s, um die Artefaktentfernungsfunktion von BrainBeats zu veranschaulichen.
2. Herzschlagevozierte Potentiale (HEP) und Oszillationen (HEO)
- Öffnen Sie die erste allgemeine Benutzeroberfläche (GUI) von BrainBeats, um die Hauptparameter auszuwählen. Klicken Sie in EEGLAB auf Tools > BrainBeats >1. Ebene (Fachebene). Wählen Sie Heartbeat-evoked potentials HEP als durchzuführende Analyse, EKG als Herzdatentyp und klicken Sie auf die Schaltfläche, um die Liste der Kanäle anzuzeigen, um den EKG-Kanal mit der Bezeichnung EKG auszuwählen (oder geben Sie ihn direkt in das Textfeld ein). Lassen Sie die Optionen Ausgaben visualisieren (empfohlen) und Ausgaben speichern ausgewählt, und klicken Sie auf OK.
- Es öffnet sich ein zweites GUI-Fenster, das auf den vorherigen Auswahlmöglichkeiten basiert (d. h. HEP-Analyse mit EKG-Daten). Wählen Sie Vorverarbeitungsparameter für EEG- und EKG-Signale aus. Deaktivieren Sie die Vorverarbeitung, indem Sie die Kontrollkästchen EKG vorverarbeiten und EEG vorverarbeiten deaktivieren, um zu sehen, ob die Daten bereits vor dem Start von BrainBeats vorverarbeitet wurden. Das Rauschen der Stromleitung wurde in der Preprocess-EEG-Sektion auf 50 Hz geändert, da diese EEG-Daten in Russland aufgezeichnet wurden. Klicken Sie zum Starten auf OK.
- Es wird eine Warnmeldung angezeigt, in der Sie um Bestätigung gebeten werden, den erkannten PPG-Kanal zu entfernen. Dies liegt daran, dass die Toolbox nicht dafür ausgelegt ist, sowohl EKG als auch PPG gleichzeitig (oder andere Hilfskanäle) zu analysieren, und dass die Speicherung im Datensatz zu schwerwiegenden Fehlern führt (z. B. Entfernung von Artefakten, schlechte ICA-Zerlegung usw.). Klicken Sie auf Ja. BrainBeats beginnt mit der Durchführung einiger Überprüfungen, dem Festlegen einiger Standardparameter und trennt das EKG von den EEG-Daten, um das EKG-Signal vorzuverarbeiten und die RR-Intervalle zu berechnen.
- EKG- und RR-Zeitreihen werden mit validierten Algorithmen aus der Physionet Cardiovascular Signal Toolbox46,64 vorverarbeitet, die an die Datenformatierung von BrainBeats angepasst sind, die Klarheit, die Parameterabstimmung und die Rechenzeit erhöhen (siehe Referenzen zur Validierung der Algorithmen). Das Plugin gibt die RR-Intervalle, Zeitstempel, das gefilterte EKG-Signal, die R-Spitzenindizes und die Herzfrequenz (HF) aus. Optimieren Sie diese Parameter über die GUI oder die Befehlszeile.
HINWEIS: Das EKG-Signal wird unter Verwendung eines angepassten, validierten Filters (1-30 Hz) bandpassgefiltert und scannt das Signal, um den QRS-Komplex und die R-Peaks unter Verwendung der Pan-Tompkins (P&T) Methode65 zu identifizieren, wobei einige Signalverarbeitungsoperationen implementiert werden, einschließlich Differenzierung, Quadratierung, Integration und Glättung für beste Leistung. Der P&T-Energieschwellenwert wird auf der Grundlage der Abtastrate und der geglätteten EKG-Werte geschätzt, um Störungen durch große Unebenheiten zu vermeiden. Wenn die Variabilität des RR-Intervalls das 1,5-fache des Medians überschreitet, wird nach verpassten Spitzen gesucht. Das mittlere R-Peak-Vorzeichen wird über 30 s-Segmente berechnet, und die Peakpunkte werden durch eine Überprüfung der Refraktärperiode verfeinert, um die flachen Bedingungen zu verwalten und eine konsistente Detektion zu gewährleisten. - Als nächstes identifiziert BrainBeats abnormale RR-Intervalle oder Spitzen innerhalb von RR-Intervallen mithilfe einer Vorwärts-Rückwärts-Suche und physiologischer Schwellenwerte. Der Signalqualitätsindex (SQI) wird berechnet46; Aktivieren Sie diese Option, wenn das System Warnungen anzeigt, bei denen mehr als 20 % der RR-Zeitreihe RR-Artefakte enthalten (außerhalb der physiologischen Grenzen oder mit einem SQI unter 0,9). Ein Diagramm zeigt das gefilterte EKG-Signal, identifizierte R-Peaks, NN-Intervalle und interpolierte Artefakte (siehe Abbildung 2).
HINWEIS: Die RR-Artefakte werden standardmäßig mit der formerhaltenden stückweisen kubischen Methode interpoliert, um die Normal-zu-Normal-Intervalle (NN) zu erhalten, sie jedoch zu entfernen (nicht empfohlen) oder bei Bedarf eine andere Interpolationsmethode (linear, kubisch, nächster Nachbar, vorheriger/nächster Nachbar, Spline, kubische Faltung oder modifizierte kubische Akima-Methode) zu verwenden. Wenn mehrere EKG-Kanäle vorhanden sind, werden die RR-Intervalle für jeden Kanal geschätzt, und der Kanal mit der geringsten Anzahl von RR-Artefakten wird für die folgenden Schritte ausgewählt. - Scrollen Sie durch vergrößerte 30 s-Fenster der R-Peaks, um sie genauer zu betrachten, indem Sie die Pfeile nach rechts/links drücken. Wenn die Daten mehrere EKG/PPG-Kanäle enthalten, verwenden Sie den Kanal mit dem besten Signalqualitätsindex für die RR-Intervalle. BrainBeats unterstützt derzeit nicht sowohl EKG- als auch PPG-Signale gleichzeitig. Zu Tutorialzwecken haben wir ein Beispieldataset ausgewählt, das beide Datentypen enthält.
- Sobald das EKG-Signal fertig ist, filtert der BrainBeats-Bandpass die EEG-Daten bei 1-40 Hz standardmäßig mit einem nichtlinearen kausalen Minimum-Phasen-FIR-Filter, um die Verschmierungsaktivität zwischen Vor- und Nach-Herzschlag zu reduzieren, die Kausalität zu erhalten und unerwünschte Gruppenverzögerungen zu vermeiden44. Dies ist besonders wichtig für Benutzer, die die Periode vor dem Herzschlag untersuchen. Wenn der Tiefpassfilter auf einen Wert oberhalb der Netzfrequenz eingestellt ist (z. B. 80 Hz Tiefpass mit Netzfrequenz bei 50 Hz), verwenden Sie einen scharfen Kerbfilter, um das Leitungsrauschen zu entfernen. Die EEG-Daten werden dann mit dem REST-Algorithmus (am besten geeignet für die HEP-Analyse49) auf unendlich referenziert, es sei denn, es werden weniger als 30 Kanäle erkannt (in diesem Fall können sie nicht zuverlässig erneut referenziert werden, und es wird eine Warnung generiert, um den Benutzer darüber zu informieren).
- BrainBeats erkennt, entfernt und interpoliert dann abnormale EEG-Kanäle (Abbildung 3). Überprüfen Sie, ob die Standardparameter flache Linien größer als 5 s (clean_flatlines-Algorithmus), eine maximale Standardabweichung des hochfrequenten Rauschens von 10, eine Fensterlänge von 5 s (zur besseren Erfassung von langsamen Frequenzartefakten52), eine minimale Korrelation zwischen benachbarten Kanälen von 0,65 und ein maximal tolerierter Anteil von 33 % (clean_channels Algorithmus) sind.
HINWEIS: Die Anzahl der RANSAC-Samples ist standardmäßig auf 500 eingestellt, um die Konvergenz und Reproduzierbarkeit der Ablehnung fehlerhafter Kanäle zu erhöhen (obwohl dies die Berechnungszeit erhöht). - Fügen Sie als Nächstes R-Peaks als Ereignismarker in die EEG-Daten ein, um jeden Herzschlag zu markieren, und segmentieren Sie die Daten um diese Marker herum, ohne dass die Ausgangswerte entfernt werden müssen (gemäß den Richtlinien43,66; Abbildung 4). Da NN-Intervalle unterschiedlich lang sind und das EEG mit einer konstanten Länge segmentiert werden muss, schätzen Sie den minimalen Epochengrößen-Cutoff nach R-Peak-Ereignissen anhand des 5. Perzentils der Interbeat-Interval (IBI)-Daten (d. h. den Wert, unter den 5 % der kürzesten IBIs fallen, dargestellt als gestrichelte rote Linie in einem Histogramm; siehe Abbildung 5).
HINWEIS: Dieser 5. Perzentilwert ist ein guter Kompromiss, um so viele Epochen wie möglich zu erhalten und gleichzeitig sicherzustellen, dass sie nicht zu kurz sind, da der für die HEP/HEO-Analyse relevante Zeitraum 200-600 ms nach Heartbeat49,67 beträgt. - Segmentieren Sie EEG-Daten von -300 ms vor den R-Peaks bis zum 5. Perzentilwert nach R-Peak, wobei der R-Peak zum Zeitpunkt 0 ist. Lehnen Sie Epochen ab, die kürzer als 550 ms sind oder mehr als einen R-Peak enthalten (was das ERP/ERSP verzerren würde), gemäß den Richtlinien49,67. Epochen mit großen EEG-Artefakten werden unter Verwendung von RMS- (Mean-Square) und Signal-Rausch-Verhältnis (SNR) sowie der islier-Funktion von MATLAB erkannt (Abbildung 6). Künstliche Epochen werden entfernt.
- Führen Sie eine blinde Quellentrennung mit dem standardmäßigen Infomax-Algorithmus durch, wobei der Datenrang für die beste Leistung53 berücksichtigt wird. Falls gewünscht, wählen Sie den vorkonditionierten PICARD-Algorithmus (Independent Component Analysis) für die schnelle Berechnung54,54, indem Sie die Option Picard (schnell) wählen, oder den modifizierten Infomax-Algorithmus, indem Sie die Option Modifizierter Infomax (lang, aber replizierbar) für das Feld ICA-Methode wählen (obwohl dies wesentlich längere Rechenzeiten erfordert). Verwenden Sie das ICLabel-Plug-in56, um ICs automatisch zu klassifizieren, um Artefakte außerhalb des Gehirns zu extrahieren (okuläre Komponenten werden mit einer Zuverlässigkeit von 90 % entfernt, während Muskel-, Zeilenrauschen und Kanalrauschen mit einer Zuverlässigkeit von 99 % entfernt werden; Abbildung 7).
- Lassen Sie die Option Ausgaben visualisieren im ersten GUI-Fenster ausgewählt, um die endgültige EEG-Zeitreihe, den großen durchschnittlichen HEP (Abbildung 8 oben), den HEP für jeden Herzschlag (Abbildung 8 unten) und die großen durchschnittlichen Herzschlag-evozillationen (HEO; Abbildung 9). Untersuchen Sie HEOs in Bezug auf ereigniskorrelierte spektrale Störungen (ERSP, d.h. Änderungen der EEG-Leistung über Herzschläge; Abbildung 9 oben) und Phasenkohärenz zwischen Studien (ITC, d. h. Konsistenz des Phasenwinkels über Herzschläge; Abbildung 9 unten).
HINWEIS: ERSP wird unter Verwendung eines standardmäßigen 3-Zyklus-Wavelets berechnet (mit einem Hanning-konischen Fenster, Pad-Verhältnis von 2) und mit der Anzahl der Zyklen in den Wavelets, die für höhere Frequenzen verwendet werden, die langsam auf bis zu 20 % der Anzahl der Zyklen im entsprechenden FFT-Fenster bei der höchsten Frequenz (1 minus 0,8) anwächst. Dies steuert die Formen der einzelnen Fenster, die von der Funktion gemessen werden, und deren Formen in den resultierenden Zeit-/Häufigkeitsbereichen. Eine willkürliche Basislinie wird zu Illustrationszwecken entfernt, und ERSP wird für Frequenzen von 7 bis 25 Hz berechnet, um den in der Literatur beschriebenen typischen HEO-Effekt zu erfassen, nämlich 300 bis 450 ms nach dem Herzschlag im Alpha-Band (8-13 Hz) über frontozentrale Elektroden 17,67. Niedrigere Frequenzen können aufgrund der kurzen Epochengröße, die durch die Schwebungsintervalle definiert wird, nicht geschätzt werden. Nichtparametrische (Permutations-)Statistiken werden angewendet, um den HEO für einen p-Wert von 0,05 zu visualisieren, korrigiert um die Falscherkennungsrate (FDR, d. h. Fehler vom Typ 1 oder familienbezogener Fehler). Diese Diagramme werden zu Tutorial-Zwecken oder zur Analyse einzelner Versuche generiert. - Es werden Vorverarbeitungsdiagramme erstellt, um die verschiedenen Schritte zu visualisieren. Um es zu deaktivieren, deaktivieren Sie das Kontrollkästchen Vorverarbeitungen visualisieren. Die endgültigen EEG-Daten (bereinigt und segmentiert um die R-Peaks) enthalten keine EKG-Daten, da dies die ERP/ERSP-Analyse verzerren würde. Um den Herzkanal in der endgültigen Ausgabe beizubehalten, aktivieren Sie das Kontrollkästchen Herzkanal beibehalten.
HINWEIS: Pausieren Sie hier bei Bedarf, bevor Sie die nächste Datei verarbeiten (nächste Bedingung oder nächster Teilnehmer). - BrainBeats unterstützt die Verlaufsfunktion von EEGLAB. Geben Sie am Ende aller Vorgänge eegh in das Befehlsfenster von MATLAB ein, um die Befehlszeile zu drucken und alle oben genannten Schritte über eine einzige Befehlszeile zu wiederholen, mit den Parametern, die manuell in der GUI ausgewählt wurden, was eine einfache Automatisierung ermöglicht. Suchen Sie nach Vorverarbeitungsausgaben (z. B. Signalqualitätsindex der kardiovaskulären Zeitreihen, NN-Intervalle, entfernte EEG-Kanäle, Segmente und Komponenten usw.) in der EEGLAB-Struktur: EEG.brainbeats.preprocessings. Alle Parameter werden auch in EEG.brainbeats.parameters exportiert.
- Für fortgeschrittene Benutzer führen Sie alle oben genannten Schritte mit Standardparametern mit den folgenden Befehlszeilen aus (weitere Optionen finden Sie im Tutorial-Skript im BrainBeats-Repository):
eeglab; schließen; % EEGLAB ohne GUI starten
main_path = Dateiteile(welche('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('Dateiname','Datensatz.Set',
'filepath',fullfile(main_path, 'sample_data')); %Laden Sie das Beispieldataset
EEG = brainbeats_process(EEG, 'Analyse',
'hep','heart_signal','EKG', 'heart_channels',
{'EKG'},'clean_eeg',wahr); % Starten Sie BrainBeats auf 1. Ebene, um die Datei für die HEP-Analyse mit Standardparametern zu verarbeiten - In den obigen Schritten wurde HEP/HEO aus dem EKG-Signal durchgeführt. Führen Sie die folgenden Schritte für das PPG-Signal aus.
- Führen Sie in den folgenden Schritten die gleichen Vorgänge aus, jedoch mit einem PPG-Signal. Laden Sie denselben Datensatz erneut (siehe Schritt 1.5), da er von den vorherigen Operationen überschrieben wurde, und öffnen Sie die erste GUI von BrainBeats erneut, um die Hauptparameter auszuwählen. Klicken Sie auf Tools > BrainBeats >1. Ebene (Fachebene). Wählen Sie Heartbeat-evoked potentials (HEP) als auszuführende Analyse, PPG als Herzdatentyp aus und klicken Sie auf die Schaltfläche, um die Liste der Kanäle anzuzeigen, um den PPG-Kanal auszuwählen. Lassen Sie die Optionen Ausgaben visualisieren (empfohlen) und Ausgaben speichern ausgewählt und klicken Sie auf OK.
- Das zweite GUI-Fenster öffnet sich ähnlich wie in Schritt 2.2 . Der einzige Unterschied besteht darin, dass wir den Parameter jetzt so abstimmen können, dass er das PPG-Signal anstelle des EKG-Signals verarbeitet. Klicken Sie auf OK , um die Ausführung mit den Standardparametern durchzuführen.
- Es wird eine Warnmeldung angezeigt, in der Sie um Bestätigung gebeten werden, den erkannten zusätzlichen EKG-Kanal zu entfernen. Auch dies ist zu erwarten. Klicken Sie auf Ja. Standardmäßig verarbeitet die Toolbox das PPG-Signal vor, erkennt die Pulswellen, um die RR-Intervalle zu erhalten, identifiziert RR-Artefakte, falls vorhanden, und interpoliert sie (Abbildung 10). Schritte 2.7. bis 2.12. Es werden die gleichen Diagramme und Ausgaben generiert, jedoch basierend auf den R-Wellen, die vom PPG-Signal detektiert werden (siehe Abbildung 11 und Abbildung 12).
HINWEIS: R-Wellen werden anhand der Steigung des Signals innerhalb eines bestimmten Fensters erkannt. Potenzielle Impulse werden dann gekennzeichnet, wenn die Steigung einen dynamischen Schwellenwert überschreitet, der auf der Grundlage der Erkennungshistorie und der Signaleigenschaften angepasst wird. Der Algorithmus sucht dann innerhalb einer Augenschlussphase, um die maximale Steigung zu bestimmen, und anschließend wird der Beginn der Pulswelle durch Schwellenwerte bestimmt. Die R-Wellenspitzen werden als die Täler in der Nähe des Anfangs identifiziert und ihre Positionen werden aufgezeichnet. Der Algorithmus iteriert durch das gesamte Signal, passt die Detektionsschwellen kontinuierlich an und identifiziert R-Wellen-Spitzen, die dann zur Berechnung der RR-Intervalle verwendet werden. - Für fortgeschrittene Benutzer führen Sie alle oben genannten Schritte mit Standardparametern mit den folgenden Befehlszeilen aus (weitere Optionen finden Sie im Tutorial-Skript im BrainBeats-Repository):
eeglab; schließen; % EEGLAB ohne GUI starten
main_path = Dateiteile(welche('eegplugin_
BrainBeats.m')); cd(main_path);
EEG = pop_loadset('Dateiname','Datensatz.Set',
'filepath',fullfile(main_path, 'sample_data')); %Laden Sie das Beispieldataset
EEG = brainbeats_process(EEG, 'Analyse',
'hep','heart_signal','ppg', 'heart_channels',
{'PPG'},'clean_eeg',wahr); % Starten Sie BrainBeats auf 1. Ebene, um die Datei für die HEP-Analyse mit Standardparametern zu verarbeiten
3. Extraktion von EEG- und HRV-Merkmalen
- Laden Sie den gleichen Datensatz erneut (siehe Schritt 1.5; Klicken Sie auf Datei > Vorhandenen Datensatz laden > dataset.set auswählen), da er durch die vorherigen Operationen überschrieben wurde, und öffnen Sie die Haupt-GUI erneut, um die Hauptparameter auszuwählen (Schritt 2.1; Klicken Sie auf Tools > BrainBeats >1. Ebene). Wählen Sie EEG- und HRV-Merkmale extrahieren für den Analysetyp, EKG für den Herzsignaltyp und wählen Sie EKG in der Liste der Elektrodenetiketten. Klicken Sie auf OK.
- Das zweite GUI-Fenster öffnet sich wie in Schritt 2.2, jedoch mit anderen Parametern für die EEG-Vorverarbeitung und die Extraktion von HRV- und EEG-Merkmalen. Stellen Sie diese Parameter wie unten beschrieben ein. Klicken Sie auf OK , um mit den Standardparametern zu starten.
- Klicken Sie im Abschnitt HRV auf die Schaltfläche freq. Optionen , um die Methode zur Berechnung der HRV-Leistung auszuwählen (Standardeinstellung auf normalisiertes Lomb-Scargle-Periodogramm), die Fensterüberlappung (Standardeinstellung auf 25%) und für die Durchführung einer Normalisierung der zweiten Ebene (standardmäßig nicht festgelegt; siehe Hinweis unten für weitere Details).
- Klicken Sie im Abschnitt EEG-Funktionen auf die Schaltfläche freq. Optionen , um einige Parameter fein abzustimmen, wie z. B. den Gesamtfrequenzbereich, auf dem die spektrale Leistungsdichte berechnet werden soll (PSD; Standard = 1-40 Hz), die Einheiten (Dezibel, μV2/Hz oder normiert durch die Gesamtleistung), den Fenstertyp (Standard = Hamming), die Fensterüberlappung (Standard = 50%), die Fensterlänge (Standard = 2 s), und die Arten von Frequenzgrenzen für jedes Band.
HINWEIS: Die HRV-Leistung wird standardmäßig anhand des normalisierten Lomb-Scargle-Periodogramms berechnet, das kein erneutes Sampling erfordert (daher die ursprünglichen Informationen besser erhalten) und am besten mit ungleichmäßig abgetasteten Daten, fehlenden Daten und Rauschen (typisch für NN-Intervalle) umgehen kann68. Die normalisierte Version skaliert die Leistung um die Varianz des Signals und liefert Ergebnisse, die weniger empfindlich auf unterschiedliche Rauschpegel reagieren, sich mehr auf die relative Stärke periodischer Komponenten konzentrieren und über verschiedene Aufnahmen oder Probanden hinweg vergleichbarer sind. Weitere verfügbare Methoden sind das nicht-normalisierte Lomb-Scargle-Periodogramm, die Welch-Methode und die Fast-Fourier-Transformation (FFT). Für die Welch- und die FFT-Methode wird automatisch ein Resampling durchgeführt, um die erforderlichen regelmäßig abgetasteten Zeitreihen zu erstellen. Eine Normalisierung der zweiten Ebene kann angewendet werden, indem die Leistung jedes Frequenzbands durch die Gesamtleistung dividiert wird, wodurch ein intuitiveres Maß für den relativen Beitrag jeder Frequenzkomponente zur Gesamtleistung ermöglicht wird. Sie ist standardmäßig deaktiviert, da sie nur dann sinnvoll ist, wenn alle vier Bänder verfügbar sind und ein Signal von mindestens 24 Stunden erforderlich ist. Diese Algorithmen sind aus der Physionet Cardiovascular Signal Processing Toolbox46 adaptiert.
- Es wird dieselbe Warnmeldung angezeigt, in der Sie um Bestätigung gebeten werden, um den zusätzlichen PPG-Kanal zu entfernen, der erkannt wurde. Auch dies ist zu erwarten. Klicken Sie auf Ja. BrainBeats beginnt mit der Vorverarbeitung der EKG-Daten und der Extraktion der NN-Intervalle identisch, wie in Schritt 2.4. Anschließend extrahiert es Merkmale der Herzfrequenzvariabilität (HRV) aus den NN-Intervallen in den Bereichen Zeit (SDNN, RMSSD, pNN50), Frequenz (ULF, VLF, LF, HF, LF:HF-Verhältnis, Gesamtleistung) und nichtlinearen (Poincaré, phasengleichgerichtete Signalmittelung, Fuzzy-Entropie und fraktale Dimension).
HINWEIS: BrainBeats überprüft automatisch die Dateilänge, um sicherzustellen, dass die Mindestanforderungen erfüllt sind (z. B. erfordert die ULF-HRV-Leistung 24 Stunden Daten), sendet Warnmeldungen, wenn dies nicht der Fall ist, und exportiert diese Funktionen nicht, um unzuverlässige Schätzungen zu vermeiden. BrainBeats befolgt die Richtlinien und Empfehlungen zur Schätzung von HRV-Metriken 27,45. - BrainBeats verarbeitet die EEG-Daten wie in Schritt 2.7 vor. (siehe Abbildung 3). Große Artefakte werden in den kontinuierlichen Daten automatisch mit dem ASR-Algorithmus (Artifact Subspace Reconstruction Algorithmus)50,52 erkannt (das Standard-SD-Kriterium ist auf 30 festgelegt und verwendet 80 % des verfügbaren RAM, um die Geschwindigkeit zu erhöhen). Diese großen Artefakte werden aus den EEG-Daten entfernt (siehe Abbildung 13). Um diese Parameter in der GUI anzupassen, wählen Sie die Felder Schwellenwert zum Ablehnen fehlerhafter Segmente mit ASR und Verfügbarer RAM zur Verwendung für ASR aus.
HINWEIS: Das EEG und die kardiovaskulären Zeitreihen müssen für den Merkmalsmodus nicht zeitsynchronisiert werden, da die Merkmale für jedes Signal separat geschätzt werden. Somit können EEG-Artefakte direkt aus den EEG-Daten entfernt werden (in rot, Abbildung 13), im Gegensatz zum HEP-Modus, bei dem Epochen, die Artefakte enthalten, für beide Zeitreihen abgelehnt wurden, da die Zeitsynchronisation mit ms-Genauigkeit für diese Methode unerlässlich ist. - Führen Sie die ICA mit den gleichen Algorithmen und Parametern wie für HEP durch (siehe Schritt 2.11.), mit der Ausnahme, dass diesmal die Herzkomponenten entfernt werden, wenn sie mit einer Sicherheit von 99% erkannt werden (sie wurden für HEP/HEO konserviert, da wir keine relevanten kardialen Signale entfernen möchten).
- Das Kontrollkästchen Frequenzbereich ist in Schritt 3.2 aktiviert, daher extrahiert BrainBeats die folgenden Merkmale des Frequenzbereichs: die durchschnittliche spektrale Leistungsdichte (PSD) für die Frequenzbänder Delta (1-3 Hz), Theta (3-7 Hz), Alpha (8-13 Hz), Beta (13-30 Hz) und Gamma (30+ Hz), die individuelle Alpha-Frequenz (IAF) und die Alpha-Asymmetrie auf allen verfügbaren (symmetrischen) Elektrodenpaaren. Überprüfen Sie diese Funktionen.
HINWEIS: PSD, umgerechnet in Dezibel (dB), erleichtert den Vergleich der Ergebnisse zwischen Aufnahmen und Motiven. Die Frequenzgrenzen können auf die konventionellen Grenzen (z. B. vordefinierte 8-13 Hz für das Alpha-Band) oder auf die individualisierten Grenzen gesetzt werden, die aus der Verteilung der spektralen Leistungsdichte erfasst werden, um interindividuelle Unterschiede69 zu berücksichtigen (z. B. 7,3-12,6 Hz für das Alpha-Band). Der Algorithmus wurde für das Alpha-Band entwickelt und funktioniert für andere Bänder nicht so gut, insbesondere wenn keine Spitzen in der spektralen Leistungsverteilung vorhanden sind. Die individuelle Alpha-Frequenz (IAF) wird unter Verwendung des Alpha-Schwerpunkts geschätzt, um besser mit Split-Peaks oder mehrdeutigen Peaks umgehenzu können 69. Die Alpha-Asymmetrie wird nach Richtlinien berechnet (2-s-Hamming-Fenster mit 50% Überlappung, der Logarithmus der Alpha-Leistung aus dem linken Kanal minus Logarithmus der Alpha-Leistung aus dem rechten Kanal)47. Daher weisen positive Werte auf eine größere Alpha-Leistung von links als rechts hin und umgekehrt. Die Alpha-Asymmetrie kann normalisiert werden, indem die Alpha-Leistung von jeder Elektrode durch die Alpha-Leistung dividiert wird, die über alle Elektroden47 summiert wird. Die symmetrischen Paare werden unter Verwendung von Theta-Abständen erhalten, wobei die EEG-Daten Elektrodenkoordinaten enthalten müssen. - Das Kontrollkästchen Nichtlinearer Bereich ist in Schritt 3.2 aktiviert, BrainBeats extrahiert die Fuzzy-Entropie und die fraktale Dimension für jeden EEG-Kanal.
ANMERKUNG: Es wird angenommen, dass nichtlineare Domänenmerkmale nichtlineare, komplexe Dynamiken des Gehirns erfassen, die von spektralen Messungen übersehen werden und besonders vielversprechend für die Untersuchung von Wechselwirkungen zwischen verschiedenen Körpersystemen sind 70,71,72. Fuzzy-Entropie ist zuverlässiger und robuster als ihre Alternativen (Sample- und approximative Entropie), erfordert aber längere Rechenzeiten (insbesondere bei langen EEG-Zeitreihen mit hohen Abtastraten). Um dieses Problem zu beheben, werden EEG-Signale, die länger als 2 Minuten sind und eine Abtastrate von mehr als 100 Hz aufweisen, automatisch auf 90 Hz heruntergerechnet (oder dezimiert, wenn der Faktor keine ganze Zahl ist) (d. h. entsprechend einer Nyquist-Frequenz von 45 Hz, um dem Standard-Tiefpassfilter zu entsprechen und Zeilenrauschartefakte so weit wie möglich zu vermeiden). Darüber hinaus ist das parallele Rechnen bei der Schätzung von EEG-Merkmalen standardmäßig aktiviert, was die Rechenzeit reduziert, insbesondere wenn viele EEG-Kanäle verfügbar sind. - Wählen Sie in der ersten GUI die Option Ausgaben visualisieren (siehe Schritt 3.1.), um ein Diagramm zu erstellen, das die spektrale Leistungsdichte (PSD) für HRV- und EEG-Daten anzeigt (Abbildung 14) zusammen mit den Kopfhauttopographien, die einige EEG-Merkmale anzeigen (Abbildung 15).
HINWEIS: Sie können auch einige Vorverarbeitungsausgaben in EEG.brainbeats.preprocessing und alle verwendeten Parameter in EEG.brainbeats.parameters finden. Wir ermutigen die Nutzer, diese Ergebnisse in wissenschaftlichen Publikationen zu melden, um die Replizierbarkeit der Ergebnisse zu erhöhen. - Lassen Sie das Kontrollkästchen Ausgaben speichern im ersten GUI-Fenster aktiviert, um alle Features zu speichern, die in die EEGLAB .set-Datei in EEG.brainbeats.features exportiert wurden, und speichern Sie sie in einer .mat-Datei im selben Ordner, in den der Datensatz geladen wurde.
- BrainBeats unterstützt die Verlaufsfunktion von EEGLAB. Geben Sie am Ende aller Vorgänge eegh in das Befehlsfenster von MATLAB ein, um die Befehlszeile zu drucken, die es Ihnen ermöglicht, alle oben genannten Schritte über eine einzige Befehlszeile zu wiederholen, mit den Parametern, die manuell in der GUI ausgewählt wurden, was eine einfache Automatisierung und Replikation der Operationen ermöglicht.
- Für fortgeschrittene Benutzer führen Sie alle oben genannten Schritte mit dem folgenden Befehl aus:
eeglab; schließen; % EEGLAB ohne GUI starten
main_path = Dateiteile(welche('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('Dateiname','Datensatz.Set',
'filepath',fullfile(main_path, 'sample_data')); % Laden Sie das Beispieldataset
EEG = brainbeats_process(EEG, 'Analyse',
'Funktionen','heart_signal','EKG', 'heart_channels',{'EKG'},'clean_eeg',true); - In den vorherigen Schritten wurden HRV-Merkmale aus dem EKG-Signal extrahiert. Führen Sie die folgenden Schritte aus, um HRV-Merkmale aus dem PPG-Signal zu extrahieren (EEG-Merkmale sind identisch).
- Laden Sie den gleichen Datensatz erneut (Schritt 1.5.), da er von den Operationen überschrieben wurde, und öffnen Sie die Haupt-GUI erneut (Schritt 2.5.). Wählen Sie EEG- und HRV-Merkmale extrahieren für die Analyse und wählen Sie PPG für den Herzsignaltyp und PPG für den Kanalnamen. Klicken Sie auf OK.
- Das 2. GUI-Fenster zeigt nun die Parameter für die Vorverarbeitung von PPG und für die Extraktion der HRV- und EEG-Merkmale an. Klicken Sie auf OK , um die Ausführung mit den Standardparametern durchzuführen. Die Parameter werden in Schritt 2.17 beschrieben.
- Es erscheint eine Warnmeldung, in der Sie um Bestätigung gebeten werden, den erkannten EKG-Kanal zu entfernen. Dies ist zu erwarten, da die Toolbox nicht darauf ausgelegt ist, sowohl EKG als auch PPG gleichzeitig (oder andere Hilfskanäle) zu analysieren, und die Speicherung im Datensatz zu schwerwiegenden Fehlern führt (z. B. Entfernung von Artefakten, schlechte ICA-Zerlegung usw.). Klicken Sie auf Ja.
- BrainBeats verarbeitet das PPG-Signal vor und schätzt die NN-Intervalle wie in Schritt 2.5. Überprüfen Sie die extrahierten HRV-Merkmale aus den NN-Intervallen, genau wie in Schritt 3.2 . mit der Ausnahme, dass die NN-Intervalle jetzt aus dem PPG-Signal abgerufen wurden. Die EEG-Signale werden wie in Schritt 3.2 vorverarbeitet. BrainBeats zeichnet die PSD auf (Abbildung 16). Der einzige Unterschied besteht hier in der PSD, die aus den NN-Intervallen aus dem PPG im Gegensatz zum EKG geschätzt wird.
- Für fortgeschrittene Benutzer führen Sie alle oben genannten Schritte mit dem folgenden Befehl aus:
eeglab; schließen; % EEGLAB ohne GUI starten
main_path = Dateiteile(welche('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('Dateiname','Datensatz.Set','Dateipfad',
fullfile(main_path, 'sample_data')); %Laden Sie das Beispieldataset
EEG = brainbeats_process(EEG, 'Analyse',
'Funktionen','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. Extrahieren Sie Herzartefakte aus EEG-Signalen.
- Laden Sie den Beispieldatensatz (siehe Schritt 1.5.).
- Öffnen Sie das Hauptfenster der Benutzeroberfläche, indem Sie auf Tools > BrainBeats > 1. Ebene (Subjektebene) klicken und Herzartefakte aus EEG-Signalen extrahieren für den Analysetyp, EKG für den Herzsignaltyp und EKG in der Liste der Elektrodenbeschriftungen auswählen. Klicken Sie auf OK.
- Das 2. GUI-Fenster zeigt die Vorverarbeitungsparameter. Stellen Sie das Rauschen der Stromleitung auf 50 Hz (Europa) ein, bearbeiten Sie bei Bedarf das Konfidenzniveau, aktivieren Sie das Kontrollkästchen Boost-Modus (Beta) und klicken Sie auf OK , um mit den Standardparametern zu arbeiten, da die EEG-Signale aus dem Beispieldatensatz nicht vorverarbeitet werden.
HINWEIS: Das Konfidenzniveau für die Erkennung von Herzkomponenten ist standardmäßig auf 80 % eingestellt, was für einige Datensätze zu niedrig oder zu hoch sein kann. Eine Erhöhung dieses Wertes erhöht die Wahrscheinlichkeit, Herzkomponenten zu erkennen, verringert jedoch die Zuverlässigkeit dieser Erkennung. Der Boost-Modus (Beta) ist optional und zielt darauf ab, die Klassifikationsleistung zu verbessern, indem das Herzsignal in die EEG-Signale eingeschmiert wird. - Es wird eine Warnmeldung angezeigt, in der Sie um Bestätigung gebeten werden, den erkannten zusätzlichen PPG-Kanal zu entfernen. Dies ist zu erwarten, da die Toolbox nicht darauf ausgelegt ist, sowohl EKG als auch PPG gleichzeitig (oder andere Hilfskanäle) zu analysieren, und die Speicherung im Datensatz zu schwerwiegenden Fehlern führt (z. B. Entfernung von Artefakten, schlechte ICA-Zerlegung usw.). Klicken Sie auf Ja.
- Das EKG-Signal ist bandpassgefiltert, um langsame Frequenzdrifts unter 1 Hz und hochfrequentes Rauschen über 20 Hz zu entfernen (mit einem nicht-kausalen Nullphasen-FIR-Filter). Verarbeiten Sie die EEG-Signale wie in Schritt 3.4 vor.
- Führen Sie eine unabhängige Komponentenanalyse (ICA) mit dem vorkonditionierten ICA for Real Data Algorithmus (PICARD) durch. Um diese Option zu ändern, wählen Sie in Schritt 4.3 den Standard-Infomax-Algorithmus oder den modifizierten Infomax-Algorithmus für die Replikation aus der GUI aus. Klassifizieren Sie dann die unabhängigen Komponenten automatisch mit ICLabel. Wenn eine Komponente mit einer Konfidenz von 80 % als Herzkomponente klassifiziert wird, wird sie standardmäßig automatisch erkannt und aus den EEG-Daten extrahiert.
- Behalten Sie das Feld Visualize-Ausgaben in der ersten Haupt-GUI (Schritt 4.2.) bei, um die Kopfhauttopographie der entfernten Komponente (Abbildung 17 links) und die endgültige EEG-Zeitreihe (in blau, Abbildung 17 rechts) nach der Extraktion der Herzkomponente (in rot, Abbildung 17 rechts) zu visualisieren.
HINWEIS: Der EKG-Kanal wird für die Visualisierung beibehalten, um die Extraktion von EKG-bezogenen Komponenten zu bestätigen, aber er wird nach diesem Schritt entfernt, da er keine relevanten Informationen mehr enthält. - Führen Sie für fortgeschrittene Benutzer die folgenden Schritte mit den folgenden Befehlszeilen aus:
eeglab; schließen; % EEGLAB ohne GUI starten
main_path = Dateiteile(welche('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('Dateiname','Datensatz.Set','Dateipfad',
fullfile(main_path, 'sample_data'));
EEG = brainbeats_process(EEG,'Analyse','rm_heart',
'heart_signal','EKG', ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true);
Ergebnisse
Zunächst wurde das BrainBeats-Plugin verwendet, um EEG- und EKG-Daten vorzuverarbeiten, Artefakte zu identifizieren und zu entfernen sowie herzschlagevozierte Potentiale (HEP) und Oszillationen (HEO) zu analysieren. BrainBeats erkannte erfolgreich die RR-Intervalle aus dem EKG-Signal und einige RR-Artefakte (Abbildung 2). BrainBeats berichtete im Befehlsfenster auch, dass 11/305 (3,61%) der Heartbeats als Artefakte markiert und interpoliert wurden. Der durchschnittliche Signalqualitätsinde...
Diskussion
Kritische Schritte im Protokoll
Kritische Schritte werden in den Schritten 1.1-1.4 beschrieben. Warnungen und Fehlermeldungen sind an verschiedenen Stellen in der Toolbox implementiert, um den Benutzern zu helfen, zu verstehen, warum sie auf Probleme stoßen können (z. B. Elektrodenpositionen, die nicht in die EEG-Daten geladen sind, Dateilänge, die zu kurz ist, um ein zuverlässiges Maß für die ultraniederfrequente HRV zu berechnen, Signalqualität, die für eine zuverlässige Analyse zu niedrig ...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Das Institut für Noetische Wissenschaften unterstützte diese Forschung. Wir danken den Entwicklern der ursprünglichen Open-Source-Algorithmen, die angepasst wurden, um einige der Algorithmen von BrainBeats zu entwickeln.
Materialien
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Referenzen
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten