A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
BrainBeats כתוסף EEGLAB בקוד פתוח לניתוח משותף של אותות EEG ואותות לב וכלי דם
In This Article
Summary
ארגז הכלים של BrainBeats הוא תוסף EEGLAB בקוד פתוח שנועד לנתח במשותף אותות EEG ולב וכלי דם (ECG/PPG). הוא כולל הערכת פוטנציאלים מעוררי פעימות לב (HEP), ניתוח מבוסס תכונות וחילוץ חפצי לב מאותות EEG. הפרוטוקול יסייע בחקר יחסי גומלין מוח-לב באמצעות שתי עדשות (HEP ותכונות), וישפר את יכולת השחזור והנגישות.
Abstract
יחסי הגומלין בין המוח למערכת הלב וכלי הדם זוכים לתשומת לב מוגברת בשל הפוטנציאל שלהם לקדם את הבנתנו את הפיזיולוגיה האנושית ולשפר את התוצאות הבריאותיות. עם זאת, הניתוח הרב-מודאלי של אותות אלה מאתגר בשל היעדר הנחיות, עיבוד אותות סטנדרטיים וכלים סטטיסטיים, ממשקי משתמש גרפיים (GUI) ואוטומציה לעיבוד מערכי נתונים גדולים או הגדלת יכולת השחזור. חלל נוסף קיים בשיטות מיצוי סטנדרטיות של תכונות EEG והשתנות קצב לב (HRV), המערערות את האבחון הקליני או את החוסן של מודלים של למידת מכונה (ML). בתגובה למגבלות אלה, אנו מציגים את ארגז הכלים של BrainBeats. BrainBeats, המיושמת כתוסף EEGLAB בקוד פתוח, משלבת שלושה פרוטוקולים עיקריים: 1) פוטנציאלים מעוררי פעימות לב (HEP) ותנודות (HEO) להערכת יחסי גומלין בין מוח ללב נעולים בזמן בדיוק של אלפיות השנייה; 2) מיצוי תכונות EEG ו- HRV לבחינת קשרים/הבדלים בין מדדי מוח ולב שונים או לבניית מודלים חזקים מבוססי תכונות ML; 3) חילוץ אוטומטי של ממצאי לב מאותות EEG כדי להסיר כל זיהום קרדיווסקולרי פוטנציאלי בעת ביצוע ניתוח EEG. אנו מספקים הדרכה שלב אחר שלב ליישום שלוש שיטות אלה על מערך נתונים בקוד פתוח המכיל אותות EEG, ECG ו- PPG בו-זמנית של 64 ערוצים. משתמשים יכולים לכוונן בקלות פרמטרים כדי להתאים אישית את צרכי המחקר הייחודיים שלהם באמצעות ממשק המשתמש הגרפי (GUI) או שורת הפקודה. BrainBeats אמור להפוך את מחקר יחסי הגומלין בין המוח ללב לנגיש יותר וניתן לשחזור.
Introduction
במשך זמן רב, הגישה הרדוקציוניסטית שלטה במחקר המדעי בפיזיולוגיה ובקוגניציה האנושית. גישה זו כללה ניתוח תהליכים גופניים ומנטליים מורכבים למרכיבים קטנים וקלים יותר לניהול, מה שאיפשר לחוקרים להתמקד במערכות בודדות בבידוד. אסטרטגיה זו התעוררה בשל האתגרים בחקר הטבע המורכב והמקושר של הגוף והנפש האנושיים1. רדוקציוניזם סייע בהבנת תת-מערכות בודדות בבידוד, כגון הבהרת תפקידן של תעלות יונים ופוטנציאלי פעולה עבור תקשורת עצבית2 או לבבית3 . עם זאת, נותר פער משמעותי בהבנתנו כיצד מערכות מבודדות אלה מתקשרות בקנה מידה מרחבי וזמני גדול יותר. המסגרת הרב-מודאלית (אינטגרטיבית או אקולוגית) רואה בגוף האדם מערכת רב-ממדית מורכבת, שבה הנפש אינה נתפסת כתוצר של המוח אלא כפעילות של היצור החי, פעילות המשלבת את המוח בתפקודים היומיומיים של גוף האדם4. הגישות הרב-מודאליות והרדוקציוניסטיות אינן בלעדיות, בדיוק כפי שאיננו יכולים לחקור נוירון אחד ללא המוח כולו או המוח כולו ללא הבנת תכונות נוירונים בודדים. יחד, הם סוללים את הדרך להבנה מקיפה וסינרגטית יותר של בריאות האדם, פתולוגיה, קוגניציה, פסיכולוגיה ותודעה. השיטה הנוכחית נועדה להקל על החקירה הרב-מודאלית של יחסי הגומלין בין המוח ללב על ידי מתן ניתוח משותף של אלקטרואנצפלוגרפיה (EEG) ואותות קרדיווסקולריים, כלומר אלקטרוקרדיוגרפיה (ECG) ופוטופלטיסמוגרפיה (PPG). ארגז כלים זה, המיושם כתוסף EEGLAB ב- MATLAB, נותן מענה למגבלות מתודולוגיות קיימות ונעשה קוד פתוח כדי להקל על נגישות ושחזור בתחום המדעי. הוא מיישם את ההנחיות וההמלצות העדכניות ביותר בפרמטרי העיצוב וברירת המחדל שלו כדי לעודד משתמשים לעקוב אחר שיטות עבודה מומלצות ידועות. ארגז הכלים המוצע צריך להיות משאב רב ערך עבור חוקרים וקלינאים המעוניינים 1) לחקור פוטנציאלים מעוררי פעימות לב, 2) חילוץ תכונות מאותות EEG ו- ECG/PPG, או 3) הסרת ממצאי לב מאותות EEG.
חקר הלב-מוח
הקשר בין הלב למוח נחקר היסטורית באמצעות שיטות דימות מוחי כגון דימות תהודה מגנטית תפקודי (fMRI) וטומוגרפיית פליטת פוזיטרונים (PET). באמצעות כלים אלה, החוקרים הדגישו כמה אזורי מוח הקשורים לשליטה קרדיווסקולרית (למשל, מניפולציה של קצב הלב ולחץ דם5), הראו את ההשפעה של קצב הלב על אות BOLD6, או זיהו מסלולים פוטנציאליים במוח-גוף התורמים למחלות לב כליליות (כלומר, לחץ דם המעורר מתח7). בעוד שמחקרים אלה קידמו באופן משמעותי את הבנתנו את יחסי הגומלין המורכבים בין מערכת העצבים המרכזית (CNS) לבין תפקוד הלב וכלי הדם, טכניקות דימות מוחי אלה יקרות, זמינותן מוגבלת, והן מוגבלות לסביבות מעבדה מבוקרות, המגבילות את המעשיות שלהן ליישומים בעולם האמיתי ובקנה מידה גדול.
לעומת זאת, EEG ו- ECG/PPG הם כלים זולים וניידים יותר המציעים פוטנציאל לחקר אינטראקציות מוח-לב בסביבות ואוכלוסיות מגוונות יותר או על פני תקופות ארוכות יותר, ומספקים הזדמנויות חדשות. אק"ג מודד את האותות החשמליים הנוצרים על ידי כל פעימת לב כאשר הלב מתכווץ ונרגע באמצעות אלקטרודות המונחות על העור (בדרך כלל על החזה או הזרועות)8. PPG מודד שינויים בנפח הדם ברקמות כלי הדם (כלומר, זרימת הדם וקצב הדופק) באמצעות מקור אור (למשל, LED) וגלאי אור (בדרך כלל ממוקם על קצה האצבע, פרק כף היד או המצח), בהסתמך על האופן שבו הדם סופג יותר אור מאשר הרקמה שמסביב9. שתי השיטות מספקות מידע רב ערך על תפקוד הלב וכלי הדם, אך משרתות מטרות שונות ומציעות סוגי נתונים נפרדים. בדומה לאק"ג, EEG רושם את השדות החשמליים הנוצרים על-ידי הפעילות המסונכרנת של אלפי תאי עצב בקליפת המוח המתפשטים דרך המטריצה, הרקמות, הגולגולת והקרקפת החוץ-תאיות עד שהם מגיעים לאלקטרודות הממוקמות על פני הקרקפת10. ככזה, השימוש ב-EEG ובאק"ג/PPG טומן בחובו הבטחה גדולה לקידום הבנתנו את התהליכים הפיזיולוגיים, הקוגניטיביים והרגשיים העומדים בבסיס אינטראקציות מוח-לב והשלכותיהם על בריאות האדם ורווחתו. לכן, לכידת יחסי לב-מוח מ-EEG, אותות אק"ג/PPG באמצעות ארגז הכלים של BrainBeats עשויה להיות שימושית במיוחד בתחומים המדעיים הבאים: אבחון וחיזוי קליניים, למידת מכונה של ביג דאטה (ML), ניטור עצמי בעולם האמיתי11 והדמיית מוח/גוף ניידת (MoBI)12,13.
שתי גישות לניתוח משותף של אותות EEG ו- ECG
ישנן שתי גישות עיקריות לחקר אינטראקציות בין EEG לבין אותות קרדיווסקולריים:
הפוטנציאלים מעוררי פעימות הלב (HEP) בתחום הזמן: פוטנציאלים הקשורים לאירועים (ERP), והתנודות המעוררות פעימות לב (HEO) בתחום תדר הזמן: הפרעות ספקטרליות הקשורות לאירועים (ERSP) וקוהרנטיות בין-ניסויית (ITC). גישה זו בוחנת כיצד המוח מעבד כל פעימת לב. עם דיוק של אלפיות השנייה (ms), שיטה זו דורשת ששתי סדרות הזמן יהיו מסונכרנות באופן מושלם ופעימות הלב יסומנו באותות EEG. גישה זו זוכה להתעניינות בשנים האחרונות 14,15,16,17,18,19.
גישה מבוססת תכונות: גישה זו מחלצת תכונות EEG והשתנות קצב לב (HRV) מאותות רציפים ובוחנת קשרים ביניהם. זה נעשה באופן עצמאי עבור EEG (המכונה לעתים קרובות EEG כמותי או qEEG20), ECG 21,22,23, ו- PPG 24,25,26. גישה זו מציגה יישומים מבטיחים על ידי לכידת משתנים הקשורים למדינה ולתכונות כאחד. שימו לב שגם עבור אותות EEG וגם עבור אותות קרדיווסקולריים, ככל שההקלטה ארוכה יותר, כך משתנה התכונה 27,28,29 דומיננטי יותר. לפיכך, היישומים תלויים בפרמטרים ההקלטה. ניתוחים מבוססי תכונות צוברים עניין הולך וגובר, ומספקים מדדים כמותיים חדשים לחיזוי התפתחות הפרעות נפשיות ונוירולוגיות, תגובת טיפול או הישנות30,31,32,33,34,35. גישה זו משכנעת במיוחד עם מערכי נתונים גדולים בעולם האמיתי (למשל, מרפאה, ניטור מרחוק), אשר ניתן להשיג בקלות רבה יותר הודות לחידושים האחרונים בנוירוטכנולוגיה לבישה11. יישום פחות נחקר הוא זיהוי קשרים בין תכונות ספציפיות של המוח והלב, תוך הדגשת הפוטנציאל העומד בבסיס הדינמיקה של מערכת העצבים המרכזית. ניתן לחשב את השתנות הדופק (HRV) הן מאותות אק"ג והן מאותות PPG. הוא מספק מידע על מערכת העצבים האוטונומית (ANS) על ידי מדידת השינויים במרווחי הזמן בין פעימות הלב (כלומר, המרווחים הרגילים לנורמליים)27. פעילות סימפתטית מוגברת (SNS) (למשל, במהלך מתח או פעילות גופנית) בדרך כלל מפחיתה HRV, בעוד פעילות פאראסימפתטית (PNS) (למשל, במהלך הרפיה) מגבירה אותו. קצב נשימה איטי יותר בדרך כלל מגביר את HRV עקב פעילות מוגברת של מערכת העצבים הפארא-סימפתטית, במיוחד עבור הקלטות קצרות (<10 דקות)27. ציוני HRV גבוהים יותר מצביעים בדרך כלל על ANS גמיש יותר וניתן להסתגלות, בעוד HRV נמוך יותר יכול להצביע על מתח, עייפות או בעיות בריאות בסיסיות. הקלטות HRV ארוכות (כלומר, לפחות 24 שעות) מספקות פרוגנוזה מנבאת למצבים בריאותיים שונים, כולל מחלות לב וכלי דם, מתח, חרדה וכמה מצבים נוירולוגיים27. מדדים כמו לחץ דם, קצב לב או רמות כולסטרול מספקים מידע על מצב מערכת הלב וכלי הדם. לעומת זאת, HRV מוסיף היבט דינמי, המראה כיצד הלב מגיב ללחץ ומתאושש ממנו.
היתרונות של BrainBeats על פני השיטות הקיימות
בעוד שקיימים כלים, כפי שיסוקר להלן, לעיבוד אותות לב וכלי דם ואותות EEG בנפרד זה מזה, לא ניתן לנתח אותם במשותף. יתר על כן, רוב האמצעים הזמינים לעיבוד אותות קרדיווסקולריים כרוכים ברישוי יקר, אינם מאפשרים עיבוד אוטומטי (מועיל במיוחד עבור מערכי נתונים גדולים), בעלי אלגוריתמים קנייניים המונעים שקיפות ושחזור, או דורשים מיומנויות תכנות מתקדמות בכך שאינם מספקים ממשק משתמש גרפי (GUI)36. למיטב ידיעתנו, ארבעה ארגזי כלים של MATLAB בקוד פתוח תומכים בניתוח HEP/HEO באמצעות ממשק משתמש גרפי: ארגז הכלים של ערכת האק"ג37, צינור BeMoBIL38, תוסף HEPLAB EEGLAB39 וארגז הכלים CARE-rCortex40. בעוד HEPLAB, BeMoBIL וערכת אק"ג מסייעים לניתוח HEP על ידי זיהוי פעימות לב וסימונם באותות EEG, הם אינם מספקים ניתוח סטטיסטי או מוגבלים לתחום הזמן (כלומר, HEP). תוסף CARE-rCortex טיפל בבעיות אלה על ידי תמיכה באותות אק"ג ונשימה, ניתוח תחום תדר זמן, סטטיסטיקה ושיטות מתקדמות לנורמליזציה ותיקון בסיסיים המותאמות לניתוח HEP/HEO. עם זאת, היא משתמשת בשיטת בונפרוני לתיקון סטטיסטי של שגיאה מסוג 1 (כלומר, תוצאות חיוביות שגויות), שהיא שמרנית מדי ולא מבוססת פיזיולוגית עבור יישומי EEG, מה שמוביל לעלייה בשגיאות מסוג II (כלומר, שליליות שגויות)41. יתר על כן, ארגז הכלים אינו מציע גישה לשורת הפקודה עבור אוטומציה. לבסוף, מחקרים אחרונים ממליצים שלא להשתמש בשיטות תיקון בסיסיות 42,43,44, מכיוון שהן מפחיתות את יחס האות לרעש (SNR) והן מיותרות ובלתי רצויות מבחינה סטטיסטית.
כדי להתמודד עם מגבלות אלה, אנו מציגים את ארגז הכלים של BrainBeats, המיושם כיום כתוסף EEGLAB בקוד פתוח בסביבת MATLAB. הוא משלב את היתרונות הבאים על פני שיטות קודמות:
1) ממשק משתמש גרפי קל לשימוש ויכולות שורת פקודה (למתכנתים השואפים לבצע עיבוד אוטומטי). 2) אלגוריתמים, פרמטרים והנחיות מתוקפים לעיבוד אותות קרדיווסקולריים, כגון זיהוי פסגות R, אינטרפולציה של ממצאי RR וחישוב מדדי HRV (למשל, הנחיות להשתלת חלוניות, דגימה מחדש, נורמליזציה וכו '27,45,46). זה חשוב מכיוון ש- Vest et al. הראו כיצד הבדלים צנועים בשלבי עיבוד אלה יכולים להוביל לתוצאות שונות, ולתרום לחוסר יכולת השחזור והישימות הקלינית של מדדי HRV46. 3) אלגוריתמים מאומתים, פרמטרי ברירת מחדל והנחיות לעיבוד אותות EEG, כולל סינון וחלון44,47, הפניה מחדש48,49, הסרת תעלות חריגות וממצאים 50,51,52, פירוק אופטימלי של ICA וסיווג רכיבים בלתי תלויים 53,54,55,56. המשתמשים יכולים לכוונן את כל פרמטרי העיבוד המקדים או אפילו לעבד מראש את נתוני ה-EEG שלהם בשיטה המועדפת עליהם לפני השימוש בארגז הכלים שיתאים לצרכיהם (למשל, עם תוסף EEGLAB clean_rawdata50,52, צינור BeMoBIL38, צינור PREP57 וכו'). 4) פוטנציאלים מעוררי פעימות לב (HEP, כלומר, תחום זמן) ותנודות (HEO; הפרעות ספקטרליות הקשורות לאירועים בשיטות wavelet או FFT, וקוהרנטיות בין ניסויים זמינים באמצעות תוכנת EEGLAB סטנדרטית) מאותות ECG. סטטיסטיקה פרמטרית ולא פרמטרית עם תיקונים לשגיאות מסוג 1 זמינה באמצעות התוכנה הסטנדרטית של EEGLAB. סטטיסטיקות לא פרמטריות כוללות סטטיסטיקות פרמוטציה ותיקונים מרחביים-זמניים עבור השוואות מרובות (למשל, אשכולות מרחביים-זמניים או שיפור אשכולות ללא סף)58,59. משתמשים יכולים להשתמש בתוסף LIMO-EEG כדי ליישם מידול ליניארי היררכי, המביא בחשבון היטב שונות בתוך ובין נושאים ומיישם גישה חד-משתנית מסה נטולת הנחות עם בקרה חזקה עבור שגיאות מסוג I ו- II60,61. ניתן לבצע את הניתוחים הסטטיסטיים של נתוני HEP/HEO בתחומי הערוץ והרכיבים הבלתי תלויים. 5) ניתוח HEP/HEO ו-HRV מאותות PPG (לראשונה עבור HEP/HEO). 6) תומך בחילוץ משותף של תכונות EEG ו- HRV בפעם הראשונה. 7) ארגז הכלים מספק הדמיות נתונים שונות לבדיקת אותות בשלבי עיבוד נחוצים שונים ופלטים ברמת הנושא.
| שיטת | זיהוי פסגות R מאק"ג | זיהוי גלי R מ-PPG | HEP/HEO | תכונות EEG ו-HRV | הסרת חפצי לב מ- EEG | ממשק משתמש גרפי | שורת הפקודה |
| ערכת אק"ג | X | X | X | X | |||
| BeMoBIL | X | X | X | ||||
| חפלאב | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| פעימות מוח | X | X | X | X | X | X | X |
טבלה 1: חידושים שהביאה BrainBeats ביחס לשיטות קיימות ודומות.
מידע שיעזור לקוראים להחליט אם השיטה מתאימה להם
ארגז כלים זה מתאים לכל חוקר או קלינאי עם נתוני EEG ו- ECG/PPG. התוסף עדיין אינו תומך בייבוא אותות EEG ו-ECG/PPG מקבצים נפרדים (אם כי תכונה זו תהיה זמינה בקרוב). ארגז הכלים מתאים לכל מי שמטרתו לבצע ניתוח HEP/HEO, לחלץ תכונות EEG ו / או HRV בשיטות סטנדרטיות, או פשוט להסיר ממצאי לב מאותות EEG. ראו איור 1 עבור דיאגרמת בלוקים המסכמת את הזרימה הכוללת של BrainBeats ואת השיטות.
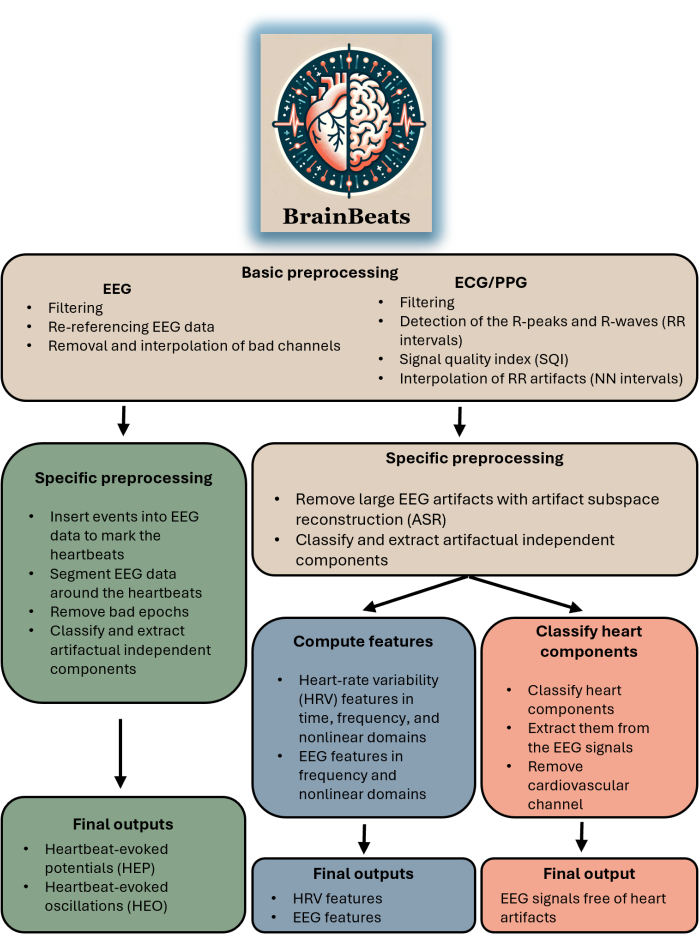
איור 1. דיאגרמת בלוקים המסכמת את הארכיטקטורה והזרימה הכוללת של BrainBeats. הפעולות הנפוצות בשלוש השיטות הן חומות. ניתוחים ספציפיים לפוטנציאלים מעוררי פעימות לב (HEP) ותנודות (HEO) הם ירוקים. פעולות ספציפיות לחילוץ תכונות EEG ו- HRV הן כחולות. ניתוחים ספציפיים להסרת חפצי לב מאותות EEG הם אדומים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
Protocol
הסכמה מדעת התקבלה מכל משתתף, וועדת האתיקה של האוניברסיטה הפדרלית של אורל אישרה את פרוטוקול הניסוי.
1. דרישות BrainBeats
- התקינו את MATLAB ואת EEGLAB במחשב. ניתן להוריד EEGLAB ב-https://github.com/sccn/eeglab ולפתוח את הרוכסן (או לשכפל אותו עבור משתמשי Git) בכל מקום במחשב. עיין בדף GitHub לקבלת פרטים נוספים על ההתקנה.
- הוסיפו את הנתיב לתיקייה EEGLAB בלוח הבית של MATLAB על ידי לחיצה על כפתור Set Path . בחר את תיקיית eeglab עם הקובץ unzipped ולחץ להציל > סגור.
- הפעל את EEGLAB על ידי הקלדת eeglab בחלון הפקודה של MATLAB.
- התקן את תוסף BrainBeats על ידי לחיצה על File > Manage EEGLAB extensions. הקלד brainbeats בסרגל החיפוש, בחר את תוסף BrainBeats ברשימה ולחץ על התקן/עדכן.
- טען את ערכת הנתונים לדוגמה לתוך EEGLAB. לחץ על קובץ > טען נתונים קיימים. נווט לתיקייה EEGLAB, עבור לתיקיית התוספים, עבור לתיקייה BrainBeats ופתח לתיקייה sample_data. בחר את הקובץ dataset.set.
הערה: ערכת נתונים זו תואמת לתת-032 (מצב מנוחה בעיניים פקוחות) מתוך ערכת נתונים מרובת נושאיםבקוד פתוח 62,63. זה נבחר מכיוון שהוא מכיל EEG בו זמנית (64 ערוצים), אק"ג (ערוץ אחד) ו- PPG (ערוץ אחד), בקצב דגימה של 1000 הרץ. אותות א.ק.ג. ו-PPG נאספו באמצעות קלטי העזר של מערכת actiCHamp. נתוני EEG נרשמו עם אלקטרודות פעילות שהונחו על פי מערכת 10-20, עם FCz כייחוס המקוון ו- Fpz כאלקטרודת הקרקע, ועם העכבה שנשמרה מתחת ל- 25 kOm. אק"ג נרשם באמצעות אלקטרודה פעילה אחת שהונחה על שורש כף יד ימין, אלקטרודת הייחוס על שורש כף יד שמאל, והקרקע על האמה הפנימית השמאלית במרחק של 3 ס"מ מהמרפק. PPG נרשם מהאצבע המורה השמאלית. נתוני EEG, ECG ו-PPG מסונכרנים בזמן מכיוון שהם הוקלטו בו זמנית עם אותו מגבר. ראה הפניות 62,63 לפרטים נוספים. - סמן את תיבת הסימון שמור פלטים כדי לשמור הכל בקובץ .set המתאים באותו מיקום כמו הקובץ המקורי שנטען ל- EEGLAB.
הערה: עבור ערכת לימוד זו, מיזגנו את נתוני EEG, ECG ו- PPG לערכת נתונים אחת של EEGLAB, טענו את קואורדינטות האלקטרודות של שיטת אלמנט הגבול התלת-ממדי (BEM) ודגמנו את האות ל- 250 הרץ כדי להפחית את משקל הקובץ (להורדת משתמש) ולהאיץ את זמן המחשוב. מכיוון שלמערך הנתונים הזה לא היה ערוץ EEG חריג, שינינו באופן מלאכותי את ערוץ TP9 כדי להמחיש את אלגוריתם זיהוי והסרת הערוצים הגרועים של BrainBeats. באופן דומה, אנו מדמים באופן מלאכותי חפץ EEG גדול בתחילת הקובץ וחפץ דמוי שריר בתדר גבוה בתעלות הרקתיות בין 3-6 שניות כדי להמחיש את תכונת הסרת החפץ של BrainBeats.
2. פוטנציאלים מעוררי פעימות לב (HEP) ותנודות (HEO)
- פתח את ממשק המשתמש הכללי (GUI) הראשון של BrainBeats כדי לבחור את הפרמטרים העיקריים. ב- EEGLAB, לחץ על כלים > BrainBeats > 1st level (רמת נושא). בחר פוטנציאלים מעוררי פעימות לב HEP כניתוח להפעלה, אק"ג כסוג נתוני הלב, ולחץ על הכפתור כדי להציג את רשימת הערוצים כדי לבחור את ערוץ האק"ג שכותרתו אק"ג (או הקלד אותו ישירות בתיבת הטקסט). השאר את האפשרויות הצג פלט באופן חזותי (מומלץ) ושמור פלטים מסומנים ולחץ על אישור.
- חלון GUI שני מוקפץ בהתבסס על האפשרויות הקודמות (כלומר, ניתוח HEP עם נתוני אק"ג). בחר פרמטרים של עיבוד מקדים עבור אותות EEG ו- ECG. בטל את העיבוד המקדים על-ידי ביטול הסימון של התיבות Preprocess ECG ו - Preprocess EEG כדי לראות אם הנתונים כבר עובדו מראש לפני הפעלת BrainBeats. שנה את רעש קו החשמל ל- 50 הרץ בסעיף EEG טרום תהליך מכיוון שנתוני EEG אלה נרשמו ברוסיה. לחץ על אישור כדי להפעיל.
- תופיע הודעת אזהרה המבקשת אישור להסרת ערוץ PPG שזוהה. הסיבה לכך היא שארגז הכלים אינו מיועד לנתח הן אק"ג והן PPG בו זמנית (או ערוצי עזר אחרים) והשארתו במערך הנתונים תוביל לטעויות חמורות (למשל, הסרת חפצים, פירוק לקוי של האגודה למלחמה בסרטן וכו'). לחץ על כן. BrainBeats מתחילה לבצע כמה בדיקות, להגדיר כמה פרמטרים ברירת מחדל, ומפריד את האק"ג מנתוני EEG כדי לעבד מראש את אות האק"ג ולחשב את מרווחי RR.
- סדרות הזמן ECG ו-RR מעובדות מראש באמצעות אלגוריתמים מאומתים מארגז הכלים Physionet Cardiovascular Signal46,64 המותאמים לעיצוב הנתונים של BrainBeats, להגברת הבהירות, לכוונון הפרמטרים ולזמן המחשוב (ראו הפניות לאימות האלגוריתמים). התוסף מפיק את מרווחי RR, חותמות זמן, אות א.ק.ג מסונן, מדדי פסגות R ודופק (HR). כוונן פרמטרים אלה באמצעות ממשק המשתמש הגרפי או שורת הפקודה.
הערה: אות האק"ג מסונן באמצעות פסים באמצעות מסנן מותאם אישית ומאומת (1-30 הרץ) וסורק את האות כדי לזהות את קומפלקס QRS ואת פסגות ה-R באמצעות שיטת Pan-Tompkins (P&T)65, תוך יישום כמה פעולות עיבוד אותות, כולל בידול, ריבוע, אינטגרציה והחלקה לקבלת הביצועים הטובים ביותר. סף האנרגיה P&T מוערך בהתבסס על קצב הדגימה וערכי האק"ג המוחלקים כדי למנוע הפרעה מבליטות גדולות. אם השתנות מרווח RR עולה על פי 1.5 מהחציון, הוא מחפש פסגות שהוחמצו. סימן השיא הממוצע R מחושב על פני מקטעים של 30 שניות, ונקודות השיא מעודנות באמצעות בדיקת תקופה עקשנית, ניהול תנאי קו שטוח והבטחת זיהוי עקבי. - לאחר מכן, BrainBeats מזהה מרווחי RR חריגים או קפיצות בתוך מרווחי RR באמצעות חיפוש קדימה-אחורה וספים פיזיולוגיים. מדד איכות האות (SQI) מחושב46; בדוק זאת אם המערכת מציגה אזהרות כאשר יותר מ -20% מסדרת RR times מכילה ממצאים RR (מחוץ למגבלות פיזיולוגיות או עם SQI מתחת ל -0.9). תרשים מציג את אות האק"ג המסונן, פסגות R מזוהות, מרווחי NN וארטיפקטים שעברו אינטרפולציה (ראו איור 2).
הערה: תוצרי RR עוברים אינטרפולציה כברירת מחדל באמצעות השיטה המעוקבת משמרת הצורה כדי לקבל את המרווחים הרגילים לנורמליים (NN) אך להסיר אותם (לא מומלץ) או להשתמש בשיטת אינטרפולציה אחרת (ליניארית, מעוקבת, השכן הקרוב ביותר, השכן הקודם/הבא, ספלין, פיתול מעוקב או אקימה מעוקבת שונה) במידת הצורך. כאשר קיימים מספר ערוצי אק"ג, מרווחי RR מוערכים עבור כל אחד מהם, והערוץ עם המספר הנמוך ביותר של תוצרי RR נבחר עבור השלבים הבאים. - גלול בין חלונות מוגדלים של 30 שניות של פסגות R לבדיקה מדוקדקת יותר על-ידי לחיצה על החצים ימינה/שמאלה. אם הנתונים מכילים מספר ערוצי אק"ג/PPG, השתמש בערוץ עם מדד איכות האות הטוב ביותר עבור מרווחי RR. BrainBeats אינו תומך הן באותות אק"ג והן באותות PPG בו זמנית כרגע. למטרות ערכת לימוד, בחרנו ערכת נתונים לדוגמה המכילה את שני סוגי הנתונים.
- לאחר סיום השימוש באות האק"ג, BrainBeats bandpass מסנן את נתוני ה-EEG ב-1-40 הרץ באמצעות מסנן FIR סיבתי לא ליניארי בעל פאזה מינימלית כברירת מחדל כדי להפחית את פעילות המריחה בין תקופות לפני ואחרי פעימות הלב, לשמר סיבתיות ולמנוע עיכובים קבוצתיים בלתי רצויים44. זה חשוב במיוחד עבור משתמשים הבוחנים את התקופה שלפני פעימות הלב. אם מסנן המעבר הנמוך מוגדר לערך מעל תדר קו החשמל (לדוגמה, מעבר נמוך של 80 הרץ עם תדר קו חשמל של 50 הרץ), השתמש במסנן חריץ חד כדי להסיר את לכלוץ רעש הקו. לאחר מכן נתוני EEG מופנים מחדש לאינסוף באמצעות אלגוריתם REST (המתאים ביותר לניתוח HEP49), אלא אם כן מזוהים פחות מ-30 ערוצים (ובמקרה זה לא ניתן להפנות אליהם מחדש באופן אמין, ונוצרת אזהרה כדי ליידע את המשתמשים).
- לאחר מכן, BrainBeats מזהה, מסיר ומבצע אינטרפולציה של ערוצי EEG חריגים (איור 3). בדקו שהפרמטרים המוגדרים כברירת מחדל הם קווים שטוחים גדולים מ-5 שניות (אלגוריתם clean_flatlines), סטיית תקן מקסימלית לרעש בתדר גבוה של 10, אורך חלון של 5 שניות (כדי ללכוד טוב יותר תוצרים בתדר איטי52), מתאם מינימלי בין ערוצים שכנים של 0.65 וחלק נסבל מקסימלי של 33% (אלגוריתם clean_channels).
הערה: מספר דגימות RANSAC מוגדר ל- 500 כברירת מחדל כדי להגדיל את ההתכנסות והשכפול של דחיית ערוצים פגומים (אם כי זה מאריך את זמן החישוב). - לאחר מכן, הוסף פסגות R כסמני אירוע לנתוני EEG כדי לסמן כל פעימת לב ולפלח את הנתונים סביב סמנים אלה ללא הסרה בסיסית (לפי הנחיות43,66; איור 4). מכיוון שלמרווחי NN יש אורכים שונים ויש לפלח EEG באורך קבוע, הערך את חיתוך גודל התקופה המינימלי לאחר אירועי שיא R באמצעות האחוזוןהחמישי של נתוני מרווח הפעימות (IBI) (כלומר, הערך שמתחתיו נופלים 5% מ- IBIs הקצרים ביותר, המוצג כקו אדום מקווקו בהיסטוגרמה; ראה איור 5).
הערה: ערךאחוזון 5 זה הוא פשרה טובה כדי לשמר תקופות רבות ככל האפשר תוך הבטחה שהן לא קצרות מדי מכיוון שתקופת הריבית לניתוח HEP/HEO היא 200-600 אלפיות השנייה לאחר פעימות הלב49,67. - פלח נתוני EEG מ- -300 מילישניות לפני פסגות R לערךאחוזון 5 לאחר שיא R, כאשר שיא R בזמן 0. דחה תקופות קצרות מ- 550 אלפיות השנייה או המכילות יותר משיא R אחד (מה שעלול להטות את ה- ERP/ERSP), לפי הנחיות49,67. תקופות המכילות תוצרי EEG גדולים מזוהות באמצעות מדדי שורש-ממוצע-ריבוע (RMS) ויחס אות לרעש (SNR) והפונקציה isoutlier של MATLAB (איור 6). תקופות ארטיפטריות מוסרות.
- בצע הפרדת מקורות עיוורת באמצעות אלגוריתם ברירת המחדל של Infomax, תוך התחשבות בדירוג הנתונים לקבלת הביצועים הטובים ביותר53. אם תרצה, בחר באלגוריתם ניתוח רכיבים עצמאיים מותנה מראש (PICARD) לחישוב מהיר54,54 על ידי בחירה באפשרות Picard (מהיר) או אלגוריתם Infomax שהשתנה על ידי בחירת האפשרות Modified Infomax (ארוך אך ניתן לשכפול) עבור שיטת ICA בשדה (אם כי זה כרוך בזמני חישוב ארוכים בהרבה). השתמש בתוסף ICLabel56 כדי לסווג באופן אוטומטי מעגלים משולבים כדי לחלץ ממצאים שאינם מוחיים (רכיבים עיניים מוסרים בביטחון של 90%, בעוד שרעשי שרירים, קווים ורעשי ערוץ מוסרים בביטחון של 99%; איור 7).
- השאר את האפשרות הצג פלט באופן חזותי שנבחר בחלון ממשק המשתמש הגרפי הראשון כדי להציג באופן חזותי את סדרת הזמן הסופית של EEG, את ממוצע ה- HEP הכולל (איור 8 למעלה), HEP עבור כל פעימת לב (איור 8 למטה) ואת התנודות המתעוררות בדופק הממוצע הגדול (HEO; איור 9). לבחון HEOs במונחים של הפרעות ספקטרליות הקשורות לאירועים (ERSP, כלומר, שינויים בעוצמת EEG על פני פעימות הלב; איור 9 למעלה) וקוהרנטיות שלב בין ניסויים (ITC, כלומר, עקביות זווית הפאזה על פני פעימות הלב; איור 9 למטה).
הערה: ERSP מחושב באמצעות wavelet ברירת מחדל של 3 מחזורים (עם חלון מחודד Hanning, יחס pad של 2) ועם מספר המחזורים בגלי המשמשים לתדרים גבוהים יותר המתרחב באיטיות עד 20% ממספר המחזורים בחלון FFT המקביל בתדר הגבוה ביותר שלו (1 פחות 0.8). פעולה זו שולטת בצורות של החלונות הבודדים הנמדדים על-ידי הפונקציה ובצורות שלהם בחלוניות הזמן/תדירות המתקבלות. קו בסיס שרירותי מוסר למטרות המחשה, ו- ERSP מחושב עבור תדרים 7-25 הרץ כדי ללכוד את אפקט HEO הטיפוסי המתואר בספרות, כלומר 300-450 מילישניות לאחר פעימות לב בפס אלפא (8-13 הרץ) מעל אלקטרודות פרונטו-מרכזיות17,67. לא ניתן להעריך תדרים נמוכים יותר בשל גודל התקופה הקצרה המוגדר על ידי מרווחי הפעימות. סטטיסטיקה לא פרמטרית (פרמוטציה) מיושמת כדי להמחיש את ה- HEO עבור ערך p של 0.05, מתוקן עבור שיעור גילוי שגוי (FDR, כלומר, שגיאה מסוג 1 או שגיאה מבחינת המשפחה). חלקות אלה נוצרות למטרות הדרכה או ניתוח ניסויים בודדים. - עלילות עיבוד מראש נוצרות כדי להמחיש את השלבים השונים. כדי לבטל אפשרות זו, בטל את סימון התיבה הצג באופן חזותי תהליכים מקדימים. נתוני ה-EEG הסופיים (מנוקים ומפולחים סביב פסגות ה-R) אינם כוללים את נתוני האק"ג מכיוון שהם מטים את ניתוח ה-ERP/ERSP. כדי לשמר את ערוץ הלב בפלט הסופי, סמן את התיבה שמור על ערוץ הלב.
הערה: השתהה כאן במידת הצורך לפני עיבוד הקובץ הבא (התנאי הבא או המשתתף הבא). - BrainBeats תומכת בפונקציית ההיסטוריה של EEGLAB. בסוף כל הפעולות, הקלד eegh בחלון הפקודות של MATLAB כדי להדפיס את שורת הפקודה כדי לחזור על כל השלבים לעיל באמצעות שורת פקודה אחת, עם הפרמטרים שנבחרו ידנית בממשק המשתמש הגרפי, המאפשר אוטומציה קלה. מצא פלטי עיבוד מקדים (למשל, מדד איכות האות של סדרת הזמן הקרדיווסקולרית, מרווחי NN, ערוצי EEG שהוסרו, מקטעים ורכיבים וכו ') במבנה EEGLAB: EEG.brainbeats.preprocessings. כל הפרמטרים מיוצאים גם ב- EEG.brainbeats.parameters.
- עבור משתמשים מתקדמים, בצע את כל השלבים לעיל עם פרמטרי ברירת המחדל באמצעות שורות הפקודה הבאות (עיין בסקריפט ערכת הלימוד במאגר BrainBeats לקבלת אפשרויות נוספות):
EEGLAB; קרוב; % השקת EEGLAB ללא ממשק המשתמש הגרפי
main_path = fileparts(which('eegplugin_BrainBeats.m')); תקליטור(main_path);
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); %טען את ערכת הנתונים לדוגמה
EEG = brainbeats_process(EEG,'ניתוח',
'hep','heart_signal','ECG', 'heart_channels',
{'אק"ג'},'clean_eeg',נכון); %הפעל את BrainBeats 1 st level כדי לעבד את הקובץ לניתוח HEP עם פרמטרי ברירת מחדל - השלבים לעיל בוצעו HEP/HEO מאות האק"ג. בצע את השלבים הבאים עבור אות PPG.
- בשלבים הבאים, בצע את אותן פעולות אך באמצעות אות PPG. טען שוב את אותו מערך נתונים (ראה שלב 1.5) מאז שהפעולות הקודמות החליפו אותו, ופתח שוב את ממשק המשתמש הגרפי הראשון של BrainBeats כדי לבחור את הפרמטרים העיקריים. לחץ על כלים > BrainBeats > רמהראשונה (רמת נושא). בחר פוטנציאלים מעוררי פעימות לב (HEP) כניתוח להפעלה, PPG כסוג נתוני הלב, ולחץ על הכפתור כדי להציג את רשימת הערוצים לבחירת ערוץ PPG. השאר את האפשרויות הצג פלט באופן חזותי (מומלץ) ושמור פלטים מסומנים ולחץ על הלחצן 'אשר'.
- חלון ממשק המשתמש הגרפי השני מוקפץ באופן דומה לשלב 2.2 . ההבדל היחיד הוא שאנו יכולים כעת לכוונן את הפרמטר כדי לעבד את אות PPG במקום את אות האק"ג. לחץ על אישור כדי לפעול עם פרמטרי ברירת המחדל.
- מופיעה הודעת אזהרה המבקשת אישור להסרת ערוץ האק"ג הנוסף שזוהה. שוב, זה צפוי. לחץ על כן. כברירת מחדל, ארגז הכלים יעבד מראש את אות ה-PPG, יזהה את גלי הדופק כדי לקבל את מרווחי RR, יזהה ממצאים של RR, אם בכלל, ויבצע אינטרפולציה ביניהם (איור 10). שלבים 2.7. ל-2.12. מבוצעים, ואותן חלקות ויציאות נוצרות אך מבוססות על גלי R שזוהו מאות PPG (ראו איור 11 ואיור 12).
הערה: גלי R מזוהים באמצעות שיפוע האות בחלון שצוין. פולסים פוטנציאליים מסומנים לאחר מכן כאשר השיפוע חורג מסף דינמי, אשר מותאם בהתבסס על היסטוריית הזיהוי ומאפייני האות. לאחר מכן האלגוריתם מחפש בתוך תקופת עצימת עיניים כדי לאתר את השיפוע המקסימלי, ולאחר מכן, תחילת גל הדופק נקבעת באמצעות סף. פסגות גלי ה-R מזוהות כעמקים הסמוכים לתחילתו, ומיקומם מתועד. האלגוריתם חוזר על האות כולו, מתאים ברציפות את ספי הזיהוי ומזהה פסגות גל R, המשמשות לאחר מכן לחישוב מרווחי RR. - עבור משתמשים מתקדמים, בצע את כל השלבים לעיל עם פרמטרי ברירת המחדל באמצעות שורות הפקודה הבאות (עיין בסקריפט ערכת הלימוד במאגר BrainBeats לקבלת אפשרויות נוספות):
EEGLAB; קרוב; % השקת EEGLAB ללא ממשק המשתמש הגרפי
main_path = fileparts(which('eegplugin_
BrainBeats.m')); תקליטור(main_path);
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); %טען את ערכת הנתונים לדוגמה
EEG = brainbeats_process(EEG,'ניתוח',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',true); %הפעל את BrainBeats 1 st level כדי לעבד את הקובץ לניתוח HEP עם פרמטרי ברירת מחדל
3. חילוץ תכונות EEG ו- HRV
- טען שוב את אותה ערכת נתונים (ראה שלב 1.5; לחץ על קובץ > טען ערכת נתונים קיימת > בחר dataset.set) מכיוון שהוא הוחלף על ידי הפעולות הקודמות ופתח שוב את ממשק המשתמש הגרפי הראשי כדי לבחור את הפרמטרים העיקריים (שלב 2.1; לחץ על כלים > BrainBeats > 1st level). בחר חלץ תכונות EEG ו- HRV עבור סוג הניתוח, אק"ג עבור סוג אות הלב ובחר אק"ג ברשימת תוויות האלקטרודות. לחץ על אישור.
- חלון ה- GUI השני צץ כמו בשלב 2.2 אך עם פרמטרים שונים עבור עיבוד מקדים של EEG וחילוץ תכונות HRV ו- EEG. הגדר פרמטרים אלה כמתואר להלן. לחץ על אישור כדי להפעיל עם פרמטרי ברירת מחדל.
- בקטע HRV, לחץ על כפתור freq. אפשרויות לבחירת השיטה לחישוב כוח HRV (ברירת המחדל מוגדרת לפריודוגרמה מנורמלת של Lomb-Scargle), חפיפת החלון (ברירת המחדל מוגדרת ל- 25%), ולביצוע נורמליזציה ברמה שנייה (לא מוגדרת כברירת מחדל; ראה הערה להלן לפרטים נוספים).
- במקטע תכונות EEG, לחץ על לחצן freq. אפשרויות לכוונון עדין של פרמטרים מסוימים, כגון טווח התדרים הכולל שבו יש לחשב את הצפיפות הספקטרלית של הספק (PSD; ברירת מחדל = 1-40 הרץ), היחידות (דציבלים, μV2/Hz, או מנורמל בהספק כולל), סוג החלון (ברירת מחדל = hamming), חפיפת החלון (ברירת מחדל = 50%), אורך החלון (ברירת מחדל = 2 שניות), וסוגי גבולות התדרים עבור כל רצועה.
הערה: עוצמת HRV מחושבת כברירת מחדל באמצעות פריודוגרמה מנורמלת של Lomb-Scargle, שאינה דורשת דגימה מחדש (ולכן שומרת טוב יותר על מידע מקורי) ומטפלת בצורה הטובה ביותר בנתונים שנדגמו באופן לא אחיד, נתונים חסרים ורעש (אופייני עם מרווחי NN)68. הגרסה המנורמלת משנה את העוצמה לפי השונות של האות, ומספקת תוצאות רגישות פחות לרמות רעש משתנות, ממוקדות יותר בחוזק היחסי של רכיבים מחזוריים, וניתנות להשוואה רבה יותר בין הקלטות או נושאים שונים. שיטות אחרות הזמינות כוללות פריודוגרמה לא מנורמלת של לומב-סקרגל, שיטת וולש והתמרת פורייה מהירה (FFT). דגימה מחדש מבוצעת באופן אוטומטי עבור שיטות Welch ו- FFT כדי ליצור את סדרות הזמן הדרושות שנדגמו באופן קבוע. נורמליזציה ברמה שנייה יכולה להיות מיושמת על ידי חלוקת הכוח של כל רצועת תדרים בעוצמה הכוללת, מתן מדד אינטואיטיבי יותר של התרומה היחסית של כל רכיב תדר להספק הכולל. הוא מושבת כברירת מחדל מכיוון שהוא משמעותי רק כאשר כל ארבעת הפסים זמינים, ודורשים לפחות 24 שעות של אות. אלגוריתמים אלה מותאמים מארגז הכלים לעיבוד אותות קרדיווסקולרייםPhysionet 46.
- אותה הודעת אזהרה מופיעה, ומבקשת אישור להסיר את ערוץ PPG הנוסף שזוהה. שוב, זה צפוי. לחץ על כן. BrainBeats תתחיל לעבד מראש את נתוני האק"ג ולחלץ את מרווחי ה-NN באופן זהה, כמו בשלב 2.4. לאחר מכן, הוא מחלץ תכונות השתנות קצב לב (HRV) ממרווחי NN בזמן (SDNN, RMSSD, pNN50), תדר (ULF, VLF, LF, HF, LF:HF יחס, הספק כולל) ולא ליניארי (פואנקרה, ממוצע אות מתוקן פאזה, אנטרופיה מטושטשת וממד פרקטלי).
הערה: BrainBeats בודקת באופן אוטומטי את אורך הקובץ כדי לוודא עמידה בדרישות המינימום (לדוגמה, הספק ULF-HRV דורש 24 שעות של נתונים), שולחת הודעות אזהרה אם לא, ואינה מייצאת תכונות אלה כדי למנוע הערכות לא אמינות. BrainBeats עוקבת אחר הנחיות והמלצות להערכת מדדי HRV27,45. - BrainBeats מעבד מראש את נתוני EEG כמו בשלב 2.7 . (ראה איור 3). תוצרים גדולים מזוהים באופן אוטומטי בנתונים הרציפים באמצעות אלגוריתם שחזור תת-מרחב (ASR)50,52 (קריטריון SD המוגדר כברירת מחדל ל-30 ושימוש ב-80% מה-RAM הזמין להגברת המהירות). ממצאים גדולים אלה מוסרים מנתוני ה-EEG (ראו איור 13). כדי להתאים פרמטרים אלה בממשק המשתמש הגרפי, בחר את השדות סף כדי לדחות מקטעים פגומים עם ASR ו- RAM זמין לשימוש עבור ASR.
הערה: סדרות הזמן EEG ו-Cardiovascular אינן צריכות להיות מסונכרנות בזמן עבור מצב התכונות מכיוון שהתכונות מוערכות בכל אות בנפרד. לפיכך, ניתן להסיר תוצרי EEG ישירות מנתוני EEG (באדום, איור 13), שלא כמו במצב HEP, שבו תקופות המכילות ממצאים נדחו עבור שתי סדרות הזמן מכיוון שסנכרון זמן עם דיוק MS חיוני לשיטה זו. - בצע ICA באמצעות אותם אלגוריתמים ופרמטרים כמו עבור HEP (ראה שלב 2.11.), אלא שהפעם רכיבי הלב מוסרים אם זוהו בביטחון של 99% (הם נשמרו עבור HEP / HEO מכיוון שאיננו רוצים להסיר אותות רלוונטיים הקשורים ללב).
- תחום התדרים בתיבה מסומן בשלב 3.2., כך ש-BrainBeats מחלצת את התכונות הבאות של תחום התדרים: הצפיפות הספקטרלית הממוצעת בהספק (PSD) עבור הדלתא (1-3 הרץ), תטא (3-7 הרץ), אלפא (8-13 הרץ), בטא (13-30 הרץ) וגמא (30+ הרץ), תדרי אלפא בודדים (IAF) וא-סימטריה אלפא בכל זוגות האלקטרודות (סימטריים) הזמינים. בדוק תכונות אלה.
הערה: PSD מומר לדציבלים (dB) מאפשר השוואת תוצאות בין הקלטות ונושאים. ניתן להגדיר את גבולות התדר לגבולות המקובלים (למשל, מוגדרים מראש 8-13 הרץ עבור פס אלפא) או לגבולות האינדיבידואליים, אשר מזוהים מתוך התפלגות הצפיפות הספקטרלית של העוצמה כדי להסביר הבדלים בין אינדיבידואלים69 (למשל, 7.3-12.6 הרץ עבור פס אלפא). האלגוריתם תוכנן עבור פס אלפא ואינו מתפקד טוב עבור להקות אחרות, במיוחד כאשר פסגות אינן קיימות בהתפלגות ספקטרלית העוצמה. תדר אלפא בודד (IAF) מוערך באמצעות מרכז הכובד אלפא כדי להתמודד טוב יותר עם פסגות מפוצלות או פסגות דו-משמעיות69. אסימטריה אלפא מחושבת לפי קווים מנחים (חלון המינג של 2 שניות עם חפיפה של 50%, הלוגריתם של כוח אלפא מהערוץ השמאלי פחות לוגריתם של כוח אלפא מהערוץ הימני)47. לפיכך, ערכים חיוביים מצביעים על עוצמת אלפא גדולה יותר משמאל לימין ולהיפך. ניתן לנרמל אסימטריה אלפא על ידי חלוקת כוח אלפא מכל אלקטרודה בכוח אלפא המסוכם על פני כל האלקטרודות47. הזוגות הסימטריים מתקבלים באמצעות מרחקי תטא, הדורשים מנתוני EEG להכיל קואורדינטות אלקטרודות. - התיבה תחום לא ליניארי מסומנת בשלב 3.2., BrainBeats מחלצת את האנטרופיה המטושטשת ואת הממד הפרקטלי עבור כל ערוץ EEG.
הערה: תכונות בתחום הלא-ליניארי נחשבות ללוכדות דינמיקה לא לינארית ומורכבת של המוח המוחמצת על ידי מדדים ספקטרליים ומראות הבטחה מיוחדת לחקר אינטראקציות בין מערכות גוף שונות 70,71,72. אנטרופיה מטושטשת אמינה וחזקה יותר מהחלופות שלה (מדגם ואנטרופיה משוערת) אך דורשת זמני חישוב ארוכים יותר (במיוחד עם סדרות זמן EEG ארוכות עם שיעורי דגימה גבוהים). כדי לטפל בבעיה זו, כאשר אותות EEG ארוכים מ-2 דקות עם קצב דגימה גדול מ-100 הרץ, הם עוברים דגימת הפחתה אוטומטית (או מושמדים כאשר הגורם אינו מספר שלם) ל-90 הרץ (כלומר, בהתאם לתדר Nyquist של 45 הרץ, כדי להתאים למסנן ברירת המחדל של מעבר נמוך ולהימנע ככל האפשר מארטיפקטים של רעשי קו). יתר על כן, מחשוב מקבילי מופעל כברירת מחדל בעת הערכת תכונות EEG, מה שמקצר את זמן המחשוב, במיוחד כאשר ערוצי EEG רבים זמינים. - בחר Visualize outputs בממשק המשתמש הגרפי הראשון (ראה שלב 3.1.), כדי ליצור תרשים המציג את הצפיפות הספקטרלית של הספק (PSD) עבור נתוני HRV ו-EEG (איור 14), יחד עם טופוגרפיות קרקפת המציגות תכונות EEG מסוימות (איור 15).
הערה: תוכל גם למצוא כמה פלטי עיבוד מקדים ב- EEG.brainbeats.preprocessing ואת כל הפרמטרים המשמשים ב- EEG.brainbeats.parameters. אנו מעודדים משתמשים לדווח על תפוקות אלה בפרסומים מדעיים כדי להגדיל את יכולת השכפול של הממצאים. - השאר את התיבה שמור פלטים מסומנת בחלון ממשק המשתמש הגרפי הראשון כדי לשמור את כל התכונות המיוצאות לקובץ .set של EEGLAB ב- EEG.brainbeats.features ולשמור אותן בקובץ .mat באותה תיקיה שבה נטען ערכת הנתונים.
- BrainBeats תומכת בפונקציית ההיסטוריה של EEGLAB. בסוף כל הפעולות, הקלד eegh בחלון הפקודה של MATLAB כדי להדפיס את שורת הפקודהשתשתאפשר לך לחזור על כל השלבים הנ"ל באמצעות שורת פקודה אחת, עם הפרמטרים שנבחרו ידנית בממשק המשתמש הגרפי, המאפשר אוטומציה קלה ושכפול של הפעולות.
- עבור משתמשים מתקדמים, בצע את כל השלבים לעיל באמצעות הפקודה הבאה:
EEGLAB; קרוב; % השקת EEGLAB ללא ממשק המשתמש הגרפי
main_path = fileparts(which('eegplugin_BrainBeats.m')); תקליטור(main_path);
EEG = pop_loadset('filename','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); % טען את ערכת הנתונים לדוגמה
EEG = brainbeats_process(EEG,' ניתוח',
'תכונות','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - השלבים הקודמים חילצו תכונות HRV מאות אק"ג. בצע את השלבים הבאים כדי לחלץ תכונות HRV מאות PPG (תכונות EEG זהות).
- טען שוב את אותה ערכת נתונים (שלב 1.5.) מכיוון שהיא הוחלפה על-ידי הפעולות ופתח שוב את ממשק המשתמש הגרפי הראשי (שלב 2.5). בחר חלץ תכונות EEG ו- HRV עבור הניתוח ובחר PPG עבור סוג אות הלב ו - PPG עבור שם הערוץ. לחץ על אישור.
- חלון ממשק המשתמש הגרפיהשני מציג כעת את הפרמטרים לעיבוד מראש של PPG ולחילוץ תכונות HRV ו- EEG. לחץ על אישור כדי לפעול עם פרמטרי ברירת המחדל. הפרמטרים מתוארים בשלב 2.17.
- תופיע הודעת אזהרה המבקשת אישור להסרת ערוץ האק"ג שזוהה. זה צפוי מכיוון שארגז הכלים אינו מיועד לנתח הן אק"ג והן PPG בו זמנית (או ערוצי עזר אחרים) והשארתו במערך הנתונים תוביל לטעויות חמורות (למשל, הסרת חפצים, פירוק לקוי של ICA וכו'). לחץ על כן.
- BrainBeats מעבד מראש את אות ה-PPG ומעריך את מרווחי ה-NN כמו בשלב 2.5. בדוק את תכונות HRV שחולצו מרווחי הזמן של NN, כמו בשלב 3.2 . אלא שמרווחי ה-NN התקבלו כעת מהאות PPG. אותות ה-EEG מעובדים מראש כמו בשלב 3.2. BrainBeats משרטטת את ה-PSD (איור 16). ההבדל היחיד כאן הוא PSD המוערך מתוך מרווחי NN המתקבלים PPG לעומת ECG.
- עבור משתמשים מתקדמים, בצע את כל השלבים לעיל באמצעות הפקודה הבאה:
EEGLAB; קרוב; % השקת EEGLAB ללא ממשק המשתמש הגרפי
main_path = fileparts(which('eegplugin_BrainBeats.m')); תקליטור(main_path);
EEG = pop_loadset('filename','dataset.set','filepath',
קובץ מלא(main_path, 'sample_data')); %טען את ערכת הנתונים לדוגמה
EEG = brainbeats_process(EEG,'ניתוח',
'תכונות','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. חלצו ממצאי לב מאותות EEG.
- טען את ערכת הנתונים לדוגמה (ראה שלב 1.5).
- פתח את חלון ממשק המשתמש הגרפי הראשי על-ידי לחיצה על כלים > BrainBeats > 1 st level (רמת הנושא) ובחר חלץ תוצרי לב מאותות EEG עבור סוג הניתוח, אק"ג עבור סוג אות הלב ובחר אק"ג ברשימת תוויות האלקטרודות. לחץ על אישור.
- חלון ממשק המשתמש הגרפיהשני מציג את פרמטרי העיבוד המקדים. הגדר את רעש קו החשמל ל- 50 הרץ (אירופה), ערוך את רמת הביטחון במידת הצורך, סמן את התיבה מצב הגברת (ביתא) ולחץ על אישור כדי לפעול עם פרמטרי ברירת המחדל מכיוון שאותות ה- EEG מערך הנתונים לדוגמה אינם מעובדים מראש.
הערה: רמת הביטחון לזיהוי רכיבי לב מוגדרת כברירת מחדל ל- 80%, שעשויה להיות נמוכה מדי או גבוהה מדי עבור ערכות נתונים מסוימות. הגדלת ערך זה תגדיל את הסיכוי לגילוי רכיבי לב אך תקטין את אמינות הזיהוי. מצב Boost (בטא) הוא אופציונלי ומטרתו לשפר את ביצועי הסיווג על ידי מריחת אות הלב לתוך אותות EEG. - תופיע הודעת אזהרה המבקשת אישור להסרת ערוץ PPG נוסף שזוהה. זה צפוי מכיוון שארגז הכלים אינו מיועד לנתח הן אק"ג והן PPG בו זמנית (או ערוצי עזר אחרים) והשארתו במערך הנתונים תוביל לטעויות חמורות (למשל, הסרת חפצים, פירוק לקוי של ICA וכו'). לחץ על כן.
- אות האק"ג מסונן באמצעות פסים כדי להסיר סטיות בתדר איטי מתחת ל-1 הרץ ורעש בתדר גבוה מעל 20 הרץ (עם מסנן FIR אפס-פאזי לא סיבתי). עבד מראש את אותות ה- EEG כמו בשלב 3.4.
- ביצוע ניתוח רכיבים בלתי תלוי (ICA) באמצעות אלגוריתם ICA for real data (PICARD) המותנה מראש. כדי לשנות אפשרות זו, בחר באלגוריתם Infomax רגיל או באלגוריתם Infomax ששונה לשכפול מממשק המשתמש הגרפי בשלב 4.3 . לאחר מכן, סווגו את הרכיבים הבלתי תלויים באופן אוטומטי עם כיל. אם רכיב מסווג כרכיב לב בביטחון של 80%, הוא מזוהה, כברירת מחדל, ומופק אוטומטית מנתוני ה- EEG.
- שמור את התיבה הצג פלט באופן חזותי בממשק המשתמש הגרפי הראשי הראשון (שלב 4.2.) כדי להמחיש את טופוגרפיית הקרקפת של הרכיב שהוסר (איור 17 משמאל) ואת סדרת הזמן הסופית של EEG (בכחול, איור 17 מימין) לאחר חילוץ רכיב הלב (באדום, איור 17 מימין).
הערה: ערוץ האק"ג נשמר לצורך הדמיה כדי לאשר את החילוץ של רכיבים הקשורים לאק"ג, אך הוא מוסר לאחר שלב זה מכיוון שאינו מכיל עוד מידע רלוונטי. - עבור משתמשים מתקדמים, בצע שלבים אלה באמצעות שורות הפקודה הבאות:
EEGLAB; קרוב; % השקת EEGLAB ללא ממשק המשתמש הגרפי
main_path = fileparts(which('eegplugin_BrainBeats.m')); תקליטור(main_path);
EEG = pop_loadset('filename','dataset.set','filepath',
קובץ מלא(main_path, 'sample_data'));
EEG = brainbeats_process(EEG,'ניתוח','rm_heart',
'heart_signal','א.ק.ג',...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'דחיפה',נכון);
תוצאות
ראשית, תוסף BrainBeats שימש לעיבוד מוקדם של נתוני EEG ואק"ג, זיהוי והסרה של ממצאים וניתוח פוטנציאלים מעוררי פעימות לב (HEP) ותנודות (HEO). BrainBeats זיהו בהצלחה את מרווחי ה-RR מאות האק"ג וכמה ממצאים של RR (איור 2). BrainBeats דיווחה גם בחלון הפקודה כי 11/305 (3.61%) מפעימות הלב סומנו כממצאים ועברו אינטרפו?...
Discussion
שלבים קריטיים בפרוטוקול
שלבים קריטיים מתוארים בשלבים 1.1-1.4. אזהרות והודעות שגיאה מיושמות במקומות שונים בארגז הכלים כדי לעזור למשתמשים להבין מדוע הם עלולים להיתקל בבעיות (למשל, מיקומי אלקטרודות שאינם נטענים בנתוני EEG, אורך הקובץ קצר מדי לחישוב מדד אמין של HRV בתדר אולטרה נמוך, איכו...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
המכון למדעי הנוטיקה תמך במחקר זה. אנו מודים למפתחי אלגוריתמי הקוד הפתוח המקוריים שהותאמו לפיתוח חלק מהאלגוריתמים של BrainBeats.
Materials
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
References
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved