このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
BrainBeatsは、脳波と心血管の信号を共同で分析するためのオープンソースのEEGLABプラグインです
要約
BrainBeatsツールボックスは、EEGと心血管(ECG / PPG)信号を共同で分析するために設計されたオープンソースのEEGLABプラグインです。これには、心拍誘発電位(HEP)評価、特徴ベースの分析、およびEEG信号からの心臓アーチファクト抽出が含まれます。このプロトコルは、2つのレンズ(HEPと機能)を通じて脳と心臓の相互作用を研究するのに役立ち、再現性とアクセシビリティを向上させます。
要約
脳と心血管系の相互作用は、人間の生理学の理解を深め、健康状態を改善する可能性が注目されています。しかし、これらの信号のマルチモーダル解析は、ガイドライン、標準化された信号処理と統計ツール、グラフィカルユーザーインターフェース(GUI)、大規模なデータセットの処理や再現性の向上のための自動化が不足しているため、困難です。標準化されたEEGおよび心拍変動(HRV)特徴抽出法には、さらに空白が存在し、臨床診断や機械学習(ML)モデルの堅牢性を損ないます。これらの制限に対応して、BrainBeats ツールボックスを紹介します。オープンソースのEEGLEBプラグインとして実装されたBrainBeatsは、3つの主要なプロトコルを統合しています:1)ミリ秒の精度で時間ロックされた脳と心臓の相互作用を評価するための心拍誘発電位(HEP)と振動(HEO)。2)さまざまな脳と心臓の指標間の関連性/違いを調べるため、または堅牢な機能ベースのMLモデルを構築するためのEEGおよびHRV特徴抽出。3)脳波信号から心臓のアーチファクトを自動的に抽出し、脳波分析を行いながら潜在的な心血管汚染を除去します。これら3つの方法を、64チャンネルのEEG、ECG、およびPPG信号を同時に含むオープンソースのデータセットに適用するためのステップバイステップのチュートリアルを提供します。ユーザーは、グラフィカルユーザーインターフェース(GUI)またはコマンドラインを使用して、独自の研究ニーズに合わせてパラメーターを簡単に微調整できます。BrainBetsは、脳と心臓の相互作用の研究をよりアクセスしやすく、再現性のあるものにするはずです。
概要
長い間、還元主義的アプローチは、人間の生理学と認知における科学的調査を支配してきました。このアプローチでは、複雑な身体および精神的なプロセスを、より小さく、より管理しやすいコンポーネントに分解することで、研究者は個々のシステムに単独で集中することができました。この戦略は、人間の身体と心の複雑で相互に関連し合った性質を研究する際の課題から生じました1。還元主義は、神経2 または心臓3 のコミュニケーションにおけるイオンチャネルと活動電位の役割を解明するなど、個々のサブシステムを単独で理解するのに役立ちました。しかし、これらの孤立したシステムがより大きな空間的および時間的スケールでどのように相互作用するかについての理解には、大きなギャップが残っています。マルチモーダル(統合的または生態学的)フレームワークは、人体を複雑な多次元システムと見なし、心は脳の産物ではなく、生物の活動、つまり脳を人体の日常的な機能に統合する活動として見なされます4。マルチモーダルアプローチと還元主義的アプローチは排他的ではなく、脳全体なしでは1つのニューロンを研究できないか、個々のニューロンの特性を理解せずに脳全体を研究できないのと同じです。彼らは一緒になって、人間の健康、病理、認知、心理学、意識について、より包括的で相乗的な理解への道を切り開きます。本手法は、脳波(EEG)と心血管信号、すなわち心電図(ECG)と光電式容積脈波(PPG)の共同解析を提供することにより、脳と心臓の相互作用のマルチモーダル調査を容易にすることを目的としています。このツールボックスは、MATLAB の EEGLAB プラグインとして実装され、既存の方法論の限界に対処し、科学分野でのアクセシビリティと再現性を促進するためにオープンソース化されています。最新のガイドラインと推奨事項を設計と既定のパラメーターに実装して、ユーザーが既知のベスト プラクティスに従うことを奨励します。提案されたツールボックスは、1)心拍誘発電位の研究、2)EEGおよびECG / PPG信号からの特徴の抽出、または3)EEG信号からの心臓アーチファクトの除去に関心のある研究者や臨床医にとって貴重なリソースになるはずです。
心脳研究
心臓と脳の関係は、機能的磁気共鳴画像法(fMRI)や陽電子放出断層撮影法(PET)などの神経画像法によって歴史的に研究されてきました。これらのツールを使用して、研究者は心血管制御に関連するいくつかの脳領域(心拍数や血圧の操作など)を強調したり、BOLD信号6に対する心拍数の影響を示したり、冠状動脈性心疾患(ストレス誘発性血圧7)に寄与する可能性のある脳-体経路を特定したりしました).これらの研究により、中枢神経系(CNS)と心血管機能との間の複雑な相互作用についての理解が大幅に進歩しましたが、これらのニューロイメージング技術は高価で、利用可能性が限られており、制御された実験室環境に限定されているため、実世界および大規模なアプリケーションへの実用性が制限されています。
対照的に、EEGとECG / PPGは、より手頃な価格でポータブルなツールであり、より多様な環境や集団、または長期間にわたって脳と心臓の相互作用を研究する可能性を提供し、新しい機会を提供します。心電図は、心臓が収縮および弛緩するときに、皮膚(通常は胸部または腕)に配置された電極を介して、各心拍によって生成される電気信号を測定します8。PPGは、光源(例えば、LED)および光検出器(通常は指先、手首、または額に配置される)を使用して、微小血管組織の血液量の変化(すなわち、血流および脈拍数)を測定するが、これは、血液が周囲の組織よりも多くの光を吸収する方法に依存している9。どちらの方法も心血管機能に関する貴重な情報を提供しますが、目的も異なり、データタイプも異なります。ECGと同様に、EEGは、細胞外マトリックス、組織、頭蓋骨、および頭皮を伝播して頭皮の表面10に着目するまで伝播する数千の皮質ニューロンの同期活動によって生成される電場を記録する。このように、EEGとECG/PPGの使用は、脳と心臓の相互作用の根底にある生理学的、認知的、感情的なプロセスと、それらが人間の健康と幸福に与える影響についての理解を深める上で大きな期待を寄せています。したがって、BrainBeatsツールボックスを使用してEEG、ECG/PPG信号から心と脳の相互作用をキャプチャすることは、臨床診断および予測、ビッグデータ機械学習(ML)、実世界の自己モニタリング11、およびモバイル脳/身体イメージング(MoBI)12,13の科学分野で特に役立つ可能性があります。
脳波信号とECG信号を共同で解析するための2つのアプローチ
EEGと心血管信号の間の相互作用を研究するには、主に2つのアプローチがあります。
時間領域の心拍誘発電位(HEP):イベント関連電位(ERP)、および時間-周波数領域の心拍誘発振動(HEO):イベント関連スペクトル摂動(ERSP)と試行間コヒーレンス(ITC)。このアプローチでは、脳が各心拍をどのように処理するかを調べます。ミリ秒(ms)の精度で、この方法では、両方の時系列が完全に同期され、心拍がEEG信号にマークされる必要があります。このアプローチは、近年14,15,16,17,18,19で関心を集めています。
特徴ベースのアプローチ: このアプローチでは、連続的な信号から EEG と心拍変動 (HRV) の特徴を抽出し、それらの間の関連性を調べます。これは、EEG(定量的EEGまたはqEEG20と呼ばれることが多い)、ECG 21,22,23、およびPPG 24,25,26とは独立して行われています。このアプローチは、状態と形質に関連する変数の両方を捕捉することにより、有望なアプリケーションを提供します。EEG信号と心血管信号の両方について、記録が長いほど、形質変数27,28,29がより支配的になることに注意してください。したがって、アプリケーションは記録パラメータに依存します。特徴量ベースの分析はますます関心を集めており、精神障害および神経障害の発症、治療反応、または再発を予測するための新しい定量的指標を提供しています30,31,32,33,34,35。このアプローチは、ウェアラブルニューロテクノロジー11の最近の革新のおかげでより簡単に取得できる大規模な現実世界のデータセット(例:クリニック、リモートモニタリング)で特に魅力的です。あまり探求されていないアプリケーションは、特定の脳と心臓の特徴との間の関連を特定し、中枢神経系のダイナミクスの根底にある可能性を強調することです。心拍変動(HRV)は、ECG信号とPPG信号の両方から計算できます。これは、心拍間の時間間隔(すなわち、正常から正常への間隔)の変動を測定することにより、自律神経系(ANS)に関する情報を提供する27。交感神経(SNS)活動の増加(ストレスや運動中など)は通常、HRVを減少させ、副交感神経(PNS)活動(リラクゼーション中など)はHRVを増加させます。呼吸数が遅いと、特に短い録音(<10分)の場合、PNS活動が増加するため、一般にHRVが増加します27。HRVスコアが高いほど、一般的にANSの回復力と適応性が高いことを示唆し、HRVが低い場合は、ストレス、疲労、または根本的な健康問題を示している可能性があります。長いHRV記録(すなわち、少なくとも24時間)は、心血管疾患、ストレス、不安、およびいくつかの神経学的状態を含むさまざまな健康状態の予測予後を提供する27。血圧、心拍数、コレステロール値などの測定値は、心血管系の状態に関する情報を提供します。対照的に、HRVはダイナミックな側面を追加し、心臓がストレスにどのように反応し、ストレスから回復するかを示します。
既存の方法に対するBrainBeatsの利点
以下で検討するように、心血管信号と脳波信号を互いに独立して処理するツールは存在しますが、それらを一緒に分析することはできません。さらに、心血管信号を処理するための利用可能なほとんどの手段は、高価なライセンスを伴い、自動処理を許可せず(特に大規模なデータセットに有益)、透明性と再現性を妨げる独自のアルゴリズムを持っているか、またはグラフィカルユーザーインターフェース(GUI)を提供しないことにより高度なプログラミングスキルを必要とする36。私たちの知る限り、4つのオープンソースのMATLABツールボックス(ecg-kitツールボックス37、BeMoBILパイプライン38、HEPLAB EEGLABプラグイン39、およびCARE-rCortexツールボックス40)がGUIによるHEP/HEO解析をサポートしています。HEPLAB、BeMoBIL、およびecg-kitは、心拍を検出し、それらをEEG信号にマーキングすることでHEP分析を容易にしますが、統計分析を提供しないか、時間領域(つまりHEP)に限定されます。CARE-rCortexプラグインは、ECGおよび呼吸信号、時間-周波数領域分析、統計、およびHEP/HEO分析に適応した高度なベースライン正規化および補正方法をサポートすることにより、これらの問題に対処しました。しかし、それは、タイプ1エラー(すなわち、偽陽性)の統計的修正のためにボンフェローニ法を使用しており、これはあまりにも保守的であり、EEGアプリケーションにとっては生理学的に健全ではなく、タイプIIエラー(すなわち、偽陰性)の増加につながる41。さらに、ツールボックスは自動化のためのコマンドラインアクセスを提供していません。最後に、最近の研究では、ベースライン補正法42,43,44は、信号対雑音比(SNR)を低下させ、統計的に不必要で望ましくないため、推奨していません。
これらの制限に対処するために、現在 MATLAB 環境にオープンソースの EEGLAB プラグインとして実装されている BrainBeats ツールボックスを紹介します。これには、以前の方法に比べて次の利点があります。
1) 使いやすいGUIとコマンドライン機能(自動処理を目指すプログラマー向け)2)Rピークの検出、RRアーティファクトの補間、HRVメトリックの計算など、心血管信号を処理するための検証済みのアルゴリズム、パラメータ、およびガイドライン(例えば、ウィンドウ処理、リサンプリング、正規化などの埋め込みガイドライン27,45,46)。Vestらは、これらの処理ステップのわずかな違いが結果の違いを招き、HRVメトリクスの再現性と臨床的適用性の欠如に寄与することを示したため、これは重要です46。3)フィルタリングとウィンドウ処理44,47、再参照48,49、異常なチャネルとアーティファクトの除去50,51,52、最適化されたICA分解と独立したコンポーネントの分類53,54,55,56を含む、EEG信号を処理するための検証済みのアルゴリズム、デフォルトパラメータ、およびガイドライン.ユーザーは、自分のニーズに合わせてツールボックスを使用する前に、すべての前処理パラメータを微調整したり、好みの方法でEEGデータを前処理したりすることができます(例:EEGLAB clean_rawdataプラグイン50,52、BeMoBILパイプライン38、PREPパイプライン57など)。4)心電図信号から、心拍誘発電位(HEP、すなわち時間領域)および振動(HEO;ウェーブレットまたはFFT法によるイベント関連のスペクトル摂動、および試行間コヒーレンス)は、標準のEEGLABソフトウェアを通じて利用可能です。タイプ1エラーの補正を含むパラメトリックおよびノンパラメトリック統計は、EEGLABの標準ソフトウェアを介して利用できます。ノンパラメトリック統計には、多重比較のための順列統計と時空間補正が含まれます(例:時空間クラスタリングまたはしきい値なしクラスター強化)58,59。ユーザーは、LIMO-EEGプラグインを使用して、被験者内および被験者間の分散を適切に説明し、タイプIおよびIIエラー60,61のロバスト制御を備えた仮定のない質量単変量アプローチを実装する階層線形モデリングを実装できます。HEP/HEOデータの統計解析は、チャネルドメインと独立コンポーネントドメインで行うことができます。5) PPG信号からのHEP/HEOおよびHRV解析(HEP/HEOでは初めて)。6) 脳波とHRVの特徴の共同抽出を初めてサポートします。7) このツールボックスは、さまざまな必要な処理ステップで信号を検査し、被験者レベルでの出力を生成するためのさまざまなデータ視覚化を提供します。
| 方式 | ECGからのRピークの検出 | PPGからのR波の検出 | HEP/HEO | EEG & HRV 機能 | 脳波から心臓のアーチファクトを取り除く | GUIの | コマンドライン |
| ECGキット | X | X | X | X | |||
| ベモビル | X | X | X | ||||
| ヘプラブ | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| ブレインビート | X | X | X | X | X | X | X |
表1:BrainBeatsが既存の類似の方法と比較してもたらした新規性。
読者がその方法が自分に適しているかどうかを判断するのに役立つ情報
このツールボックスは、脳波およびECG/PPGデータを持つ研究者や臨床医に適しています。このプラグインは、EEG信号とECG/PPG信号を別々のファイルからインポートすることにはまだ対応していません(ただし、この機能は近日中に利用可能になります)。このツールボックスは、HEP/HEO分析の実行、標準化された方法でのEEGおよび/またはHRV特徴の抽出、または単にEEG信号からの心臓アーチファクトの除去を目指す人に適しています。 図 1 は、BrainBeats の全体的なフローとメソッドをまとめたブロック図です。
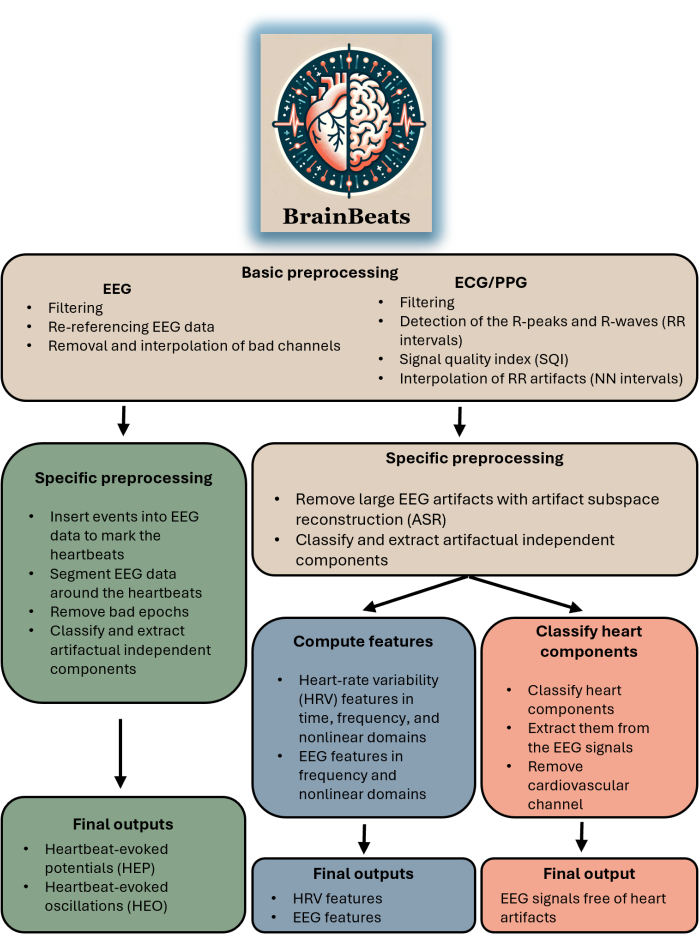
図 1.BrainBeatsの全体的なアーキテクチャとフローをまとめたブロック図。3 つの方法に共通する操作は茶色です。心拍誘発電位(HEP)と振動(HEO)に固有の操作は緑色です。EEGおよびHRV特徴の抽出に固有の操作は青色です。EEG信号から心臓のアーチファクトを除去するための操作は赤です。 この図の拡大版を表示するには、ここをクリックしてください。
プロトコル
各参加者からインフォームドコンセントが得られ、ウラル連邦大学の倫理委員会が実験プロトコルを承認しました。
1. BrainBeatsの要件
- MATLABとEEGLABをコンピューターにインストールします。EEGLABは https://github.com/sccn/eeglab でダウンロードでき、コンピューター上の任意の場所で解凍(またはGitユーザーの場合はクローン化)できます。インストールの詳細については、GitHub ページを参照してください。
- MATLAB のホーム パネルで EEGLAB フォルダーへのパスを追加するには、[ パスの設定 ] ボタンをクリックします。解凍したファイルがあるeeglabフォルダを選択し、[ 保存]をクリックして>[閉じる]をクリックします。
- MATLAB のコマンド ウィンドウで 「eeglab 」と入力して EEGLAB を起動します。
- 「 File」>「Manage EEGLAB拡張機能」をクリックして、BrainBeatsプラグインをインストールします。検索バーに 「brainbeats 」と入力し、リストから BrainBeats プラグインを選択して、[ インストール/更新]をクリックします。
- サンプルデータセットをEEGLABに読み込みます。 [ファイル] > [既存のデータをロード] をクリックします。EEGLABフォルダに移動し、pluginsフォルダに移動し、BrainBeatsフォルダに移動して、sample_dataフォルダを開きます。 ファイル dataset.set を選択します。
注:このデータセットは、オープンソースのマルチサブジェクトデータセット62,63のsub-032(目を開けて安静状態)に対応します。これは、1000 Hz のサンプリング レートで、EEG (64 チャネル)、ECG (1 チャネル)、PPG (1 チャネル) の同時機能が含まれているため、選択されました。ECG信号とPPG信号は、actiCHampシステムの補助入力を使用して収集されました。脳波データは、10-20システムに従って配置されたアクティブ電極、オンラインリファレンスとしてFCz、接地電極としてFpzを使用し、インピーダンスを25kOm未満に維持して記録しました。ECG は、右手首に 1 つのアクティブ電極を配置し、参照電極を左手首に配置し、肘から遠位 3 cm の左前腕の内側に地面を配置して記録しました。PPGは左の人差し指から記録しました。EEG、ECG、およびPPGデータは、同じアンプで同時に記録されたため、時間同期しています。詳細については、参考文献62,63を参照してください。 - Save Outputsチェックボックスにチェックを入れると、対応する.setファイルのすべてがEEGLABにロードされた元のファイルと同じ場所に保存されます。
注:このチュートリアルでは、EEG、ECG、およびPPGデータを1つのEEGLABデータセットに統合し、3D境界要素法(BEM)電極座標をロードし、信号を250 Hzにダウンサンプリングして、ファイル重量(ユーザーダウンロード用)を減らし、計算時間を短縮しました。このデータセットには異常な脳波チャネルがなかったため、BrainBeatsの不良チャネル検出および除去アルゴリズムを示すために、チャネルTP9を人為的に変更しました。同様に、BrainBeatsのアーティファクト除去機能を説明するために、ファイルの先頭にある大きなEEGアーティファクトと、3〜6秒の時間チャネルの高周波筋肉様アーティファクトを人工的にシミュレートします。
2. 心拍誘発電位(HEP)と振動(HEO)
- BrainBeatsの最初の一般的なユーザーインターフェイス(GUI)を開いて、メインパラメータを選択します。EEGLABで、 Tools > BrainBeats >1st level (subject level)をクリックします。実行する分析として [Heartbeat-evoked potentials HEP ]を選択し、心臓のデータタイプとして [ECG ]を選択し、ボタンをクリックしてチャネルのリストを表示し、ECGとラベル付けされたECGチャネルを選択します(またはテキストボックスに直接入力します)。 「出力の視覚化 (推奨)」 オプションと「 出力の保存 」オプションを選択したまま、「 OK」をクリックします。
- 前の選択に基づいて、2番目のGUIウィンドウがポップアップ表示されます(つまり、ECGデータを使用したHEP分析)。EEG信号とECG信号の両方の前処理パラメータを選択します。BrainBeatsを起動する前に、Preprocess ECGとPreprocess EEGのチェックボックスをオフにして、データがすでに前処理されているかどうかを確認します。これらのEEGデータはロシアで記録されたため、EEGの前処理セクションで電力線ノイズを50Hzに変更します。[OK] をクリックして起動します。
- 警告メッセージが表示され、検出されたPPGチャネルを削除するように確認を求められます。これは、ツールボックスが ECG と PPG の両方 (または他の補助チャネル) を同時に解析するように設計されておらず、データセットに保持すると重大なエラー (アーティファクトの除去、ICA 分解の不良など) が発生するためです。 [はい] をクリックします。BrainBeatsは、いくつかのチェックの実行を開始し、いくつかのデフォルトパラメータを設定し、ECGをEEGデータから分離して、ECG信号を前処理し、RR間隔を計算します。
- ECGおよびRR時系列は、BrainBeatsのデータフォーマット、明瞭度の向上、パラメータ調整、および計算時間に適合するように適合したPhysionet Cardiovascular Signalツールボックス46,64の検証済みアルゴリズムを使用して前処理されます(アルゴリズムの検証については参考文献を参照)。プラグインは、RR間隔、タイムスタンプ、フィルタリングされたECG信号、Rピークインデックス、および心拍数(HR)を出力します。これらのパラメータは、GUIまたはコマンドラインで調整します。
注:ECG信号は、カスタマイズされた検証済みフィルター(1〜30 Hz)を使用してバンドパスフィルタリングされ、Pan-Tompkins(P&T)メソッド65を使用してQRSプレックスとRピークを識別するために信号をスキャンします。最高のパフォーマンスを得るために、微分、二乗、積分、平滑化などの信号処理操作を実装します。P&T エネルギーのしきい値は、サンプルレートと平滑化された ECG 値に基づいて推定され、大きなバンプによる中断を回避します。RR間隔の変動が中央値の1.5倍を超える場合、欠落したピークが検索されます。平均 R ピーク符号は 30 秒のセグメントで計算され、ピーク点は不応期チェックを通じて精製され、フラットライン条件を管理し、一貫した検出を確保します。 - 次に、BrainBeatsは、前方後方検索と生理学的閾値を使用して、異常なRR間隔またはRR間隔内のスパイクを特定します。信号品質指数(SQI)は46計算されます。RR タイム シリーズの 20% を超える RR アーチファクト (生理学的限界の範囲外、または SQI が 0.9 未満) に警告が表示される場合は、これを確認します。プロットには、フィルタリングされたECG信号、同定されたRピーク、NN間隔、および補間されたアーチファクトが表示されます( 図2を参照)。
注: RR アーティファクトは、デフォルトで形状保持区分的 3 次法を使用して内挿され、法線から法線 (NN) 間隔を取得しますが、必要に応じてそれらを削除する (推奨されません) か、別の補間法 (線形、3 次、最近傍、前/次近傍、スプライン、3 次畳み込み、または修正 Akima 3 次) を使用します。複数の ECG チャネルが存在する場合、RR 間隔がそれぞれに推定され、RR アーティファクトの数が最も少ないチャネルが次の手順で選択されます。 - Rピークのズームインされた30秒のウィンドウをスクロールして、 右/左矢印を押して詳しく調べます。データに複数の ECG/PPG チャネルが含まれている場合は、RR 間隔の信号品質インデックスが最適なチャネルを使用します。BrainBeatsは、現時点ではECG信号とPPG信号の両方を同時にサポートしていません。チュートリアルの目的で、両方のデータ型を含むサンプル データセットを選択しました。
- ECG信号の処理が完了すると、BrainBeatsバンドパスは、デフォルトで非線形因果最小位相FIRフィルタを使用してEEGデータを1〜40Hzでフィルタリングし、心拍前と心拍後の期間の間のスミアリング活動を減らし、因果関係を維持し、望ましくないグループ遅延を回避します44。これは、ハートビート前の期間を調べるユーザーにとって特に重要です。ローパスフィルタが電力線周波数よりも高い値に設定されている場合(たとえば、電力線周波数が50Hzの80Hzローパス)、シャープなノッチフィルタを使用してラインノイズアーチファクトを除去します。その後、EEGデータは、30チャネル未満が検出されない限り、RESTアルゴリズム(HEP分析49に最適)を使用して無限に再参照されます(この場合、チャネルを確実に再参照することができず、ユーザーに知らせるために警告が生成されます)。
- その後、BrainBets は異常な脳波チャネルを検出、除去、補間します(図 3)。デフォルトのパラメータが、5秒より大きいフラットライン(clean_flatlinesアルゴリズム)、最大高周波ノイズ標準偏差10、ウィンドウ長5秒(低速周波数アーティファクト52をより適切にキャプチャするため)、隣接するチャネル間の最小相関が.65、および最大許容部分が33%(clean_channelsアルゴリズム)であることを確認します。
注:RANSACサンプルの数は、不良チャネル除去の収束性と再現性を高めるために、デフォルトで500に設定されています(ただし、計算時間は長くなります)。 - 次に、RピークをイベントマーカーとしてEEGデータに挿入して、各心拍をマークし、ベースラインを除去せずにこれらのマーカーの周りにデータをセグメント化します(ガイドライン43,66による;図4)。NNインターバルは長さが異なり、EEGは一定の長さでセグメント化する必要があるため、心拍インターバル(IBI)データの5パーセンタイルを使用して、Rピークイベント後の最小エポックサイズカットオフを推定します(つまり、最短IBIの5%がそれ以下に収まる値は、ヒストグラムに赤い破線で表示されます;図5を参照)。
注:この5パーセンタイル値は、HEP / HEO分析の関心のある期間がハートビート49,67後200〜600ミリ秒であるため、エポックが短すぎないようにしながら、できるだけ多くのエポックを保持するための良い妥協点です。 - 脳波データを R ピークの -300 ミリ秒前から R ピーク後の5 パーセンタイル値までセグメント化し、R ピークを時間 0 にします。ガイドライン49,67に従って、550 msより短いエポック、または複数のRピークを含むエポック(ERP/ERSPに偏りがある)はリジェクトします。大きなEEGアーチファクトを含むエポックは、二乗平均平方根(RMS)と信号対雑音比(SNR)のメトリクスとMATLABのアイソアウトライアー関数を使用して検出されます(図6)。アーティファクト エポックは削除されます。
- デフォルトのInfomaxアルゴリズムを使用してブラインドソース分離を実行し、最高のパフォーマンス53のデータランクを考慮します。必要に応じて、高速計算のための前処理付き独立成分分析(PICARD)アルゴリズムを選択するか、オプションPicard(高速)を選択することにより、またはフィールドICA法に対してオプションModified Infomax(長いが複製可能)を選択することにより、修正Infomaxアルゴリズムを選択する(ただし、これにははるかに長い計算時間が含まれる)。ICLabelプラグイン56を使用して、ICを自動的に分類し、非脳アーティファクトを抽出します(眼球成分は90%の信頼度で除去されますが、筋肉、ラインノイズ、チャネルノイズは99%の信頼度で除去されます。図 7)。
- 最初のGUIウィンドウで[出力の視覚化]オプションを選択したままにして、最終的なEEG時系列、総平均HEP(図8上)、各心拍のHEP(図8下)、および総平均心拍誘発振動(HEO;図9)。イベント関連のスペクトル摂動(ERSP、つまり、心拍間のEEGパワーの変化;図9上)と試行間位相コヒーレンス(ITC、つまり、心拍間の位相角の一貫性。図 9 下)。
注 : ERSP は、既定の 3 サイクル ウェーブレット (ハニング テーパー ウィンドウが適用され、パディング比 2) を使用して計算され、高周波数に使用されるウェーブレットのサイクル数は、最高周波数 (1 から 0.8) での同等の FFT ウィンドウのサイクル数の 20% までゆっくりと拡大します。これにより、関数によって測定された個々のウィンドウの形状と、結果の時間/周波数ペインでのそれらの形状が制御されます。任意のベースラインは、説明のために削除され、ERSPは、文献に記載されている典型的なHEO効果、すなわち、前部中心電極17,67上のアルファバンド(8-13Hz)の心拍後300-450ミリ秒を捕捉するために、周波数7-25Hzに対して計算される。より低い周波数は、ビート間間隔によって定義されるエポックサイズが短いため、推定できません。ノンパラメトリック(順列)統計を適用して、p値0.05のHEOを視覚化し、偽発見率(FDR、つまりタイプ1エラーまたはファミリーワイズエラー)を補正します。これらのプロットは、チュートリアル目的または単一試行解析用に生成されます。 - さまざまなステップを視覚化するために、前処理プロットが生成されます。オフにするには、[ 前処理を視覚化する] ボックスのチェックを外します。最終的なEEGデータ(クリーニングされ、Rピークを中心にセグメント化)には、ERP / ERSP分析に偏りがあるため、ECGデータは含まれません。最終出力でハートチャンネルを保持するには、[ ハートチャンネルを保持する]チェックボックスをオンにします。
注: 必要に応じて、次のファイル (次の条件または参加先) を処理する前に、ここで一時停止します。 - BrainBeatsはEEGLABの履歴機能をサポートしています。すべての操作の最後に、MATLABのコマンドウィンドウに eegh と入力してコマンドラインを印刷し、GUIで手動で選択したパラメーターを使用して、上記のすべての手順を1つのコマンドラインで繰り返すため、自動化が容易になります。前処理の出力(心血管時系列の信号品質指数、NN間隔、削除された脳波チャネル、セグメント、コンポーネントなど)をEEGLAB構造体で見つけます:EEG.brainbeats.preprocessings。すべてのパラメータはEEG.brainbeats.parametersにもエクスポートされます。
- 上級ユーザーの場合は、以下のコマンドラインを使用して、デフォルトのパラメータを使用して上記のすべての手順を実行します(その他のオプションについては、BrainBeatsリポジトリのチュートリアルスクリプトを参照してください)。
EEGLABの;閉める;% GUIなしでEEGLABを起動
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
EEG = pop_loadset('ファイル名','dataset.set',
'ファイルパス',fullfile(main_path, 'sample_data'));%サンプルデータセットを読み込みます
脳波 = brainbeats_process(脳波,'分析',
'hep'、'heart_signal'、'ECG'、'heart_channels'、
{'ECG'},'clean_eeg',true);% BrainBeats 1st レベルを起動して、デフォルトのパラメータでHEP分析用のファイルを処理します - 上記の手順では、ECG信号からHEP / HEOを実行しました。PPG 信号に対して、以下の手順を使用します。
- 次の手順では、同じ操作を PPG 信号を使用して実行します。前の操作で上書きされた同じデータセットを再度読み込み(ステップ1.5を参照)、BrainBeatsの最初のGUIを再度開いてメインパラメータを選択します。 「Tools」> BrainBeats>1st レベル(サブジェクトレベル)をクリックします。実行する分析として 心拍誘発電位(HEP) を選択し、心臓のデータタイプとして PPG を選択し、ボタンをクリックしてチャネルのリストを表示し、PPGチャネルを選択します。 「出力の視覚化 (推奨)」 オプションと「 出力の保存 」オプションを選択したまま、「OK」をクリックします。
- 2番目のGUIウィンドウは、手順2.2と同様の方法でポップアップします。唯一の違いは、ECG信号の代わりにPPG信号を処理するようにパラメーターを微調整できるようになったことです。 [OK ] をクリックして、デフォルトのパラメータで実行します。
- 警告メッセージが表示され、検出された余分なECGチャネルを削除するように確認を求められます。繰り返しになりますが、これは予想通りです。 [はい] をクリックします。デフォルトでは、ツールボックスは PPG 信号を前処理し、パルス波を検出して RR 間隔を取得し、RR アーティファクト (存在する場合) を特定して補間します (図 10)。ステップ 2.7.を 2.12 に設定します。が実行され、同じプロットと出力が生成されますが、PPG信号から検出されたR波に基づいています( 図11 および 図12を参照)。
注:R波は、指定されたウィンドウ内の信号の傾きを使用して検出されます。その後、スロープが動的スレッショルドを超えると、潜在的なパルスにフラグが立てられ、検出履歴と信号特性に基づいて調整されます。次に、アルゴリズムは目を閉じる時間内に検索して最大傾きを特定し、その後、パルス波の開始をしきい値化によって決定します。R波のピークは、開始近くの谷として識別され、その位置が記録されます。このアルゴリズムは、信号全体を反復処理し、検出しきい値を連続的に調整して R 波のピークを特定し、RR 間隔の計算に使用されます。 - 上級ユーザーの場合は、以下のコマンドラインを使用して、デフォルトのパラメータを使用して上記のすべての手順を実行します(その他のオプションについては、BrainBeatsリポジトリのチュートリアルスクリプトを参照してください)。
EEGLABの;閉める;% GUIなしでEEGLABを起動
main_path = fileparts(which('eegplugin_
BrainBeats.m'));cd(main_path);
EEG = pop_loadset('ファイル名','dataset.set',
'ファイルパス',fullfile(main_path, 'sample_data'));%サンプルデータセットを読み込みます
脳波 = brainbeats_process(脳波,'分析',
'hep'、'heart_signal'、'PPG'、'heart_channels'、
{'PPG'},'clean_eeg',true);% BrainBeats 1st レベルを起動して、デフォルトのパラメータでHEP分析用のファイルを処理します
3. 脳波・HRV特徴量の抽出
- 同じデータセットを再度読み込みます (手順 1.5 を参照)。前の操作で上書きされたため、[ファイル] > [既存のデータセットをロード] > [dataset.set] を選択) をクリックし、メイン GUI を再度開いてメイン パラメーターを選択します (ステップ 2.1;[ツール] > [BrainBeats] > 1レベル) をクリックします。解析タイプとして「Extract EEG & HRV features」を、心臓信号タイプとして「ECG」を選択し、電極ラベルのリストで「ECG」を選択します。[OK] をクリックします。
- 2番目のGUIウィンドウは、ステップ2.2と同様にポップアップしますが、EEGの前処理とHRVおよびEEG特徴の抽出のための異なるパラメータがあります。これらのパラメータは、以下で説明するように設定します。 「OK 」をクリックして、デフォルトのパラメータで起動します。
- HRVセクションで、HRVパワーの計算方法(デフォルトでは正規化されたLomb-Scargleピリオドグラムに設定)、ウィンドウのオーバーラップ(デフォルトは25%に設定)、およびセカンドレベルの正規化の実行方法(デフォルトでは設定されていません。詳細については以下の注を参照)を選択するための「 周波数オプション 」ボタンをクリックします。
- EEG機能セクションで、ボタン freq.オプション をクリックして、パワースペクトル密度を計算する全体的な周波数範囲(PSD、デフォルト= 1-40 Hz)、単位(デシベル、μV2 / Hz、または総電力で正規化)、ウィンドウタイプ(デフォルト=ハミング)、ウィンドウのオーバーラップ(デフォルト= 50%)、ウィンドウの長さ(デフォルト= 2秒)、 各バンドの周波数境界のタイプ。
注:HRVパワーは、デフォルトでは正規化されたLomb-Scargleピリオドグラムを使用して計算され、リサンプリングを必要とせず(したがって、元の情報をよりよく保存する)、不均一にサンプリングされたデータ、欠損データ、およびノイズ(NN間隔で典型的)を最適に処理します68。正規化されたバージョンは、信号の分散によって電力をスケーリングし、ノイズレベルの変化に対する感度が低く、周期成分の相対的な強度に重点を置き、異なる録音や被験者間でより比較可能な結果を提供します。その他の方法としては、非正規化Lomb-Scargleピリオドグラム、ウェルチ法、高速フーリエ変換(FFT)などがあります。ウェルチ法とFFT法では、必要な定期的にサンプリングされた時系列を作成するために、リサンプリングが自動的に実行されます。第 2 レベルの正規化は、各周波数帯域の電力を総電力で割ることによって適用でき、各周波数成分の全体的な電力に対する相対的な寄与をより直感的に測定できます。これは、4つのバンドすべてが利用可能で、少なくとも24時間の信号が必要な場合にのみ意味があるため、デフォルトでは無効になっています。これらのアルゴリズムは、Physionet Cardiovascular signal processing toolbox46から適応される。
- 同じ警告メッセージが表示され、検出された余分なPPGチャネルを削除するように確認を求められます。繰り返しになりますが、これは予想通りです。 [はい] をクリックします。BrainBeatsは、ステップ2.4と同様に、ECGデータの前処理とNN間隔の抽出を開始します。次に、時間領域(SDNN、RMSSD、pNN50)、周波数領域(ULF、VLF、LF、HF、LF:HF比、総電力)、非線形領域(Poincare、位相整流信号平均化、ファジーエントロピー、フラクタル次元)のNN間隔から心拍変動(HRV)の特徴を抽出します。
注:BrainBeatsは、ファイルの長さを自動的にチェックして、最小要件が満たされていることを確認し(たとえば、ULF-HRV電源には24時間のデータが必要)、満たされていない場合は警告メッセージを送信し、信頼性の低い推定を防ぐためにこれらの機能をエクスポートしません。BrainBeatsは、HRVメトリック27,45を推定するためのガイドラインと推奨事項に従います。 - BrainBeatsは、ステップ2.7のようにEEGデータを前処理します。(図3を参照)。大きなアーティファクトは、アーティファクト部分空間再構成(ASR)アルゴリズム50,52(デフォルトのSD基準は30に設定され、使用可能なRAMの80%を使用して速度を上げる)を使用して、連続データで自動的に検出されます。これらの大きなアーティファクトは、EEGデータから削除されます(図13を参照)。GUI でこれらのパラメータを調整するには、[Threshold to reject bad segments with ASR] フィールドと [Available RAM to use for ASR] フィールドを選択します。
注:EEGと心血管の時系列は、各信号で個別に推定されるため、特徴モードの時間を同期する必要はありません。したがって、EEGアーティファクトはEEGデータから直接除去できます( 赤、図13)が、アーティファクトを含むエポックが両方の時系列で拒否されたHEPモードとは異なり、その方法にはms精度との時間同期が不可欠であるためです。 - HEPと同じアルゴリズムとパラメータを使用してICAを実行します(ステップ2.11を参照)が、今回は99%の信頼度で検出された場合に心臓成分が除去されます(関連する心臓関連の信号を除去したくないため、HEP/HEO用に保持されました)。
- ステップ3.2で 「周波数領域 」にチェックを入れると、BrainBeatsは、デルタ(1-3 Hz)、シータ(3-7 Hz)、アルファ(8-13 Hz)、ベータ(13-30 Hz)、ガンマ(30+ Hz)周波数帯域の平均パワースペクトル密度(PSD)、個々のアルファ周波数(IAF)、および利用可能なすべての(対称)電極ペアのアルファ非対称性を抽出します。これらの機能を確認してください。
注:PSDをデシベル(dB)に変換すると、録音と被写体間での結果の比較が容易になります。周波数境界は、従来の境界(例えば、アルファバンドのための事前定義された8〜13Hz)または、個人間差69 (例えば、アルファバンドのための7.3〜12.6Hz)を説明するためにパワースペクトル密度の分布から検出される個別化された境界に設定することができる。このアルゴリズムはアルファバンド用に設計されており、特にパワースペクトル分布にピークが存在しない場合、他のバンドではあまり機能しません。個々のアルファ周波数(IAF)は、分割されたピークまたはあいまいなピーク69によりよく対処するために、アルファ重心を使用して推定される。アルファ非対称性は、ガイドライン(50%オーバーラップの2秒ハミングウィンドウ、左チャネルからのアルファパワーの対数から右チャネルからのアルファパワーの対数を引いたもの)に従って計算されます47。したがって、正の値は右よりも左のアルファパワーが大きいことを示し、その逆も同様です。アルファ非対称性は、各電極からのアルファパワーを、すべての電極47をまたいで合計したアルファパワーで割ることによって正規化することができる。対称ペアはシータ距離を使用して取得されるため、EEGデータには電極座標が含まれている必要があります。 - ステップ3.2で 「非線形領域 」にチェックを入れると、BrainBeatsは各脳波チャネルのファジーエントロピーとフラクタル次元を抽出します。
注:非線形領域の特徴は、スペクトル測定では見逃される脳の非線形で複雑なダイナミクスを捉えると考えられており、さまざまな身体システム間の相互作用を調査するために特に有望である70,71,72。ファジーエントロピーは、他の選択肢(サンプルエントロピーと近似エントロピー)よりも信頼性と堅牢性に優れていますが、計算時間が長くなります(特に、高いサンプリングレートでEEG時系列が長い場合)。この問題に対処するために、EEG信号の長さが2分より長く、サンプリングレートが100Hzを超える場合、EEG信号は自動的に90Hzにダウンサンプリングされます(係数が整数でない場合はデシメーションされます)つまり、デフォルトのローパスフィルタに一致させ、ラインノイズアーティファクトを可能な限り回避するために、45Hzのナイキスト周波数に対応します)。さらに、EEGの特徴を推定する際には、並列計算がデフォルトでアクティブになっているため、特に多くのEEGチャネルが利用可能な場合に計算時間を短縮します。 - 最初のGUIで [Visualize outputs ]を選択し(ステップ3.1を参照)、HRVおよびEEGデータ(図14)のパワースペクトル密度(PSD)と、一部のEEG特徴を表示する頭皮トポグラフィー(図15)を表示するプロットを生成します。
注:一部の前処理出力はEEG.brainbeats.preprocessingにあり、使用されているすべてのパラメータはEEG.brainbeats.parametersにあります。私たちは、発見の再現性を高めるために、これらの成果を科学出版物で報告することをユーザーに推奨しています。 - 最初のGUIウィンドウで 「出力を保存 」チェックボックスをオンのままにして、EEGLABの.setファイルにエクスポートされたすべての特徴をEEG.brainbeats.featuresに保存し、データセットがロードされたのと同じフォルダの.matファイルに保存します。
- BrainBeatsはEEGLABの履歴機能をサポートしています。すべての操作の最後に、MATLABのコマンドウィンドウに eegh と入力してコマンドラインを印刷しますこれにより、GUIで手動で選択したパラメーターを使用して、単一のコマンドラインで上記のすべての手順を繰り返すことができるため、操作の自動化と複製が容易になります。
- 上級ユーザーの場合は、次のコマンドを使用して上記のすべての手順を実行します。
EEGLABの;閉める;% GUIなしでEEGLABを起動
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
EEG = pop_loadset('ファイル名','dataset.set',
'ファイルパス',fullfile(main_path, 'sample_data'));% サンプル データセットを読み込みます
脳波 = brainbeats_process(脳波,' 解析',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - 前の手順では、ECG 信号から HRV 特徴を抽出しました。次の手順を使用して、PPG信号からHRV特徴を抽出します(EEG特徴は同じです)。
- 操作によって上書きされた同じデータセットを再度読み込み(手順1.5)、メインGUIを再度開きます(手順2.5)。解析には 「Extract EEG & HRV features 」を選択し、心臓信号の種類には 「PPG 」を、チャンネル名には 「PPG 」を選択します。 [OK] をクリックします。
- 2番目のGUIウィンドウには、PPGの前処理とHRVおよびEEG特徴の抽出のためのパラメータが表示されます。[OK] をクリックして、デフォルトのパラメータで実行します。パラメータについては、ステップ 2.17 で説明します。
- 警告メッセージが表示され、検出されたECGチャネルを削除するかどうかの確認を求められます。ツールボックスは ECG と PPG の両方 (または他の補助チャネル) を同時に解析するようには設計されておらず、データセットに保持すると重大なエラー (アーティファクトの除去、ICA 分解の不良など) が発生するため、これは予想されることです。 [はい] をクリックします。
- BrainBeatsは、ステップ2.5と同様にPPG信号を前処理し、NN間隔を推定します。NN間隔から抽出されたHRV特徴量を、手順3.2と同様に確認します。ただし、NN 間隔は PPG 信号から取得されています。EEG信号は、ステップ3.2と同様に前処理されます。BrainBeatsはPSDをプロットします(図16)。ここでの唯一の違いは、ECGとは対照的に、PPGから得られたNN間隔から推定されたPSDです。
- 上級ユーザーの場合は、次のコマンドを使用して上記のすべての手順を実行します。
EEGLABの;閉める;% GUIなしでEEGLABを起動
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
EEG = pop_loadset('ファイル名','dataset.set','ファイルパス',
fullfile(main_path, 'sample_data'));%サンプルデータセットを読み込みます
脳波 = brainbeats_process(脳波,'分析',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4.脳波信号から心臓のアーチファクトを抽出します。
- サンプルデータセットを読み込みます(ステップ1.5を参照)。
- BrainBeats >1st level(サブジェクトレベル)>ツールをクリックしてメインGUIウィンドウを開き、分析タイプとしてEEG信号から心臓アーティファクトを抽出し、心臓信号タイプとしてECGを選択し、電極ラベルのリストでECGを選択します。[OK] をクリックします。
- 2 番目の GUI ウィンドウには、前処理パラメータが表示されます。[電力線ノイズ] を 50 Hz (ヨーロッパ) に設定し、必要に応じて信頼水準を編集し、[ブースト モード (ベータ)] チェック ボックスをオンにし、[OK] をクリックして、サンプル データセットからの EEG 信号が前処理されていないため、デフォルトのパラメーターで実行します。
注: 心臓の成分を検出するための信頼度はデフォルトで 80% に設定されていますが、データセットによっては低すぎたり高すぎたりすることがあります。この値を大きくすると、心臓の成分が検出される可能性は高くなりますが、検出の信頼性は低下します。ブーストモード(ベータ)はオプションで、心拍信号を脳波信号に塗りつけることで分類性能を向上させることを目的としています。 - 警告メッセージが表示され、検出された余分なPPGチャネルを削除するように確認を求められます。ツールボックスは ECG と PPG の両方 (または他の補助チャネル) を同時に解析するようには設計されておらず、データセットに保持すると重大なエラー (アーティファクトの除去、ICA 分解の不良など) が発生するため、これは予想されることです。 [はい] をクリックします。
- ECG 信号はバンドパス フィルタ処理され、1 Hz 未満の低速周波数ドリフトと 20 Hz を超える高周波ノイズが除去されます (非因果的なゼロフェーズ FIR フィルタを使用)。ステップ3.4のようにEEG信号を前処理します。
- プレコンディショニングされた実データアルゴリズム(PICARD)を使用して、独立成分分析(ICA)を実行します。このオプションを変更するには、ステップ 4.3 で GUI からレプリケーション用の標準 Infomax アルゴリズムまたは変更された Infomax アルゴリズムを選択します。次に、ICLabel を使用して独立したコンポーネントを自動的に分類します。コンポーネントが80%の信頼度で心臓コンポーネントとして分類されている場合、デフォルトでは、EEGデータから自動的に検出および抽出されます。
- 最初のメインGUI(ステップ4.2)でボックスのVisualize出力を保持して、心臓コンポーネントを抽出した後の除去されたコンポーネントの頭皮トポグラフィー(図17左)と最終的なEEG時系列(青、図17右)を視覚化します(赤、図17右)。
注:ECGチャネルは、ECG関連コンポーネントの抽出を確認するために視覚化するために保持されますが、関連情報が含まれなくなったため、この手順の後は削除されます。 - 上級ユーザーの場合は、次のコマンド ラインを使用して次の手順を実行します。
EEGLABの;閉める;% GUIなしでEEGLABを起動
main_path = fileparts(which('eegplugin_BrainBeats.m'));cd(main_path);
EEG = pop_loadset('ファイル名','dataset.set','ファイルパス',
fullfile(main_path, 'sample_data'));
脳波 = brainbeats_process(脳波,'分析','rm_heart',
'heart_signal'、'心電図'、...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true);
結果
まず、BrainBeatsプラグインを使用して、EEGおよびECGデータの前処理、アーティファクトの特定と除去、心拍誘発電位(HEP)と振動(HEO)の分析を行いました。BrainBetsは、ECG信号と一部のRRアーティファクトからRR間隔を正常に検出しました(図2)。また、BrainBeatsはコマンドウィンドウで、心拍の11/305(3.61%)がアーティファクトとしてフラグが立てられ、補間されたと報告しました?...
ディスカッション
プロトコルの重要なステップ
重要な手順については、手順 1.1 から 1.4 で説明します。警告とエラーメッセージは、ツールボックスのさまざまな場所に実装されており、ユーザーが問題が発生する理由を理解するのに役立ちます(例:電極の位置がEEGデータにロードされていない、ファイルの長さが超低周波HRVの信頼性の高い測定値を計算するには短すぎる、信号品質が低すぎて?...
開示事項
著者は何も開示していません。
謝辞
この研究を支援したのが、ノエティック科学研究所です。BrainBeatsの一部のアルゴリズムを開発するために適応されたオリジナルのオープンソースアルゴリズムの開発者に感謝します。
資料
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
参考文献
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved