È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
BrainBeats come plug-in EEGLAB open source per analizzare congiuntamente i segnali EEG e cardiovascolari
In questo articolo
Riepilogo
Il toolbox BrainBeats è un plug-in EEGLAB open source progettato per analizzare congiuntamente i segnali EEG e cardiovascolari (ECG/PPG). Include la valutazione dei potenziali evocati dal battito cardiaco (HEP), l'analisi basata sulle caratteristiche e l'estrazione di artefatti cardiaci dai segnali EEG. Il protocollo aiuterà a studiare l'interazione cervello-cuore attraverso due lenti (HEP e caratteristiche), migliorando la riproducibilità e l'accessibilità.
Abstract
L'interazione tra il cervello e il sistema cardiovascolare sta attirando una maggiore attenzione per il suo potenziale di far progredire la nostra comprensione della fisiologia umana e migliorare i risultati di salute. Tuttavia, l'analisi multimodale di questi segnali è impegnativa a causa della mancanza di linee guida, elaborazione dei segnali standardizzata e strumenti statistici, interfacce utente grafiche (GUI) e automazione per l'elaborazione di grandi set di dati o l'aumento della riproducibilità. Esiste un ulteriore vuoto nei metodi standardizzati di estrazione delle caratteristiche EEG e variabilità della frequenza cardiaca (HRV), che minano la diagnostica clinica o la robustezza dei modelli di apprendimento automatico (ML). In risposta a queste limitazioni, introduciamo la cassetta degli attrezzi BrainBeats. Implementato come plug-in EEGLAB open-source, BrainBeats integra tre protocolli principali: 1) potenziali evocati dal battito cardiaco (HEP) e oscillazioni (HEO) per valutare l'interazione cervello-cuore bloccata nel tempo con una precisione del millisecondo; 2) estrazione di caratteristiche EEG e HRV per esaminare associazioni/differenze tra varie metriche cerebrali e cardiache o per costruire robusti modelli di ML basati su funzionalità; 3) Estrazione automatizzata di artefatti cardiaci dai segnali EEG per rimuovere qualsiasi potenziale contaminazione cardiovascolare durante l'esecuzione dell'analisi EEG. Forniamo un tutorial passo passo per applicare questi tre metodi a un set di dati open source contenente segnali EEG, ECG e PPG simultanei a 64 canali. Gli utenti possono facilmente mettere a punto i parametri per personalizzare le loro esigenze di ricerca uniche utilizzando l'interfaccia utente grafica (GUI) o la riga di comando. BrainBeats dovrebbe rendere la ricerca sull'interazione cervello-cuore più accessibile e riproducibile.
Introduzione
Per molto tempo, l'approccio riduzionista ha dominato l'indagine scientifica in fisiologia e cognizione umana. Questo approccio ha comportato la sezionazione di processi corporei e mentali complessi in componenti più piccoli e più gestibili, consentendo ai ricercatori di concentrarsi sui singoli sistemi in isolamento. Questa strategia è nata a causa delle sfide nello studio della natura intricata e interconnessa del corpo e della mente umana1. Il riduzionismo è stato determinante per comprendere i singoli sottosistemi in isolamento, come chiarire il ruolo dei canali ionici e dei potenziali d'azione per la comunicazione neurale2 o cardiaca3 . Tuttavia, rimane una lacuna significativa nella nostra comprensione di come questi sistemi isolati interagiscono su una scala spaziale e temporale più ampia. Il quadro multimodale (integrativo o ecologico) considera il corpo umano un sistema multidimensionale complesso, dove la mente è vista non come un prodotto del cervello ma come un'attività dell'essere vivente, un'attività che integra il cervello all'interno delle funzioni quotidiane del corpo umano4. Gli approcci multimodali e riduzionisti non sono esclusivi, proprio come non possiamo studiare un neurone senza l'intero cervello o l'intero cervello senza comprendere le proprietà dei singoli neuroni. Insieme, aprono la strada a una comprensione più completa e sinergica della salute umana, della patologia, della cognizione, della psicologia e della coscienza. Il presente metodo mira a facilitare l'indagine multimodale dell'interazione tra il cervello e il cuore fornendo un'analisi congiunta dell'elettroencefalografia (EEG) e dei segnali cardiovascolari, in particolare l'elettrocardiografia (ECG) e la fotopletismografia (PPG). Questo toolbox, implementato come plugin EEGLAB in MATLAB, affronta i limiti metodologici esistenti ed è reso open source per facilitare l'accessibilità e la riproducibilità nell'area scientifica. Implementa le linee guida e le raccomandazioni più recenti nella sua progettazione e nei parametri predefiniti per incoraggiare gli utenti a seguire le migliori pratiche note. Il toolbox proposto dovrebbe essere una risorsa preziosa per i ricercatori e i medici interessati a 1) studiare i potenziali evocati dal battito cardiaco, 2) estrarre caratteristiche dai segnali EEG ed ECG/PPG, o 3) rimuovere gli artefatti cardiaci dai segnali EEG.
Ricerca cuore-cervello
La relazione tra il cuore e il cervello è stata storicamente studiata attraverso metodi di neuroimaging come la risonanza magnetica funzionale (fMRI) e la tomografia a emissione di positroni (PET). Utilizzando questi strumenti, i ricercatori hanno evidenziato alcune regioni cerebrali associate al controllo cardiovascolare (ad esempio, la manipolazione della frequenza cardiaca e della pressione sanguigna5), hanno mostrato l'influenza della frequenza cardiaca sul segnale BOLD6 o hanno identificato potenziali percorsi cervello-corpo che contribuiscono alla malattia coronarica (ad esempio, la pressione sanguigna evocata dallo stress7). Sebbene questi studi abbiano fatto progredire in modo significativo la nostra comprensione della complessa interazione tra il sistema nervoso centrale (SNC) e la funzione cardiovascolare, queste tecniche di neuroimaging sono costose, hanno una disponibilità limitata e sono confinate in ambienti di laboratorio controllati, il che limita la loro praticità per applicazioni nel mondo reale e su larga scala.
Al contrario, l'EEG e l'ECG/PPG sono strumenti più convenienti e portatili che offrono il potenziale per studiare le interazioni cervello-cuore in contesti e popolazioni più diversificati o per periodi più lunghi, fornendo nuove opportunità. L'ECG misura i segnali elettrici generati da ogni battito cardiaco quando il cuore si contrae e si rilassa tramite elettrodi posizionati sulla pelle (di solito sul torace o sulle braccia)8. Il PPG misura le variazioni del volume del sangue nei tessuti microvascolari (ad esempio, il flusso sanguigno e la frequenza cardiaca) utilizzando una sorgente luminosa (ad esempio, LED) e un fotorilevatore (comunemente posizionato sulla punta del dito, sul polso o sulla fronte), basandosi su come il sangue assorbe più luce rispetto al tessuto circostante9. Entrambi i metodi forniscono informazioni preziose sulla funzione cardiovascolare, ma servono a scopi diversi e offrono tipi di dati distinti. Come l'ECG, l'EEG registra i campi elettrici generati dall'attività sincronizzata di migliaia di neuroni corticali che si propagano attraverso la matrice extracellulare, i tessuti, il cranio e il cuoio capelluto fino a raggiungere gli elettrodi posti sulla superficie del cuoio capelluto10. Pertanto, l'uso dell'EEG e dell'ECG/PPG è molto promettente per far progredire la nostra comprensione dei processi fisiologici, cognitivi ed emotivi alla base delle interazioni cervello-cuore e delle loro implicazioni per la salute e il benessere umano. Pertanto, l'acquisizione dell'interazione cuore-cervello da segnali EEG, ECG/PPG con il toolbox BrainBeats può essere particolarmente utile per le seguenti aree scientifiche: diagnostica clinica e previsione, apprendimento automatico dei big data (ML), automonitoraggio del mondo reale11 e imaging mobile cervello/corpo (MoBI)12,13.
Due approcci per l'analisi congiunta dei segnali EEG ed ECG
Esistono due approcci principali per studiare le interazioni tra EEG e segnali cardiovascolari:
I potenziali evocati dal battito cardiaco (HEP) nel dominio del tempo: potenziali correlati agli eventi (ERP) e le oscillazioni evocate dal battito cardiaco (HEO) nel dominio della frequenza temporale: perturbazioni spettrali correlate agli eventi (ERSP) e coerenza inter-trial (ITC). Questo approccio esamina il modo in cui il cervello elabora ogni battito cardiaco. Con una precisione di millisecondi (ms), questo metodo richiede che entrambe le serie temporali siano perfettamente sincronizzate e che i battiti cardiaci siano marcati nei segnali EEG. Questo approccio ha guadagnato interesse negli ultimi anni 14,15,16,17,18,19.
Approccio basato sulle funzionalità: questo approccio estrae le caratteristiche dell'EEG e della variabilità della frequenza cardiaca (HRV) dai segnali continui ed esamina le associazioni tra di loro. Questo è stato fatto in modo indipendente per l'EEG (spesso chiamato EEG quantitativo o qEEG20), ECG 21,22,23 e PPG 24,25,26. Questo approccio presenta applicazioni promettenti catturando variabili relative allo stato e ai tratti. Si noti che, sia per i segnali EEG che cardiovascolari, più lunga è la registrazione, più dominante è la variabile di tratto 27,28,29. Pertanto, le applicazioni dipendono dai parametri di registrazione. Le analisi basate sulle caratteristiche stanno guadagnando un crescente interesse, fornendo nuove metriche quantitative per prevedere lo sviluppo di disturbi mentali e neurologici, la risposta al trattamento o la ricaduta 30,31,32,33,34,35. Questo approccio è particolarmente interessante con set di dati di grandi dimensioni e del mondo reale (ad esempio, clinica, monitoraggio remoto), che possono essere ottenuti più facilmente grazie alle recenti innovazioni nella neurotecnologia indossabile11. Un'applicazione meno esplorata è l'identificazione di associazioni tra specifiche caratteristiche del cervello e del cuore, evidenziando le potenziali dinamiche sottostanti del sistema nervoso centrale. La variabilità della frequenza cardiaca (HRV) può essere calcolata sia dai segnali ECG che PPG. Fornisce informazioni sul sistema nervoso autonomo (ANS) misurando le variazioni degli intervalli di tempo tra i battiti cardiaci (cioè gli intervalli da normale a normale)27. L'aumento dell'attività simpatica (SNS) (ad esempio, durante lo stress o l'esercizio fisico) in genere riduce l'HRV, mentre l'attività parasimpatica (PNS) (ad esempio, durante il rilassamento) la aumenta. Una frequenza respiratoria più lenta generalmente aumenta l'HRV a causa di una maggiore attività del PNS, soprattutto per registrazioni brevi (<10 min)27. Punteggi HRV più alti generalmente suggeriscono un ANS più resiliente e adattabile, mentre un HRV più basso può indicare stress, affaticamento o problemi di salute sottostanti. Registrazioni HRV lunghe (cioè almeno 24 ore) forniscono una prognosi predittiva per varie condizioni di salute, tra cui malattie cardiovascolari, stress, ansia e alcune condizioni neurologiche27. Misure come la pressione sanguigna, la frequenza cardiaca o i livelli di colesterolo forniscono informazioni sullo stato del sistema cardiovascolare. Al contrario, l'HRV aggiunge un aspetto dinamico, mostrando come il cuore risponde e si riprende dallo stress.
I vantaggi di BrainBeats rispetto ai metodi esistenti
Sebbene esistano strumenti, come esaminato di seguito, per elaborare i segnali cardiovascolari ed EEG indipendentemente l'uno dall'altro, non possono essere analizzati congiuntamente. Inoltre, la maggior parte dei mezzi disponibili per elaborare i segnali cardiovascolari comporta licenze costose, non consente l'elaborazione automatizzata (particolarmente vantaggiosa per set di dati di grandi dimensioni), dispone di algoritmi proprietari che impediscono la trasparenza e la riproducibilità o richiede competenze di programmazione avanzate non fornendo un'interfaccia utente grafica (GUI)36. Per quanto ne sappiamo, quattro toolbox MATLAB open source supportano l'analisi HEP/HEO con un'interfaccia grafica: il toolbox37 del kit ecg, il pipeline38 di BeMoBIL, il plugin EEGLAB39 di HEPLAB e il toolbox40 di CARE-rCortex. Sebbene HEPLAB, BeMoBIL ed ecg-kit facilitino l'analisi HEP rilevando i battiti cardiaci e marcandoli nei segnali EEG, non forniscono analisi statistiche o sono limitati al dominio del tempo (cioè HEP). Il plug-in CARE-rCortex ha affrontato questi problemi supportando l'ECG e i segnali respiratori, l'analisi nel dominio del tempo-frequenza, le statistiche e i metodi avanzati di normalizzazione e correzione della linea di base adattati all'analisi HEP/HEO. Tuttavia, utilizza il metodo Bonferroni per la correzione statistica dell'errore di tipo 1 (cioè i falsi positivi), che è troppo conservativo e non fisiologicamente valido per le applicazioni EEG, portando ad un aumento degli errori di tipo II (cioè i falsi negativi)41. Inoltre, il toolbox non offre l'accesso alla riga di comando per l'automazione. Infine, studi recenti sconsigliano i metodi di correzione della linea di base 42,43,44, in quanto riducono il rapporto segnale/rumore (SNR) e sono statisticamente inutili e indesiderabili.
Per risolvere queste limitazioni, introduciamo il toolbox BrainBeats, attualmente implementato come plug-in EEGLAB open source nell'ambiente MATLAB. Incorpora i seguenti vantaggi rispetto ai metodi precedenti:
1) Una GUI facile da usare e funzionalità a riga di comando (per i programmatori che mirano a eseguire l'elaborazione automatizzata). 2) Algoritmi, parametri e linee guida convalidati per l'elaborazione dei segnali cardiovascolari, come il rilevamento dei picchi R, l'interpolazione degli artefatti RR e il calcolo delle metriche HRV (ad esempio, l'impianto di linee guida per windowing, ricampionamento, normalizzazione, ecc.27,45,46). Questo è importante perché Vest et al. hanno dimostrato come modeste differenze in queste fasi di elaborazione possano portare a risultati divergenti, contribuendo alla mancanza di riproducibilità e applicabilità clinica delle metriche HRV46. 3) Algoritmi convalidati, parametri predefiniti e linee guida per l'elaborazione dei segnali EEG, inclusi filtraggio e windowing44,47, rireferenziazione48,49, rimozione di canali anomali e artefatti 50,51,52, decomposizione ICA ottimizzata e classificazione di componenti indipendenti 53,54,55,56. Gli utenti possono mettere a punto tutti i parametri di pre-elaborazione o persino pre-elaborare i propri dati EEG con il metodo preferito prima di utilizzare il toolbox per soddisfare le proprie esigenze (ad esempio, con il plug-in EEGLAB clean_rawdata50,52, la pipeline BeMoBIL38, la pipeline PREP57, ecc.). 4) Potenziali evocati dal battito cardiaco (HEP, cioè nel dominio del tempo) e oscillazioni (HEO; perturbazioni spettrali correlate agli eventi con metodi wavelet o FFT e coerenza inter-trial sono disponibili attraverso il software standard EEGLAB) dai segnali ECG. Le statistiche parametriche e non parametriche con correzioni per errori di tipo 1 sono disponibili tramite il software standard di EEGLAB. Le statistiche non parametriche includono statistiche di permutazione e correzioni spazio-temporali per confronti multipli (ad esempio, clustering spazio-temporale o miglioramento del cluster senza soglia)58,59. Gli utenti possono utilizzare il plug-in LIMO-EEG per implementare la modellazione lineare gerarchica, che tiene conto della varianza all'interno e tra i soggetti e implementa un approccio univariato di massa privo di ipotesi con un controllo robusto per gli errori di tipo I e II60,61. Le analisi statistiche dei dati HEP/HEO possono essere eseguite nei domini del canale e dei componenti indipendenti. 5) Analisi HEP/HEO e HRV da segnali PPG (per la prima volta per HEP/HEO). 6) Supporta per la prima volta l'estrazione congiunta delle caratteristiche EEG e HRV. 7) Il toolbox fornisce varie visualizzazioni di dati per ispezionare i segnali nelle varie fasi di elaborazione necessarie e gli output a livello di soggetto.
| Metodo | Rileva i picchi R dall'ECG | Rileva le onde R da PPG | HEP/HEO | Funzioni EEG e HRV | Rimuovere gli artefatti cardiaci dall'EEG | Interfaccia grafica | Riga di comando |
| kit ECG | X | X | X | X | |||
| BeMoBIL | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| BrainBeats | X | X | X | X | X | X | X |
TABELLA 1: Novità apportate da BrainBeats rispetto a metodi preesistenti e simili.
Informazioni per aiutare i lettori a decidere se il metodo è appropriato per loro
Questo toolbox è adatto a qualsiasi ricercatore o clinico con dati EEG ed ECG/PPG. Il plug-in non supporta ancora l'importazione di segnali EEG ed ECG/PPG da file separati (anche se questa funzione sarà presto disponibile). Il toolbox è adatto a chiunque intenda eseguire analisi HEP/HEO, estrarre le caratteristiche EEG e/o HRV con metodi standardizzati o semplicemente rimuovere gli artefatti cardiaci dai segnali EEG. Vedere la Figura 1 per un diagramma a blocchi che riassume il flusso e i metodi complessivi di BrainBeats.
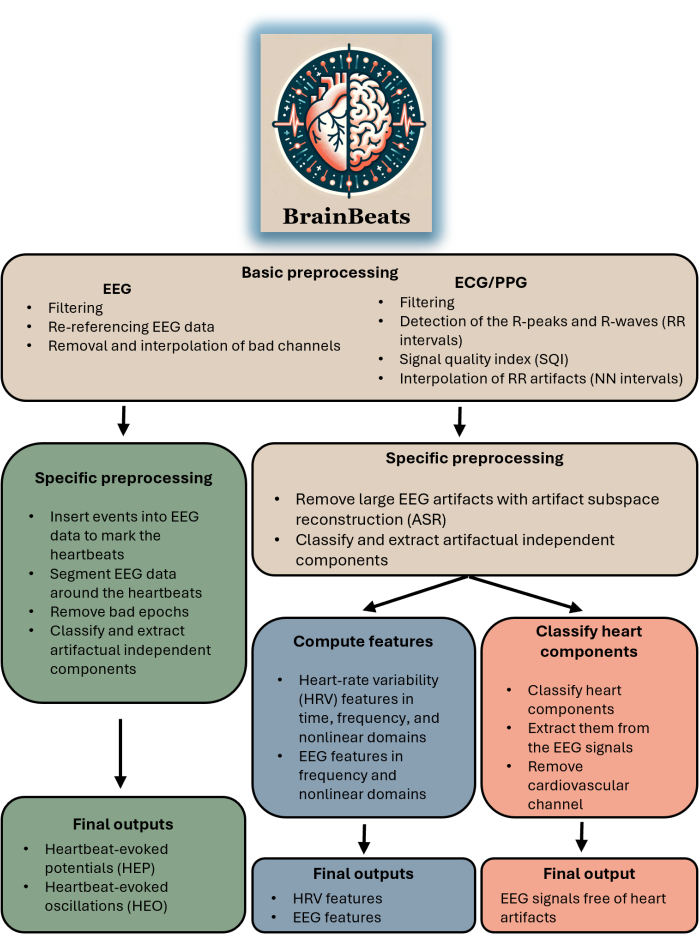
FIGURA 1. Diagramma a blocchi che riassume l'architettura e il flusso complessivi di BrainBeats. Le operazioni comuni ai tre metodi sono marroni. Le operazioni specifiche per i potenziali evocati dal battito cardiaco (HEP) e le oscillazioni (HEO) sono in verde. Le operazioni specifiche per l'estrazione delle caratteristiche EEG e HRV sono blu. Le operazioni specifiche per la rimozione degli artefatti cardiaci dai segnali EEG sono in rosso. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
Il consenso informato è stato ottenuto da ciascun partecipante e il comitato etico dell'Università Federale degli Urali ha approvato il protocollo sperimentale.
1. Requisiti di BrainBeats
- Installa MATLAB ed EEGLAB sul computer. EEGLAB può essere scaricato su https://github.com/sccn/eeglab e decompresso (o clonato per gli utenti Git) ovunque sul computer. Per ulteriori dettagli sull'installazione, vedere la pagina GitHub.
- Aggiungi il percorso alla cartella EEGLAB nel pannello home di MATLAB facendo clic sul pulsante Imposta percorso . Seleziona la cartella eeglab con il file decompresso e fai clic su Salva > Chiudi.
- Avvia EEGLAB digitando eeglab nella finestra di comando di MATLAB.
- Installa il plug-in BrainBeats facendo clic su File > Gestisci estensioni EEGLAB. Digita brainbeats nella barra di ricerca, seleziona il plug-in BrainBeats nell'elenco e fai clic su Installa/Aggiorna.
- Caricare il set di dati di esempio in EEGLAB. Fare clic su File > caricare i dati esistenti. Andate alla cartella EEGLAB, andate alla cartella dei plugin, andate alla cartella BrainBeats e aprite la cartella sample_data. Selezionare il file dataset.set.
NOTA: questo set di dati corrisponde al sub-032 (stato di riposo con gli occhi aperti) da un set di dati multi-soggetto open source62,63. Questa opzione è stata selezionata perché contiene EEG (64 canali), ECG (un canale) e PPG (un canale) simultanei, a una frequenza di campionamento di 1000 Hz. I segnali ECG e PPG sono stati raccolti utilizzando gli ingressi ausiliari del sistema actiCHamp. I dati EEG sono stati registrati con elettrodi attivi posizionati secondo il sistema 10-20, con FCz come riferimento online e Fpz come elettrodo di terra, e con l'impedenza mantenuta al di sotto di 25 kOm. L'ECG è stato registrato utilizzando un elettrodo attivo posizionato sul polso destro, l'elettrodo di riferimento sul polso sinistro e il suolo sull'avambraccio interno sinistro a 3 cm distalmente dal gomito. Il PPG è stato registrato dall'indice sinistro. I dati EEG, ECG e PPG sono sincronizzati nel tempo poiché sono stati registrati contemporaneamente con lo stesso amplificatore. Vedere i riferimenti62,63 per maggiori dettagli. - Seleziona la casella di controllo Salva output per salvare tutto nel file .set corrispondente nella stessa posizione del file originale che è stato caricato in EEGLAB.
NOTA: Per questo tutorial, abbiamo unito i dati EEG, ECG e PPG in un unico set di dati EEGLAB, caricato le coordinate degli elettrodi del metodo degli elementi limite 3D (BEM) e sottocampionato il segnale a 250 Hz per ridurre il peso del file (per il download da parte dell'utente) e accelerare il tempo di calcolo. Poiché questo set di dati non aveva un canale EEG anomalo, abbiamo modificato artificialmente il canale TP9 per illustrare l'algoritmo di rilevamento e rimozione del canale difettoso di BrainBeats. Allo stesso modo, simuliamo artificialmente un grande artefatto EEG all'inizio del file e un artefatto muscolare ad alta frequenza nei canali temporali da 3 a 6 s per illustrare la funzione di rimozione degli artefatti di BrainBeats.
2. Potenziali evocati dal battito cardiaco (HEP) e oscillazioni (HEO)
- Apri la prima interfaccia utente generale (GUI) di BrainBeats per selezionare i parametri principali. In EEGLAB, fare clic su Strumenti > BrainBeats > livello 1( livello materia). Selezionare Heartbeat-evocaked potentials HEP come analisi da eseguire, ECG come tipo di dati cardiaci e fare clic sul pulsante per visualizzare l'elenco dei canali per selezionare il canale ECG etichettato ECG (o digitarlo direttamente nella casella di testo). Mantieni selezionate le opzioni Visualizza output (consigliato) e Salva output e fai clic su Ok.
- Viene visualizzata una seconda finestra GUI in base alle scelte precedenti (ad esempio, analisi HEP con dati ECG). Selezionare i parametri di pre-elaborazione per i segnali EEG ed ECG. Disattiva la pre-elaborazione deselezionando le caselle Pre-elaborazione ECG e Pre-elaborazione EEG per verificare se i dati sono già stati pre-elaborati prima dell'avvio di BrainBeats. Modificare il rumore della linea elettrica a 50 Hz nella sezione EEG di pre-elaborazione poiché questi dati EEG sono stati registrati in Russia. Fare clic su OK per avviare.
- Verrà visualizzato un messaggio di avviso che richiede la conferma della rimozione del canale PPG rilevato. Questo perché il toolbox non è progettato per analizzare contemporaneamente sia l'ECG che il PPG (o altri canali ausiliari) e mantenerlo nel set di dati porterà a gravi errori (ad esempio, rimozione di artefatti, scarsa decomposizione ICA, ecc.). Fare clic su Sì. BrainBeats inizia a eseguire alcuni controlli, impostando alcuni parametri predefiniti e separa l'ECG dai dati EEG per pre-elaborare il segnale ECG e calcolare gli intervalli RR.
- Le serie temporali ECG e RR sono pre-elaborate utilizzando algoritmi convalidati dal toolbox Physionet Cardiovascular Signal46,64 adattati per adattarsi alla formattazione dei dati di BrainBeats, aumentare la chiarezza, la regolazione dei parametri e il tempo di calcolo (vedere i riferimenti per la convalida degli algoritmi). Il plug-in emette gli intervalli RR, i timestamp, il segnale ECG filtrato, gli indici dei picchi R e la frequenza cardiaca (FC). Regola questi parametri tramite la GUI o la riga di comando.
NOTA: Il segnale ECG viene filtrato passa-banda utilizzando un filtro personalizzato e convalidato (1-30 Hz) e scansiona il segnale per identificare il complesso QRS e i picchi R utilizzando il metodo Pan-Tompkins (P&T)65, implementando alcune operazioni di elaborazione del segnale tra cui differenziazione, quadratura, integrazione e livellamento per ottenere le migliori prestazioni. La soglia di energia P&T viene stimata in base alla frequenza di campionamento e ai valori ECG levigati per evitare interruzioni dovute a grandi protuberanze. Se la variabilità dell'intervallo RR supera 1,5 volte la mediana, cerca i picchi mancati. Il segno medio del picco R viene calcolato su segmenti di 30 s e i punti di picco vengono perfezionati attraverso un controllo del periodo refrattario, gestendo le condizioni di linea piatta e garantendo un rilevamento coerente. - Successivamente, BrainBeats identifica gli intervalli RR anomali o i picchi all'interno degli intervalli RR utilizzando una ricerca avanti-indietro e soglie fisiologiche. L'indice di qualità del segnale (SQI) è calcolato46; verificare questa opzione se il sistema visualizza avvisi in cui più del 20% delle serie temporali RR contiene artefatti RR (al di fuori dei limiti fisiologici o con un SQI inferiore a 0,9). Un grafico mostra il segnale ECG filtrato, i picchi R identificati, gli intervalli NN e gli artefatti interpolati (vedi Figura 2).
NOTA: gli artefatti RR vengono interpolati per impostazione predefinita utilizzando il metodo cubico a tratti che preserva la forma per ottenere gli intervalli da normale a normale (NN), ma rimuoverli (non consigliato) o utilizzare un altro metodo di interpolazione (lineare, cubico, vicino più prossimo, vicino precedente/successivo, spline, convoluzione cubica o cubico di Akima modificato) se necessario. Quando sono presenti più canali ECG, gli intervalli RR vengono stimati per ciascuno e viene selezionato il canale con il minor numero di artefatti RR per i passaggi successivi. - Scorrere le finestre ingrandite di 30 s dei picchi R per un'ispezione più ravvicinata premendo le frecce destra/sinistra. Se i dati contengono più canali ECG/PPG, utilizzare il canale con il miglior indice di qualità del segnale per gli intervalli RR. Al momento, BrainBeats non supporta contemporaneamente i segnali ECG e PPG. A scopo di esercitazione, è stato scelto un set di dati di esempio che contiene entrambi i tipi di dati.
- Una volta terminato il segnale ECG, BrainBeats filtra i dati EEG a 1-40 Hz utilizzando un filtro FIR causale a fase minima non lineare per ridurre l'attività di striscio tra i periodi pre e post battito cardiaco, preservare la causalità ed evitare ritardi di gruppo indesiderati44. Ciò è particolarmente importante per gli utenti che esaminano il periodo pre-heartbeat. Se il filtro passa-basso è impostato su un valore superiore alla frequenza della linea di alimentazione (ad esempio, passa-basso a 80 Hz con frequenza della linea di alimentazione a 50 Hz), utilizzare un filtro a tacca affilata per rimuovere l'artefatto del rumore di linea. I dati EEG vengono quindi rireferenziati all'infinito utilizzando l'algoritmo REST (più adatto per l'analisi HEP49) a meno che non vengano rilevati meno di 30 canali (nel qual caso non possono essere rireferenziati in modo affidabile e viene generato un avviso per informare gli utenti).
- BrainBeats rileva, rimuove e interpola quindi i canali EEG anomali (Figura 3). Verificare che i parametri predefiniti siano linee piatte superiori a 5 s (algoritmo clean_flatlines), una deviazione standard massima del rumore ad alta frequenza di 10, una lunghezza della finestra di 5 s (per catturare meglio gli artefatti a bassa frequenza52), una correlazione minima tra canali vicini di 0,65 e una parte massima tollerata del 33% (algoritmo clean_channels).
NOTA: Il numero di campioni RANSAC è impostato su 500 per impostazione predefinita per aumentare la convergenza e la replicabilità della reiezione del canale errato (sebbene aumenti il tempo di calcolo). - Successivamente, inserire i picchi R come marcatori di eventi nei dati EEG per contrassegnare ogni battito cardiaco e segmentare i dati attorno a questi marcatori senza rimozione al basale (secondo le linee guida43,66; Figura 4). Poiché gli intervalli NN hanno lunghezze diverse e l'EEG deve essere segmentato a una lunghezza costante, stimare il limite minimo della dimensione dell'epoca a seguito di eventi di picco R utilizzando il 5° percentile dei dati dell'intervallo interbeat (IBI) (cioè, il valore al di sotto del quale cade il 5% degli IBI più brevi, visualizzato come una linea rossa tratteggiata su un istogramma; vedi Figura 5).
NOTA: Questo valore del 5° percentile è un buon compromesso per preservare il maggior numero possibile di epoche, garantendo al contempo che non siano troppo brevi, poiché il periodo di interesse per l'analisi HEP/HEO è di 200-600 ms dopo l'heartbeat49,67. - Segmentare i dati EEG da -300 ms prima dei picchi R al valore del 5° percentile dopo il picco R, con il picco R al tempo 0. Rifiuta epoche inferiori a 550 ms o contenenti più di un picco R (che distorcerebbe l'ERP/ERSP), secondo le linee guida49,67. Le epoche contenenti grandi artefatti EEG vengono rilevate utilizzando le metriche RMS (Root-Mean-Square) e SNR (Signal-to-Noise Ratio) e la funzione isoutlier di MATLAB (Figura 6). Le epoche artefattiche sono state rimosse.
- Esegui la separazione cieca della fonte utilizzando l'algoritmo Infomax predefinito, tenendo conto della classificazione dei dati per ottenere le migliori prestazioni53. Se lo si desidera, scegliere l'algoritmo di analisi dei componenti indipendenti precondizionati (PICARD) per il calcolo veloce54,54 scegliendo l'opzione Picard (veloce) o l'algoritmo Infomax modificato scegliendo l'opzione Infomax modificato (lungo ma replicabile) per il metodo ICA di campo (anche se questo comporta tempi di calcolo molto più lunghi). Utilizzare il plug-in ICLabel56 per classificare automaticamente gli IC per estrarre artefatti non cerebrali (i componenti oculari vengono rimossi con una confidenza del 90%, mentre il rumore muscolare, di linea e di canale vengono rimossi con una confidenza del 99%); Figura 7).
- Mantenere selezionata l'opzione Visualizza uscite nella prima finestra della GUI per visualizzare la serie temporale EEG finale, l'HEP medio generale (Figura 8 in alto), l'HEP per ogni battito cardiaco (Figura 8 in basso) e le oscillazioni evocate dal battito cardiaco medio generale (HEO; Figura 9). Esaminare gli HEO in termini di perturbazione spettrale correlata agli eventi (ERSP, ovvero cambiamenti nella potenza dell'EEG attraverso i battiti cardiaci; Figura 9 in alto) e la coerenza della fase inter-trial (ITC, cioè la coerenza dell'angolo di fase tra i battiti cardiaci; Figura 9 in basso).
NOTA: L'ERSP viene calcolato utilizzando una wavelet predefinita a 3 cicli (con una finestra conica di Hanning applicata, rapporto pad di 2) e con il numero di cicli nelle wavelet utilizzate per le frequenze più alte che si espande lentamente fino al 20% del numero di cicli nella finestra FFT equivalente alla sua frequenza più alta (1 meno 0,8). Controlla le forme delle singole finestre misurate dalla funzione e le relative forme nei riquadri di tempo/frequenza risultanti. Una linea di base arbitraria viene rimossa a scopo illustrativo e l'ERSP viene calcolato per frequenze 7-25 Hz per catturare il tipico effetto HEO descritto in letteratura, vale a dire 300-450 ms dopo il battito cardiaco nella banda alfa (8-13 Hz) su elettrodi frontocentrali17,67. Le frequenze più basse non possono essere stimate a causa della breve dimensione dell'epoca definita dagli intervalli di interbeat. Le statistiche non parametriche (permutazione) vengono applicate per visualizzare l'HEO per un valore p di 0,05, corretto per il tasso di false scoperte (FDR, cioè errore di tipo 1 o errore per famiglia). Questi grafici vengono generati a scopo di esercitazione o di analisi di prove singole. - Vengono generati grafici di pre-elaborazione per visualizzare i diversi passaggi. Per disattivarlo, deseleziona la casella Visualizza pre-elaborazioni. I dati EEG finali (puliti e segmentati attorno ai picchi R) non includono i dati ECG poiché distorcerebbero l'analisi ERP/ERSP. Per mantenere il canale del cuore nell'output finale, selezionare la casella Mantieni canale del cuore.
NOTA: Mettere in pausa qui, se necessario, prima di elaborare il file successivo (condizione o partecipante successivo). - BrainBeats supporta la funzione di cronologia di EEGLAB. Al termine di tutte le operazioni, digitare eegh nella finestra di comando di MATLAB per stampare la riga di comando in modo da ripetere tutti i passaggi precedenti tramite un'unica riga di comando, con i parametri selezionati manualmente nella GUI, consentendo una facile automazione. Trova gli output di pre-elaborazione (ad esempio, l'indice di qualità del segnale delle serie temporali cardiovascolari, gli intervalli NN, i canali EEG rimossi, i segmenti e i componenti, ecc.) nella struttura EEGLAB: EEG.brainbeats.preprocessings. Tutti i parametri vengono esportati anche in EEG.brainbeats.parameters.
- Per gli utenti esperti, eseguire tutti i passaggi precedenti con i parametri predefiniti con le seguenti righe di comando (vedere lo script del tutorial nel repository BrainBeats per ulteriori opzioni):
eeglab; chiudere; % Avvia EEGLAB senza la GUI
main_path = fileparts(quale('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome file','dataset.set',
'percorsofile',file completo(main_path, 'sample_data')); %Carica il set di dati di esempio
EEG = brainbeats_process(EEG,'analisi',
'hep','heart_signal', 'ECG', 'heart_channels',
{'ECG'},'clean_eeg',true); % Avvia BrainBeats 1° livello per elaborare il file per l'analisi HEP con parametri predefiniti - I passaggi precedenti hanno eseguito HEP/HEO dal segnale ECG. Utilizzare i seguenti passaggi per il segnale PPG.
- Nei passaggi seguenti, eseguire le stesse operazioni ma utilizzando un segnale PPG. Carica nuovamente lo stesso set di dati (vedi passaggio 1.5) poiché le operazioni precedenti lo hanno sovrascritto e apri nuovamente la prima GUI di BrainBeats per selezionare i parametri principali. Fare clic su Strumenti > BrainBeats > 1° livello (livello materia). Selezionare Potenziali evocati dal battito cardiaco (HEP) come analisi da eseguire, PPG come tipo di dati cardiaci e fare clic sul pulsante per visualizzare l'elenco dei canali per selezionare il canale PPG. Mantenere selezionate le opzioni Visualizza output (scelta consigliata) e Salva output e fare clic su OK.
- La seconda finestra della GUI viene visualizzata in modo simile al passaggio 2.2 . L'unica differenza è che ora possiamo mettere a punto il parametro per elaborare il segnale PPG invece del segnale ECG. Fare clic su OK per eseguire l'esecuzione con i parametri predefiniti.
- Viene visualizzato un messaggio di avviso che richiede la conferma per rimuovere il canale ECG aggiuntivo rilevato. Ancora una volta, questo è previsto. Fare clic su Sì. Per impostazione predefinita, il toolbox pre-elabora il segnale PPG, rileva le onde del polso per ottenere gli intervalli RR, identifica eventuali artefatti RR e li interpola (Figura 10). Passaggi 2.7. al punto 2.12. e vengono generati gli stessi grafici e gli stessi output, ma in base alle onde R rilevate dal segnale PPG (vedere Figura 11 e Figura 12).
NOTA: Le onde R vengono rilevate utilizzando la pendenza del segnale all'interno di una finestra specificata. Gli impulsi potenziali vengono quindi contrassegnati quando la pendenza supera una soglia dinamica, che viene regolata in base alla cronologia dei rilevamenti e alle caratteristiche del segnale. L'algoritmo cerca quindi all'interno di un periodo di chiusura dell'occhio per individuare la pendenza massima e, successivamente, l'inizio dell'onda del polso viene determinato attraverso la soglia. I picchi delle onde R sono identificati come le valli vicine all'inizio e le loro posizioni sono registrate. L'algoritmo scorre l'intero segnale, regolando continuamente le soglie di rilevamento e identificando i picchi delle onde R, che vengono poi utilizzati per calcolare gli intervalli RR. - Per gli utenti esperti, eseguire tutti i passaggi precedenti con i parametri predefiniti con le seguenti righe di comando (vedere lo script del tutorial nel repository BrainBeats per ulteriori opzioni):
eeglab; chiudere; % Avvia EEGLAB senza la GUI
main_path = fileparts(quale('eegplugin_
BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome file','dataset.set',
'percorsofile',file completo(main_path, 'sample_data')); %Carica il set di dati di esempio
EEG = brainbeats_process(EEG,'analisi',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',true); % Avvia BrainBeats 1° livello per elaborare il file per l'analisi HEP con parametri predefiniti
3. Estrazione delle caratteristiche EEG e HRV
- Caricare nuovamente lo stesso set di dati (vedere il passaggio 1.5; Fare clic su File > Carica set di dati esistente > Seleziona dataset.set) poiché è stato sovrascritto dalle operazioni precedenti e aprire nuovamente la GUI principale per selezionare i parametri principali (passaggio 2.1; Fare clic su Strumenti > BrainBeats > 1° livello). Selezionare Estrai funzioni EEG e HRV per il tipo di analisi, ECG per il tipo di segnale cardiaco e selezionare ECG nell'elenco delle etichette degli elettrodi. Fare clic su OK.
- Viene visualizzata la seconda finestra dell'interfaccia grafica come nel passaggio 2.2, ma con parametri diversi per la pre-elaborazione EEG e l'estrazione delle funzioni HRV ed EEG. Impostare questi parametri come descritto di seguito. Fare clic su OK per avviare con i parametri predefiniti.
- Nella sezione HRV, fare clic sul pulsante opzioni frequenti per selezionare il metodo di calcolo della potenza HRV (impostazione predefinita sul periodogramma di Lomb-Scargle normalizzato), la sovrapposizione della finestra (impostazione predefinita al 25%) e per l'esecuzione di una normalizzazione di secondo livello (non impostata per impostazione predefinita; vedere la nota sotto per maggiori dettagli).
- Nella sezione Caratteristiche EEG, cliccare sul pulsante frequenzimetro opzioni per mettere a punto alcuni parametri, come l'intervallo di frequenza complessivo su cui calcolare la densità spettrale di potenza (PSD; default = 1-40 Hz), le unità (decibel, μV2/Hz, o normalizzate dalla potenza totale), il tipo di finestra (default = hamming), la sovrapposizione della finestra (default = 50%), la lunghezza della finestra (default = 2 s), e i tipi di limiti di frequenza per ciascuna banda.
NOTA: La potenza HRV viene calcolata per impostazione predefinita utilizzando il periodogramma Lomb-Scargle normalizzato, che non richiede il ricampionamento (quindi preserva meglio le informazioni originali) e si occupa meglio di dati campionati in modo non uniforme, dati mancanti e rumore (tipico con intervalli NN)68. La versione normalizzata scala la potenza in base alla varianza del segnale, fornendo risultati meno sensibili ai livelli di rumore variabili, più focalizzati sulla forza relativa dei componenti periodici e più comparabili tra diverse registrazioni o soggetti. Altri metodi disponibili includono il periodogramma di Lomb-Scargle non normalizzato, il metodo Welch e la trasformata di Fourier veloce (FFT). Il ricampionamento viene eseguito automaticamente per i metodi Welch e FFT per creare le serie temporali campionate regolarmente necessarie. Una normalizzazione di secondo livello può essere applicata dividendo la potenza di ciascuna banda di frequenza per la potenza totale, fornendo una misura più intuitiva del contributo relativo di ciascun componente di frequenza alla potenza complessiva. È disabilitato per impostazione predefinita poiché è significativo solo quando tutte e quattro le bande sono disponibili, richiedendo almeno 24 ore di segnale. Questi algoritmi sono adattati dal toolbox di elaborazione del segnale cardiovascolare Physionet46.
- Viene visualizzato lo stesso messaggio di avviso, che richiede la conferma di rimuovere il canale PPG aggiuntivo rilevato. Ancora una volta, questo è previsto. Fare clic su Sì. BrainBeats inizierà la pre-elaborazione dei dati ECG e l'estrazione degli intervalli NN in modo identico, come nel passaggio 2.4. Quindi, estrae le caratteristiche di variabilità della frequenza cardiaca (HRV) dagli intervalli NN nel tempo (SDNN, RMSSD, pNN50), nella frequenza (ULF, VLF, LF, HF, LF:HF ratio, potenza totale) e non lineari (Poincaré, media del segnale rettificata in fase, entropia fuzzy e dimensione frattale).
NOTA: BrainBeats controlla automaticamente la lunghezza del file per garantire che i requisiti minimi siano soddisfatti (ad esempio, l'alimentazione ULF-HRV richiede 24 ore di dati), invia messaggi di avviso in caso contrario e non esporta queste funzioni per evitare stime inaffidabili. BrainBeats segue le linee guida e le raccomandazioni per la stima delle metriche HRV27,45. - BrainBeats preelabora i dati EEG come nel passaggio 2.7 . (vedi Figura 3). Gli artefatti di grandi dimensioni vengono rilevati automaticamente nei dati continui utilizzando l'algoritmo di ricostruzione del sottospazio degli artefatti (ASR)50,52 (criterio SD predefinito impostato su 30 e utilizzando l'80% della RAM disponibile per aumentare la velocità). Questi grandi artefatti vengono rimossi dai dati EEG (vedi Figura 13). Per regolare questi parametri nella GUI, selezionare i campi Soglia per rifiutare i segmenti danneggiati con ASR e RAM disponibile da utilizzare per ASR.
NOTA: Non è necessario sincronizzare l'ora delle serie temporali EEG e cardiovascolari per la modalità funzioni, poiché le caratteristiche sono stimate separatamente su ciascun segnale. Pertanto, gli artefatti EEG possono essere rimossi direttamente dai dati EEG (in rosso, Figura 13), a differenza della modalità HEP, dove le epoche contenenti artefatti sono state rifiutate per entrambe le serie temporali perché la sincronizzazione temporale con l'accuratezza ms è essenziale per quel metodo. - Eseguire l'ICA utilizzando gli stessi algoritmi e parametri dell'HEP (vedere il passaggio 2.11.), tranne per il fatto che questa volta i componenti cardiaci vengono rimossi se rilevati con un'affidabilità del 99% (sono stati conservati per HEP/HEO poiché non vogliamo rimuovere i segnali cardiaci rilevanti).
- La casella Dominio della frequenza è selezionata nel passaggio 3.2., quindi BrainBeats estrae le seguenti caratteristiche del dominio della frequenza: la densità spettrale di potenza media (PSD) per le bande di frequenza delta (1-3 Hz), theta (3-7 Hz), alfa (8-13 Hz), beta (13-30 Hz) e gamma (30+ Hz), la frequenza alfa individuale (IAF) e l'asimmetria alfa su tutte le coppie di elettrodi (simmetriche) disponibili. Controlla queste funzionalità.
NOTA: La PSD convertita in decibel (dB) facilita il confronto dei risultati tra registrazioni e soggetti. I limiti di frequenza possono essere impostati sui limiti convenzionali (ad esempio, 8-13 Hz predefiniti per la banda alfa) o sui limiti individualizzati, che vengono rilevati dalla distribuzione della densità spettrale di potenza per tenere conto delle differenze interindividuali69 (ad esempio, 7,3-12,6 Hz per la banda alfa). L'algoritmo è stato progettato per la banda alfa e non funziona altrettanto bene per altre bande, soprattutto quando i picchi non sono presenti nella distribuzione spettrale di potenza. La frequenza alfa individuale (IAF) viene stimata utilizzando il baricentro alfa per gestire meglio i picchi divisi o i picchi ambigui69. L'asimmetria alfa viene calcolata seguendo le linee guida (finestra di Hamming di 2 secondi con sovrapposizione del 50%, logaritmo della potenza alfa dal canale sinistro meno logaritmo della potenza alfa dal canale destro)47. Quindi, valori positivi indicano una maggiore potenza alfa sinistra che destra e viceversa. L'asimmetria alfa può essere normalizzata dividendo la potenza alfa di ciascun elettrodo per la potenza alfa sommata tra tutti gli elettrodi47. Le coppie simmetriche sono ottenute utilizzando le distanze theta, richiedendo che i dati EEG contengano le coordinate degli elettrodi. - La casella Dominio non lineare è selezionata nel passaggio 3.2., BrainBeats estrae l'entropia fuzzy e la dimensione frattale per ogni canale EEG.
NOTA: Si ritiene che le caratteristiche del dominio non lineare catturino le dinamiche complesse e non lineari del cervello che non vengono rilevate dalle misure spettrali e mostrino una particolare promessa per lo studio delle interazioni tra vari sistemi corporei 70,71,72. L'entropia fuzzy è più affidabile e robusta delle sue alternative (entropia campionaria e approssimativa) ma richiede tempi di calcolo più lunghi (specialmente con lunghe serie temporali EEG con alte frequenze di campionamento). Per risolvere questo problema, quando i segnali EEG durano più di 2 minuti con una frequenza di campionamento superiore a 100 Hz, vengono automaticamente sottocampionati (o decimati quando il fattore non è un numero intero) a 90 Hz (cioè corrispondenti a una frequenza di Nyquist di 45 Hz, per corrispondere al filtro passa-basso predefinito ed evitare il più possibile artefatti di rumore di linea). Inoltre, il calcolo parallelo è attivato per impostazione predefinita durante la stima delle caratteristiche EEG, il che riduce il tempo di calcolo, soprattutto quando sono disponibili molti canali EEG. - Selezionare Visualizza output nella prima GUI (vedere il passaggio 3.1.), per generare un grafico che mostra la densità spettrale di potenza (PSD) per i dati HRV ed EEG (Figura 14) insieme alle topografie del cuoio capelluto che mostrano alcune caratteristiche EEG (Figura 15).
NOTA: È possibile trovare anche alcuni output di pre-elaborazione in EEG.brainbeats.preprocessing e tutti i parametri utilizzati in EEG.brainbeats.parameters. Incoraggiamo gli utenti a riportare questi risultati in pubblicazioni scientifiche per aumentare la replicabilità dei risultati. - Mantenere selezionata la casella Salva output nella prima finestra della GUI per salvare tutte le funzionalità esportate nel file .set EEGLAB in EEG.brainbeats.features e salvarle in un file .mat nella stessa cartella in cui è stato caricato il set di dati.
- BrainBeats supporta la funzione di cronologia di EEGLAB. Al termine di tutte le operazioni, digitare eegh nella finestra di comando di MATLAB per stampare la riga di comando che permetterà di ripetere tutti i passaggi precedenti tramite un'unica riga di comando, con i parametri che sono stati selezionati manualmente nella GUI, consentendo una facile automazione e replica delle operazioni.
- Per gli utenti avanzati, eseguire tutti i passaggi precedenti con il seguente comando:
eeglab; chiudere; % Avvia EEGLAB senza la GUI
main_path = fileparts(quale('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome file','dataset.set',
'percorsofile',file completo(main_path, 'sample_data')); % Caricare il set di dati di esempio
EEG = brainbeats_process(EEG,' analisi',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - I passaggi precedenti hanno estratto le caratteristiche HRV dal segnale ECG. Utilizzare i seguenti passaggi per estrarre le funzioni HRV dal segnale PPG (le funzioni EEG sono le stesse).
- Caricare nuovamente lo stesso set di dati (passaggio 1.5.) poiché è stato sovrascritto dalle operazioni e aprire nuovamente la GUI principale (passaggio 2.5.). Selezionare Estrai funzioni EEG e HRV per l'analisi e selezionare PPG per il tipo di segnale cardiaco e PPG per il nome del canale. Fare clic su OK.
- La seconda finestra dell'interfaccia grafica mostra ora i parametri per la pre-elaborazione del PPG e per l'estrazione delle funzioni HRV ed EEG. Fare clic su OK per eseguire l'esecuzione con i parametri predefiniti. I parametri sono descritti nel passaggio 2.17.
- Apparirà un messaggio di avviso, che richiede la conferma di rimuovere il canale ECG rilevato. Questo è prevedibile poiché il toolbox non è progettato per analizzare contemporaneamente sia l'ECG che il PPG (o altri canali ausiliari) e mantenerlo nel set di dati porterà a gravi errori (ad esempio, rimozione di artefatti, scarsa decomposizione ICA, ecc.). Fare clic su Sì.
- BrainBeats preelabora il segnale PPG e stima gli intervalli NN come nel passaggio 2.5. Controllare le caratteristiche HRV estratte dagli intervalli NN, come nel passaggio 3.2. tranne per il fatto che gli intervalli NN sono stati ottenuti dal segnale PPG. I segnali EEG vengono pre-elaborati come al punto 3.2. BrainBeats traccia il PSD (Figura 16). L'unica differenza qui è la PSD stimata dagli intervalli NN ottenuti dal PPG rispetto all'ECG.
- Per gli utenti avanzati, eseguire tutti i passaggi precedenti con il seguente comando:
eeglab; chiudere; % Avvia EEGLAB senza la GUI
main_path = fileparts(quale('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome file','dataset.set','percorso file',
fullfile(main_path, 'sample_data')); %Carica il set di dati di esempio
EEG = brainbeats_process(EEG,'analisi',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. Estrarre gli artefatti cardiaci dai segnali EEG.
- Caricare il set di dati di esempio (vedere il passaggio 1.5.).
- Aprire la finestra principale della GUI facendo clic su Strumenti > BrainBeats > 1° livello (livello soggetto) e selezionare Estrai artefatti cardiaci dai segnali EEG per il tipo di analisi, ECG per il tipo di segnale cardiaco e selezionare ECG nell'elenco delle etichette degli elettrodi. Fare clic su OK.
- La seconda finestra GUI mostra i parametri di pre-elaborazione. Impostare il rumore della linea elettrica su 50 Hz (Europa), modificare il livello di confidenza se necessario, selezionare la casella Modalità boost (beta) e fare clic su OK per eseguire con i parametri predefiniti poiché i segnali EEG del set di dati campione non sono pre-elaborati.
NOTA: il livello di confidenza per rilevare i componenti cardiaci è impostato su 80% per impostazione predefinita, che potrebbe essere troppo basso o troppo alto per alcuni set di dati. L'aumento di questo valore aumenterà le possibilità di rilevare i componenti del cuore, ma diminuirà l'affidabilità di tale rilevamento. La modalità Boost (beta) è opzionale e mira a migliorare le prestazioni di classificazione spalmando il segnale cardiaco nei segnali EEG. - Verrà visualizzato un messaggio di avviso che richiede la conferma per rimuovere il canale PPG aggiuntivo che è stato rilevato. Questo è prevedibile poiché il toolbox non è progettato per analizzare contemporaneamente sia l'ECG che il PPG (o altri canali ausiliari) e mantenerlo nel set di dati porterà a gravi errori (ad esempio, rimozione di artefatti, scarsa decomposizione ICA, ecc.). Fare clic su Sì.
- Il segnale ECG è filtrato passa-banda per rimuovere le derive di frequenza lente inferiori a 1 Hz e il rumore ad alta frequenza superiore a 20 Hz (con un filtro FIR a fase zero non causale). Pre-elaborare i segnali EEG come al punto 3.4.
- Esegui l'analisi dei componenti indipendenti (ICA) utilizzando l'algoritmo PICARD (ICA for real data ) precondizionato. Per modificare questa opzione, scegliere l'algoritmo Infomax standard o l'algoritmo Infomax modificato per la replica dalla GUI nel passaggio 4.3. Quindi, classificare automaticamente i componenti indipendenti con ICLabel. Se un componente è classificato come componente cardiaco con un'affidabilità dell'80%, per impostazione predefinita viene rilevato ed estratto automaticamente dai dati EEG.
- Conservare la casella Visualizza output nella prima GUI principale (passaggio 4.2.) per visualizzare la topografia del cuoio capelluto del componente rimosso (Figura 17 a sinistra) e la serie temporale EEG finale (in blu, Figura 17 a destra) dopo l'estrazione del componente cardiaco (in rosso, Figura 17 a destra).
NOTA: il canale ECG viene conservato per la visualizzazione per confermare l'estrazione dei componenti correlati all'ECG, ma viene rimosso dopo questo passaggio poiché non contiene più alcuna informazione rilevante. - Per gli utenti esperti, eseguire questi passaggi utilizzando le seguenti righe di comando:
eeglab; chiudere; % Avvia EEGLAB senza la GUI
main_path = fileparts(quale('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome file','dataset.set','percorso file',
fullfile(main_path, 'sample_data'));
EEG = brainbeats_process(EEG,'analisi','rm_heart',
«heart_signal», «ECG», ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true);
Risultati
In primo luogo, il plug-in BrainBeats è stato utilizzato per preelaborare i dati EEG ed ECG, identificare e rimuovere gli artefatti e analizzare i potenziali evocati dal battito cardiaco (HEP) e le oscillazioni (HEO). BrainBeats ha rilevato con successo gli intervalli RR dal segnale ECG e alcuni artefatti RR (Figura 2). BrainBeats ha anche segnalato nella finestra di comando che 11/305 (3,61%) dei battiti cardiaci sono stati contrassegnati come artefatti e interpolati. L'indice medio di qua...
Discussione
Passaggi critici nel protocollo
I passaggi critici sono descritti nei passaggi 1.1-1.4. Avvisi e messaggi di errore sono implementati in vari punti della cassetta degli attrezzi per aiutare gli utenti a capire perché potrebbero riscontrare problemi (ad esempio, posizioni degli elettrodi non caricate nei dati EEG, lunghezza del file troppo corta per calcolare una misura affidabile di HRV a frequenza ultrabassa, qualità del segnale troppo bassa per qualsiasi analisi affidabile, ecc.). Ogni funzione è...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
L'Istituto di Scienze Noetiche ha sostenuto questa ricerca. Ringraziamo gli sviluppatori degli algoritmi open source originali che sono stati adattati per sviluppare alcuni degli algoritmi di BrainBeats.
Materiali
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Riferimenti
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon