Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
BrainBeats como plugin de EEGLAB de código abierto para analizar conjuntamente señales EEG y cardiovasculares
En este artículo
Resumen
La caja de herramientas BrainBeats es un complemento de EEGLAB de código abierto diseñado para analizar conjuntamente señales de EEG y cardiovasculares (ECG/PPG). Incluye la evaluación de potenciales evocados por latidos cardíacos (HEP), análisis basado en características y extracción de artefactos cardíacos a partir de señales de EEG. El protocolo ayudará a estudiar la interacción cerebro-corazón a través de dos lentes (HEP y características), mejorando la reproducibilidad y la accesibilidad.
Resumen
La interacción entre el cerebro y los sistemas cardiovasculares está atrayendo cada vez más atención por su potencial para avanzar en nuestra comprensión de la fisiología humana y mejorar los resultados de salud. Sin embargo, el análisis multimodal de estas señales es un desafío debido a la falta de pautas, herramientas estadísticas y de procesamiento de señales estandarizadas, interfaces gráficas de usuario (GUI) y automatización para procesar grandes conjuntos de datos o aumentar la reproducibilidad. Existe un vacío adicional en los métodos estandarizados de extracción de características de EEG y variabilidad de la frecuencia cardíaca (HRV), lo que socava el diagnóstico clínico o la solidez de los modelos de aprendizaje automático (ML). En respuesta a estas limitaciones, presentamos la caja de herramientas BrainBeats. Implementado como un complemento de EEGLAB de código abierto, BrainBeats integra tres protocolos principales: 1) Potenciales evocados por latidos cardíacos (HEP) y oscilaciones (HEO) para evaluar la interacción cerebro-corazón bloqueada en el tiempo con una precisión de milisegundos; 2) Extracción de características de EEG y HRV para examinar asociaciones/diferencias entre varias métricas cerebrales y cardíacas o para construir modelos sólidos de ML basados en características; 3) Extracción automatizada de artefactos cardíacos a partir de señales de EEG para eliminar cualquier posible contaminación cardiovascular mientras se realiza el análisis de EEG. Proporcionamos un tutorial paso a paso para aplicar estos tres métodos a un conjunto de datos de código abierto que contiene señales simultáneas de EEG, ECG y PPG de 64 canales. Los usuarios pueden ajustar fácilmente los parámetros para adaptarlos a sus necesidades de investigación únicas utilizando la interfaz gráfica de usuario (GUI) o la línea de comandos. BrainBeats debería hacer que la investigación sobre la interacción entre el cerebro y el corazón sea más accesible y reproducible.
Introducción
Durante mucho tiempo, el enfoque reduccionista ha dominado la investigación científica en fisiología y cognición humanas. Este enfoque implicó diseccionar procesos corporales y mentales complejos en componentes más pequeños y manejables, lo que permitió a los investigadores centrarse en sistemas individuales de forma aislada. Esta estrategia surgió debido a los desafíos en el estudio de la naturaleza intrincada e interconectada del cuerpo y la mente humanos1. El reduccionismo ha sido fundamental para comprender los subsistemas individuales de forma aislada, como dilucidar el papel de los canales iónicos y los potenciales de acción para la comunicación neuronal2 o cardíaca3 . Sin embargo, sigue habiendo una brecha significativa en nuestra comprensión de cómo interactúan estos sistemas aislados en una escala espacial y temporal más amplia. El marco multimodal (integrador o ecológico) considera al cuerpo humano como un sistema multidimensional complejo, donde la mente no es vista como un producto del cerebro sino como una actividad del ser vivo, una actividad que integra el cerebro dentro de las funciones cotidianas del cuerpo humano4. Los enfoques multimodal y reduccionista no son excluyentes, al igual que no podemos estudiar una neurona sin todo el cerebro o todo el cerebro sin comprender las propiedades individuales de las neuronas. Juntos, allanan el camino para una comprensión más completa y sinérgica de la salud, la patología, la cognición, la psicología y la conciencia humanas. El presente método tiene como objetivo facilitar la investigación multimodal de la interacción entre el cerebro y el corazón al proporcionar análisis conjuntos de electroencefalografía (EEG) y señales cardiovasculares, a saber, electrocardiografía (ECG) y fotopletismografía (PPG). Esta caja de herramientas, implementada como un complemento de EEGLAB en MATLAB, aborda las limitaciones metodológicas existentes y se ha hecho de código abierto para facilitar la accesibilidad y la reproducibilidad en el área científica. Implementa las últimas directrices y recomendaciones en su diseño y parámetros predeterminados para animar a los usuarios a seguir las mejores prácticas conocidas. La caja de herramientas propuesta debería ser un recurso valioso para los investigadores y médicos interesados en 1) estudiar los potenciales evocados por los latidos del corazón, 2) extraer características de las señales de EEG y ECG/PPG, o 3) eliminar los artefactos cardíacos de las señales de EEG.
Investigación corazón-cerebro
La relación entre el corazón y el cerebro se ha estudiado históricamente a través de métodos de neuroimagen como la resonancia magnética funcional (fMRI) y la tomografía por emisión de positrones (PET). Utilizando estas herramientas, los investigadores destacaron algunas regiones del cerebro asociadas con el control cardiovascular (por ejemplo, la manipulación de la frecuencia cardíaca y la presión arterial5), mostraron la influencia de la frecuencia cardíaca en la señal BOLD6 o identificaron posibles vías cerebro-cuerpo que contribuyen a la enfermedad coronaria (es decir, la presión arterial provocada por el estrés7). Si bien estos estudios han avanzado significativamente nuestra comprensión de la compleja interacción entre el sistema nervioso central (SNC) y la función cardiovascular, estas técnicas de neuroimagen son costosas, tienen una disponibilidad limitada y se limitan a entornos de laboratorio controlados, lo que restringe su practicidad para aplicaciones en el mundo real y a gran escala.
Por el contrario, el EEG y el ECG/PPG son herramientas más asequibles y portátiles que ofrecen el potencial de estudiar las interacciones cerebro-corazón en entornos y poblaciones más diversas o durante períodos más largos, lo que brinda nuevas oportunidades. El ECG mide las señales eléctricas generadas por cada latido del corazón cuando el corazón se contrae y se relaja a través de electrodos colocados en la piel (generalmente en el pecho o los brazos)8. La PPG mide los cambios en el volumen sanguíneo en los tejidos microvasculares (es decir, el flujo sanguíneo y la frecuencia del pulso) utilizando una fuente de luz (por ejemplo, LED) y un fotodetector (comúnmente colocado en la punta del dedo, la muñeca o la frente), dependiendo de cómo la sangre absorbe más luz que el tejido circundante9. Ambos métodos proporcionan información valiosa sobre la función cardiovascular, pero sirven para diferentes propósitos y ofrecen distintos tipos de datos. Al igual que el ECG, el EEG registra los campos eléctricos generados por la actividad sincronizada de miles de neuronas corticales que se propagan a través de la matriz extracelular, los tejidos, el cráneo y el cuero cabelludo hasta llegar a los electrodos colocados enla superficie del cuero cabelludo. Como tal, el uso de EEG y ECG/PPG es muy prometedor para avanzar en nuestra comprensión de los procesos fisiológicos, cognitivos y emocionales que subyacen a las interacciones cerebro-corazón y sus implicaciones para la salud y el bienestar humanos. Por lo tanto, la captura de la interacción corazón-cerebro de las señales de EEG, ECG/PPG con la caja de herramientas BrainBeats puede ser particularmente útil para las siguientes áreas científicas: diagnóstico y pronóstico clínico, aprendizaje automático (ML) de big data, autocontrol del mundo real11 e imágenes móviles de cerebro/cuerpo (MoBI)12,13.
Dos enfoques para analizar conjuntamente las señales de EEG y ECG
Existen dos enfoques principales para estudiar las interacciones entre el EEG y las señales cardiovasculares:
Los potenciales evocados por latidos cardíacos (HEP) en el dominio del tiempo: potenciales relacionados con eventos (ERP), y las oscilaciones evocadas por latidos cardíacos (HEO) en el dominio tiempo-frecuencia: perturbaciones espectrales relacionadas con eventos (ERSP) y coherencia entre ensayos (ITC). Este enfoque examina cómo el cerebro procesa cada latido del corazón. Con una precisión de milisegundos (ms), este método requiere que ambas series temporales estén perfectamente sincronizadas y que los latidos del corazón se marquen en las señales de EEG. Este enfoque ha ganado interés en los últimos años 14,15,16,17,18,19.
Enfoque basado en características: este enfoque extrae las características del EEG y la variabilidad de la frecuencia cardíaca (VFC) de las señales continuas y examina las asociaciones entre ellas. Esto se ha hecho de forma independiente para el EEG (a menudo denominado EEG cuantitativo o qEEG20), ECG 21,22,23 y PPG 24,25,26. Este enfoque presenta aplicaciones prometedoras al capturar variables relacionadas con el estado y los rasgos. Obsérvese que, tanto para el EEG como para las señales cardiovasculares, cuanto más largo es el registro, más dominante es la variable rasgo 27,28,29. Por lo tanto, las aplicaciones dependen de los parámetros de grabación. Los análisis basados en características están ganando cada vez más interés, proporcionando nuevas métricas cuantitativas para pronosticar el desarrollo de trastornos mentales y neurológicos, tratamiento-respuesta o recaída 30,31,32,33,34,35. Este enfoque es especialmente convincente con grandes conjuntos de datos del mundo real (por ejemplo, clínica, monitoreo remoto), que se pueden obtener más fácilmente gracias a las recientes innovaciones en neurotecnología portátil11. Una aplicación menos explorada es la identificación de asociaciones entre características específicas del cerebro y el corazón, lo que pone de relieve las posibles dinámicas subyacentes del sistema nervioso central. La variabilidad de la frecuencia cardíaca (VFC) se puede calcular a partir de señales de ECG y PPG. Proporciona información sobre el sistema nervioso autónomo (SNA) midiendo las variaciones en los intervalos de tiempo entre los latidos del corazón (es decir, los intervalos entre normales y normales)27. El aumento de la actividad simpática (SNS) (p. ej., durante el estrés o el ejercicio) generalmente reduce la VFC, mientras que la actividad parasimpática (SNP) (p. ej., durante la relajación) la aumenta. Una frecuencia respiratoria más lenta generalmente aumenta la VFC debido a una mayor actividad del SNP, especialmente para registros cortos (<10 min)27. Las puntuaciones más altas de VFC generalmente sugieren un SNA más resistente y adaptable, mientras que una VFC más baja puede indicar estrés, fatiga o problemas de salud subyacentes. Los registros largos de VFC (es decir, al menos 24 h) proporcionan un pronóstico predictivo para diversas condiciones de salud, incluidas las enfermedades cardiovasculares, el estrés, la ansiedad y algunas afecciones neurológicas27. Medidas como la presión arterial, la frecuencia cardíaca o los niveles de colesterol proporcionan información sobre el estado del sistema cardiovascular. Por el contrario, la VFC añade un aspecto dinámico, mostrando cómo el corazón responde y se recupera del estrés.
Ventajas de BrainBeats sobre los métodos existentes
Si bien existen herramientas, como se revisa a continuación, para procesar las señales cardiovasculares y EEG de forma independiente entre sí, no se pueden analizar conjuntamente. Además, la mayoría de los medios disponibles para procesar señales cardiovasculares implican licencias costosas, no permiten el procesamiento automatizado (especialmente beneficioso para grandes conjuntos de datos), tienen algoritmos propietarios que impiden la transparencia y la reproducibilidad, o requieren habilidades de programación avanzadas al no proporcionar una interfaz gráfica de usuario (GUI)36. Hasta donde sabemos, cuatro toolboxes de MATLAB de código abierto soportan el análisis HEP/HEO con una GUI: la caja de herramientas del kit de ecg37, la pipeline BeMoBIL38, el plugin HEPLAB EEGLAB39 y la caja de herramientas CARE-rCortex40. Si bien HEPLAB, BeMoBIL y el kit de ECG facilitan el análisis de HEP al detectar los latidos del corazón y marcarlos en las señales de EEG, no proporcionan análisis estadístico o se limitan al dominio del tiempo (es decir, HEP). El complemento CARE-rCortex abordó estos problemas al admitir señales respiratorias y de ECG, análisis en el dominio del tiempo-frecuencia, estadísticas y métodos avanzados de normalización y corrección de línea de base adaptados al análisis HEP/HEO. Sin embargo, utiliza el método de Bonferroni para la corrección estadística del error de tipo 1 (es decir, falsos positivos), que es demasiado conservador y no fisiológicamente sólido para las aplicaciones de EEG, lo que conduce a un aumento de los errores de tipo II (es decir, falsos negativos)41. Además, la toolbox no ofrece acceso a la línea de comandos para la automatización. Por último, estudios recientes desaconsejan los métodos de corrección basal 42,43,44, ya que reducen la relación señal-ruido (SNR) y son estadísticamente innecesarios e indeseables.
Para abordar estas limitaciones, presentamos la caja de herramientas BrainBeats, actualmente implementada como un complemento de EEGLAB de código abierto en el entorno de MATLAB. Incorpora las siguientes ventajas respecto a los métodos anteriores:
1) Una interfaz gráfica de usuario fácil de usar y capacidades de línea de comandos (para programadores que buscan realizar un procesamiento automatizado). 2) Algoritmos, parámetros y directrices validados para el procesamiento de señales cardiovasculares, como la detección de picos R, la interpolación de artefactos RR y el cálculo de métricas de VFC (p. ej., implantación de directrices para ventanas, remuestreo, normalización, etc.27,45,46). Esto es importante porque Vest et al. demostraron cómo diferencias modestas en estos pasos de procesamiento pueden conducir a resultados divergentes, lo que contribuye a la falta de reproducibilidad y aplicabilidad clínica de las métricas de VFC46. 3) Algoritmos validados, parámetros predeterminados y pautas para el procesamiento de señales de EEG, incluido el filtrado y la creación de ventanas44,47, la rereferenciación48,49, la eliminación de canales y artefactos anormales50,51,52, la descomposición ICA optimizada y la clasificación de componentes independientes53,54,55,56. Los usuarios pueden ajustar todos los parámetros de preprocesamiento o incluso preprocesar sus datos de EEG con su método preferido antes de utilizar la caja de herramientas para satisfacer sus necesidades (por ejemplo, con el complemento clean_rawdata EEGLAB50,52, la tubería BeMoBIL38, la tubería PREP57, etc.). 4) Los potenciales evocados por los latidos del corazón (HEP, es decir, el dominio del tiempo) y las oscilaciones (HEO; perturbaciones espectrales relacionadas con eventos con métodos wavelet o FFT, y coherencia entre ensayos están disponibles a través del software estándar EEGLAB) a partir de señales de ECG. Las estadísticas paramétricas y no paramétricas con correcciones para errores de tipo 1 están disponibles a través del software estándar de EEGLAB. Los estadísticos no paramétricos incluyen estadísticos de permutación y correcciones espacio-temporales para comparaciones múltiples (p. ej., agrupamiento espacio-temporal o mejora de conglomerados sin umbral)58,59. Los usuarios pueden utilizar el complemento LIMO-EEG para implementar un modelado lineal jerárquico, que tiene en cuenta bien la varianza dentro y entre sujetos e implementa un enfoque univariado de masa libre de suposiciones con un control robusto de los errores de tipo I y II60,61. Los análisis estadísticos de datos HEP/HEO se pueden realizar en los dominios de canal y componentes independientes. 5) Análisis HEP/HEO y HRV a partir de señales PPG (por primera vez para HEP/HEO). 6) Admite la extracción conjunta de las características de EEG y HRV por primera vez. 7) La caja de herramientas proporciona varias visualizaciones de datos para inspeccionar señales en varios pasos de procesamiento necesarios y salidas a nivel de sujeto.
| Método | Detección de picos R a partir de ECG | Detección de ondas R de PPG | HEP/HEO | Características de EEG y HRV | Eliminar los artefactos cardíacos del EEG | Interfaz gráfica de usuario | Línea de comandos |
| kit de electrocardiograma | X | X | X | X | |||
| BeMoBIL | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CUIDADO-rCortex | X | X | X | X | |||
| Latidos cerebrales | X | X | X | X | X | X | X |
TABLA 1: Novedades aportadas por BrainBeats en relación con métodos similares preexistentes.
Información para ayudar a los lectores a decidir si el método es apropiado para ellos
Esta caja de herramientas es apropiada para cualquier investigador o clínico con datos de EEG y ECG/PPG. El plugin aún no admite la importación de señales de EEG y ECG/PPG desde archivos separados (aunque esta función estará disponible pronto). La caja de herramientas es apropiada para cualquier persona que desee realizar análisis HEP/HEO, extraer características de EEG y/o HRV con métodos estandarizados, o simplemente eliminar artefactos cardíacos de las señales de EEG. Consulte la Figura 1 para ver un diagrama de bloques que resume el flujo general y los métodos de BrainBeats.
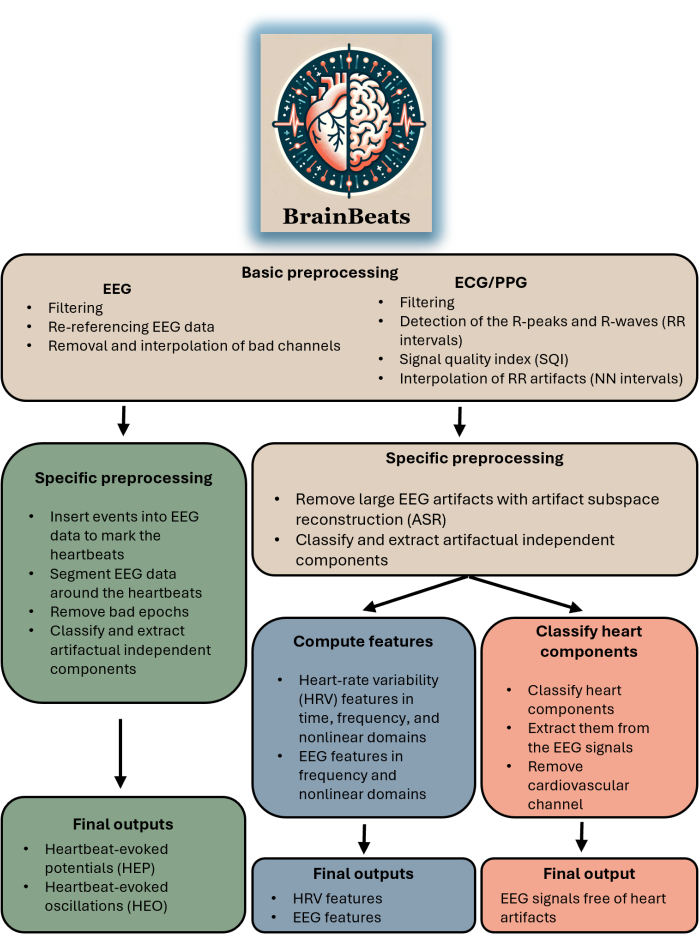
FIGURA 1. Diagrama de bloques que resume la arquitectura y el flujo general de BrainBeats. Las operaciones que son comunes a los tres métodos son marrones. Las operaciones específicas de los potenciales evocados por latidos cardíacos (HEP) y las oscilaciones (HEO) son verdes. Las operaciones específicas de la extracción de las características del EEG y la VFC son azules. Las operaciones específicas para eliminar artefactos cardíacos de las señales de EEG son rojas. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Se obtuvo el consentimiento informado de cada participante y el comité de ética de la Universidad Federal de los Urales aprobó el protocolo experimental.
1. Requisitos de BrainBeats
- Instale MATLAB y EEGLAB en el equipo. EEGLAB se puede descargar en https://github.com/sccn/eeglab y descomprimir (o clonar para usuarios de Git) en cualquier lugar de la computadora. Consulte la página de GitHub para obtener más detalles sobre la instalación.
- Agregue la ruta a la carpeta EEGLAB en el panel de inicio de MATLAB haciendo clic en el botón Set Path . Seleccione la carpeta eeglab con el archivo descomprimido y haga clic en Guardar > cerrar.
- Inicie EEGLAB escribiendo eeglab en la ventana de comandos de MATLAB.
- Instala el plugin BrainBeats haciendo clic en Archivo > Administrar extensiones de EEGLAB. Escriba brainbeats en la barra de búsqueda, seleccione el complemento BrainBeats en la lista y haga clic en Instalar/Actualizar.
- Cargue el conjunto de datos de muestra en EEGLAB. Haga clic en Archivo > Cargar datos existentes. Vaya a la carpeta EEGLAB, vaya a la carpeta de complementos, vaya a la carpeta BrainBeats y abra la carpeta sample_data. Seleccione el archivo dataset.set.
NOTA: Este conjunto de datos corresponde al sub-032 (estado de reposo con los ojos abiertos) de un conjunto de datos multitema de código abierto62,63. Se seleccionó porque contiene EEG (64 canales), ECG (un canal) y PPG (un canal) simultáneos, a una frecuencia de muestreo de 1000 Hz. Las señales de ECG y PPG se recogieron utilizando las entradas auxiliares del sistema actiCHamp. Los datos de EEG se registraron con electrodos activos colocados según el sistema 10-20, con FCz como referencia en línea y Fpz como electrodo de tierra, y con la impedancia mantenida por debajo de 25 kOm. El ECG se registró utilizando un electrodo activo colocado en la muñeca derecha, el electrodo de referencia en la muñeca izquierda y el suelo en el antebrazo interno izquierdo a 3 cm distalmente del codo. La PPG se registró desde el dedo índice izquierdo. Los datos de EEG, ECG y PPG están sincronizados en el tiempo, ya que se registraron simultáneamente con el mismo amplificador. Véanse las referencias62,63 para más detalles. - Marque la casilla de verificación Guardar salidas para guardar todo en el archivo .set correspondiente en la misma ubicación que el archivo original que se cargó en EEGLAB.
NOTA: Para este tutorial, fusionamos los datos de EEG, ECG y PPG en un conjunto de datos de EEGLAB, cargamos las coordenadas del electrodo del método de elementos de contorno (BEM) 3D y redujimos la resolución de la señal a 250 Hz para reducir el peso del archivo (para descarga del usuario) y acelerar el tiempo de cómputo. Debido a que este conjunto de datos no tenía un canal de EEG anormal, modificamos artificialmente el canal TP9 para ilustrar el algoritmo de detección y eliminación de canales defectuosos de BrainBeats. Del mismo modo, simulamos artificialmente un gran artefacto de EEG al principio del archivo y un artefacto de alta frecuencia similar a un músculo en los canales temporales de 3 a 6 s para ilustrar la función de eliminación de artefactos de BrainBeats.
2. Potenciales evocados por latidos cardíacos (HEP) y oscilaciones (HEO)
- Abra la primera interfaz de usuario general (GUI) de BrainBeats para seleccionar los parámetros principales. En EEGLAB, haga clic en Herramientas > BrainBeats >1er nivel (nivel de sujeto). Seleccione Potenciales evocados por latidos HEP como el análisis a ejecutar, ECG como el tipo de datos cardíacos y haga clic en el botón para mostrar la lista de canales para seleccionar el canal de ECG etiquetado como ECG (o escríbalo directamente en el cuadro de texto). Mantenga seleccionadas las opciones Visualizar salidas (recomendado) y Guardar salidas y haga clic en Aceptar.
- Aparece una segunda ventana de GUI basada en las opciones anteriores (es decir, análisis HEP con datos de ECG). Seleccione los parámetros de preprocesamiento para las señales de EEG y ECG. Desactiva el preprocesamiento desmarcando las casillas Preprocesar ECG y Preprocesar EEG para ver si los datos ya se han preprocesado antes de iniciar BrainBeats. Cambie el ruido de la línea eléctrica a 50 Hz en la sección de EEG previo al procesamiento , ya que estos datos de EEG se registraron en Rusia. Haga clic en Aceptar para iniciar.
- Aparecerá un mensaje de advertencia, solicitando confirmación para eliminar el canal PPG que se ha detectado. Esto se debe a que la toolbox no está diseñada para analizar ECG y PPG simultáneamente (u otros canales auxiliares) y mantenerla en el conjunto de datos dará lugar a errores graves (por ejemplo, eliminación de artefactos, mala descomposición de ICA, etc.). Haga clic en Sí. BrainBeats comienza a realizar algunas comprobaciones, establece algunos parámetros predeterminados y separa el ECG de los datos del EEG para preprocesar la señal del ECG y calcular los intervalos RR.
- Las series temporales de ECG y RR se preprocesan utilizando algoritmos validados de la caja de herramientas Physionet Cardiovascular Signal 46,64 adaptados para adaptarse al formato de datos de BrainBeats, aumentar la claridad, el ajuste de parámetros y el tiempo de cálculo (consulte las referencias para la validación de los algoritmos). El plugin emite los intervalos RR, las marcas de tiempo, la señal de ECG filtrada, los índices de picos R y la frecuencia cardíaca (FC). Ajuste estos parámetros a través de la GUI o la línea de comandos.
NOTA: La señal de ECG se filtra de paso de banda utilizando un filtro personalizado y validado (1-30 Hz) y escanea la señal para identificar el complejo QRS y los picos R utilizando el método Pan-Tompkins (P&T)65, implementando algunas operaciones de procesamiento de señales que incluyen diferenciación, cuadratura, integración y suavizado para obtener el mejor rendimiento. El umbral de energía de P&T se estima en función de la frecuencia de muestreo y los valores de ECG suavizados para evitar interrupciones por grandes protuberancias. Si la variabilidad del intervalo RR supera 1,5 veces la mediana, busca los picos perdidos. La señal media del pico R se calcula en segmentos de 30 s, y los puntos de pico se refinan a través de una verificación del período refractario, gestionando las condiciones de línea plana y asegurando una detección consistente. - A continuación, BrainBeats identifica intervalos RR anormales o picos dentro de los intervalos RR mediante una búsqueda hacia adelante y hacia atrás y umbrales fisiológicos. El índice de calidad de la señal (SQI) se calcula46; compruébelo si el sistema muestra avisos en los que más del 20% de la serie temporal RR contiene artefactos RR (fuera de los límites fisiológicos o con un ICS inferior a 0,9). Un gráfico muestra la señal de ECG filtrada, los picos R identificados, los intervalos NN y los artefactos interpolados (consulte la Figura 2).
NOTA: Los artefactos RR se interpolan de forma predeterminada utilizando el método cúbico por partes que conserva la forma para obtener los intervalos de normal a normal (NN), pero se eliminan (no se recomienda) o se utiliza otro método de interpolación (lineal, cúbico, vecino más cercano, vecino anterior/siguiente, spline, convolución cúbica o cúbico Akima modificado) si es necesario. Cuando hay varios canales de ECG, se estiman los intervalos RR para cada uno y se selecciona el canal con el menor número de artefactos RR para los pasos siguientes. - Desplácese por las ventanas ampliadas de 30 segundos de los picos R para una inspección más cercana presionando las flechas derecha/izquierda. Si los datos contienen varios canales de ECG/PPG, utilice el canal con el mejor índice de calidad de señal para los intervalos RR. BrainBeats no admite señales de ECG y PPG simultáneamente en este momento. Para fines del tutorial, elegimos un conjunto de datos de muestra que contiene ambos tipos de datos.
- Una vez hecho con la señal de ECG, el paso de banda de BrainBeats filtra los datos de EEG a 1-40 Hz utilizando un filtro FIR de fase mínima causal no lineal de forma predeterminada para reducir la actividad de frotis entre los períodos previos y posteriores a los latidos del corazón, preservar la causalidad y evitar retrasos grupales no deseados44. Esto es particularmente importante para los usuarios que examinan el período previo a los latidos del corazón. Si el filtro de paso bajo está configurado en un valor por encima de la frecuencia de la línea de alimentación (por ejemplo, paso bajo de 80 Hz con frecuencia de línea de alimentación a 50 Hz), use un filtro de muesca afilada para eliminar el artefacto de ruido de línea. A continuación, los datos de EEG se vuelven a referenciar hasta el infinito utilizando el algoritmo REST (el más adecuado para el análisis HEP49) a menos que se detecten menos de 30 canales (en cuyo caso no se pueden volver a referenciar de forma fiable, y se genera una advertencia para informar a los usuarios).
- Luego, BrainBeats detecta, elimina e interpola los canales de EEG anormales (Figura 3). Compruebe que los parámetros predeterminados son líneas planas superiores a 5 s (algoritmo de clean_flatlines), una desviación estándar máxima de ruido de alta frecuencia de 10, una longitud de ventana de 5 s (para capturar mejor los artefactos de frecuencia lenta52), una correlación mínima entre canales vecinos de 0,65 y una porción máxima tolerada del 33% (algoritmo de clean_channels).
NOTA: El número de muestras RANSAC se establece en 500 de forma predeterminada para aumentar la convergencia y la replicabilidad del rechazo de canal incorrecto (aunque aumenta el tiempo de cálculo). - A continuación, inserte los picos R como marcadores de eventos en los datos del EEG para marcar cada latido del corazón y segmente los datos en torno a estos marcadores sin eliminar la línea de base (según las pautas43,66; Figura 4). Dado que los intervalos NN tienen diferentes longitudes y el EEG debe segmentarse a una longitud constante, estime el límite mínimo del tamaño de la época después de los eventos de pico R utilizandoel percentil 5 de los datos del intervalo de interlatación (IBI) (es decir, el valor por debajo del cual caen el 5% de los IBI más cortos, que se muestra como una línea roja discontinua en un histograma; consulte la Figura 5).
NOTA: Este valordel percentil 5 es un buen compromiso para preservar tantas épocas como sea posible y garantizar que no sean demasiado cortas, ya que el período de interés para el análisis HEP/HEO es de 200-600 ms después del latido49,67. - Segmente los datos de EEG desde -300 ms antes de los picos R hasta el valordel percentil 5 después del pico R, con el pico R en el tiempo 0. Rechace las épocas inferiores a 550 ms o que contengan más de un pico R (lo que sesgaría el ERP/ERSP), según las directrices49,67. Las épocas que contienen grandes artefactos de EEG se detectan utilizando métricas de raíz cuadrada media (RMS) y relación señal-ruido (SNR) y la función isoutlier de MATLAB (Figura 6). Se eliminan las épocas artifactuales.
- Realice la separación de fuentes ciegas utilizando el algoritmo predeterminado de Infomax, teniendo en cuenta el rango de datos para obtener el mejor rendimiento53. Si lo desea, elija el algoritmo de análisis de componentes independientes precondicionados (PICARD) para el cálculo rápido54,54 eligiendo la opción Picard (rápido) o el algoritmo Infomax modificado eligiendo la opción Infomax modificado (largo pero replicable) para el método ICA de campo (aunque esto implica tiempos de cálculo mucho más largos). Utilice el complemento ICLabel56 para clasificar automáticamente los circuitos integrados para extraer artefactos no cerebrales (los componentes oculares se eliminan con un 90% de confianza, mientras que los músculos, el ruido de línea y el ruido de canal se eliminan con un 99% de confianza; Figura 7).
- Mantenga la opción Visualizar salidas seleccionada en la primera ventana de la GUI para visualizar la serie temporal final de EEG, el HEP promedio general (Figura 8 arriba), HEP para cada latido (Figura 8 abajo) y las oscilaciones evocadas por latidos cardíacos promedio (HEO; Figura 9). Examinar los HEO en términos de perturbación espectral relacionada con eventos (ERSP, es decir, cambios en la potencia del EEG a través de los latidos del corazón; Figura 9 arriba) y la coherencia de fase entre ensayos (ITC, es decir, la consistencia del ángulo de fase a través de los latidos del corazón; Figura 9 abajo).
NOTA: El ERSP se calcula utilizando una ondícula predeterminada de 3 ciclos (con una ventana cónica de Hanning aplicada, relación de almohadilla de 2) y con el número de ciclos en las ondículas utilizadas para frecuencias más altas que se expanden lentamente hasta el 20% del número de ciclos en la ventana FFT equivalente en su frecuencia más alta (1 menos 0,8). Esto controla las formas de las ventanas individuales medidas por la función y sus formas en los paneles de tiempo/frecuencia resultantes. Se elimina una línea de base arbitraria con fines ilustrativos y se calcula el ERSP para las frecuencias 7-25 Hz para capturar el efecto HEO típico descrito en la literatura, es decir, 300-450 ms después del latido del corazón en la banda alfa (8-13 Hz) sobre electrodos frontocentrales17,67. Las frecuencias más bajas no se pueden estimar debido al corto tamaño de la época definido por los intervalos entre tiempos. Se aplican estadísticos no paramétricos (permutación) para visualizar el HEO para un valor p de 0,05, corregido por la tasa de descubrimiento falso (FDR, es decir, error de tipo 1 o error por familia). Estos gráficos se generan con fines tutoriales o análisis de ensayos individuales. - Se generan gráficos de preprocesamiento para visualizar los diferentes pasos. Para desactivarlo, desmarque la casilla Visualizar preprocesamientos. Los datos finales del EEG (limpios y segmentados alrededor de los picos R) no incluyen los datos del ECG, ya que sesgarían el análisis ERP/ERSP. Para conservar el canal cardíaco en la salida final, marque la casilla Mantener canal cardíaco.
NOTA: Haga una pausa aquí si es necesario antes de procesar el siguiente archivo (siguiente condición o participante). - BrainBeats es compatible con la función de historial de EEGLAB. Al final de todas las operaciones, escriba eegh en la ventana de comandos de MATLAB para imprimir la línea de comandos y repetir todos los pasos anteriores a través de una sola línea de comandos, con los parámetros que se seleccionaron manualmente en la GUI, lo que permite una fácil automatización. Encuentre las salidas de preprocesamiento (por ejemplo, el índice de calidad de la señal de las series temporales cardiovasculares, los intervalos NN, los canales, segmentos y componentes del EEG eliminados, etc.) en la estructura del EEGLAB: EEG.brainbeats.preprocessings. Todos los parámetros también se exportan en EEG.brainbeats.parameters.
- Para usuarios avanzados, realice todos los pasos anteriores con parámetros predeterminados con las siguientes líneas de comando (consulte el script del tutorial en el repositorio de BrainBeats para obtener más opciones):
eeglab; cerrar; % Iniciar EEGLAB sin la GUI
main_path = partes de archivo(que('eegplugin_BrainBeats.m')); CD(main_path);
EEG = pop_loadset('nombre de archivo','conjunto de datos.conjunto',
'filepath',fullfile(main_path, 'sample_data')); %Cargar el conjunto de datos de ejemplo
EEG = brainbeats_process(EEG,'análisis',
'hep','heart_signal','ECG', 'heart_channels',
{'ECG'},'clean_eeg',verdadero); % Inicie BrainBeats1er nivel para procesar el archivo para el análisis HEP con parámetros predeterminados - Los pasos anteriores realizaron HEP/HEO a partir de la señal de ECG. Utilice los siguientes pasos para la señal PPG.
- En los pasos siguientes, realice las mismas operaciones pero utilizando una señal PPG. Cargue el mismo conjunto de datos nuevamente (consulte el paso 1.5) ya que las operaciones anteriores lo sobrescribieron y vuelva a abrir la primera GUI de BrainBeats para seleccionar los parámetros principales. Haga clic en Herramientas > BrainBeats > 1er nivel (nivel de materia). Seleccione Potenciales evocados por latidos (HEP) como el análisis a ejecutar, PPG como el tipo de datos cardíacos y haga clic en el botón para mostrar la lista de canales para seleccionar el canal PPG. Mantenga seleccionadas las opciones Visualizar salidas (recomendado) y Guardar salidas y haga clic en Aceptar.
- La segunda ventana de la GUI aparece de manera similar al paso 2.2 . La única diferencia es que ahora podemos ajustar el parámetro para procesar la señal PPG en lugar de la señal ECG. Haga clic en Aceptar para ejecutar con los parámetros predeterminados.
- Aparece un mensaje de advertencia en el que se solicita confirmación para eliminar el canal de ECG adicional que se ha detectado. De nuevo, esto es lo que se espera. Haga clic en Sí. De forma predeterminada, la caja de herramientas preprocesará la señal PPG, detectará las ondas de pulso para obtener los intervalos RR, identificará los artefactos RR, si los hay, y los interpolará (Figura 10). Pasos 2.7. a 2.12. y se generan los mismos gráficos y salidas, pero basados en las ondas R detectadas a partir de la señal PPG (ver Figura 11 y Figura 12).
NOTA: Las ondas R se detectan utilizando la pendiente de la señal dentro de una ventana especificada. A continuación, se marcan los pulsos potenciales cuando la pendiente supera un umbral dinámico, que se ajusta en función del historial de detección y las características de la señal. A continuación, el algoritmo busca dentro de un período de cierre para determinar la pendiente máxima y, posteriormente, se determina el inicio de la onda de pulso mediante el umbral. Los picos de la onda R se identifican como los valles cercanos al inicio y se registran sus ubicaciones. El algoritmo recorre toda la señal, ajustando continuamente los umbrales de detección e identificando los picos de la onda R, que luego se utilizan para calcular los intervalos RR. - Para usuarios avanzados, realice todos los pasos anteriores con parámetros predeterminados con las siguientes líneas de comando (consulte el script del tutorial en el repositorio de BrainBeats para obtener más opciones):
eeglab; cerrar; % Iniciar EEGLAB sin la GUI
main_path = partes de archivo(que('eegplugin_
BrainBeats.m')); CD(main_path);
EEG = pop_loadset('nombre de archivo','conjunto de datos.conjunto',
'filepath',fullfile(main_path, 'sample_data')); %Cargar el conjunto de datos de ejemplo
EEG = brainbeats_process(EEG,'análisis',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',verdadero); % Inicie BrainBeats1er nivel para procesar el archivo para el análisis HEP con parámetros predeterminados
3. Extracción de las características del EEG y la VFC
- Cargue de nuevo el mismo conjunto de datos (consulte el paso 1.5; Haga clic en Archivo > Cargar conjunto de datos existente > Seleccionar dataset.set) ya que se sobrescribió por las operaciones anteriores y vuelva a abrir la GUI principal para seleccionar los parámetros principales (paso 2.1; Haga clic en Herramientas > BrainBeats >1er nivel). Seleccione Extraer características de EEG y HRV para el tipo de análisis, ECG para el tipo de señal cardíaca y seleccione ECG en la lista de etiquetas de electrodos. Haga clic en Aceptar.
- Aparece la segunda ventana de la GUI como en el paso 2.2, pero con diferentes parámetros para el preprocesamiento de EEG y la extracción de las características de HRV y EEG. Establezca estos parámetros como se describe a continuación. Haga clic en Aceptar para iniciar con los parámetros predeterminados.
- En la sección HRV, haga clic en el botón opciones frecuentes para seleccionar el método para calcular la potencia de HRV (establecido por defecto en el periodograma normalizado de Lomb-Scargle), la superposición de ventanas (por defecto establecido en 25%) y para realizar una normalización de segundo nivel (no establecido por defecto; véase la nota a continuación para más detalles).
- En la sección de características del EEG, haga clic en el botón opciones de frecuencia para ajustar algunos parámetros, como el rango de frecuencia general en el que calcular la densidad espectral de potencia (PSD; por defecto = 1-40 Hz), las unidades (decibelios, μV2/Hz, o normalizado por la potencia total), el tipo de ventana (por defecto = hamming), la superposición de la ventana (por defecto = 50%), la longitud de la ventana (por defecto = 2 s), y los tipos de límites de frecuencia para cada banda.
NOTA: La potencia de la VFC se calcula de forma predeterminada utilizando el periodograma normalizado de Lomb-Scargle, que no requiere remuestreo (por lo tanto, conserva mejor la información original) y se ocupa mejor de los datos muestreados de manera no uniforme, los datos faltantes y el ruido (típico de los intervalos NN)68. La versión normalizada escala la potencia según la varianza de la señal, lo que proporciona resultados que son menos sensibles a los niveles de ruido variables, más centrados en la fuerza relativa de los componentes periódicos y más comparables entre diferentes grabaciones o sujetos. Otros métodos disponibles incluyen el periodograma de Lomb-Scargle no normalizado, el método de Welch y la transformada rápida de Fourier (FFT). El remuestreo se realiza automáticamente para los métodos Welch y FFT para crear las series temporales necesarias muestreadas regularmente. Se puede aplicar una normalización de segundo nivel dividiendo la potencia de cada banda de frecuencia por la potencia total, proporcionando una medida más intuitiva de la contribución relativa de cada componente de frecuencia a la potencia total. Está desactivado de forma predeterminada, ya que solo tiene sentido cuando las cuatro bandas están disponibles, lo que requiere al menos 24 h de señal. Estos algoritmos son una adaptación de la caja de herramientas de procesamiento de señales cardiovasculares de Physionet46.
- Aparece el mismo mensaje de advertencia, solicitando confirmación para eliminar el canal PPG adicional que se ha detectado. De nuevo, esto es lo que se espera. Haga clic en Sí. BrainBeats comenzará a preprocesar los datos de ECG y a extraer los intervalos NN de forma idéntica, como en el paso 2.4. A continuación, extrae las características de la variabilidad de la frecuencia cardíaca (HRV) de los intervalos NN en los dominios de tiempo (SDNN, RMSSD, pNN50), frecuencia (ULF, VLF, LF, HF, relación lf:hf, potencia total) y no lineales (Poincaré, promediación de señales rectificadas en fase, entropía difusa y dimensión fractal).
NOTA: BrainBeats verifica automáticamente la longitud del archivo para asegurarse de que se cumplan los requisitos mínimos (por ejemplo, la alimentación ULF-HRV requiere 24 horas de datos), envía mensajes de advertencia si no es así y no exporta estas funciones para evitar estimaciones poco confiables. BrainBeats sigue las pautas y recomendaciones para estimar las métricas de VFC27,45. - BrainBeats preprocesa los datos de EEG como en el paso 2.7 . (ver Figura 3). Los artefactos grandes se detectan automáticamente en los datos continuos utilizando el algoritmo de reconstrucción de subespacio de artefactos (ASR)50,52 (criterio SD predeterminado establecido en 30 y utilizando el 80% de la RAM disponible para aumentar la velocidad). Estos grandes artefactos se eliminan de los datos del EEG (véase la figura 13). Para ajustar estos parámetros en la GUI, seleccione los campos Umbral para rechazar segmentos defectuosos con ASR y RAM disponible para usar para ASR.
NOTA: No es necesario sincronizar el tiempo del EEG y las series temporales cardiovasculares para el modo de características, ya que las características se estiman en cada señal por separado. Por lo tanto, los artefactos de EEG se pueden eliminar directamente de los datos de EEG (en rojo, Figura 13), a diferencia del modo HEP, donde las épocas que contienen artefactos se rechazaron para ambas series temporales porque la sincronización del tiempo con la precisión de ms es esencial para ese método. - Realice la ACI utilizando los mismos algoritmos y parámetros que para la HEP (consulte el paso 2.11.), excepto que esta vez los componentes del corazón se eliminan si se detectan con un 99% de confianza (se conservaron para la HEP/HEO ya que no queremos eliminar las señales relevantes relacionadas con el corazón).
- La casilla Dominio de la frecuencia está marcada en el paso 3.2., por lo que BrainBeats extrae las siguientes características del dominio de la frecuencia: la densidad espectral de potencia media (PSD) para las bandas de frecuencia delta (1-3 Hz), theta (3-7 Hz), alfa (8-13 Hz), beta (13-30 Hz) y gamma (30+ Hz), la frecuencia alfa individual (IAF) y la asimetría alfa en todos los pares de electrodos (simétricos) disponibles. Compruebe estas características.
NOTA: La PSD convertida a decibelios (dB) facilita la comparación de los resultados entre grabaciones y sujetos. Los límites de frecuencia se pueden establecer en los límites convencionales (por ejemplo, 8-13 Hz predefinidos para la banda alfa) o en los límites individualizados, que se detectan a partir de la distribución de la densidad espectral de potencia para tener en cuenta las diferencias interindividuales69 (por ejemplo, 7,3-12,6 Hz para la banda alfa). El algoritmo fue diseñado para la banda alfa y no funciona tan bien para otras bandas, especialmente cuando los picos no están presentes en la distribución espectral de potencia. La frecuencia alfa individual (IAF) se estima utilizando el centro de gravedad alfa para tratar mejor los picos divididos o los picos ambiguos69. La asimetría alfa se calcula siguiendo pautas (ventana de Hamming de 2 s con 50% de superposición, el logaritmo de la potencia alfa del canal izquierdo menos el logaritmo de la potencia alfa del canal derecho)47. Por lo tanto, los valores positivos indican una mayor potencia alfa a la izquierda que a la derecha y viceversa. La asimetría alfa se puede normalizar dividiendo la potencia alfa de cada electrodo por la potencia alfa sumada entre todos los electrodos47. Los pares simétricos se obtienen utilizando distancias theta, lo que requiere que los datos del EEG contengan coordenadas de electrodos. - La casilla Dominio no lineal está marcada en el paso 3.2., BrainBeats extrae la entropía difusa y la dimensión fractal para cada canal de EEG.
NOTA: Se cree que las características de dominio no lineal capturan dinámicas complejas no lineales del cerebro que se pasan por alto por las medidas espectrales y son particularmente prometedoras para investigar las interacciones entre varios sistemas corporales 70,71,72. La entropía difusa es más fiable y robusta que sus alternativas (entropía de muestra y aproximada), pero requiere tiempos de cálculo más largos (especialmente con series temporales de EEG largas con altas tasas de muestreo). Para solucionar este problema, cuando las señales de EEG duran más de 2 minutos con una frecuencia de muestreo superior a 100 Hz, se reducen automáticamente (o se diezman cuando el factor no es un número entero) a 90 Hz (es decir, correspondiente a una frecuencia de Nyquist de 45 Hz, para que coincida con el filtro de paso bajo predeterminado y evite los artefactos de ruido de línea tanto como sea posible). Además, la computación paralela está activada de forma predeterminada cuando se estiman las características del EEG, lo que reduce el tiempo de cálculo, especialmente cuando hay muchos canales de EEG disponibles. - Seleccione Visualizar salidas en la primera GUI (consulte el paso 3.1) para generar una gráfica que muestre la densidad espectral de potencia (PSD) para los datos de HRV y EEG (Figura 14) junto con topografías del cuero cabelludo que muestren algunas características del EEG (Figura 15).
NOTA: También puede encontrar algunas salidas de preprocesamiento en EEG.brainbeats.preprocessing y todos los parámetros utilizados en EEG.brainbeats.parameters. Alentamos a los usuarios a informar de estos resultados en publicaciones científicas para aumentar la replicabilidad de los hallazgos. - Mantenga marcada la casilla Guardar salidas en la primera ventana de la GUI para guardar todas las características exportadas al archivo .set de EEGLAB en EEG.brainbeats.features y guardarlas en un archivo .mat en la misma carpeta donde se cargó el conjunto de datos.
- BrainBeats es compatible con la función de historial de EEGLAB. Al final de todas las operaciones, escriba eegh en la ventana de comandos de MATLAB para imprimir la línea de comandos que le permitirá repetir todos los pasos anteriores a través de una sola línea de comandos, con los parámetros que se seleccionaron manualmente en la GUI, lo que permite una fácil automatización y replicación de las operaciones.
- Para usuarios avanzados, realice todos los pasos anteriores con el siguiente comando:
eeglab; cerrar; % Iniciar EEGLAB sin la GUI
main_path = partes de archivo(que('eegplugin_BrainBeats.m')); CD(main_path);
EEG = pop_loadset('nombre de archivo','conjunto de datos.conjunto',
'filepath',fullfile(main_path, 'sample_data')); % Cargar el conjunto de datos de muestra
EEG = brainbeats_process(EEG'análisis',
'características','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - Los pasos anteriores extrajeron las características de la VFC de la señal de ECG. Utilice los siguientes pasos para extraer las características de HRV de la señal PPG (las características de EEG son las mismas).
- Cargue de nuevo el mismo conjunto de datos (paso 1.5.) ya que fue sobrescrito por las operaciones y vuelva a abrir la GUI principal (paso 2.5.). Seleccione Extraer características de EEG y HRV para el análisis y seleccione PPG para el tipo de señal cardíaca y PPG para el nombre del canal. Haga clic en Aceptar.
- La2ª ventana de la GUI muestra ahora los parámetros para el preprocesamiento de PPG y para la extracción de las características HRV y EEG. Haga clic en Aceptar para ejecutar con los parámetros predeterminados. Los parámetros se describen en el paso 2.17.
- Aparecerá un mensaje de advertencia, solicitando confirmación para eliminar el canal de ECG detectado. Esto es de esperar, ya que la toolbox no está diseñada para analizar ECG y PPG simultáneamente (u otros canales auxiliares) y mantenerla en el conjunto de datos conducirá a errores graves (por ejemplo, eliminación de artefactos, descomposición deficiente de ICA, etc.). Haga clic en Sí.
- BrainBeats preprocesa la señal PPG y estima los intervalos NN como en el paso 2.5. Verifique las características HRV extraídas de los intervalos NN, lo mismo que en el paso 3.2 . excepto que los intervalos NN se han obtenido ahora de la señal PPG. Las señales de EEG se preprocesan como en el paso 3.2. BrainBeats traza el PSD (Figura 16). La única diferencia aquí es la PSD estimada a partir de los intervalos NN obtenidos de la PPG en comparación con el ECG.
- Para usuarios avanzados, realice todos los pasos anteriores con el siguiente comando:
eeglab; cerrar; % Iniciar EEGLAB sin la GUI
main_path = partes de archivo(que('eegplugin_BrainBeats.m')); CD(main_path);
EEG = pop_loadset('nombre de archivo','conjunto de datos.conjunto','ruta de archivo',
archivo completo(main_path, 'sample_data')); %Cargar el conjunto de datos de ejemplo
EEG = brainbeats_process(EEG,'análisis',
'características','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. Extraiga los artefactos cardíacos de las señales de EEG.
- Cargue el conjunto de datos de muestra (consulte el paso 1.5.).
- Abra la ventana principal de la GUI haciendo clic en Herramientas > BrainBeats >1er nivel (nivel de sujeto) y seleccione Extraer artefactos cardíacos de señales de EEG para el tipo de análisis, ECG para el tipo de señal cardíaca y seleccione ECG en la lista de etiquetas de electrodos. Haga clic en Aceptar.
- La2ª ventana de la GUI muestra los parámetros de preprocesamiento. Establezca el ruido de la línea eléctrica en 50 Hz (Europa), edite el nivel de confianza si es necesario, marque la casilla Modo de refuerzo (beta) y haga clic en Aceptar para ejecutar con los parámetros predeterminados, ya que las señales de EEG del conjunto de datos de muestra no se preprocesan.
NOTA: el nivel de confianza para detectar componentes cardíacos se establece en 80% de forma predeterminada, que puede ser demasiado bajo o demasiado alto para algunos conjuntos de datos. El aumento de este valor aumentará las posibilidades de detectar componentes cardíacos, pero disminuirá la confiabilidad de esa detección. El modo Boost (beta) es opcional y tiene como objetivo mejorar el rendimiento de la clasificación mediante la difuminación de la señal cardíaca en las señales de EEG. - Aparecerá un mensaje de advertencia, solicitando confirmación para eliminar el canal PPG adicional que se ha detectado. Esto es de esperar, ya que la toolbox no está diseñada para analizar ECG y PPG simultáneamente (u otros canales auxiliares) y mantenerla en el conjunto de datos conducirá a errores graves (por ejemplo, eliminación de artefactos, descomposición deficiente de ICA, etc.). Haga clic en Sí.
- La señal de ECG se filtra por paso de banda para eliminar las derivas de frecuencia lentas por debajo de 1 Hz y el ruido de alta frecuencia por encima de 20 Hz (con un filtro FIR de fase cero no causal). Preprocese las señales de EEG como en el paso 3.4.
- Realice análisis de componentes independientes (ICA) utilizando el algoritmo precondicionado ICA para datos reales (PICARD). Para cambiar esta opción, elija el algoritmo Infomax estándar o el algoritmo Infomax modificado para la replicación desde la GUI en el paso 4.3 . A continuación, clasifique los componentes independientes automáticamente con ICLabel. Si un componente se clasifica como componente cardíaco con un 80% de confianza, se detecta y extrae automáticamente de los datos del EEG de forma predeterminada.
- Mantenga la casilla Visualizar salidas en la primera GUI principal (paso 4.2.) para visualizar la topografía del cuero cabelludo del componente extraído (Figura 17 a la izquierda) y la serie temporal final del EEG (en azul, Figura 17 a la derecha) después de extraer el componente cardíaco (en rojo, Figura 17 a la derecha).
NOTA: el canal de ECG se conserva para la visualización para confirmar la extracción de los componentes relacionados con el ECG, pero se elimina después de este paso ya que ya no contiene ninguna información relevante. - Para usuarios avanzados, realice estos pasos mediante las siguientes líneas de comandos:
eeglab; cerrar; % Iniciar EEGLAB sin la GUI
main_path = partes de archivo(que('eegplugin_BrainBeats.m')); CD(main_path);
EEG = pop_loadset('nombre de archivo','conjunto de datos.conjunto','ruta de archivo',
archivo completo(main_path, 'sample_data'));
EEG = brainbeats_process(EEG,'análisis','rm_heart',
'heart_signal','ECG', ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'impulso',verdadero);
Resultados
En primer lugar, se utilizó el plugin BrainBeats para preprocesar los datos de EEG y ECG, identificar y eliminar artefactos, y analizar los potenciales evocados por los latidos del corazón (HEP) y las oscilaciones (HEO). BrainBeats detectó con éxito los intervalos RR de la señal de ECG y algunos artefactos RR (Figura 2). BrainBeats también informó en la ventana de comandos que 11/305 (3,61%) de los latidos del corazón se marcaron como artefactos e interpolaron. El índice medio de ca...
Discusión
Pasos críticos en el protocolo
Los pasos críticos se describen en los pasos 1.1-1.4. Las advertencias y los mensajes de error se implementan en varios lugares de la caja de herramientas para ayudar a los usuarios a comprender por qué pueden encontrar problemas (por ejemplo, ubicaciones de los electrodos no cargadas en los datos de EEG, longitud del archivo demasiado corta para calcular una medida confiable de HRV de frecuencia ultrabaja, calidad de la señal demasiado baja para cualquier análisis ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El Instituto de Ciencias Noéticas apoyó esta investigación. Agradecemos a los desarrolladores de los algoritmos originales de código abierto que se adaptaron para desarrollar algunos de los algoritmos de BrainBeats.
Materiales
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Referencias
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados