Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
BrainBeats как плагин EEGLAB с открытым исходным кодом для совместного анализа сигналов ЭЭГ и сердечно-сосудистой системы
В этой статье
Резюме
BrainBeats toolbox — это плагин EEGLAB с открытым исходным кодом, предназначенный для совместного анализа сигналов ЭЭГ и сердечно-сосудистой системы (ЭКГ/ФПГ). Он включает в себя оценку вызванных сердцебиением потенциалов (HEP), анализ на основе признаков и извлечение сердечных артефактов из сигналов ЭЭГ. Протокол поможет в изучении взаимодействия мозга и сердца через две линзы (HEP и функции), повышая воспроизводимость и доступность.
Аннотация
Взаимодействие между мозгом и сердечно-сосудистой системой привлекает все большее внимание из-за его потенциала для улучшения нашего понимания физиологии человека и улучшения состояния здоровья. Тем не менее, мультимодальный анализ этих сигналов является сложной задачей из-за отсутствия руководящих принципов, стандартизированных инструментов обработки сигналов и статистики, графических пользовательских интерфейсов (GUI) и автоматизации обработки больших наборов данных или повышения воспроизводимости. Еще один пробел существует в стандартизированных методах извлечения признаков ЭЭГ и вариабельности сердечного ритма (ВСР), что подрывает клиническую диагностику или надежность моделей машинного обучения (ML). В ответ на эти ограничения мы представляем набор инструментов BrainBeats. BrainBeats, реализованный в виде плагина EEGLAB с открытым исходным кодом, интегрирует три основных протокола: 1) Вызванные сердцебиением потенциалы (HEP) и осцилляции (HEO) для оценки заблокированного во времени взаимодействия мозга и сердца с точностью до миллисекунды; 2) Извлечение признаков ЭЭГ и ВСР для изучения ассоциаций/различий между различными показателями мозга и сердца или для построения надежных моделей машинного обучения на основе признаков; 3) Автоматизированное извлечение артефактов сердца из сигналов ЭЭГ для устранения любых потенциальных сердечно-сосудистых загрязнений при проведении анализа ЭЭГ. Мы предоставляем пошаговое руководство по применению этих трех методов к набору данных с открытым исходным кодом, содержащему одновременные 64-канальные сигналы ЭЭГ, ЭКГ и ППГ. Пользователи могут легко настраивать параметры в соответствии со своими уникальными исследовательскими потребностями с помощью графического интерфейса пользователя (GUI) или командной строки. BrainBeats должен сделать исследования взаимодействия мозга и сердца более доступными и воспроизводимыми.
Введение
В течение долгого времени редукционистский подход доминировал в научных исследованиях в области физиологии и познания человека. Этот подход включал в себя препарирование сложных телесных и психических процессов на более мелкие, более управляемые компоненты, что позволило исследователям сосредоточиться на отдельных системах в отдельности. Эта стратегия возникла из-за трудностей в изучении сложной и взаимосвязанной природы человеческого тела и разума1. Редукционизм сыграл важную роль в понимании отдельных подсистем по отдельности, например, в выяснении роли ионных каналов и потенциалов действиядля нейронной коммуникации2 или сердечной 3. Тем не менее, остается значительный пробел в нашем понимании того, как эти изолированные системы взаимодействуют в более широком пространственном и временном масштабе. Мультимодальная (интегративная или экологическая) структура рассматривает человеческое тело как сложную многомерную систему, в которой разум рассматривается не как продукт мозга, а как деятельность живого существа, деятельность, которая интегрирует мозг в повседневные функциичеловеческого тела. Мультимодальный и редукционистский подходы не являются исключительными, точно так же, как мы не можем изучать один нейрон без всего мозга или весь мозг без понимания свойств отдельных нейронов. Вместе они прокладывают путь к более всестороннему, синергетическому пониманию человеческого здоровья, патологии, познания, психологии и сознания. Настоящий метод направлен на облегчение мультимодального исследования взаимодействия между мозгом и сердцем путем обеспечения совместного анализа электроэнцефалографии (ЭЭГ) и сердечно-сосудистых сигналов, а именно электрокардиографии (ЭКГ) и фотоплетизмографии (ФПГ). Этот инструментарий, реализованный в виде плагина EEGLAB в MATLAB, устраняет существующие методологические ограничения и сделан открытым исходным кодом для обеспечения доступности и воспроизводимости в научной области. Он внедряет последние руководства и рекомендации в свой дизайн и параметры по умолчанию, чтобы побудить пользователей следовать известным передовым практикам. Предлагаемый инструментарий должен стать ценным ресурсом для исследователей и клиницистов, заинтересованных в 1) изучении вызванных сердцебиением потенциалов, 2) извлечении особенностей из сигналов ЭЭГ и ЭКГ/ФПГ или 3) удалении сердечных артефактов из сигналов ЭЭГ.
Исследования сердца и мозга
Взаимосвязь между сердцем и мозгом исторически изучалась с помощью методов нейровизуализации, таких как функциональная магнитно-резонансная томография (фМРТ) и позитронно-эмиссионная томография (ПЭТ). Используя эти инструменты, исследователи выделили некоторые области мозга, связанные с контролем сердечно-сосудистой системы (например, управление частотой сердечных сокращений и артериальным давлением), показали влияние частоты сердечных сокращений на сигнал BOLD6 или определили потенциальные пути мозг-тело, способствующие ишемической болезни сердца (т.е. артериальное давление, вызванноестрессом7). Несмотря на то, что эти исследования значительно продвинули наше понимание сложного взаимодействия между центральной нервной системой (ЦНС) и сердечно-сосудистой функцией, эти методы нейровизуализации дороги, имеют ограниченную доступность и ограничены контролируемыми лабораторными условиями, что ограничивает их практичность для реальных и крупномасштабных приложений.
Напротив, ЭЭГ и ЭКГ/ФПГ являются более доступными и портативными инструментами, которые предлагают потенциал для изучения взаимодействий мозга и сердца в более разнообразных условиях и популяциях или в течение более длительных периодов времени, предоставляя новые возможности. ЭКГ измеряет электрические сигналы, генерируемые каждым ударом сердца, когда сердце сокращается и расслабляется с помощью электродов, размещенных на коже (обычно на груди или руках)8. PPG измеряет изменения объема крови в микрососудистых тканях (т.е. кровоток и частоту пульса) с помощью источника света (например, светодиода) и фотодетектора (обычно размещаемого на кончике пальца, запястья или лбу), полагаясь на то, как кровь поглощает больше света, чем окружающиеткани. Оба метода предоставляют ценную информацию о сердечно-сосудистой функции, но служат разным целям и предлагают разные типы данных. Как и ЭКГ, ЭЭГ записывает электрические поля, генерируемые синхронизированной активностью тысяч корковых нейронов, которые распространяются через внеклеточный матрикс, ткани, череп и кожу головы, пока не достигнут электродов, размещенныхна поверхности кожи головы. Таким образом, использование ЭЭГ и ЭКГ/ФПГ имеет большие перспективы для улучшения нашего понимания физиологических, когнитивных и эмоциональных процессов, лежащих в основе взаимодействия мозга и сердца, и их последствий для здоровья и благополучия человека. Таким образом, захват взаимодействия сердца и мозга из сигналов ЭЭГ, ЭКГ/ППГ с помощью инструментария BrainBeats может быть особенно полезен в следующих научных областях: клиническая диагностика и прогнозирование, машинное обучение больших данных (ML), самоконтроль в реальноммире11 и мобильная визуализация мозга/тела (MoBI)12,13.
Два подхода к совместному анализу сигналов ЭЭГ и ЭКГ
Существует два основных подхода к изучению взаимодействия между ЭЭГ и сердечно-сосудистыми сигналами:
Вызванные сердцебиением потенциалы (HEP) во временной области: потенциалы, связанные с событиями (ERP), и вызванные сердцебиением осцилляции (HEO) во временной частотной области: спектральные возмущения, связанные с событиями (ERSP) и межсудебная когерентность (ITC). Этот подход изучает, как мозг обрабатывает каждое сердцебиение. С точностью до миллисекунды (мс) этот метод требует, чтобы оба временных ряда были идеально синхронизированы, а сердцебиения были отмечены в сигналах ЭЭГ. Этот подход вызвал интерес в последние годы 14,15,16,17,18,19.
Подход, основанный на функциях: этот подход извлекает особенности ЭЭГ и вариабельности сердечного ритма (ВСР) из непрерывных сигналов и изучает связи между ними. Это было сделано независимо для ЭЭГ (часто называемой количественной ЭЭГ или кЭЭГ20), ЭКГ 21,22,23 и ППГ 24,25,26. Этот подход представляет собой многообещающие приложения, захватывая переменные, связанные как с состоянием, так и с типажем. Обратите внимание, что как для ЭЭГ, так и для сердечно-сосудистых сигналов, чем длиннее запись, тем более доминирующей является переменная черт 27,28,29. Таким образом, приложения зависят от параметров записи. Анализ, основанный на признаках, вызывает все больший интерес, предоставляя новые количественные метрики для прогнозирования развития психических и неврологических расстройств, реакции на лечение или рецидива 30,31,32,33,34,35. Этот подход особенно привлекателен для больших и реальных наборов данных (например, клиника, удаленный мониторинг), которые могут быть более легко получены благодаря последним инновациям в носимыхнейротехнологиях. Менее изученным применением является выявление связей между конкретными особенностями мозга и сердца, что подчеркивает потенциал, лежащий в основе динамики центральной нервной системы. Вариабельность сердечного ритма (ВСР) может быть рассчитана по сигналам ЭКГ и ФПГ. Он предоставляет информацию об автономной нервной системе (ВНС) путем измерения вариаций временных интервалов между сердечными сокращениями (т.е. интервалов от нормы к норме)27. Повышенная симпатическая активность (например, во время стресса или физических упражнений) обычно снижает ВСР, в то время как парасимпатическая активность (ПНС) (например, во время релаксации) увеличивает ее. Более медленная частота дыхания обычно увеличивает ВСР из-за повышенной активности ПНС, особенно при коротких записях (<10 минут)27. Более высокие показатели ВСР, как правило, указывают на более устойчивую и адаптируемую ВНС, в то время как более низкая ВСР может указывать на стресс, усталость или основные проблемы со здоровьем. Длительные записи ВСР (т.е. не менее 24 часов) обеспечивают прогностический прогноз для различных состояний здоровья, включая сердечно-сосудистые заболевания, стресс, тревогу и некоторые неврологическиесостояния. Такие показатели, как артериальное давление, частота сердечных сокращений или уровень холестерина, дают информацию о состоянии сердечно-сосудистой системы. Напротив, ВСР добавляет динамический аспект, показывая, как сердце реагирует на стресс и восстанавливается после него.
Преимущества BrainBeats перед существующими методами
Несмотря на то, что существуют инструменты, рассмотренные ниже, для обработки сердечно-сосудистых и ЭЭГ-сигналов независимо друг от друга, они не могут быть совместно проанализированы. Кроме того, большинство доступных средств обработки сердечно-сосудистых сигналов требуют дорогостоящего лицензирования, не позволяют автоматизировать обработку (что особенно выгодно для больших наборов данных), имеют собственные алгоритмы, препятствующие прозрачности и воспроизводимости, или требуют продвинутых навыков программирования, не предоставляя графического пользовательского интерфейса (GUI)36. Насколько нам известно, четыре набора инструментов MATLAB с открытым исходным кодом поддерживают анализ HEP/HEO с графическим интерфейсом пользователя: набор инструментовECG-kit 37, конвейер BeMoBIL38, плагин HEPLAB EEGLAB39 и набор инструментов CARE-rCortex40. Несмотря на то, что HEPLAB, BeMoBIL и набор для ЭКГ облегчают анализ HEP, обнаруживая сердцебиение и отмечая его в сигналах ЭЭГ, они не обеспечивают статистический анализ или ограничены временной областью (т.е. HEP). Плагин CARE-rCortex решает эти проблемы за счет поддержки ЭКГ и дыхательных сигналов, анализа во временной и частотной области, статистики и передовых методов нормализации и коррекции исходного уровня, адаптированных к анализу HEP/HEO. Тем не менее, он использует метод Бонферрони для статистической коррекции ошибки 1-го типа (т.е. ложноположительных), которая является слишком консервативной и физиологически не обоснованной для приложений ЭЭГ, что приводит к увеличению ошибок II типа (т.е. ложноотрицательных результатов)41. Кроме того, набор инструментов не предлагает доступ к командной строке для автоматизации. Наконец, недавние исследования не рекомендуют использовать методы коррекцииисходного уровня 42,43,44, так как они снижают отношение сигнал/шум (SNR) и являются статистически ненужными и нежелательными.
Чтобы обойти эти ограничения, мы представляем набор инструментов BrainBeats, который в настоящее время реализован в виде плагина EEGLAB с открытым исходным кодом в среде MATLAB. Он включает в себя следующие преимущества по сравнению с предыдущими методами:
1) Простой в использовании графический интерфейс и возможности командной строки (для программистов, стремящихся выполнять автоматизированную обработку). 2) Валидированные алгоритмы, параметры и рекомендации по обработке сердечно-сосудистых сигналов, такие как обнаружение пиков R, интерполяция артефактов RR и вычисление метрик ВСР (например, рекомендации по имплантации окон, ресамплизации, нормализации и т. д.27,45,46). Это важно, потому что Vest et al. продемонстрировали, как незначительные различия в этих этапах обработки могут привести к расходящимся результатам, способствуя отсутствию воспроизводимости и клинической применимости метрик ВСР46. 3) Валидированные алгоритмы, стандартные параметры и рекомендации по обработке сигналов ЭЭГ, включая фильтрацию и окноирование44,47, повторные ссылки48,49, удаление аномальных каналов и артефактов 50,51,52, оптимизированную декомпозицию ICA и классификацию независимых компонентов 53,54,55,56. Пользователи могут точно настроить все параметры предварительной обработки или даже предварительно обработать свои данные ЭЭГ с помощью предпочитаемого метода, прежде чем использовать набор инструментов в соответствии со своими потребностями (например, с помощью плагина50,52 EEGLAB clean_rawdata, конвейера BeMoBIL38, конвейера PREP57 и т.д.). 4) Вызванные сердцебиением потенциалы (HEP, т.е. во временной области) и осцилляции (HEO; спектральные возмущения, связанные с событиями с помощью вейвлет-методов или БПФ, а также межпробная когерентность доступны с помощью стандартного программного обеспечения EEGLAB) из сигналов ЭКГ. Параметрическая и непараметрическая статистика с поправками на ошибки 1-го типа доступна в стандартном программном обеспечении EEGLAB. Непараметрическая статистика включает статистику перестановок и пространственно-временные поправки для множественных сравнений (например, пространственно-временная кластеризация или беспороговое кластерное усиление)58,59. Пользователи могут использовать плагин LIMO-EEG для реализации иерархического линейного моделирования, которое хорошо учитывает дисперсию внутри и между субъектами и реализует массово-одномерный подход без предположений с надежным контролем ошибок I и II типа60,61. Статистический анализ данных HEP/HEO может быть выполнен в канальной и независимой компонентной областях. 5) HEP/HEO и анализ ВСР по сигналам PPG (впервые для HEP/HEO). 6) Впервые поддерживает совместное извлечение признаков ЭЭГ и ВСР. 7) Инструментарий предоставляет различные визуализации данных для проверки сигналов на различных необходимых этапах обработки и выходных данных на уровне предмета.
| Метод | Определение R-пиков по данным ЭКГ | Обнаружение R-волн от PPG | HEP/HEO | Особенности ЭЭГ и ВСР | Удаление артефактов сердца с ЭЭГ | Графический интерфейс пользователя | Командная строка |
| ЭКГ-комплект | X | X | X | X | |||
| БеМоБИЛ | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| БрейнБитс | X | X | X | X | X | X | X |
Таблица 1: Новшества, привнесенные BrainBeats по сравнению с ранее существовавшими аналогичными методами.
Информация, которая поможет читателям решить, подходит ли им этот метод
Этот набор инструментов подходит для любого исследователя или клинициста с данными ЭЭГ и ЭКГ/ФПГ. Плагин пока не поддерживает импорт сигналов ЭЭГ и ЭКГ/ФПГ из отдельных файлов (хотя эта функция будет доступна в ближайшее время). Набор инструментов подходит для тех, кто хочет провести анализ HEP/HEO, извлечь особенности ЭЭГ и/или ВСР с помощью стандартизированных методов или просто удалить сердечные артефакты из сигналов ЭЭГ. На рисунке 1 показана блок-схема, обобщающая общий поток и методы BrainBeats.
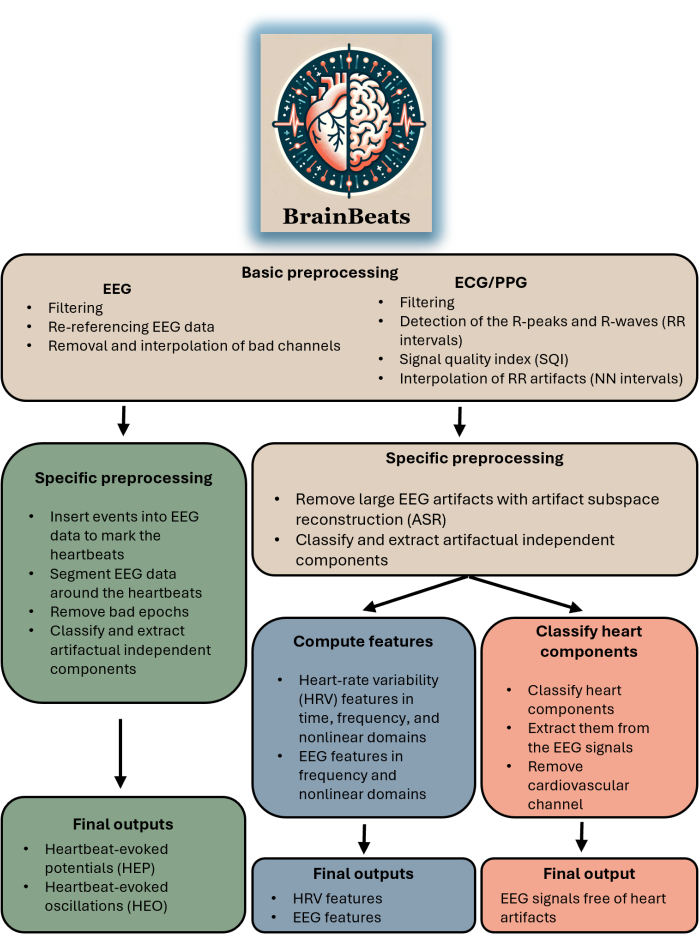
РИСУНОК 1. Блок-схема, обобщающая общую архитектуру и поток BrainBeats. Операции, общие для всех трех методов, выделены коричневым цветом. Операции, специфичные для вызванных сердцебиением потенциалов (HEP) и колебаний (HEO), выделены зеленым цветом. Операции, специфичные для извлечения признаков ЭЭГ и ВСР, выделены синим цветом. Операции, специфичные для удаления сердечных артефактов из сигналов ЭЭГ, выделены красным цветом. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
протокол
От каждого участника было получено информированное согласие, а этический комитет Уральского федерального университета утвердил протокол эксперимента.
1. Требования BrainBeats
- Установите MATLAB и EEGLAB на компьютер. EEGLAB можно скачать по адресу https://github.com/sccn/eeglab и распаковать (или клонировать для пользователей Git) в любом месте компьютера. Дополнительные сведения об установке см. на странице GitHub.
- Добавьте путь к папке EEGLAB на главной панели MATLAB, нажав кнопку «Задать путь ». Выберите папку eeglab с распакованным файлом и нажмите «Сохранить» > «Закрыть».
- Запустите EEGLAB, набрав eeglab в командном окне MATLAB.
- Установите плагин BrainBeats, нажав «Файл» > «Управление расширениями EEGLAB». Введите brainbeats в строку поиска, выберите в списке плагин BrainBeats и нажмите «Установить/обновить».
- Загрузите образец набора данных в EEGLAB. Нажмите кнопку Файл > Загрузить существующие данные. Перейдите в папку EEGLAB, перейдите в папку плагинов, перейдите в папку BrainBeats и откройте папку sample_data. Выберите файл dataset.set.
ПРИМЕЧАНИЕ: Этот набор данных соответствует sub-032 (состояние покоя с открытыми глазами) из набора данных с открытым исходным кодом62,63. Этот вариант был выбран потому, что он содержит одновременные ЭЭГ (64 канала), ЭКГ (один канал) и ФПГ (один канал) с частотой дискретизации 1000 Гц. Сигналы ЭКГ и ФПГ собирали с помощью вспомогательных входов системы actiCHamp. Данные ЭЭГ регистрировали с помощью активных электродов, размещенных по системе 10-20, с FCz в качестве онлайн-эталона и Fpz в качестве заземляющего электрода, с поддержанием импеданса ниже 25 кОм. ЭКГ регистрировали с помощью одного активного электрода, размещенного на правом запястье, электрода сравнения на левом запястье и земли на левом внутреннем предплечье на расстоянии 3 см от локтя. ППГ регистрировалась с указательного пальца левой руки. Данные ЭЭГ, ЭКГ и ФПГ синхронизированы по времени, так как они были записаны одновременно с помощью одного и того же усилителя. Более подробную информацию см. в ссылках62,63. - Установите флажок «Сохранить выходы», чтобы сохранить все данные в соответствующем файле .set в том же месте, что и исходный файл, который был загружен в EEGLAB.
ПРИМЕЧАНИЕ: В этом уроке мы объединили данные ЭЭГ, ЭКГ и PPG в один набор данных EEGLAB, загрузили координаты электродов методом 3D граничных элементов (BEM) и уменьшили частоту сигнала до 250 Гц, чтобы уменьшить вес файла (для загрузки пользователем) и ускорить время вычислений. Поскольку в этом наборе данных не было аномального канала ЭЭГ, мы искусственно изменили канал TP9, чтобы проиллюстрировать алгоритм обнаружения и удаления плохих каналов BrainBeats. Аналогичным образом, мы искусственно моделируем большой артефакт ЭЭГ в начале файла и высокочастотный мышечный артефакт в височных каналах от 3 до 6 с, чтобы проиллюстрировать функцию удаления артефактов в BrainBeats.
2. Вызванные сердцебиением потенциалы (HEP) и колебания (HEO)
- Откройте первый общий пользовательский интерфейс (GUI) BrainBeats, чтобы выбрать основные параметры. В EEGLAB нажмите «Инструменты» > BrainBeats >1-м уровне (уровень предмета). Выберите Heartbeat-induced potentials HEP в качестве анализа для выполнения, ЭКГ в качестве типа данных сердца и нажмите на кнопку, чтобы отобразить список каналов, чтобы выбрать канал ЭКГ, помеченный как ECG (или введите его непосредственно в текстовое поле). Оставьте выбранными опции Визуализировать выходные данные (рекомендуется) и Сохранить выходные данные и нажмите Ok.
- Второе окно графического интерфейса пользователя открывается на основе предыдущих вариантов (т.е. анализ HEP с данными ЭКГ). Выберите параметры предварительной обработки для сигналов ЭЭГ и ЭКГ. Отключите предварительную обработку, сняв флажки «Предварительная обработка ЭКГ » и « Предварительная обработка ЭЭГ », чтобы проверить, были ли данные уже предварительно обработаны перед запуском BrainBeats. Измените шум линии питания на 50 Гц в секции предварительной обработки ЭЭГ , так как эти данные ЭЭГ были зарегистрированы в России. Нажмите OK для запуска.
- Появится предупреждающее сообщение с просьбой подтвердить удаление обнаруженного канала PPG. Это связано с тем, что инструментарий не предназначен для одновременного анализа ЭКГ и ФПГ (или других вспомогательных каналов), и сохранение его в наборе данных приведет к серьезным ошибкам (например, удалению артефактов, плохой декомпозиции ICA и т. д.). Нажмите кнопку Да. BrainBeats начинает выполнять некоторые проверки, устанавливая некоторые параметры по умолчанию, и отделяет ЭКГ от данных ЭЭГ для предварительной обработки сигнала ЭКГ и вычисления интервалов RR.
- Временные ряды ЭКГ и RR предварительно обрабатываются с использованием проверенных алгоритмов из набора инструментов Physionet Cardiovascular Signal46,64, адаптированных к форматированию данных BrainBeats, повышению ясности, настройке параметров и времени вычислений (см. ссылки для проверки алгоритмов). Плагин выводит интервалы RR, временные метки, отфильтрованный сигнал ЭКГ, индексы R-пиков и частоту сердечных сокращений (ЧСС). Настройте эти параметры с помощью графического интерфейса пользователя или командной строки.
ПРИМЕЧАНИЕ: Сигнал ЭКГ фильтруется по полосе пропускания с использованием специализированного, проверенного фильтра (1-30 Гц) и сканирует сигнал для идентификации комплекса QRS и R-пиков с использованием метода Пана-Томпкинса (P&T)65, реализуя некоторые операции обработки сигнала, включая дифференцирование, возведение в квадрат, интегрирование и сглаживание для достижения наилучших результатов. Энергетический порог P&T оценивается на основе частоты дискретизации и сглаженных значений ЭКГ, чтобы избежать разрушения из-за больших неровностей. Если вариабельность интервала RR превышает медиану в 1,5 раза, выполняется поиск пропущенных пиков. Средний знак R-пика рассчитывается по сегментам 30 с, а пиковые точки уточняются с помощью проверки рефрактерного периода, управления плоскими условиями и обеспечения согласованного обнаружения. - Затем BrainBeats идентифицирует аномальные интервалы RR или пики в интервалах RR, используя прямой и обратный поиск и физиологические пороги. Индекс качества сигнала (SQI) рассчитываетсякак 46; проверьте это, если система отображает предупреждения, в которых более 20% временного ряда RR содержат артефакты RR (за пределами физиологических пределов или с SQI ниже 0,9). На графике отображается отфильтрованный сигнал ЭКГ, идентифицированные R-пики, интервалы NN и интерполированные артефакты (см. рис. 2).
ПРИМЕЧАНИЕ: Артефакты RR интерполируются по умолчанию с использованием кусочно-кубического метода с сохранением формы для получения интервалов от нормали к нормали (NN), но удаляют их (не рекомендуется) или используют другой метод интерполяции (линейный, кубический, ближайший сосед, предыдущий/следующий сосед, сплайн, кубическая свертка или модифицированный кубический Akima), если это необходимо. При наличии нескольких каналов ЭКГ для каждого из них оцениваются интервалы RR, а для следующих этапов выбирается канал с наименьшим числом артефактов RR. - Прокрутите увеличенные 30-секундные окна R-пиков для более близкого рассмотрения, нажимая стрелки вправо/влево. Если данные содержат несколько каналов ЭКГ/ФПГ, используйте канал с наилучшим индексом качества сигнала для интервалов RR. В настоящее время BrainBeats не поддерживает сигналы ЭКГ и ФПГ одновременно. В качестве учебника мы выбрали пример набора данных, который содержит оба типа данных.
- После обработки сигнала ЭКГ полоса BrainBeats фильтрует данные ЭЭГ на частоте 1-40 Гц, используя нелинейный причинно-следственный фильтр минимальной фазы КИХ, который по умолчанию снижает активность смазывания между периодами до и после сердцебиения, сохраняет причинно-следственную связь и позволяет избежатьнежелательных групповых задержек. Это особенно важно для пользователей, изучающих период до сердцебиения. Если фильтр нижних частот установлен на значение выше частоты сети питания (например, 80 Гц при частоте сети питания 50 Гц), используйте фильтр с резким режектором для удаления артефакта шума линии. Затем данные ЭЭГ повторно привязываются до бесконечности с использованием алгоритма REST (лучше всего подходит для анализа HEP49), если не обнаружено менее 30 каналов (в этом случае они не могут быть надежно повторно привязаны, и генерируется предупреждение, чтобы сообщить об этом пользователям).
- Затем BrainBeats обнаруживает, удаляет и интерполирует аномальные каналы ЭЭГ (рис. 3). Убедитесь, что параметрами по умолчанию являются плоские линии больше 5 с (алгоритм clean_flatlines), максимальное стандартное отклонение высокочастотного шума 10, длина окна 5 с (для лучшего захвата низкочастотных артефактов52), минимальная корреляция между соседними каналами 0,65 и максимально допустимая доля 33% (алгоритм clean_channels).
ПРИМЕЧАНИЕ: Количество выборок RANSAC по умолчанию установлено равным 500 для повышения сходимости и воспроизводимости плохого отклонения канала (хотя это увеличивает время вычислений). - Затем вставьте R-пики в качестве маркеров событий в данные ЭЭГ, чтобы отметить каждый пульс, и сегментируйте данные вокруг этих маркеров без удаления исходного уровня (согласно рекомендациям43,66; Рисунок 4). Поскольку интервалы NN имеют разную длину и ЭЭГ должна быть сегментирована с постоянной длиной, оцените минимальный предел размера эпохи после событий R-пика, используя5-й процентиль данных межтактового интервала (IBI) (т.е. значение, ниже которого опускаются 5% самых коротких IBI, отображается пунктирной красной линией на гистограмме; см. рис. 5).
ПРИМЕЧАНИЕ: Это значение5-го процентиля является хорошим компромиссом для сохранения как можно большего количества эпох, гарантируя, что они не будут слишком короткими, поскольку период интереса для анализа HEP/HЭО составляет 200-600 мс после пульса 49,67. - Сегментируйте данные ЭЭГ от -300 мс до R-пика до5-го процентиля после R-пика, с R-пиком в момент времени 0. Отклоняйте эпохи короче 550 мс или содержащие более одного R-пика (что приведет к смещению ERP/ESP), согласно рекомендациям49,67. Эпохи, содержащие большие артефакты ЭЭГ, обнаруживаются с помощью метрик среднеквадратичного значения (RMS) и отношения сигнал/шум (SNR), а также функции изовыброса MATLAB (рис. 6). Искусственные эпохи удалены.
- Выполнение слепого разделения источников с использованием стандартного алгоритма Infomax с учетом ранга данных для достижения наилучшей производительности53. При желании выберите алгоритм предобусловленного анализа независимых компонент (PICARD) для быстрого вычисления 54,54, выбрав опцию Picard (быстрый), или модифицированный алгоритм Infomax, выбрав опцию Modified Infomax (длинный, но воспроизводимый) для метода ICA поля (хотя это требует гораздо более длительного времени вычислений). Используйте плагин ICLabel56 для автоматической классификации ИС для извлечения артефактов, не относящихся к мозгу (глазные компоненты удаляются с 90% достоверностью, тогда как мышечные, линейные шумы и шумы каналов удаляются с 99% достоверностью; Рисунок 7).
- Оставьте выбранный параметр Визуализировать выходы в первом окне графического интерфейса, чтобы визуализировать окончательный временной ряд ЭЭГ, большое среднее значение HEP (Рисунок 8 вверху), HEP для каждого сердцебиения (Рисунок 8 внизу) и большое среднее количество колебаний, вызванных сердцебиением (HEO; Рисунок 9). Исследуйте ВЭО с точки зрения спектральных возмущений, связанных с событиями (ERSP, т.е. изменения мощности ЭЭГ в течение сердечных сокращений; Рисунок 9 вверху) и когерентность фазы между испытаниями (ITC, т.е. постоянство фазового угла в течение сердечных сокращений; Рисунок 9 внизу).
ПРИМЕЧАНИЕ: ERSP вычисляется с использованием стандартного 3-тактового вейвлета (с примененным коническим окном Хеннинга, отношением прокладок 2) и с числом циклов в вейвлетах, используемых для более высоких частот, медленно расширяющимся до 20% от числа циклов в эквивалентном окне БПФ на его самой высокой частоте (1 минус 0,8). Это позволяет управлять формами отдельных окон, измеряемых функцией, и их формами в результирующих стеклах времени/частоты. Произвольная базовая линия удаляется в иллюстративных целях, и ERSP вычисляется для частот 7-25 Гц, чтобы зафиксировать типичный эффект HEO, описанный в литературе, а именно 300-450 мс после сердцебиения в альфа-диапазоне (8-13 Гц) над лобноцентральными электродами17,67. Более низкие частоты не могут быть оценены из-за короткого размера эпохи, определяемого интервалами между тактами. Непараметрическая статистика (перестановка) применяется для визуализации HEO для p-значения 0,05, скорректированного на частоту ложных открытий (FDR, т.е. ошибка 1-го типа или ошибка семейства). Эти графики генерируются в учебных целях или для анализа в отдельных испытаниях. - Графики предварительной обработки генерируются для визуализации различных этапов. Чтобы отключить его, снимите флажок Визуализировать препроцессирование. Окончательные данные ЭЭГ (очищенные и сегментированные вокруг R-пиков) не включают данные ЭКГ, поскольку это может привести к смещению анализа ERP/ESP. Чтобы сохранить сердечный канал на конечном выходе, установите флажок Сохранить сердечный канал.
ПРИМЕЧАНИЕ: При необходимости сделайте паузу перед обработкой следующего файла (следующего условия или участника). - BrainBeats поддерживает функцию истории EEGLAB. В конце всех операций введите eegh в командном окне MATLAB, чтобы распечатать командную строку и повторить все вышеуказанные шаги через одну командную строку, с параметрами, которые были выбраны вручную в графическом интерфейсе, что позволяет легко автоматизировать. Найдите выходы предварительной обработки (например, индекс качества сигнала сердечно-сосудистых временных рядов, интервалы NN, удаленные каналы ЭЭГ, сегменты и компоненты и т. д.) в структуре EEGLAB: EEG.brainbeats.preprocessings. Все параметры также экспортируются в EEG.brainbeats.parameters.
- Для продвинутых пользователей выполните все вышеуказанные шаги с параметрами по умолчанию с помощью следующих командных строк (дополнительные параметры см. в учебном скрипте в репозитории BrainBeats):
eeglab; закрывать; % Запуск EEGLAB без графического интерфейса
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('имя_файла','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); %Загрузить образец набора данных
ЭЭГ = brainbeats_process(ЭЭГ,'анализ',
'hep','heart_signal','ECG', 'heart_channels',
{'ECG'},'clean_eeg',true); % Запустите BrainBeats1-го уровня для обработки файла для анализа HEP с параметрами по умолчанию - На описанных выше этапах выполняется HEP/HEO по сигналу ЭКГ. Выполните следующие действия для сигнала PPG.
- В следующих шагах выполните те же операции, но с использованием сигнала PPG. Загрузите тот же набор данных снова (см. шаг 1.5), так как предыдущие операции перезаписали его, и снова откройте первый графический интерфейс BrainBeats, чтобы выбрать основные параметры. Нажмите Инструменты > BrainBeats >1-м уровне (уровень предмета). Выберите Heartbeat-induced potentials (HEP) в качестве анализа для выполнения, PPG в качестве типа данных о сердце, и нажмите на кнопку, чтобы отобразить список каналов, чтобы выбрать канал PPG. Оставьте выбранными параметры Визуализировать выходные данные (рекомендуется) и Сохранить выходные данные и нажмите OK.
- Второе окно с графическим интерфейсом пользователя появляется таким же образом, как и на шаге 2.2. Единственное отличие заключается в том, что теперь мы можем тонко настроить параметр для обработки сигнала PPG вместо сигнала ЭКГ. Нажмите OK , чтобы запустить с параметрами по умолчанию.
- Появится предупреждающее сообщение с просьбой подтвердить удаление обнаруженного дополнительного канала ЭКГ. Опять же, это ожидаемо. Нажмите кнопку Да. По умолчанию набор инструментов выполняет предварительную обработку сигнала PPG, обнаруживает пульсовые волны для получения интервалов RR, идентифицирует артефакты RR, если таковые имеются, и интерполирует их (рис. 10). Шаги 2.7. к 2.12. и генерируются те же графики и выходные данные, но на основе волн R, обнаруженных по сигналу PPG (см. Рисунок 11 и Рисунок 12).
ПРИМЕЧАНИЕ: Волны R обнаруживаются с помощью наклона сигнала в пределах указанного окна. Затем потенциальные импульсы помечаются, когда наклон превышает динамический порог, который корректируется на основе истории обнаружения и характеристик сигнала. Затем алгоритм выполняет поиск в течение периода закрытия глаза, чтобы точно определить максимальный наклон, а затем начало пульсовой волны определяется с помощью порогового значения. Пики R-волны идентифицируются как впадины вблизи начала, и их местоположение записывается. Алгоритм перебирает весь сигнал, непрерывно корректируя пороги обнаружения и определяя пики R-волны, которые затем используются для вычисления интервалов RR. - Для продвинутых пользователей выполните все вышеуказанные шаги с параметрами по умолчанию с помощью следующих командных строк (дополнительные параметры см. в учебном скрипте в репозитории BrainBeats):
eeglab; закрывать; % Запуск EEGLAB без графического интерфейса
main_path = fileparts(which('eegplugin_
BrainBeats.m')); cd(main_path);
EEG = pop_loadset('имя_файла','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); %Загрузить образец набора данных
ЭЭГ = brainbeats_process(ЭЭГ,'анализ',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',true); % Запустите BrainBeats1-го уровня для обработки файла для анализа HEP с параметрами по умолчанию
3. Извлечение особенностей ЭЭГ и ВСР
- Загрузите тот же набор данных еще раз (см. шаг 1.5; Нажмите File > Load existing dataset > Select dataset.set), так как он был перезаписан предыдущими операциями, и снова откройте основной графический интерфейс для выбора основных параметров (шаг 2.1; Нажмите «Инструменты» > BrainBeats >1-м уровне). Выберите Извлечь признаки ЭЭГ и ВСР для типа анализа, ЭКГ для типа сердечного сигнала и выберите ЭКГ в списке меток электродов. Нажмите OK.
- Второе окно с графическим интерфейсом появляется, как и в шаге 2.2, но с другими параметрами для предварительной обработки ЭЭГ и извлечения характеристик ВСР, и ЭЭГ. Установите эти параметры, как описано ниже. Нажмите OK для запуска с параметрами по умолчанию.
- В разделе «ВСР» нажмите на кнопку « Параметры частоты», чтобы выбрать метод вычисления мощности ВСР (по умолчанию установлено нормализованная периодограмма Ломба-Скаргла), перекрытие окон (по умолчанию установлено значение 25%) и для выполнения нормализации второго уровня (по умолчанию не установлено; см. примечание ниже для получения более подробной информации).
- В разделе «Функции ЭЭГ» нажмите на кнопку «Параметры частоты», чтобы точно настроить некоторые параметры, такие как общий диапазон частот, для которого вычисляется спектральная плотность мощности (PSD; по умолчанию = 1-40 Гц), единицы измерения (децибелы, μВ2/Гц или нормализованные по общей мощности), тип окна (по умолчанию = хамминг), перекрытие окон (по умолчанию = 50%), длина окна (по умолчанию = 2 с), и типы частотных границ для каждого диапазона.
ПРИМЕЧАНИЕ: Мощность ВСР рассчитывается по умолчанию с использованием нормализованной периодограммы Ломба-Скаргла, которая не требует повторной дискретизации (следовательно, лучше сохраняет исходную информацию) и лучше всего справляется с неравномерно дискретизированными данными, недостающими данными и шумом (типично для интервалов NN)68. Нормализованная версия масштабирует мощность в зависимости от дисперсии сигнала, обеспечивая результаты, которые менее чувствительны к изменяющимся уровням шума, более сосредоточены на относительной силе периодических составляющих и более сопоставимы между различными записями или объектами. Другие доступные методы включают ненормализованную периодограмму Ломба-Скаргла, метод Уэлча и быстрое преобразование Фурье (БПФ). Для методов Уэлча и БПФ для создания необходимых регулярных временных рядов выполняется повторная выборка. Нормализация второго уровня может быть применена путем деления мощности каждой полосы частот на общую мощность, что обеспечивает более интуитивную меру относительного вклада каждой частотной составляющей в общую мощность. По умолчанию он отключен, так как имеет смысл только при наличии всех четырех диапазонов, требующих не менее 24 часов сигнала. Эти алгоритмы адаптированы из набора инструментов46 для обработки сердечно-сосудистых сигналов Physionet.
- Появляется то же предупреждающее сообщение с просьбой подтвердить удаление обнаруженного дополнительного канала PPG. Опять же, это ожидаемо. Нажмите кнопку Да. BrainBeats начнет предварительную обработку данных ЭКГ и извлечение интервалов NN идентично, как на шаге 2.4. Затем он извлекает характеристики вариабельности сердечного ритма (ВСР) из интервалов NN во временной (SDNN, RMSSD, pNN50), частотной (УНЧ, ОНЧ, НЧ, ВЧ, отношение НЧ:ВЧ, общая мощность) и нелинейной (Пуанкаре, фазовыпрямленное усреднение сигнала, нечеткая энтропия и фрактальная размерность) областях.
ПРИМЕЧАНИЕ: BrainBeats автоматически проверяет длину файла, чтобы убедиться в соблюдении минимальных требований (например, для питания ULF-HRV требуется 24 часа данных), отправляет предупреждающие сообщения, если это не так, и не экспортирует эти функции для предотвращения ненадежных оценок. BrainBeats следует указаниям и рекомендациям по оценке показателей ВСР27,45. - BrainBeats предварительно обрабатывает данные ЭЭГ, как показано на шаге 2.7. (см. Рисунок 3). Большие артефакты обнаруживаются автоматически в непрерывных данных с использованием алгоритма реконструкции подпространства артефактов (ASR) 50,52 (критерий SD по умолчанию установлен на 30 и использование 80% доступной оперативной памяти для увеличения скорости). Эти большие артефакты удаляются из данных ЭЭГ (см. рисунок 13). Чтобы настроить эти параметры в графическом интерфейсе, выберите поля Порог для отклонения плохих сегментов с ASR и Доступная оперативная память для использования для ASR.
ПРИМЕЧАНИЕ: Временные ряды ЭЭГ и сердечно-сосудистой системы не нуждаются в синхронизации по времени для режима функций, поскольку особенности оцениваются по каждому сигналу отдельно. Таким образом, артефакты ЭЭГ могут быть удалены непосредственно из данных ЭЭГ (выделены красным цветом, рис. 13), в отличие от режима HEP, где эпохи, содержащие артефакты, были отвергнуты для обоих временных рядов, поскольку для этого метода важна синхронизация времени с точностью мс. - Выполняйте ВСА с использованием тех же алгоритмов и параметров, что и для HEP (см. шаг 2.11.), за исключением того, что на этот раз компоненты сердца удаляются при обнаружении с вероятностью 99% (они были сохранены для HEP/HEO, поскольку мы не хотим удалять соответствующие сердечные сигналы).
- На шаге 3.2 отмечен флажок «Частотная область », поэтому BrainBeats извлекает следующие характеристики частотной области: среднюю спектральную плотность мощности (PSD) для полос частот дельта (1-3 Гц), тета (3-7 Гц), альфа (8-13 Гц), бета (13-30 Гц) и гамма (30+ Гц), индивидуальная альфа-частота (IAF) и альфа-асимметрия на всех доступных (симметричных) парах электродов. Проверьте эти функции.
ПРИМЕЧАНИЕ: PSD, преобразованный в децибелы (дБ), облегчает сравнение результатов по записям и объектам. Частотные границы могут быть установлены в соответствии с условными границами (например, предопределенными 8-13 Гц для альфа-диапазона) или в соответствии с индивидуализированными границами, которые обнаруживаются по распределению спектральной плотности мощности для учета межиндивидуальных различий69 (например, 7,3-12,6 Гц для альфа-диапазона). Алгоритм был разработан для альфа-диапазона и не работает так же хорошо для других диапазонов, особенно когда пики отсутствуют в спектральном распределении мощности. Индивидуальная альфа-частота (IAF) оценивается с использованием альфа-центра тяжести для лучшей работы с расщепленными пиками или неоднозначными пиками69. Альфа-асимметрия вычисляется в соответствии с рекомендациями (2-секундное окно Хамминга с 50% перекрытием, логарифм альфа-мощности из левого канала минус логарифм альфа-мощности из правого канала)47. Следовательно, положительные значения указывают на большую альфа-мощность слева, чем справа, и наоборот. Альфа-асимметрия может быть нормализована путем деления альфа-мощности от каждого электрода на альфа-мощность, суммированную по всем электродам47. Симметричные пары получаются с использованием тета-расстояний, требующих, чтобы данные ЭЭГ содержали координаты электродов. - На шаге 3.2 отмечен флажок Нелинейная область , BrainBeats извлекает нечеткую энтропию и фрактальную размерность для каждого канала ЭЭГ.
Примечание: Считается, что функции нелинейной области улавливают нелинейную, сложную динамику мозга, которая не учитывается при спектральных измерениях, и демонстрируют особые перспективы для исследования взаимодействий между различными системами организма 70,71,72. Нечеткая энтропия более надежна и устойчива, чем ее альтернативы (выборочная и приближенная энтропия), но требует более длительного времени вычислений (особенно при длинных временных рядах ЭЭГ с высокой частотой дискретизации). Чтобы решить эту проблему, когда сигналы ЭЭГ длятся более 2 минут с частотой дискретизации более 100 Гц, они автоматически понижаются (или уменьшаются, если множитель не является целым числом) до 90 Гц (т. е. соответствуют частоте Найквиста 45 Гц, чтобы соответствовать фильтру нижних частот по умолчанию и максимально избегать артефактов линейного шума). Кроме того, параллельные вычисления активируются по умолчанию при оценке характеристик ЭЭГ, что сокращает время вычислений, особенно при наличии большого количества каналов ЭЭГ. - Выберите Визуализировать выходные данные в первом графическом интерфейсе пользователя (см. шаг 3.1.), чтобы создать график, отображающий спектральную плотность мощности (PSD) для данных ВСР и ЭЭГ (рисунок 14), а также топографии кожи головы, отображающие некоторые особенности ЭЭГ (рисунок 15).
ПРИМЕЧАНИЕ: Вы также можете найти некоторые выходные данные предварительной обработки в EEG.brainbeats.preprocessing и все параметры, используемые в EEG.brainbeats.parameters. Мы призываем пользователей сообщать об этих результатах в научных публикациях, чтобы повысить воспроизводимость результатов. - Установите флажок Сохранять выходные данные в первом окне графического интерфейса, чтобы сохранить все функции, экспортированные в файл EEGLAB .set в EEG.brainbeats.features, и сохранить их в файле .mat в той же папке, где был загружен набор данных.
- BrainBeats поддерживает функцию истории EEGLAB. В конце всех операций введите eegh в командном окне MATLAB, чтобы распечатать командную строку, которая позволит вам повторить все вышеуказанные шаги через одну командную строку, с параметрами, которые были выбраны вручную в графическом интерфейсе, что позволяет легко автоматизировать и повторить операции.
- Для продвинутых пользователей выполните все описанные выше действия с помощью следующей команды:
eeglab; закрывать; % Запуск EEGLAB без графического интерфейса
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('имя_файла','dataset.set',
'filepath',fullfile(main_path, 'sample_data')); % Загрузить образец набора данных
ЭЭГ = brainbeats_process(ЭЭГ,' анализ',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - На предыдущих шагах из сигнала ЭКГ извлекались особенности ВСР. Выполните следующие действия, чтобы извлечь признаки ВСР из сигнала PPG (функции ЭЭГ те же).
- Загрузите тот же набор данных снова (шаг 1.5.), так как он был перезаписан операциями, и снова откройте основной графический интерфейс (шаг 2.5.). Выберите «Извлечь характеристики ЭЭГ и ВСР» для анализа, а затем выберите PPG для типа сердечного сигнала и PPG для названия канала. Нажмите OK.
- Во2-м окне графического интерфейса теперь отображаются параметры для предварительной обработки PPG и извлечения признаков ВСР и ЭЭГ. Нажмите OK , чтобы запустить с параметрами по умолчанию. Параметры описаны в шаге 2.17.
- Появится предупреждающее сообщение с просьбой подтвердить удаление обнаруженного канала ЭКГ. Этого следовало ожидать, так как инструментарий не предназначен для одновременного анализа ЭКГ и ФПГ (или других вспомогательных каналов) и сохранение его в наборе данных приведет к серьезным ошибкам (например, удалению артефактов, плохому разложению ICA и т. д.). Нажмите кнопку Да.
- BrainBeats предварительно обрабатывает сигнал PPG и оценивает интервалы NN как на шаге 2.5. Проверьте извлеченные признаки ВСР из интервалов NN, как и на шаге 3.2. за исключением того, что интервалы NN теперь получены из сигнала PPG. Сигналы ЭЭГ предварительно обрабатываются, как показано на шаге 3.2. BrainBeats строит график PSD (рис. 16). Единственное различие здесь заключается в том, что ПСД оценивается по интервалам NN, полученным из ППГ, в отличие от ЭКГ.
- Для продвинутых пользователей выполните все описанные выше действия с помощью следующей команды:
eeglab; закрывать; % Запуск EEGLAB без графического интерфейса
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('имя файла','dataset.set','filepath',
fullfile(main_path, 'sample_data')); %Загрузить образец набора данных
ЭЭГ = brainbeats_process(ЭЭГ,'анализ',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. Извлекайте сердечные артефакты из сигналов ЭЭГ.
- Загрузите образец набора данных (см. шаг 1.5.).
- Откройте главное окно графического интерфейса, нажав Инструменты > BrainBeats >1-м уровне (тематический уровень) и выберите тип анализа Извлечь артефакты сердца из сигналов ЭЭГ, тип сигнала сердца — ЭКГ и выберите ЭКГ в списке меток электродов. Нажмите OK.
- Во2-м окне графического интерфейса отображаются параметры предварительной обработки. Установите шум линии электропередачи на частоту 50 Гц (Европа), при необходимости отредактируйте уровень достоверности, установите флажок Режим усиления (бета) и нажмите OK , чтобы запустить с параметрами по умолчанию, поскольку сигналы ЭЭГ из примера набора данных не обрабатываются заранее.
ПРИМЕЧАНИЕ: уровень достоверности для обнаружения компонентов сердца по умолчанию установлен на уровне 80%, что может быть слишком низким или слишком высоким для некоторых наборов данных. Увеличение этого значения увеличит шансы обнаружения компонентов сердца, но снизит надежность этого обнаружения. Режим Boost (бета) является опциональным и направлен на улучшение характеристик классификации путем размазывания сердечного сигнала в сигналы ЭЭГ. - Появится предупреждающее сообщение с просьбой подтвердить удаление обнаруженного дополнительного канала PPG. Этого следовало ожидать, так как инструментарий не предназначен для одновременного анализа ЭКГ и ФПГ (или других вспомогательных каналов) и сохранение его в наборе данных приведет к серьезным ошибкам (например, удалению артефактов, плохому разложению ICA и т. д.). Нажмите кнопку Да.
- Сигнал ЭКГ фильтруется полосой пропускания для удаления медленных дрейфов частот ниже 1 Гц и высокочастотных шумов выше 20 Гц (с помощью непричинного фильтра нулевой фазы КИХ). Предварительная обработка сигналов ЭЭГ, как показано на шаге 3.4.
- Выполнение независимого компонентного анализа (ICA) с использованием предварительно подготовленного алгоритма ICA для реальных данных (PICARD). Чтобы изменить эту опцию, выберите стандартный алгоритм Infomax или модифицированный алгоритм Infomax для репликации из графического интерфейса пользователя на шаге 4.3. Затем автоматически классифицируйте независимые компоненты с помощью ICLabel. Если компонент классифицируется как компонент сердца с 80% достоверностью, он по умолчанию обнаруживается и извлекается автоматически из данных ЭЭГ.
- Оставьте поле Визуализировать выходы в первом основном графическом интерфейсе пользователя (шаг 4.2.), чтобы визуализировать топографию кожи головы удаленного компонента (рисунок 17 слева) и окончательный временной ряд ЭЭГ (синим цветом, рисунок 17 справа) после извлечения сердечного компонента (красным, рисунок 17 справа).
ПРИМЕЧАНИЕ: канал ЭКГ сохраняется для визуализации с целью подтверждения извлечения компонентов, связанных с ЭКГ, но после этого шага он удаляется, так как больше не содержит никакой релевантной информации. - Для опытных пользователей выполните следующие действия с помощью следующих командных строк:
eeglab; закрывать; % Запуск EEGLAB без графического интерфейса
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('имя файла','dataset.set','filepath',
fullfile(main_path, 'sample_data'));
ЭЭГ = brainbeats_process(ЭЭГ,'анализ','rm_heart',
'heart_signal','ЭКГ',...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true);
Результаты
Во-первых, плагин BrainBeats использовался для предварительной обработки данных ЭЭГ и ЭКГ, выявления и удаления артефактов, а также анализа вызванных сердцебиением потенциалов (HEP) и колебаний (HEO). BrainBeats успешно обнаружил интервалы RR по сигналу ЭКГ и некоторые артефакты RR (рис. 2
Обсуждение
Критические шаги в протоколе
Критические шаги описаны в шагах 1.1-1.4. Предупреждения и сообщения об ошибках реализованы в различных местах набора инструментов, чтобы помочь пользователям понять, почему они могут столкнуться с проблемами (например, расположение электродов не ?...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Институт ноэтических наук поддержал это исследование. Мы благодарим разработчиков оригинальных алгоритмов с открытым исходным кодом, которые были адаптированы для разработки некоторых алгоритмов BrainBeats.
Материалы
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Ссылки
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены