É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
BrainBeats como um plug-in EEGLAB de código aberto para analisar conjuntamente sinais cardiovasculares e de EEG
Neste Artigo
Resumo
A caixa de ferramentas BrainBeats é um plug-in EEGLAB de código aberto projetado para analisar em conjunto sinais de EEG e cardiovasculares (ECG/PPG). Inclui avaliação de potenciais evocados por batimentos cardíacos (HEP), análise baseada em recursos e extração de artefatos cardíacos de sinais de EEG. O protocolo ajudará a estudar a interação cérebro-coração através de duas lentes (HEP e recursos), aumentando a reprodutibilidade e acessibilidade.
Resumo
A interação entre o cérebro e os sistemas cardiovasculares está atraindo cada vez mais atenção por seu potencial de avançar nossa compreensão da fisiologia humana e melhorar os resultados de saúde. No entanto, a análise multimodal desses sinais é desafiadora devido à falta de diretrizes, processamento de sinais padronizado e ferramentas estatísticas, interfaces gráficas de usuário (GUIs) e automação para processar grandes conjuntos de dados ou aumentar a reprodutibilidade. Existe uma lacuna adicional nos métodos padronizados de extração de recursos de EEG e variabilidade da frequência cardíaca (VFC), prejudicando o diagnóstico clínico ou a robustez dos modelos de aprendizado de máquina (ML). Em resposta a essas limitações, apresentamos a caixa de ferramentas BrainBeats. Implementado como um plug-in EEGLAB de código aberto, o BrainBeats integra três protocolos principais: 1) Potenciais evocados por batimentos cardíacos (HEP) e oscilações (HEO) para avaliar a interação cérebro-coração bloqueada no tempo com precisão de milissegundos; 2) Extração de recursos de EEG e VFC para examinar associações/diferenças entre várias métricas cerebrais e cardíacas ou para construir modelos robustos de ML baseados em recursos; 3) Extração automatizada de artefatos cardíacos de sinais de EEG para remover qualquer contaminação cardiovascular potencial durante a realização de análises de EEG. Fornecemos um tutorial passo a passo para aplicar esses três métodos a um conjunto de dados de código aberto contendo sinais simultâneos de EEG, ECG e PPG de 64 canais. Os usuários podem ajustar facilmente os parâmetros para adaptar suas necessidades exclusivas de pesquisa usando a interface gráfica do usuário (GUI) ou a linha de comando. O BrainBeats deve tornar a pesquisa de interação cérebro-coração mais acessível e reprodutível.
Introdução
Por muito tempo, a abordagem reducionista dominou a investigação científica em fisiologia e cognição humanas. Essa abordagem envolveu a dissecação de processos corporais e mentais complexos em componentes menores e mais gerenciáveis, permitindo que os pesquisadores se concentrassem em sistemas individuais isoladamente. Essa estratégia surgiu devido aos desafios em estudar a natureza intrincada e interconectada do corpo e da mente humanos1. O reducionismo tem sido fundamental para a compreensão de subsistemas individuais isoladamente, como elucidar o papel dos canais iônicos e potenciais de ação para a comunicação neural2 ou cardíaca3 . No entanto, uma lacuna significativa permanece em nossa compreensão de como esses sistemas isolados interagem em uma escala espacial e temporal maior. A estrutura multimodal (integrativa ou ecológica) considera o corpo humano um sistema multidimensional complexo, onde a mente é vista não como um produto do cérebro, mas como uma atividade do ser vivo, uma atividade que integra o cérebro nas funções cotidianas do corpo humano4. As abordagens multimodais e reducionistas não são exclusivas, assim como não podemos estudar um neurônio sem todo o cérebro ou todo o cérebro sem entender as propriedades individuais dos neurônios. Juntos, eles abrem caminho para uma compreensão mais abrangente e sinérgica da saúde humana, patologia, cognição, psicologia e consciência. O presente método visa facilitar a investigação multimodal da interação entre o cérebro e o coração, fornecendo análise conjunta de eletroencefalografia (EEG) e sinais cardiovasculares, ou seja, eletrocardiografia (ECG) e fotopletismografia (PPG). Esta caixa de ferramentas, implementada como um plugin EEGLAB no MATLAB, aborda as limitações metodológicas existentes e é feita de código aberto para facilitar a acessibilidade e reprodutibilidade na área científica. Ele implementa as diretrizes e recomendações mais recentes em seu design e parâmetros padrão para incentivar os usuários a seguir as práticas recomendadas conhecidas. A caixa de ferramentas proposta deve ser um recurso valioso para pesquisadores e médicos interessados em 1) estudar potenciais evocados por batimentos cardíacos, 2) extrair características de sinais de EEG e ECG / PPG ou 3) remover artefatos cardíacos de sinais de EEG.
Pesquisa coração-cérebro
A relação entre o coração e o cérebro tem sido historicamente estudada por meio de métodos de neuroimagem, como ressonância magnética funcional (fMRI) e tomografia por emissão de pósitrons (PET). Usando essas ferramentas, os pesquisadores destacaram algumas regiões cerebrais associadas ao controle cardiovascular (por exemplo, manipulação da frequência cardíaca e da pressão arterial5), mostraram a influência da frequência cardíaca no sinal BOLD6 ou identificaram possíveis vias cérebro-corpo que contribuem para a doença cardíaca coronária (ou seja, pressão arterial evocada por estresse7). Embora esses estudos tenham avançado significativamente nossa compreensão da complexa interação entre o sistema nervoso central (SNC) e a função cardiovascular, essas técnicas de neuroimagem são caras, têm disponibilidade limitada e estão confinadas a ambientes laboratoriais controlados, o que restringe sua praticidade para aplicações no mundo real e em larga escala.
Em contraste, EEG e ECG / PPG são ferramentas mais acessíveis e portáteis que oferecem o potencial para estudar as interações cérebro-coração em ambientes e populações mais diversos ou por períodos mais longos, proporcionando novas oportunidades. O ECG mede os sinais elétricos gerados por cada batimento cardíaco quando o coração se contrai e relaxa por meio de eletrodos colocados na pele (geralmente no tórax ou nos braços)8. O PPG mede as alterações do volume sanguíneo nos tecidos microvasculares (ou seja, fluxo sanguíneo e pulsação) usando uma fonte de luz (por exemplo, LED) e um fotodetector (comumente colocado na ponta do dedo, pulso ou testa), dependendo de como o sangue absorve mais luz do que o tecido circundante9. Ambos os métodos fornecem informações valiosas sobre a função cardiovascular, mas servem a propósitos diferentes e oferecem tipos de dados distintos. Assim como o ECG, o EEG registra os campos elétricos gerados pela atividade sincronizada de milhares de neurônios corticais que se propagam pela matriz extracelular, tecidos, crânio e couro cabeludo até atingirem os eletrodos colocados na superfície do couro cabeludo10. Como tal, o uso de EEG e ECG / PPG é uma grande promessa para avançar nossa compreensão dos processos fisiológicos, cognitivos e emocionais subjacentes às interações cérebro-coração e suas implicações para a saúde e o bem-estar humanos. Portanto, capturar a interação coração-cérebro a partir de sinais de EEG, ECG / PPG com a caixa de ferramentas BrainBeats pode ser particularmente útil para as seguintes áreas científicas: diagnóstico clínico e previsão, aprendizado de máquina de big data (ML), automonitoramento do mundo real11 e imagens móveis do cérebro / corpo (MoBI) 12 , 13 .
Duas abordagens para analisar conjuntamente sinais de EEG e ECG
Existem duas abordagens principais para estudar as interações entre EEG e sinais cardiovasculares:
Os potenciais evocados por batimentos cardíacos (HEP) no domínio do tempo: potenciais relacionados a eventos (ERP) e as oscilações evocadas por batimentos cardíacos (HEO) no domínio da frequência de tempo: perturbações espectrais relacionadas a eventos (ERSP) e coerência entre tentativas (ITC). Essa abordagem examina como o cérebro processa cada batimento cardíaco. Com precisão de milissegundos (ms), esse método requer que ambas as séries temporais estejam perfeitamente sincronizadas e que os batimentos cardíacos sejam marcados nos sinais de EEG. Essa abordagem ganhou interesse nos últimos anos 14,15,16,17,18,19.
Abordagem baseada em recursos: esta abordagem extrai características de EEG e variabilidade da frequência cardíaca (VFC) de sinais contínuos e examina associações entre eles. Isso foi feito independentemente para EEG (muitas vezes denominado EEG quantitativo ou qEEG20), ECG 21,22,23 e PPG 24,25,26. Essa abordagem apresenta aplicações promissoras ao capturar variáveis relacionadas ao estado e às características. Ressalta-se que, tanto para o EEG quanto para os sinais cardiovasculares, quanto maior o registro, mais dominante é a variável traço 27,28,29. Assim, as aplicações dependem dos parâmetros de gravação. As análises baseadas em recursos estão ganhando interesse crescente, fornecendo novas métricas quantitativas para prever o desenvolvimento de transtornos mentais e neurológicos, resposta ao tratamento ou recaída 30,31,32,33,34,35. Essa abordagem é especialmente atraente com conjuntos de dados grandes e do mundo real (por exemplo, clínica, monitoramento remoto), que podem ser obtidos mais facilmente graças às recentes inovações em neurotecnologia vestível11. Uma aplicação menos explorada é a identificação de associações entre características específicas do cérebro e do coração, destacando a dinâmica subjacente do sistema nervoso central. A variabilidade da frequência cardíaca (VFC) pode ser calculada a partir de sinais de ECG e PPG. Ele fornece informações sobre o sistema nervoso autônomo (SNA) medindo as variações nos intervalos de tempo entre os batimentos cardíacos (ou seja, os intervalos normais a normais)27. O aumento da atividade simpática (SNS) (por exemplo, durante o estresse ou exercício) normalmente reduz a VFC, enquanto a atividade parassimpática (PNS) (por exemplo, durante o relaxamento) a aumenta. Uma frequência respiratória mais lenta geralmente aumenta a VFC devido à atividade aprimorada do SNP, especialmente para gravações curtas (<10 min)27. Pontuações mais altas de VFC geralmente sugerem um SNA mais resiliente e adaptável, enquanto uma VFC mais baixa pode indicar estresse, fadiga ou problemas de saúde subjacentes. Registros longos de VFC (ou seja, pelo menos 24 h) fornecem um prognóstico preditivo para várias condições de saúde, incluindo doenças cardiovasculares, estresse, ansiedade e algumas condições neurológicas27. Medidas como pressão arterial, frequência cardíaca ou níveis de colesterol fornecem informações sobre o estado do sistema cardiovascular. Em contraste, a VFC adiciona um aspecto dinâmico, mostrando como o coração responde e se recupera do estresse.
Vantagens do BrainBeats sobre os métodos existentes
Embora existam ferramentas, conforme revisado abaixo, para processar sinais cardiovasculares e de EEG independentemente um do outro, eles não podem ser analisados em conjunto. Além disso, a maioria dos meios disponíveis para processar sinais cardiovasculares envolve licenciamento caro, não permite processamento automatizado (especialmente benéfico para grandes conjuntos de dados), possui algoritmos proprietários que impedem a transparência e a reprodutibilidade ou requer habilidades avançadas de programação por não fornecer uma interface gráfica do usuário (GUI)36. Até onde sabemos, quatro caixas de ferramentas MATLAB de código aberto suportam análise HEP/HEO com uma GUI: a caixa de ferramentas ecg-kit37, o pipeline BeMoBIL38, o plug-in HEPLAB EEGLAB39 e a caixa de ferramentas CARE-rCortex40. Embora o HEPLAB, o BeMoBIL e o kit de ecg facilitem a análise HEP detectando batimentos cardíacos e marcando-os nos sinais de EEG, eles não fornecem análise estatística ou estão limitados ao domínio do tempo (ou seja, HEP). O plug-in CARE-rCortex abordou esses problemas suportando ECG e sinais respiratórios, análise no domínio da frequência de tempo, estatísticas e métodos avançados de normalização e correção de linha de base adaptados à análise HEP/HEO. No entanto, utiliza o método de Bonferroni para correção estatística do erro tipo 1 (ou seja, falsos positivos), que é muito conservador e não fisiologicamente sólido para aplicações de EEG, levando a um aumento nos erros do tipo II (ou seja, falsos negativos)41. Além disso, a caixa de ferramentas não oferece acesso à linha de comando para automação. Por fim, estudos recentes não recomendam métodos de correção basal 42,43,44, pois reduzem a relação sinal-ruído (RSR) e são estatisticamente desnecessários e indesejáveis.
Para resolver essas limitações, apresentamos a caixa de ferramentas BrainBeats, atualmente implementada como um plugin EEGLAB de código aberto no ambiente MATLAB. Ele incorpora as seguintes vantagens em relação aos métodos anteriores:
1) Uma GUI fácil de usar e recursos de linha de comando (para programadores que desejam realizar processamento automatizado). 2) Algoritmos, parâmetros e diretrizes validados para o processamento de sinais cardiovasculares, como detecção de picos de R, interpolação de artefatos de RR e cálculo de métricas de VFC (por exemplo, implantação de diretrizes para janelas, reamostragem, normalização, etc.27,45,46). Isso é importante porque Vest et al. demonstraram como diferenças modestas nessas etapas de processamento podem levar a resultados divergentes, contribuindo para a falta de reprodutibilidade e aplicabilidade clínica das métricas de VFC46. 3) Algoritmos validados, parâmetros padrão e diretrizes para processamento de sinais de EEG, incluindo filtragem e janelamento44,47, re-referenciamento48,49, remoção de canais e artefatos anormais 50,51,52, decomposição otimizada de ICA e classificação de componentes independentes 53,54,55,56. Os usuários podem ajustar todos os parâmetros de pré-processamento ou até mesmo pré-processar seus dados de EEG com seu método preferido antes de usar a caixa de ferramentas para atender às suas necessidades (por exemplo, com o plug-in EEGLAB clean_rawdata50,52, o pipeline BeMoBIL38, o pipeline PREP57, etc.). 4) Potenciais evocados por batimentos cardíacos (HEP, ou seja, domínio do tempo) e oscilações (HEO; perturbações espectrais relacionadas a eventos com métodos wavelet ou FFT e coerência entre tentativas estão disponíveis através do software EEGLAB padrão) a partir de sinais de ECG. Estatísticas paramétricas e não paramétricas com correções para erros do tipo 1 estão disponíveis por meio do software padrão da EEGLAB. As estatísticas não paramétricas incluem estatísticas de permutação e correções espaço-temporais para comparações múltiplas (por exemplo, agrupamento espaço-temporal ou aprimoramento de agrupamento livre de limiar) 58 , 59 . Os usuários podem usar o plug-in LIMO-EEG para implementar modelagem linear hierárquica, que leva em conta bem a variância dentro e entre os sujeitos e implementa uma abordagem univariada em massa livre de suposições com controle robusto para erros tipo I e II60,61. As análises estatísticas dos dados HEP/HEO podem ser realizadas nos domínios do canal e do componente independente. 5) Análise HEP/HEO e HRV a partir de sinais PPG (pela primeira vez para HEP/HEO). 6) Suporta a extração conjunta de recursos de EEG e HRV pela primeira vez. 7) A caixa de ferramentas fornece várias visualizações de dados para inspecionar sinais em várias etapas de processamento necessárias e saídas no nível do assunto.
| Método | Detectar picos R do ECG | Detectar ondas R de PPG | HEP/HEO | Características do EEG e da VFC | Remova artefatos cardíacos do EEG | GUI | Linha de comando |
| kit de ecg | X | X | X | X | |||
| Bônus | X | X | X | ||||
| HEPLAB | X | X | X | X | |||
| CARE-rCortex | X | X | X | X | |||
| Batidas cerebrais | X | X | X | X | X | X | X |
TABELA 1: Novidades trazidas pelo BrainBeats em relação a métodos semelhantes pré-existentes.
Informações para ajudar os leitores a decidir se o método é apropriado para eles
Esta caixa de ferramentas é apropriada para qualquer pesquisador ou clínico com dados de EEG e ECG/PPG. O plug-in ainda não suporta a importação de sinais de EEG e ECG/PPG de arquivos separados (embora esse recurso esteja disponível em breve). A caixa de ferramentas é apropriada para qualquer pessoa que pretenda realizar análises HEP/HEO, extrair características de EEG e/ou VFC com métodos padronizados ou simplesmente remover artefatos cardíacos dos sinais de EEG. Consulte a Figura 1 para obter um diagrama de blocos resumindo o fluxo e os métodos gerais do BrainBeats.
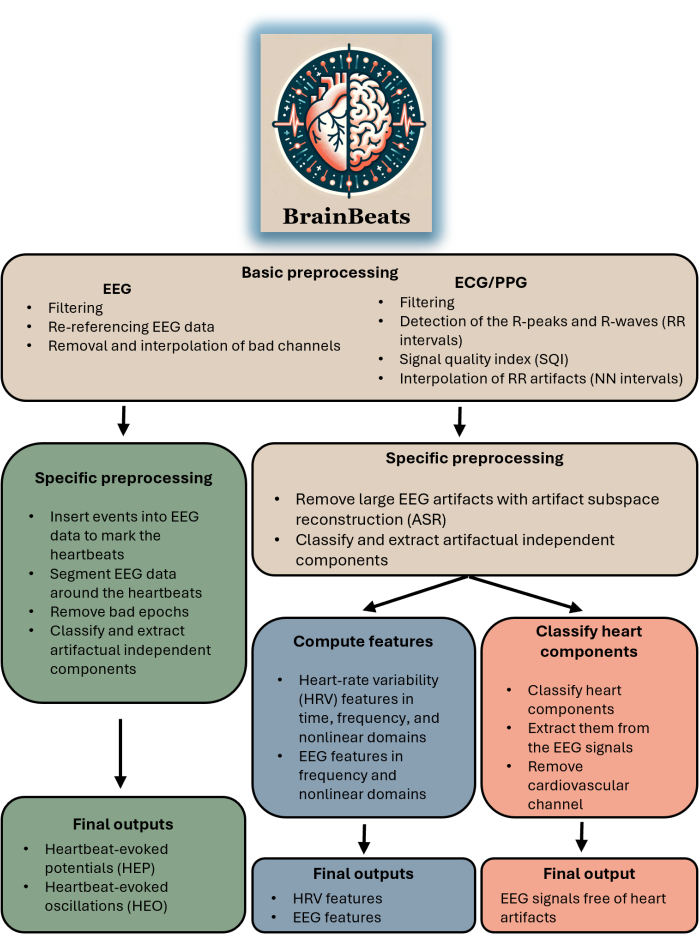
FIGURA 1. Diagrama de blocos resumindo a arquitetura e o fluxo geral do BrainBeats. As operações comuns entre os três métodos são marrons. As operações específicas para potenciais evocados por batimentos cardíacos (HEP) e oscilações (HEO) são verdes. As operações específicas para a extração de características de EEG e VFC são azuis. As operações específicas para remover artefatos cardíacos dos sinais de EEG são vermelhas. Clique aqui para ver uma versão maior desta figura.
Protocolo
O consentimento informado foi obtido de cada participante, e o comitê de ética da Universidade Federal dos Urais aprovou o protocolo experimental.
1. Requisitos do BrainBeats
- Instale o MATLAB e o EEGLAB no computador. O EEGLAB pode ser baixado em https://github.com/sccn/eeglab e descompactado (ou clonado para usuários do Git) em qualquer lugar do computador. Consulte a página do GitHub para obter mais detalhes sobre a instalação.
- Adicione o caminho para a pasta EEGLAB no painel inicial do MATLAB clicando no botão Definir caminho . Selecione a pasta eeglab com o arquivo descompactado e clique em Salvar > Fechar.
- Inicie o EEGLAB digitando eeglab na janela de comando do MATLAB.
- Instale o plug-in do BrainBeats clicando em Arquivo > Gerenciar extensões EEGLAB. Digite brainbeats na barra de pesquisa, selecione o plug-in BrainBeats na lista e clique em Instalar/Atualizar.
- Carregue o conjunto de dados de amostra no EEGLAB. Clique em Arquivo > Carregar dados existentes. Navegue até a pasta EEGLAB, vá para a pasta plugins, vá para a pasta BrainBeats e abra para a pasta sample_data. Selecione o arquivo dataset.set.
NOTA: Este conjunto de dados corresponde ao sub-032 (estado de repouso com os olhos abertos) de um conjunto de dados multissujeito de código aberto62,63. Este foi selecionado porque contém EEG simultâneo (64 canais), ECG (um canal) e PPG (um canal), a uma taxa de amostragem de 1000 Hz. Os sinais de ECG e PPG foram coletados usando as entradas auxiliares do sistema actiCHamp. Os dados de EEG foram registrados com eletrodos ativos posicionados de acordo com o sistema 10-20, com FCz como referência on-line e Fpz como eletrodo terra, e com a impedância mantida abaixo de 25 kOm. O ECG foi registrado usando um eletrodo ativo colocado no punho direito, o eletrodo de referência no punho esquerdo e o solo no antebraço interno esquerdo a 3 cm distalmente do cotovelo. O PPG foi registrado a partir do dedo indicador esquerdo. Os dados de EEG, ECG e PPG são sincronizados no tempo, pois foram gravados simultaneamente com o mesmo amplificador. Consulte as referências62,63 para obter mais detalhes. - Marque a caixa de seleção Salvar saídas para salvar tudo no arquivo .set correspondente no mesmo local do arquivo original que foi carregado no EEGLAB.
NOTA: Para este tutorial, mesclamos os dados de EEG, ECG e PPG em um conjunto de dados EEGLAB, carregamos as coordenadas de eletrodo do método de elemento de contorno 3D (BEM) e reduzimos a amostragem do sinal para 250 Hz para reduzir o peso do arquivo (para download do usuário) e acelerar o tempo de computação. Como esse conjunto de dados não tinha canal de EEG anormal, modificamos artificialmente o canal TP9 para ilustrar o algoritmo de detecção e remoção de canais ruins do BrainBeats. Da mesma forma, simulamos artificialmente um grande artefato de EEG no início do arquivo e um artefato semelhante a um músculo de alta frequência nos canais temporais de 3 a 6 s para ilustrar o recurso de remoção de artefatos do BrainBeats.
2. Potenciais evocados por batimentos cardíacos (HEP) e oscilações (HEO)
- Abra a primeira interface de usuário (GUI) geral do BrainBeats para selecionar os parâmetros principais. No EEGLAB, clique em Ferramentas > BrainBeats >1º nível (nível de assunto). Selecione Potenciais evocados por batimentos cardíacos HEP como a análise a ser executada, ECG como o tipo de dados cardíacos e clique no botão para exibir a lista de canais para selecionar o canal de ECG rotulado ECG (ou digite-o diretamente na caixa de texto). Mantenha as opções Visualizar saídas (recomendado) e Salvar saídas selecionadas e clique em Ok.
- Uma segunda janela GUI aparece com base nas escolhas anteriores (ou seja, análise HEP com dados de ECG). Selecione os parâmetros de pré-processamento para sinais de EEG e ECG. Desative o pré-processamento desmarcando as caixas Pré-processar ECG e Pré-processar EEG para ver se os dados já foram pré-processados antes de iniciar o BrainBeats. Altere o ruído da linha de energia para 50 Hz na seção EEG de pré-processamento , pois esses dados de EEG foram registrados na Rússia. Clique em OK para iniciar.
- Uma mensagem de aviso aparecerá, solicitando confirmação para remover o canal PPG que foi detectado. Isso ocorre porque a caixa de ferramentas não foi projetada para analisar ECG e PPG simultaneamente (ou outros canais auxiliares) e mantê-la no conjunto de dados levará a erros graves (por exemplo, remoção de artefatos, decomposição ruim de ICA, etc.). Clique em Sim. O BrainBeats começa a realizar algumas verificações, definindo alguns parâmetros padrão e separa o ECG dos dados do EEG para pré-processar o sinal de ECG e calcular os intervalos RR.
- As séries temporais de ECG e RR são pré-processadas usando algoritmos validados da caixa de ferramentas Physionet Cardiovascular Signal46,64 adaptados para se adequar à formatação de dados do BrainBeats, aumentar a clareza, o ajuste de parâmetros e o tempo de computação (consulte as referências para validação dos algoritmos). O plug-in gera os intervalos RR, carimbos de data/hora, sinal de ECG filtrado, índices de picos R e frequência cardíaca (FC). Ajuste esses parâmetros por meio da GUI ou da linha de comando.
NOTA: O sinal de ECG é filtrado por passagem de banda usando um filtro personalizado e validado (1-30 Hz) e varre o sinal para identificar o complexo QRS e os picos R usando o método Pan-Tompkins (P & T)65, implementando algumas operações de processamento de sinal, incluindo diferenciação, quadratura, integração e suavização para melhor desempenho. O limite de energia P&T é estimado com base na taxa de amostragem e nos valores de ECG suavizados para evitar interrupções causadas por grandes saliências. Se a variabilidade do intervalo RR exceder 1,5 vezes a mediana, ele procura picos perdidos. O sinal médio do pico R é calculado em segmentos de 30 s, e os pontos de pico são refinados por meio de uma verificação do período refratário, gerenciando as condições de linha plana e garantindo uma detecção consistente. - Em seguida, o BrainBeats identifica intervalos RR anormais ou picos dentro dos intervalos RR usando uma pesquisa para frente e para trás e limiares fisiológicos. O índice de qualidade do sinal (SQI) é calculado46; verifique isso se o sistema exibir avisos em que mais de 20% da série temporal RR contém artefatos RR (fora dos limites fisiológicos ou com um SQI abaixo de 0,9). Um gráfico exibe o sinal de ECG filtrado, os picos R identificados, os intervalos NN e os artefatos interpolados (consulte a Figura 2).
NOTA: Os artefatos RR são interpolados por padrão usando o método cúbico por partes com preservação de forma para obter os intervalos normais a normais (NN), mas removê-los (não recomendado) ou usar outro método de interpolação (linear, cúbico, vizinho mais próximo, vizinho anterior/próximo, spline, convolução cúbica ou Akima cúbico modificado), se necessário. Quando vários canais de ECG estão presentes, os intervalos RR são estimados para cada um e o canal com o menor número de artefatos RR é selecionado para as etapas a seguir. - Percorra as janelas ampliadas de 30 s dos picos R para uma inspeção mais detalhada pressionando as setas para a direita/esquerda. Se os dados contiverem vários canais de ECG/PPG, use o canal com o melhor índice de qualidade de sinal para os intervalos RR. O BrainBeats não suporta sinais de ECG e PPG simultaneamente no momento. Para fins de tutorial, escolhemos um conjunto de dados de amostra que contém os dois tipos de dados.
- Uma vez feito com o sinal de ECG, a passagem de banda do BrainBeats filtra os dados de EEG em 1-40 Hz usando um filtro FIR de fase mínima causal não linear por padrão para reduzir a atividade de esfregaço entre os períodos pré e pós-batimento cardíaco, preservar a causalidade e evitar atrasos indesejados do grupo44. Isso é particularmente importante para usuários que examinam o período pré-batimento cardíaco. Se o filtro passa-baixa estiver definido para um valor acima da frequência da linha de energia (por exemplo, passa-baixa de 80 Hz com frequência da linha de energia em 50 Hz), use um filtro de entalhe agudo para remover o artefato de ruído da linha. Os dados de EEG são então referenciados novamente ao infinito usando o algoritmo REST (mais adequado para análise HEP49), a menos que menos de 30 canais sejam detectados (caso em que eles não podem ser referenciados de forma confiável e um aviso é gerado para informar os usuários).
- O BrainBeats então detecta, remove e interpola canais de EEG anormais (Figura 3). Verifique se os parâmetros padrão são linhas planas maiores que 5 s (algoritmo clean_flatlines), um desvio padrão máximo de ruído de alta frequência de 10, um comprimento de janela de 5 s (para capturar melhor artefatos de baixa frequência52), uma correlação mínima entre canais vizinhos de 65 e uma porção máxima tolerada de 33% (clean_channels algoritmo).
NOTA: O número de amostras RANSAC é definido como 500 por padrão para aumentar a convergência e a replicabilidade da rejeição de canal inválido (embora aumente o tempo de computação). - Em seguida, insira R-peaks como marcadores de eventos nos dados de EEG para marcar cada batimento cardíaco e segmentar os dados em torno desses marcadores sem remoção da linha de base (de acordo com as diretrizes43,66; Figura 4). Como os intervalos NN têm comprimentos diferentes e o EEG deve ser segmentado em um comprimento constante, estime o limite mínimo do tamanho da época após eventos de pico R usando o5º percentil dos dados do intervalo interbatido (IBI) (ou seja, o valor abaixo do qual 5% dos IBIs mais curtos caem, exibido como uma linha vermelha tracejada em um histograma; veja a Figura 5).
NOTA: Este valor do5º percentil é um bom compromisso para preservar o maior número possível de épocas, garantindo que elas não sejam muito curtas, pois o período de interesse para a análise HEP/HEO é de 200-600 ms após o batimento cardíaco49,67. - Segmente os dados de EEG de -300 ms antes dos picos R até o valor do5º percentil pós-pico R, com o pico R no tempo 0. Rejeite épocas menores que 550 ms ou contendo mais de um pico R (o que influenciaria o ERP/ERSP), de acordo com as diretrizes49,67. Épocas contendo grandes artefatos de EEG são detectadas usando métricas de quadratura média (RMS) e relação sinal-ruído (SNR) e a função isoutlier do MATLAB (Figura 6). Épocas artificiais são removidas.
- Execute a separação cega da fonte usando o algoritmo Infomax padrão, contabilizando a classificação dos dados para melhor desempenho53. Se desejar, escolha o algoritmo de análise de componentes independentes pré-condicionado (PICARD) para computação rápida 54,54 escolhendo a opção Picard (rápido) ou o algoritmo Infomax modificado escolhendo a opção Infomax modificado (longo, mas replicável) para o método ICA de campo (embora isso envolva tempos de computação muito mais longos). Use o plug-in ICLabel56 para classificar automaticamente os CIs para extrair artefatos não cerebrais (os componentes oculares são removidos com 90% de confiança, enquanto os musculares, o ruído de linha e o ruído do canal são removidos com 99% de confiança; Figura 7).
- Mantenha a opção Visualizar saídas selecionada na primeira janela da GUI para visualizar a série temporal final do EEG, a média geral HEP (Figura 8 superior), HEP para cada batimento cardíaco (Figura 8 inferior) e as oscilações evocadas por batimentos cardíacos médios gerais (HEO; Figura 9). Examine os HEOs em termos de perturbação espectral relacionada a eventos (ERSP, ou seja, mudanças na potência do EEG entre os batimentos cardíacos; Figura 9 acima) e coerência de fase inter-ensaio (ITC, ou seja, consistência do ângulo de fase entre os batimentos cardíacos; Figura 9 abaixo).
NOTA: O ERSP é calculado usando uma wavelet padrão de 3 ciclos (com uma janela cônica de Hanning aplicada, proporção de pad de 2) e com o número de ciclos nas wavelets usadas para frequências mais altas expandindo lentamente até 20% do número de ciclos na janela FFT equivalente em sua frequência mais alta (1 menos 0.8). Isso controla as formas das janelas individuais medidas pela função e suas formas nos painéis de tempo/frequência resultantes. Uma linha de base arbitrária é removida para fins ilustrativos, e o ERSP é calculado para frequências de 7-25 Hz para capturar o efeito HEO típico descrito na literatura, ou seja, 300-450 ms pós-batimento cardíaco na banda alfa (8-13 Hz) sobre eletrodos frontocentrais17,67. As frequências mais baixas não podem ser estimadas devido ao tamanho curto da época definido pelos intervalos entre batimentos. Estatísticas não paramétricas (permutação) são aplicadas para visualizar o HEO para um valor-p de 0,05, corrigido para a taxa de descoberta falsa (FDR, ou seja, erro tipo 1 ou erro familiar). Esses gráficos são gerados para fins tutoriais ou análise de ensaios únicos. - Gráficos de pré-processamento são gerados para visualizar as diferentes etapas. Para desativá-lo, desmarque a caixa Visualizar pré-processamentos. Os dados finais de EEG (limpos e segmentados em torno dos picos R) não incluem os dados de ECG, pois isso influenciaria a análise ERP/ERSP. Para preservar o canal cardíaco na saída final, marque a caixa Manter canal cardíaco.
NOTA: Faça uma pausa aqui, se necessário, antes de processar o próximo arquivo (próxima condição ou participante). - O BrainBeats suporta a função de histórico do EEGLAB. Ao final de todas as operações, digite eegh na janela de comando do MATLAB para imprimir a linha de comando para repetir todas as etapas acima por meio de uma única linha de comando, com os parâmetros que foram selecionados manualmente na GUI, permitindo fácil automação. Encontre saídas de pré-processamento (por exemplo, índice de qualidade de sinal da série temporal cardiovascular, intervalos NN, canais de EEG removidos, segmentos e componentes, etc.) na estrutura do EEGLAB: EEG.brainbeats.preprocessings. Todos os parâmetros também são exportados em EEG.brainbeats.parameters.
- Para usuários avançados, execute todas as etapas acima com parâmetros padrão com as seguintes linhas de comando (consulte o script do tutorial no repositório do BrainBeats para obter mais opções):
eeglab; fechar; % Iniciar EEGLAB sem a GUI
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome do arquivo','dataset.set',
'caminho do arquivo',fullfile(main_path, 'sample_data')); %Carregar o conjunto de dados de amostra
EEG = brainbeats_process(EEG,'análise',
«hep»,«heart_signal»,«ECG», «heart_channels»,
{'ECG'},'clean_eeg',true); % Inicie o BrainBeats1º nível para processar o arquivo para análise HEP com parâmetros padrão - As etapas acima executaram HEP/HEO a partir do sinal de ECG. Use as etapas a seguir para o sinal PPG.
- Nas etapas a seguir, execute as mesmas operações, mas usando um sinal PPG. Carregue o mesmo conjunto de dados novamente (consulte a etapa 1.5), pois as operações anteriores o substituíram, e abra a primeira GUI do BrainBeats novamente para selecionar os parâmetros principais. Clique em Ferramentas > BrainBeats >1º nível (nível do assunto). Selecione Potenciais evocados por batimentos cardíacos (HEP) como a análise a ser executada, PPG como o tipo de dados cardíaco e clique no botão para exibir a lista de canais para selecionar o canal PPG. Mantenha as opções Visualizar saídas (recomendado) e Salvar saídas selecionadas e clique em OK.
- A segunda janela da GUI aparece de maneira semelhante à etapa 2.2 . A única diferença é que agora podemos ajustar o parâmetro para processar o sinal PPG em vez do sinal ECG. Clique em OK para executar com os parâmetros padrão.
- Uma mensagem de aviso é exibida, solicitando confirmação para remover o canal de ECG extra que foi detectado. Novamente, isso é esperado. Clique em Sim. Por padrão, a caixa de ferramentas pré-processará o sinal PPG, detectará as ondas de pulso para obter os intervalos RR, identificará artefatos RR, se houver, e os interpolará (Figura 10). Passos 2.7. para 2.12. são realizados, e os mesmos gráficos e saídas são gerados, mas com base nas ondas R detectadas a partir do sinal PPG (ver Figura 11 e Figura 12).
NOTA: As ondas R são detectadas usando a inclinação do sinal dentro de uma janela especificada. Os pulsos potenciais são então sinalizados quando a inclinação excede um limite dinâmico, que é ajustado com base no histórico de detecção e nas características do sinal. O algoritmo então procura dentro de um período de fechamento de olhos para identificar a inclinação máxima e, posteriormente, o início da onda de pulso é determinado por meio de limiar. Os picos da onda R são identificados como os vales próximos ao início e suas localizações são registradas. O algoritmo itera por todo o sinal, ajustando continuamente os limites de detecção e identificando os picos da onda R, que são usados para calcular os intervalos RR. - Para usuários avançados, execute todas as etapas acima com parâmetros padrão com as seguintes linhas de comando (consulte o script do tutorial no repositório do BrainBeats para obter mais opções):
eeglab; fechar; % Iniciar EEGLAB sem a GUI
main_path = fileparts(which('eegplugin_
BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome do arquivo','dataset.set',
'caminho do arquivo',fullfile(main_path, 'sample_data')); %Carregar o conjunto de dados de amostra
EEG = brainbeats_process(EEG,'análise',
'hep','heart_signal','PPG', 'heart_channels',
{'PPG'},'clean_eeg',true); % Inicie o BrainBeats1º nível para processar o arquivo para análise HEP com parâmetros padrão
3. Extraindo recursos de EEG e HRV
- Carregue o mesmo conjunto de dados novamente (consulte a etapa 1.5; Clique em Arquivo > Carregar conjunto de dados existente > Selecionar dataset.set) uma vez que foi substituído pelas operações anteriores e abra a GUI principal novamente para selecionar os parâmetros principais (etapa 2.1; Clique em Ferramentas > BrainBeats >1º nível). Selecione Extrair recursos de EEG e VFC para o tipo de análise, ECG para o tipo de sinal cardíaco e selecione ECG na lista de rótulos de eletrodos. Clique em OK.
- A segunda janela da GUI aparece como na etapa 2.2, mas com parâmetros diferentes para pré-processamento de EEG e extração de recursos de VFC e EEG. Defina esses parâmetros conforme descrito abaixo. Clique em OK para iniciar com os parâmetros padrão.
- Na seção VFC, clique no botão freq. opções para selecionar o método para calcular a potência da VFC (padrão definido para periodograma de Lomb-Scargle normalizado), a sobreposição da janela (padrão definido como 25%) e para realizar uma normalização de segundo nível (não definida por padrão; veja a nota abaixo para mais detalhes).
- Na seção de recursos do EEG, clique no botão freq. opções para ajustar alguns parâmetros, como a faixa de frequência geral na qual calcular a densidade espectral de potência (PSD; padrão = 1-40 Hz), as unidades (decibéis, μV2/Hz ou normalizado pela potência total), o tipo de janela (padrão = hamming), a sobreposição da janela (padrão = 50%), o comprimento da janela (padrão = 2 s), e os tipos de limites de frequência para cada banda.
NOTA: A potência da VFC é calculada por padrão usando o periodograma de Lomb-Scargle normalizado, que não requer reamostragem (portanto, preservando melhor as informações originais) e lida melhor com dados amostrados de maneira não uniforme, dados ausentes e ruído (típico com intervalos NN) 68 . A versão normalizada dimensiona a potência pela variação do sinal, fornecendo resultados menos sensíveis a níveis de ruído variáveis, mais focados na força relativa de componentes periódicos e mais comparáveis em diferentes gravações ou assuntos. Outros métodos disponíveis incluem o periodograma de Lomb-Scargle não normalizado, o método de Welch e a Transformada Rápida de Fourier (FFT). A reamostragem é realizada automaticamente para os métodos Welch e FFT para criar as séries temporais regularmente amostradas necessárias. Uma normalização de segundo nível pode ser aplicada dividindo a potência de cada banda de frequência pela potência total, fornecendo uma medida mais intuitiva da contribuição relativa de cada componente de frequência para a potência geral. Ele é desabilitado por padrão, pois só é significativo quando todas as quatro bandas estão disponíveis, exigindo pelo menos 24 h de sinal. Esses algoritmos são adaptados da caixa de ferramentas de processamento de sinais Physionet Cardiovascular46.
- A mesma mensagem de aviso é exibida, solicitando confirmação para remover o canal PPG extra que foi detectado. Novamente, isso é esperado. Clique em Sim. O BrainBeats começará a pré-processar os dados de ECG e extrair os intervalos NN de forma idêntica, como na etapa 2.4. Em seguida, ele extrai as características da variabilidade da frequência cardíaca (VFC) dos intervalos NN nos domínios de tempo (SDNN, RMSSD, pNN50), frequência (ULF, VLF, LF, HF, relação LF:HF, potência total) e não lineares (Poincaré, média de sinal retificado por fase, entropia difusa e dimensão fractal).
NOTA: O BrainBeats verifica automaticamente o comprimento do arquivo para garantir que os requisitos mínimos sejam atendidos (por exemplo, a alimentação ULF-HRV requer 24 h de dados), envia mensagens de aviso caso contrário e não exporta esses recursos para evitar estimativas não confiáveis. O BrainBeats segue diretrizes e recomendações para estimar as métricas de VFC 27,45. - O BrainBeats pré-processa os dados de EEG como na etapa 2.7. (veja a Figura 3). Artefatos grandes são detectados automaticamente nos dados contínuos usando o algoritmo de reconstrução de subespaço de artefato (ASR)50,52 (critério SD padrão definido como 30 e usando 80% da RAM disponível para aumentar a velocidade). Esses grandes artefatos são removidos dos dados do EEG (veja a Figura 13). Para ajustar esses parâmetros na GUI, selecione os campos Limite para rejeitar segmentos defeituosos com ASR e RAM disponível para usar para ASR.
NOTA: O EEG e as séries temporais cardiovasculares não precisam ser sincronizados com o tempo para o modo de recursos, pois os recursos são estimados em cada sinal separadamente. Assim, os artefatos de EEG podem ser removidos diretamente dos dados de EEG (em vermelho, Figura 13), ao contrário do modo HEP, onde as épocas contendo artefatos foram rejeitadas para ambas as séries temporais porque a sincronização de tempo com precisão ms é essencial para esse método. - Realize a ACI usando os mesmos algoritmos e parâmetros da HEP (consulte a etapa 2.11.), exceto que desta vez os componentes do coração são removidos se detectados com 99% de confiança (eles foram preservados para HEP / HEO, pois não queremos remover sinais cardíacos relevantes).
- A caixa Domínio da frequência está marcada na etapa 3.2., portanto, o BrainBeats extrai os seguintes recursos do domínio da frequência: a densidade espectral de potência média (PSD) para as bandas de frequência delta (1-3 Hz), (3-7 Hz), alfa (8-13 Hz), beta (13-30 Hz) e gama (30+ Hz), a frequência alfa individual (IAF) e a assimetria alfa em todos os pares de eletrodos (simétricos) disponíveis. Verifique esses recursos.
NOTA: O PSD convertido em decibéis (dB) facilita a comparação dos resultados entre gravações e assuntos. Os limites de frequência podem ser definidos para os limites convencionais (por exemplo, 8-13 Hz predefinidos para a banda alfa) ou para os limites individualizados, que são detectados a partir da distribuição da densidade espectral de potência para levar em conta as diferenças interindividuais69 (por exemplo, 7,3-12,6 Hz para a banda alfa). O algoritmo foi projetado para a banda alfa e não funciona tão bem para outras bandas, especialmente quando os picos não estão presentes na distribuição espectral de potência. A frequência alfa individual (IAF) é estimada usando o centro de gravidade alfa para lidar melhor com picos divididos ou picos ambíguos69. A assimetria alfa é calculada seguindo as diretrizes (janela de hamming de 2 s com 50% de sobreposição, o logaritmo da potência alfa do canal esquerdo menos o logaritmo da potência alfa do canal direito) 47 . Portanto, valores positivos indicam maior potência alfa esquerda do que direita e vice-versa. A assimetria alfa pode ser normalizada dividindo a potência alfa de cada eletrodo pela potência alfa somada em todos os eletrodos47. Os pares simétricos são obtidos usando distâncias, exigindo que os dados de EEG contenham coordenadas de eletrodos. - A caixa Domínio não linear é marcada na etapa 3.2., o BrainBeats extrai a entropia difusa e a dimensão fractal para cada canal de EEG.
NOTA: Acredita-se que as características de domínio não linear capturem dinâmicas complexas e não lineares do cérebro que são perdidas por medidas espectrais e mostram uma promessa particular para investigar interações entre vários sistemas do corpo 70,71,72. A entropia difusa é mais confiável e robusta do que suas alternativas (amostra e entropia aproximada), mas requer tempos de computação mais longos (especialmente com longas séries temporais de EEG com altas taxas de amostragem). Para resolver esse problema, quando os sinais de EEG têm mais de 2 minutos de duração com uma taxa de amostragem superior a 100 Hz, eles são automaticamente reduzidos (ou dizimados quando o fator não é um número inteiro) para 90 Hz (ou seja, correspondendo a uma frequência de Nyquist de 45 Hz, para corresponder ao filtro passa-baixa padrão e evitar artefatos de ruído de linha tanto quanto possível). Além disso, a computação paralela é ativada por padrão ao estimar os recursos do EEG, o que reduz o tempo de computação, especialmente quando muitos canais de EEG estão disponíveis. - Selecione Visualizar saídas na primeira GUI (consulte a etapa 3.1.), para gerar um gráfico exibindo a densidade espectral de potência (PSD) para dados de VFC e EEG (Figura 14) junto com topografias do couro cabeludo exibindo alguns recursos de EEG (Figura 15).
NOTA: Você também pode encontrar algumas saídas de pré-processamento em EEG.brainbeats.preprocessing e todos os parâmetros usados em EEG.brainbeats.parameters. Incentivamos os usuários a relatar esses resultados em publicações científicas para aumentar a replicabilidade das descobertas. - Mantenha a caixa Salvar saídas marcada na primeira janela da GUI para salvar todos os recursos exportados para o arquivo EEGLAB .set em EEG.brainbeats.features e salve-os em um arquivo .mat na mesma pasta em que o conjunto de dados foi carregado.
- O BrainBeats suporta a função de histórico do EEGLAB. Ao final de todas as operações, digite eegh na janela de comando do MATLAB para imprimir a linha de comandoque permitirá repetir todas as etapas acima por meio de uma única linha de comando, com os parâmetros que foram selecionados manualmente na GUI, permitindo fácil automação e replicação das operações.
- Para usuários avançados, execute todas as etapas acima com o seguinte comando:
eeglab; fechar; % Iniciar EEGLAB sem a GUI
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome do arquivo','dataset.set',
'caminho do arquivo',fullfile(main_path, 'sample_data')); % Carregar o conjunto de dados de amostra
EEG = brainbeats_process(EEG', análise',
'features','heart_signal','ECG', 'heart_channels',{'ECG'},'clean_eeg',true); - As etapas anteriores extraíram as características da VFC do sinal de ECG. Use as etapas a seguir para extrair os recursos da VFC do sinal PPG (os recursos do EEG são os mesmos).
- Carregue o mesmo conjunto de dados novamente (etapa 1.5.), pois ele foi substituído pelas operações e abra a GUI principal novamente (etapa 2.5.). Selecione Extrair recursos de EEG e VFC para a análise e selecione PPG para o tipo de sinal cardíaco e PPG para o nome do canal. Clique em OK.
- A2ª janela GUI agora mostra os parâmetros para pré-processamento de PPG e para extrair os recursos HRV e EEG. Clique em OK para executar com os parâmetros padrão. Os parâmetros são descritos na etapa 2.17.
- Uma mensagem de aviso aparecerá, solicitando confirmação para remover o canal de ECG detectado. Isso é esperado, pois a caixa de ferramentas não foi projetada para analisar ECG e PPG simultaneamente (ou outros canais auxiliares) e mantê-la no conjunto de dados levará a erros graves (por exemplo, remoção de artefatos, decomposição ruim de ICA, etc.). Clique em Sim.
- O BrainBeats pré-processa o sinal PPG e estima os intervalos NN como na etapa 2.5. Verifique os recursos extraídos da VFC dos intervalos NN, o mesmo que na etapa 3.2. exceto que os intervalos NN agora foram obtidos a partir do sinal PPG. Os sinais de EEG são pré-processados como na etapa 3.2. O BrainBeats plota o PSD (Figura 16). A única diferença aqui é o PSD estimado a partir dos intervalos NN obtidos do PPG em oposição ao ECG.
- Para usuários avançados, execute todas as etapas acima com o seguinte comando:
eeglab; fechar; % Iniciar EEGLAB sem a GUI
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome do arquivo','dataset.set','caminho do arquivo',
fullfile(main_path, 'sample_data')); %Carregar o conjunto de dados de amostra
EEG = brainbeats_process(EEG,'análise',
'features','heart_signal', 'PPG','heart_channels',{'PPG'},'clean_eeg',true);
4. Extraia artefatos cardíacos dos sinais de EEG.
- Carregue o conjunto de dados de amostra (consulte a etapa 1.5.).
- Abra a janela principal da GUI clicando em Ferramentas > BrainBeats >1º nível (nível do assunto) e selecione Extrair artefatos cardíacos de sinais de EEG para o tipo de análise, ECG para tipo de sinal cardíaco e selecione ECG na lista de rótulos de eletrodos. Clique em OK.
- A2ª janela da GUI mostra os parâmetros de pré-processamento. Defina o ruído da linha de energia para 50 Hz (Europa), edite o nível de confiança, se necessário, marque a caixa Modo de reforço (beta) e clique em OK para executar com os parâmetros padrão, pois os sinais de EEG do conjunto de dados de amostra não são pré-processados.
NOTA: o nível de confiança para detectar componentes cardíacos é definido como 80% por padrão, o que pode ser muito baixo ou muito alto para alguns conjuntos de dados. Aumentar esse valor aumentará as chances de detectar componentes cardíacos, mas diminuirá a confiabilidade dessa detecção. O modo Boost (beta) é opcional e visa melhorar o desempenho da classificação, espalhando o sinal cardíaco nos sinais de EEG. - Uma mensagem de aviso aparecerá, solicitando confirmação para remover o canal PPG extra que foi detectado. Isso é esperado, pois a caixa de ferramentas não foi projetada para analisar ECG e PPG simultaneamente (ou outros canais auxiliares) e mantê-la no conjunto de dados levará a erros graves (por exemplo, remoção de artefatos, decomposição ruim de ICA, etc.). Clique em Sim.
- O sinal de ECG é filtrado por passagem de banda para remover desvios de frequência lentos abaixo de 1 Hz e ruído de alta frequência acima de 20 Hz (com um filtro FIR de fase zero não causal). Pré-processe os sinais de EEG como na etapa 3.4.
- Realize a análise de componentes independentes (ICA) usando o algoritmo pré-condicionado ICA para dados reais (PICARD). Para alterar essa opção, escolha o algoritmo Infomax padrão ou o algoritmo Infomax modificado para replicação da GUI na etapa 4.3 . Em seguida, classifique os componentes independentes automaticamente com ICLabel. Se um componente for classificado como um componente cardíaco com 80% de confiança, ele será, por padrão, detectado e extraído automaticamente dos dados do EEG.
- Mantenha a caixa Visualize as saídas na primeira GUI principal (etapa 4.2.) para visualizar a topografia do couro cabeludo do componente removido (Figura 17 à esquerda) e a série temporal final do EEG (em azul, Figura 17 à direita) após a extração do componente cardíaco (em vermelho, Figura 17 à direita).
NOTA: o canal de ECG é preservado para visualização para confirmar a extração de componentes relacionados ao ECG, mas é removido após esta etapa, pois não contém mais nenhuma informação relevante. - Para usuários avançados, execute estas etapas usando as seguintes linhas de comando:
eeglab; fechar; % Iniciar EEGLAB sem a GUI
main_path = fileparts(which('eegplugin_BrainBeats.m')); cd(main_path);
EEG = pop_loadset('nome do arquivo','dataset.set','caminho do arquivo',
fullfile(main_path, 'sample_data'));
EEG = brainbeats_process(EEG,'análise','rm_heart',
'heart_signal','ECG', ...
'heart_channels',{'ECG'},'clean_eeg',true,'vis_cleaning',false,...
'conf_thresh',.8,'boost',true);
Resultados
Primeiro, o plug-in BrainBeats foi usado para pré-processar dados de EEG e ECG, identificar e remover artefatos e analisar potenciais evocados por batimentos cardíacos (HEP) e oscilações (HEO). O BrainBeats detectou com sucesso os intervalos RR do sinal de ECG e alguns artefatos RR (Figura 2). O BrainBeats também relatou na janela de comando que 11/305 (3,61%) dos batimentos cardíacos foram sinalizados como artefatos e interpolados. O índice médio de qualidade do sinal (SQI) dos inte...
Discussão
Etapas críticas no protocolo
As etapas críticas são descritas nas etapas 1.1-1.4. Avisos e mensagens de erro são implementados em vários locais da caixa de ferramentas para ajudar os usuários a entender por que podem encontrar problemas (por exemplo, locais de eletrodos não carregados nos dados de EEG, comprimento do arquivo muito curto para calcular uma medida confiável de VFC de frequência ultrabaixa, qualidade do sinal muito baixa para qualquer análise confiável, etc.). Cada função é ...
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
O Instituto de Ciências Noéticas apoiou esta pesquisa. Agradecemos aos desenvolvedores dos algoritmos originais de código aberto que foram adaptados para desenvolver alguns dos algoritmos do BrainBeats.
Materiais
| Name | Company | Catalog Number | Comments |
| EEGLAB | Swartz Center for Computational Neuroscience (SCCN) | Free/Open-source | |
| MATLAB | The Mathworks, Inc. | Requires a license | |
| Windows PC | Lenovo, Inc. |
Referências
- von Bertalanffy, L. . General system theory Foundations, development, applications. , (1968).
- Hodgkin, A. L., Huxley, A. F. A quantitative description of membrane current and its application to conduction and excitation in nerve. J Physiol. 117 (4), 500-544 (1952).
- Bean, B. P. Nitrendipine block of cardiac calcium channels: high-affinity binding to the inactivated state. Proc Natl Acad Sci U SA. 81 (20), 6388-6392 (1984).
- Fuchs, T. . Ecology of the brain: The phenomenology and biology of the embodied mind. , (2017).
- Napadow, V., et al. Brain correlates of autonomic modulation: Combining heart rate variability with fMRI. NeuroImage. 42 (1), 169-177 (2008).
- Chang, C., Cunningham, J. P., Glover, G. H. Influence of heart rate on the BOLD signal: The cardiac response function. NeuroImage. 44 (3), 857-869 (2009).
- Gianaros, P. J., Sheu, L. K. A review of neuroimaging studies of stressor-evoked blood pressure reactivity: Emerging evidence for a brain-body pathway to coronary heart disease risk. NeuroImage. 47 (3), 922-936 (2009).
- Burch, G. E., DePasquale, N. P. . A history of electrocardiography. No 1. , (1990).
- Allen, J. Photoplethysmography and its application in clinical physiological measurement. Physiol Meas. 28 (3), 1 (2007).
- Cohen, M. X. Where does EEG come from and what does it mean. Trends Neurosci. 40 (4), 208-218 (2017).
- Cannard, C., Brandmeyer, T., Wahbeh, H., Delorme, A. Self-health monitoring and wearable neurotechnologies. Handb Clin Neurol. 168, 207-232 (2020).
- Gramann, K., Ferris, D. P., Gwin, J., Makeig, S. Imaging natural cognition in action. Int. J. Psychophysiol. 91 (1), 22-29 (2014).
- Jungnickel, E., Gehrke, L., Klug, M., Gramann, K. Chapter 10 - MoBI-Mobile brain/body imaging. Neuroergonomics. , 59-63 (2019).
- Al, E., et al. Heart-brain interactions shape somatosensory perception and evoked potentials. Proc Natl Acad Sci. 117 (19), 10575-10584 (2020).
- Banellis, L., Cruse, D. Skipping a beat: Heartbeat-evoked potentials reflect predictions during interoceptive-exteroceptive integration. Cereb Cortex Commun. 1 (1), (2020).
- Baranauskas, M., Grabauskaitė, A., Griškova-Bulanova, I., Lataitytė-Šimkevičienė, B., Stanikūnas, R. Heartbeat evoked potentials (HEP) capture brain activity affecting subsequent heartbeat. Biomed Signal Process. Cont. 68, 102731 (2021).
- Candia-Rivera, D., et al. Neural responses to heartbeats detect residual signs of consciousness during resting state in postcomatose patients. J Neurosci. 41 (24), 5251-5262 (2021).
- Jiang, H., et al. Brain-heart interactions underlying traditional Tibetan buddhist meditation. Cereb cortex. 30 (2), 439-450 (2020).
- Kumral, D., et al. Attenuation of the heartbeat-evoked potential in patients with atrial fibrillation. JACC Clin Electrophysiol. 8 (10), 1219-1230 (2022).
- Thakor, N. V., Tong, S. Advances in quantitative electroencephalogram analysis methods. Annu Rev Biomed Eng. 6, 453-495 (2004).
- Thayer, J. F., Åhs, F., Fredrikson, M., Sollers, J. J., Wager, T. D. A meta-analysis of heart rate variability and neuroimaging studies: Implications for heart rate variability as a marker of stress and health. Neurosci Biobehav Rev. 36 (2), 747-756 (2012).
- Mather, M., Thayer, J. F. How heart rate variability affects emotion regulation brain networks. Curr Opin Beha. Sci. 19, 98-104 (2018).
- Kemp, A. H., Quintana, D. S. The relationship between mental and physical health: Insights from the study of heart rate variability. Int J Psychophysiol. 89 (3), 288-296 (2013).
- Daneshi Kohan, M., Motie Nasrabadi, A., Shamsollahi, M. B., Sharifi, A. EEG/PPG effective connectivity fusion for analyzing deception in interview. Signal Image Video Process. 14 (5), 907-914 (2020).
- Übeyli, E. D., Cvetkovic, D., Cosic, I. Analysis of human PPG, ECG and EEG signals by eigenvector methods. Digit Signal Process. 20 (3), 956-963 (2010).
- Zambrana-Vinaroz, D., Vicente-Samper, J. M., Manrique-Cordoba, J., Sabater-Navarro, J. M. Wearable epileptic seizure prediction system based on machine learning techniques using ECG, PPG and EEG signals. Sensors. 22 (23), 9372 (2022).
- Shaffer, F., Ginsberg, J. P. An overview of heart rate variability metrics and norms. Front Public Health. 5, 258 (2017).
- Coan, J. A., Allen, J. J. B. The state and trait nature of frontal EEG asymmetry in emotion. The asymmetrical brain. , 565-615 (2003).
- Hagemann, D., Hewig, J., Seifert, J., Naumann, E., Bartussek, D. The latent state-trait structure of resting EEG asymmetry: replication and extension. Psychophysiology. 42 (6), 740-752 (2005).
- Widge, A. S., et al. Electroencephalographic biomarkers for treatment response prediction in major depressive illness: A meta-analysis. Am J Psychiatry. 176 (1), 44-56 (2019).
- Olbrich, S., Arns, M. EEG biomarkers in major depressive disorder: Discriminative power and prediction of treatment response. Int Rev Psychiatry. 25 (5), 604-618 (2013).
- Kumar, Y., Dewal, M. L., Anand, R. S. Epileptic seizures detection in EEG using DWT-based ApEn and artificial neural network. Signal Image Video Process. 8, 1323-1334 (2014).
- Acharya, U. R., et al. Automated diagnosis of epileptic EEG using entropies. Biomed Signal Process Control. 7 (4), 401-408 (2012).
- de Aguiar Neto, F. S., Rosa, J. L. G. Depression biomarkers using non-invasive EEG: A review. Neurosci Biobehav Rev. 105, 83-93 (2019).
- Cannard, C., Wahbeh, H., Delorme, A. Electroencephalography correlates of well-being using a low-cost wearable system. Front Hum Neurosci. 15, 736 (2021).
- Tarvainen, M. P., Niskanen, J. P., Lipponen, J. A., Ranta-aho, P. O., Karjalainen, P. A. Kubios HRV - Heart rate variability analysis software. Comput. Methods Programs Biomed. 113 (1), 210-220 (2014).
- Demski, A. J., Soria, M. L. ECG-kit: a Matlab toolbox for cardiovascular signal processing. Journal of Open Research Software. 4 (1), e8 (2016).
- Klug, M., et al. The BeMoBIL pipeline for automated analyses of multimodal mobile brain and body imaging data. BioRxiv. , (2022).
- Perakakis, P. . HEPLAB. , (2023).
- Grosselin, F., Navarro-Sune, X., Raux, M., Similowski, T., Chavez, M. CARE-rCortex: A Matlab toolbox for the analysis of CArdio-REspiratory-related activity in the Cortex. J Neurosci Meth. 308, 309-316 (2018).
- Luck, S. J., Gaspelin, N. How to get statistically significant effects in any ERP experiment (and why you shouldn't). Psychophysiology. 54 (1), 146-157 (2017).
- Alday, P. M. How much baseline correction do we need in ERP research? Extended GLM model can replace baseline correction while lifting its limits. Psychophysiology. 56 (12), e13451 (2019).
- Delorme, A. EEG is better left alone. Sci Rep. 13 (1), 2372 (2023).
- Widmann, A., Schröger, E., Maess, B. Digital filter design for electrophysiological data - a practical approach. J Neurosci Methods. 250, 34-46 (2015).
- Pham, T., Lau, Z. J., Chen, S. H. A., Makowski, D. Heart Rate Variability in Psychology: A Review of HRV Indices and an Analysis Tutorial. Sensors. 21 (12), 3998 (2021).
- Vest, A. N., et al. An open source benchmarked toolbox for cardiovascular waveform and interval analysis. Physiol Meas. 39 (10), 105004 (2018).
- Smith, E. E., Reznik, S. J., Stewart, J. L., Allen, J. J. B. Assessing and conceptualizing frontal EEG asymmetry: An updated primer on recording, processing, analyzing, and interpreting frontal alpha asymmetry. Int J Psychophysiol Off J Int Organ Psychophysiol. 111, 98-114 (2017).
- Dong, L., et al. MATLAB toolboxes for reference electrode standardization technique (REST) of scalp EEG. Front Neurosci. 11, 601 (2017).
- Candia-Rivera, D., Catrambone, V., Valenza, G. The role of electroencephalography electrical reference in the assessment of functional brain-heart interplay: From methodology to user guidelines. J Neurosci Methods. 360, 109269 (2021).
- Mullen, T. R., et al. Real-time Neuroimaging and cognitive monitoring using wearable dry EEG. IEEE Trans Biomed Eng Spec Issue Wearable Technol. 62 (11), 2553-2567 (2015).
- Chang, C. Y., Hsu, S. H., Pion-Tonachini, L., Jung, T. P. Evaluation of artifact subspace reconstruction for automatic EEG artifact removal. 40th Ann Int Conf IEEE Eng Med Biol Soc. , 1242-1245 (2018).
- Miyakoshi, M. Artifact subspace reconstruction: a candidate for a dream solution for EEG studies, sleep or awake. Sleep. 46 (12), 241 (2023).
- Kim, H., et al. ICA's bug: How ghost ICs emerge from effective rank deficiency caused by EEG electrode interpolation and incorrect re-referencing. Front Signal Process. 3, 1064138 (2023).
- Frank, G., Makeig, S., Delorme, A. A Framework to evaluate independent component analysis applied to EEG signal: testing on the Picard algorithm. ArXiv. , (2022).
- Ablin, P., Cardoso, J. F., Gramfort, A. Faster independent component analysis by preconditioning with Hessian approximations. IEEE Trans Signal Process. 66 (15), 4040-4049 (2018).
- Pion-Tonachini, L., Kreutz-Delgado, K., Makeig, S. ICLabel: An automated electroencephalographic independent component classifier, dataset, and website. NeuroImage. 198, 181-197 (2019).
- Bigdely-Shamlo, N., Mullen, T., Kothe, C., Su, K. M., Robbins, K. A. The PREP pipeline: standardized preprocessing for large-scale EEG analysis. Front Neuroinformatics. 9, 12 (2015).
- Maris, E., Oostenveld, R. Nonparametric statistical testing of EEG- and MEG-data. J Neurosci Methods. 164 (1), 177-190 (2007).
- Pernet, C. R., Latinus, M., Nichols, T. E., Rousselet, G. A. Cluster-based computational methods for mass univariate analyses of event-related brain potentials/fields: A simulation study. J Neurosci Methods. 250, 85-93 (2015).
- Pernet, C. R., Chauveau, N., Gaspar, C., Rousselet, G. A. LIMO EEG: A toolbox for hierarchical LInear MOdeling of ElectroEncephaloGraphic data. Comput Intell Neurosci. 2011, 1-11 (2011).
- Pernet, C., et al. Electroencephalography robust statistical linear modelling using a single weight per trial. Aperture Neuro. 2022 (7), 51 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I., Busch, N. A. Pupillometry and electroencephalography in the digit span task. Sci. Data. 9 (1), 325 (2022).
- Pavlov, Y. G., Kasanov, D., Kosachenko, A. I., Kotyusov, A. I. EEG, pupillometry, ECG and photoplethysmography, and behavioral data in the digit span task and rest. OpenNeuro. , (2024).
- Clifford, G. . Signal processing methods for heart rate variability. , (2002).
- Pan, J., Tompkins, W. J. A real-time QRS detection algorithm. IEEE Trans. Biomed. Eng. 3, 230-236 (1985).
- Maess, B., Schröger, E., Widmann, A. Highpass filters and baseline correction in M/EEG analysis. Commentary on: "How inappropriate highpass filters can produce artefacts and incorrect conclusions in ERP studies of language and cognition. J. Neurosci. Methods. 266, 164-165 (2016).
- Park, H. D., Blanke, O. Heartbeat-evoked cortical responses: Underlying mechanisms, functional roles, and methodological considerations. NeuroImage. 197, 502-511 (2019).
- Lomb, N. R. Least-squares frequency analysis of unequally spaced data. Astrophys Space Sci. 39, 447-462 (1976).
- Corcoran, A. W., Alday, P. M., Schlesewsky, M., Bornkessel-Schlesewsky, I. Toward a reliable, automated method of individual alpha frequency (IAF) quantification. Psychophysiology. 55 (7), e13064 (2018).
- Chen, W., Zhuang, J., Yu, W., Wang, Z. Measuring complexity using FuzzyEn, ApEn, and SampEn. Med Eng Phys. 31 (1), 61-68 (2009).
- Cannard, C., Delorme, A. . An open-source EEGLAB plugin for computing entropy-based measures on MEEG signals. , (2022).
- Lau, Z. J., Pham, T., Chen, S. H. A., Makowski, D. Brain entropy, fractal dimensions and predictability: A review of complexity measures for EEG in healthy and neuropsychiatric populations. Eur J Neurosci. 56 (7), 5047-5069 (2022).
- Costa, M., Goldberger, A. L., Peng, C. -. K. Multiscale entropy analysis of biological signals. Phys Rev E Stat Nonlin Soft Matter Phys. 71 (2), 021906 (2005).
- Humeau-Heurtier, A. multiscale entropy approaches and their applications. Entropy. 22 (6), 644 (2020).
- Armañac-Julián, P., et al. Reliability of pulse photoplethysmography sensors: Coverage using different setups and body locations. Front Electron. 3, 906324 (2022).
- Catrambone, V., Greco, A., Vanello, N., Scilingo, E. P., Valenza, G. Time-resolved directional brain-heart interplay measurement through synthetic data generation models. Ann Biomed Eng. 47, 1479-1489 (2019).
- Georgieva-Tsaneva, G., Gospodinova, E., Gospodinov, M., Cheshmedzhiev, K. Portable sensor system for registration, processing and mathematical analysis of PPG signals. Appl Sci. 10 (3), 1051 (2020).
- Kim, J. H., Park, S. E., Jeung, G. W., Kim, K. S. Detection of R-peaks in ECG signal by adaptive linear neuron (ADALINE) artificial neural network. MATEC Web Conf. 54, 10001 (2016).
- Lei, R., Ling, B. W. K., Feng, P., Chen, J. Estimation of heart rate and respiratory rate from PPG signal using complementary ensemble empirical mode decomposition with both independent component analysis and non-negative matrix factorization. Sensors. 20 (11), 3238 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados