需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Research Article
通过适应性强的分子克隆课程本科生研究体验提高学生成绩
* 这些作者具有相同的贡献
摘要
适应性强的 Gibson Assembly 分子克隆模块以基于课程的本科生研究经验 (CURE) 格式为分子生物学实验室课程学生采用。对学生学习成果的评估表明,完成 CURE 后对分子克隆的理解和信心有所提高,并克隆了新型质粒用于天然产物生物合成研究。
摘要
分子生物学技术的不断进步要求定期完善分子生物学课程,以有效地为学生以现代能力进入劳动力市场做好准备。特别是,Gibson Assembly 的出现是一种高度可定制和适应性强的分子克隆技术,推动了众多研究环境中分子克隆的前景。因此,我们创建了一个 Gibson Assembly 克隆模块,用于部署在加州理工州立大学圣路易斯奥比斯波分校的分子生物学实验室课程中,并评估了该模块的学生学习成果。在该课程的三个迭代中,学生们参加了一个基于实验的独立项目,该项目涉及克隆三个独特的质粒文库,以支持天然产物生物合成的研究项目。学生接受了前后问卷调查,以评估他们对分子克隆的理解以及他们对分子生物学术语和技术的信心。学生的回答显示,在学习分子克隆概念和自我报告的对分子克隆术语和技术的信心方面都有显著增加。该模块框架可以推广到教授各种应用的 Gibson 组装,为教师提供了一个工具包,用于教授适应性强的紧急克隆技术,同时推进他们的研究项目。
引言
对本科生进行基本分子生物学概念和实验室技术培训对于他们的科学和专业发展至关重要,因为这些方法在各种研究环境中都很常见,包括学术界和工业界。因此,加州理工州立大学圣路易斯奥比斯波分校 (Cal Poly) 生物学(分子和细胞生物学专业)和生物化学专业的学生需要参加高年级分子生物学实验室课程来学习和应用这些主题 (CHEM/BIO 475)。本课程的基础课程之前已经开发,其中学生进行基于拓扑异构酶 (TOPO) 的克隆以组装由酵母互补 DNA (cDNA) 模板1 制备的含肌动蛋白的质粒。学生根据模仿真实研究假设的问题设计实验,提高他们对实验室实践和探究式学习的熟悉程度。分子生物学领域的不断进步要求相应的课程进行调整,以培养具有现代能力的学生进入劳动力市场。特别是,Gibson Assembly 的使用在科学界变得更加普遍;虽然该方法最初是为了合成人工染色体2 而建立的,但在本报告发布时,已有 5,000 多篇出版物引用了 Gibson 等人的原始工作。与传统克隆方法相比,Gibson Assembly 是独一无二的:它是高度可定制的,可以轻松连接多个线性 DNA 片段,而无需限制性位点来产生连接。因此,我们看到了改进 CHEM/BIO 475 课程的机会,以纳入现代分子克隆技术并改进基于探究的课程模式。
已经确定,学生的研究经历有助于提高概念理解、技能发展和对科学的毅力3,但并非所有本科生都有机会直接参与研究实验室。为了应对研究实验室学生能力有限的挑战,已经开发并采用基于课程的本科生研究体验 (CURE),以通过在课堂上进行真实的研究来提高科学的可及性。虽然 CURE 的实施方式各不相同,但已经建立了解决科学研究五项原则的常见做法。在精心设计的 CURE 中,学生将 1) 使用科学实践,2) 在研究项目中合作,3) 尝试取得新发现,4) 为课堂外相关的工作做出贡献,以及 5) 在实验失败的情况下重新评估和修改假设和方法4.与学生在实验室中的传统研究经历类似,CURE 已被证明可以增强学生对科学、科学技能、项目所有权以及科学、技术、工程和数学 (STEM) 的毅力的信心5。虽然之前已经报道了涉及分子克隆的 CURE 6,7,8,9,10,11,12,13,但我们不知道任何强调 Gibson Assembly 的适应性以制作真实研究质粒文库的报道。
在这里,我们报告了加州理工州立大学圣路易斯奥比斯波分校当前基于探究的 CHEM/BIO 475 课程的扩展,其中有两项主要改进:使用 Gibson Assembly 的实践经验和学生参与 CURE,该研究为美国国家科学基金会资助的研究项目(NSF-1708919 和 NSF-2300890)提供了原始质粒构建体。在该课程的三个实施过程中,学生为两个不同的研究项目做出了贡献,这些项目专注于放线菌产生的生物活性分子的天然产物生物合成。天然产物通常含有具有抗生素、抗真菌和/或抗癌活性的药效团,因此这些小分子在药物发现工作中具有重要意义,并具有潜在的临床相关性14。这项研究需要创建质粒文库,以便能够研究细菌生物合成酶的功能和工程潜力。在这个 CURE 中,学生设计并进行了 Gibson Assembly 实验,以克隆与这些研究项目相关的独特质粒文库(图 1)。此外,该模块的格式和设计非常独特,因为它很容易适应为其他研究项目生成任何感兴趣的质粒。
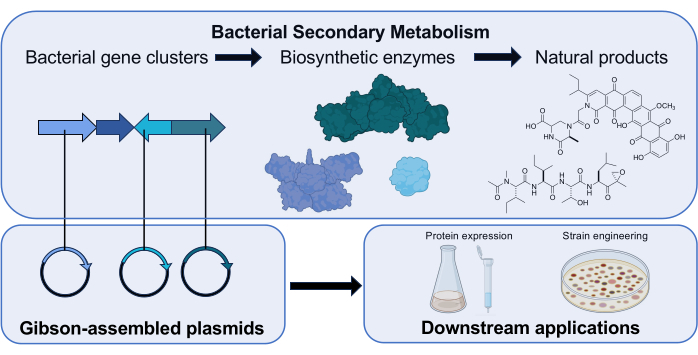
图 1:Gibson Assembly 在我们研究实验室中的作用概述。 放线菌使用编码生物合成酶的基因簇生产具有临床相关生物活性的小分子天然产物。在我们的研究中,含有生物合成基因的质粒通过 Gibson Assembly 组装,用于编码酶功能的下游研究。来自 Biorender.com 的科学图标。请单击此处查看此图的较大版本。
实验室概述
分子生物学实验室是生物化学专业和分子与细胞生物学专业的高年级课程。欢迎其他满足先决条件的学生将该课程作为高年级选修课。该课程由加州理工学院的化学和生物化学系以及生物系共同列出。两个系的教师每学期轮流教授该课程(两个季度的生物,一个季度的化学和生物化学)。
该课程每周在实验室开会两次,每次 170 分钟,每周一次,每次 50 分钟的讲座。实验部分最多包含 16 名学生,每个季度提供 2-3 个实验部分。该课程为期 10 周,期末考试在第 10 周的最后一次会议上进行。讲座时间用于讨论实验室中进行的许多实验技术背后的理论,以及实验室中未涵盖的分子生物学的当前主题。实验室的核心课程包括从酵母1 克隆肌动蛋白基因的过程,大约需要 7 周(13-14 次实验室会议)。技术包括微量移液、酵母 RNA 分离、使用逆转录聚合酶链反应 (RT-PCR) 扩增酵母基因、TOPO 克隆、蓝白斑筛选、质粒分离和通过限制性酶切和 PCR 验证插入片段、克隆 的计算机 分析和 DNA 序列分析。课程最后 3 周的课程由教师自行决定,但涉及学生完成一个结果不确定的“独立项目”。
实验概述
我们研究小组的一个重点是放线菌门的生物合成途径。在设计独立项目时,我们设想让学生通过 Gibson Assembly 创建质粒,用于我们研究天然产物生物合成的研究项目。虽然这里评估的课程模块的迭代特定于允许作生物合成途径的质粒,但 Gibson Assembly 工作流程非常适用于其他分子克隆项目(图 2)。工作流程分为三个不同的实验(A、B 和 C),分别在两个讲座时段和六个实验时段(共 3 周)内完成(参见 补充文件 1 和 补充文件 2)。实验之前有工作表,以支持学生准备和评估学生的理解(补充文件 3、 补充文件 4 和 补充文件 5)。工作流程以灵活的格式呈现,以适应教师的需求和兴趣。

图 2:Gibson Assembly 模块工作流程。 第 1 天和第 4 天是讲座时间,学生在其中完成 计算机序列 分析和实验设计。第 2-3 天和第 5-8 天是实验室会议,其中进行通过 Gibson 组装克隆新型质粒的步骤,然后进行分离和筛选。图解流程图按学生进行的三个实验(A、B 和 C)分组。更详细的说明和协议可以在补充 文件 1 和 补充文件 2 分别提供的教师手册和学生手册中找到。来自 Biorender.com 的科学图标。请单击此处查看此图的较大版本。
Gibson Assembly 独立项目模块于 2019 年春季季度在 CHEM/BIO 475 中首次试行。在 2020 年和 2021 年,由于 SARS-CoV-2 大流行,该课程改为在线授课。当 2022 年和 2023 年春季恢复面对面教学时,该课程的学生被邀请参加一项研究,评估 Gibson Assembly 独立项目的学习成果,其中将克隆原始的、与研究相关的质粒。2019 年,学生创建了一个质粒库,该质粒由来自 棘孢微单孢 菌 ATCC 15837 基因组的基因盒组成,这些基因盒被克隆到 pKC1132 中(图 3)。该质粒文库在我们的研究实验室中用于灭活天然产物 TLN-0522015 的推定生物合成基因簇中的目标基因。为了补充我们的基因失活研究,学生在 2022 年将推定的 TLN-05220 基因簇中的小型基因库克隆到 pUC19 中(图3);我们的研究小组利用这些质粒将基因亚克隆到表达载体中,包括 pET28b,用于蛋白质过表达和纯化。2023 届学生为正在进行的环氧霉素合成酶16 生物合成工程项目的工作做出了贡献。在 3-4 人的团队中,学生将非核糖体肽合成酶17 模块的工程结构域克隆到各种蛋白质表达载体中,以优化我们研究实验室中这些酶的过表达和纯化(图 3)。冗余已内置到每个队列的克隆计划中。例如,2019 年的队列包含 44 名学生,并将 15 个质粒分配给该班级进行克隆。因此,尝试克隆每个质粒 2 或 3 次。
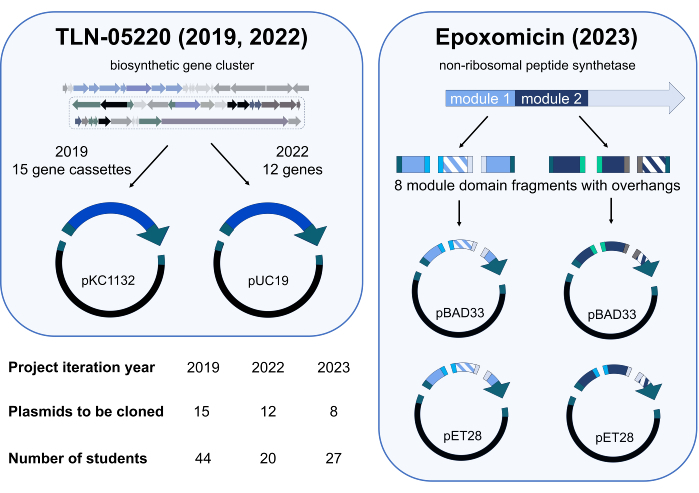
图 3:2019 年、2022 年和 2023 年独立项目迭代期间克隆的质粒和学生参与者的总结。 Gibson Assembly 项目已部署三次。在每项产品中,学生参与者克隆了不同的质粒库,用于探索生物合成途径的研究项目。2019 年和 2022 年的项目支持了我们正在进行的天然产物 TLN-0522015 的工作,该产物具有两个片段(一个基因盒或基因和一个载体)Gibson Assembly 反应。2023 年的项目涉及参与环氧霉素生物合成的非核糖体肽合成酶 (NRPS) 酶模块 1 和 2 内的结构域交换16。阴影片段表示交换结构域的两个不同突变体,纯色表示未交换的结构域。总共生成了 8 个不同的基因片段 (模块 1 4 个,模块 2 4 个),其突出端与 Gibson 组装兼容。对于每个模块,将三个基因片段的两种不同组合与两种不同载体(pBAD33 和 pET28,每个组装总共四个片段)中的一种组装在一起,以有可能产生 8 个工程化的 NRPS 质粒。来自 Biorender.com 的科学图标。请单击此处查看此图的较大版本。
评估概述
在 Gibson Assembly 独立项目开始前至少 1 周,2022 年和 2023 年的学生被邀请参加学习成果调查,其设计类似于在 CalPoly 18 的生物化学实验室课程中进行的研究。学生参与者在独立项目开始前的实验室会议上完成了多项选择题的预问卷,并在最后的实验室会议期间(即在他们完成 Gibson Assembly 模块之后)完成了多项选择后问卷。问卷前和问卷后包括 28 个相同的问题,问卷后还有两个额外的问题(共 30 个)。编写了 10 个内容问题,以评估学生对分子克隆所涉及的酶和机制(例如,聚合酶链反应 [PCR]、Gibson 组装、转化、蓝白斑筛选)的了解。以下 7 个问题要求学生自我评估他们对分子克隆术语(例如 DNA 聚合酶、核酸外切酶、连接酶)的熟悉程度。接下来的 10 个问题让学生能够自我评估他们执行分子克隆技术(例如 DNA 序列分析、限制性消化反应、琼脂糖凝胶电泳)的能力。学生们还报告了根据他们对分子克隆技术的了解,他们是否愿意从事分子生物学方面的职业。问卷后还包括两个额外的问题,供学生自我评估他们对课程中学习的承诺以及该课程是否是一次宝贵的学习经历(补充文件 6 和 补充文件 7)。来自 2022 年和 2023 年学生问卷前和问卷后回答的所有数据被合并进行分析,并可在 补充表 S1 中找到。
研究方案
涉及人类参与者的研究由加州理工学院研究机构审查委员会 (2022-113-CP (IRB)) 的人类受试者审查和批准。参与者提供了参与本研究的书面知情同意书。
以下协议概述了教师准备(步骤 1.1-1.3)、学生对三实验教学模块的作,该模块包括 PCR 以获得线性片段(步骤 2.1-2.7)、Gibson 组装、转化和克隆选择(步骤 3.1-3.5)、质粒分离和筛选(步骤 4.1-4.4)以及学习成果评估(5.1-5)5.2). 讲师准备描述了引物设计和计算机中所需质粒图谱制备的一个代表性示例。该方案的所有部分都适用于其他所需的质粒。每个学生实验分为两个 3 小时的实验室会议。
1. 教练准备
- 使用 Benchling Assembly Wizard 的 Gibson Assembly 引物设计
- 确定基因插入片段的 DNA 模板来源,例如基因组 DNA、质粒和合成 DNA。
注意: 对于将从复杂基因组 DNA 模板中扩增的基因,建议首先使用美国国家生物技术信息中心 (NCBI) 基本局部比对搜索工具 (BLAST)21 确定基因组 DNA 模板中对目标基因具有特异性的引物的独特结合位点,如 补充文件 1 中所述。对于简单的模板,如质粒或合成 DNA,请继续下一步。 - 检索基因插入片段和载体的 DNA 序列(例如,参见 补充文件 8 中作为 GenBank 文件提供的聚酮羟化酶基因序列和 pUC19 序列)。将所需的插入序列和所需的载体序列导入 Benchling,每个序列都作为新的 DNA 序列文件。打开要包含在 Gibson Assembly 反应中的每个导入序列。
注意: 可以导入多种格式的 DNA 序列文件,包括 GenBank 和 FASTA。也可以直接从带有登录号的数据库中导入序列,或者粘贴从另一个文件或查看器复制的核苷酸序列。 - 在屏幕底部,找到 Assembly Wizard 工具。单击 Assembly Wizard,然后选择 Create New Assembly。从提供的选项中,选择 Gibson 并单击 Start 开始组装。
- 在向量序列窗口中,从向量主干中选择要包含在程序集中的所有碱基。从基因插入片段 3' 端的核苷酸位置开始选择(例如,pUC19 的 657 位置),然后选择要包含的所有剩余核苷酸(例如,通过位置 656 包括整个 pUC19 序列)。选择后,单击屏幕底部的 Backbone 选项卡|Set Fragment。
注意: 在载体的 Sequence Map 和 Plasmid 视图之间切换,以选择圆形模板的所有所需碱基。或者,如果计划通过限制性酶切(而不是 PCR)制备用于 Gibson 组装的载体片段,请使用限制性内切酶切割位点(例如 XbaI)对骨架序列进行线性化。为此,请单击 剪切站点,按住 Shift 键,然后再次单击 剪切站点 。然后单击 Set Fragment 以设置主干。请注意,如果骨架在 组装向导中使用限制性内切酶线性化,则 Benchling 不会生成载体特异性引物。 - 在 插入序列 窗口中,选择要包含在装配体中的所有插入基体。选择后,单击屏幕底部的 Insert 选项卡 |设置 Fragment。
- 如果有多个基因插入片段,请单击 Assembly Wizard 右侧的 + 按钮。在插入序列窗口中,选择要包含在装配体中的所有插入基体。选择后,单击屏幕底部的 Insert 选项卡 |设置 Fragment。
- 设置完所有片段后,将组装重命名为所需的质粒名称,然后单击组装向导右侧的 Assemble。为 Sequence Folder 和 Primer Folder 选择所需的文件夹位置。单击 Create 以组装重组质粒序列。
- 打开组装的质粒,然后单击 Assembly History 以查看基本质粒图谱和衍生序列的概述。确保每个片段的核苷酸位置与所需的设计相匹配。
- 在 Assembly Parameters 选项卡中,查看为程序集设计的引物的名称。请注意,每个插入片段将设计两个引物(正向和反向),引物名称将从 DNA 序列文件的标题中得出。还要确保引物熔解温度和建议的退火温度在此窗口中兼容(参见 补充文件 8,代表性引物)。
- 如果引物熔解温度或退火温度不理想,请在引物序列文件中手动调整引物序列(例如,去除核苷酸以降低熔解温度)。或者,单击 Re-open 并点击组装向导中 Assemble 按钮旁边的 Enzyme/Primer Settings 按钮来调整引物设置。然后,重复组装步骤并重新检查引物序列。
- 如果要克隆质粒文库,并将许多不同的基因克隆到同一载体骨架中,
- 在每种情况下,对载体使用相同的引物对。为此,请从载体引物中手动去除所有核苷酸,这些引物将安装插入片段特异性的突出端。这些核苷酸位于引物的 5' 端。去除后,确保引物熔解温度和退火温度兼容(参见 补充文件 8,代表性引物)。
- 确保所有插入引物对具有非常相似的熔解温度和退火温度。
注意: 这将减少对每对退火温度优化的需要,并允许使用相同的热循环仪程序同时循环所有学生 PCR 反应。
- 所需质粒的引物设计完成后,单击 Assembly Wizard(组装向导)中的 Finalize(完成)。从 DNA 合成公司订购引物,并检索用于插入片段和载体的 DNA 模板样品。
- 使用 DNA 模板的梯度 PCR 实验,测试插入片段和载体特异性引物对的最佳退火温度。最初测试 Benchling 推荐的退火温度± 3 °C。
- 确定退火温度后,为学生制备引物溶液、DNA 模板和 PCR 反应所需试剂的等分试样。
- 确定基因插入片段的 DNA 模板来源,例如基因组 DNA、质粒和合成 DNA。
- 在实验之前,向学生提供工作表以支持准备和评估理解(补充文件 4、补充 5 和 补充 6)。
- 学习成果数据收集
注意: 如果有兴趣从 Gibson Assembly 模块收集学习成果数据并发布结果,请根据该机构的政策获得批准对人类受试者进行研究。- 邀请学生在 Gibson Assembly 独立项目开始前至少 1 周参与学习成果调查。
- 让学生参与者在独立项目开始前的实验会议上完成多项选择题预问卷(补充文件 6)。
2. 学生实验 A:PCR 获得线性片段
- 从讲师处获得用于 PCR 的引物和模板 DNA 溶液。
- 根据学生手册中的说明,移液 25 μL 反应物,包括 DNA 聚合酶 MasterMix、DNA 模板、正向和反向引物以及无核酸酶的水(参见 补充文件 2)。
- 根据 补充文件 2 中的学生手册在热循环仪中循环反应。确保反应的退火温度适合引物,并且延伸时间适合所需扩增子的长度。有关详细说明,请参阅 补充文件 2 和 补充文件 3 。
- 将 PCR 反应物储存在 4 °C 或 -20 °C 直至下一个实验室阶段。
- 通过琼脂糖凝胶电泳分析每个反应的 5 μL。当凝胶电泳时,向使用质粒 DNA 作为模板的每个反应中加入 1 μL DpnI 限制性内切酶。将此混合物在 37 °C 下孵育 1 小时。
注意: DpnI 将降解任何剩余的环状质粒 DNA 模板,使其在转化步骤中获得假阳性的可能性更小。 - 对凝胶进行成像以确认 PCR 成功并实现正确的扩增。使用市售的 PCR 纯化试剂盒纯化任何成功的 PCR 反应。使用超微量分光光度计测量纯化 PCR 产物的浓度 (ng/μL),用于后续的 Gibson 组装计算。
- 在下一次班会之前,设计 Gibson Assembly 的反应配方(参见 补充文件 2 和 补充文件 4)。
3. 学生实验 B:克隆的 Gibson 组装、转化和选择
- 根据设计的配方移液 Gibson Assembly 反应。将反应物在 50 °C 下孵育 15 分钟。在反应孵育时,通过在冰上解冻化学感受态 的大肠杆菌 细胞来准备转化。
- 通过热休克将 2 μL 的 Gibson 组件转化为化学感受态细胞。在两个选择板上吸取 100 μL 转化的细胞,并用消毒的珠子铺展。在 Luria-Bertani (LB) 琼脂上制备剩余细胞的 10-4、10-5 和 10-6 稀释液。将 100 μL 的每种连续稀释液接种在 LB(无选择)上,并用消毒的珠子铺展。
注意: 此步骤允许计算转换效率。 - 将板在 37 °C 下孵育过夜。将板在 4 °C 下储存至下一个课程。
- 对所有平板上的菌落进行计数,并按照 补充文件 2 中的说明计算转化效率。用来自任一选择性板的四个菌落重新划线一个选择性板,并将该板在 37 °C 下孵育过夜。
注意: 这用作阳性克隆的备用培养物。 - 使用记号笔,在选择板上选择、圈出并标记四个不同的菌落,并带有学生的姓名首字母和数字(例如,ABC1)。检索含有抗生素的新 LB 琼脂平板进行选择,使用标记物将平板分成象限(即四象限),然后使用每个菌落的 ~1/2 重新划线到各自标记的象限上,另外 1/2 接种 5 mL 液体 LB 培养物为选定的四个菌落中的每个象限。确保用相应的菌落身份(例如 ABC1)标记试管,并添加正确浓度的抗生素进行选择。将液体培养物在 37 °C 的振荡培养箱中孵育过夜,并将琼脂平板在 37 °C 的静态培养箱中孵育;将液体培养物和板储存在 4 °C 直至下一个课程期。
4. 学生实验 C:质粒分离与筛选
- 使用小量制备试剂盒从实验 B 的液体培养物中分离质粒 DNA。测量分离质粒的浓度(以 ng/μL 为单位)。
- 设计限制性酶切或 PCR 筛选以分析分离的质粒(参见 补充文件 2 中的详细说明和 补充文件 5 中的报告预期)。
- 移液限制性酶切反应或根据设计的配方进行反应,并在先前设计的温度和持续时间下孵育。
- 通过琼脂糖凝胶电泳分析结果。
5. 评估
- 让学生参与者在 Gibson Assembly 模块完成后完成实验会议中的多项选择后问卷(补充文件 7)。
- 合并问卷前和问卷后回答中的所有数据进行分析。
- 使用双尾配对 t 检验和 Cohen 的 d 效应大小检查问卷之间每个单独内容问题的平均分增加的统计显着性。
- 评估每个单独术语和技巧的学生置信度增加,以确定变化的程度及其统计意义。
- 通过使用标准化学习增益 (NLG) 和未配对的 t 检验,确定学生背景和学术因素对学生学习的影响。
- 通过评估平均分来评估学生对 项目经历和分子生物学职业的态度。
结果
学生克隆的成功
在 Gibson Assembly 模块的每次迭代(2019 年、2022 年和 2023 年)中,学生被要求准备一份报告,总结他们的发现。2019 年,44 名学生中有 36 名 (81.8%) 报告说,他们根据为实验 C 设计的筛选结果成功克隆了质粒。20 名学生中有 14 名 (70.0%) 报告在 2022 年成功克隆了他们指定的构建体,而 2023 年的团队项目有 27 名学生中有 12 名(44.4% 或 8 组中?...
讨论
在这里,我们提出了一个可调节的本科课堂实验室项目,在基于课程的研究环境中通过 Gibson Assembly 教授学生分子克隆。本科生在课堂环境中总共克隆了 28 个新型质粒。在三个队列中,个体学生报告的成功率从 44.4% 到 81.8% 不等,总体克隆成功率为 80%(总共 35 个分配的质粒中有 28 个成功克隆)。2023 年克隆成功率较低可能是由于两个复合因素。首先,用非常密切相关的?...
披露声明
作者声明他们没有竞争性的经济利益或其他利益冲突。
致谢
作者衷心感谢 Andrea Laubscher 的技术支持,以及 Michael Black、Sandi Clement 和 Javin Oza 对教学实验室实施和学习成果评估的有益讨论。作者衷心感谢所有在 2019 年、2022 年和 2023 年参加学习成果研究的学生,以及协助为教学实验室实施准备试剂的研究学生 Nathan Kuhn 和 Aayushi Adettiwar。作者还感谢 William 和 Linda Frost 基金、生物技术应用中心的 Chevron 生物技术应用研究捐赠基金和美国国家科学基金会(NSF-1708919 和 NSF-2300890)的资助。
材料
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
参考文献
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。