Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Research Article
Améliorer les résultats des étudiants grâce à une expérience de recherche de premier cycle basée sur un cours adaptable de clonage moléculaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Un module adaptable de clonage moléculaire Gibson Assembly a été utilisé dans le cadre d’un programme d’expérience de recherche de premier cycle (CURE) pour les étudiants du cours de biologie moléculaire. L’évaluation des résultats d’apprentissage des élèves a montré une meilleure compréhension et une meilleure confiance dans le clonage moléculaire après l’achèvement de l’étude CURE, et de nouveaux plasmides ont été clonés pour la recherche sur la biosynthèse des produits naturels.
Résumé
L’avancement continu des techniques de biologie moléculaire exige que les programmes de biologie moléculaire soient régulièrement affinés afin de préparer efficacement les étudiants à entrer sur le marché du travail avec des compétences modernes. En particulier, l’émergence de Gibson Assembly, une technique de clonage moléculaire hautement personnalisable et adaptative, a fait progresser le paysage du clonage moléculaire dans de nombreux environnements de recherche. Ainsi, nous avons créé un module de clonage d’assemblage Gibson pour le déploiement dans un cours de laboratoire de biologie moléculaire à l’Université d’État polytechnique de Californie, à San Luis Obispo, et évalué les résultats d’apprentissage des étudiants à partir du module. Au cours de trois itérations du cours, les étudiants ont participé à un projet indépendant basé sur l’expérimentation qui impliquait le clonage de trois banques de plasmides uniques pour soutenir des projets de recherche sur la biosynthèse des produits naturels. Les étudiants ont reçu des questionnaires avant et après leur compréhension du clonage moléculaire et leur confiance dans les termes et les techniques de biologie moléculaire. Les réponses des étudiants ont montré une augmentation significative à la fois de l’apprentissage des concepts de clonage moléculaire et de la confiance autodéclarée avec les termes et les techniques de clonage moléculaire. Ce cadre de module peut être généralisé pour enseigner l’assemblage Gibson pour diverses applications, fournissant aux instructeurs une boîte à outils pour enseigner une technologie de clonage adaptable et émergente tout en faisant avancer leurs projets de recherche.
Introduction
La formation des étudiants de premier cycle aux concepts fondamentaux de la biologie moléculaire et aux techniques de laboratoire est cruciale pour leur développement scientifique et professionnel, car ces méthodologies sont courantes dans divers contextes de recherche, y compris le milieu universitaire et l’industrie. En tant que tels, les étudiants en biologie (concentration en biologie moléculaire et cellulaire) et en biochimie à l’Université d’État polytechnique de Californie, San Luis Obispo (Cal Poly) sont tenus de suivre un cours de laboratoire de biologie moléculaire de division supérieure pour apprendre et appliquer ces sujets (CHEM/BIO 475). Un programme de base pour ce cours a déjà été élaboré dans lequel les étudiants effectuent un clonage à base de topoisomérase (TOPO) pour assembler un plasmide contenant de l’actine préparé à partir d’un modèle d’ADN complémentaire de levure (ADNc)1. Les étudiants conçoivent des expériences basées sur des questions qui imitent des hypothèses de recherche authentiques, augmentant ainsi leur familiarité avec les pratiques de laboratoire et l’apprentissage basé sur l’enquête. L’avancement continu dans le domaine de la biologie moléculaire exige que les programmes d’études correspondants s’adaptent pour préparer les étudiants dotés de compétences modernes à la main-d’œuvre. En particulier, l’utilisation de l’assemblage Gibson est devenue plus prédominante dans la communauté scientifique ; Bien que la méthode ait été établie à l’origine pour synthétiser des chromosomes artificiels2, plus de 5 000 publications au moment de la rédaction de ce rapport ont fait référence aux travaux originaux de Gibson et al. Gibson Assembly est unique par rapport aux méthodologies de clonage traditionnelles : il est hautement personnalisable et peut facilement ligaturer plusieurs fragments d’ADN linéaires sans avoir besoin de sites de restriction pour produire les jonctions. Ainsi, nous avons vu l’opportunité de réorganiser le programme CHEM/BIO 475 pour intégrer des techniques modernes de clonage moléculaire et améliorer le modèle de cours basé sur l’enquête.
Il a été établi que les expériences de recherche des étudiants contribuent à une meilleure compréhension conceptuelle, au développement des compétences et à la persévérance en sciences3, mais tous les étudiants de premier cycle n’ont pas la possibilité de participer directement à un laboratoire de recherche. Pour relever le défi de la capacité limitée des étudiants dans les laboratoires de recherche, des expériences de recherche de premier cycle basées sur des cours (CURE) ont été développées et utilisées pour accroître l’accessibilité des sciences grâce à des recherches authentiques en classe. Bien que la mise en œuvre des CURE varie, des pratiques communes qui abordent les cinq principes de la recherche scientifique ont été établies. Dans le cadre d’un CURE bien conçu, les élèves devront 1) utiliser des pratiques scientifiques, 2) collaborer à un projet de recherche, 3) tenter de faire de nouvelles découvertes, 4) contribuer à des travaux pertinents en dehors de la salle de classe et 5) réévaluer et réviser des hypothèses et des méthodes en cas d’échec expérimental4. À l’instar des expériences de recherche traditionnelles des étudiants en laboratoire, il a été démontré que les CURE renforcent la confiance des étudiants dans les sciences, les compétences scientifiques, l’appropriation du projet et la persévérance dans les sciences, la technologie, l’ingénierie et les mathématiques (STIM)5. Bien que des CURE impliquant le clonage moléculaire aient été signalés précédemment 6,7,8,9,10,11,12,13, nous n’en connaissons aucun qui souligne l’adaptabilité de Gibson Assembly pour créer une bibliothèque de plasmides de recherche authentiques.
Ici, nous rapportons une expansion du programme actuel basé sur l’enquête CHEM/BIO 475 à l’Université d’État polytechnique de Californie, San Luis Obispo, avec deux améliorations majeures : l’expérience pratique de l’utilisation de Gibson Assembly et la participation d’étudiants à un CURE, qui a fourni des constructions plasmidiques originales pour des projets de recherche financés par la National Science Foundation (NSF-1708919 et NSF-2300890). Au cours des trois mises en œuvre de ce programme, les étudiants ont contribué à deux projets de recherche distincts axés sur la biosynthèse de produits naturels de molécules bioactives produites par Actinomycetota. Les produits naturels contiennent souvent des pharmacophores ayant des activités antibiotiques, antifongiques et/ou anticancéreuses, ce qui confère à ces petites molécules une importance dans les efforts de découverte de médicaments et un potentiel de pertinence clinique14. Cette recherche nécessite la création de banques de plasmides pour permettre d’étudier à la fois la fonction et le potentiel d’ingénierie des enzymes biosynthétiques bactériennes. Dans le cadre de ce CURE, les étudiants ont conçu et réalisé des expériences d’assemblage de Gibson pour cloner les bibliothèques de plasmides uniques pertinentes à ces projets de recherche (Figure 1). De plus, le format et la conception du module se distinguent par le fait qu’il est facilement adaptable pour générer des plasmides d’intérêt pour d’autres projets de recherche.
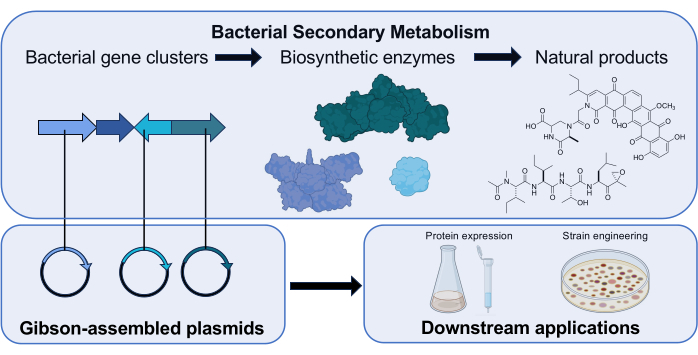
Figure 1 : Vue d’ensemble du rôle de Gibson Assembly dans notre laboratoire de recherche. Actinomycetota produit des produits naturels à petites molécules avec des bioactivités cliniquement pertinentes en utilisant des groupes de gènes qui codent pour des enzymes biosynthétiques. Dans nos recherches, des plasmides contenant un gène biosynthétique sont assemblés via l’assemblage de Gibson pour des études en aval de la fonction de l’enzyme codée. Icônes scientifiques de Biorender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vue d’ensemble du laboratoire
Le laboratoire de biologie moléculaire est un cours de division supérieure requis pour les majors en biochimie et les majors en biologie avec une concentration en biologie moléculaire et cellulaire. Les autres étudiants qui remplissent les conditions préalables sont invités à suivre le cours en tant que cours facultatif de division supérieure. Le cours est co-répertorié entre le département de chimie et de biochimie et le département de biologie de Cal Poly. Les professeurs des deux départements se relaient pour dispenser le cours chaque trimestre (deux trimestres en biologie, un trimestre en chimie et en biochimie).
La classe se réunit dans le laboratoire deux fois par semaine pendant des périodes de 170 minutes et une fois par semaine pour un cours de 50 minutes. Les sections de laboratoire contiennent un maximum de 16 étudiants et 2 à 3 sections de laboratoire sont proposées chaque trimestre. Le cours dure 10 semaines et l’examen final est administré lors de la dernière réunion de la semaine 10. Le temps de cours est consacré à la discussion de la théorie derrière de nombreuses techniques expérimentales menées en laboratoire, ainsi que des sujets d’actualité en biologie moléculaire qui ne sont pas couverts en laboratoire. Le programme de base du laboratoire englobe le processus de clonage du gène de l’actine à partir de la levure1, qui prend environ 7 semaines (13 à 14 réunions de laboratoire). Les techniques comprennent le micropipetage, l’isolement de l’ARN de levure, l’amplification d’un gène de levure à l’aide de la transcription inverse et de la réaction en chaîne par polymérase (RT-PCR), le clonage TOPO, le criblage bleu-blanc, l’isolement des plasmides et la vérification de l’insert par digestion de restriction et PCR, l’analyse in silico des clones et l’analyse de séquences d’ADN. Le programme pour les 3 dernières semaines du cours est à la discrétion de l’instructeur, mais implique que les étudiants réalisent un « projet indépendant » avec des résultats indéfinis.
Présentation de l’expérience
L’un des objectifs de notre groupe de recherche est les voies de biosynthèse chez Actinomycetota. Lors de la conception du projet indépendant, nous avons envisagé de demander aux étudiants de créer des plasmides à l’aide de Gibson Assembly pour les utiliser dans nos projets de recherche sur la biosynthèse des produits naturels. Alors que les itérations du module de cours qui sont évaluées ici étaient spécifiques aux plasmides qui permettent la manipulation des voies de biosynthèse, le flux de travail de l’assemblage Gibson est énormément adaptable à d’autres projets de clonage moléculaire (Figure 2). Le flux de travail a été divisé en trois expériences différentes (A, B et C) qui ont été réalisées sur deux périodes de cours et six périodes de laboratoire (3 semaines au total) (voir le fichier supplémentaire 1 et le fichier supplémentaire 2). Les expériences ont été précédées de feuilles de travail pour aider les élèves à se préparer et à évaluer leur compréhension (Fichier supplémentaire 3, Fichier supplémentaire 4 et Fichier supplémentaire 5). Le flux de travail est présenté dans un format qui s’adapte aux besoins et aux intérêts de l’instructeur.

Figure 2 : flux de travail du module d’assemblage Gibson. Le jour 1 et le jour 4 sont des périodes de cours où les étudiants effectuent une analyse séquentielle in silico et une conception expérimentale. Les jours 2 et 3 et 5 à 8 sont des réunions de laboratoire au cours desquelles les étapes de clonage de nouveaux plasmides via Gibson Assembly sont effectuées, suivies d’un isolement et d’un dépistage. L’organigramme illustré est regroupé en fonction des trois expériences réalisées par les élèves (A, B et C). Des directives et des protocoles plus détaillés se trouvent dans les manuels de l’instructeur et de l’étudiant fournis dans le dossier supplémentaire 1 et le fichier supplémentaire 2, respectivement. Icônes scientifiques de Biorender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le module de projet indépendant Gibson Assembly a été testé pour la première fois au printemps 2019 dans CHEM/BIO 475. En 2020 et 2021, le cours a été dispensé en ligne en raison de la pandémie de SARS-CoV-2. Lorsque l’enseignement en personne a repris au printemps 2022 et 2023, les étudiants du cours ont été invités à participer à une étude évaluant les résultats d’apprentissage d’un projet indépendant de Gibson Assembly où des plasmides originaux et pertinents pour la recherche seraient clonés. En 2019, les étudiants ont créé une bibliothèque de plasmides composée de cassettes de gènes du génome de Micromonospora echinospora ATCC 15837 qui ont été clonées dans pKC1132 (Figure 3). Cette bibliothèque de plasmides est utilisée dans notre laboratoire de recherche pour inactiver les gènes d’intérêt dans un groupe de gènes biosynthétiques présumés pour le produit naturel TLN-0522015. Pour compléter nos études d’inactivation génique, les étudiants ont cloné en 2022 une petite bibliothèque de gènes du groupe de gènes présumé TLN-05220 dans pUC19 (Figure3) ; Notre groupe de recherche a utilisé ces plasmides pour subcloner des gènes dans des vecteurs d’expression, y compris pET28b, pour la surexpression et la purification des protéines. Les étudiants de la cohorte 2023 ont contribué aux travaux en cours sur un projet d’ingénierie biosynthétique sur l’époxomicine synthétase16. En équipes de 3 à 4, les étudiants ont cloné des domaines modifiés de modules de peptide synthétase17 non ribosomique dans divers vecteurs d’expression protéique afin d’optimiser la surexpression et la purification de ces enzymes dans notre laboratoire de recherche (Figure 3). La redondance a été intégrée dans le plan de clonage pour chaque cohorte. Par exemple, la cohorte de 2019 contenait 44 étudiants, et 15 plasmides ont été attribués à la classe, pour le clonage. Ainsi, le clonage de chaque plasmide a été tenté deux ou trois fois.
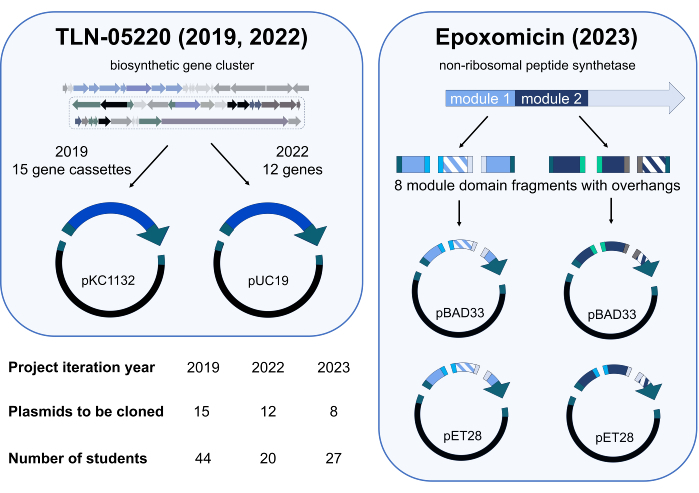
Figure 3 : Résumé des plasmides clonés et des participants étudiants au cours des itérations indépendantes du projet en 2019, 2022 et 2023. Le projet Gibson Assembly a été déployé trois fois. Dans chaque offrande, les étudiants participants ont cloné une bibliothèque différente de plasmides à utiliser dans des projets de recherche explorant les voies de biosynthèse. Les projets de 2019 et 2022 ont soutenu nos travaux en cours sur le produit naturel TLN-0522015 avec deux réactions d’assemblage Gibson fragment (une cassette de gènes ou un gène et un vecteur). Le projet de 2023 a impliqué l’échange de domaines au sein des modules 1 et 2 d’une enzyme peptide synthétase non ribosomique (NRPS) impliquée dans la biosynthèse de l’époxomicine16. Les fragments hachurés représentent deux mutants différents du domaine échangé, et les couleurs unies représentent des domaines qui n’ont pas été échangés. Au total, huit fragments de gènes différents (quatre pour le module 1 et quatre pour le module 2) ont été générés avec des porte-à-faux compatibles avec l’assemblage Gibson. Pour chaque module, deux combinaisons différentes de trois fragments de gènes ont été assemblées avec l’un des deux vecteurs différents (pBAD33 et pET28, quatre fragments au total par assemblage), afin de générer huit plasmides NRPS modifiés. Icônes scientifiques de Biorender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Aperçu de l’évaluation
Au moins 1 semaine avant le début du projet indépendant Gibson Assembly, les étudiants des cohortes 2022 et 2023 ont été invités à participer à une enquête sur les résultats d’apprentissage, conçue de la même manière qu’une étude réalisée dans le cadre d’un cours d’enquête sur le laboratoire de biochimie à Cal Poly18. Les étudiants participants ont rempli un pré-questionnaire à choix multiples lors de la réunion de laboratoire avant le début du projet indépendant et un post-questionnaire à choix multiples lors de la réunion finale du laboratoire (c’est-à-dire après avoir terminé le module d’assemblage Gibson). Le pré-questionnaire et le post-questionnaire comprenaient 28 questions identiques, avec deux questions supplémentaires dans le post-questionnaire (30 au total). Dix questions de contenu ont été rédigées pour évaluer les connaissances des élèves sur les enzymes et les mécanismes impliqués dans le clonage moléculaire (p. ex., réaction en chaîne par polymérase [PCR], assemblage de Gibson, transformation, criblage bleu-blanc). Les sept questions suivantes demandaient aux élèves d’auto-évaluer leur familiarité avec les termes du clonage moléculaire (p. ex., ADN polymérase, exonucléase, ligase). Les 10 questions suivantes ont permis aux étudiants d’auto-évaluer leur capacité à effectuer des techniques de clonage moléculaire (par exemple, analyse de séquences d’ADN, réactions de digestion par restriction, électrophorèse sur gel d’agarose). Les étudiants ont également indiqué s’ils seraient à l’aise de poursuivre une carrière en biologie moléculaire en fonction de leurs connaissances des techniques de clonage moléculaire. Deux questions supplémentaires ont été incluses dans le post-questionnaire pour permettre aux étudiants d’auto-évaluer leur engagement à apprendre dans le cours et de déterminer si le cours a été une expérience d’apprentissage précieuse (Fichier supplémentaire 6 et Fichier supplémentaire 7). Toutes les données des réponses des élèves avant et après le questionnaire de 2022 et de 2023 ont été combinées aux fins d’analyse et sont disponibles dans le tableau supplémentaire S1.
Protocole
Les études impliquant des participants humains ont été examinées et approuvées par des sujets humains dans le Research Institutional Review Board de Cal Poly (2022-113-CP (IRB)). Les participants ont donné leur consentement éclairé écrit à participer à cette étude.
Le protocole suivant décrit la préparation de l’instructeur (étapes 1.1 à 1.3), les actions des étudiants pour un module d’enseignement de trois expériences qui comprend la PCR pour obtenir des fragments linéaires (étapes 2.1 à 2.7), l’assemblage, la transformation et la sélection de clones de Gibson (étapes 3.1 à 3.5), l’isolement et le criblage des plasmides (étapes 4.1 à 4.4) et l’évaluation des résultats d’apprentissage (5.1 à 2,7).5.2). La préparation de l’instructeur décrit un exemple représentatif de conception d’amorces et de préparation d’une carte plasmidique souhaitée in silico. Toutes les sections du protocole sont adaptables à d’autres plasmides souhaités. Chaque expérience d’étudiant est divisée en deux réunions de laboratoire de 3 heures.
1. Préparation de l’instructeur
- Conception d’apprêt Gibson Assembly à l’aide de l’assistant d’assemblage Benchling
- Déterminez la source de matrice d’ADN pour les inserts de gènes, tels que l’ADN génomique, le plasmide et l’ADN synthétique.
NOTE: Pour les gènes qui seront amplifiés à partir de matrices d’ADN génomique complexes, il est conseillé de déterminer d’abord des sites de liaison uniques pour les amorces dans la matrice d’ADN génomique qui sont spécifiques pour le ou les gènes d’intérêt, à l’aide de l’outil de recherche d’alignement local de base (BLAST) du National Center for Biotechnology Information (NCBI)21, comme détaillé dans le fichier supplémentaire 1. Pour les matrices simples telles que les plasmides ou l’ADN synthétique, passez à l’étape suivante. - Récupérez les séquences d’ADN de l’insert ou des inserts de gène et du vecteur (par exemple, voir la séquence du gène de la polycétide hydroxylase et la séquence pUC19 fournies sous forme de fichiers GenBank dans le fichier supplémentaire 8). Importez la ou les séquences d’insertion souhaitées et la séquence vectorielle souhaitée dans Benchling, chacune sous la forme d’un nouveau fichier de séquence d’ADN. Ouvrez chaque séquence importée à inclure dans la réaction de l’assemblage Gibson.
NOTE: On peut importer des fichiers de séquences d’ADN dans plusieurs formats, y compris GenBank et FASTA. On peut également importer une séquence directement à partir d’une base de données avec un numéro d’accès ou coller une séquence de nucléotides copiée à partir d’un autre fichier ou d’un autre visualiseur. - En bas de l’écran, recherchez l’outil Assistant d’assemblage . Cliquez sur Assistant Assemblage, puis sélectionnez Créer un assemblage. Parmi les options proposées, sélectionnez Gibson et cliquez sur Démarrer pour commencer l’assemblage.
- Dans la fenêtre de séquence vectorielle , sélectionnez toutes les bases à inclure dans l’assemblage à partir du squelette vectoriel. Commencez la sélection à la position du nucléotide à l’extrémité 3' de l’insert du gène (par exemple, position 657 de pUC19), et sélectionnez tous les nucléotides restants à inclure (par exemple, jusqu’à la position 656 pour inclure la séquence entière de pUC19). Une fois sélectionné, cliquez sur l’onglet Backbone en bas de l’écran | Définir le fragment.
NOTE: Basculez entre les vues Carte de séquence et Plasmide du vecteur pour sélectionner toutes les bases souhaitées d’un modèle circulaire. Par ailleurs, si vous prévoyez de préparer le fragment de vecteur par digestion de restriction (au lieu de PCR) pour l’assemblage de Gibson, linéarisez la séquence du squelette avec un site de coupe de l’enzyme de restriction (par exemple, XbaI). Pour ce faire, cliquez sur le site de coupe, maintenez la touche Maj enfoncée, puis cliquez à nouveau sur le site de coupe . Cliquez ensuite sur Définir le fragment pour définir le backbone. N’oubliez pas que les amorces spécifiques au vecteur ne seront pas générées par Benchling si le squelette est linéarisé avec une enzyme de restriction dans l’assistant d’assemblage. - Dans la fenêtre de séquence d’insertion , sélectionnez toutes les bases de l’insert à inclure dans l’assemblage. Une fois sélectionné, cliquez sur l’onglet Insertion en bas de l’écran | Définir le fragment.
- Dans le cas d’insertions de plusieurs gènes, cliquez sur le bouton + sur le côté droit de l’assistant d’assemblage. Dans la fenêtre de séquence d’insertion , sélectionnez toutes les bases d’insertion à inclure dans l’assemblage. Une fois sélectionné, cliquez sur l’onglet Insertion en bas de l’écran | Définir le fragment.
- Une fois tous les fragments définis, renommez l’assemblage avec le nom de plasmide souhaité et cliquez sur Assembler à droite de l’assistant d’assemblage. Sélectionnez l’emplacement de dossier souhaité pour le dossier de séquence et le dossier d’amorce. Cliquez sur Créer pour assembler la séquence plasmidique recombinante.
- Ouvrez le plasmide assemblé et cliquez sur Historique de l’assemblage pour afficher une carte de plasmide de base et une vue d’ensemble des séquences dont il est dérivé. Assurez-vous que les positions des nucléotides de chaque fragment correspondent à la conception souhaitée.
- Dans l’onglet Paramètres d’assemblage , affichez les noms des amorces conçues pour l’assemblage. Notez qu’il y aura deux amorces conçues pour chaque insert (une avant et une arrière), et que les noms des amorces seront dérivés des titres des fichiers de séquence d’ADN. Assurez-vous également que les températures de fusion de l’amorce et les températures de recuit suggérées sont compatibles dans cette fenêtre (voir le fichier supplémentaire 8, Amorces représentatives).
- Si les températures de fusion ou de recuit de l’amorce ne sont pas idéales, ajustez manuellement les séquences d’amorces (par exemple, en supprimant les nucléotides pour réduire la température de fusion) dans les fichiers de séquences d’amorces. Vous pouvez également cliquer sur Rouvrir et ajuster les paramètres de l’amorce en cliquant sur le bouton Paramètres de l’enzyme/de l’amorce à côté du bouton Assembler dans l’assistant d’assemblage. Ensuite, répétez l’étape d’assemblage et réexaminez les séquences d’amorces.
- Si une bibliothèque de plasmides doit être clonée avec de nombreux gènes différents clonés dans le même squelette vecteur,
- Utilisez la même paire d’amorces pour le vecteur dans chaque cas. Pour ce faire, retirez manuellement tous les nucléotides des amorces vectorielles qui installeraient les surplombs spécifiques à l’insert. Ces nucléotides se trouveraient à l’extrémité 5' de l’amorce . Après le retrait, assurez-vous que les températures de fusion de l’apprêt et les températures de recuit sont compatibles (voir le fichier supplémentaire 8, Amorces représentatives).
- Assurez-vous que toutes les paires d’amorces d’insertion ont des températures de fusion et de recuit très similaires.
NOTE: Cela réduira le besoin d’optimiser la température de recuit de chaque paire et permettra à toutes les réactions PCR des étudiants d’être cyclées en même temps à l’aide du même programme de thermocycleur.
- Une fois la conception de l’amorce terminée pour les plasmides souhaités, cliquez sur Finaliser dans l’assistant d’assemblage. Commandez les amorces auprès d’une entreprise de synthèse d’ADN et récupérez des échantillons des matrices d’ADN pour les inserts et les vecteurs.
- Testez des paires d’amorces spécifiques à l’insert et au vecteur pour des températures de recuit optimales à l’aide d’une expérience de PCR à gradient avec les matrices d’ADN. Testez initialement les températures de recuit recommandées par Benchling ± 3 °C.
- Une fois que les températures de recuit ont été déterminées, créez des aliquotes de solutions d’amorces, des matrices d’ADN et les réactifs nécessaires aux réactions PCR pour les étudiants.
- Déterminez la source de matrice d’ADN pour les inserts de gènes, tels que l’ADN génomique, le plasmide et l’ADN synthétique.
- Avant les expériences, fournissez des feuilles de travail aux élèves pour les aider à se préparer et à évaluer la compréhension (Dossier supplémentaire 4, Dossier supplémentaire 5 et Dossier supplémentaire 6).
- Collecte de données sur les résultats d’apprentissage
NOTE: Si vous souhaitez collecter des données sur les résultats d’apprentissage à partir du module Gibson Assembly et publier les résultats, obtenir l’approbation de la recherche avec des sujets humains conformément aux politiques de l’établissement.- Invitez les élèves à participer à une enquête sur les résultats d’apprentissage au moins 1 semaine avant le début du projet indépendant Gibson Assembly.
- Demandez aux étudiants participants de remplir le pré-questionnaire à choix multiples (fichier supplémentaire 6) lors de la réunion de laboratoire avant le début du projet indépendant.
2. Expérience d’étudiant A : PCR pour obtenir des fragments linéaires
- Obtenez des amorces et des solutions d’ADN pour la PCR auprès de l’instructeur.
- Pipette pour des réactions de 25 μL, y compris l’ADN polymérase MasterMix, la matrice d’ADN, les amorces avant et inverse et l’eau sans nucléases conformément aux instructions du manuel de l’étudiant (voir le fichier supplémentaire 2).
- Effectuez le cycle de la réaction dans un thermocycleur conformément au manuel de l’étudiant dans le fichier supplémentaire 2. Assurez-vous que la température de recuit de la réaction est appropriée pour les amorces et que le temps d’extension est approprié pour la longueur de l’amplicon souhaité. Reportez-vous aux fichiers supplémentaires 2 et 3 pour des instructions détaillées.
- Conservez les réactions de PCR à 4 °C ou à -20 °C jusqu’à la période de laboratoire suivante.
- Analyser 5 μL de chaque réaction par électrophorèse sur gel d’agarose. Pendant que le gel fonctionne, ajoutez 1 μL d’enzyme de restriction DpnI à chaque réaction qui a utilisé l’ADN plasmidique comme modèle. Incuber ce mélange pendant 1 h à 37 °C.
NOTE: DpnI dégradera toute matrice d’ADN plasmidique circulaire restante, ce qui la rendra moins susceptible d’obtenir des faux positifs lors de l’étape de transformation. - Imagez le gel pour confirmer que la PCR a réussi et que l’amplification correcte a été obtenue. Purifiez toutes les réactions PCR réussies avec un kit de purification PCR disponible dans le commerce. Mesurez la concentration (ng/μL) du produit PCR purifié à l’aide d’un spectrophotomètre à microvolume pour l’utiliser dans les calculs d’assemblage ultérieurs de Gibson.
- Avant la prochaine réunion de classe, concevez la recette de réaction pour l’assemblage Gibson (voir le fichier supplémentaire 2 et le fichier supplémentaire 4).
3. Expérience B de l’étudiant : Assemblage de Gibson, transformation et sélection de clones
- Pipeter la réaction de l’assemblage Gibson selon la recette conçue. Incuber la réaction à 50 °C pendant 15 min. Pendant que les réactions incubent, préparez-vous à la transformation en décongelant des cellules d’Escherichia coli chimiquement compétentes sur de la glace.
- Transformez 2 μL de l’assemblage Gibson en cellules chimiquement compétentes par choc thermique. Pipeter 100 μL des cellules transformées sur deux plaques de sélection et étaler avec des billes stérilisées. Préparez 10-4, 10-5 et 10-6 dilutions des cellules restantes sur de la gélose Luria-Bertani (LB). Plaque de 100 μL de chaque dilution en série sur LB (pas de sélection) et étalage avec des billes stérilisées.
NOTE: Cette étape permet de calculer l’efficacité de la transformation. - Incuber les plaques pendant la nuit à 37 °C. Conserver les plaques à 4 °C jusqu’à la prochaine période de cours.
- Comptez les colonies sur toutes les plaques et calculez l’efficacité de la transformation comme indiqué dans le fichier supplémentaire 2. Striez de nouveau une plaque sélective avec quatre colonies de l’une ou l’autre plaque sélective et incubez cette plaque à 37 °C pendant la nuit.
NOTE: Cela sert de culture de sauvegarde de vos clones positifs. - À l’aide d’un marqueur, sélectionnez, encerclez et étiquetez quatre colonies distinctes sur la plaque de sélection avec les initiales de l’élève et un chiffre (p. ex., ABC1). Récupérez une nouvelle plaque de gélose LB contenant l’antibiotique pour la sélection, utilisez un marqueur pour diviser la plaque en quadrants (c’est-à-dire des quartes), puis utilisez ~1/2 de chaque colonie pour retracer sur le quadrant respectivement marqué et l’autre 1/2 pour inoculer une culture liquide de LB de 5 mL pour chacune des quatre colonies sélectionnées. Assurez-vous d’étiqueter les tubes avec l’identité de la colonie respective (par exemple, ABC1) et d’ajouter la concentration correcte d’antibiotique pour la sélection. Incuber les cultures liquides dans un incubateur à agitation à 37 °C pendant la nuit et la plaque de gélose dans un incubateur statique à 37 °C ; Conserver les cultures liquides et les plaques à 4 °C jusqu’à la période de classe suivante.
4. Expérience d’étudiant C : Isolement et criblage de plasmides
- Isolez l’ADN plasmidique des cultures liquides de l’expérience B à l’aide d’un kit miniprep. Mesurer la concentration du plasmide isolé (en ng/μL).
- Concevoir un test de synthèse de restriction ou un dépistage par PCR pour analyser les plasmides isolés (voir les instructions détaillées dans le fichier supplémentaire 2 et les attentes en matière de rapports dans le fichier supplémentaire 5).
- Pipeter la restriction, digérer les réactions ou les réactions selon les recettes conçues et incuber à des températures et des durées conçues précédemment.
- Analysez les résultats par électrophorèse sur gel d’agarose.
5. Appréciation
- Demandez aux étudiants participants de remplir le post-questionnaire à choix multiples (fichier supplémentaire 7) lors de la réunion de laboratoire après la fin du module d’assemblage Gibson.
- Combinez toutes les données des réponses avant et après le questionnaire pour l’analyse.
- Examinez la signification statistique de l’augmentation du score moyen de chaque question de contenu individuelle entre les questionnaires à l’aide de tests t appariés bilatéraux et de l’ampleur de l’effet d de Cohen.
- Évaluez l’augmentation de la confiance des élèves pour chaque terme et chaque technique afin de déterminer l’ampleur du changement et sa signification statistique.
- Déterminez l’impact des antécédents et des facteurs scolaires de l’élève sur l’apprentissage des élèves à l’aide du gain d’apprentissage normalisé (NLG) et des tests t non appariés.
- Évaluez les attitudes des étudiants à l’égard de l’expérience du projet et des carrières en biologie moléculaire en évaluant les notes moyennes.
Résultats
Réussite des élèves dans le clonage
Dans chaque itération du module Gibson Assembly (2019, 2022 et 2023), les étudiants ont été invités à préparer un rapport résumant leurs conclusions. En 2019, 36 des 44 étudiants (81,8 %) ont déclaré avoir réussi à cloner leurs plasmides en se basant sur les résultats du criblage qu’ils ont conçu pour l’expérience C. Au total, 14 étudiants sur 20 (70,0 %) ont déclaré avoir réussi à cloner les concepts qui...
Discussion
Ici, nous proposons un projet de laboratoire de premier cycle ajustable qui enseigne aux étudiants le clonage moléculaire par Gibson Assembly dans un cadre de recherche basé sur un cours. Au total, 28 nouveaux plasmides ont été clonés par des étudiants de premier cycle dans une salle de classe. Le succès individuel rapporté par les élèves variait de 44,4 % à 81,8 % sur trois cohortes, et le succès global du clonage était de 80 % (28 des 35 plasmides attribués ont été clo...
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Les auteurs remercient chaleureusement Andrea Laubscher pour son soutien technique, ainsi que Michael Black, Sandi Clement et Javin Oza pour leurs discussions utiles sur la mise en œuvre du laboratoire d’enseignement et l’évaluation des résultats d’apprentissage. Les auteurs remercient tous les étudiants qui ont participé à l’étude sur les résultats d’apprentissage dans les cohortes de 2019, 2022 et 2023, ainsi que les étudiants Nathan Kuhn et Aayushi Adettiwar qui ont aidé à la préparation des réactifs pour la mise en œuvre du laboratoire d’enseignement. Les auteurs remercient également le soutien financier du Fonds William et Linda Frost, de la subvention de dotation Chevron Biotechnology Applied Research du Center for Applications in Biotechnology et de la National Science Foundation (NSF-1708919 et NSF-2300890).
matériels
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Références
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon