JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Research Article
Adaptable Molecular Cloning 과정 기반 학부 연구 경험을 통한 학생 결과 개선
* 이 저자들은 동등하게 기여했습니다
요약
적응형 Gibson Assembly 분자 복제 모듈은 분자 생물학 실험실 과정 학생들을 위한 과정 기반 CURE(학부 연구 경험) 형식으로 사용되었습니다. 학생 학습 결과에 대한 평가는 CURE 완료 후 분자 복제에 대한 이해와 자신감이 향상되었음을 보여주었으며 천연물 생합성 연구를 위해 새로운 플라스미드를 복제했습니다.
초록
분자생물학 기술의 지속적인 발전은 학생들이 현대적인 역량을 가지고 직장에 진입할 수 있도록 효과적으로 준비할 수 있도록 분자생물학 커리큘럼을 정기적으로 개선해야 합니다. 특히, 고도로 사용자 정의 가능하고 적응력이 뛰어난 분자 복제 기술인 Gibson Assembly의 출현은 수많은 연구 환경에서 분자 복제의 지평을 발전시켰습니다. 따라서 우리는 San Luis Obispo에 있는 California Polytechnic State University의 분자 생물학 실험실 과정에 배포하기 위한 Gibson Assembly 클로닝 모듈을 만들고 이 모듈에서 학생들의 학습 결과를 평가했습니다. 이 과정을 세 번 반복하는 동안 학생들은 천연 제품 생합성 연구 프로젝트를 지원하기 위해 3개의 고유한 플라스미드 라이브러리를 클로닝하는 것과 관련된 실험 기반의 독립 프로젝트에 참여했습니다. 학생들은 분자 복제에 대한 이해와 분자 생물학 용어 및 기술에 대한 자신감을 평가하기 위해 사전 및 사후 설문지를 받았습니다. 학생들의 응답은 분자 복제 개념을 학습하는 것과 분자 복제 용어 및 기술에 대한 자기 보고 신뢰도 모두에서 상당한 증가를 보여주었습니다. 이 모듈 프레임워크는 다양한 응용 분야에 대해 Gibson Assembly를 가르치기 위해 일반화할 수 있으며, 강사에게 연구 프로젝트를 진행하면서 적응 가능하고 새로운 복제 기술을 가르치기 위한 툴킷을 제공합니다.
서문
학부생에게 기본적인 분자 생물학 개념 및 실험실 기술을 교육하는 것은 학계 및 산업을 포함한 다양한 연구 환경에서 일반적이기 때문에 학부생의 과학 및 전문성 개발에 매우 중요합니다. 따라서 캘리포니아 폴리테크닉 주립대학교(California Polytechnic State University, San Luis Obispo, Cal Poly)에서 생물학(분자 및 세포 생물학 집중) 및 생화학 전공 학생들은 이러한 주제(CHEM/BIO 475)를 배우고 적용하기 위해 상위 부문 분자 생물학 실험실 과정을 이수해야 합니다. 이 과정의 기본 커리큘럼은 이전에 개발되었으며, 학생들은 효모 상보적 DNA(cDNA) 주형1에서 준비된 액틴 함유 플라스미드를 조립하기 위해 토포이소머라아제 기반(TOPO) 클로닝을 수행합니다. 학생들은 실제 연구 가설을 모방한 질문을 기반으로 실험을 설계하여 실험실 실습 및 탐구 기반 학습에 대한 친숙도를 높입니다. 분자 생물학 분야의 지속적인 발전은 학생들이 노동 인구를 위한 현대적인 역량을 갖출 수 있도록 해당 커리큘럼을 조정해야 합니다. 특히, Gibson Assembly의 사용은 과학계에서 더욱 우세해졌습니다. 이 방법은 원래 인공 염색체2를 합성하기 위해 확립되었지만, 이 보고서 작성 당시 5,000개 이상의 출판물에서 Gibson et al.의 원본 연구를 참조했습니다. Gibson Assembly는 기존 클로닝 방법론과 비교할 때 독특합니다: 사용자 정의가 가능하며 접합부를 생성하기 위한 제한 부위 없이 여러 선형 DNA 단편을 쉽게 접합할 수 있습니다. 따라서 우리는 CHEM/BIO 475 커리큘럼을 수정하여 현대 분자 복제 기술을 통합하고 탐구 기반 과정 모델을 개선할 수 있는 기회를 보았습니다.
학생의 연구경험이 과학에 대한 개념 이해, 기술 개발 및 끈기 향상에 기여하는 것으로 확인되었지만3 모든 학부생이 연구실에 직접 참여할 수 있는 기회를 갖는 것은 아닙니다. 연구 실험실에서 제한된 학생 수용 능력의 문제를 해결하기 위해 강의 기반 학부 연구 경험(CURE)이 개발되고 채택되어 교실에서의 진정한 연구를 통해 과학적 접근성을 높입니다. CURE는 구현에 있어 다양하지만 과학 연구의 다섯 가지 원칙을 다루는 일반적인 관행이 확립되었습니다. 잘 설계된 CURE에서 학생들은 1) 과학적 관행을 사용하고, 2) 연구 프로젝트에 협력하고, 3) 새로운 발견을 시도하고, 4) 교실 밖에서 관련 작업에 기여하고, 5) 실험 실패의 경우 가설과 방법을 재평가하고 수정합니다4. 실험실에서의 전통적인 학생 연구 경험과 유사하게, CURE는 과학, 과학적 기술, 프로젝트 소유권 및 과학, 기술, 공학 및 수학(STEM)에 대한 인내심에 대한 학생의 자신감을 강화하는 것으로 나타났습니다5. 분자 클로닝과 관련된 CURE는 이전에 보고되었지만, 6,7,8,9,10,11,12,13, 진정한 연구 플라스미드 라이브러리를 만들기 위한 Gibson Assembly의 적응성을 강조하는 사례는 없습니다.
여기에서는 San Luis Obispo에 있는 California Polytechnic State University의 현재 탐구 기반 CHEM/BIO 475 커리큘럼을 두 가지 주요 개선 사항으로 확장한 것을 보고합니다: Gibson Assembly를 사용한 실습 경험과 National Science Foundation(NSF-1708919 및 NSF-2300890)에서 자금을 지원하는 연구 프로젝트를 위한 독창적인 플라스미드 구조를 제공한 CURE에 대한 학생 참여. 이 커리큘럼을 세 번 이상 구현한 학생들은 방선균에 의해 생성된 생체 활성 분자의 천연 제품 생합성에 초점을 맞춘 두 가지 별개의 연구 프로젝트에 기여했습니다. 천연물에는 항생제, 항진균제 및/또는 항암 활성을 가진 약전이 함유되어 있는 경우가 많기 때문에 이러한 소분자는 신약 개발 노력에 중요한 역할을 하고 임상적 관련성을 가질 수 있다14. 이 연구는 박테리아 생합성 효소의 기능과 엔지니어링 잠재력을 모두 조사할 수 있도록 플라스미드 라이브러리를 생성해야 합니다. 이 CURE에서 학생들은 이러한 연구 프로젝트와 관련된 고유한 플라스미드 라이브러리를 복제하기 위해 Gibson Assembly 실험을 설계하고 수행했습니다(그림 1). 또한 모듈의 형식과 디자인은 다른 연구 프로젝트에서 관심 있는 플라스미드를 생성하도록 쉽게 조정할 수 있기 때문에 독특합니다.
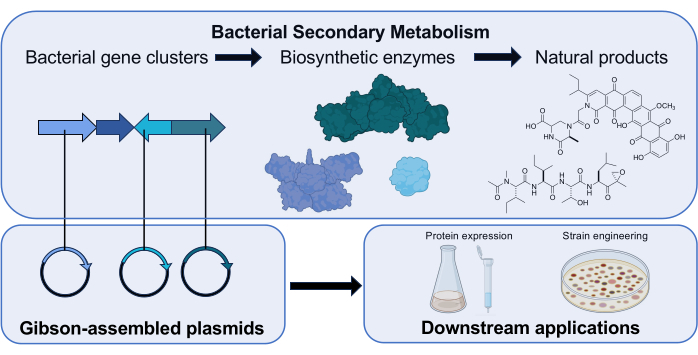
그림 1: 우리 연구소에서 Gibson Assembly의 역할에 대한 개요. 방선균(actinomycetota)은 생합성 효소를 암호화하는 유전자 클러스터를 사용하여 임상적으로 관련된 생체 활성을 가진 저분자 천연 제품을 생산합니다. 본 연구에서는 암호화된 효소의 기능에 대한 다운스트림 조사를 위해 Gibson Assembly를 통해 생합성 유전자를 포함하는 플라스미드를 조립합니다. Biorender.com 의 과학 아이콘. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
연구실 개요
분자생물학 연구실은 분자 및 세포 생물학에 집중하는 생화학 전공 및 생물학 전공자에게 필요한 상위 부문 과정입니다. 전제 조건을 충족하는 다른 학생들은 상위 부문 선택 과목으로 과정을 수강할 수 있습니다. 이 과정은 Cal Poly의 화학 및 생화학과와 생물학과가 공동으로 연계되어 있습니다. 두 학과의 교수진이 매 학기마다 교대로 과정을 가르칩니다(생물학 2/4, 화학 및 생화학 1/4).
수업은 매주 두 번 실험실에서 170분 동안 만나고 일주일에 한 번 50분 강의를 위해 만납니다. 랩 섹션에는 최대 16명의 학생이 포함되며 매 분기마다 2-3개의 랩 섹션이 제공됩니다. 이 과정은 10주 동안 진행되며 기말고사는 10주 마지막 회의에서 시행됩니다. 강의 시간은 실험실에서 수행되는 많은 실험 기술의 이면에 있는 이론과 실험실에서 다루지 않는 분자 생물학의 최신 주제에 대해 토론하는 데 사용됩니다. 실험실의 핵심 커리큘럼은 효모1에서 액틴 유전자를 복제하는 과정을 포함하며, 이는 약 7주(13-14회의 실험실 회의)가 소요됩니다. 이 기술에는 마이크로피펫팅, 효모 RNA 분리, 역전사-중합효소 연쇄 반응(RT-PCR)을 사용한 효모 유전자 증폭, TOPO 클로닝, 청백색 스크리닝, 플라스미드 분리 및 제한 분해 및 PCR에 의한 인서트 검증, 클론의 인실리코 분석, DNA 염기서열 분석 등이 포함됩니다. 과정의 마지막 3주 동안의 커리큘럼은 강사의 재량에 달려 있지만 정의되지 않은 결과로 "독립적인 프로젝트"를 완료하는 학생과 관련이 있습니다.
실험 개요
우리 연구 그룹의 초점 중 하나는 방선균의 생합성 경로입니다. 독립적인 프로젝트를 설계할 때 우리는 학생들이 천연 제품 생합성을 조사하는 연구 프로젝트에 사용하기 위해 Gibson Assembly를 통해 플라스미드를 만드는 것을 구상했습니다. 여기에서 평가된 교육과정 모듈의 반복은 생합성 경로의 조작을 가능하게 하는 플라스미드에 국한된 것이지만, Gibson Assembly 워크플로우는 다른 분자 클로닝 프로젝트에도 매우 적합합니다(그림 2). 워크플로우는 3개의 서로 다른 실험(A, B, C)으로 나뉘어 2개의 강의 기간과 6개의 실험 기간(총 3주)에 걸쳐 완료되었습니다( 보충 파일 1 및 보충 파일 2 참조). 실험에 앞서 학생의 준비를 지원하고 학생의 이해도를 평가하기 위한 워크시트가 있었습니다(보충 파일 3, 보충 파일 4 및 보충 파일 5). 워크플로우는 강사의 필요와 관심에 유연하게 맞는 형식으로 제공됩니다.

그림 2: Gibson Assembly 모듈 워크플로우. 1일차와 4일차는 학생들이 인실리코(in silico ) 염기서열 분석 및 실험 설계를 완료하는 강의 시간입니다. 2-3일차와 5-8일차는 실험실 회의로, Gibson Assembly를 통해 새로운 플라스미드를 복제하는 단계를 수행한 후 분리 및 스크리닝을 수행합니다. 그림 순서도는 학생들이 수행하는 세 가지 실험(A, B, C)으로 그룹화됩니다. 자세한 지침과 프로토콜은 각각 Supplemental File 1 및 Supplemental File 2 로 제공된 강사 및 학생 매뉴얼에서 찾을 수 있습니다. Biorender.com 의 과학 아이콘. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Gibson Assembly 독립 프로젝트 모듈은 2019년 봄 분기에 CHEM/BIO 475에서 처음 시범 운영되었습니다. 2020년과 2021년에는 SARS-CoV-2 전염병으로 인해 이 과정이 온라인으로 진행되었습니다. 2022년과 2023년 봄에 대면 수업이 재개되었을 때, 이 과정에 참여한 학생들은 연구 관련 플라스미드를 복제하는 Gibson Assembly 독립 프로젝트의 학습 결과를 평가하는 연구에 참여하도록 초대되었습니다. 2019년에 학생들은 pKC1132로 복제된 Micromonospora echinospora ATCC 15837 게놈의 유전자 카세트로 구성된 플라스미드 라이브러리를 만들었습니다(그림 3). 이 플라스미드 라이브러리는 천연 산물 TLN-0522015에 대한 추정되는 생합성 유전자 클러스터에서 관심 유전자를 비활성화하기 위해 당사 연구실에서 활용되고 있습니다. 유전자 불활성화 연구를 보완하기 위해 2022년 학생들은 추정되는 TLN-05220 유전자 클러스터의 작은 유전자 라이브러리를 pUC19(Figure 3)로 복제했습니다. 우리 연구 그룹은 단백질 과발현 및 정제를 위해 pET28b를 포함한 발현 벡터로 유전자를 subcloning하기 위해 이러한 플라스미드를 활용했습니다. 2023년 코호트의 학생들은 에폭소미신 합성효소16에 대한 생합성 공학 프로젝트의 지속적인 작업에 기여했습니다. 3-4명으로 구성된 팀으로 학생들은 연구실에서 이러한 효소의 과발현 및 정제를 최적화하기 위해 비리보솜 펩타이드 synthetase17 모듈의 엔지니어링된 도메인을 다양한 단백질 발현 벡터로 복제했습니다(그림 3). 중복성은 각 코호트에 대한 복제 계획에 구축되었습니다. 예를 들어, 2019년 코호트에는 44명의 학생이 포함되어 있었고 15개의 플라스미드가 클로닝을 위해 수업에 할당되었습니다. 따라서, 각 플라스미드의 클로닝을 2회 또는 3회 시도하였다.
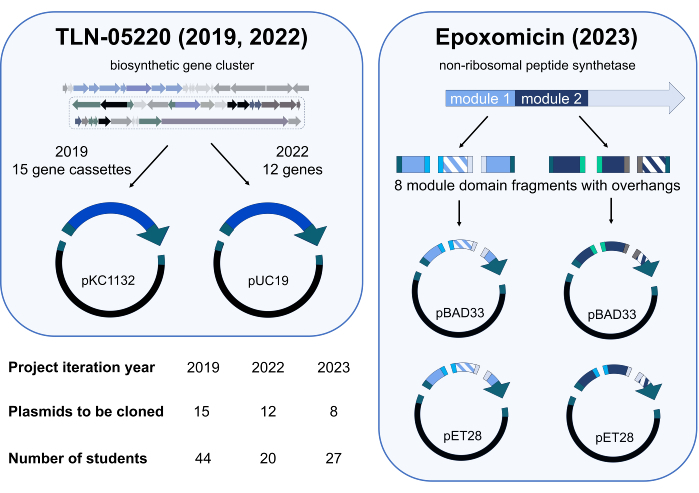
그림 3: 2019년, 2022년 및 2023년 독립 프로젝트 반복 동안 복제된 플라스미드 및 학생 참가자 요약. Gibson Assembly 프로젝트는 세 번 배포되었습니다. 각 제안에서 학생 참가자들은 생합성 경로를 탐구하는 연구 프로젝트에 활용하기 위해 서로 다른 플라스미드 라이브러리를 복제했습니다. 2019년과 2022년의 프로젝트는 두 개의 단편(하나의 유전자 카세트 또는 유전자와 벡터) Gibson Assembly 반응을 포함하는 천연 제품 TLN-0522015 에 대한 지속적인 작업을 지원했습니다. 2023년 프로젝트에는 에폭소마이신 생합성에 관여하는 비리보솜 펩타이드 합성효소(NRPS) 효소의 모듈 1 및 2 내 도메인 스와핑이 포함되었습니다16. 부화한 조각은 스왑된 도메인의 두 개의 서로 다른 돌연변이를 나타내고 단색은 스왑되지 않은 도메인을 나타냅니다. 총 8개의 서로 다른 유전자 단편(모듈 1에 4개, 모듈 2에 4개)이 Gibson Assembly와 호환되는 돌출부로 생성되었습니다. 각 모듈에 대해 3개의 유전자 단편의 2가지 다른 조합을 2개의 서로 다른 벡터(pBAD33 및 pET28, 조립당 총 4개의 단편) 중 하나로 조립하여 8개의 엔지니어링된 NRPS 플라스미드를 생성할 수 있었습니다. Biorender.com 의 과학 아이콘. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
평가 개요
Gibson Assembly 독립 프로젝트가 시작되기 최소 1주일 전에 2022년 및 2023년 코호트의 학생들은 Cal Poly18의 생화학 실험실 과정 조사에서 수행된 연구와 유사하게 설계된 학습 결과 조사에 참여하도록 초대되었습니다. 학생 참가자들은 독립 프로젝트가 시작되기 전에 실험실 회의에서 객관식 사전 설문지를 작성했고, 최종 실험실 회의(즉, Gibson Assembly 모듈을 완료한 후)에서 객관식 사후 설문지를 작성했습니다. 사전 및 사후 설문지는 28개의 동일한 질문으로 구성되었으며 사후 설문지에 2개의 추가 질문(총 30개)이 있었습니다. 분자 복제와 관련된 효소 및 메커니즘(예: 중합효소 연쇄 반응[PCR], Gibson Assembly, 형질전환, 청백색 스크리닝)에 대한 학생들의 지식을 평가하기 위해 10개의 내용 질문이 작성되었습니다. 다음 7개의 질문은 학생들에게 분자 복제 용어(예: DNA 중합효소, 엑소뉴클레아제, 리가아제)에 대한 친숙도를 스스로 평가하도록 요청했습니다. 다음 10개의 질문을 통해 학생들은 분자 복제 기술(예: DNA 염기서열 분석, 제한 분해 반응, 아가로스 젤 전기영동)을 수행할 수 있는 능력을 스스로 평가할 수 있었습니다. 학생들은 또한 분자 클로닝 기술에 대한 지식을 바탕으로 분자 생물학 분야에서 경력을 쌓는 것이 편한지 여부를 보고했습니다. 학생들이 강좌 학습에 대한 자신의 헌신과 강좌가 가치 있는 학습 경험이었는지 스스로 평가할 수 있도록 사후 설문지에 두 개의 추가 질문이 포함되었습니다(보충 파일 6 및 보충 파일 7). 2022년 및 2023년 학생 사전 및 사후 설문지 응답의 모든 데이터는 분석을 위해 결합되었으며 보충 표 S1에서 사용할 수 있습니다.
프로토콜
인간 참가자를 대상으로 한 연구는 Cal Poly의 Research Institutional Review Board(2022-113-CP (IRB))에서 인간 피험자에 의해 검토 및 승인되었습니다. 참가자들은 본 연구에 참여하기 위해 서면 동의서를 제공했습니다.
다음 프로토콜은 강사 준비(1.1–1.3단계), 선형 단편을 얻기 위한 PCR(2.1–2.7단계), Gibson 조립, 형질전환 및 클론 선택(3.1–3.5단계), 플라스미드 분리 및 스크리닝(4.1–4.4단계), 학습 결과 평가(5.1–)5.2). 강사 준비는 프라이머 설계 및 원하는 플라스미드 맵의 in silico 준비의 대표적인 예를 설명합니다. 프로토콜의 모든 섹션은 원하는 다른 플라스미드에 적용할 수 있습니다. 각 학생 실험은 두 번의 3시간 랩 회의로 나뉩니다.
1. 강사 준비
- Benchling Assembly Wizard를 사용한 Gibson Assembly 프라이머 설계
- 게놈 DNA, 플라스미드 및 합성 DNA와 같은 유전자 삽입에 대한 DNA 템플릿 소스를 결정합니다.
메모: 복잡한 게놈 DNA 주형에서 증폭될 유전자의 경우, 먼저 보충 파일 1에 자세히 설명된 바와 같이 NCBI(National Center for Biotechnology Information) BLAST(Basic Local Alignment Search Tool)21을 사용하여 관심 유전자에 특이적인 게놈 DNA 주형 내 프라이머에 대한 고유한 결합 부위를 결정하는 것이 좋습니다. 플라스미드 또는 합성 DNA와 같은 간단한 템플릿의 경우 다음 단계로 진행합니다. - 유전자 삽입물 및 벡터의 DNA 염기서열을 검색합니다(예: 보충 파일 8에서 GenBank 파일로 제공된 폴리케타이드 하이드록실라제 유전자 서열 및 pUC19 서열 참조). 원하는 삽입 염기서열과 원하는 벡터 염기서열을 각각 새로운 DNA 염기서열 파일로 Benchling으로 가져옵니다. 가져온 각 시퀀스를 Gibson Assembly 반응에 포함하도록 엽니다.
메모: GenBank 및 FASTA를 포함한 여러 형식의 DNA 염기서열 파일을 가져올 수 있습니다. 또한 식별 번호가 있는 데이터베이스에서 직접 염기서열을 가져오거나 다른 파일 또는 뷰어에서 복사한 뉴클레오티드 염기서열에 붙여넣을 수 있습니다. - 화면 하단에서 Assembly Wizard 도구를 찾습니다. Assembly Wizard(어셈블리 마법사)를 클릭한 다음 Create New Assembly(새 어셈블리 생성)를 선택합니다. 제공된 옵션에서 Gibson을 선택하고 시작을 클릭하여 어셈블리를 시작합니다.
- 벡터 시퀀스 창에서 벡터 백본에서 어셈블리에 포함할 모든 베이스를 선택합니다. 유전자 삽입물의 3' 말단에 있는 뉴클레오티드 위치(예: pUC19의 위치 657)에서 선택을 시작하고, 포함할 나머지 뉴클레오티드를 모두 선택합니다(예: 전체 pUC19 염기서열을 포함하기 위해 위치 656까지). 선택한 후 화면 하단의 Backbone 탭을 클릭합니다. | Set Fragment.
메모: 벡터의 Sequence Map 과 Plasmid 보기 사이를 전환하여 원형 템플릿의 원하는 모든 베이스를 선택합니다. 또는 Gibson Assembly를 위해 PCR 대신 제한 분해물을 통해 벡터 단편을 준비하려는 경우 제한 효소 절단 부위(예: XbaI)로 백본 염기서열을 선형화합니다. 이렇게 하려면 잘라낸 부위를 클릭하고 Shift 키를 누른 상태에서 잘라낸 부위 를 다시 클릭합니다. 그런 다음 Set Fragment 를 클릭하여 백본을 설정합니다. 벡터별 primer는 backbone이 Assembly Wizard에서 restriction enzyme으로 선형화된 경우 Benchling에 의해 생성되지 않습니다. - 삽입 순서 창에서 어셈블리에 포함할 삽입의 모든 베이스를 선택합니다. 선택한 후 화면 하단의 Insert 탭을 클릭합니다. 프래그먼트를 설정합니다.
- 유전자 삽입이 여러 개인 경우 Assembly Wizard의 오른쪽에 있는 + 버튼을 클릭합니다. 삽입 순서 창에서 어셈블리에 포함할 모든 삽입 베이스를 선택합니다. 선택한 후 화면 하단의 Insert 탭을 클릭합니다. 프래그먼트를 설정합니다.
- 모든 조각이 설정되면 어셈블리의 이름을 원하는 플라스미드 이름으로 바꾸고 Assembly Wizard의 오른쪽에 있는 Assemble을 클릭합니다. Sequence Folder와 Primer Folder에 대해 원하는 폴더 위치를 선택합니다. Create를 클릭하여 재조합 플라스미드 염기서열을 조합합니다.
- 조립된 플라스미드를 열고 Assembly History 를 클릭하여 기본 플라스미드 맵과 플라스미드가 파생된 염기서열에 대한 개요를 확인합니다. 각 단편의 뉴클레오티드 위치가 원하는 설계와 일치하는지 확인합니다.
- Assembly Parameters 탭에서, 어셈블리를 위해 설계된 프라이머의 이름을 봅니다. 각 삽입물을 위해 디자인된 2개의 뇌관이 (앞으로와 뒤로) 있을 것이라는 점을 주지하고, 뇌관 이름은 DNA 순서 파일의 제목에서 파생될 것이다. 또한 프라이머 용융 온도와 제안된 어닐링 온도가 이 창에서 호환되는지 확인하십시오(보충 파일 8, 대표 프라이머 참조).
- 프라이머 용융 온도 또는 어닐링 온도가 이상적이지 않은 경우, 프라이머 염기서열 파일 내에서 프라이머 염기서열을 수동으로 조정(예: 용융 온도를 낮추기 위해 뉴클레오티드 제거)합니다. 또는 Re-open을 클릭하고 Assembly Wizard의 Assemble 버튼 옆에 있는 Enzyme/Primer Settings 버튼을 클릭하여 프라이머 설정을 조정합니다. 그런 다음 조립 단계를 반복하고 프라이머 염기서열을 다시 검사합니다.
- plasmids 라이브러리가 동일한 vector backbone에 복제된 많은 다른 유전자로 복제되는 경우,
- 각 경우에 벡터에 대해 동일한 프라이머 쌍을 사용합니다. 이렇게 하려면 삽입물에 특이적인 돌출부를 설치할 벡터 프라이머에서 모든 뉴클레오티드를 수동으로 제거합니다. 이러한 뉴클레오티드는 프라이머의 5' 말단에서 발견됩니다. 제거 후 프라이머 용융 온도와 어닐링 온도가 호환되는지 확인하십시오( 보충 파일 8, 대표 프라이머 참조).
- 모든 인서트 프라이머 쌍이 매우 유사한 용융 온도와 어닐링 온도를 갖는지 확인하십시오.
메모: 이를 통해 모든 쌍의 어닐링 온도 최적화의 필요성이 줄어들고 동일한 thermocycler 프로그램을 사용하여 모든 student PCR 반응을 동시에 순환할 수 있습니다.
- 원하는 플라스미드에 대한 primer design이 완료되면 Assembly Wizard에서 Finalize를 클릭합니다. DNA 종합 회사에서 프라이머를 주문하고 삽입 및 벡터에 대한 DNA 템플릿의 샘플을 검색합니다.
- DNA 템플릿과 함께 gradient PCR 실험을 사용하여 최적의 어닐링 온도에 대한 insert- 및 vector-specific primer pair를 모두 테스트할 수 있습니다. 처음에 3 °C± Benchling에서 권장하는 어닐링 온도를 테스트하십시오.
- 어닐링 온도가 결정되면 프라이머 용액, DNA 템플릿 및 학생의 PCR 반응에 필요한 시약의 부분 표본을 생성합니다.
- 게놈 DNA, 플라스미드 및 합성 DNA와 같은 유전자 삽입에 대한 DNA 템플릿 소스를 결정합니다.
- 실험을 시작하기 전에 학생들에게 워크시트를 제공하여 준비를 지원하고 이해도를 평가합니다(보충 파일 4, 보충 5 및 보충 6).
- 학습 성과에 대한 데이터 수집
메모: Gibson Assembly 모듈에서 학습 결과 데이터를 수집하고 결과를 게시하는 데 관심이 있는 경우 기관의 정책에 따라 인간 피험자를 대상으로 한 연구에 대한 승인을 받으십시오.- 학생들에게 Gibson Assembly 독립 프로젝트가 시작되기 최소 1주일 전에 학습 결과 조사에 참여하도록 초대합니다.
- 학생 참가자들이 독립 프로젝트를 시작하기 전에 실험실 회의에서 객관식 사전 설문지(보충 파일 6)를 완료하도록 합니다.
2. 학생 실험 A: 선형 단편을 얻기 위한 PCR
- 강사로부터 PCR에 대한 프라이머와 템플릿 DNA 솔루션을 얻습니다.
- 학생 교재의 지침에 따라 DNA 중합효소 MasterMix, DNA 템플릿, 정방향 및 역방향 프라이머, 뉴클레아제가 없는 물을 포함한 25μL 반응 피펫.
- Supplemental File 2의 학생 설명서에 따라 thermocycler에서 반응을 순환합니다. 반응의 어닐링 온도가 프라이머에 적합하고 확장 시간이 원하는 앰플리콘의 길이에 적합한지 확인합니다. 자세한 지침은 Supplemental File 2 및 Supplemental File 3을 참조하십시오.
- PCR 반응은 다음 실험실 기간까지 4 °C 또는 -20 °C에서 보관합니다.
- 아가로스 젤 전기영동을 통해 각 반응의 5 μL를 분석합니다. 겔이 작동하는 동안 플라스미드 DNA를 주형으로 사용하는 각 반응에 1μL의 DpnI 제한 효소를 추가합니다. 이 혼합물을 37 ° C에서 1 시간 동안 배양합니다.
메모: DpnI는 남아 있는 원형 플라스미드 DNA 템플릿을 분해하여 형질전환 단계에서 위양성을 얻을 가능성을 줄입니다. - PCR이 성공적이고 올바른 증폭이 이루어졌는지 확인하기 위해 겔을 이미지화합니다. 상업적으로 이용 가능한 PCR 정제 키트로 성공적인 PCR 반응을 정제하십시오. 후속 Gibson 어셈블리 계산에 사용하기 위해 미량 분광 광도계를 사용하여 정제된 PCR 산물의 농도(ng/μL)를 측정합니다.
- 다음 수업 회의 전에 Gibson Assembly에 대한 반응 레시피를 설계합니다( 보충 파일 2 및 보충 파일 4 참조).
3. 학생 실험 B: Gibson 조립, 변형 및 클론 선택
- 설계된 레시피에 따라 Gibson Assembly 반응을 피펫팅합니다. 50 °C에서 15분 동안 반응을 배양합니다. 반응이 배양하는 동안 화학적으로 유능한 대장균 세포를 얼음에서 해동하여 형질전환을 준비합니다.
- 열 충격을 통해 2μL의 Gibson 어셈블리를 화학적으로 적합한 셀로 변환합니다. 형질전환된 세포 100μL를 두 개의 선택 플레이트에 피펫팅하고 멸균된 비드로 도포합니다. Luria-Bertani(LB) 한천에 남아 있는 세포의 10-4, 10-5 및 10-6 희석액을 준비합니다. LB(선택 없음)에 각 직렬 희석액을 100μL씩 플레이트화하고 멸균된 비드로 펴 바릅니다.
메모: 이 단계를 통해 변환 효율성을 계산할 수 있습니다. - 37 °C에서 밤새 플레이트를 배양하고 다음 수업 시간까지 플레이트를 4 °C에서 보관하십시오.
- 모든 플레이트의 콜로니를 세고 보충 파일 2의 지시에 따라 변형 효율을 계산합니다. 선택 플레이트에서 4개의 콜로니가 있는 하나의 선택적 플레이트를 다시 연결하고 이 플레이트를 37 °C에서 하룻밤 동안 배양합니다.
메모: 이것은 포지티브 클론의 백업 문화로 작용합니다. - 마커를 사용하여 선택 플레이트에서 4개의 고유한 군체를 선택하고, 동그라미를 치고, 학생의 이니셜과 숫자(예: ABC1)로 레이블을 지정합니다. 항생제가 포함된 새로운 LB-한천 플레이트를 검색하고, 마커를 사용하여 플레이트를 사분면(즉, 4분)으로 나눈 다음, 각 콜로니의 ~1/2을 사용하여 각각 라벨링된 사분면에 다시 줄무늬를 적용하고 다른 1/2을 사용하여 선택한 4개의 콜로니 각각에 대해 5mL 액체 LB 배양을 접종합니다. 튜브에 해당 콜로니 정체성(예: ABC1)을 표시하고 올바른 농도의 항생제를 선택하여 선택해야 합니다. 액체 배양물을 37 °C의 진탕 인큐베이터에서 하룻밤 동안 배양하고 한천 플레이트를 37 °C의 정적 인큐베이터에서 배양합니다. 액체 배양 및 플레이트는 다음 등급 기간까지 4 °C에서 보관하십시오.
4. 학생 실험 C: 플라스미드 분리 및 스크리닝
- miniprep 키트를 사용하여 실험 B의 액체 배양에서 플라스미드 DNA를 분리합니다. 분리된 플라스미드의 농도를 측정합니다(ng/μL 단위).
- 분리된 플라스미드를 분석하기 위한 제한 분해 또는 PCR 스크리닝을 설계합니다( 보충 파일 2 의 자세한 지침 및 보충 파일 5의 보고서 기대치 참조).
- 피펫팅: 제한 반응 또는 설계된 레시피에 따른 반응을 분해하고 이전에 설계된 온도와 지속 시간에 배양합니다.
- 아가로스 겔 전기영동을 통해 결과를 분석합니다.
5. 평가
- 학생 참가자들이 Gibson Assembly 모듈이 완료된 후 실험실 회의에서 객관식 사후 설문지(보충 파일 7)를 완료하도록 합니다.
- 분석을 위해 사전 및 사후 설문지 응답의 모든 데이터를 결합합니다.
- 양측 쌍 t-검정과 Cohen의 d 효과 크기를 사용하여 설문지 간 각 개별 내용 질문의 평균 점수 증가에 대한 통계적 유의성을 조사합니다.
- 변화의 정도와 통계적 유의성을 결정하기 위해 각 개별 용어 및 기술에 대한 학생 자신감 증가를 평가합니다.
- NLG(Normalized Learning Gain) 및 쌍을 이루지 않은 t-검정을 사용하여 학생의 배경과 학업 요인이 학생 학습에 미치는 영향을 확인합니다.
- 평균 점수를 평가하여 프로젝트 경험 및 분자 생물학 경력에 대한 학생들의 태도 를 평가합니다.
결과
복제에 대한 학생의 성공
Gibson Assembly 모듈(2019, 2022, 2023)의 각 반복에서 학생들은 자신의 결과를 요약한 보고서를 준비하도록 요청받았습니다. 2019년에는 학생 44명 중 36명(81.8%)이 실험 C를 위해 설계한 스크리닝 결과를 기반으로 플라스미드를 성공적으로 복제했다고 보고했습니다. 2022년 학생 20명 중 14명(70.0%)이 할당된 구조를 복제하는 데 성공했다고 보?...
토론
여기에서는 과정 기반 연구 환경에서 Gibson Assembly를 통해 학생들에게 분자 복제를 가르치는 조정 가능한 학부 교실 실험실 프로젝트를 제안합니다. 총 28개의 새로운 플라스미드가 학부생들에 의해 교실 환경에서 복제되었습니다. 개별 학생이 보고한 성공률은 3개 코호트에서 44.4%에서 81.8% 사이였으며 전체 클로닝 성공률은 80%였습니다(할당된 총 35개의 플라스미드 중 28?...
공개
저자는 경쟁하는 재정적 이해관계나 다른 이해상충이 없음을 선언합니다.
감사의 말
저자들은 기술 지원을 해준 안드레아 라우브셔(Andrea Laubscher)와 교수 실습 구현 및 학습 결과 평가에 대한 유용한 토론을 해준 마이클 블랙(Michael Black), 샌디 클레멘트(Sandi Clement), 제이빈 오자(Javin Oza)에게 감사의 뜻을 전한다. 저자는 2019년, 2022년, 2023년 코호트에서 학습 결과 연구에 참여한 모든 학생과 교육 실험실 구현을 위한 시약 준비를 도운 연구 학생 Nathan Kuhn과 Aayushi Adettiwar에게 감사의 뜻을 전합니다. 저자는 또한 William and Linda Frost Fund, Center for Applications in Biotechnology의 Chevron Biotechnology Applied Research Endowment Grant 및 National Science Foundation(NSF-1708919 및 NSF-2300890)의 자금 지원을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
참고문헌
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유