Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Research Article
Mejorar los resultados de los estudiantes con una experiencia de investigación de pregrado basada en cursos de clonación molecular adaptable
* Estos autores han contribuido por igual
En este artículo
Resumen
Se empleó un módulo de clonación molecular adaptable de Gibson Assembly en un formato de experiencia de investigación de pregrado (CURE) basado en cursos para estudiantes de cursos de laboratorio de biología molecular. La evaluación de los resultados de aprendizaje de los estudiantes mostró una mejor comprensión y confianza en la clonación molecular después de completar el CURE, y se clonaron nuevos plásmidos para la investigación de biosíntesis de productos naturales.
Resumen
El avance continuo de las técnicas de biología molecular requiere que los planes de estudio de biología molecular se refinen regularmente para preparar de manera efectiva a los estudiantes para ingresar a la fuerza laboral con competencias modernas. En particular, la aparición de Gibson Assembly, una técnica de clonación molecular altamente personalizable y adaptable, ha hecho avanzar el panorama de la clonación molecular en numerosos entornos de investigación. Por lo tanto, creamos un módulo de clonación de Gibson Assembly para su implementación en un curso de laboratorio de biología molecular en la Universidad Estatal Politécnica de California, San Luis Obispo, y evaluamos los resultados de aprendizaje de los estudiantes del módulo. A lo largo de tres iteraciones del curso, los estudiantes participaron en un proyecto independiente basado en experimentos que implicó la clonación de tres bibliotecas de plásmidos únicas para apoyar proyectos de investigación en biosíntesis de productos naturales. Los estudiantes recibieron cuestionarios previos y posteriores para evaluar su comprensión de la clonación molecular y su confianza en los términos y técnicas de biología molecular. Las respuestas de los estudiantes mostraron un aumento significativo tanto en el aprendizaje de los conceptos de clonación molecular como en la confianza autoinformada con los términos y técnicas de clonación molecular. Este marco de módulos se puede generalizar para enseñar Gibson Assembly para diversas aplicaciones, proporcionando a los instructores un conjunto de herramientas para enseñar una tecnología de clonación adaptable y emergente mientras avanzan en sus proyectos de investigación.
Introducción
La formación de los estudiantes universitarios en conceptos fundamentales de biología molecular y técnicas de laboratorio es crucial para su desarrollo científico y profesional, ya que estas metodologías son comunes en diversos entornos de investigación, incluidos el mundo académico y la industria. Como tal, los estudiantes de las carreras de biología (concentración de biología molecular y celular) y bioquímica en la Universidad Estatal Politécnica de California, San Luis Obispo (Cal Poly) deben tomar un curso de laboratorio de biología molecular de la división superior para aprender y aplicar estos temas (CHEM / BIO 475). Se ha desarrollado previamente un plan de estudios básico para este curso en el que los estudiantes realizan la clonación basada en topoisomerasa (TOPO) para ensamblar un plásmido que contiene actina que se prepara a partir de una plantilla de ADN complementario (ADNc)de levadura 1. Los estudiantes diseñan experimentos basados en preguntas que imitan hipótesis de investigación auténticas, aumentando su familiaridad con las prácticas de laboratorio y el aprendizaje basado en la indagación. El avance continuo en el campo de la biología molecular requiere que los planes de estudio correspondientes se adapten para preparar a los estudiantes con competencias modernas para la fuerza laboral. En particular, el uso de Gibson Assembly se ha vuelto más predominante en la comunidad científica; Si bien el método se estableció originalmente para sintetizar cromosomas artificiales2, más de 5,000 publicaciones en el momento de este informe han hecho referencia al trabajo original de Gibson et al. Gibson Assembly es único en comparación con las metodologías de clonación tradicionales: es altamente personalizable y puede ligar fácilmente múltiples fragmentos de ADN lineal sin necesidad de sitios de restricción para producir las uniones. Por lo tanto, vimos la oportunidad de renovar el plan de estudios de CHEM/BIO 475 para incorporar técnicas modernas de clonación molecular y mejorar el modelo de curso basado en la investigación.
Se ha establecido que las experiencias de investigación de los estudiantes contribuyen a una mayor comprensión conceptual, desarrollo de habilidades y persistencia en ciencias3, sin embargo, no todos los estudiantes de pregrado tienen la oportunidad de participar directamente en un laboratorio de investigación. Para abordar el desafío de la capacidad limitada de los estudiantes en los laboratorios de investigación, se han desarrollado y empleado experiencias de investigación de pregrado (CURE) basadas en cursos para aumentar la accesibilidad a la ciencia a través de la investigación auténtica en el aula. Si bien las CURE varían en su implementación, se han establecido prácticas comunes que abordan los cinco principios de la investigación científica. En un CURE bien diseñado, los estudiantes 1) utilizarán prácticas científicas, 2) colaborarán en un proyecto de investigación, 3) intentarán hacer nuevos descubrimientos, 4) contribuirán al trabajo relevante fuera del aula, y 5) reevaluarán y revisarán hipótesis y métodos en caso de fracaso experimental4. Al igual que las experiencias tradicionales de investigación de los estudiantes en un laboratorio, se ha demostrado que los CURE fortalecen la confianza de los estudiantes en la ciencia, las habilidades científicas, la propiedad de proyectos y la persistencia en ciencia, tecnología, ingeniería y matemáticas (STEM)5. Si bien las curas que involucran clonación molecular han sido reportadas previamente 6,7,8,9,10,11,12,13, no tenemos conocimiento de ninguna que enfatice la adaptabilidad de Gibson Assembly para hacer una biblioteca de plásmidos de investigación auténticos.
Aquí, informamos de una expansión del actual plan de estudios CHEM/BIO 475 basado en la investigación en la Universidad Estatal Politécnica de California, San Luis Obispo, con dos mejoras importantes: la experiencia práctica con el ensamblaje de Gibson y la participación de los estudiantes en un CURE, que ha proporcionado construcciones plasmídicas originales para proyectos de investigación financiados por la Fundación Nacional de Ciencias (NSF-1708919 y NSF-2300890). A lo largo de tres implementaciones de este plan de estudios, los estudiantes han contribuido a dos proyectos de investigación distintos centrados en la biosíntesis de productos naturales de moléculas bioactivas producidas por Actinomycetota. Los productos naturales a menudo contienen farmacóforos con actividades antibióticas, antifúngicas y/o anticancerígenas, lo que da a estas pequeñas moléculas importancia en los esfuerzos de descubrimiento de fármacos y potencial de relevancia clínica14. Esta investigación requiere la creación de bibliotecas de plásmidos que permitan investigar tanto la función como el potencial de ingeniería de las enzimas biosintéticas bacterianas. En este CURE, los estudiantes diseñaron y realizaron experimentos de ensamblaje de Gibson para clonar las bibliotecas de plásmidos únicas pertinentes a estos proyectos de investigación (Figura 1). Además, el formato y diseño del módulo es distintivo porque es fácilmente adaptable para generar cualquier plásmido de interés para otros proyectos de investigación.
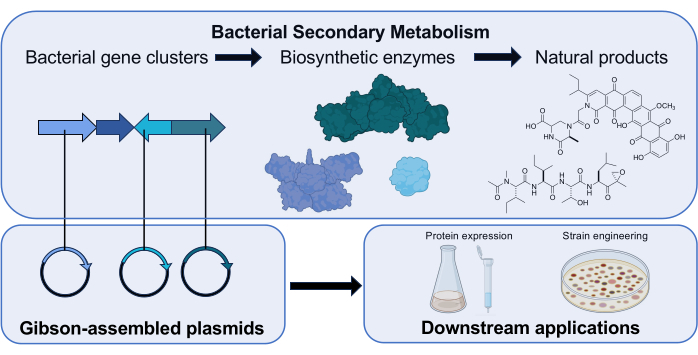
Figura 1: Descripción general del papel de Gibson Assembly en nuestro laboratorio de investigación. Actinomycetota produce productos naturales de moléculas pequeñas con bioactividades clínicamente relevantes utilizando grupos de genes que codifican enzimas biosintéticas. En nuestra investigación, los plásmidos que contienen un gen biosintético se ensamblan a través de Gibson Assembly para investigaciones posteriores de la función de la enzima codificada. Iconos de la ciencia de Biorender.com. Haga clic aquí para ver una versión más grande de esta figura.
Descripción general del laboratorio
El Laboratorio de Biología Molecular es un curso de división superior requerido para los estudiantes de bioquímica y biología con una concentración en biología molecular y celular. Otros estudiantes que cumplan con los requisitos previos son bienvenidos a tomar el curso como una materia optativa de la división superior. El curso está co-listado entre el Departamento de Química y Bioquímica y el Departamento de Biología de Cal Poly. Los profesores de ambos departamentos se turnan para impartir el curso cada trimestre (dos trimestres de Biología, un trimestre de Química y Bioquímica).
La clase se reúne en el laboratorio dos veces por semana durante períodos de 170 minutos y una vez a la semana para una conferencia de 50 minutos. Las secciones de laboratorio contienen un máximo de 16 estudiantes y se ofrecen 2-3 secciones de laboratorio cada trimestre. El curso tiene una duración de 10 semanas y el examen final se administra durante la última reunión de la semana 10. El tiempo de la conferencia se dedica a discutir la teoría detrás de muchas de las técnicas experimentales realizadas en el laboratorio, así como temas actuales en biología molecular que no se cubren en el laboratorio. El plan de estudios básico del laboratorio abarca el proceso de clonación del gen de actina de la levadura1, que dura aproximadamente 7 semanas (13-14 reuniones de laboratorio). Las técnicas incluyen el micropipeteo, el aislamiento de ARN de levadura, la amplificación de un gen de levadura mediante la reacción en cadena de la polimerasa con transcriptasa inversa (RT-PCR), la clonación de TOPO, el cribado azul-blanco, el aislamiento de plásmidos y la verificación del inserto mediante digestión de restricción y PCR, análisis in silico de clones y análisis de secuencias de ADN. El plan de estudios de las últimas 3 semanas del curso queda a discreción del instructor, pero implica que los estudiantes completen un "proyecto independiente" con resultados indefinidos.
Descripción general del experimento
Uno de los focos de nuestro grupo de investigación son las rutas biosintéticas en Actinomycetota. Al diseñar el proyecto independiente, imaginamos que los estudiantes crearan plásmidos a través de Gibson Assembly para su uso en nuestros proyectos de investigación que investigan la biosíntesis de productos naturales. Si bien las iteraciones del módulo del curso que se evalúan aquí eran particulares de los plásmidos que permiten la manipulación de vías biosintéticas, el flujo de trabajo de Gibson Assembly es enormemente adaptable para otros proyectos de clonación molecular (Figura 2). El flujo de trabajo se dividió en tres experimentos diferentes (A, B y C) que se completaron durante dos períodos de clase y seis períodos de laboratorio (3 semanas en total) (consulte el Archivo Suplementario 1 y el Archivo Suplementario 2). Los experimentos fueron precedidos por hojas de trabajo para apoyar la preparación de los estudiantes y evaluar su comprensión (Archivo Suplementario 3, Archivo Suplementario 4 y Archivo Suplementario 5). El flujo de trabajo se presenta en un formato que es flexible a las necesidades e intereses del instructor.

Figura 2: Flujo de trabajo del módulo de ensamblaje de Gibson. El día 1 y el día 4 son períodos de clase en los que los estudiantes completan el análisis de secuencias in silico y el diseño experimental. Los días 2 y 3 y 5 a 8 son reuniones de laboratorio en las que se llevan a cabo los pasos para clonar plásmidos nuevos a través de Gibson Assembly, seguidos de aislamiento y detección. El diagrama de flujo pictórico se agrupa por los tres experimentos que realizan los estudiantes (A, B y C). Se pueden encontrar instrucciones y protocolos más detallados en los Manuales del Instructor y del Estudiante que se proporcionan como Archivo Complementario 1 y Archivo Suplementario 2, respectivamente. Iconos de la ciencia de Biorender.com. Haga clic aquí para ver una versión más grande de esta figura.
El módulo de proyecto independiente Gibson Assembly se probó por primera vez en el trimestre de primavera de 2019 en CHEM/BIO 475. En 2020 y 2021, el curso se impartió en línea debido a la pandemia de SARS-CoV-2. Cuando se reanudó la enseñanza presencial en la primavera de 2022 y 2023, se invitó a los estudiantes del curso a participar en un estudio que evaluaba los resultados de aprendizaje de un proyecto independiente de Gibson Assembly en el que se clonarían plásmidos originales relevantes para la investigación. En 2019, los estudiantes crearon una biblioteca de plásmidos compuesta por casetes de genes del genoma de Micromonospora echinospora ATCC 15837 que se clonaron en pKC1132 (Figura 3). Esta biblioteca de plásmidos se está utilizando en nuestro laboratorio de investigación para inactivar genes de interés en un grupo de genes biosintéticos putativos para el producto natural TLN-0522015. Para complementar nuestros estudios de inactivación génica, los estudiantes clonaron en 2022 una pequeña biblioteca de genes del grupo de genes TLN-05220 putativo en pUC19 (figura3); nuestro grupo de investigación ha utilizado estos plásmidos para subclonar genes en vectores de expresión, incluido pET28b, para la sobreexpresión y purificación de proteínas. Los estudiantes de la cohorte de 2023 contribuyeron al trabajo en curso en un proyecto de ingeniería biosintética sobre la epoxomicinasintetasa 16. En equipos de 3 a 4 estudiantes, los estudiantes clonaron dominios de ingeniería de péptido sintetasa no ribosómico17 módulos en varios vectores de expresión de proteínas para optimizar la sobreexpresión y purificación de estas enzimas en nuestro laboratorio de investigación (Figura 3). La redundancia se incorporó en el plan de clonación de cada cohorte. Por ejemplo, la cohorte de 2019 contenía 44 estudiantes y se asignaron 15 plásmidos a la clase para su clonación. Por lo tanto, la clonación de cada plásmido se intentó dos o tres veces.
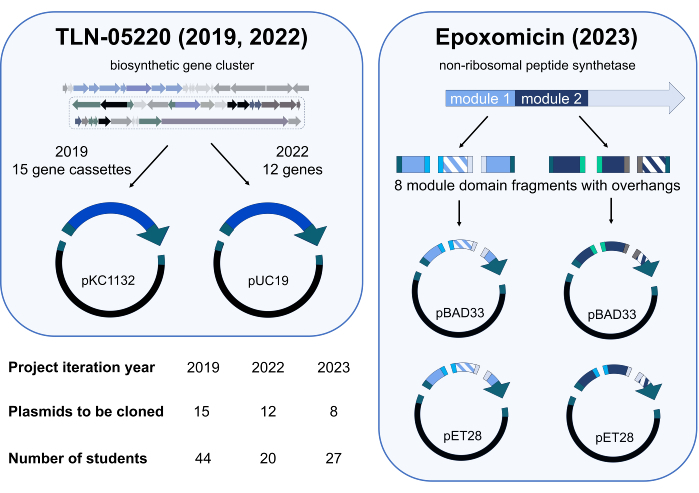
Figura 3: Resumen de plásmidos clonados y estudiantes participantes durante las iteraciones de proyectos independientes de 2019, 2022 y 2023. El proyecto de ensamblaje de Gibson se ha implementado tres veces. En cada oferta, los estudiantes participantes clonaron una biblioteca diferente de plásmidos para utilizarla en proyectos de investigación que exploran las vías biosintéticas. Los proyectos de 2019 y 2022 respaldaron nuestro trabajo en curso sobre el producto natural TLN-0522015 con reacciones de ensamblaje de Gibson de dos fragmentos (un casete de genes o un gen y un vector). El proyecto de 2023 consistió en el intercambio de dominios dentro de los módulos 1 y 2 de una enzima péptido sintetasa no ribosómico (NRPS) que participa en la biosíntesis de epoxomicina16. Los fragmentos sombreados representan dos mutantes diferentes del dominio intercambiado, y los colores sólidos representan dominios que no se intercambiaron. En total, se generaron ocho fragmentos de genes diferentes (cuatro para el módulo 1 y cuatro para el módulo 2) con voladizos compatibles con el ensamblaje de Gibson. Para cada módulo, se ensamblaron dos combinaciones diferentes de tres fragmentos de genes con uno de dos vectores diferentes (pBAD33 y pET28, cuatro fragmentos totales por ensamblaje), para el potencial de generar ocho plásmidos NRPS diseñados. Iconos de la ciencia de Biorender.com. Haga clic aquí para ver una versión más grande de esta figura.
Resumen de la evaluación
Al menos 1 semana antes del inicio del proyecto independiente de Gibson Assembly, se invitó a los estudiantes de las cohortes de 2022 y 2023 a participar en una investigación de resultados de aprendizaje, diseñada de manera similar a un estudio realizado en un curso de laboratorio de encuesta de bioquímica en Cal Poly18. Los estudiantes participantes completaron un cuestionario previo de opción múltiple en la reunión de laboratorio antes del inicio del proyecto independiente y un cuestionario posterior de opción múltiple durante la reunión final de laboratorio (es decir, después de haber completado el módulo de ensamblaje de Gibson). Los cuestionarios pre y post constaron de 28 preguntas idénticas con dos preguntas adicionales en el post-cuestionario (30 en total). Se escribieron diez preguntas de contenido para evaluar el conocimiento de los estudiantes sobre las enzimas y los mecanismos involucrados en la clonación molecular (p. ej., reacción en cadena de la polimerasa [PCR], ensamblaje de Gibson, transformación, detección azul-blanco). En las siguientes siete preguntas se pidió a los estudiantes que autoevaluaran su familiaridad con los términos de clonación molecular (p. ej., ADN polimerasa, exonucleasa, ligasa). Las siguientes 10 preguntas permitieron a los estudiantes autoevaluar su capacidad para realizar técnicas de clonación molecular (por ejemplo, análisis de secuencias de ADN, reacciones de digestión de restricción, electroforesis en gel de agarosa). Los estudiantes también informaron si se sentirían cómodos siguiendo una carrera en biología molecular basándose en su conocimiento de las técnicas de clonación molecular. Se incluyeron dos preguntas adicionales en el cuestionario posterior para que los estudiantes autoevaluaran su compromiso con el aprendizaje en el curso y si el curso fue una experiencia de aprendizaje valiosa (Archivo Suplementario 6 y Archivo Suplementario 7). Todos los datos de las respuestas de los estudiantes antes y después del cuestionario de 2022 y 2023 se combinaron para su análisis y están disponibles en la Tabla Complementaria S1.
Protocolo
Los estudios con participantes humanos fueron revisados y aprobados por sujetos humanos en la Junta de Revisión Institucional de Investigación de Cal Poly (2022-113-CP (IRB)). Los participantes dieron su consentimiento informado por escrito para participar en este estudio.
El siguiente protocolo describe la preparación del instructor (pasos 1.1-1.3), las acciones de los estudiantes para un módulo didáctico de tres experimentos que incluye PCR para obtener fragmentos lineales (pasos 2.1-2.7), ensamblaje, transformación y selección de clones de Gibson (pasos 3.1-3.5), aislamiento y cribado de plásmidos (pasos 4.1-4.4) y evaluación de los resultados de aprendizaje (5.1–5.2). La preparación del instructor describe un ejemplo representativo de diseño de cebadores y preparación de un mapa de plásmidos deseado en silico. Todas las secciones del protocolo son adaptables para otros plásmidos deseados. Cada experimento de los estudiantes se divide en dos reuniones de laboratorio de 3 horas.
1. Preparación del instructor
- Diseño de imprimación de ensamblaje Gibson con el asistente de ensamblaje de bancos
- Determine la fuente de la plantilla de ADN para las inserciones de genes, como el ADN genómico, el plásmido y el ADN sintético.
NOTA: En el caso de los genes que se amplificarán a partir de plantillas complejas de ADN genómico, se recomienda determinar primero los sitios de unión únicos para los cebadores dentro de la plantilla de ADN genómico que sean específicos para los genes de interés, utilizando la Herramienta de Búsqueda Básica de Alineación Local (BLAST) del Centro Nacional de Información Biotecnológica (NCBI)21, como se detalla en el Archivo Suplementario 1. Para plantillas simples como plásmidos o ADN sintético, continúe con el siguiente paso. - Recupere las secuencias de ADN de las inserciones de genes y el vector (por ejemplo, consulte la secuencia del gen de la policétido hidroxilasa y la secuencia pUC19 proporcionadas como archivos GenBank en el Archivo Suplementario 8). Importe la(s) secuencia(s) de inserción deseada(s) y la secuencia vectorial deseada a Benchling, cada una como un nuevo archivo de secuencia de ADN. Abra cada secuencia importada para incluirla en la reacción del ensamblaje de Gibson.
NOTA: Se pueden importar archivos de secuencias de ADN en varios formatos, incluidos GenBank y FASTA. También se puede importar una secuencia directamente desde una base de datos con un número de accesión o pegar una secuencia de nucleótidos copiada de otro archivo o visor. - En la parte inferior de la pantalla, localice la herramienta Asistente para ensamblajes . Haga clic en Asistente para ensamblaje y, a continuación, seleccione Crear nuevo ensamblaje. De las opciones proporcionadas, seleccione Gibson y haga clic en Iniciar para comenzar el ensamblaje.
- En la ventana de secuencia vectorial , seleccione todas las bases que se incluirán en el ensamblaje de la columna vertebral vectorial. Comience la selección en la posición del nucleótido en el extremo 3' del inserto del gen (por ejemplo, la posición 657 de pUC19) y seleccione todos los nucleótidos restantes que se incluirán (por ejemplo, a través de la posición 656 para incluir la secuencia completa de pUC19). Una vez seleccionado, haga clic en la pestaña Backbone en la parte inferior de la pantalla| Establecer fragmento.
NOTA: Alterne entre las vistas Mapa de secuencia y Plásmido del vector para seleccionar todas las bases deseadas de una plantilla circular. Alternativamente, si planea preparar el fragmento de vector a través de un resumen de restricción (en lugar de PCR) para el ensamblaje de Gibson, linealice la secuencia principal con un sitio de corte de enzima de restricción (por ejemplo, XbaI). Para ello, haga clic en el sitio de corte, mantenga presionada la tecla Mayús y vuelva a hacer clic en el sitio de corte . A continuación, haga clic en Establecer fragmento para establecer la columna vertebral. Tenga en cuenta que Benchling no generará cebadores específicos de vectores si la columna vertebral está linealizada con una enzima de restricción en el Asistente para ensamblaje. - En la ventana de secuencia de inserción , seleccione todas las bases de la plaquita que se incluirán en el ensamblaje. Una vez seleccionado, haga clic en la pestaña Insertar en la parte inferior de la pantalla | Establecer fragmento.
- En el caso de inserciones de varios genes, haga clic en el botón + en el lado derecho del Asistente de ensamblaje. En la ventana de secuencia de inserción , seleccione todas las bases de inserción que se incluirán en el ensamblaje. Una vez seleccionado, haga clic en la pestaña Insertar en la parte inferior de la pantalla | Establecer fragmento.
- Una vez que se hayan establecido todos los fragmentos, cambie el nombre del ensamblaje al nombre de plásmido deseado y haga clic en Ensamblar a la derecha del Asistente para ensamblaje. Seleccione la ubicación de carpeta deseada tanto para la carpeta de secuencia como para la carpeta de cebador. Haga clic en Crear para ensamblar la secuencia de plásmido recombinante.
- Abra el plásmido ensamblado y haga clic en Historial de ensamblaje para ver un mapa de plásmido básico y una descripción general de las secuencias de las que se derivó. Asegúrese de que las posiciones de los nucleótidos de cada fragmento coincidan con el diseño deseado.
- En la pestaña Parámetros de ensamblaje , vea los nombres de los cebadores diseñados para el ensamblaje. Tenga en cuenta que habrá dos cebadores diseñados para cada inserción (uno hacia adelante y otro hacia atrás), y los nombres de los cebadores se derivarán de los títulos de los archivos de secuencia de ADN. También asegúrese de que las temperaturas de fusión del cebador y las temperaturas de recocido sugeridas sean compatibles en esta ventana (consulte el Archivo Suplementario 8, Imprimaciones representativas).
- Si las temperaturas de fusión del cebador o del recocido no son ideales, ajuste manualmente las secuencias del cebador (por ejemplo, eliminando nucleótidos para reducir la temperatura de fusión) dentro de los archivos de secuencia del cebador. Alternativamente, haga clic en Volver a abrir y ajuste la configuración del cebador haciendo clic en el botón Configuración de enzima/cebador junto al botón Ensamblar en el Asistente de ensamblaje. A continuación, repita el paso de montaje y vuelva a examinar las secuencias de cebadores.
- Si se va a clonar una biblioteca de plásmidos con muchos genes diferentes clonados en la misma columna vertebral del vector,
- Utilice el mismo par de cebadores para el vector en cada caso. Para ello, retire manualmente todos los nucleótidos de los cebadores vectoriales que instalarían los voladizos específicos de la inserción. Estos nucleótidos se encontrarían en el extremo 5' del cebador. Después de la extracción, asegúrese de que las temperaturas de fusión del cebador y las temperaturas de recocido sean compatibles (consulte el Archivo Suplementario 8, Imprimaciones Representativas).
- Asegúrese de que todos los pares de cebadores de plaquitas tengan temperaturas de fusión y recocido muy similares.
NOTA: Esto reducirá la necesidad de optimizar la temperatura de recocido de cada par y permite que todas las reacciones de PCR de los estudiantes se ciclen al mismo tiempo utilizando el mismo programa de termociclador.
- Una vez que se haya completado el diseño del cebador para los plásmidos deseados, haga clic en Finalizar en el Asistente de ensamblaje. Pida los cebadores a una empresa de síntesis de ADN y recupere muestras de las plantillas de ADN para los insertos y el vector.
- Pruebe los pares de cebadores específicos de insertos y vectores para obtener temperaturas óptimas de recocido mediante un experimento de PCR en gradiente con las plantillas de ADN. Pruebe inicialmente las temperaturas de recocido recomendadas por el banco ± 3 °C.
- Una vez que se hayan determinado las temperaturas de recocido, cree alícuotas de soluciones de cebadores, plantillas de ADN y reactivos necesarios para las reacciones de PCR para los estudiantes.
- Determine la fuente de la plantilla de ADN para las inserciones de genes, como el ADN genómico, el plásmido y el ADN sintético.
- Antes de los experimentos, proporcione hojas de trabajo a los estudiantes para apoyar la preparación y evaluar la comprensión (Archivo Suplementario 4, Suplementario 5 y Suplementario 6).
- Recopilación de datos sobre los resultados del aprendizaje
NOTA: Si está interesado en recopilar datos de resultados de aprendizaje del módulo Gibson Assembly y publicar los resultados, obtenga la aprobación para la investigación con sujetos humanos según las políticas de la institución.- Invite a los estudiantes a participar en una investigación de resultados de aprendizaje al menos 1 semana antes del inicio del proyecto independiente de la Asamblea Gibson.
- Pida a los estudiantes participantes que completen el cuestionario previo de opción múltiple (Archivo Suplementario 6) la reunión de laboratorio antes del inicio del proyecto independiente.
2. Experimento A: PCR para obtener fragmentos lineales
- Obtener cebadores y soluciones de ADN molde para PCR del instructor.
- Pipetee reacciones de 25 μL, incluyendo ADN polimerasa MasterMix, plantilla de ADN, cebadores directos e inversos y agua sin nucleasas de acuerdo con las instrucciones del manual del alumno (véase el Archivo complementario 2).
- Cicle la reacción en un termociclador de acuerdo con el manual del estudiante en el Archivo Suplementario 2. Asegúrese de que la temperatura de recocido de la reacción sea adecuada para los cebadores y que el tiempo de extensión sea adecuado para la longitud del amplicón deseado. Consulte el Archivo Suplementario 2 y el Archivo Suplementario 3 para obtener instrucciones detalladas.
- Almacene las reacciones de PCR a 4 °C o -20 °C hasta el siguiente período de laboratorio.
- Analice 5 μL de cada reacción mediante electroforesis en gel de agarosa. Mientras el gel está funcionando, agregue 1 μL de enzima de restricción DpnI a cada reacción que utilizó ADN plasmídico como plantilla. Incubar esta mezcla durante 1 h a 37 °C.
NOTA: DpnI degradará cualquier plantilla de ADN plasmídico circular restante, lo que hace que sea menos probable obtener falsos positivos en el paso de transformación. - Obtener una imagen del gel para confirmar que la PCR fue exitosa y que se logró la amplificación correcta. Purifique cualquier reacción de PCR exitosa con un kit de purificación de PCR disponible en el mercado. Mida la concentración (ng/μL) de un producto de PCR purificado utilizando un espectrofotómetro de microvolumen para su uso en cálculos posteriores de ensamblaje de Gibson.
- Antes de la próxima reunión de clase, diseñe la receta de reacción para el ensamblaje de Gibson (consulte el Archivo Suplementario 2 y el Archivo Suplementario 4).
3. Experimento del alumno B: Gibson Montaje, transformación y selección de clones
- Pipetear la Gibson Reacción del ensamblaje de acuerdo con la receta diseñada. Incubar la reacción a 50 °C durante 15 min. Mientras se incuban las reacciones, prepárese para la transformación descongelando células de Escherichia coli químicamente competentes en hielo.
- Transforme 2 μL del ensamblaje Gibson en celdas químicamente competentes mediante choque térmico. Pipetear 100 μL de las células transformadas en dos placas de selección y extender con perlas esterilizadas. Prepare diluciones 10-4, 10-5 y 10-6 de las células restantes en agar Luria-Bertani (LB). Plaque 100 μL de cada dilución en serie en LB (sin selección) y extienda con perlas esterilizadas.
NOTA: Este paso permite calcular la eficiencia de la transformación. - Incubar las placas durante la noche a 37 °C. Almacene las placas a 4 °C hasta el siguiente período de clase.
- Cuente las colonias en todas las placas y calcule la eficiencia de la transformación como se indica en el Archivo Suplementario 2. Vuelva a trazar una placa selectiva con cuatro colonias de cualquiera de las placas selectivas e incube esta placa a 37 °C durante la noche.
NOTA: Esto sirve como un cultivo de respaldo de sus clones positivos. - Usando un marcador, seleccione, encierre en un círculo y etiquete cuatro colonias distintas en la placa de selección con las iniciales del estudiante y un número (por ejemplo, ABC1). Recupere una nueva placa de agar LB que contenga antibiótico para la selección, use un marcador para dividir la placa en cuadrantes (es decir, cuartos) y luego use ~ 1/2 de cada colonia para volver a rayar en el cuadrante marcado respectivamente y la otra 1/2 para inocular un cultivo líquido de LB de 5 mL para cada una de las cuatro colonias seleccionadas. Asegúrese de etiquetar los tubos con la identidad de colonia respectiva (por ejemplo, ABC1) y agregue la concentración correcta de antibiótico para la selección. Incubar los cultivos líquidos en una incubadora agitadora a 37 °C durante la noche y la placa de agar en una incubadora estática a 37 °C; almacenar los cultivos líquidos y las placas a 4 °C hasta el siguiente período de clase.
4. Experimento del estudiante C: Aislamiento y cribado de plásmidos
- Aísle el ADN plásmido de los cultivos líquidos del Experimento B utilizando un kit de minipreparación. Mida la concentración de plásmido aislado (en ng/μL).
- Diseñe un resumen de restricción o una prueba de PCR para analizar los plásmidos aislados (consulte las instrucciones detalladas en el Archivo Suplementario 2 y las expectativas de informe en el Archivo Suplementario 5).
- Pipetear las reacciones de restricción, digerir o reacciones de acuerdo con recetas diseñadas e incubar a temperaturas y duraciones diseñadas anteriormente.
- Analice los resultados mediante electroforesis en gel de agarosa.
5. Apreciación
- Pida a los estudiantes participantes que completen el cuestionario posterior de opción múltiple (Archivo complementario 7) en la reunión de laboratorio después de completar el módulo de ensamblaje de Gibson.
- Combine todos los datos de las respuestas previas y posteriores al cuestionario para el análisis.
- Examinar la significación estadística del aumento en la puntuación media de cada pregunta de contenido individual entre los cuestionarios mediante el uso de pruebas t pareadas de dos colas y el tamaño del efecto d de Cohen.
- Evalúe el aumento de la confianza de los estudiantes para cada término y técnica individual para determinar el alcance del cambio y su significación estadística.
- Determine el impacto de los antecedentes y los factores académicos de los estudiantes en el aprendizaje de los estudiantes mediante el uso de la ganancia de aprendizaje normalizada (NLG) y las pruebas t no emparejadas.
- Evaluar las actitudes de los estudiantes hacia la experiencia del proyecto y las carreras de biología molecular mediante la evaluación de las puntuaciones medias.
Resultados
Éxito de los estudiantes en la clonación
En cada iteración del módulo de ensamblaje de Gibson (2019, 2022 y 2023), se les pidió a los estudiantes que prepararan un informe que resumiera sus hallazgos. En 2019, 36 de 44 estudiantes (81,8 %) informaron que clonaron con éxito sus plásmidos en función de los resultados de la pantalla que diseñaron para el Experimento C. Un total de 14 de 20 estudiantes (70,0%) informaron haber tenido éxito en la clonación de su...
Discusión
Aquí, proponemos un proyecto de laboratorio ajustable para el aula de pregrado que enseña a los estudiantes la clonación molecular a través de Gibson Assembly en un entorno de investigación basado en cursos. En total, 28 plásmidos novedosos fueron clonados por estudiantes de pregrado en un salón de clases. El éxito individual reportado por los estudiantes osciló entre el 44,4% y el 81,8% en tres cohortes, y el éxito general de la clonación fue del 80% (28 de los 35 plásmidos ...
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos ni otros conflictos de intereses.
Agradecimientos
Los autores agradecen a Andrea Laubscher por su apoyo técnico, y a Michael Black, Sandi Clement y Javin Oza por sus útiles discusiones sobre la implementación del laboratorio de enseñanza y la evaluación de los resultados del aprendizaje. Los autores agradecen a todos los estudiantes que participaron en el estudio de resultados de aprendizaje en las cohortes de 2019, 2022 y 2023, así como a los estudiantes de investigación Nathan Kuhn y Aayushi Adettiwar que ayudaron en la preparación de reactivos para la implementación en el laboratorio de enseñanza. Los autores también reconocen el apoyo financiero del Fondo William y Linda Frost, la Beca de Investigación Aplicada Chevron Biotechnology del Centro de Aplicaciones en Biotecnología y la Fundación Nacional de Ciencias (NSF-1708919 y NSF-2300890).
Materiales
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Referencias
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados