A subscription to JoVE is required to view this content. Sign in or start your free trial.
Research Article
שיפור תוצאות התלמידים עם ניסיון מחקר לתואר ראשון המבוסס על קורס שיבוט מולקולרי ניתן להתאמה
* These authors contributed equally
In This Article
Summary
מודול שיבוט מולקולרי של Gibson Assembly הניתן להתאמה הועסק בפורמט ניסיון מחקרי לתואר ראשון (CURE) מבוסס קורס לתלמידי קורס מעבדה לביולוגיה מולקולרית. הערכת תוצאות הלמידה של התלמידים הראתה שיפור בהבנה ובביטחון בשיבוט מולקולרי לאחר השלמת CURE, ופלסמידים חדשים שובטו למחקר ביוסינתזה של מוצרים טבעיים.
Abstract
ההתקדמות המתמשכת של טכניקות ביולוגיה מולקולרית דורשת כי תוכניות הלימודים ביולוגיה מולקולרית מעודנות באופן קבוע כדי להכין ביעילות את התלמידים להיכנס לכוח העבודה עם יכולות מודרניות. בפרט, הופעתה של Gibson Assembly, טכניקת שיבוט מולקולרית אדפטיבית וניתנת להתאמה אישית, קידמה את הנוף של שיבוט מולקולרי בסביבות מחקר רבות. לפיכך, יצרנו מודול שיבוט של Gibson Assembly לפריסה בקורס מעבדה לביולוגיה מולקולרית באוניברסיטה הפוליטכנית של קליפורניה, סן לואיס אוביספו והערכנו את תוצאות הלמידה של התלמידים מהמודול. במהלך שלוש חזרות של הקורס, הסטודנטים השתתפו בפרויקט עצמאי מבוסס ניסוי שכלל שיבוט שלוש ספריות פלסמיד ייחודיות לתמיכה בפרויקטי מחקר בביוסינתזה של מוצרים טבעיים. התלמידים קיבלו שאלונים לפני ואחרי כדי להעריך את הבנתם בשיבוט מולקולרי ואת ביטחונם במונחים ובטכניקות של ביולוגיה מולקולרית. תגובות התלמידים הראו עלייה משמעותית הן בלימוד מושגי שיבוט מולקולרי והן בדיווח עצמי על ביטחון עם מונחים וטכניקות שיבוט מולקולרי. ניתן להכליל מסגרת מודול זו כדי ללמד את Gibson Assembly עבור יישומים שונים, ולספק למדריכים ערכת כלים להוראת טכנולוגיית שיבוט ניתנת להתאמה ומתפתחת תוך קידום פרויקטי המחקר שלהם.
Introduction
הכשרת סטודנטים לתואר ראשון במושגי ביולוגיה מולקולרית בסיסיים ובטכניקות מעבדה חיונית להתפתחותם המדעית והמקצועית שכן מתודולוגיות אלה נפוצות במסגרות מחקר שונות, כולל אקדמיה ותעשייה. ככזה, סטודנטים בביולוגיה (ריכוז ביולוגיה מולקולרית ותאית) וביוכימיה באוניברסיטה הפוליטכנית של קליפורניה, סן לואיס אוביספו (Cal Poly) נדרשים לקחת קורס מעבדה ביולוגיה מולקולרית חטיבה עליונה כדי ללמוד וליישם נושאים אלה (CHEM / BIO 475). תוכנית לימודים בסיסית לקורס זה פותחה בעבר בה התלמידים מבצעים שיבוט מבוסס טופואיזומראז (TOPO) כדי להרכיב פלסמיד המכיל אקטין המוכן מתבנית DNA משלים של שמרים (cDNA)1. התלמידים מתכננים ניסויים המבוססים על שאלות המחקות השערות מחקר אותנטיות, ומגבירים את היכרותם עם פרקטיקות מעבדה ולמידה מבוססת חקירה. התקדמות מתמשכת בתחום הביולוגיה המולקולרית דורשת כי תוכניות הלימודים המתאימות להתאים להכין תלמידים עם יכולות מודרניות עבור כוח העבודה. בפרט, השימוש באסיפת גיבסון הפך לדומיננטי יותר בקהילה המדעית; בעוד השיטה הוקמה במקור כדי לסנתז כרומוזומים מלאכותיים2, מעל 5,000 פרסומים בזמן דו"ח זה התייחסו לעבודתם המקורית של גיבסון ואחרים. הרכבה של גיבסון ייחודית בהשוואה למתודולוגיות שיבוט מסורתיות: היא ניתנת להתאמה אישית גבוהה ויכולה בקלות לקשור מקטעי דנ"א ליניאריים מרובים ללא צורך באתרי הגבלה כדי לייצר את הצמתים. לפיכך, ראינו הזדמנות לחדש את תוכנית הלימודים של CHEM / BIO 475 כדי לשלב טכניקות שיבוט מולקולרי מודרניות ולשפר את מודל הקורס מבוסס החקירה.
נקבע כי חוויות המחקר של התלמידים תורמות להבנה מושגית מוגברת, פיתוח מיומנויות והתמדה במדע3, אך לא לכל הסטודנטים לתואר ראשון יש הזדמנות להשתתף ישירות במעבדת מחקר. כדי להתמודד עם האתגר של קיבולת מוגבלת של סטודנטים במעבדות מחקר, חוויות מחקר לתואר ראשון מבוססות קורס (CUREs) פותחו והופעלו כדי להגדיל את הנגישות המדעית באמצעות מחקר אותנטי בכיתה. בעוד CUREs משתנים ביישומם, נקבעו פרקטיקות נפוצות המתייחסות לחמשת עקרונות המחקר המדעי. ב- CURE מתוכנן היטב, התלמידים 1) ישתמשו בפרקטיקות מדעיות, 2) ישתפו פעולה בפרויקט מחקר, 3) ינסו לגלות תגליות חדשות, 4) יתרמו לעבודה רלוונטית מחוץ לכיתה, ו -5) יעריכו מחדש וישנו השערות ושיטות במקרה של כישלון ניסויי4. בדומה לחוויות מחקר מסורתיות של סטודנטים במעבדה, CUREs הוכחו כמחזקים את אמון התלמידים במדע, מיומנויות מדעיות, בעלות על פרויקטים והתמדה במדע, טכנולוגיה, הנדסה ומתמטיקה (STEM)5. בעוד שבעבר דווח על CUREs הכוללים שיבוט מולקולרי 6,7,8,9,10,11,12,13, איננו מודעים לאף אחד מהם המדגישים את יכולת ההסתגלות של אסיפת גיבסון ליצירת ספרייה של פלסמידים מחקריים אותנטיים.
כאן, אנו מדווחים על הרחבה של תוכנית הלימודים הנוכחית מבוססת חקירה CHEM / BIO 475 באוניברסיטה הפוליטכנית של קליפורניה, סן לואיס אוביספו עם שני שיפורים עיקריים: ניסיון מעשי באמצעות אסיפת גיבסון והשתתפות סטודנטים ב- CURE, שסיפקה מבני פלסמיד מקוריים לפרויקטים מחקריים במימון הקרן הלאומית למדע (NSF-1708919 ו- NSF-2300890). במהלך שלושה יישומים של תוכנית לימודים זו, התלמידים תרמו לשני פרויקטי מחקר נפרדים המתמקדים בביוסינתזה של מוצרים טבעיים של מולקולות ביו-אקטיביות המיוצרות על ידי Actinomycetota. מוצרים טבעיים מכילים לעתים קרובות פרמקופורים עם פעילויות אנטיביוטיות, אנטי פטרייתיות ו / או אנטי סרטניות, מה שמקנה למולקולות קטנות אלה חשיבות במאמצי גילוי תרופות ופוטנציאל לרלוונטיות קלינית14. מחקר זה דורש יצירת ספריות פלסמיד כדי לאפשר לחקור הן את התפקוד והן את הפוטנציאל ההנדסי של אנזימים ביוסינתטיים חיידקיים. ב-CURE, סטודנטים תכננו וביצעו ניסויי Gibson Assembly כדי לשכפל את ספריות הפלסמיד הייחודיות הרלוונטיות לפרויקטי המחקר האלה (איור 1). בנוסף, הפורמט והעיצוב של המודול הוא ייחודי כי זה בקלות להסתגל כדי ליצור כל פלסמידים של עניין עבור פרויקטים מחקר אחרים.
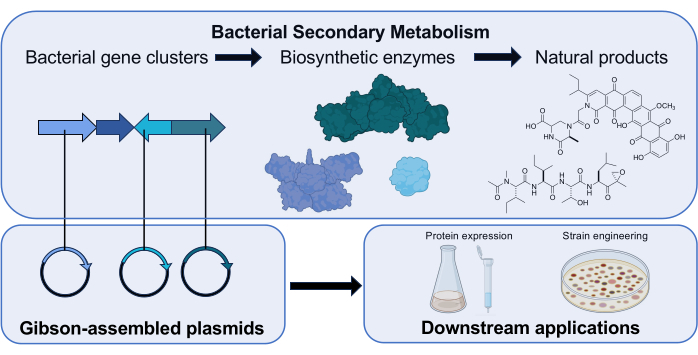
איור 1: סקירה כללית של תפקידה של אסיפת גיבסון במעבדת המחקר שלנו. Actinomycetota מייצרת מוצרים טבעיים של מולקולות קטנות עם פעילויות ביולוגיות רלוונטיות מבחינה קלינית באמצעות צבירי גנים המקודדים אנזימים ביוסינתטיים. במחקר שלנו, פלסמידים המכילים גן ביוסינתטי מורכבים באמצעות אסיפת גיבסון לחקירות במורד הזרם של תפקוד האנזים המקודד. אייקונים מדעיים משנת Biorender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
סקירה כללית של המעבדה
המעבדה לביולוגיה מולקולרית היא קורס חטיבה עליונה הנדרש למגמות ביוכימיה וביולוגיה עם ריכוז ביולוגיה מולקולרית ותאית. סטודנטים אחרים העומדים בדרישות הקדם מוזמנים ללמוד את הקורס כקורס בחירה בחטיבה העליונה. הקורס מתקיים בשיתוף המחלקה לכימיה וביוכימיה והמחלקה לביולוגיה ב-Cal Poly. חברי סגל משתי המחלקות מלמדים בתורות את הקורס בכל סמסטר (שני רבעים ביולוגיה, רבע כימיה וביוכימיה).
הכיתה נפגשת במעבדה פעמיים בשבוע לפרקי זמן של 170 דקות ופעם בשבוע להרצאה של 50 דקות. מדורי מעבדה מכילים מקסימום של 16 סטודנטים ו 2-3 קטעי מעבדה מוצעים בכל רבעון. הקורס נמשך 10 שבועות, ובחינת הגמר נערכת במפגש האחרון בשבוע 10. זמן ההרצאה מוקדש לדיון בתיאוריה שמאחורי רבות מהטכניקות הניסוייות שנערכו במעבדה, כמו גם בנושאים עכשוויים בביולוגיה מולקולרית שאינם מכוסים במעבדה. תוכנית הליבה של המעבדה כוללת את תהליך שיבוט הגן אקטין משמרים1, שנמשך כ-7 שבועות (13-14 מפגשי מעבדה). הטכניקות כוללות מיקרופיפטינג, בידוד RNA שמרים, הגברת גן שמרים באמצעות תגובת שרשרת שעתוק לאחור-פולימראז (RT-PCR), שיבוט טופו, סינון כחול-לבן, בידוד פלסמיד ואימות החדרה על ידי תקציר הגבלה ו- PCR, בניתוח סיליקו של שיבוטים, וניתוח רצף DNA. תוכנית הלימודים במשך 3 השבועות האחרונים של הקורס נתונה לשיקול דעתו של המדריך, אך כוללת תלמידים המשלימים "פרויקט עצמאי" עם תוצאות לא מוגדרות.
סקירה כללית של הניסוי
מוקד אחד של קבוצת המחקר שלנו הוא מסלולים ביוסינתטיים באקטינומיצטוטה. בעת תכנון הפרויקט העצמאי, חזינו שסטודנטים ייצרו פלסמידים באמצעות Gibson Assembly לשימוש בפרויקטי המחקר שלנו החוקרים ביוסינתזה של מוצרים טבעיים. בעוד שהאיטרציות של מודול הקורס המוערכות כאן היו מיוחדות לפלסמידים המאפשרים מניפולציה של מסלולים ביוסינתטיים, תהליך העבודה של הרכבת גיבסון ניתן להתאמה עצומה לפרויקטים אחרים של שיבוט מולקולרי (איור 2). תהליך העבודה פוצל לשלושה ניסויים שונים (A, B ו-C) שהושלמו על פני שתי תקופות הרצאה ושש תקופות מעבדה (3 שבועות בסך הכל) (ראה קובץ משלים 1 וקובץ משלים 2). לניסויים קדמו דפי עבודה לתמיכה בהכנת התלמידים ולהערכת הבנת התלמידים (קובץ משלים 3, קובץ משלים 4 וקובץ משלים 5). זרימת העבודה מוצגת בפורמט גמיש לצרכים ולתחומי העניין של המדריך.

איור 2: זרימת עבודה של מודול Gibson Assembly. יום 1 ויום 4 הם תקופות הרצאה בהן התלמידים משלימים ניתוח רצף סיליקו ועיצוב ניסויי. ימים 2-3 ו-5-8 הם מפגשי מעבדה שבהם נערכים השלבים לשיבוט פלסמידים חדשים באמצעות אסיפת גיבסון, ולאחר מכן בידוד וסינון. תרשים הזרימה הציורי מקובץ לפי שלושת הניסויים שהתלמידים מבצעים (A, B ו-C). הוראות ופרוטוקולים מפורטים יותר ניתן למצוא במדריכים למדריך ולתלמיד המסופקים כקובץ משלים 1 וקובץ משלים 2, בהתאמה. אייקונים מדעיים משנת Biorender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
מודול הפרויקט העצמאי Gibson Assembly נוסה לראשונה ברבעון האביב של 2019 ב- CHEM/BIO 475. בשנים 2020 ו -2021, הקורס נלמד באופן מקוון עקב מגיפת SARS-CoV-2. כאשר חודשה ההוראה הפרונטלית באביב 2022 ו-2023, סטודנטים בקורס הוזמנו להשתתף במחקר להערכת תוצאות למידה של פרויקט עצמאי של Gibson Assembly שבו ישובטו פלסמידים מקוריים ורלוונטיים למחקר. בשנת 2019, סטודנטים יצרו ספרייה של פלסמידים המורכבת מקלטות גנים מהגנום של Micromonospora echinospora ATCC 15837 ששובטו לתוך pKC1132 (איור 3). ספריית פלסמיד זו משמשת במעבדת המחקר שלנו כדי להשבית גנים בעלי עניין בצביר גנים ביוסינתטי משוער עבור המוצר הטבעי TLN-0522015. כדי להשלים את מחקרי השבתת הגנים שלנו, סטודנטים בשנת 2022 שיבטו ספרייה קטנה של גנים מאשכול הגנים המשוער TLN-05220 לתוך pUC19 (Figure 3); קבוצת המחקר שלנו השתמשה בפלסמידים אלה לשיבוט גנים לווקטורי ביטוי, כולל pET28b, לביטוי יתר וטיהור חלבונים. סטודנטים במחזור 2023 תרמו לעבודה מתמשכת על פרויקט הנדסה ביוסינתטית על epoxomicin synthetase16. בצוותים של 3-4, סטודנטים שיבטו תחומים מהונדסים של מודולי סינתטאז17 פפטיד לא ריבוזומלי לתוך וקטורים שונים של ביטוי חלבונים כדי למטב את ביטוי היתר והטיהור של האנזימים האלה במעבדת המחקר שלנו (איור 3). יתירות נבנתה בתוכנית השיבוט עבור כל קבוצה. לדוגמה, מחזור 2019 הכיל 44 תלמידים, ו -15 פלסמידים הוקצו לכיתה, לשיבוט. לפיכך, נעשה ניסיון שיבוט של כל פלסמיד פעמיים או שלוש.
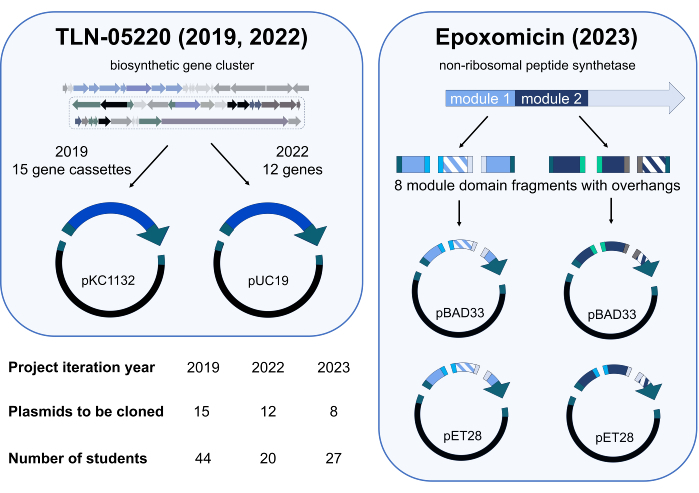
תרשים 3: סיכום של פלסמידים משובטים וסטודנטים משתתפים במהלך חזרות פרויקטים עצמאיים בשנים 2019, 2022 ו-2023. פרויקט Gibson Assembly נפרס שלוש פעמים. בכל הצעה, הסטודנטים שיבטו ספרייה אחרת של פלסמידים שישמשו בפרויקטים מחקריים הבוחנים מסלולים ביוסינתטיים. פרויקטים בשנים 2019 ו-2022 תמכו בעבודתנו השוטפת על המוצר הטבעי TLN-0522015 עם שתי תגובות מקטע (קלטת גן אחת או גן אחד וקטור) Gibson Assembly. הפרויקט בשנת 2023 כלל החלפת תחומים בתוך מודולים 1 ו-2 של אנזים סינטטאז פפטידי לא ריבוזומלי (NRPS) המעורב בביוסינתזה של אפוקסומיצין16. השברים שבקעו מייצגים שני מוטנטים שונים של התחום שהוחלף, וצבעים אחידים מייצגים תחומים שלא הוחלפו. בסך הכל, שמונה מקטעי גנים שונים (ארבעה עבור מודול 1 וארבעה עבור מודול 2) נוצרו עם שלוחות תואמות עבור Gibson Assembly. עבור כל מודול, שני שילובים שונים של שלושה מקטעי גנים הורכבו עם אחד משני וקטורים שונים (pBAD33 ו-pET28, ארבעה מקטעים בסך הכל לכל הרכבה), עבור הפוטנציאל ליצור שמונה פלסמידים NRPS מהונדסים של NRPS. אייקונים מדעיים משנת Biorender.com. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
סקירה כללית של הערכה
לפחות שבוע לפני תחילת הפרויקט העצמאי של Gibson Assembly, סטודנטים במחזורי 2022 ו-2023 הוזמנו להשתתף בחקירת תוצאות למידה, שתוכננה בדומה למחקר שנערך בקורס מעבדה לביוכימיה ב-Cal Poly18. הסטודנטים מילאו שאלון מקדים רב-ברירה בישיבת המעבדה לפני תחילת הפרויקט העצמאי ופוסט-שאלון רב-ברירה במהלך פגישת המעבדה הסופית (כלומר, לאחר שהשלימו את מודול הרכבת גיבסון). השאלונים לפני ואחרי השאלון כללו 28 שאלות זהות ושתי שאלות נוספות לאחר השאלון (30 בסך הכל). עשר שאלות תוכן נכתבו כדי להעריך את הידע של התלמידים על אנזימים ומנגנונים המעורבים בשיבוט מולקולרי (למשל, תגובת שרשרת פולימראז [PCR], הרכבת גיבסון, טרנספורמציה, סינון כחול-לבן). שבע השאלות הבאות ביקשו מהתלמידים להעריך בעצמם את היכרותם עם מונחי שיבוט מולקולרי (למשל, DNA פולימראז, אקסונוקלאז, ליגאז). 10 השאלות הבאות אפשרו לתלמידים להעריך את יכולתם לבצע טכניקות שיבוט מולקולרי (למשל, ניתוח רצף DNA, תגובות עיכול הגבלה, אלקטרופורזה של ג'ל אגרוז). הסטודנטים גם דיווחו אם יהיה להם נוח להמשיך בקריירה בביולוגיה מולקולרית בהתבסס על הידע שלהם בטכניקות שיבוט מולקולרי. שתי שאלות נוספות נכללו לאחר השאלון כדי לאפשר לסטודנטים להעריך את מחויבותם ללמידה בקורס והאם הקורס היה חוויה לימודית בעלת ערך (קובץ משלים 6 ותיק משלים 7). כל הנתונים מתשובות התלמידים לשנים 2022 ו-2023 לפני ואחרי השאלון שולבו לניתוח וזמינים בטבלה משלימה S1.
Protocol
המחקרים שכללו משתתפים אנושיים נסקרו ואושרו על ידי נבדקים אנושיים במועצת הסקירה המוסדית למחקר ב- Cal Poly (2022-113-CP (IRB)). המשתתפים נתנו את הסכמתם מדעת בכתב להשתתף במחקר זה.
הפרוטוקול הבא מתאר את הכנת המדריך (שלבים 1.1-1.3), פעולות התלמידים למודול הוראה בן שלושה ניסויים הכולל PCR להשגת מקטעים ליניאריים (שלבים 2.1-2.7), הרכבה של גיבסון, טרנספורמציה ובחירה של שיבוטים (שלבים 3.1-3.5), בידוד פלסמיד וסינון (שלבים 4.1-4.4), והערכת תוצאות למידה (5.1 –5.2). הכנת המדריך מתארת דוגמה מייצגת אחת של תכנון פריימר והכנת מפת פלסמיד רצויה בסיליקו. כל חלקי הפרוטוקול ניתנים להתאמה עבור פלסמידים רצויים אחרים. כל ניסוי סטודנטים מחולק לשני מפגשי מעבדה של 3 שעות.
1. הכנת המדריך
- עיצוב פריימר של Gibson Assembly באמצעות Benchling Assembly Wizard
- קבע את מקור תבנית ה- DNA עבור תוספות הגנים, כגון DNA גנומי, פלסמיד ו- DNA סינתטי.
הערה: עבור גנים שיוגדלו מתבניות DNA גנומיות מורכבות, מומלץ לקבוע תחילה אתרי קישור ייחודיים לפריימרים בתוך תבנית הדנ"א הגנומי הספציפיים לגנים המעניינים, באמצעות הכלי הבסיסי לחיפוש יישור מקומי (BLAST) של המרכז הלאומי למידע ביוטכנולוגי (NCBI)21, כמפורט בקובץ משלים 1. עבור תבניות פשוטות כגון פלסמידים או דנ"א סינתטי, המשך לשלב הבא. - אחזור רצפי DNA של תוספות הגן והווקטור (לדוגמה, ראה רצף הגן polyketide hydroxylase ורצף pUC19 שסופקו כקובצי GenBank בקובץ המשלים 8). ייבא את רצף ההוספה הרצוי ואת הרצף הווקטורי הרצוי ל- Benchling, כל אחד מהם כקובץ רצף DNA חדש. פתח כל רצף מיובא כדי להיכלל בתגובת אסיפת גיבסון.
הערה: ניתן לייבא קבצי רצף DNA במספר פורמטים, כולל GenBank ו- FASTA. ניתן גם לייבא רצף ישירות ממסד נתונים עם מספר הצטרפות או להדביק רצף נוקלאוטידים שהועתק מקובץ או מציג אחר. - בתחתית המסך, אתר את הכלי אשף ההרכבה . לחץ/ י על ״אשף ההרכבה״ ולאחר מכן בחר/י ״צור הרכבה חדשה״. מתוך האפשרויות המוצגות, בחר Gibson ולחץ על התחל כדי להתחיל בהרכבה.
- בחלון הרצף הווקטורי , בחר את כל הבסיסים שייכללו בהרכבה מעמוד השדרה הווקטורי. התחל את הבחירה במיקום הנוקלאוטידים בקצה 3' של עלון הגן (לדוגמה, מיקום 657 של pUC19), ובחר את כל הנוקלאוטידים הנותרים שייכללו (למשל, דרך מיקום 656 כדי לכלול רצף pUC19 שלם). לאחר שנבחר, לחץ על הכרטיסיה עמוד שדרה בתחתית המסך| הגדר מקטע.
הערה: עברו בין תצוגת 'מפת רצף ' לתצוגת 'פלסמיד ' של הווקטור לבחירת כל הבסיסים הרצויים בתבנית מעגלית. לחלופין, אם אתם מתכננים להכין את המקטע הווקטורי באמצעות תקציר הגבלה (במקום PCR) עבור Gibson Assembly, בצעו ליניאריזציה של רצף עמוד השדרה עם אתר חיתוך אנזים הגבלה (למשל, XbaI). לשם כך, לחץ על האתר שנחתך, החזק את מקש Shift לחוץ ולחץ שוב על האתר החתוך . לאחר מכן לחץ על הגדר מקטע כדי להגדיר את עמוד השדרה. שים לב שפריימרים ספציפיים לווקטורים לא ייווצרו על ידי Benchling אם עמוד השדרה הוא ליניארי עם אנזים הגבלה באשף ההרכבה. - בחלון הוספת רצף , בחר בכל בסיסי ההוספה שייכללו בהרכבה. לאחר שנבחר, לחץ על הכרטיסיה הוספה בתחתית המסך | סט פרגמנט.
- במקרה של תוספות גנים מרובות, לחץ על לחצן + בצד ימין של אשף ההרכבה. בחלון הוספת רצף , בחר בכל בסיסי ההוספה שייכללו בהרכבה. לאחר שנבחר, לחץ על הכרטיסיה הוספה בתחתית המסך | סט פרגמנט.
- לאחר הגדרת כל המקטעים, שנה את שם ההרכבה לשם הפלסמיד הרצוי ולחץ על הרכיב מימין לאשף ההרכבה. בחר את מיקום התיקייה הרצוי הן לתיקיית רצף והן לתיקיית פריימר. לחצו על 'צור' להרכבת רצף הפלסמיד הרקומביננטי.
- פתח את הפלסמיד שהורכב ולחץ על היסטוריית הרכבה כדי להציג מפת פלסמיד בסיסית וסקירה כללית של הרצפים שמהם הוא נגזר. ודא שמיקומי הנוקלאוטידים עבור כל מקטע תואמים לעיצוב הרצוי.
- בכרטיסיה פרמטרים של הרכבה , הצג את שמות הפריימרים המיועדים להרכבה. שימו לב שיהיו שני פריימרים המיועדים לכל תוספת (קדימה ואחורה), ושמות הפריימרים ייגזרו מהכותרות של קבצי רצף הדנ"א. כמו כן, ודא שטמפרטורות ההתכה של הפריימר וטמפרטורות החישול המומלצות תואמות בחלון זה (ראה קובץ משלים 8, פריימרים מייצגים).
- אם טמפרטורות ההתכה של פריימר או חישול אינן אידיאליות, התאימו ידנית רצפי פריימר (לדוגמה, הסרת נוקלאוטידים כדי להפחית את טמפרטורת ההתכה) בתוך קובצי רצף הפריימר. לחלופין, לחץ על פתיחה מחדש והתאם את הגדרות הפריימר על ידי לחיצה על כפתור הגדרות אנזים/פריימר לצד כפתור ההרכבה באשף ההרכבה. לאחר מכן, חזור על שלב ההרכבה ובחן מחדש רצפי פריימר.
- אם רוצים לשכפל ספרייה של פלסמידים עם גנים רבים ושונים המשובטים לאותו עמוד שדרה וקטורי,
- השתמש באותו זוג פריימר עבור הווקטור בכל מקרה. לשם כך, הסר ידנית את כל הנוקלאוטידים מהפריימרים הווקטוריים שיתקינו את התלבטות הספציפיות לעל. נוקלאוטידים אלה יימצאו בקצה ה-5' של הפריימר. לאחר ההסרה, ודא שטמפרטורות ההתכה של הפריימר וטמפרטורות החישול תואמות (ראה קובץ משלים 8, פריימרים מייצגים).
- ודא שלכל זוגות הפריימרים יש טמפרטורות התכה וחישול דומות מאוד.
הערה: זה יפחית את הצורך באופטימיזציה של טמפרטורת החישול של כל זוג ויאפשר לכל תגובות ה- PCR של התלמידים להיות מחזוריות בו זמנית באמצעות אותה תוכנית thermocycler.
- לאחר השלמת עיצוב הפריימר עבור פלסמידים רצויים, לחץ על סיום באשף ההרכבה. הזמינו את הפריימרים מחברת סינתזת DNA ושלפו דגימות של תבניות הדנ"א עבור תוספות ווקטור.
- בדוק זוגות פריימר ספציפיים להוספה ולווקטור לטמפרטורות חישול אופטימליות באמצעות ניסוי PCR הדרגתי עם תבניות DNA. בדוק את טמפרטורות החישול המומלצות על ידי Benchling ± 3 ° C בתחילה.
- לאחר קביעת טמפרטורות החישול, צור אליציטוטים של תמיסות פריימר, תבניות DNA וריאגנטים נחוצים לתגובות PCR לתלמידים.
- קבע את מקור תבנית ה- DNA עבור תוספות הגנים, כגון DNA גנומי, פלסמיד ו- DNA סינתטי.
- לפני הניסויים, ספקו לתלמידים דפי עבודה לתמיכה בהכנה ולהערכת ההבנה (קובץ משלים 4, נספח 5 ונספח 6).
- איסוף נתונים על תוצרי למידה
הערה: אם אתם מעוניינים לאסוף נתוני תוצאות למידה ממודול Gibson Assembly ולפרסם את התוצאות, קבלו אישור למחקר עם נבדקים אנושיים בהתאם למדיניות המוסד.- הזמינו את התלמידים להשתתף בחקירת תוצאות למידה לפחות שבוע לפני תחילת הפרויקט העצמאי של אסיפת גיבסון.
- בקשו מהסטודנטים המשתתפים למלא את השאלון המקדים רב ברירה (קובץ משלים 6) את מפגש המעבדה לפני תחילת הפרויקט העצמאי.
2. ניסוי תלמידים א': PCR להשגת מקטעים ליניאריים
- השג פריימרים ותבניות DNA עבור PCR מהמדריך.
- תגובות פיפטה 25 μL כולל DNA פולימראז MasterMix, תבנית DNA, פריימרים קדימה ואחורה, ומים ללא נוקלאז בהתאם להוראות במדריך לתלמיד (ראה קובץ משלים 2).
- מחזור התגובה בתרמוסייקלר בהתאם למדריך לתלמיד בקובץ משלים 2. ודא שטמפרטורת החישול של התגובה מתאימה לפריימרים, ושזמן ההארכה מתאים לאורך המגבר הרצוי. עיין בקובץ משלים 2 ובקובץ משלים 3 לקבלת הוראות מפורטות.
- אחסן תגובות PCR ב 4 ° C או -20 ° C עד תקופת המעבדה הבאה.
- לנתח 5 μL של כל תגובה באמצעות אלקטרופורזה ג'ל agarose. בזמן שהג'ל פועל, הוסיפו 1 μL של אנזים הגבלת DpnI לכל תגובה שהשתמשה בדנ"א פלסמיד כתבנית. לדגור על תערובת זו במשך 1 שעה ב 37 ° C.
הערה: DpnI יפגע בכל תבנית DNA פלסמיד מעגלית שנותרה, מה שמקטין את הסיכוי לקבל תוצאות חיוביות שגויות בשלב הטרנספורמציה. - צלם את הג'ל כדי לאשר כי PCR היה מוצלח והגברה נכונה הושגה. טהרו תגובות PCR מוצלחות בעזרת ערכת טיהור PCR הזמינה באופן מסחרי. מדוד את הריכוז (ng/μL) של מוצר PCR מטוהר באמצעות ספקטרופוטומטר מיקרו-נפח לשימוש בחישובי הרכבה עוקבים של Gibson.
- לפני פגישת הכיתה הבאה, עצבו את מתכון התגובה לאסיפת גיבסון (ראו קובץ משלים 2 וקובץ משלים 4).
3. ניסוי תלמידים ב': הרכבה של גיבסון, טרנספורמציה ובחירה של שיבוטים
- פיפטה תגובת אסיפת גיבסון על פי המתכון המעוצב. לדגור על התגובה ב 50 ° C במשך 15 דקות. בזמן שהתגובות דוגרות, התכוננו לשינוי על ידי הפשרת תאי Escherichia coli בעלי כשירות כימית על קרח.
- הפוך 2 μL של מכלול גיבסון לתאים בעלי כשירות כימית באמצעות הלם חום. פיפטה 100 μL של התאים שהשתנו על שני לוחות בחירה והתפשט עם חרוזים מעוקרים. הכינו 10-4, 10-5 ו-10-6 דילולים של התאים הנותרים על אגר לוריא-ברטאני (LB). צלחת 100 μL של כל דילול סדרתי על LB (אין בחירה) ולהפיץ עם חרוזים מעוקרים.
הערה: שלב זה מאפשר חישוב יעילות הטרנספורמציה. - לדגור את הצלחות לילה ב 37 ° C. לאחסן את הצלחות ב 4 ° C עד תקופת השיעור הבאה.
- ספור מושבות על כל הלוחות וחשב את יעילות הטרנספורמציה בהתאם להוראות בקובץ משלים 2. פזרו מחדש צלחת סלקטיבית אחת עם ארבע מושבות מכל צלחת סלקטיבית ודגרו על צלחת זו בטמפרטורה של 37 °C למשך הלילה.
הערה: זה משמש כתרבות גיבוי של השיבוטים החיוביים שלך. - באמצעות טוש, בחר, הקף ותייג ארבע מושבות נפרדות על לוח הבחירה עם ראשי התיבות של התלמיד ומספר (לדוגמה, ABC1). שלפו צלחת LB-אגר חדשה המכילה אנטיביוטיקה לבחירה, השתמשו בטוש כדי לחלק את הצלחת לרבעים (כלומר, רביעיות), ולאחר מכן השתמשו ב~1/2 מכל מושבה כדי להתפשט מחדש לרביע המסומן בהתאמה וב-1/2 האחר כדי לחסן תרבית LB נוזלית של 5 מ"ל עבור כל אחת מארבע המושבות שנבחרו. הקפד לתייג את הצינורות עם זהות המושבה המתאימה (למשל, ABC1) ולהוסיף את הריכוז הנכון של אנטיביוטיקה לבחירה. לדגור על תרביות נוזליות באינקובטור מטלטל ב 37 מעלות צלזיוס לילה ואת צלחת האגר באינקובטור סטטי ב 37 מעלות צלזיוס; יש לאחסן תרביות וצלחות נוזליות בטמפרטורה של 4 °C עד לשיעור הבא.
4. ניסוי תלמידים ג': בידוד פלסמיד וסינון
- בודדו דנ"א פלסמיד מהתרביות הנוזליות מניסוי ב' באמצעות ערכת מיניפרפ. למדוד את ריכוז הפלסמיד המבודד (ב-ng/μL).
- תכנן תקציר הגבלה או מסך PCR לניתוח הפלסמידים המבודדים (ראה הוראות מפורטות בקובץ משלים 2 ודווח על ציפיות בקובץ משלים 5).
- פיפטה, ההגבלה, מעכלת תגובות או תגובות על פי מתכונים מתוכננים ודוגרת בטמפרטורות ומשכי זמן שתוכננו קודם לכן.
- ניתוח התוצאות באמצעות אלקטרופורזה ג'ל אגרוז.
5. הערכה
- בקש מהסטודנטים המשתתפים למלא את השאלון הרב-ברירה (קובץ משלים 7) את פגישת המעבדה לאחר השלמת מודול ההרכבה של גיבסון.
- שלב את כל הנתונים מהתשובות לפני ואחרי השאלון לניתוח.
- לבחון את המובהקות הסטטיסטית של העלייה בציון הממוצע של כל שאלת תוכן בנפרד בין השאלונים באמצעות מבחני t זוגיים דו-זנביים וגודל אפקט d של כהן.
- להעריך את עליית הביטחון של התלמידים עבור כל מונח וטכניקה בודדים כדי לקבוע את מידת השינוי ואת המשמעות הסטטיסטית שלו.
- לקבוע את ההשפעה של רקע התלמידים וגורמים אקדמיים על למידת התלמידים באמצעות רווח למידה מנורמל (NLG) ומבחני t לא מזווגים.
- להעריך את עמדות התלמידים כלפי ניסיון הפרויקט ואת הקריירה ביולוגיה מולקולרית על ידי הערכת הציונים הממוצעים.
תוצאות
הצלחת התלמידים בשיבוט
בכל איטרציה של מודול הרכבת גיבסון (2019, 2022 ו-2023), התלמידים התבקשו להכין דו"ח המסכם את ממצאיהם. בשנת 2019, 36 מתוך 44 סטודנטים (81.8%) דיווחו כי הם שיבטו בהצלחה את הפלסמידים שלהם בהתבסס על תוצאות המסך שהם תכננו עבור ניסוי C. 14 מתוך 20 סטודנטים (70.0%) דיווח...
Discussion
כאן, אנו מציעים פרויקט מעבדה מתכוונן בכיתה לתואר ראשון המלמד סטודנטים שיבוט מולקולרי באמצעות Gibson Assembly במסגרת מחקר מבוססת קורס. בסך הכל, 28 פלסמידים חדשים שובטו על ידי סטודנטים לתואר ראשון במסגרת כיתתית. ההצלחה שדווחה על ידי תלמידים בודדים נעה בין 44.4% ל-81.8% על פני שלוש קבוצ?...
Disclosures
המחברים מצהירים כי אין להם אינטרסים כלכליים מתחרים או ניגודי עניינים אחרים.
Acknowledgements
המחברים מודים לאנדריאה לאובשר על התמיכה הטכנית, ולמייקל בלאק, סנדי קלמנט וג'אווין אוזה על דיונים מועילים על יישום מעבדת הוראה והערכה של תוצאות למידה. המחברים מודים בהערכה לכל התלמידים שהשתתפו במחקר תוצאות הלמידה במחזורי 2019, 2022 ו-2023, כמו גם לתלמידי המחקר נתן קון ואיושי אדטיוואר שסייעו בהכנת ריאגנטים ליישום מעבדת ההוראה. המחברים מודים גם על תמיכת מימון מקרן ויליאם ולינדה פרוסט, המרכז ליישומים בביוטכנולוגיה של שברון מענק מחקר יישומי יישומי והקרן הלאומית למדע (NSF-1708919 ו- NSF-2300890).
Materials
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
References
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved