É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Research Article
Melhorando os resultados dos alunos com uma experiência de pesquisa de graduação baseada em curso de clonagem molecular adaptável
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Um módulo de clonagem molecular adaptável da Gibson Assembly foi empregado em um formato de experiência de pesquisa de graduação (CURE) baseado em curso para alunos do curso de laboratório de biologia molecular. A avaliação dos resultados de aprendizagem dos alunos mostrou melhor compreensão e confiança na clonagem molecular após a conclusão do CURE, e novos plasmídeos foram clonados para pesquisa de biossíntese de produtos naturais.
Resumo
O avanço contínuo das técnicas de biologia molecular exige que os currículos de biologia molecular sejam regularmente refinados para preparar efetivamente os alunos para entrar no mercado de trabalho com competências modernas. Em particular, o surgimento da Gibson Assembly, uma técnica de clonagem molecular altamente personalizável e adaptável, avançou o cenário da clonagem molecular em vários ambientes de pesquisa. Assim, criamos um módulo de clonagem Gibson Assembly para implantação em um curso de laboratório de biologia molecular na California Polytechnic State University, San Luis Obispo e avaliamos os resultados de aprendizagem dos alunos do módulo. Ao longo de três iterações do curso, os alunos participaram de um projeto independente baseado em experimentos que envolveu a clonagem de três bibliotecas de plasmídeos exclusivas para apoiar projetos de pesquisa em biossíntese de produtos naturais. Os alunos receberam pré e pós-questionários para avaliar sua compreensão da clonagem molecular e sua confiança nos termos e técnicas de biologia molecular. As respostas dos alunos mostraram um aumento significativo tanto no aprendizado de conceitos de clonagem molecular quanto na confiança auto-relatada com termos e técnicas de clonagem molecular. Esta estrutura de módulo pode ser generalizada para ensinar Gibson Assembly para várias aplicações, fornecendo aos instrutores um kit de ferramentas para ensinar uma tecnologia de clonagem adaptável e emergente enquanto avançam em seus projetos de pesquisa.
Introdução
Treinar alunos de graduação em conceitos fundamentais de biologia molecular e técnicas laboratoriais é crucial para seu desenvolvimento científico e profissional, pois essas metodologias são comuns em vários ambientes de pesquisa, incluindo academia e indústria. Como tal, os alunos dos cursos de biologia (concentração de biologia molecular e celular) e bioquímica da California Polytechnic State University, San Luis Obispo (Cal Poly) são obrigados a fazer um curso de laboratório de biologia molecular de divisão superior para aprender e aplicar esses tópicos (CHEM / BIO 475). Um currículo básico para este curso foi desenvolvido anteriormente, no qual os alunos realizam clonagem baseada em topoisomerase (TOPO) para montar um plasmídeo contendo actina que é preparado a partir de um modelo de DNA complementar de levedura (cDNA)1. Os alunos projetam experimentos com base em perguntas que imitam hipóteses de pesquisa autênticas, aumentando sua familiaridade com as práticas de laboratório e o aprendizado baseado em investigação. O avanço contínuo no campo da biologia molecular exige que os currículos correspondentes se adaptem para preparar os alunos com competências modernas para a força de trabalho. Em particular, o uso de Gibson Assembly tornou-se mais predominante na comunidade científica; embora o método tenha sido originalmente estabelecido para sintetizar cromossomos artificiais2, mais de 5.000 publicações no momento deste relatório fizeram referência ao trabalho original de Gibson et al. O Gibson Assembly é único em comparação com as metodologias tradicionais de clonagem: é altamente personalizável e pode facilmente ligar vários fragmentos lineares de DNA sem a necessidade de locais de restrição para produzir as junções. Assim, vimos a oportunidade de reformular o currículo CHEM / BIO 475 para incorporar técnicas modernas de clonagem molecular e melhorar o modelo de curso baseado em investigação.
Foi estabelecido que as experiências de pesquisa dos alunos contribuem para aumentar a compreensão conceitual, o desenvolvimento de habilidades e a persistência na ciência3, mas nem todos os alunos de graduação têm a oportunidade de participar diretamente de um laboratório de pesquisa. Para enfrentar o desafio da capacidade limitada dos alunos em laboratórios de pesquisa, experiências de pesquisa de graduação baseadas em cursos (CUREs) foram desenvolvidas e empregadas para aumentar a acessibilidade à ciência por meio de pesquisas autênticas em sala de aula. Embora os CUREs variem em sua implementação, práticas comuns que abordam os cinco princípios da pesquisa científica foram estabelecidas. Em um CURE bem projetado, os alunos irão 1) usar práticas científicas, 2) colaborar em um projeto de pesquisa, 3) tentar fazer novas descobertas, 4) contribuir para o trabalho relevante fora da sala de aula e 5) reavaliar e revisar hipóteses e métodos em caso de falha experimental4. Semelhante às experiências tradicionais de pesquisa dos alunos em um laboratório, os CUREs demonstraram fortalecer a confiança dos alunos na ciência, nas habilidades científicas, na propriedade do projeto e na persistência em ciência, tecnologia, engenharia e matemática (STEM)5. Embora CUREs envolvendo clonagem molecular tenham sido relatados anteriormente 6,7,8,9,10,11,12,13, não temos conhecimento de nenhum que enfatize a adaptabilidade da Gibson Assembly para fazer uma biblioteca de plasmídeos de pesquisa autênticos.
Aqui, relatamos uma expansão do atual currículo CHEM / BIO 475 baseado em investigação na California Polytechnic State University, San Luis Obispo, com duas grandes melhorias: experiência prática usando Gibson Assembly e participação dos alunos em um CURE, que forneceu construções de plasmídeo originais para projetos de pesquisa financiados pela National Science Foundation (NSF-1708919 e NSF-2300890). Ao longo de três implementações deste currículo, os alunos contribuíram para dois projetos de pesquisa distintos focados na biossíntese de produtos naturais de moléculas bioativas produzidas por Actinomycetota. Os produtos naturais geralmente contêm farmacoforos com atividades antibióticas, antifúngicas e/ou anticancerígenas, dando a essas pequenas moléculas importância nos esforços de descoberta de medicamentos e potencial de relevância clínica14. Esta pesquisa requer a criação de bibliotecas de plasmídeos para permitir investigações da função e do potencial de engenharia das enzimas biossintéticas bacterianas. Neste CURE, os alunos projetaram e realizaram experimentos de montagem Gibson para clonar as bibliotecas de plasmídeos exclusivas pertinentes a esses projetos de pesquisa (Figura 1). Além disso, o formato e o design do módulo são distintos porque são facilmente adaptáveis para gerar quaisquer plasmídeos de interesse para outros projetos de pesquisa.
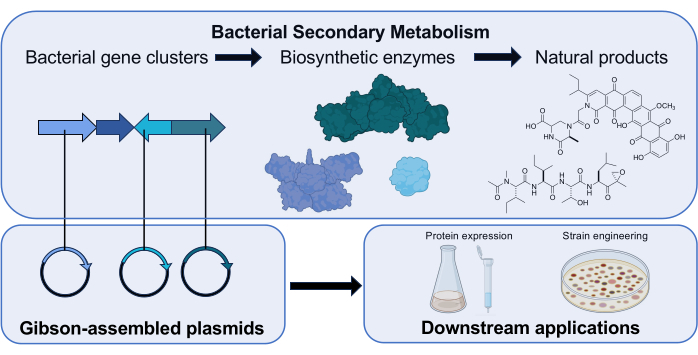
Figura 1: Visão geral do papel da Gibson Assembly em nosso laboratório de pesquisa. Actinomycetota produz produtos naturais de pequenas moléculas com bioatividades clinicamente relevantes usando clusters de genes que codificam enzimas biossintéticas. Em nossa pesquisa, plasmídeos contendo um gene biossintético são montados via Gibson Assembly para investigações a jusante da função da enzima codificada. Ícones da ciência de Biorender.com. Clique aqui para ver uma versão maior desta figura.
Visão geral do laboratório
O Laboratório de Biologia Molecular é um curso de divisão superior necessário para graduados em bioquímica e biologia com concentração em biologia molecular e celular. Outros alunos que atendam aos pré-requisitos são bem-vindos para fazer o curso como uma eletiva de divisão superior. O curso é co-listado entre o Departamento de Química e Bioquímica e o Departamento de Biologia da Cal Poly. O corpo docente de ambos os departamentos se reveza no ensino do curso a cada semestre (dois trimestres de Biologia, um quarto de Química e Bioquímica).
A turma se reúne no laboratório duas vezes por semana por períodos de 170 minutos e uma vez por semana para uma palestra de 50 minutos. As seções de laboratório contêm no máximo 16 alunos e 2 a 3 seções de laboratório são oferecidas a cada trimestre. O curso dura 10 semanas e o exame final é administrado durante a última reunião da semana 10. O tempo de aula é gasto discutindo a teoria por trás de muitas das técnicas experimentais conduzidas no laboratório, bem como tópicos atuais em biologia molecular que não são abordados no laboratório. O currículo básico do laboratório abrange o processo de clonagem do gene da actina da levedura1, que leva aproximadamente 7 semanas (13 a 14 reuniões de laboratório). As técnicas incluem micropipetagem, isolamento de RNA de levedura, amplificação de um gene de levedura usando reação em cadeia da polimerase com transcrição reversa (RT-PCR), clonagem TOPO, triagem azul-branca, isolamento de plasmídeo e verificação de inserção por digestão de restrição e PCR, análise in silico de clones e análise de sequência de DNA. O currículo das últimas 3 semanas do curso fica a critério do instrutor, mas envolve os alunos concluindo um "projeto independente" com resultados indefinidos.
Visão geral do experimento
Um foco do nosso grupo de pesquisa são as vias biossintéticas em Actinomycetota. Ao projetar o projeto independente, imaginamos que os alunos criassem plasmídeos por meio da Gibson Assembly para uso em nossos projetos de pesquisa que investigam a biossíntese de produtos naturais. Embora as iterações do módulo do curso avaliadas aqui sejam específicas para plasmídeos que permitem a manipulação de vias biossintéticas, o fluxo de trabalho da Gibson Assembly é extremamente adaptável para outros projetos de clonagem molecular (Figura 2). O fluxo de trabalho foi dividido em três experimentos diferentes (A, B e C) que foram concluídos em dois períodos de aula e seis períodos de laboratório (3 semanas no total) (consulte o Arquivo Suplementar 1 e o Arquivo Suplementar 2). Os experimentos foram precedidos por planilhas para apoiar a preparação do aluno e avaliar a compreensão do aluno (Arquivo Suplementar 3, Arquivo Suplementar 4 e Arquivo Suplementar 5). O fluxo de trabalho é apresentado em um formato flexível às necessidades e interesses de um instrutor.

Figura 2: Fluxo de trabalho do módulo Gibson Assembly. O dia 1 e o dia 4 são períodos de aula em que os alunos concluem a análise de sequência in silico e o projeto experimental. Os dias 2 a 3 e 5 a 8 são reuniões de laboratório onde são realizadas as etapas para clonar novos plasmídeos por meio da Gibson Assembly, seguidas de isolamento e triagem. O fluxograma pictórico é agrupado pelos três experimentos que os alunos realizam (A, B e C). Instruções e protocolos mais detalhados podem ser encontrados nos Manuais do Instrutor e do Aluno fornecidos como Arquivo Suplementar 1 e Arquivo Suplementar 2, respectivamente. Ícones da ciência de Biorender.com. Clique aqui para ver uma versão maior desta figura.
O módulo de projeto independente da Gibson Assembly foi testado pela primeira vez no trimestre da primavera de 2019 no CHEM/BIO 475. Em 2020 e 2021, o curso foi ministrado online devido à pandemia de SARS-CoV-2. Quando as aulas presenciais foram retomadas na primavera de 2022 e 2023, os alunos do curso foram convidados a participar de um estudo avaliando os resultados de aprendizagem de um projeto independente da Gibson Assembly, onde plasmídeos originais e relevantes para a pesquisa seriam clonados. Em 2019, os alunos criaram uma biblioteca de plasmídeos consistindo em de genes do genoma de Micromonospora echinospora ATCC 15837 que foram clonados em pKC1132 (Figura 3). Esta biblioteca de plasmídeos está sendo utilizada em nosso laboratório de pesquisa para inativar genes de interesse em um suposto cluster de genes biossintéticos para o produto natural TLN-0522015. Para complementar nossos estudos de inativação gênica, os alunos em 2022 clonaram uma pequena biblioteca de genes do suposto cluster de genes TLN-05220 em pUC19 (Figura3); nosso grupo de pesquisa utilizou esses plasmídeos para subclonar genes em vetores de expressão, incluindo pET28b, para superexpressão e purificação de proteínas. Os alunos da coorte de 2023 contribuíram para o trabalho em andamento em um projeto de engenharia biossintética sobre a epoxomicina sintetase16. Em equipes de 3 a 4, os alunos clonaram domínios de engenharia de módulos de peptídeo sintatética17 não ribossômico em vários vetores de expressão de proteínas para otimizar a superexpressão e purificação dessas enzimas em nosso laboratório de pesquisa (Figura 3). A redundância foi incorporada ao plano de clonagem para cada coorte. Por exemplo, a coorte de 2019 continha 44 alunos e 15 plasmídeos foram designados para a turma para clonagem. Assim, a clonagem de cada plasmídeo foi tentada duas ou três vezes.
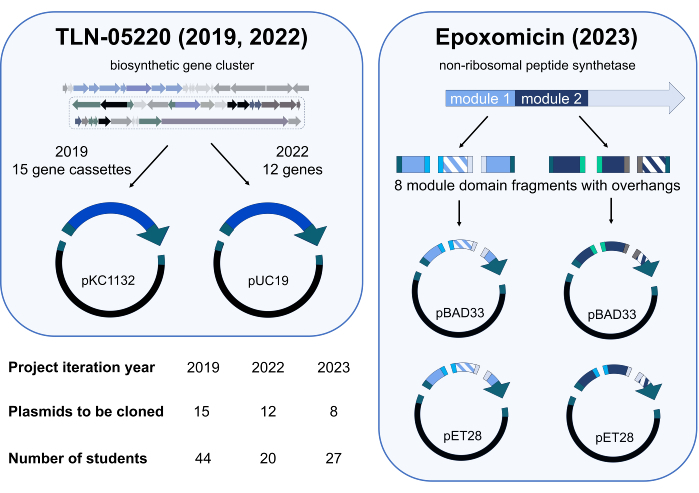
Figura 3: Resumo dos plasmídeos clonados e dos alunos participantes durante as iterações independentes do projeto de 2019, 2022 e 2023. O projeto Gibson Assembly foi implantado três vezes. Em cada oferta, os alunos participantes clonaram uma biblioteca diferente de plasmídeos para serem utilizados em projetos de pesquisa que exploram vias biossintéticas. Projetos em 2019 e 2022 apoiaram nosso trabalho contínuo no produto natural TLN-0522015 com duas reações de fragmento (um de genes ou gene e um vetor) Gibson Assembly. O projeto de 2023 envolveu a troca de domínio nos módulos 1 e 2 de uma enzima peptídeo sintetase não ribossômica (NRPS) que está envolvida na biossíntese de epoxomicina16. Os fragmentos hachurados representam dois mutantes diferentes do domínio trocado e as cores sólidas representam domínios que não foram trocados. No total, oito fragmentos de genes diferentes (quatro para o módulo 1 e quatro para o módulo 2) foram gerados com saliências compatíveis para Gibson Assembly. Para cada módulo, duas combinações diferentes de três fragmentos de genes foram montadas com um dos dois vetores diferentes (pBAD33 e pET28, quatro fragmentos totais por conjunto), para o potencial de gerar oito plasmídeos NRPS projetados. Ícones da ciência de Biorender.com. Clique aqui para ver uma versão maior desta figura.
Visão geral da avaliação
Pelo menos 1 semana antes do início do projeto independente Gibson Assembly, os alunos das coortes de 2022 e 2023 foram convidados a participar de uma investigação de resultados de aprendizagem, projetada de forma semelhante a um estudo realizado em uma pesquisa do curso de laboratório de bioquímica na Cal Poly18. Os alunos participantes preencheram um pré-questionário de múltipla escolha na reunião de laboratório antes do início do projeto independente e um pós-questionário de múltipla escolha durante a reunião final do laboratório (ou seja, depois de terem concluído o módulo Gibson Assembly). Os pré e pós-questionários consistiram em 28 questões idênticas com duas perguntas adicionais no pós-questionário (30 no total). Dez questões de conteúdo foram escritas para avaliar o conhecimento dos alunos sobre enzimas e mecanismos envolvidos na clonagem molecular (por exemplo, reação em cadeia da polimerase [PCR], Gibson Assembly, transformação, triagem azul-branco). As sete perguntas a seguir pediram aos alunos que autoavaliassem sua familiaridade com os termos de clonagem molecular (por exemplo, DNA polimerase, exonuclease, ligase). As próximas 10 perguntas permitiram que os alunos autoavaliassem sua capacidade de realizar técnicas de clonagem molecular (por exemplo, análise de sequência de DNA, reações de digestão de restrição, eletroforese em gel de agarose). Os alunos também relataram se se sentiriam confortáveis em seguir uma carreira em biologia molecular com base em seu conhecimento de técnicas de clonagem molecular. Duas perguntas adicionais foram incluídas no pós-questionário para que os alunos autoavaliassem seu compromisso com o aprendizado no curso e se o curso foi uma experiência de aprendizado valiosa (Arquivo Suplementar 6 e Arquivo Suplementar 7). Todos os dados das respostas pré e pós-questionário dos alunos de 2022 e 2023 foram combinados para análise e estão disponíveis na Tabela Suplementar S1.
Protocolo
Os estudos envolvendo participantes humanos foram revisados e aprovados por sujeitos humanos no Conselho de Revisão Institucional de Pesquisa da Cal Poly (2022-113-CP (IRB)). Os participantes forneceram seu consentimento informado por escrito para participar deste estudo.
O protocolo a seguir descreve a preparação do instrutor (etapas 1.1–1.3), as ações dos alunos para um módulo de ensino de três experimentos que inclui PCR para obter fragmentos lineares (etapas 2.1–2.7), montagem, transformação e seleção de clones de Gibson (etapas 3.1–3.5), isolamento e triagem de plasmídeo (etapas 4.1–4.4) e avaliação dos resultados de aprendizagem (5.1–5.2). A preparação do instrutor descreve um exemplo representativo de projeto de primer e preparação de um mapa de plasmídeo desejado in silico. Todas as seções do protocolo são adaptáveis para outros plasmídeos desejados. Cada experimento do aluno é dividido em duas reuniões de laboratório de 3 h.
1. Preparação do instrutor
- Projeto de primer de montagem Gibson usando o Benchling Assembly Wizard
- Determine a fonte do molde de DNA para a(s) inserção(ões) do gene, como DNA genômico, plasmídeo e DNA sintético.
NOTA: Para genes que serão amplificados a partir de modelos complexos de DNA genômico, é aconselhável primeiro determinar locais de ligação exclusivos para primers dentro do modelo de DNA genômico que são específicos para o(s) gene(s) de interesse, usando a Ferramenta de Pesquisa de Alinhamento Local Básico (BLAST) do National Center for Biotechnology Information (NCBI)21, conforme detalhado no Arquivo Suplementar 1. Para modelos simples, como plasmídeos ou DNA sintético, prossiga para a próxima etapa. - Recupere sequências de DNA da(s) inserção(ões) do gene e do vetor (por exemplo, consulte a sequência do gene da policetídeo hidroxilase e a sequência pUC19 fornecidas como arquivos GenBank no Arquivo Suplementar 8). Importe a(s) sequência(s) de inserção desejada(s) e a sequência vetorial desejada para o Benchling, cada uma como um novo arquivo de sequência de DNA. Abra cada sequência importada a ser incluída na reação de montagem de Gibson.
NOTA: Pode-se importar arquivos de sequência de DNA em vários formatos, incluindo GenBank e FASTA. Também é possível importar uma sequência diretamente de um banco de dados com um número de acesso ou colar uma sequência de nucleotídeos copiada de outro arquivo ou visualizador. - Na parte inferior da tela, localize a ferramenta Assistente de montagem . Clique em Assistente de montagem e selecione Criar nova montagem. Nas opções fornecidas, selecione Gibson e clique em Iniciar para iniciar a montagem.
- Na janela de sequência vetorial , selecione todas as bases a serem incluídas na montagem a partir do backbone vetorial. Inicie a seleção na posição do nucleotídeo na extremidade 3' da inserção do gene (por exemplo, posição 657 de pUC19) e selecione todos os nucleotídeos restantes a serem incluídos (por exemplo, através da posição 656 para incluir toda a sequência de pUC19). Uma vez selecionado, clique na guia Backbone na parte inferior da tela| Definir fragmento.
NOTA: Alterne entre as visualizações Mapa de sequência e Plasmídeo do vetor para selecionar todas as bases desejadas de um modelo circular. Alternativamente, se estiver planejando preparar o fragmento vetorial por meio de digestão de restrição (em vez de PCR) para Gibson Assembly, linearize a sequência de backbone com um local de corte de enzima de restrição (por exemplo, XbaI). Para fazer isso, clique no local de corte, mantenha pressionada a tecla Shift e clique no local de corte novamente. Em seguida, clique em Definir fragmento para definir o backbone. Lembre-se de que os primers específicos do vetor não serão gerados pelo Benchling se o backbone for linearizado com uma enzima de restrição no Assistente de montagem. - Na janela de sequência de inserção , selecione todas as bases da pastilha a serem incluídas na montagem. Uma vez selecionado, clique na guia Inserir na parte inferior da tela | Fragmento de conjunto.
- No caso de várias inserções de genes, clique no botão + no lado direito do Assistente de montagem. Na janela Sequência de inserção , selecione todas as bases de pastilhas a serem incluídas na montagem. Uma vez selecionado, clique na guia Inserir na parte inferior da tela | Fragmento de conjunto.
- Depois que todos os fragmentos estiverem definidos, renomeie a montagem para o nome de plasmídeo desejado e clique em Montar à direita do Assistente de montagem. Selecione o local da pasta desejada para a Pasta de sequência e a Pasta do Primer. Clique em Criar para montar a sequência de plasmídeo recombinante.
- Abra o plasmídeo montado e clique em Histórico de montagem para visualizar um mapa básico de plasmídeo e uma visão geral das sequências das quais ele foi derivado. Certifique-se de que as posições dos nucleotídeos para cada fragmento correspondam ao design desejado.
- Na guia Parâmetros de montagem , visualize os nomes dos primers projetados para a montagem. Observe que haverá dois primers projetados para cada inserção (um direto e um reverso), e os nomes dos primers serão derivados dos títulos dos arquivos de sequência de DNA. Certifique-se também de que as temperaturas de fusão do primer e as temperaturas de recozimento sugeridas sejam compatíveis nesta janela (consulte o Arquivo Suplementar 8, Primers Representativos).
- Se as temperaturas de fusão do primer ou as temperaturas de recozimento não forem ideais, ajuste manualmente as sequências do primer (por exemplo, removendo nucleotídeos para reduzir a temperatura de fusão) nos arquivos de sequência do primer. Como alternativa, clique em Reabrir e ajuste as configurações do primer clicando no botão Configurações de enzima/primer ao lado do botão Montar no Assistente de montagem. Em seguida, repita a etapa de montagem e reexamine as sequências de primer.
- Se uma biblioteca de plasmídeos for clonada com muitos genes diferentes clonados na mesma estrutura vetorial,
- Utilize o mesmo par de primers para o vetor em cada caso. Para fazer isso, remova manualmente todos os nucleotídeos dos primers vetoriais que instalariam as saliências específicas da inserção. Esses nucleotídeos seriam encontrados na extremidade 5 ' do primer. Após a remoção, certifique-se de que as temperaturas de fusão do primer e as temperaturas de recozimento sejam compatíveis (consulte o Arquivo Suplementar 8, Primers Representativos).
- Certifique-se de que todos os pares de primers de pastilhas tenham temperaturas de fusão e temperaturas de recozimento muito semelhantes.
NOTA: Isso reduzirá a necessidade de otimização da temperatura de recozimento de cada par e permitirá que todas as reações de PCR dos alunos sejam cicladas ao mesmo tempo usando o mesmo programa de termociclador.
- Quando o projeto do primer estiver concluído para os plasmídeos desejados, clique em Finalizar no Assistente de montagem. Encomende os primers de uma empresa de síntese de DNA e recupere amostras dos modelos de DNA para inserção(ões) e vetor.
- Teste os pares de primers específicos para inserção e vetor para temperaturas ideais de recozimento usando um experimento de PCR gradiente com os modelos de DNA. Teste as temperaturas de recozimento recomendadas pela bancada ± 3 °C inicialmente.
- Uma vez que as temperaturas de recozimento tenham sido determinadas, crie alíquotas de soluções de primer, modelos de DNA e reagentes necessários para reações de PCR para os alunos.
- Determine a fonte do molde de DNA para a(s) inserção(ões) do gene, como DNA genômico, plasmídeo e DNA sintético.
- Antes dos experimentos, forneça planilhas aos alunos para apoiar a preparação e avaliar a compreensão (Arquivo Suplementar 4, Suplementar 5 e Suplementar 6).
- Recolha de dados sobre os resultados de aprendizagem
NOTA: Se estiver interessado em coletar dados de resultados de aprendizagem do módulo Gibson Assembly e publicar os resultados, obtenha aprovação para pesquisas com seres humanos de acordo com as políticas da instituição.- Peça aos alunos que participem de uma investigação de resultados de aprendizagem pelo menos 1 semana antes do início do projeto independente da Gibson Assembly.
- Peça aos alunos participantes que preencham o pré-questionário de múltipla escolha (Arquivo Suplementar 6) na reunião de laboratório antes do início do projeto independente.
2. Experiência A: PCR para obtenção de fragmentos lineares
- Obtenha primers e soluções de DNA modelo para PCR com o instrutor.
- Pipete reações de 25 μL, incluindo DNA polimerase MasterMix, molde de DNA, primers diretos e reversos e água livre de nuclease de acordo com as instruções do manual do aluno (consulte o Arquivo Suplementar 2).
- Alterne a reação em um termociclador de acordo com o manual do aluno no Arquivo Suplementar 2. Certifique-se de que a temperatura de recozimento da reação seja apropriada para os primers e que o tempo de extensão seja apropriado para o comprimento do amplicon desejado. Consulte o Arquivo Suplementar 2 e o Arquivo Suplementar 3 para obter instruções detalhadas.
- Armazene as reações de PCR a 4 ° C ou -20 ° C até o período de laboratório seguinte.
- Analise 5 μL de cada reação por eletroforese em gel de agarose. Enquanto o gel estiver funcionando, adicione 1 μL de enzima de restrição DpnI a cada reação que utilizou DNA de plasmídeo como molde. Incubar esta mistura durante 1 h a 37 °C.
NOTA: O DpnI degradará qualquer molde de DNA de plasmídeo circular restante, tornando menos provável a obtenção de falsos positivos na etapa de transformação. - Visualize o gel para confirmar que a reação em cadeia da polimerase foi bem-sucedida e a amplificação correta foi alcançada. Purifique todas as reações de PCR bem-sucedidas com um kit de purificação de PCR disponível comercialmente. Meça a concentração (ng/μL) do produto de PCR purificado usando um espectrofotômetro de microvolume para uso em cálculos de montagem Gibson subsequentes.
- Antes da próxima reunião de classe, projete a receita de reação para o Gibson Assembly (consulte o Arquivo Suplementar 2 e o Arquivo Suplementar 4).
3. Experiência B do aluno: Montagem de Gibson, transformação e seleção de clones
- Pipete a reação do conjunto Gibson de acordo com a receita projetada. Incubar a reação a 50 °C durante 15 min. Enquanto as reações incubam, prepare-se para a transformação descongelando células de Escherichia coli quimicamente competentes no gelo.
- Transforme 2 μL do conjunto Gibson em células quimicamente competentes por meio de choque térmico. Pipetar 100 μL das células transformadas em duas placas de seleção e espalhar com esferas esterilizadas. Prepare diluições 10-4, 10-5 e 10-6 das células restantes em ágar Luria-Bertani (LB). Colocar 100 μL de cada diluição seriada em LB (sem seleção) e espalhar com esferas esterilizadas.
NOTA: Esta etapa permite o cálculo da eficiência da transformação. - Incubar as placas durante a noite a 37 °C. Conservar as placas a 4 °C até ao próximo período de aula.
- Conte as colônias em todas as placas e calcule a eficiência da transformação conforme instruído no Arquivo Suplementar 2. Voltar a riscar uma placa selectiva com quatro colónias de uma das placas selectivas e incubar esta placa a 37 °C durante a noite.
NOTA: Isso serve como uma cultura de backup de seus clones positivos. - Usando um marcador, selecione, circule e rotule quatro colônias distintas na placa de seleção com as iniciais do aluno e um número (por exemplo, ABC1). Recupere uma nova placa de ágar LB contendo antibiótico para seleção, use um marcador para dividir a placa em quadrantes (ou seja, quartos) e, em seguida, use ~ 1/2 de cada colônia para re-streak no quadrante respectivamente marcado e o outro 1/2 para inocular uma cultura líquida de LB de 5 mL para cada uma das quatro colônias selecionadas. Certifique-se de rotular os tubos com a respectiva identidade da colônia (por exemplo, ABC1) e adicione a concentração correta de antibiótico para seleção. Incubar as culturas líquidas numa incubadora agitada a 37 °C durante a noite e a placa de ágar numa incubadora estática a 37 °C; armazenar as culturas líquidas e as placas a 4 °C até ao período de aula seguinte.
4. Experiência do aluno C: Isolamento e triagem de plasmídeos
- Isole o DNA do plasmídeo das culturas líquidas do Experimento B usando um kit de minipreparação. Medir a concentração do plasmídeo isolado (em ng/μl).
- Projete um resumo de restrição ou triagem de PCR para analisar os plasmídeos isolados (consulte as instruções detalhadas no Arquivo Suplementar 2 e relate as expectativas no Arquivo Suplementar 5).
- Pipetar as reações de digestão de restrição ou reações de acordo com as receitas projetadas e incubar em temperaturas e durações projetadas anteriormente.
- Analise os resultados por eletroforese em gel de agarose.
5. Apreciação
- Peça aos alunos participantes que preencham o pós-questionário de múltipla escolha (Arquivo Suplementar 7) na reunião de laboratório após a conclusão do módulo Gibson Assembly.
- Combine todos os dados das respostas pré e pós-questionário para análise.
- Examine a significância estatística do aumento na pontuação média de cada questão de conteúdo individual entre os questionários usando testes t pareados bicaudais e o tamanho do efeito d de Cohen.
- Avalie o aumento da confiança do aluno para cada termo e técnica individual para determinar a extensão da mudança e sua significância estatística.
- Determine o impacto do histórico do aluno e dos fatores acadêmicos na aprendizagem do aluno usando ganho de aprendizagem normalizado (NLG) e testes t não pareados.
- Avalie as atitudes dos alunos em relação à experiência do projeto e às carreiras de biologia molecular, avaliando as pontuações médias.
Resultados
Sucesso do aluno na clonagem
Em cada iteração do módulo Gibson Assembly (2019, 2022 e 2023), os alunos foram solicitados a preparar um relatório resumindo suas descobertas. Em 2019, 36 dos 44 alunos (81,8%) relataram que clonaram com sucesso seus plasmídeos com base nos resultados da tela que projetaram para o Experimento C. Um total de 14 em cada 20 alunos (70,0%) relataram sucesso na clonagem de seus construtos atribuídos em 2022, enquanto o projeto baseado em...
Discussão
Aqui, propomos um projeto de laboratório de sala de aula de graduação ajustável que ensina aos alunos a clonagem molecular por meio da Gibson Assembly em um ambiente de pesquisa baseado em curso. No total, 28 novos plasmídeos foram clonados por alunos de graduação em sala de aula. O sucesso individual relatado pelos alunos variou de 44,4% a 81,8% em três coortes, e o sucesso geral da clonagem foi de 80% (28 do total de 35 plasmídeos atribuídos foram clonados com sucesso). O men...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Os autores agradecem a Andrea Laubscher pelo suporte técnico e Michael Black, Sandi Clement e Javin Oza pelas discussões úteis sobre a implementação do laboratório de ensino e a avaliação dos resultados da aprendizagem. Os autores agradecem a todos os alunos que participaram do estudo de resultados de aprendizagem nas coortes de 2019, 2022 e 2023, bem como aos alunos de pesquisa Nathan Kuhn e Aayushi Adettiwar, que ajudaram na preparação de reagentes para a implementação do laboratório de ensino. Os autores também reconhecem o apoio financeiro do William and Linda Frost Fund, do Chevron Biotechnology Applied Research Endowment Grant do Center for Applications in Biotechnology e da National Science Foundation (NSF-1708919 e NSF-2300890).
Materiais
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Referências
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados