Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Research Article
Verbesserung der Studienergebnisse mit einer anpassungsfähigen kursbasierten Forschungserfahrung im Grundstudium zum molekularen Klonen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Ein anpassungsfähiges Gibson Assembly Modul zur molekularen Klonierung wurde in einem kursbasierten CURE-Format (Undergraduate Research Experience) für Studenten von Molekularbiologie-Laborkursen eingesetzt. Die Bewertung der Lernergebnisse der Schüler zeigte ein verbessertes Verständnis und Vertrauen in die molekulare Klonierung nach Abschluss des CURE, und neuartige Plasmide wurden für die Forschung zur Biosynthese von Naturstoffen kloniert.
Zusammenfassung
Die kontinuierliche Weiterentwicklung molekularbiologischer Techniken erfordert, dass die Lehrpläne der Molekularbiologie regelmäßig verfeinert werden, um die Studierenden effektiv auf den Eintritt in die Arbeitswelt mit modernen Kompetenzen vorzubereiten. Insbesondere das Aufkommen der Gibson-Assemblierung, einer hochgradig anpassbaren und anpassungsfähigen molekularen Klonierungstechnik, hat die Landschaft des molekularen Klonens in zahlreichen Forschungsumgebungen vorangebracht. Aus diesem Grund haben wir ein Gibson-Assembly-Klonierungsmodul für den Einsatz in einem molekularbiologischen Laborkurs an der California Polytechnic State University, San Luis Obispo, entwickelt und die Lernergebnisse der Studierenden aus dem Modul ausgewertet. In drei Iterationen des Kurses nahmen die Studierenden an einem experimentellen, unabhängigen Projekt teil, bei dem drei einzigartige Plasmidbibliotheken kloniert wurden, um Forschungsprojekte in der Biosynthese von Naturstoffen zu unterstützen. Die Studierenden erhielten Vor- und Nachfragebögen, um ihr Verständnis des molekularen Klonens und ihr Vertrauen in molekularbiologische Begriffe und Techniken zu bewerten. Die Antworten der Studierenden zeigten eine signifikante Zunahme sowohl beim Erlernen von molekularen Klonierungskonzepten als auch bei der Selbsteinschätzung des Vertrauens in Begriffe und Techniken des molekularen Klonens. Dieses Modul-Framework kann verallgemeinert werden, um Gibson Assembly für verschiedene Anwendungen zu unterrichten und den Dozenten ein Toolkit für die Vermittlung einer anpassungsfähigen und aufstrebenden Klonierungstechnologie zur Verfügung zu stellen, während sie ihre Forschungsprojekte vorantreiben.
Einleitung
Die Ausbildung von Studenten in grundlegenden molekularbiologischen Konzepten und Labortechniken ist für ihre wissenschaftliche und berufliche Entwicklung von entscheidender Bedeutung, da diese Methoden in verschiedenen Forschungsbereichen, einschließlich Hochschulen und Industrie, üblich sind. Daher müssen Studierende der Hauptfächer Biologie (Konzentration Molekular- und Zellbiologie) und Biochemie an der California Polytechnic State University, San Luis Obispo (Cal Poly) einen molekularbiologischen Laborkurs der Oberklasse belegen, um diese Themen zu erlernen und anzuwenden (CHEM/BIO 475). Zuvor wurde ein Basiscurriculum für diesen Kurs entwickelt, in dem die Studierenden eine Topoisomerase-basierte (TOPO) Klonierung durchführen, um ein Aktin-haltiges Plasmid zusammenzusetzen, das aus einer Hefe-Komplementär-DNA (cDNA)-Vorlagehergestellt wird 1. Die Schülerinnen und Schüler entwerfen Experimente auf der Grundlage von Fragen, die authentische Forschungshypothesen nachahmen, und erhöhen so ihre Vertrautheit mit Laborpraktiken und forschendem Lernen. Die kontinuierliche Weiterentwicklung auf dem Gebiet der Molekularbiologie erfordert eine Anpassung der entsprechenden Lehrpläne, um die Studierenden mit modernen Kompetenzen auf die Arbeitswelt vorzubereiten. Insbesondere die Verwendung der Gibson-Assembly hat sich in der wissenschaftlichen Gemeinschaft immer mehr durchgesetzt; Während die Methode ursprünglich zur Synthese künstlicher Chromosomen2 etabliert wurde, haben zum Zeitpunkt dieses Berichts über 5.000 Veröffentlichungen auf die ursprüngliche Arbeit von Gibson et al. verwiesen. Die Gibson-Assemblierung ist im Vergleich zu herkömmlichen Klonierungsmethoden einzigartig: Sie ist hochgradig anpassbar und kann problemlos mehrere lineare DNA-Fragmente ligieren, ohne dass Restriktionsstellen zur Herstellung der Verbindungen erforderlich sind. Daher sahen wir die Möglichkeit, das CHEM/BIO 475-Curriculum zu überarbeiten, um moderne molekulare Klonierungstechniken einzubeziehen und das forschungsbasierte Kursmodell zu verbessern.
Es wurde festgestellt, dass studentische Forschungserfahrungen zu einem verbesserten konzeptionellen Verständnis, zur Entwicklung von Fähigkeiten und zu einem besseren Durchhaltevermögen in der Wissenschaftbeitragen 3, aber nicht alle Studenten im Grundstudium haben die Möglichkeit, direkt an einem Forschungslabor teilzunehmen. Um der Herausforderung der begrenzten studentischen Kapazitäten in Forschungslabors zu begegnen, wurden kursbasierte Forschungserfahrungen (CUREs) entwickelt und eingesetzt, um den Zugang zu Wissenschaft durch authentische Forschung im Klassenzimmer zu verbessern. Während CUREs in ihrer Umsetzung unterschiedlich sind, haben sich gemeinsame Praktiken etabliert, die sich mit den fünf Grundsätzen der wissenschaftlichen Forschung befassen. In einem gut konzipierten CURE werden die Studierenden 1) wissenschaftliche Praktiken anwenden, 2) an einem Forschungsprojekt mitarbeiten, 3) versuchen, neue Entdeckungen zu machen, 4) zu einer außerhalb des Klassenzimmers relevanten Arbeit beitragen und 5) Hypothesen und Methoden im Falle eines experimentellen Scheiterns neu bewerten und überarbeiten4. Ähnlich wie bei herkömmlichen Forschungserfahrungen von Schülern in einem Labor hat sich gezeigt, dass CUREs das Vertrauen der Schüler in die Wissenschaft, die wissenschaftlichen Fähigkeiten, die Projektverantwortung und die Beharrlichkeit in den Bereichen Mathematik, Informatik, Naturwissenschaften und Technik (MINT) stärken5. Während CUREs, die molekulare Klonierung beinhalten, bereits zuvor berichtet wurden 6,7,8,9,10,11,12,13, sind uns keine bekannt, die die Anpassungsfähigkeit der Gibson-Assemblierung zur Erstellung einer Bibliothek authentischer Forschungsplasmide betonen.
Hier berichten wir über eine Erweiterung des aktuellen forschungsbasierten CHEM/BIO 475-Curriculums an der California Polytechnic State University, San Luis Obispo, mit zwei wesentlichen Verbesserungen: praktische Erfahrungen mit der Gibson Assembly und studentische Teilnahme an einem CURE, das originelle Plasmidkonstrukte für Forschungsprojekte bereitgestellt hat, die von der National Science Foundation finanziert wurden (NSF-1708919 und NSF-2300890). In drei Implementierungen dieses Curriculums haben die Studierenden zu zwei unterschiedlichen Forschungsprojekten beigetragen, die sich auf die Naturstoffbiosynthese von bioaktiven Molekülen konzentrieren, die von Actinomycetota produziert werden. Naturstoffe enthalten häufig Pharmakophore mit antibiotischen, antimykotischen und/oder krebshemmenden Wirkungen, was diesen kleinen Molekülen Bedeutung für die Wirkstoffforschung und Potenzial für klinische Relevanz verleiht14. Diese Forschung erfordert die Erstellung von Plasmidbibliotheken, um sowohl die Funktion als auch das technische Potenzial bakterieller biosynthetischer Enzyme untersuchen zu können. In diesem CURE entwarfen und führten die Studierenden Gibson-Assembly-Experimente durch, um die einzigartigen Plasmidbibliotheken zu klonen, die für diese Forschungsprojekte relevant sind (Abbildung 1). Darüber hinaus zeichnen sich das Format und das Design des Moduls dadurch aus, dass es leicht anpassbar ist, um beliebige Plasmide zu erzeugen, die für andere Forschungsprojekte von Interesse sind.
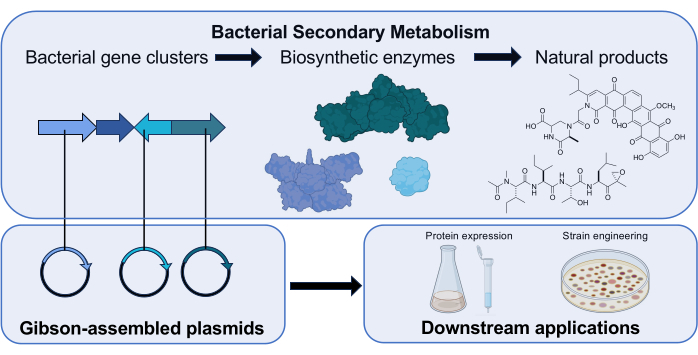
Abbildung 1: Überblick über die Rolle der Gibson-Montage in unserem Forschungslabor. Actinomycetota stellen niedermolekulare Naturstoffe mit klinisch relevanten Bioaktivitäten her, indem sie Gencluster verwenden, die für biosynthetische Enzyme kodieren. In unserer Forschung werden Plasmide, die ein biosynthetisches Gen enthalten, über Gibson-Assemblierung assembliert, um die Funktion des kodierten Enzyms nachgelagert zu untersuchen. Wissenschaftsikonen aus Biorender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Übersicht über das Labor
Das Molecular Biology Laboratory ist ein Oberstufenkurs, der für Biochemie-Hauptfächer und Biologie-Hauptfächer mit einem Schwerpunkt auf Molekular- und Zellbiologie erforderlich ist. Andere Studierende, die die Voraussetzungen erfüllen, sind herzlich eingeladen, den Kurs als Wahlfach der Oberstufe zu belegen. Der Kurs ist gemeinsam mit dem Fachbereich Chemie und Biochemie und dem Fachbereich Biologie an der Cal Poly gelistet. Dozenten beider Fachbereiche wechseln sich in jedem Semester ab (zwei Viertel Biologie, ein Viertel Chemie und Biochemie).
Die Klasse trifft sich zweimal pro Woche für 170 Minuten und einmal pro Woche für eine 50-minütige Vorlesung im Labor. Laborabschnitte umfassen maximal 16 Studierende und jedes Quartal werden 2-3 Laborabschnitte angeboten. Der Kurs dauert 10 Wochen, und die Abschlussprüfung wird während des letzten Termins in Woche 10 durchgeführt. In der Vorlesungszeit wird die Theorie hinter vielen der im Labor durchgeführten experimentellen Techniken sowie aktuelle Themen der Molekularbiologie diskutiert, die im Labor nicht behandelt werden. Der Kernlehrplan des Labors umfasst den Prozess der Klonierung des Aktin-Gens aus Hefe1, der etwa 7 Wochen (13-14 Laborsitzungen) dauert. Zu den Techniken gehören Mikropipettieren, Hefe-RNA-Isolierung, Amplifikation eines Hefegens mittels reverser Transkriptions-Polymerase-Kettenreaktion (RT-PCR), TOPO-Klonierung, Blau-Weiß-Screening, Plasmidisolierung und Verifizierung des Inserts durch Restriktionsverdau und PCR, In-silico-Analyse von Klonen und DNA-Sequenzanalyse. Der Lehrplan für die letzten 3 Wochen des Kurses liegt im Ermessen des Dozenten, beinhaltet jedoch die Durchführung eines "unabhängigen Projekts" mit undefinierten Ergebnissen.
Übersicht über Experimente
Ein Schwerpunkt unserer Arbeitsgruppe sind die Biosynthesewege in Actinomycetota. Bei der Konzeption des unabhängigen Projekts sahen wir vor, dass die Studenten Plasmide durch Gibson Assembly herstellen sollten, die in unseren Forschungsprojekten zur Untersuchung der Biosynthese von Naturstoffen verwendet werden sollten. Während die Iterationen des Kursmoduls, die hier bewertet werden, speziell für Plasmide waren, die die Manipulation von Biosynthesewegen ermöglichen, ist der Gibson-Assembly-Workflow enorm anpassbar für andere molekulare Klonierungsprojekte (Abbildung 2). Der Arbeitsablauf wurde in drei verschiedene Experimente (A, B und C) aufgeteilt, die über zwei Vorlesungsstunden und sechs Laborstunden (insgesamt 3 Wochen) durchgeführt wurden (siehe Ergänzungsdatei 1 und Ergänzungsdatei 2). Den Experimenten gingen Arbeitsblätter voraus, um die Vorbereitung der Schüler zu unterstützen und das Verständnis der Schüler zu bewerten (Ergänzende Akte 3, Ergänzende Akte 4 und Ergänzende Akte 5). Der Workflow wird in einem Format dargestellt, das flexibel auf die Bedürfnisse und Interessen eines Kursleiters zugeschnitten ist.

Abbildung 2: Arbeitsablauf des Gibson Assembly-Moduls. Tag 1 und Tag 4 sind Vorlesungszeiten, in denen die Studierenden die In-silico-Sequenzanalyse und die Versuchsplanung durchführen. Die Tage 2–3 und 5–8 sind Laborbesprechungen, in denen die Schritte zur Klonierung neuartiger Plasmide mittels Gibson-Assemblierung durchgeführt werden, gefolgt von Isolierung und Screening. Das bildliche Flussdiagramm ist nach den drei Experimenten gruppiert, die die Schülerinnen und Schüler durchführen (A, B und C). Ausführlichere Anweisungen und Protokolle finden Sie in den Dozenten- und Schülerhandbüchern, die als ergänzende Datei 1 bzw. ergänzende Datei 2 bereitgestellt werden. Wissenschaftsikonen aus Biorender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Das unabhängige Projektmodul Gibson Assembly wurde erstmals im Frühjahrsquartal 2019 in der CHEM/BIO 475 pilotiert. In den Jahren 2020 und 2021 wurde der Kurs aufgrund der SARS-CoV-2-Pandemie online unterrichtet. Als der Präsenzunterricht im Frühjahr 2022 und 2023 wieder aufgenommen wurde, wurden die Studierenden des Kurses eingeladen, an einer Studie teilzunehmen, in der die Lernergebnisse eines unabhängigen Projekts der Gibson Assembly bewertet wurden, bei dem originale, forschungsrelevante Plasmide kloniert werden sollten. Im Jahr 2019 erstellten die Studierenden eine Bibliothek von Plasmiden, die aus Genkassetten aus dem Genom von Micromonospora echinospora ATCC 15837 bestand, die in pKC1132 kloniert wurden (Abbildung 3). Diese Plasmidbibliothek wird in unserem Forschungslabor verwendet, um Gene von Interesse in einem mutmaßlichen biosynthetischen Gencluster für den Naturstoff TLN-0522015 zu inaktivieren. Um unsere Geninaktivierungsstudien zu ergänzen, klonierten die Studierenden im Jahr 2022 eine kleine Bibliothek von Genen aus dem mutmaßlichen TLN-05220-Gencluster in pUC19 (Abbildung3); Unsere Forschungsgruppe hat diese Plasmide zur Subklonierung von Genen in Expressionsvektoren, einschließlich pET28b, für die Überexpression und Reinigung von Proteinen verwendet. Die Studierenden des Jahrgangs 2023 trugen zu den laufenden Arbeiten an einem Biosynthese-Engineering-Projekt zur Epoxomicin-Synthetase16 bei. In Teams von 3 bis 4 Klonierten die Studierenden manipulierte Domänen von nicht-ribosomalen Peptidsynthetase17-Modulen in verschiedene Proteinexpressionsvektoren, um die Überexpression und Reinigung dieser Enzyme in unserem Forschungslabor zu optimieren (Abbildung 3). Redundanz wurde in den Klonierungsplan für jede Kohorte eingebaut. Zum Beispiel enthielt die Kohorte 2019 44 Studenten, und 15 Plasmide wurden der Klasse für die Klonierung zugewiesen. Daher wurde die Klonierung jedes Plasmids zwei- oder dreimal versucht.
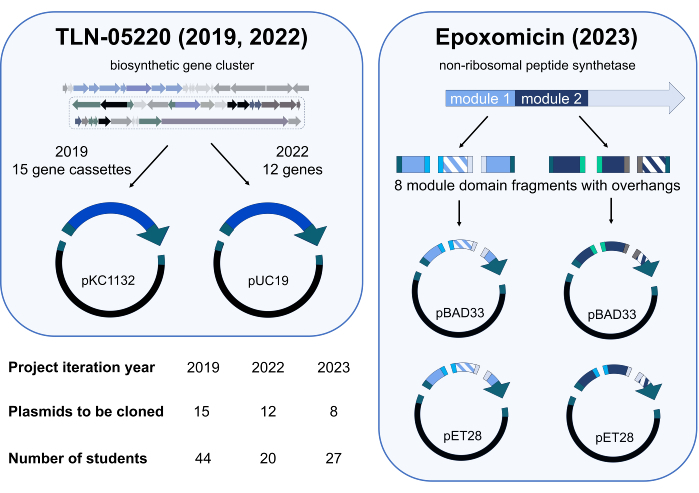
Abbildung 3: Zusammenfassung der klonierten Plasmide und der teilnehmenden Studenten während der unabhängigen Projektiterationen 2019, 2022 und 2023. Das Gibson Assembly-Projekt wurde bereits dreimal eingesetzt. In jedem Angebot klonierten die studentischen Teilnehmer eine andere Bibliothek von Plasmiden, die in Forschungsprojekten zur Erforschung von Biosynthesewegen verwendet werden sollen. Projekte in den Jahren 2019 und 2022 unterstützten unsere laufenden Arbeiten am Naturstoff TLN-0522015 mit zwei Fragmenten (eine Genkassette bzw. ein Gen und ein Vektor) Gibson Assemblierungsreaktionen. Das Projekt aus dem Jahr 2023 befasste sich mit dem Domänentausch innerhalb der Module 1 und 2 eines nicht-ribosomalen Peptidsynthetase (NRPS)-Enzyms, das an der Epoxomicin-Biosynthese beteiligt ist16. Die schraffierten Fragmente stellen zwei verschiedene Mutanten der vertauschten Domäne dar, und Volltonfarben stellen Domänen dar, die nicht vertauscht wurden. Insgesamt wurden acht verschiedene Genfragmente (vier für Modul 1 und vier für Modul 2) mit Überhängen erzeugt, die für die Gibson-Assemblierung kompatibel sind. Für jedes Modul wurden zwei verschiedene Kombinationen von drei Genfragmenten mit einem von zwei verschiedenen Vektoren (pBAD33 und pET28, insgesamt vier Fragmente pro Assemblierung) assembliert, um das Potenzial zu haben, acht manipulierte NRPS-Plasmide zu erzeugen. Wissenschaftsikonen aus Biorender.com. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Übersicht über die Bewertung
Mindestens 1 Woche vor Beginn des unabhängigen Gibson Assembly-Projekts wurden die Studierenden der Kohorten 2022 und 2023 eingeladen, an einer Lernergebnisuntersuchung teilzunehmen, die ähnlich wie eine Studie konzipiert war, die im Rahmen einer Umfrage im Biochemie-Laborkurs an der Cal Poly18 durchgeführt wurde. Die studentischen Teilnehmer füllten einen Multiple-Choice-Fragebogen im Lab-Meeting vor Beginn des unabhängigen Projekts und einen Multiple-Choice-Post-Fragebogen während des abschließenden Lab-Meetings (d.h. nachdem sie das Gibson-Assembly-Modul absolviert hatten) aus. Die Vor- und Nachfragebögen bestanden aus 28 identischen Fragen mit zwei zusätzlichen Fragen im Nachfragebogen (insgesamt 30). Es wurden zehn inhaltliche Fragen verfasst, um das Wissen der Schülerinnen und Schüler über Enzyme und Mechanismen zu bewerten, die an der molekularen Klonierung beteiligt sind (z. B. Polymerase-Kettenreaktion [PCR], Gibson-Assemblierung, Transformation, Blau-Weiß-Screening). In den folgenden sieben Fragen wurden die Studierenden aufgefordert, ihre Vertrautheit mit Begriffen der molekularen Klonierung (z. B. DNA-Polymerase, Exonuklease, Ligase) selbst einzuschätzen. Die nächsten 10 Fragen ermöglichten es den Studierenden, ihre Fähigkeit zur Durchführung molekularer Klonierungstechniken (z. B. DNA-Sequenzanalyse, Restriktionsverdaureaktionen, Agarosegelelektrophorese) selbst einzuschätzen. Die Studierenden berichteten auch, ob sie aufgrund ihres Wissens über molekulare Klonierungstechniken eine Karriere in der Molekularbiologie anstreben würden. Im Nachfragebogen wurden zwei zusätzliche Fragen gestellt, mit denen die Studierenden selbst beurteilen konnten, wie sehr sie sich für das Lernen im Kurs engagieren und ob der Kurs eine wertvolle Lernerfahrung war (Ergänzende Akte 6 und Ergänzende Akte 7). Alle Daten aus den Vor- und Nachfrageantworten der Studierenden aus den Jahren 2022 und 2023 wurden für die Analyse kombiniert und sind in der ergänzenden Tabelle S1 verfügbar.
Protokoll
Die Studien mit menschlichen Teilnehmern wurden von menschlichen Probanden im Research Institutional Review Board an der Cal Poly (2022-113-CP (IRB)) überprüft und genehmigt. Die Teilnehmer gaben ihre schriftliche Einverständniserklärung zur Teilnahme an dieser Studie ab.
Das folgende Protokoll beschreibt die Vorbereitung des Dozenten (Schritte 1.1–1.3), die Aktionen der Studierenden für ein Lehrmodul mit drei Experimenten, das PCR zur Gewinnung linearer Fragmente (Schritte 2.1–2.7), Gibson-Assemblierung, Transformation und Auswahl von Klonen (Schritte 3.1–3.5), Plasmidisolierung und -screening (Schritte 4.1–4.4) und die Bewertung der Lernergebnisse (5.1–5.2). Die Vorbereitung des Dozenten beschreibt ein repräsentatives Beispiel für das Design von Primern und die Erstellung einer gewünschten Plasmidkarte in silico. Alle Abschnitte des Protokolls sind für andere gewünschte Plasmide anpassbar. Jedes Schülerexperiment ist in zwei 3-stündige Laborsitzungen unterteilt.
1. Vorbereitung auf den Tauchlehrer
- Gibson Assembly Primer Design mit dem Benchling Assembly Wizard
- Bestimmen Sie die DNA-Template-Quelle für das/die Geninsert(s), z. B. genomische DNA, Plasmid und synthetische DNA.
ANMERKUNG: Für Gene, die aus komplexen genomischen DNA-Templates amplifiziert werden sollen, wird empfohlen, zunächst einzigartige Bindungsstellen für Primer innerhalb der genomischen DNA-Template zu bestimmen, die spezifisch für das/die interessierende(n) Gen(e) sind, und zwar unter Verwendung des Basic Local Alignment Search Tool (BLAST)21 des National Center for Biotechnology Information (NCBI), wie in Supplemental File 1 beschrieben. Für einfache Vorlagen wie Plasmide oder synthetische DNA fahren Sie mit dem nächsten Schritt fort. - Abrufen von DNA-Sequenzen des Geninserts/der Geninserts und des Vektors (siehe z. B. die Polyketid-Hydroxylase-Gensequenz und die pUC19-Sequenz, die als GenBank-Dateien in der ergänzenden Datei 8 bereitgestellt werden). Importieren Sie die gewünschte(n) Insert-Sequenz(en) und die gewünschte Vektorsequenz in Benchling, jeweils als neue DNA-Sequenzdatei. Öffnen Sie jede importierte Sequenz, die in die Reaktion Gibson Assembly einbezogen werden soll.
ANMERKUNG: DNA-Sequenzdateien können in verschiedenen Formaten importiert werden, darunter GenBank und FASTA. Man kann auch eine Sequenz direkt aus einer Datenbank mit einer Zugangsnummer importieren oder eine Nukleotidsequenz einfügen, die aus einer anderen Datei oder einem anderen Viewer kopiert wurde. - Suchen Sie am unteren Rand des Bildschirms das Werkzeug Montage-Assistent . Klicken Sie auf Baugruppen-Assistent, und wählen Sie dann Neue Baugruppe erstellen aus. Wählen Sie aus den bereitgestellten Optionen Gibson aus, und klicken Sie auf Start , um mit der Montage zu beginnen.
- Wählen Sie im Vektorsequenzfenster alle Basen aus, die in die Baugruppe aus dem Vektor-Backbone aufgenommen werden sollen. Beginnen Sie die Selektion an der Nukleotidposition am 3' -Ende des Geninserts (z. B. Position 657 von pUC19) und wählen Sie alle verbleibenden Nukleotide aus, die eingeschlossen werden sollen (z. B. durch Position 656, um die gesamte pUC19-Sequenz einzuschließen). Klicken Sie nach der Auswahl unten auf dem Bildschirm auf die Registerkarte Backbone | Fragment festlegen.
ANMERKUNG: Wechseln Sie zwischen der Sequenz-Map - und der Plasmid-Ansicht des Vektors, um alle gewünschten Basen einer kreisförmigen Vorlage auszuwählen. Wenn Sie planen, das Vektorfragment über einen Restriktionsverdau (anstelle von PCR) für die Gibson-Assemblierung vorzubereiten, linearisieren Sie die Rückgratsequenz mit einer Restriktionsenzym-Schnittstelle (z. B. XbaI). Klicken Sie dazu auf die ausgeschnittene Site, halten Sie die Umschalttaste gedrückt und klicken Sie erneut auf die ausgeschnittene Site . Klicken Sie dann auf Fragment festlegen , um das Backbone festzulegen. Beachten Sie, dass vektorspezifische Primer von Benchling nicht generiert werden, wenn das Rückgrat mit einem Restriktionsenzym im Assemblierungsassistenten linearisiert wird. - Wählen Sie im Fenster Einfügesequenz alle Basen der Einfügesequenz aus, die in die Baugruppe aufgenommen werden sollen. Klicken Sie nach der Auswahl auf die Registerkarte Einfügen am unteren Bildschirmrand | Fragment festlegen.
- Im Falle mehrerer Geninsertionen klicken Sie auf die Schaltfläche + auf der rechten Seite des Assemblierungsassistenten. Wählen Sie im Fenster Einfügesequenz alle Einfügebasen aus, die in die Baugruppe aufgenommen werden sollen. Klicken Sie nach der Auswahl auf die Registerkarte Einfügen am unteren Bildschirmrand | Fragment festlegen.
- Wenn alle Fragmente festgelegt sind, benennen Sie die Assemblierung in den gewünschten Plasmidnamen um, und klicken Sie rechts neben dem Assemblierungsassistenten auf Assemblieren. Wählen Sie den gewünschten Ordnerspeicherort sowohl für den Sequenzordner als auch für den Primerordner aus. Klicken Sie auf Erstellen, um die rekombinante Plasmidsequenz zusammenzubauen.
- Öffnen Sie das assemblierte Plasmid, und klicken Sie auf Assemblierungsverlauf , um eine grundlegende Plasmid-Map und eine Übersicht über die Sequenzen anzuzeigen, von denen es abgeleitet wurde. Stellen Sie sicher, dass die Nukleotidpositionen für jedes Fragment mit dem gewünschten Design übereinstimmen.
- Zeigen Sie auf der Registerkarte Baugruppenparameter die Namen der Primer an, die für die Baugruppe entworfen wurden. Beachten Sie, dass für jedes Insert zwei Primer entworfen werden (ein Vorwärts- und ein Rückwärts-Primer), und die Primernamen aus den Titeln der DNA-Sequenzdateien abgeleitet werden. Stellen Sie außerdem sicher, dass die Schmelztemperaturen der Grundierung und die empfohlenen Glühtemperaturen in diesem Fenster kompatibel sind (siehe Zusatzdatei 8, Repräsentative Grundierungen).
- Wenn die Schmelztemperaturen des Primers oder die Glühtemperaturen nicht ideal sind, passen Sie die Primersequenzen manuell in den Primersequenzdateien an (z. B. Entfernen von Nukleotiden zur Reduzierung der Schmelztemperatur). Alternativ können Sie auf "Erneut öffnen" klicken und die Primer-Einstellungen anpassen, indem Sie im Montageassistenten auf die Schaltfläche "Enzym-/Primer-Einstellungen" neben der Schaltfläche "Zusammenbauen" klicken. Wiederholen Sie dann den Assemblierungsschritt und überprüfen Sie die Primersequenzen erneut.
- Wenn eine Bibliothek von Plasmiden kloniert werden soll, bei der viele verschiedene Gene in dasselbe Vektorrückgrat kloniert werden,
- Verwenden Sie jeweils das gleiche Primerpaar für den Vektor. Entfernen Sie dazu manuell alle Nukleotide von den Vektorprimern, die die für den Einsatz spezifischen Überhänge installieren würden. Diese Nukleotide befinden sich am 5'- Ende des Primers. Nach der Entnahme ist darauf zu achten, dass die Schmelztemperaturen der Grundierung und die Glühtemperaturen kompatibel sind (siehe Zusatzdatei 8, Repräsentative Grundierungen).
- Stellen Sie sicher, dass alle Einsatzprimerpaare sehr ähnliche Schmelztemperaturen und Glühtemperaturen aufweisen.
ANMERKUNG: Dies reduziert den Bedarf an einer Temperaturoptimierung für jedes Paar und ermöglicht es, alle PCR-Reaktionen der Schüler gleichzeitig mit demselben Thermocycler-Programm durchzuführen.
- Sobald das Primer-Design für die gewünschten Plasmide abgeschlossen ist, klicken Sie im Montage-Assistenten auf Abschließen. Bestellen Sie die Primer bei einem DNA-Syntheseunternehmen und entnehmen Sie Proben der DNA-Templates für Insert(s) und Vektor.
- Testen Sie sowohl insert- als auch vektorspezifische Primerpaare auf optimale Annealing-Temperaturen unter Verwendung eines Gradienten-PCR-Experiments mit den DNA-Templates. Testen Sie zunächst die von Benchling empfohlenen Glühtemperaturen ± 3 °C.
- Sobald die Glühtemperaturen bestimmt wurden, erstellen Sie Aliquots von Primerlösungen, DNA-Templates und notwendigen Reagenzien für PCR-Reaktionen für Studenten.
- Bestimmen Sie die DNA-Template-Quelle für das/die Geninsert(s), z. B. genomische DNA, Plasmid und synthetische DNA.
- Stellen Sie den Schülerinnen und Schülern vor den Experimenten Arbeitsblätter zur Verfügung, um die Vorbereitung zu unterstützen und das Verständnis zu beurteilen (Ergänzende Datei 4, Ergänzung 5 und Ergänzung 6).
- Datenerhebung zu Lernergebnissen
ANMERKUNG: Wenn Sie daran interessiert sind, Lernergebnisdaten aus dem Gibson Assembly-Modul zu sammeln und die Ergebnisse zu veröffentlichen, sollten Sie die Genehmigung für die Forschung mit menschlichen Probanden gemäß den Richtlinien der Institution einholen.- Laden Sie die Schülerinnen und Schüler ein, mindestens 1 Woche vor Beginn des unabhängigen Projekts der Gibson Assembly an einer Untersuchung der Lernergebnisse teilzunehmen.
- Lassen Sie die studentischen Teilnehmer den Multiple-Choice-Vorfragebogen (Supplemental File 6) des Labortreffens vor Beginn des unabhängigen Projekts ausfüllen.
2. Schülerexperiment A: PCR zur Gewinnung linearer Fragmente
- Beziehen Sie Primer und DNA-Template-Lösungen für die PCR vom Dozenten.
- Pipettieren Sie 25 μl Reaktionen einschließlich DNA-Polymerase MasterMix, DNA-Template, Vorwärts- und Rückwärtsprimer und nukleasefreies Wasser gemäß den Anweisungen im Studentenhandbuch (siehe Ergänzungsdatei 2).
- Durchlaufen Sie die Reaktion in einem Thermocycler gemäß dem Schülerhandbuch in der Zusatzdatei 2. Es ist sicherzustellen, dass die Glühtemperatur der Reaktion für die Primer und die Verlängerungszeit für die Länge des gewünschten Amplikons geeignet ist. Detaillierte Anweisungen finden Sie in der Zusatzdatei 2 und der Zusatzdatei 3 .
- Lagern Sie PCR-Reaktionen bei 4 °C oder -20 °C bis zum folgenden Laborzeitraum.
- Analysieren Sie 5 μl jeder Reaktion mittels Agarosegelelektrophorese. Während das Gel läuft, fügen Sie 1 μl DpnI-Restriktionsenzym zu jeder Reaktion hinzu, bei der Plasmid-DNA als Matrize verwendet wird. Inkubieren Sie diese Mischung 1 h lang bei 37 °C.
ANMERKUNG: DpnI baut alle verbleibenden zirkulären Plasmid-DNA-Matrizen ab, wodurch die Wahrscheinlichkeit verringert wird, dass im Transformationsschritt falsch positive Ergebnisse erzielt werden. - Bilde das Gel, um zu bestätigen, dass die PCR erfolgreich war und eine korrekte Amplifikation erreicht wurde. Reinigen Sie alle erfolgreichen PCR-Reaktionen mit einem kommerziell erhältlichen PCR-Aufreinigungskit. Messen Sie die Konzentration (ng/μl) des gereinigten PCR-Produkts mit einem Mikrovolumen-Spektralphotometer zur Verwendung in nachfolgenden Gibson-Assemblierungsberechnungen.
- Entwerfen Sie vor dem nächsten Klassentreffen das Reaktionsrezept für die Gibson-Baugruppe (siehe Zusatzdatei 2 und Zusatzdatei 4).
3. Schülerexperiment B: Gibson Assemblierung, Transformation und Auswahl von Klonen
- Pipettieren Sie die Gibson-Assemblierungsreaktion gemäß dem entworfenen Rezept. Inkubieren Sie die Reaktion 15 Minuten lang bei 50 °C. Während die Reaktionen inkubieren, bereiten Sie sich auf die Umwandlung vor, indem Sie chemisch kompetente Escherichia coli-Zellen auf Eis auftauen.
- Verwandeln Sie 2 μl der Gibson-Assembly durch Hitzeschock in chemisch kompetente Zellen. 100 μl der transformierten Zellen auf zwei Selektionsplatten pipettieren und mit sterilisierten Kügelchen verteilen. Bereiten Sie 10-4, 10-5 und 10-6 Verdünnungen der verbleibenden Zellen auf Luria-Bertani (LB)-Agar vor. Platte 100 μl jeder seriellen Verdünnung auf LB (keine Auswahl) und mit sterilisierten Kügelchen bestreichen.
ANMERKUNG: Dieser Schritt ermöglicht die Berechnung der Transformationseffizienz. - Inkubieren Sie die Platten über Nacht bei 37 °C. Lagern Sie die Platten bis zur nächsten Unterrichtsstunde bei 4 °C.
- Zählen Sie die Kolonien auf allen Platten und berechnen Sie die Transformationseffizienz gemäß den Anweisungen in der Zusatzdatei 2. Streichen Sie eine selektive Platte mit vier Kolonien aus einer der beiden selektiven Platten erneut und inkubieren Sie diese Platte über Nacht bei 37 °C.
ANMERKUNG: Dies dient als Backup-Kultur Ihrer positiven Klone. - Wählen Sie mit einem Marker vier verschiedene Kolonien auf der Auswahltafel aus, kreisen Sie sie ein und beschriften Sie sie mit den Initialen des Schülers und einer Zahl (z. B. ABC1). Entnehmen Sie eine neue LB-Agar-Platte, die ein Antibiotikum zur Selektion enthält, verwenden Sie einen Marker, um die Platte in Quadranten (d. h. Viertel) zu unterteilen, und verwenden Sie dann ~1/2 jeder Kolonie, um erneut auf den jeweils markierten Quadranten zu streifen, und die andere Hälfte zur Inokulation einer 5-ml-Flüssig-LB-Kultur für jede der vier ausgewählten Kolonien. Achten Sie darauf, die Röhrchen mit der jeweiligen Kolonieidentität (z. B. ABC1) zu beschriften und die richtige Konzentration des Antibiotikums für die Auswahl hinzuzufügen. Flüssigkulturen werden über Nacht in einem Schüttelbrutator bei 37 °C und die Agarplatte in einem statischen Inkubator bei 37 °C inkubiert. Flüssigkulturen und Platten bis zur folgenden Unterrichtsstunde bei 4 °C lagern.
4. Schülerexperiment C: Plasmidisolierung und Screening
- Isolieren Sie Plasmid-DNA aus den Flüssigkulturen aus Experiment B mit einem Miniprep-Kit. Messen Sie die Konzentration des isolierten Plasmids (in ng/μL).
- Entwerfen Sie einen Restriktionsverdau oder ein PCR-Screening, um die isolierten Plasmide zu analysieren (siehe detaillierte Anweisungen in Supplemental File 2 und Berichte zu den Erwartungen in Supplemental File 5).
- Pipettieren Sie die Restriktionsverdaureaktionen oder Reaktionen nach entworfenen Rezepten und inkubieren Sie bei früher festgelegten Temperaturen und Dauern.
- Analysieren Sie die Ergebnisse mittels Agarose-Gelelektrophorese.
5. Würdigung
- Lassen Sie die studentischen Teilnehmer den Multiple-Choice-Fragebogen nach Abschluss des Labor-Meetings (Ergänzende Datei 7) ausfüllen, nachdem das Gibson Assembly-Modul abgeschlossen ist.
- Kombinieren Sie alle Daten aus den Antworten vor und nach dem Fragebogen für die Analyse.
- Untersuchen Sie die statistische Signifikanz des Anstiegs der durchschnittlichen Punktzahl jeder einzelnen Inhaltsfrage zwischen den Fragebögen mit Hilfe von zweiseitigen gepaarten t-Tests und der Cohen-d-Effektgröße.
- Bewerten Sie die Steigerung des Selbstvertrauens der Schüler für jedes einzelne Semester und jede einzelne Technik, um das Ausmaß der Veränderung und ihre statistische Signifikanz zu bestimmen.
- Bestimmen Sie den Einfluss des studentischen Hintergrunds und akademischer Faktoren auf das Lernen der Studenten, indem Sie normalisierten Lernzuwachs (NLG) und ungepaarte t-Tests verwenden.
- Bewerten Sie die Einstellung der Studierenden zur Projekterfahrung und zu Karrieren in der Molekularbiologie, indem Sie die Durchschnittsnoten auswerten.
Ergebnisse
Erfolg der Schüler beim Klonen
In jeder Iteration des Gibson Assembly-Moduls (2019, 2022 und 2023) wurden die Studierenden gebeten, einen Bericht zu erstellen, in dem sie ihre Ergebnisse zusammenfassen. Im Jahr 2019 gaben 36 von 44 Studierenden (81,8%) an, dass sie ihre Plasmide erfolgreich kloniert haben, basierend auf den Ergebnissen des Screenings, das sie für Experiment C entworfen haben. Insgesamt 14 von 20 Studierenden (70,0 %) berichteten im Jahr 2022 über E...
Diskussion
Hier schlagen wir ein anpassbares Laborprojekt für Studenten vor, das den Studenten das molekulare Klonen durch Gibson Assembly in einer kursbasierten Forschungsumgebung beibringt. Insgesamt wurden 28 neuartige Plasmide von Studenten im Grundstudium in einem Klassenzimmer kloniert. Der von den einzelnen Studierenden berichtete Erfolg lag zwischen 44,4 % und 81,8 % über drei Kohorten, und der Gesamterfolg der Klonierung betrug 80 % (28 von insgesamt 35 zugewiesenen Plasmiden wurden erfo...
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen oder sonstige Interessenkonflikte haben.
Danksagungen
Die Autoren danken Andrea Laubscher für die technische Unterstützung und Michael Black, Sandi Clement und Javin Oza für hilfreiche Diskussionen über die Implementierung von Lehrlaboren und die Bewertung von Lernergebnissen. Die Autoren danken allen Studierenden, die in den Kohorten 2019, 2022 und 2023 an der Lernergebnisstudie teilgenommen haben, sowie den Forschungsstudenten Nathan Kuhn und Aayushi Adettiwar, die bei der Vorbereitung der Reagenzien für die Implementierung des Lehrlabors geholfen haben. Die Autoren danken auch für die finanzielle Unterstützung durch den William and Linda Frost Fund, den Chevron Biotechnology Applied Research Endowment Grant des Center for Applications in Biotechnology und die National Science Foundation (NSF-1708919 und NSF-2300890).
Materialien
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
Referenzen
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten