このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Research Article
適応性のある分子クローニングコースベースの学部研究経験による学生の成果の向上
* これらの著者は同等に貢献しました
要約
適応性のあるGibson Assembly分子クローニングモジュールは、分子生物学研究室の学生向けのコースベースの学部研究経験(CURE)形式で採用されました。学生の学習成果の評価では、CUREの完了後に分子クローニングに対する理解と自信が向上したことが示され、天然物生合成研究のために新規プラスミドがクローニングされました。
要約
分子生物学技術の継続的な進歩には、分子生物学のカリキュラムを定期的に改良して、学生が最新の能力で労働力に参入するための効果的な準備が必要です。特に、高度にカスタマイズ可能で適応性の高い分子クローニング技術であるGibson Assemblyの登場により、多くの研究環境における分子クローニングの状況が進歩しました。そこで、カリフォルニア・ポリテクニック州立大学サンルイスオビスポ校の分子生物学研究室に展開するためのGibson Assemblyクローニングモジュールを作成し、このモジュールによる学生の学習成果を評価しました。コースの3回の反復で、学生は、天然物生合成の研究プロジェクトを支援するために、3つのユニークなプラスミドライブラリをクローニングする実験ベースの独立したプロジェクトに参加しました。学生には、分子クローニングに関する理解度と分子生物学の用語と技術に対する自信を評価するための事前および事後アンケートが行われました。学生の回答は、分子クローニングの概念を学ぶことと、分子クローニングの用語と技術に対する自己申告の自信の両方で有意に増加したことを示しました。このモジュールフレームワークは、さまざまなアプリケーション向けのGibson Assemblyを教えるために一般化することができ、研究プロジェクトを進めながら適応性のある創発的なクローニング技術を教えるためのツールキットをインストラクターに提供します。
概要
基本的な分子生物学の概念と実験技術について学部生を訓練することは、これらの方法論が学界や産業界を含むさまざまな研究環境で一般的であるため、彼らの科学的および専門的な開発にとって非常に重要です。そのため、カリフォルニア工科大学サンルイスオビスポ校(Cal Poly)の生物学(分子細胞生物学集中)および生化学専攻の学生は、これらのトピックを学び応用するために、上位の分子生物学実験室コースを受講する必要があります(CHEM/BIO 475)。このコースの基本カリキュラムは、学生がトポイソメラーゼベース(TOPO)クローニングを行い、酵母相補的DNA(cDNA)テンプレート1から調製されたアクチン含有プラスミドを組み立てるというものです。学生は、本物の研究仮説を模倣した問題に基づいて実験を設計し、実験室での実践と探究型学習への親しみを深めます。分子生物学の分野における継続的な進歩には、対応するカリキュラムが、労働力のための現代的な能力を持つ学生を準備するために適応する必要があります。特に、Gibson Assemblyの使用は、科学界でより支配的になっています。この方法はもともと人工染色体2を合成するために確立されましたが、このレポートの時点で5,000を超える出版物がGibsonらの元の研究を参照しています。Gibson Assemblyは、従来のクローニング法と比較して独特で、高度にカスタマイズ可能で、ジャンクションを生成するための制限部位を必要とせずに、複数の直鎖状DNAフラグメントを簡単にライゲーションできます。そこで、CHEM/BIO 475のカリキュラムを刷新し、最新の分子クローニング技術を取り入れ、探究型コースモデルを改善する機会を得ました。
学生の研究経験が、科学における概念的理解、スキル開発、および持続性の向上に貢献することが確立されていますが3、すべての学部生が研究室に直接参加する機会があるわけではありません。研究室での学生の能力が限られているという課題に対処するために、コースベースの学部研究体験(CURE)が開発され、採用され、教室での本格的な研究を通じて科学のアクセシビリティが向上しています。CUREはその実施方法によって異なりますが、科学研究の5つの信条に対応する共通の実践が確立されています。適切に設計されたCUREでは、学生は1)科学的実践を使用し、2)研究プロジェクトに協力し、3)新しい発見を試みる、4)教室の外で関連する仕事に貢献し、5)実験が失敗した場合に仮説と方法を再評価および修正します4。従来の学生の研究室での研究経験と同様に、CUREは、科学、科学スキル、プロジェクトのオーナーシップ、科学、技術、工学、数学(STEM)に対する学生の自信、プロジェクトのオーナーシップ、粘り強さを強化することが示されています5。分子クローニングを含む硬化は以前に報告されていますが、6,7,8,9,10,11,12,13 本ですが、本物の研究プラスミドのライブラリーを作成するための Gibson Assembly の適応性を強調するものは認識されていません。
ここでは、カリフォルニア州立工科大学サンルイスオビスポ校で現在行われている調査ベースのCHEM/BIO 475カリキュラムの拡張について報告します。Gibson Assemblyを使用した実践的な体験と、全米科学財団(NSF-1708919およびNSF-2300890)が資金提供する研究プロジェクトにオリジナルのプラスミドコンストラクトを提供したCUREへの学生の参加という2つの主要な改善が加えられています。このカリキュラムの3回の実施を通じて、学生はActinomycetotaによって生成される生理活性分子の天然物生合成に焦点を当てた2つの異なる研究プロジェクトに貢献しました。天然物には、抗生物質、抗真菌剤、および/または抗がん作用を持つファーマコフォアが含まれていることが多く、これらの小分子は創薬活動において重要であり、臨床的関連性の可能性を示しています14。この研究では、細菌の生合成酵素の機能と工学的可能性の両方を調べるために、プラスミドライブラリーの作製が必要です。このCUREでは、学生はこれらの研究プロジェクトに関連するユニークなプラスミドライブラリをクローニングするためのGibson Assembly実験を設計および実施しました(図1)。さらに、モジュールの形式とデザインは、他の研究プロジェクトで関心のあるプラスミドを生成するために容易に適応できるという点で特徴的です。
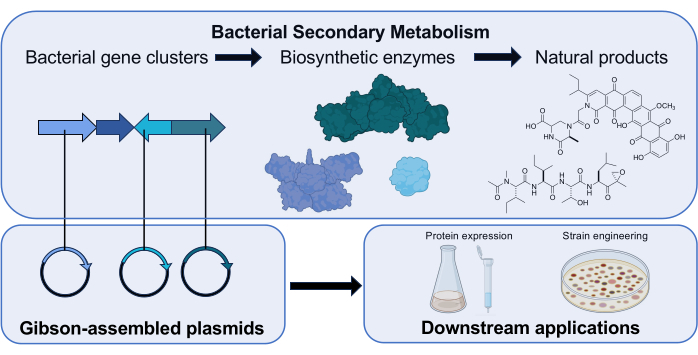
図1:当研究室におけるGibson Assemblyの役割の概要。 放線菌は、生合成酵素をコードする遺伝子クラスターを使用して、臨床的に関連する生物活性を持つ低分子天然物を生成します。私たちの研究では、生合成遺伝子を含むプラスミドをGibson Assemblyを介してアセンブルし、コードされた酵素の機能をダウンストリームで調べます。Biorender.com の科学アイコン。この図の拡大版を表示するには、ここをクリックしてください。
研究室概要
分子生物学研究室は、分子生物学と細胞生物学を集中して学ぶ生化学専攻と生物学専攻に必要な上位課程です。前提条件を満たす他の学生は、上位部門の選択科目としてコースを受講することを歓迎します。このコースは、Cal Polyの化学および生化学部門と生物学部門の間で共同リストされています。両学部の教員が各学期(生物学2学期、化学と生化学4学期)に交代で授業を行います。
クラスは、週に2回、170分、週に1回、50分の講義で研究室で開催されます。ラボセクションには最大16人の学生が含まれ、四半期ごとに2〜3のラボセクションが提供されます。コースは10週間で、最終試験は10週目の最後の会議で実施されます。講義時間は、研究室で実施されている多くの実験技術の背後にある理論や、研究室でカバーされていない分子生物学の現在のトピックについて議論するのに費やされます。ラボのコアカリキュラムには、酵母1からアクチン遺伝子をクローニングするプロセスが含まれており、これには約7週間(13〜14回のラボミーティング)かかります。マイクロピペッティング、酵母RNA単離、逆転写ポリメラーゼ連鎖反応(RT-PCR)を用いた酵母遺伝子の増幅、TOPOクローニング、ブルーホワイトスクリーニング、プラスミド単離、制限酵素消化およびPCRによるインサートの検証、クローンの in silico 解析、DNA配列解析などの技術があります。コースの最後の3週間のカリキュラムはインストラクターの裁量に委ねられていますが、学生は未定義の結果を持つ「独立したプロジェクト」を完了する必要があります。
実験の概要
私たちの研究グループでは、放線菌の生合成経路に焦点を当てています。独立したプロジェクトを設計する際には、学生がGibson Assemblyを通じてプラスミドを作成し、天然物の生合成を研究する研究プロジェクトで使用することを想定していました。ここで評価するコースモジュールの反復は、生合成経路の操作を可能にするプラスミドに特有のものでしたが、Gibson Assemblyのワークフローは、他の分子クローニングプロジェクトにも非常に適応可能です(図2)。ワークフローは3つの異なる実験(A、B、C)に分割され、2つの講義期間と6つのラボ期間(合計3週間)で完了しました( 補足ファイル1 および 補足ファイル2を参照)。実験の前には、生徒の準備をサポートし、生徒の理解度を評価するためのワークシート(補足ファイル3、 補足ファイル4、 補足ファイル5)が行われました。ワークフローは、インストラクターのニーズと関心に柔軟に対応できる形式で提供されます。

図2:Gibson Assemblyモジュールのワークフロー。 1日目と4日目は講義期間で、 学生はin silico 配列解析と実験デザインを完成させます。2〜3日目と5〜8日目は研究室のミーティングで、Gibson Assemblyを介して新規プラスミドをクローニングする手順が行われ、その後、単離とスクリーニングが行われます。絵のフローチャートは、学生が実行する3つの実験(A、B、C)によってグループ化されています。より詳細な指示とプロトコルは、それぞれ 補足ファイル1 と補足 ファイル2 として提供されるインストラクターと生徒のマニュアルに記載されています。Biorender.com の科学アイコン。この図の拡大版を表示するには、ここをクリックしてください。
Gibson Assemblyの独立プロジェクトモジュールは、2019年春四半期にCHEM/BIO 475で初めて試験運用されました。2020年と2021年には、SARS-CoV-2のパンデミックにより、コースはオンラインで教えられました。2022年と2023年の春に対面式の指導が再開されたとき、コース内の学生は、研究に関連するオリジナルのプラスミドをクローニングするGibson Assembly独立プロジェクトの学習成果を評価する研究に参加するよう招待されました。2019年、学生たちは 、Micromonospora echinospora ATCC 15837のゲノムから得られた遺伝子カセットをpKC1132にクローニングしたプラスミドのライブラリを作成しました(図3)。このプラスミドライブラリーは、天然物TLN-0522015の推定生合成遺伝子クラスターで目的の遺伝子を不活性化するために、私たちの研究室で利用されています。私たちの遺伝子不活性化研究を補完するために、2022年の学生は、推定TLN-05220遺伝子クラスターからpUC19に遺伝子の小さなライブラリをクローニングしました(Figure 3)。私たちの研究グループでは、これらのプラスミドを利用して、タンパク質の過剰発現と精製のために、pET28bを含む発現ベクターに遺伝子をサブクローニングしています。2023年のコホートの学生は、エポキソマイシン合成酵素16の生合成工学プロジェクトの進行中の研究に貢献しました。3〜4人のチームで、学生は非リボソームペプチドシンテターゼ17 モジュールの遺伝子操作ドメインをさまざまなタンパク質発現ベクターにクローニングし、私たちの研究室でこれらの酵素の過剰発現と精製を最適化しました(図3)。冗長性は、各コホートのクローニング計画に組み込まれていました。例えば、2019年のコホートには44人の学生が含まれ、クローニングのために15のプラスミドがクラスに割り当てられました。したがって、各プラスミドのクローニングを2回または3回試みました。
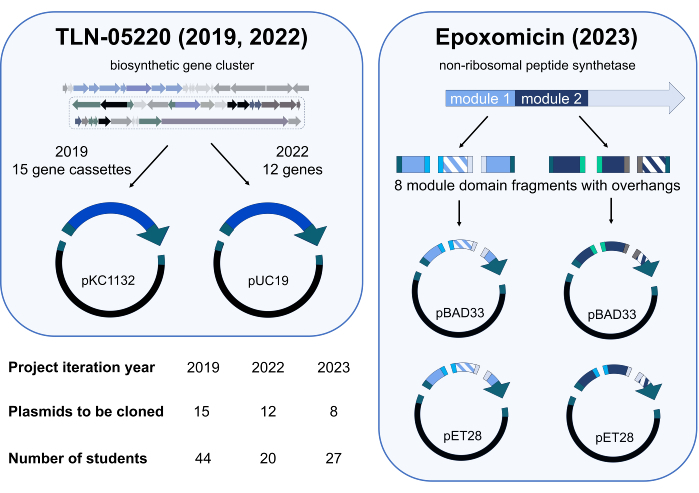
図3:2019年、2022年、2023年の独立プロジェクト反復におけるプラスミドクローニングと学生参加者のまとめ。 Gibson Assemblyプロジェクトは3回展開されています。各オファリングでは、学生参加者は、生合成経路を探索する研究プロジェクトで利用するために、プラスミドの異なるライブラリをクローニングしました。2019年と2022年のプロジェクトは、2つのフラグメント(1つの遺伝子カセットまたは遺伝子とベクター)のギブソンアセンブリ反応を用いた天然物TLN-0522015 の進行中の研究を支援しました。2023年のプロジェクトでは、エポキソマイシン生合成に関与する非リボソームペプチド合成酵素(NRPS)のモジュール1および2内のドメインスワッピングを行いました16。ハッチングされたフラグメントは、スワップされたドメインの2つの異なる変異体を表し、ソリッドカラーはスワップされなかったドメインを表します。合計で8つの異なる遺伝子フラグメント(モジュール1に4つ、モジュール2に4つ)が生成され、ギブソンアセンブリに適合するオーバーハングがありました。各モジュールについて、3つの遺伝子フラグメントの2つの異なる組み合わせを、2つの異なるベクター(pBAD33およびpET28、アセンブリあたり合計4つのフラグメント)のいずれかとアセンブルし、8つの遺伝子改変NRPSプラスミドを生成する可能性を秘めました。Biorender.com の科学アイコン。この図の拡大版を表示するには、ここをクリックしてください。
評価の概要
Gibson Assemblyの独立プロジェクト開始の少なくとも1週間前に、2022年と2023年のコホートの学生は、Cal Poly18の生化学ラボコースの調査で実施された研究と同様に設計された学習成果調査に参加するよう招待されました。学生の参加者は、独立したプロジェクトの開始前にラボミーティングで多肢選択式の事前アンケートを完了し、最終ラボミーティング中(つまり、Gibson Assemblyモジュールを完了した後)に多肢選択式の事後アンケートを完了しました。事前アンケートと事後アンケートは、28の同一の質問で構成され、事後アンケートには2つの追加質問(合計30)が含まれていました。分子クローニングに関与する酵素とメカニズムに関する学生の知識を評価するために、10のコンテンツ問題が作成されました(例:ポリメラーゼ連鎖反応[PCR]、ギブソンアセンブリ、形質転換、青白スクリーニング)。次の7つの質問では、分子クローニング用語(DNAポリメラーゼ、エキソヌクレアーゼ、リガーゼなど)への精通度を自己評価するよう学生に求めました。次の10問では、分子クローニング技術(DNA配列解析、制限消化反応、アガロースゲル電気泳動など)を行う能力を自己評価することができました。また、学生は、分子クローニング技術に関する知識に基づいて、分子生物学のキャリアを追求することに抵抗がないかどうかも報告しました。事後アンケートには、学生がコースでの学習への取り組みと、コースが貴重な学習経験であったかどうかを自己評価するための2つの追加の質問が含まれていました(補足ファイル6 および 補足ファイル7)。2022年と2023年の学生のアンケート前後の回答から得られたすべてのデータは、分析のために組み合わせられ、 補足表S1で入手できます。
プロトコル
ヒトの参加者を対象とした研究は、Cal Poly の Research Institutional Review Board (2022-113-CP (IRB)) でヒト被験者によってレビューされ、承認されました。参加者は、この研究に参加するための書面によるインフォームド コンセントを提供しました。
以下のプロトコルは、インストラクターの準備(ステップ1.1–1.3)、線状フラグメントを取得するためのPCRを含む3つの実験教育モジュールに対する学生の行動 (ステップ2.1–2.7)、ギブソンアセンブリ、形質転換、クローンの選択(ステップ3.1–3.5)、プラスミドの単離とスクリーニング(ステップ4.1–4.4)、および学習成果の評価(5.1–5.2). インストラクターによる調製では、プライマー設計の代表的な例と、in silicoでの目的とするプラスミドマップの調製について説明しています。プロトコルのすべてのセクションは、他の目的のプラスミドに適応可能です。各学生の実験は、2つの3時間のラボミーティングに分かれています。
1. 講師の準備
- Benchling Assembly Wizard を使用した Gibson Assembly プライマー設計
- ゲノムDNA、プラスミド、合成DNAなど、遺伝子挿入物のDNAテンプレートソースを決定します。
手記: 複雑なゲノムDNAテンプレートから増幅される遺伝子については、まず、National Center for Biotechnology Information(NCBI)のBasic Local Alignment Search Tool(BLAST)21を使用して、目的の遺伝子に特異的なゲノムDNAテンプレート内のプライマーのユニークな結合部位を決定することが推奨される(補足 ファイル1に詳述されている)。プラスミドや合成DNAなどの単純なテンプレートの場合は、次のステップに進みます。 - 遺伝子インサートとベクターのDNA配列を取得します(例えば、 Supplemental File 8のGenBankファイルとして提供されているポリケチドヒドロキシラーゼ遺伝子配列とpUC19配列を参照)。目的のインサート配列と目的のベクター配列を、それぞれ新しいDNA配列ファイルとしてBenchlingにインポートします。Gibson Assembly反応に含める各インポートされたシーケンスを開きます。
手記: GenBankやFASTAなど、いくつかの形式のDNA配列ファイルをインポートできます。また、アクセッション番号を使用してデータベースから直接配列をインポートしたり、別のファイルやビューアからコピーしたヌクレオチド配列に貼り付けたりすることもできます。 - 画面の下部で、 アセンブリ ウィザード ツールを見つけます。 「アセンブリ・ウィザード」をクリックし、「 新規アセンブリの作成」を選択します。表示されたオプションから「 Gibson 」を選択し、「 開始 」をクリックしてアセンブリを開始します。
- ベクトルシーケンスウィンドウで、アセンブリに含めるすべての塩基をベクトルバックボーンから選択します。遺伝子インサートの3'末端(例えば、pUC19の位置657)のヌクレオチド位置から選択を開始し、含めるべき全ての残りのヌクレオチドを選択する(例えば、位置656までで、pUC19配列全体を含む)。選択したら、画面下部の [Backbone] タブをクリックして [Set Fragment] をクリックします。
手記: ベクターの Sequence Map ビューと Plasmid ビューを切り替えて、環状テンプレートの必要な塩基をすべて選択します。あるいは、Gibson Assemblyのために(PCRではなく)制限酵素消化物を介してベクターフラグメントを調製する予定の場合は、制限酵素切断部位(XbaIなど)でバックボーン配列を直鎖化します。これを行うには、 切り取りサイトをクリックし、 Shift キーを押しながら 切り取りサイト を再度クリックします。次に、「 フラグメントを設定 」をクリックしてバックボーンを設定します。 Assembly Wizardでバックボーンが制限酵素で直鎖化されている場合、ベクター特異的プライマーはBenchlingによって生成されないことに注意してください。 - 挿入シーケンスウィンドウで、アセンブリに含めるすべての挿入のベースを選択します。選択したら、画面下部の[挿入]タブをクリックします|フラグメントを設定します。
- 複数の遺伝子を挿入する場合は、Assembly Wizardの右側にある+ボタンをクリックします。挿入シーケンスウィンドウで、アセンブリに含めるすべての挿入ベースを選択します。選択したら、画面下部の[挿入]タブをクリックします|フラグメントを設定します。
- すべてのフラグメントを設定したら、アセンブリの名前を目的のプラスミド名に変更し、Assembly Wizardの右側にあるAssembleをクリックします。Sequence Folder と Primer Folder の両方に目的のフォルダーの場所を選択します。Createをクリックして、組換えプラスミド配列をアセンブルします。
- アセンブルされたプラスミドを開き、[ Assembly History ]をクリックして、基本的なプラスミドマップと、その派生元となった配列の概要を表示します。各フラグメントのヌクレオチド位置が目的のデザインと一致していることを確認してください。
- Assembly Parametersタブで、アセンブリ用に設計されたプライマーの名前を表示します。各インサート(フォワードとリバース)に対して2つのプライマーが設計され、プライマー名はDNA配列ファイルのタイトルから導出されることに注意してください。また、プライマーの融解温度と推奨アニーリング温度がこのウィンドウで互換性があることも確認してください(補足ファイル8、代表的なプライマーを参照)。
- プライマーの融解温度またはアニーリング温度が理想的でない場合は、プライマー配列ファイル内でプライマー配列を手動で調整します(例:ヌクレオチドを除去して融解温度を下げる)。または、Re-openをクリックし、Assembly WizardのAssembleボタンの隣にあるEnzyme/Primer Settingsボタンをクリックして、プライマー設定を調整します。次に、アセンブリステップを繰り返し、プライマー配列を再検討します。
- プラスミドのライブラリーを、同じベクターバックボーンにクローニングされた多数の異なる遺伝子でクローニングする場合、
- いずれの場合も、ベクターに同じプライマーペアを使用します。これを行うには、インサートに特異的なオーバーハングを取り付けるベクタープライマーからすべてのヌクレオチドを手動で除去します。これらのヌクレオチドは、プライマーの5' 末端に見られます。除去後、プライマーの融解温度とアニーリング温度が適合していることを確認してください( 補足ファイル8、代表的なプライマーを参照)。
- すべてのインサートプライマーペアの融解温度とアニーリング温度が非常に類似していることを確認してください。
手記: これにより、すべてのペアのアニーリング温度最適化の必要性が減り、同じサーモサイクラープログラムを使用してすべての学生PCR反応を同時にサイクルできるようになります。
- 目的のプラスミドのプライマー設計が完了したら、Assembly WizardでFinalizeをクリックします。DNA合成会社にプライマーを注文し、インサートとベクター用のDNAテンプレートのサンプルを取り出します。
- DNAテンプレートを用いたグラジエントPCR実験を用いて、インサート特異的プライマーとベクター特異的プライマーの両方のペアを最適なアニーリング温度で試験します。Benchlingが推奨するアニーリング温度を最初に3 °C±テストします。
- アニーリング温度が決定したら、プライマー溶液、DNAテンプレート、および学生用のPCR反応に必要な試薬のアリコートを作成します。
- ゲノムDNA、プラスミド、合成DNAなど、遺伝子挿入物のDNAテンプレートソースを決定します。
- 実験の前に、準備をサポートし、理解度を評価するためのワークシートを生徒に提出します(補足ファイル4、補足5、 補足6)。
- 学習成果に関するデータ収集
手記: Gibson Assemblyモジュールから学習成果データを収集し、その結果を公開することに関心がある場合は、機関のポリシーに従って、人間を対象とする研究の承認を得てください。- ギブソンアセンブリの独立プロジェクトの開始の少なくとも1週間前に、学習成果の調査に参加するように学生を招待します。
- 学生参加者に、自主プロジェクト開始前に研究室会議で多肢選択式事前アンケート(補足ファイル6)に回答してもらいます。
2. 学生実験A:線状フラグメントを得るためのPCR
- PCR用のプライマーとテンプレートDNA溶液をインストラクターから入手してください。
- 学生用マニュアル( 補足ファイル2を参照)の指示に従って、DNAポリメラーゼMasterMix、DNAテンプレート、フォワードおよびリバースプライマー、およびヌクレアーゼフリー水を含む25 μL反応をピペットで使用します。
- サーモサイクラーで反応をサイクル化し、 補足ファイル2の学生マニュアルに従って行います。反応のアニーリング温度がプライマーに適切であること、および伸長時間が目的のアンプリコンの長さに対して適切であることを確認してください。詳細な手順については、 補足ファイル2 および 補足ファイル3 を参照してください。
- PCR反応液は、次のラボ期間まで4 °Cまたは-20 °Cで保存してください。
- 各反応の5 μLをアガロースゲル電気泳動で分析します。ゲルが泳動している間に、プラスミドDNAをテンプレートとして利用した各反応に1 μLのDpnI制限酵素を加えます。この混合物を37 °Cで1時間インキュベートします。
手記: DpnIは、残っている環状プラスミドDNAテンプレートを分解するため、形質転換ステップで偽陽性を得る可能性が低くなります。 - ゲルを画像化して、PCRが成功し、正しい増幅が達成されたことを確認します。成功したPCR反応は、市販のPCR精製キットで精製します。その後のギブソンアセンブリ計算で使用するために、微量分光光度計を使用して精製PCR産物の濃度(ng/μL)を測定します。
- 次回のクラスミーティングの前に、Gibson Assemblyの反応レシピを設計してください( 補足ファイル2 および 補足ファイル4を参照)。
3.学生実験B:ギブソン クローンの組み立て、形質転換、選択
- 設計されたレシピに従ってギブソンアセンブリの反応をピペットで掴みます。反応液を50 °Cで15分間インキュベートします。反応がインキュベートしている間に、化学的にコンピテントな 大腸菌 細胞を氷上で解凍することにより、形質転換の準備をします。
- 2 μLのGibsonアセンブリをヒートショックにより化学的にコンピテントな細胞に形質転換します。形質転換細胞100μLを2つの選択プレートにピペットで移動させ、滅菌したビーズで広げます。残りの細胞の10-4、10-5、および10-6希釈液をLuria-Bertani(LB)寒天上に調製します。各段階希釈液100μLをLBでプレートし(選択なし)、滅菌したビーズで広げます。
手記: このステップにより、変換効率を計算できます。 - プレートを37 °Cで一晩インキュベートし、次のクラス期間までプレートを4 °Cで保存します。
- すべてのプレート上のコロニーをカウントし、 補足ファイル2の指示に従って形質転換効率を計算します。いずれかの選択プレートから4つのコロニーを含む1つの選択プレートに再ストリークし、このプレートを37 °Cで一晩インキュベートします。
手記: これは、ポジティブクローンのバックアップカルチャーとして機能します。 - マーカーを使用して、選択プレート上の4つの異なるコロニーを選択し、丸で囲み、学生のイニシャルと番号(ABC1など)でラベルを付けます。抗生物質を含む新しいLB寒天プレートを取り出して選択し、マーカーを使用してプレートを象限(つまり、4分の1)に分割し、次に各コロニーの~1/2を使用して、それぞれ標識された象限に再ストリークし、残りの1/2を使用して、選択した4つのコロニーのそれぞれに5mLの液体LB培養物を接種します。チューブにはそれぞれのコロニー同一性(ABC1など)をラベル付けし、選択のために正しい濃度の抗生物質を追加してください。液体培養物を振とうインキュベーターで37 °Cで一晩インキュベートし、寒天プレートを37 °Cの静的インキュベーターでインキュベートします。液体培養物とプレートは、次のクラス期間まで4 °Cで保存してください。
4. 学生実験C:プラスミドの単離とスクリーニング
- ミニプレップキットを使用して、実験Bの液体培養物からプラスミドDNAを単離します。単離されたプラスミドの濃度を測定します(単位:ng/μL)。
- 単離されたプラスミドを解析するための制限消化物または PCR スクリーニングを設計します( 補足ファイル 2 の詳細な手順と 補足ファイル 5 の期待値を報告します)。
- 制限酵素消化反応または設計されたレシピに従って反応をピペットでピペットで移動し、以前に設計された温度と持続時間でインキュベートします。
- アガロースゲル電気泳動による結果の解析。
5. 評価
- Gibson Assemblyモジュールが完了した後、学生の参加者に多肢選択式の事後アンケート(補足ファイル7)をラボミーティングに記入させます。
- アンケート前後の回答から得られたすべてのデータを組み合わせて分析します。
- アンケート間の各個々のコンテンツ質問の平均スコアの増加の統計的有意性を、両側対応のt検定とCohenのd効果サイズを使用して調べます。
- 個々の学期と手法ごとに生徒の自信の増加を評価して、変化の程度とその統計的有意性を判断します。
- 正規化学習ゲイン (NLG) と対応のない t 検定を使用して、学生のバックグラウンドと学問的要因が学生の学習に与える影響を判断します。
- 平均スコアを評価することにより、プロジェクトの経験と分子生物学のキャリアに対する学生の態度 を評価します。
結果
クローン作成における学生の成功
Gibson Assemblyモジュールの各反復(2019年、2022年、2023年)で、学生は調査結果をまとめたレポートを作成するよう求められました。2019年には、44人中36人(81.8%)が、実験Cで設計したスクリーニングの結果に基づいてプラスミドのクローニングに成功したと報告しています。2022年には、20人の学生のうち合計14人(70.0%)が割り当...
ディスカッション
ここでは、コースベースの研究環境でGibson Assemblyを通じて学生に分子クローニングを教える調整可能な学部教室の実験室プロジェクトを提案します。合計で28種類の新規プラスミドが、学部生によって教室でクローニングされました。個々の学生が報告した成功率は、3つのコホートで44.4%から81.8%の範囲であり、全体のクローニング成功率は80%でした(割り当てられた?...
開示事項
著者は、競合する金銭的利益またはその他の利益相反がないことを宣言します。
謝辞
著者は、技術サポートを提供してくれたAndrea Laubscher氏、およびラボの実施と学習成果の評価に関する有益な議論を提供してくれたMichael Black氏、Sandi Clement氏、Javin Oza氏に感謝します。著者は、2019年、2022年、2023年のコホートで学習成果研究に参加したすべての学生、およびティーチングラボの実装のための試薬の準備を支援した研究生のNathan KuhnとAayushi Adettiwarに感謝します。著者はまた、ウィリアムとリンダフロスト基金、バイオテクノロジーのアプリケーションセンター、シェブロンバイオテクノロジー応用研究寄付助成金、および国立科学財団(NSF-1708919およびNSF-2300890)からの資金援助を認めています。
資料
| Name | Company | Catalog Number | Comments |
| Deoxyribonucleotide triphosphate (dNTPs, 10 mM) | Fisher Scientific | FERR0191 | Homemade' MasterMix component |
| Dithiothreitol (DTT) | Fisher Scientific | FERR0861 | Homemade' MasterMix component |
| DpnI | New England Biolabs | R0176S | 1000 units |
| Fisherbrand Isotemp Microbiological Incubator | Fisher Scientific | 15-103-0513 | |
| FisherBrand Isotemp Water Bath | Fisher Scientific | S28124 | |
| GelRed Nucleic Acid Gel Stain | Biotium | NC9594719 | 10,000X |
| GeneJET Gel Extraction and DNA Cleanup Micro Kit | Thermo Scientific | FERK0831 | 100 Preps |
| GeneRuler 1 kb DNA ladder | Fisher Scientific | FERSM0314 | 100 applications |
| LB Broth, Miller | Fisher BioReagents | BP9723-500 | 500 g |
| Magnesium chloride hexahydrate | Fisher Scientific | BP214-500 | Homemade' MasterMix component |
| Mastercycler nexus X2 Gradient Thermocycler | Eppendorf | 6337000027 | |
| Microfuge 16 Centrifuge | Beckman Coulter | A46474 | |
| Micromonospora echinospora bacteria | American Type Culture Collection | ATCC 15837 | |
| Microwave Oven | General Electric | 2440640 | |
| Molecular Biology Grade Agarose | Fisher BioReagents | BP160-100 | 100 g |
| Nanodrop One Microvolume Spectrophotometer | Thermo Scientific | 13-400-518 | |
| NEB 5-alpha Competent E. coli | New England Biolabs | C2987H | 20 x 0.05 mL |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621S | 10 reactions |
| New Brunswick Innova 40 Benchtop Orbital Shaker | New Brunswick | M1299-0090 | |
| Nuclease Free Water | Fisher BioReagents | BP248450 | 50 mL |
| PEG-8000 | Fisher Scientific | BP233-100 | Homemade' MasterMix component |
| Phusion DNA Polymerase | New England Biolabs | M0530 | Homemade' MasterMix component |
| Portable Balance | Ohaus | SKX123 | |
| pUC19 vector | New England Biolabs | N3041S | |
| Q5 High-Fidelity 2x Master Mix | New England Biolabs | M0492S | 100 reactions |
| T5 Exonuclease | Epicentre | T5E4111K | Homemade' MasterMix component |
| Taq DNA Ligase | New England Biolabs | M0208 | Homemade' MasterMix component |
| Tris-HCl | Fisher Scientific | AAA1137918 | Homemade' MasterMix component |
| TriTrack DNA Gel Loading Dye (6x) | Thermo Scientific | FERR1161 | 5 x 1 mL |
| Zyppy Plasmid Miniprep Kit | Zymo Research | D4019 | 100 Preps |
| β-Nicotinamide adenine dinucleotide (NAD+) | New England Biolabs | B9007S | Homemade' MasterMix component |
参考文献
- Black, M. W., Tuan, A., Jonasson, E. Cloning yeast actin cDNA leads to an investigative approach for the molecular biology laboratory. Biochem Mol Biol Educ. 36 (3), 217-224 (2008).
- Gibson, D. G., Young, L., Chuang, R. -. Y., Venter, J. C., Hutchison, C. A., Smith, H. O. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Bell, J. K., et al. CUREs in biochemistry—where we are and where we should go. Biochem Mol Biol Educ. 45 (1), 7-12 (2017).
- Auchincloss, L. C., et al. Assessment of course-based undergraduate research experiences: A meeting report. CBE Life Sci Educ. 13 (1), 29-40 (2014).
- Buchanan, A. J., Fisher, G. R. Current status and implementation of science practices in Course-based Undergraduate Research Experiences (CUREs): A systematic literature review. CBE Life Sci Educ. 21 (4), ar83 (2022).
- Verity, N., Ulm, B., Pham, K., Evangelista, B., Borgon, R. Demonstrating core molecular biology principles using GST-GFP in a semester-long laboratory course. Biochem Mol Biol Educ. 50 (1), 55-64 (2022).
- Li, G., et al. CUR(E)ating a new approach to study fungal effectors and enhance undergraduate education through authentic research. Biochem Mol Biol Educ. 52 (1), 6-14 (2024).
- Roecklein-Canfield, J. A., Lopilato, J. Tagging and purifying proteins to teach molecular biology and advanced biochemistry. Biochem Mol Biol Educ. 32 (6), 373-377 (2004).
- Li, C., et al. Directed evolution of glyphosate oxidase and a chemiluminescence system for glyphosate detection: A comprehensive practical laboratory experiment on biotechnology. Biochem Mol Biol Educ. 51 (3), 302-311 (2023).
- Wang, J. T. H., Schembri, M. A., Ramakrishna, M., Sagulenko, E., Fuerst, J. A. Immersing undergraduate students in the research experience: A practical laboratory module on molecular cloning of microbial genes. Biochem Mol Biol Educ. 40 (1), 37-45 (2012).
- Dean, D. M., Wilder, J. A. The “Frankenplasmid” lab: An investigative exercise for teaching recombinant DNA methods. Biochem Mol Biol Educ. 39 (5), 376-383 (2011).
- Bornhorst, J. A., Deibel, M. A., Mulnix, A. B. Gene amplification by PCR and subcloning into a GFP-fusion plasmid expression vector as a molecular biology laboratory course. Biochem Mol Biol Educ. 32 (3), 173-182 (2004).
- Roberts, L. A., Shell, S. S. A research program-linked, course-based undergraduate research experience that allows undergraduates to participate in current research on mycobacterial gene regulation. Front Microbiol. 13, 1025250 (2023).
- Jose, P. A., Maharshi, A., Jha, B. Actinobacteria in natural products research: Progress and prospects. Microbiol Res. 246, 126708 (2021).
- Banskota, A. H., et al. TLN-05220, TLN-05223, new Echinosporamicin-type antibiotics, and proposed revision of the structure of bravomicins. J Antibiot. 62 (10), 565-570 (2009).
- Schorn, M., et al. Genetic basis for the biosynthesis of the pharmaceutically important class of epoxyketone proteasome inhibitors. ACS Chem Biol. 9 (1), 301-309 (2014).
- Smith, H. G., Beech, M. J., Lewandowski, J. R., Challis, G. L., Jenner, M. Docking domain-mediated subunit interactions in natural product megasynth(et)ases. J Ind Microbiol Biotechnol. 48 (3-4), kuab018 (2021).
- Williams, L. C., et al. The genetic code kit: An open-source cell-free platform for biochemical and biotechnology education. Front Bioeng Biotechnol. 8, 941 (2020).
- Bloodhart, B., Balgopal, M. M., Casper, A. M. A., Sample McMeeking, L. B., Fischer, E. V. Outperforming yet undervalued: Undergraduate women in STEM. PLoS One. 15 (6), e0234685 (2020).
- Farrar, V. S., Aguayo, B. -. Y. C., Caporale, N. Gendered performance gaps in an upper-division biology course: Academic, demographic, environmental, and affective factors. CBE Life Sci Educ. 22 (4), ar55 (2023).
- Camacho, C., et al. BLAST+: Architecture and applications. BMC Bioinf. 10 (1), 421 (2009).
- . Optimization Tips for NEBuilder® HiFi DNA Assembly and NEB® Gibson Assembly Available from: https://www.neb.com/en-us/tools-and-resources/usage-guidelines/optimization-tips-for-nebuilder-hifi-dna-assembly-and-neb-gibson-assembly (2024)
- . Instruction Manual: Gibson Assembly® Master Mix / Gibson Assembly® Cloning Kit Available from: https://www.neb.com/-/media/nebus/files/manuals/manuale2611.pdf?rev=9db62577a41b4cfda071e21864a6763e (2021)
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved