需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用于定量体外成体 果蝇 结构的快速、简单的工作流程
摘要
在这里,我们提出了一种快速、低成本的工作流程,用于对成年 果蝇 眼睛进行高分辨率成像,以量化模式和生长缺陷。我们描述了通过点安装、高分辨率图像采集和图像分析进行样品制备的方案。
摘要
果蝇复眼是一种精确图案的组织,它揭示了驱动形态发生的分子机制和生物过程。它是一种重复单位眼的简单结构,称为 ommatidia,用于表征遗传相互作用和基因功能。影响眼睛结构的突变可以很容易地检测和分析;因此,该系统经常用于资源贫乏的机构。进一步的表型分析通常包括扫描电子显微镜 (SEM) 以生成适合定量分析的高放大倍率图像。然而,SEM 价格昂贵且需要昂贵的试剂;样品制备需要数天时间;而且,他们通常需要全职员工进行样品制备和仪器维护。这限制了它们在资源不足的机构或预算紧缩期间的效用。在昆虫学中,使用高分辨率数字成像技术是识别和表征物种的常见做法。本文描述了一种结合策略的方法,该方法允许使用开放式软件 ImageJ 对成年果蝇结构进行高分辨率数字成像和定量分析。该工作流程是一种快速且对学生友好的替代方案,通过一种经济高效且快速的定量表型分析方法,弥补了资金不足和资源不足的研究机构的局限性。
引言
黑腹果蝇是一种强大的遗传模式生物,几十年来一直被用于阐明分子信号通路和细胞行为。许多对多细胞发育至关重要的进化上保守的信号通路首先在果蝇中被发现,并确定了它们的作用机制。大约 65-75% 的人类疾病相关基因在果蝇中具有直系同源物 1,2。果蝇成虫眼是一种重要的模型,它允许进行无偏倚的遗传筛选,这有助于发现与人类疾病有关的重要保守基因,包括癌症 3,4、神经退行性变5 和代谢紊乱6。
果蝇的眼睛由 ~800 只单位眼组成,称为 ommatidia,它们在成人眼睛的表面精确地排列成六边形图案7。每个 ommatidium 由八个感光神经元组成,它们在不对称梯形中占据不同的位置。这些由四个非神经视锥细胞和两个初级色素细胞支持,它们分泌晶状体和伪视锥细胞,将光线聚焦到感光神经元的感光横纹肌上。相邻的 ommatidia 由单排间晶格细胞隔开,该晶格细胞由次生色素细胞、三级色素细胞和机械感应刚毛复合物组成 8,9,10。
眼睛发育的扰动在成人眼中可见,表现为眼睛大小增加或减少、晶状体或刷毛的异常丰度或结构,或者表现为"粗糙的眼睛",通常不变的六边形图案被破坏,以至于无法再在眼睛表面跟踪一排 ommatidia。这些表型可以使用解剖显微镜在大体组织水平上进行评分。表型的详细分析传统上包括扫描电子显微镜检查,然后进行定量图像分析11。然而,扫描电子显微镜需要昂贵的仪器、昂贵的试剂、需要数天的样品制备,并且通常需要全职人员来维护。
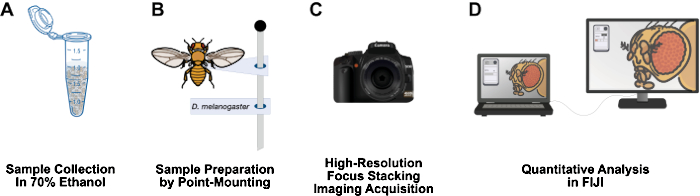
图 1:成体 果蝇 结构成像的工作流程。 (A) 在 70% 乙醇中收集和固定成体 果蝇 。(B) 通过点安装和固定在引脚上来制备用于成像的样品。(C) 通过焦点堆叠和集成获取高分辨率图像。(D) 使用 FIJI 量化图像。 请单击此处查看此图的较大版本。
本文介绍了一种相对便宜、样品制备时间短、可以在 3 英尺长的实验室工作台上轻松设置、不需要有害材料、并且可以作为 果蝇 研究实验室的长期补充(图 1)。点安装是一种昆虫学技术,用于风干和保存小型软体昆虫,例如 果蝇12。该方法依赖于将显微镜物镜与高分辨率 DSLR 相机相结合,以实现 10 倍至 1,000 倍的有效放大倍率。微距摄影固有的有限景深通过焦点堆叠来克服:将一系列图像拼接在一起,焦平面穿过感兴趣的标本13。该方法产生适用于表型定量的高分辨率图像,并且可以轻松适应其他感兴趣的结构,例如翅膀、腿部、胸部和腹部。图像分析工作流程使用免费的图像分析程序 FIJI (NIH ImageJ)。这种方法使资源贫乏机构的本科生和科学家能够进行样品制备、高分辨率成像和分析。
研究方案
1. 成体 果蝇 收集和固定
- 设置 果蝇 杂交或选择菌株,并将它们放入装有果蝇食物的小瓶中。在所需温度(通常为 25 °C)下孵育样品瓶,直到果蝇发育成熟且成虫闭合(在 25 °C 下 ~10-14 天)。
- 用 CO2 麻醉果蝇,并将它们放在 CO2 垫上。
- 用羽毛对果蝇进行分类,并选择具有所需表型 (例如,直翅) 的个体。通过修剪鹅毛以适合 1 mL 血清移液管的锥形端来制作羽毛蝇分选仪。
- 制备含 1 mL 70% 乙醇的 1.7 mL 微量离心管。将选定的果蝇放入微量离心管中并置于冰上。将微量离心管在 4 °C 下储存过夜(图 2A)。
注意:不要将果蝇在乙醇中保存超过 24 小时。苍蝇长期储存在 70% 乙醇中会导致眼睛和身体色素的损失。
2. 通过点安装进行样品制备
注意: 果蝇 是软体昆虫,风干后会变脆并塌陷;因此,该协议要求样品在安装当天进行成像。一次以 ~5 只苍蝇为一组进行工作,以防止样品损失。根据效率增加组中的样本数量。成像前需要更多时间的标本可以通过增加浓度的六甲基二硅氮烷 (HMDS) 系列进行脱水14。
- 使用专用的尖头打孔器从存档的 65 磅卡片纸上切割小三角形点 (7.1 毫米 x 2.7 毫米)。通过使用 Dumont #5 细尖镊子将尖端(最窄的 25%)弯曲成 90° 角来准备点(图 2B)。
- 使用 Dumont #5 细尖镊子,从微量离心管中取出果蝇(步骤 1.4)。用无绒实验室组织轻轻吸干果蝇,以去除多余的乙醇。在解剖显微镜下,将每只果蝇放在索引卡的左侧。
注意:通过抓住不是感兴趣区域的解剖结构从管中去除苍蝇 - 当对头部进行成像时,我们用翅膀或腿握住样品。不要握住样品的腹部,因为该结构用于将果蝇粘在卡尖上。 - 准备皮革胶,将其稠度调整到所需的粘度。将 1-2 滴皮革胶与 1-2 滴去离子 (DI) 水混合,然后用移液管在索引卡上混合。用镊子在宽端拿起准备好的卡点,然后将少量稀释的胶水浸入胶水混合物中,将其滴入弯曲的尖端(图 2C)。
注意:胶水应该是可涂抹的,但不能流动。 - 将尖端弯曲的尖端应用于右腹部前侧的腹部节段 2-3 周围(图 2C)。在胶水干燥之前,对果蝇进行轻微调整,使果蝇的前后轴垂直于尖端的弯曲尖端。
- 将 3 号安装销插入卡点的宽端(图 2D)并将其固定到昆虫固定块(图 2E)。用相应的基因型标记每个针或针行。

图 2:样品制备。 (A) 根据表型标记物对成年 果蝇 进行分类,并收集到含有 70% 乙醇的冰上标记微量离心管中。将苍蝇在 4°C 下储存过夜。(B) 通过使用一对 #5 镊子将窄端与卡片的其余部分弯曲 90° 来制备纸卡点。(C) 从试管中回收苍蝇,并短暂风干。将皮革胶涂在准备好的卡尖的小折叠端,并在腹部 2-3 节粘在成年果蝇上。(D) 将标本用识别标签固定在 #3 不锈钢昆虫针上。(E) 固定的标本存放在样品板上,直到准备好进行图像采集。 请单击此处查看此图的较大版本。
3. 高分辨率焦点堆叠成像采集
- 使用组装和定制的聚焦堆叠成像系统获取蝇眼的高分辨率照片。
- 使用带有 70-200 毫米长焦镜头的 DSLR 相机机身拍摄照片,该镜头通过 77 毫米镜头适配器连接到 20 倍 Apo 显微镜物镜。
- 确保通过扩散器用闪光灯照亮标本(图 3)。
- 使用 Stackshot 控制器和宏边栏控制 Z 轴定位。
- 将相机、闪光灯和电动载物台连接到重型阳极氧化铝三脚架上。
- 将每个点安装的样品放在通用载物台万向节上,使头部朝向,使眼睛面向镜头。用镊子轻轻移动头部,调整头部位置。
注意:大而快速的调整可能会导致意外斩首。 - 将相机联机到笔记本电脑上,在软件中调整 采集设置 。使用以下设置以 20 倍 放大倍率拍摄标本: 闪光功率 1/32、快门速度 1/200、光圈 F2.8 和 ISO 400。确保所有自动对焦和图像稳定功能均已关闭。
注意:这些设置可平衡最佳闪光亮度、快门速度和景深。它们需要针对其他放大倍率和/或镜头组合进行调整。 - 设置将生成的图像堆栈(10-50 张图像)保存到所需文件夹的位置。确保有足够的图像存储容量(每个图像 ~8.5 MB)。
- 在 Auto Distance 模式下调整 Stackshot 控制单元上的对焦堆栈设置。
- 将步长设置为 5 μm,并通过设置焦点堆栈的开始和停止位置来计算步数。
- 在 LiveView 模式下 查看标本,并在 相机处于自动拍摄模式 时查看标本,以确定开始和停止位置。
- 移动导轨,使标本最近的部分聚焦(设置 起始 位置),然后移动到最感兴趣的特征聚焦的位置(设置 结束 位置)。
- 将相机返回到 手动拍摄 模式,然后从 Stackshot 控制单元开始图像采集。
注意:图像采集时间取决于标本的大小。大型标本所需的景深越大,图像堆栈中包含的切片就越多,这将延长整体采集时间。 - 在引用的焦点堆叠软件中打开文件。通过单击堆叠 |对齐和堆叠所有 (PMax)。
- 通过单击 文件 |.tif保存输出图像。
注意:根据堆叠图像文件的分辨率和成像的标本数量,可能需要大型外部硬盘驱动器 (1 TB) 进行图像备份。在此协议中,堆叠图像在压缩之前每个图像大约为 100 MB。

图 3:图像采集。 (A) 部件标记如下的成像设备:a) DSLR 相机机身;b) 长焦镜头;c) 20 倍 Apo 显微镜物镜和适配器;d) 闪光;e) 透镜和圆顶扩散器;f) Stackshot 控制器、宏导轨和旋转平台;g) 通用载物台万向节;h) 三脚架。(B) 带有光扩散器的成像设备。(C) 已镶嵌样品在成像位置上的特写。 请单击此处查看此图的较大版本。
4. 计算成人眼部面积的 FIJI 分析工作流程
- 对于图像分析,请从 ImageJ.net 网站获取 FIJI 软件15 。
- 选择眼睛居中并对齐、光线充足且周边模糊最小、表明与相机正确对齐的图像进行分析。
- 校准图像比例尺。
- 下载与 500 μm 相关的 20 倍放大倍率的比例尺图像。或者,在获取图像时,使用相同的设置拍摄标尺。在 FIJI 软件中打开图像。
- 测量比例尺的长度(图 4A)。使用 直线工具 精确描摹线条。单击 Analyze |测量。该像素距离相当于 500 μm (图 4B)。
- 计算每微米的像素数。使用此选项可将像素测量值转换为微米测量值。
- 在 FIJI 中打开堆叠图像文件(图 4C)。
- 从工具栏中选择 Magnifying Glass(放大镜 )以扩大焦点区域。尝试用眼睛和紧邻周围的头部角质层填充屏幕(图 4D)。
- 从工具栏中选择 Freehand Select Tool 。沿着最外侧一行 ommatidia 的轮廓,尽可能接近视网膜区域的轮廓(图 4E)。要删除选区的一部分,请按住 选项 按钮并选择要删除的像素。要添加到选区,请按住 option 和 shift 按钮,然后选择要添加的像素。
- 要计算面积,请选择 Analyze | 从顶部菜单测量(图 4F)。将出现一个新窗口,其中包含 area、mean、minimum 和 maximum 参数。将这些数据复制并粘贴到电子表格中,以便进行记录和从像素到微米测量的转换。
- 执行适当的统计分析。
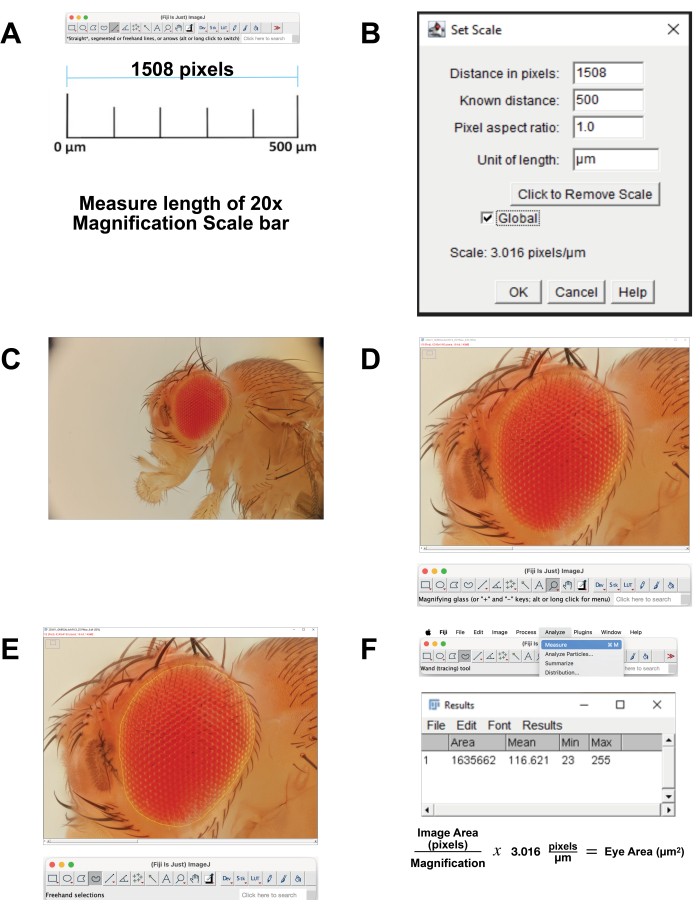
图 4:斐济的图像分析。(A) 缩放原始图像。下载校准图像并测量 500 μm 棒的长度。(B) 使用 Set Scale 功能调整缩放。(C) 打开堆叠图像。(D) 放大图像,使眼睛居中并几乎全屏。(E) 使用"徒手选择"工具在最外层的 ommatidia 行和周围角质层之间的边界处勾勒出眼睛的轮廓。(F) 测量所选区域内的面积是通过单击"分析"|"新建"来计算的测量 |面积。请单击此处查看此图的较大版本。
结果
果蝇之眼是研究组织模式、生长控制和细胞死亡的优秀模型系统。我们最近发表了一项研究,调查了细胞内 pH 值 (pHi) 如何影响组织生长。首先,我们建立了一个遗传系统,其中钠-质子交换器 DNhe2(哺乳动物 NHE1 的直系同源物)在发育中的眼睛中过表达会导致模式缺陷和增殖增加16。从酵母到哺乳动物,在物种中观察到增殖增加,pHi 更?...
讨论
在这里,我们描述了一种用于成体 果蝇 结构的样品制备、高分辨率成像和分析的方法。 果蝇 之眼是一种遗传上易于处理的模型系统,它已经对包括癌症19、神经退行性变20 和代谢疾病21 在内的疾病的分子机制产生了重要的见解。特别是,在产生携带致癌突变的转基因 果蝇 的地方会产生癌症患者?...
披露声明
作者没有需要披露的利益冲突。
致谢
作者要感谢 Grillo-Hill pHly 实验室的成员的讨论和支持。我们感谢 Tim Andriese、Randy Kirschner、Kitty (Ngoc-Huong) Nguyen、Marco Parent、Jonny Shaloub 和 Librado Veliz 的出色技术支持。这项工作得到了 NIH SC3GM132049 和 1R16GM153640 奖 (BKGH)、CSU 生物技术师生研究奖 (LM 和 BKGH) 以及圣何塞州立大学理学院和生物科学系的启动资金的支持 (FJL)。特别值得一提的是 Bernd Becker,因为他们在此过程中的足智多谋和帮助。我们感谢 BioIcons (https://bioicons.com/) 社区为我们的图表提供高质量的图标,特别是 Serviere 的移液管图标,以及 DBCLS 用于图 1 和图 2 中使用的果蝇、镊子和桌面电子显微镜图标,它们根据 CC-BY 4.0 Unported 许可。我们还要感谢 SciDraw (https://scidraw.io/) 社区为我们的图表提供高质量的图标,尤其是 Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953),它们根据知识共享 4.0 许可证 (CC-BY) 获得许可。
材料
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
参考文献
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。