Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un flujo de trabajo rápido y sencillo para la cuantificación de estructuras externas de Drosophila adulta
En este artículo
Resumen
Aquí, presentamos un flujo de trabajo rápido y de bajo costo para obtener imágenes de alta resolución de ojos de Drosophila adultas para cuantificar los patrones y los defectos de crecimiento. Describimos nuestro protocolo para la preparación de muestras mediante montaje en punto, adquisición de imágenes de alta resolución y análisis de imágenes.
Resumen
El ojo compuesto de Drosophila es un tejido con un patrón preciso que ha revelado mecanismos moleculares y procesos biológicos que impulsan la morfogénesis. Es una estructura simple de ojos unitarios repetitivos, denominada omatidios, que se utiliza para caracterizar las interacciones genéticas y las funciones de los genes. Las mutaciones que afectan a la arquitectura del ojo pueden detectarse y analizarse fácilmente; Por lo tanto, este sistema se utiliza con frecuencia en instituciones de escasos recursos. El análisis fenotípico adicional a menudo incluye un microscopio electrónico de barrido (SEM) para generar imágenes de gran aumento adecuadas para el análisis cuantitativo. Sin embargo, los SEM son caros y requieren reactivos costosos; La preparación de la muestra dura días; Y, a menudo, necesitan personal a tiempo completo para la preparación de muestras y el mantenimiento de los instrumentos. Esto limita su utilidad en instituciones de escasos recursos o durante la austeridad presupuestaria. En entomología, el uso de tecnología de imágenes digitales de alta resolución es una práctica común para la identificación y caracterización de especies. Este artículo describe un método que combina estrategias y permite la obtención de imágenes digitales de alta resolución de estructuras adultas de Drosophila y el análisis cuantitativo utilizando el software abierto ImageJ. El flujo de trabajo es una alternativa rápida y amigable para los estudiantes que remedia las limitaciones de las instalaciones de investigación con fondos y recursos insuficientes con un enfoque rápido y rentable para el análisis fenotípico cuantitativo.
Introducción
Drosophila melanogaster es un poderoso organismo modelo genético que se ha utilizado durante décadas para dilucidar las vías de señalización molecular y los comportamientos celulares. Muchas de las vías de señalización conservadas evolutivamente que son esenciales para el desarrollo multicelular se identificaron por primera vez y se definió su mecanismo de acción en Drosophila. Alrededor del 65-75% de todos los genes asociados a enfermedades humanas tienen ortólogos en Drosophila 1,2. El ojo adulto de Drosophila es un modelo importante que ha permitido realizar cribados genéticos imparciales que facilitaron el descubrimiento de importantes genes conservados implicados en enfermedades humanas, como el cáncer 3,4, la neurodegeneración5 y los trastornos metabólicos6.
El ojo de Drosophila está compuesto por ~800 ojos unitarios, denominados omatidios, que están dispuestos con precisión en un patrón hexagonal a lo largo de la superficie del ojo adulto7. Cada omatidio está compuesto por ocho neuronas fotorreceptoras que ocupan una ubicación distinta dentro de un trapecio asimétrico. Estos están respaldados por cuatro células de cono no neural y dos células pigmentarias primarias que secretan lente y pseudocono para enfocar la luz en los rabdomeros sensibles a la luz de las neuronas fotorreceptoras. Los omatidios vecinos están separados por una sola fila de células reticular interomatidiales, compuestas por células pigmentarias secundarias, células pigmentarias terciarias y complejos de cerdas mecanosensoriales 8,9,10.
Las perturbaciones en el desarrollo del ojo son visibles en los ojos de los adultos como un aumento o disminución del tamaño del ojo, una abundancia o estructura anormal de lentes o cerdas, o como un "ojo áspero" en el que el patrón hexagonal normalmente invariante se interrumpe de tal manera que ya no se puede seguir una fila de omatidios a través de la superficie del ojo. Estos fenotipos se pueden marcar a nivel de tejido macroscópico utilizando microscopios de disección. El análisis detallado de los fenotipos incluye tradicionalmente la microscopía electrónica de barrido seguida del análisis cuantitativo de imágenes11. Sin embargo, la microscopía electrónica de barrido requiere instrumentación costosa, reactivos costosos, preparación de muestras que dura días y, a menudo, personal de tiempo completo para mantenerla.
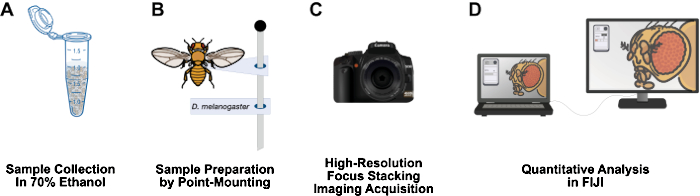
Figura 1: Flujo de trabajo para obtener imágenes de estructuras adultas de Drosophila . (A) Recolectar y fijar Drosophila adulta en etanol al 70%. (B) Prepare las muestras para la obtención de imágenes mediante el montaje en puntos y la fijación a los pines. (C) Adquirir imágenes de alta resolución a través del apilamiento de enfoque y la integración. (D) Cuantificar imágenes usando FIJI. Haga clic aquí para ver una versión más grande de esta figura.
Este documento presenta un flujo de trabajo que es relativamente económico, tiene un tiempo corto de preparación de la muestra, se puede configurar fácilmente en una mesa de laboratorio de 3 pies, no requiere materiales peligrosos y podría ser una adición duradera a los laboratorios de investigación de Drosophila (Figura 1). El montaje en punta es una técnica entomológica utilizada para secar al aire y preservar pequeños insectos de cuerpo blando, como Drosophila12. Este método se basa en la combinación de objetivos de microscopio con cámaras DSLR de alta resolución para aumentos efectivos de 10x a 1,000x. La limitada profundidad de campo inherente a la macrofotografía se supera mediante el apilamiento de enfoque: unir una serie de imágenes con el plano focal moviéndose a través del espécimen de interés13. Este método produce imágenes de alta resolución adecuadas para la cuantificación de fenotipos y podrían adaptarse fácilmente a otras estructuras de interés, como el ala, la pata, el tórax y el abdomen. El flujo de trabajo de análisis de imágenes utiliza el programa gratuito de análisis de imágenes FIJI (NIH ImageJ). Esta metodología hace que la preparación de muestras, las imágenes de alta resolución y el análisis sean accesibles para estudiantes de pregrado y científicos de instituciones de escasos recursos.
Protocolo
1. Recolección y fijación de Drosophila adulta
- Coloque cruces de Drosophila o cepas seleccionadas y colóquelas en viales que contengan alimento para moscas. Incubar los viales a la temperatura deseada (normalmente 25 °C) hasta que las moscas se hayan desarrollado y los adultos se cierren (~10-14 días a 25 °C).
- Anestesia a las moscas con CO2 y colócalas en una almohadilla de CO2 .
- Clasifique las moscas con una pluma y seleccione los individuos con el fenotipo deseado (por ejemplo, alas rectas). Haga un clasificador de moscas de plumas recortando una pluma de ganso para que quepa en el extremo cónico de una pipeta serológica de 1 mL.
- Prepare un tubo de microcentrífuga de 1,7 mL con 1 mL de etanol al 70%. Coloque las moscas seleccionadas en el tubo de microcentrífuga y póngalas en hielo. Almacene los tubos de microcentrífuga a 4 °C durante la noche (Figura 2A).
NOTA: No conserve las moscas en etanol durante más de 24 h. El almacenamiento a largo plazo de las moscas en etanol al 70% provocará la pérdida de pigmento ocular y corporal.
2. Preparación de la muestra mediante montaje puntual
NOTA: Drosophila son insectos de cuerpo blando que se vuelven quebradizos y colapsan cuando se secan al aire; Por lo tanto, este protocolo requiere que las muestras se tomen imágenes el mismo día en que se montan. Trabaje en pequeños grupos de ~ 5 moscas a la vez para evitar la pérdida de muestras. Aumente el número de muestras de un conjunto en función de la eficiencia. Las muestras que requieren más tiempo antes de la obtención de imágenes pueden deshidratarse mediante una serie de concentraciones crecientes de hexametildisilazano (HMDS)14.
- Corte pequeñas puntas triangulares (7,1 mm x 2,7 mm) de cartulina de archivo de 65 lb con un punzón especializado. Prepare los puntos doblando la punta (el 25 % más estrecho) en un ángulo de 90° con pinzas de punta fina Dumont #5 (Figura 2B).
- Con las pinzas de punta fina Dumont #5, retire las moscas de los tubos de la microcentrífuga (paso 1.4). Seque suavemente las moscas con un pañuelo de laboratorio sin pelusa para eliminar el exceso de etanol. Coloque cada mosca en su lado izquierdo en una tarjeta de índice bajo un microscopio de disección.
NOTA: Retire las moscas del tubo sujetándolas a una estructura anatómica que no sea el área de interés: al obtener imágenes de la cabeza, sostenemos las muestras por el ala o la pata. No sujete las muestras por el abdomen, ya que esa estructura se utiliza para pegar la mosca en la punta de la tarjeta. - Prepare el pegamento para pieles, ajustando su consistencia a la viscosidad deseada. Mezcle 1-2 gotas de pegamento para pieles con 1-2 gotas de agua desionizada (DI) y mezcle con una pipeta de transferencia en una ficha. Tome una punta de tarjeta preparada en el extremo ancho con pinzas y coloque una pequeña cantidad de pegamento diluido en la punta doblada de la punta frotándola en la mezcla de pegamento y agua (Figura 2C).
NOTA: El pegamento debe ser untable pero no líquido. - Aplique la punta doblada de la punta en la cara anterior del abdomen derecho alrededor de los segmentos abdominales 2-3 (Figura 2C). Antes de que el pegamento se seque, haz ligeros ajustes a la mosca de modo que el eje antero-posterior de la mosca quede perpendicular a la punta doblada de la punta.
- Inserte un pasador de montaje No. 3 en el extremo ancho de la punta de la tarjeta (Figura 2D) y asegúrelo a un bloque de clavijas para insectos (Figura 2E). Etiquete cada alfiler o fila de alfileres con el genotipo correspondiente.

Figura 2: Preparación de la muestra. (A) Las Drosophila adultas se clasifican en función de los marcadores fenotípicos y se recogen en tubos de microcentrífuga marcados que contienen un 70% de etanol en hielo. Las moscas se almacenan a 4° C durante la noche. (B) Las puntas de las tarjetas de papel se preparan doblando el extremo estrecho 90° del resto de la tarjeta con un par de pinzas #5. (C) Las moscas se recuperan de los tubos y se dejan secar brevemente al aire. El pegamento para pieles se aplica en el extremo pequeño y doblado de la punta de la tarjeta preparada y se pega a la mosca adulta en los segmentos abdominales 2-3. (D) Los especímenes se montan, con una etiqueta de identificación, en un alfiler de insectos de acero inoxidable # 3. (E) Los especímenes fijados se almacenan en una placa de muestra hasta que estén listos para la adquisición de imágenes. Haga clic aquí para ver una versión más grande de esta figura.
3. Adquisición de imágenes de apilamiento de enfoque de alta resolución
- Adquiera fotografías de alta resolución de ojos de mosca con un sistema de imágenes de apilamiento de enfoque ensamblado y personalizado, ensamblado y personalizado.
- Capture fotografías con un cuerpo de cámara DSLR con un teleobjetivo de 70-200 mm conectado a un objetivo de microscopio Apo de 20x a través de un adaptador de lente de 77 mm.
- Asegúrese de que la muestra esté iluminada con un destello a través de un difusor (Figura 3).
- Controle la posición Z mediante un controlador Stackshot y un Macro Rail.
- Conecte la cámara, el flash y la platina motorizada a un trípode de aluminio anodizado de alta resistencia.
- Coloque cada muestra montada en un punto en un cardán de etapa universal con la cabeza orientada de modo que el ojo mire hacia la lente. Realice ajustes en la posición de la cabeza moviendo suavemente la cabeza con las pinzas.
PRECAUCIÓN: Los ajustes grandes y rápidos pueden resultar en una decapitación accidental. - Con la cámara conectada a una computadora portátil, ajuste la configuración de adquisición en el software. Fotografíe muestras con un aumento de 20x con estos ajustes: Potencia del flash 1/32, Velocidad de obturación 1/200, Apertura F2.8 e ISO 400. Asegúrese de que todas las funciones de enfoque automático y estabilización de imagen estén desactivadas.
NOTA: Estos ajustes equilibran la iluminación óptima del flash, la velocidad del obturador y la profundidad de campo. Tendrían que ajustarse para otros aumentos y/o combinaciones de lentes. - Establezca la ubicación para guardar la pila de imágenes resultante (10-50 imágenes) en la carpeta de archivos deseada. Asegúrese de que haya suficiente capacidad de almacenamiento para las imágenes (~8,5 MB por imagen).
- Ajuste la configuración de la pila de enfoque en la unidad de control Stackshot en el modo de distancia automática .
- Establezca el tamaño del paso en 5 μm y calcule el número de pasos estableciendo las posiciones de inicio y parada de la pila de enfoque.
- Visualice el espécimen en el modo LiveView y con la cámara en el modo de disparo automático para identificar las posiciones de inicio y parada.
- Mueva el riel de modo que la parte más cercana del espécimen esté enfocada (establecer la posición inicial ) y, a continuación, desplácese hasta donde esté enfocada la entidad más lejana de interés (establecer la posición final ).
- Vuelva a colocar la cámara en el modo de disparo manual e inicie la adquisición de imágenes desde la unidad de control Stackshot.
NOTA: El tiempo de adquisición de la imagen depende del tamaño del espécimen. Cuanto mayor sea la profundidad de campo necesaria para muestras grandes, más cortes se incluirán en la pila de imágenes, lo que extenderá el tiempo total de adquisición. - Abra archivos en el software de apilamiento de enfoque al que se hace referencia. Genere una imagen apilada haciendo clic en Apilar | Alinear y apilar todo (PMax).
- Guarde la imagen final en el disco duro del ordenador como un archivo .tif haciendo clic en Archivo | Guarde la imagen de salida.
NOTA: Dependiendo de la resolución del archivo de imagen apilado y del número de muestras de las que se haya creado una imagen, es posible que se necesiten discos duros externos grandes (1 TB) para la copia de seguridad de la imagen. En este protocolo, las imágenes apiladas son de aproximadamente 100 MB cada una antes de ser comprimidas.

Figura 3: Adquisición de imágenes. (A) Aparato de imagen con partes etiquetadas de la siguiente manera: a) Cuerpo de la cámara DSLR; b) teleobjetivo; c) 20 objetivos y adaptadores de microscopio Apo; d) Destello; e) Difusores de lentes y cúpulas; f) Controlador de tiro apilado, Macro Rail y Etapa giratoria; g) Gimbal de escenario universal; h) Trípode. (B) Aparato de imagen con difusor de luz en su lugar. (C) Primer plano de la muestra montada en posición para la toma de imágenes. Haga clic aquí para ver una versión más grande de esta figura.
4. Flujo de trabajo de análisis FIJI para calcular el área ocular de un adulto
- Para el análisis de imágenes, obtenga el software FIJI15 en el sitio web de ImageJ.net.
- Elija imágenes para el análisis en las que el ojo esté centrado y alineado con una iluminación adecuada y un desenfoque periférico mínimo, lo que indica una alineación adecuada con la cámara.
- Calibra la escala de la imagen.
- Descargue la imagen de la barra de escala para un aumento de 20x que se correlaciona con 500 μm. Alternativamente, en el momento de la adquisición de la imagen, fotografíe una regla utilizando los mismos ajustes. Abra la imagen en el software FIJI.
- Mida la longitud de la barra de escala (Figura 4A). Usa la Herramienta Línea Recta para trazar exactamente la línea. Haga clic en Analizar | Medir. Esta distancia de píxeles es equivalente a 500 μm (Figura 4B).
- Calcule píxeles por micra. Utilícelo para convertir las mediciones de píxeles en mediciones micrométricas.
- Abra el archivo de imagen apilado en FIJI (Figura 4C).
- Seleccione Lupa en la barra de herramientas para ampliar el área de enfoque. Trate de llenar la pantalla con el ojo y la cutícula de la cabeza circundante inmediata (Figura 4D).
- Seleccione Herramienta de selección a mano alzada en la barra de herramientas. Delinee el área de la retina lo más cerca posible, siguiendo los contornos de la fila más externa de omatidios (Figura 4E). Para eliminar parte de la selección, mantenga presionado el botón de opción y seleccione los píxeles que desea eliminar. Para agregar a la selección, mantenga presionados los botones de opción y cambio y seleccione los píxeles que desea agregar.
- Para calcular el área, seleccione Analizar | Mida desde el menú superior (Figura 4F). Aparecerá una nueva ventana con los parámetros de área, media, mínimo y máximo . Copie y pegue estos datos en una hoja de cálculo para documentarlos y convertirlos de píxeles a micrómetros.
- Realizar análisis estadísticos adecuados.
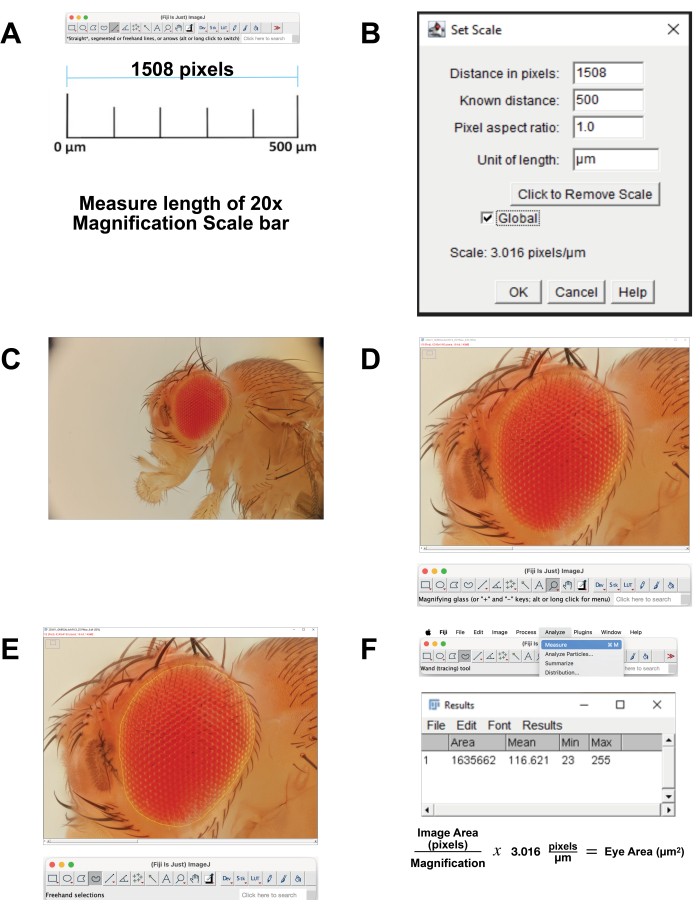
Figura 4: Análisis de imágenes en FIJI. (A) Escalar la imagen original. Descargue la imagen de calibración y mida la longitud de la barra de 500 μm. (B) Ajuste la escala usando la función Establecer escala . (C) Abra la imagen apilada. (D) Amplíe la imagen para que el ojo esté centrado y casi en pantalla completa. (E) Utilice la herramienta Selección a mano alzada para delinear el ojo en el borde entre la fila más externa de omatidios y la cutícula circundante. (F) Mida el área dentro de la región seleccionada haciendo clic en Analizar | Medir | Área. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
El ojo de Drosophila es un excelente sistema modelo para estudiar el patrón de tejidos, el control del crecimiento y la muerte celular. Recientemente hemos publicado un estudio que investiga cómo el pH intracelular (pHi) influye en el crecimiento de los tejidos. En primer lugar, establecimos un sistema genético en el que la sobreexpresión del intercambiador de sodio-protones DNhe2 (el ortólogo del NHE1 de los mamíferos) en el ojo en desarrollo causa defectos en el...
Discusión
Aquí describimos un método para la preparación de muestras, imágenes de alta resolución y análisis de estructuras adultas de Drosophila . El ojo de Drosophila es un sistema modelo genéticamente manejable que ha proporcionado información crítica sobre los mecanismos moleculares que subyacen a enfermedades como el cáncer19, la neurodegeneración20 y las enfermedades metabólicas21. En partic...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores desean agradecer a los miembros del laboratorio de pH de Grillo-Hill por las discusiones y el apoyo. Agradecemos a Tim Andriese, Randy Kirschner, Kitty (Ngoc-Huong) Nguyen, Marco Parent, Jonny Shaloub y Librado Veliz por su excelente soporte técnico. Este trabajo contó con el apoyo de los premios NIH SC3GM132049 y 1R16GM153640 (BKGH), un premio de investigación de la facultad de biotecnología de la CSU (LM y BKGH) y fondos iniciales de la Facultad de Ciencias y el Departamento de Ciencias Biológicas de la Universidad Estatal de San José (FJL). Mención especial para Bernd Becker por su ingenio y asistencia durante este proceso. Agradecemos a la comunidad de BioIcons (https://bioicons.com/) por proporcionar iconos de alta calidad para nuestras figuras y especialmente a Serviere por el icono de la pipeta, y a DBCLS por los iconos de Drosophila, fórceps y microscopio electrónico de escritorio utilizados en la Figura 1 y la Figura 2, que tienen licencia CC-BY 4.0 Unported. También agradecemos a la comunidad de SciDraw (https://scidraw.io/) por proporcionar íconos de alta calidad para nuestras figuras, especialmente Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953), que están bajo licencia Creative Commons 4.0 (CC-BY).
Materiales
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
Referencias
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados