È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un flusso di lavoro rapido e semplice per la quantificazione delle strutture esterne di Drosophila adulta
In questo articolo
Riepilogo
Qui, presentiamo un flusso di lavoro rapido e a basso costo per l'imaging ad alta risoluzione degli occhi adulti di Drosophila per quantificare il patterning e i difetti di crescita. Descriviamo il nostro protocollo per la preparazione dei campioni mediante montaggio su punti, acquisizione di immagini ad alta risoluzione e analisi delle immagini.
Abstract
L'occhio composto di Drosophila è un tessuto modellato con precisione che ha rivelato meccanismi molecolari e processi biologici che guidano la morfogenesi. Si tratta di una semplice struttura di occhi unitari ripetuti, chiamata ommatidi, che viene utilizzata per caratterizzare le interazioni genetiche e le funzioni geniche. Le mutazioni che interessano l'architettura dell'occhio possono essere facilmente rilevate e analizzate; Pertanto, questo sistema è spesso utilizzato in istituzioni con risorse insufficienti. Un'ulteriore analisi fenotipica include spesso un microscopio elettronico a scansione (SEM) per generare immagini ad alto ingrandimento adatte all'analisi quantitativa. Tuttavia, i SEM sono costosi e richiedono reagenti costosi; La preparazione del campione dura giorni; e, spesso, hanno bisogno di personale a tempo pieno per la preparazione dei campioni e la manutenzione degli strumenti. Ciò limita la loro utilità presso le istituzioni con risorse insufficienti o durante l'austerità di bilancio. In entomologia, l'uso della tecnologia di imaging digitale ad alta risoluzione è una pratica comune per l'identificazione e la caratterizzazione delle specie. Questo articolo descrive un metodo che combina strategie e consente l'imaging digitale ad alta risoluzione delle strutture adulte di Drosophila e l'analisi quantitativa utilizzando il software aperto ImageJ. Il flusso di lavoro è un'alternativa rapida e di facile utilizzo per gli studenti che pone rimedio ai limiti delle strutture di ricerca sottofinanziate e con risorse insufficienti con un approccio rapido ed economico all'analisi fenotipica quantitativa.
Introduzione
La Drosophila melanogaster è un potente organismo modello genetico che è stato utilizzato per decenni per chiarire le vie di segnalazione molecolare e i comportamenti cellulari. Molte delle vie di segnalazione evolutivamente conservate che sono essenziali per lo sviluppo multicellulare sono state identificate per la prima volta e il loro meccanismo d'azione definito in Drosophila. Circa il 65-75% di tutti i geni umani associati alla malattia ha ortologhi in Drosophila 1,2. L'occhio adulto di Drosophila è un modello importante che ha permesso di effettuare screening genetici imparziali che hanno facilitato la scoperta di importanti geni conservati implicati nelle malattie umane, tra cui il cancro 3,4, la neurodegenerazione5 e i disturbi metabolici6.
L'occhio di Drosophila è composto da ~800 occhi unitari, chiamati ommatidi, che sono disposti con precisione in uno schema esagonale sulla superficie dell'occhio adulto7. Ogni ommatidio è composto da otto neuroni fotorecettori che occupano una posizione distinta all'interno di un trapezio asimmetrico. Questi sono supportati da quattro cellule coniche non neurali e due cellule pigmentate primarie che secernono cristallino e pseudo-cono per focalizzare la luce sui rabdomeri sensibili alla luce dei neuroni fotorecettori. Gli ommatidi vicini sono separati da una singola fila di cellule reticolari interommatidiali, costituite da cellule di pigmento secondario, cellule di pigmento terziarie e complessi di setole meccanosensoriali 8,9,10.
Le perturbazioni nello sviluppo dell'occhio sono visibili negli occhi adulti come aumento o diminuzione delle dimensioni dell'occhio, abbondanza o struttura anormale di lenti o setole, o come un "occhio ruvido" in cui il modello esagonale normalmente invariante è interrotto in modo tale che una fila di ommatidi non può più essere seguita attraverso la superficie dell'occhio. Questi fenotipi possono essere valutati a livello di tessuto grossolano utilizzando microscopi da dissezione. L'analisi dettagliata dei fenotipi include tradizionalmente la microscopia elettronica a scansione seguita dall'analisi quantitativa delle immagini11. Tuttavia, la microscopia elettronica a scansione richiede strumentazione costosa, reagenti costosi, preparazione del campione che dura giorni e, spesso, personale a tempo pieno per la manutenzione.
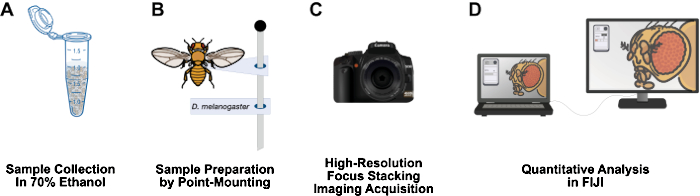
Figura 1: Flusso di lavoro per l'imaging delle strutture adulte di Drosophila . (A) Raccogliere e fissare la Drosophila adulta in etanolo al 70%. (B) Preparare i campioni per l'imaging mediante montaggio puntuale e fissaggio ai pin. (C) Acquisizione di immagini ad alta risoluzione tramite focus stacking e integrazione. (D) Quantificare le immagini utilizzando le FIJI. Clicca qui per visualizzare una versione più grande di questa figura.
Questo documento presenta un flusso di lavoro relativamente economico, con un breve tempo di preparazione del campione, può essere facilmente installato su un banco da laboratorio di 3 piedi, non richiede materiali pericolosi e potrebbe essere un'aggiunta di lunga durata ai laboratori di ricerca sulla Drosophila (Figura 1). Il montaggio a punta è una tecnica entomologica utilizzata per essiccare all'aria e conservare piccoli insetti dal corpo molle, come la Drosophila12. Questo metodo si basa sulla combinazione di obiettivi per microscopio con fotocamere DSLR ad alta risoluzione per ingrandimenti efficaci da 10x a 1.000x. La limitata profondità di campo inerente alla macrofotografia è superata dal focus stacking: cucendo insieme una serie di immagini con il piano focale che si muove attraverso il campione di interesse13. Questo metodo produce immagini ad alta risoluzione adatte alla quantificazione dei fenotipi e potrebbero essere facilmente adattate per altre strutture di interesse, come l'ala, la zampa, il torace e l'addome. Il flusso di lavoro di analisi delle immagini utilizza il programma gratuito di analisi delle immagini FIJI (NIH ImageJ). Questa metodologia rende la preparazione dei campioni, l'imaging ad alta risoluzione e l'analisi accessibili a studenti universitari e scienziati di istituzioni con risorse insufficienti.
Protocollo
1. Raccolta e fissazione della Drosophila adulta
- Prepara le croci di Drosophila o seleziona i ceppi e mettili in fiale contenenti cibo per mosche. Incubare le fiale alla temperatura desiderata (di solito 25 °C) fino a quando le mosche non si sono sviluppate e gli adulti si sono chiusi (~10-14 giorni a 25 °C).
- Anestetizzare le mosche con CO2 e posizionarle su un tampone CO2 .
- Ordina le mosche con una piuma e seleziona gli individui con il fenotipo desiderato (ad esempio, ali dritte). Crea una selezionatrice di mosche di piume tagliando una piuma d'oca per adattarla all'estremità affusolata di una pipetta sierologica da 1 ml.
- Preparare una provetta da microcentrifuga da 1,7 mL con 1 mL di etanolo al 70%. Metti le mosche selezionate nella provetta della microcentrifuga e metti sul ghiaccio. Conservare le provette per microcentrifuga a 4 °C per una notte (Figura 2A).
NOTA: Non conservare le mosche in etanolo per più di 24 ore. La conservazione a lungo termine delle mosche in etanolo al 70% comporterà la perdita di pigmento per gli occhi e il corpo.
2. Preparazione del campione mediante montaggio puntuale
NOTA: Le Drosophila sono insetti dal corpo molle che diventano fragili e collassano quando vengono essiccati all'aria; Pertanto, questo protocollo richiede che i campioni vengano ripresi lo stesso giorno in cui vengono montati. Lavorare in piccoli gruppi di ~5 mosche alla volta per evitare la perdita di campione. Aumenta il numero di campioni in un set in base all'efficienza. I campioni che richiedono più tempo prima dell'imaging possono essere disidratati attraverso una serie di concentrazioni crescenti di esametiltililazano (HMDS)14.
- Taglia piccole punte triangolari (7,1 mm x 2,7 mm) da cartoncino d'archivio da 65 libbre utilizzando un punzone specializzato. Preparare le punte piegando la punta (la più stretta del 25%) a un angolo di 90° con le pinze a punta fine Dumont #5 (Figura 2B).
- Utilizzando la pinza a punta fine Dumont #5, rimuovere le mosche dalle provette per microcentrifuga (passaggio 1.4). Tampona delicatamente le mosche con un panno di laboratorio privo di lanugine per rimuovere l'etanolo in eccesso. Posiziona ogni mosca sul lato sinistro su una scheda sotto un microscopio da dissezione.
NOTA: Rimuovere le mosche dal tubo tenendolo per una struttura anatomica che non sia l'area di interesse: durante l'imaging della testa, teniamo i campioni per l'ala o la zampa. Non tenere i campioni per l'addome, poiché quella struttura viene utilizzata per incollare la mosca sulla punta della carta. - Preparare la colla per pelli, regolandone la consistenza alla viscosità desiderata. Mescolare 1-2 gocce di colla per pelli con 1-2 gocce di acqua deionizzata (DI) e mescolare con una pipetta di trasferimento su una scheda. Raccogli una punta di carta preparata all'estremità larga con una pinza e posiziona una piccola quantità di colla diluita sulla punta piegata della punta tamponandola nella miscela di colla e acqua (Figura 2C).
NOTA: La colla deve essere spalmabile ma non liquida. - Applicare la punta piegata della punta sul lato anteriore dell'addome destro attorno ai segmenti addominali 2-3 (Figura 2C). Prima che la colla si asciughi, apportare lievi modifiche alla mosca in modo tale che l'asse antero-posteriore della mosca sia perpendicolare alla punta piegata della punta.
- Inserire un perno di montaggio n. 3 nell'estremità larga della punta della scheda (Figura 2D) e fissarlo a un blocco di fissaggio per insetti (Figura 2E). Etichetta ogni spillo o fila di spilli con il genotipo corrispondente.

Figura 2: Preparazione del campione. (A) Le Drosophila adulte vengono selezionate in base a marcatori fenotipici e raccolte in provette da microcentrifuga marcate contenenti il 70% di etanolo su ghiaccio. Le mosche vengono conservate a 4° C durante la notte. (B) I punti della carta di carta vengono preparati piegando l'estremità stretta di 90° dal resto della carta utilizzando un paio di pinze #5. (C) Le mosche vengono recuperate dai tubi e lasciate asciugare brevemente all'aria. La colla di pelle viene applicata all'estremità piccola e piegata della punta della carta preparata e incollata alla mosca adulta ai segmenti addominali 2-3. (D) Gli esemplari sono montati, con un'etichetta di identificazione, su un perno per insetti in acciaio inossidabile #3. (E) I campioni appuntati vengono conservati su una scheda campione fino al momento dell'acquisizione dell'immagine. Clicca qui per visualizzare una versione più grande di questa figura.
3. Acquisizione di immagini con focus stacking ad alta risoluzione
- Acquisisci fotografie ad alta risoluzione degli occhi delle mosche con un sistema di imaging focus stacking assemblato e personalizzato.
- Scatta fotografie con un corpo macchina DSLR con un teleobiettivo da 70-200 mm collegato a un obiettivo per microscopio Apo 20x tramite un adattatore per obiettivo da 77 mm.
- Assicurarsi che il campione sia illuminato con un flash attraverso un diffusore (Figura 3).
- Controlla il posizionamento Z utilizzando un controller Stackshot e una guida macro.
- Collega la fotocamera, il flash e il tavolino motorizzato a un treppiede in alluminio anodizzato per impieghi gravosi.
- Posiziona ogni campione montato su un gimbal a tavolino universale con la testa orientata in modo che l'occhio sia rivolto verso l'obiettivo. Effettuare le regolazioni della posizione della testa muovendo delicatamente la testa con la pinza.
ATTENZIONE: Regolazioni grandi e rapide possono provocare la decapitazione accidentale. - Con la fotocamera collegata a un computer portatile, regolare le impostazioni di acquisizione nel software. Fotografa i campioni con un ingrandimento di 20x con queste impostazioni: Potenza flash 1/32, Velocità dell'otturatore 1/200, Apertura F2.8 e ISO 400. Assicurarsi che tutte le funzioni di messa a fuoco automatica e stabilizzazione dell'immagine siano disattivate.
NOTA: Queste impostazioni bilanciano l'illuminazione ottimale del flash, la velocità dell'otturatore e la profondità di campo. Dovrebbero essere regolati per altri ingrandimenti e/o combinazioni di lenti. - Impostare la posizione per il salvataggio della pila di immagini risultante (10-50 immagini) nella cartella di file desiderata. Garantire una capacità di archiviazione sufficiente per le immagini (~8,5 MB per immagine).
- Regolare le impostazioni della sequenza di messa a fuoco sull'unità di controllo Stackshot in modalità Distanza automatica.
- Impostare la dimensione del passo su 5 μm e calcolare il numero di passi impostando le posizioni di avvio e di arresto dello stack di messa a fuoco.
- Visualizzare il campione in modalità LiveView e con la fotocamera in modalità di scatto automatico per identificare le posizioni di inizio e fine.
- Spostare il binario in modo che la parte più vicina del campione sia a fuoco (impostare la posizione iniziale ), quindi spostarsi nel punto in cui è a fuoco la caratteristica più lontana di interesse (impostare la posizione finale ).
- Riportare la fotocamera alla modalità di scatto manuale e avviare l'acquisizione dell'immagine dall'unità di controllo Stackshot.
NOTA: Il tempo di acquisizione dell'immagine dipende dalle dimensioni del campione. Maggiore è la profondità di campo necessaria per campioni di grandi dimensioni, più sezioni sono incluse nella pila di immagini, il che prolungherà il tempo di acquisizione complessivo. - Aprire i file nel software di focus stacking di riferimento. Generare un'immagine impilata facendo clic su Impila | Allinea e impila tutto (PMax).
- Salvare l'immagine finale sul disco rigido del computer come file .tif facendo clic su File | Salva l'immagine di output.
NOTA: A seconda della risoluzione del file di immagine impilato e del numero di campioni di cui è stata creata l'immagine, potrebbero essere necessari dischi rigidi esterni di grandi dimensioni (1 TB) per il backup dell'immagine. In questo protocollo, le immagini impilate sono di circa 100 MB ciascuna prima di essere compresse.

Figura 3: Acquisizione dell'immagine. (A) Apparecchio di imaging con parti etichettate come segue: a) Corpo della fotocamera DSLR; b) teleobiettivo; c) Obiettivo e adattatore per microscopio Apo 20x; d) Flash; e) Diffusori a lente e a cupola; f) Stackshot Controller, Macro Rail e Rotary Stage; g) Gimbal da palco universale; h) Treppiede. (B) Apparecchio di imaging con diffusore di luce in posizione. (C) Primo piano del campione montato in posizione per l'imaging. Clicca qui per visualizzare una versione più grande di questa figura.
4. Flusso di lavoro di analisi FIJI per calcolare l'area oculare degli adulti
- Per l'analisi delle immagini, ottenere il software FIJI15 dal sito Web ImageJ.net.
- Scegli immagini per l'analisi in cui l'occhio è centrato e allineato con un'illuminazione adeguata e una sfocatura periferica minima, indicando il corretto allineamento con la fotocamera.
- Calibrare la scala dell'immagine.
- Scarica l'immagine della barra della scala per un ingrandimento 20x correlato a 500 μm. In alternativa, al momento dell'acquisizione dell'immagine, fotografare un righello utilizzando le stesse impostazioni. Apri l'immagine nel software FIJI.
- Misurare la lunghezza della barra della scala (Figura 4A). Usa lo strumento Linea retta per tracciare esattamente la linea. Fare clic su Analizza | Misurare. Questa distanza di pixel è equivalente a 500 μm (Figura 4B).
- Calcola i pixel per micron. Utilizzare questa opzione per convertire le misurazioni dei pixel in misurazioni micrometriche.
- Aprire il file immagine impilato in FIJI (Figura 4C).
- Seleziona Lente di ingrandimento dalla barra degli strumenti per ingrandire l'area di messa a fuoco. Provare a riempire lo schermo con l'occhio e la cuticola della testa immediatamente circostante (Figura 4D).
- Seleziona lo strumento di selezione a mano libera dalla barra degli strumenti. Delineare l'area retinica il più fedelmente possibile, seguendo i contorni della fila più esterna di ommatidi (Figura 4E). Per rimuovere parte della selezione, tieni premuto il pulsante di opzione e seleziona i pixel da rimuovere. Per aggiungere alla selezione, tieni premuti i pulsanti opzione e Maiusc e seleziona i pixel da aggiungere.
- Per calcolare l'area, selezionare Analizza | Misurare dal menu in alto (Figura 4F). Apparirà una nuova finestra con i parametri area, media, minimo e massimo . Copia e incolla questi dati in un foglio di calcolo per la documentazione e la conversione da pixel a misurazioni micrometriche.
- Eseguire analisi statistiche appropriate.
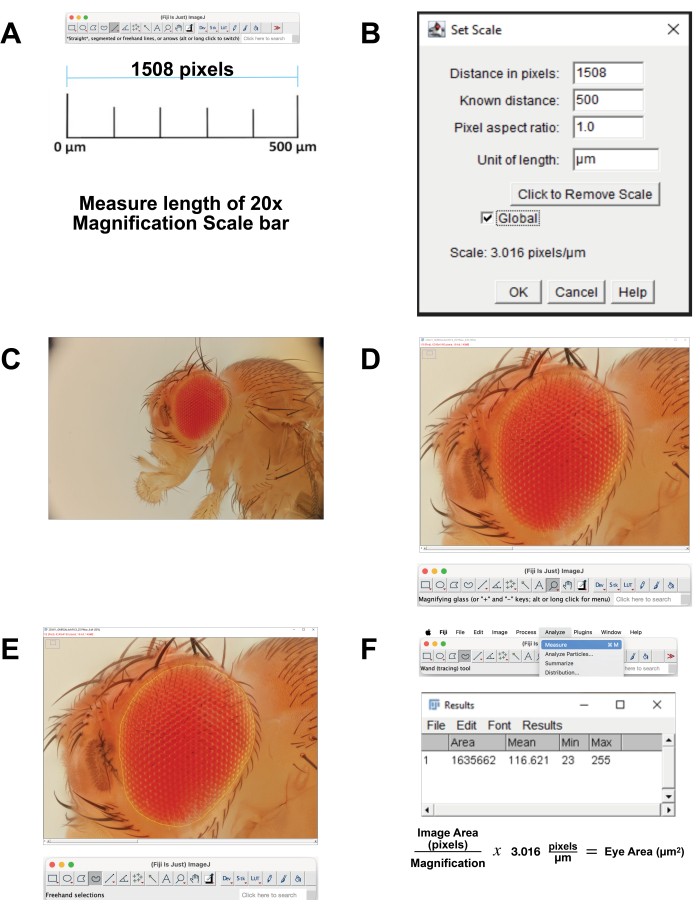
Figura 4: Analisi dell'immagine nelle FIJI. (A) Ridimensionare l'immagine originale. Scarica l'immagine di calibrazione e misura la lunghezza della barra da 500 μm. (B) Regolare la scala utilizzando la funzione Imposta scala . (C) Aprire l'immagine impilata. (D) Ingrandisci l'immagine in modo che l'occhio sia centrato e quasi a schermo intero. (E) Utilizzare lo strumento Selezione a mano libera per delineare l'occhio al confine tra la fila più esterna di ommatidi e la cuticola circostante. (F) Misurare l'area all'interno della regione selezionata viene calcolata facendo clic su Analizza | Misura | Area. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
L'occhio di Drosophila è un eccellente sistema modello per lo studio del pattern tissutale, del controllo della crescita e della morte cellulare. Di recente abbiamo pubblicato uno studio che indaga su come il pH intracellulare (pHi) influenzi la crescita dei tessuti. In primo luogo, abbiamo stabilito un sistema genetico in cui la sovraespressione dello scambiatore sodio-protone DNhe2 (l'ortologo di NHE1 nei mammiferi) nell'occhio in via di sviluppo causa difetti di pat...
Discussione
Qui descriviamo un metodo per la preparazione del campione, l'imaging ad alta risoluzione e l'analisi delle strutture adulte di Drosophila . L'occhio di Drosophila è un sistema modello geneticamente trattabile che ha prodotto informazioni critiche sui meccanismi molecolari alla base di malattie tra cui il cancro19, la neurodegenerazione20 e le malattie metaboliche21. In particolare, vengono generat...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare i membri del laboratorio di pH Grillo-Hill per le discussioni e il supporto. Ringraziamo Tim Andriese, Randy Kirschner, Kitty (Ngoc-Huong) Nguyen, Marco Parent, Jonny Shaloub e Librado Veliz per l'eccellente supporto tecnico. Questo lavoro è stato supportato dai premi NIH SC3GM132049 e 1R16GM153640 (BKGH), da un premio per la ricerca della facoltà di biotecnologia della CSU (LM e BKGH) e da fondi di avviamento del College of Science e del Dipartimento di Scienze Biologiche della San José State University (FJL). Una menzione speciale va a Bernd Becker per la sua intraprendenza e assistenza durante questo processo. Ringraziamo la comunità BioIcons (https://bioicons.com/) per aver fornito icone di alta qualità per le nostre figure e in particolare a Serviere per l'icona della pipetta, e DBCLS per le icone di Drosophila, pinze e microscopio elettronico da tavolo utilizzate in Figura 1 e Figura 2, che sono concesse in licenza sotto licenza CC-BY 4.0 Unported. Ringraziamo anche la comunità SciDraw (https://scidraw.io/) per aver fornito icone di alta qualità per le nostre figure, in particolare Diogo Losch De Oliveira (doi.org/10.5281/zenodo.3925953), che sono concesse in licenza con licenza Creative Commons 4.0 (CC-BY).
Materiali
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
Riferimenti
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -. P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317 (2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934 (2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796 (2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340 (2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041 (2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. . Insect collection and identification: Techniques for the field and laboratory. , (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10 (1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270 (2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13 (2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241 (2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon