このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
成人の ショウジョウバエ の外部構造を定量するための迅速でシンプルなワークフロー
要約
ここでは、成虫の ショウジョウバエ の眼の高解像度イメージングのための迅速で低コストのワークフローを紹介し、パターン化と成長欠陥を定量化します。ポイントマウント、高解像度画像取得、および画像分析によるサンプル調製のプロトコルについて説明します。
要約
ショウジョウバエの複眼は、形態形成を促進する分子メカニズムと生物学的プロセスを明らかにした、正確にパターン化された組織です。これは、オマチディアと呼ばれる反復単位の目の単純な構造であり、遺伝的相互作用と遺伝子機能を特徴付けるために使用されます。目の構造に影響を与える突然変異は、簡単に検出および分析できます。したがって、このシステムは、リソースが不足している機関で頻繁に使用されます。さらなる表現型解析には、定量解析に適した高倍率画像を生成するための走査型電子顕微鏡(SEM)が含まれることがよくあります。しかし、SEMは高価であり、高価な試薬が必要です。サンプル調製は数日に及びます。また、多くの場合、サンプル調製や機器のメンテナンスのためにフルタイムのスタッフが必要です。これは、リソースが不足している機関や予算の緊縮財政中にそれらの有用性を制限します。昆虫学では、高解像度のデジタルイメージング技術の使用は、種の同定と特性評価のための一般的な方法です。この論文では、戦略を組み合わせて、成虫のショウジョウバエの構造の高解像度デジタルイメージングと、オープンソフトウェアのImageJを使用した定量分析を可能にする方法について説明します。このワークフローは、定量的表現型分析への費用対効果の高い迅速なアプローチにより、資金不足やリソース不足の研究施設の制限を解消する、迅速で学生に優しい代替手段です。
概要
ショウジョウバエ・メラノガスターは、分子シグナル伝達経路や細胞の挙動を解明するために何十年にもわたって使用されてきた強力な遺伝モデル生物です。多細胞発生に不可欠な進化的に保存されたシグナル伝達経路の多くが、ショウジョウバエで初めて同定され、その作用機序が定義された。ヒトの疾患関連遺伝子の約65-75%がショウジョウバエ1,2にオルソログを持っています。成体のショウジョウバエの眼は、がん3,4、神経変性5、代謝障害6などのヒト疾患に関与する重要な保存遺伝子の発見を促進する偏りのない遺伝的スクリーニングを可能にした重要なモデルです。
ショウジョウバエの眼は、オマチディアと呼ばれる~800単位の眼で構成され、これらは成虫の眼7の表面を横切る六角形のパターンで正確に配列されています。各オマチジウムは、非対称の台形内の異なる位置を占める8つの視細胞ニューロンで構成されています。これらは、4つの非神経錐体細胞と2つの一次色素細胞によって支えられており、レンズと疑似錐体を分泌して光受容体ニューロンの光感知ラブドームに光を集中させます。隣接するommatidiaは、二次色素細胞、三次色素細胞、および機械感覚剛毛複合体8,9,10で構成される、骨間格子細胞の1列によって分離されている。
眼の発達の摂動は、成人の眼では、眼の大きさの増加または減少、レンズや剛毛の異常な存在量または構造、または通常は不変の六角形のパターンが乱され、眼の表面を横切って眼球の列をたどることができなくなる「粗い目」として見られます。これらの表現型は、解剖顕微鏡を使用して肉眼組織レベルでスコアリングできます。表現型の詳細な分析には、従来、走査型電子顕微鏡とそれに続く定量的画像分析が含まれます11。しかし、走査型電子顕微鏡には、高価な装置、高価な試薬、数日にわたるサンプル調製、そして多くの場合、フルタイムのスタッフのメンテナンスが必要です。
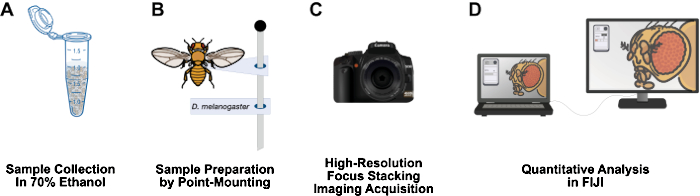
図1:成体の ショウジョウバエ の構造をイメージングするためのワークフロー。 (A)成虫の ショウジョウバエを 70%エタノールに集めて固定します。(B)イメージング用のサンプルをポイントマウントしてピンに貼り付けます。(C)焦点合成と統合により高解像度の画像を取得します。(D)FIJIを使用して画像を定量化します。 この図の拡大版を表示するには、ここをクリックしてください。
この論文では、比較的安価で、サンプル調製時間が短く、3フィートのラボベンチに簡単に設置でき、危険物を必要とせず、 ショウジョウバエ の研究ラボに長期間追加できるワークフローを紹介しています(図1)。ポイントマウントは、 ショウジョウバエ12などの小さくて柔らかい体の昆虫を風乾して保存するために使用される昆虫学的手法です。この方法は、顕微鏡の対物レンズと高解像度のデジタル一眼レフカメラを組み合わせて、10倍から1,000倍の有効倍率を実現することに依存しています。マクロ写真に固有の限られた被写界深度は、フォーカススタッキングによって克服されます:焦点面が目的の標本13を移動する一連の画像をつなぎ合わせます。この方法では、表現型の定量に適した高解像度の画像が得られ、翼、脚、胸部、腹部など、他の関心のある構造にも簡単に適応できます。画像解析ワークフローは、無料の画像解析プログラムFIJI(NIH ImageJ)を使用しています。この方法論により、サンプル調製、高解像度イメージング、および分析は、リソースが不足している機関の学部生や科学者が利用できるようになります。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1.成虫の ショウジョウバエ の収集と固定
- ショウジョウバエのクロスを設定するか、菌株を選択してフライフードの入ったバイアルに入れます。バエが成長し、成虫が閉じるまで(25°Cで~10~14日)、所望の温度(通常は25°C)でバイアルをインキュベートします。
- ハエをCO2 で麻酔し、CO2 パッドの上に置きます。
- 羽でハエを選別し、目的の表現型(例:まっすぐな翼)を持つ個体を選択します。1 mLの血清ピペットの先細りの端に収まるようにガチョウの羽をトリミングして、フェザーフライソーターを作成します。
- 1.7 mLの微量遠心チューブに1 mLの70%エタノールを調製します。選択したハエを微量遠心チューブに入れ、氷の上に置きます。微量遠心チューブは4°Cで一晩保管してください(図2A)。
注:ハエをエタノールで24時間以上保存しないでください。70%エタノールでハエを長期間保存すると、目と体の色素が失われます。
2. ポイントマウントによるサンプル調製
注: ショウジョウバエ は柔らかい体の昆虫で、風乾するともろくなり、崩壊します。したがって、このプロトコルでは、サンプルをマウントしたその日にイメージングする必要があります。一度に~5匹のフライの小さなセットで作業し、サンプルの損失を防ぎます。効率に基づいてセット内のサンプル数を増やします。イメージングまでにより多くの時間を必要とする標本は、ヘキサメチルジシラザン(HMDS)の濃度を上げることにより脱水することができます14。
- 専用のポイントパンチを使用して、アーカイブの65ポンドの厚紙から小さな三角形のポイント(7.1 mm x 2.7 mm)を切り取ります。Dumont #5 細先端鉗子を使用して、先端 (最も狭い 25%) を 90° の角度に曲げてポイントを準備します (図 2B)。
- Dumont #5細い先端の鉗子を使用して、ハエを微量遠心チューブから取り出します(ステップ1.4)。糸くずの出ない実験用ティッシュでハエをやさしく吸い取り、余分なエタノールを取り除きます。各フライをインデックスカードの左側に、解剖顕微鏡で配置します。
注:関心のある領域ではない解剖学的構造を保持して、チューブからハエを取り除きます-頭をイメージングするときは、翼または脚でサンプルを保持します。腹部にサンプルを保持しないでください、その構造はフライをカードポイントに接着するために使用されます。 - 皮のりを準備し、その一貫性を希望の粘度に調整します。1〜2滴の皮接着剤を1〜2滴の脱イオン(DI)水と混合し、インデックスカード上のトランスファーピペットと混合します。鉗子で幅の広い端に準備されたカードポイントを拾い上げ、接着剤と水の混合物に軽くたたいて、ポイントの曲がった先端に希釈した接着剤を少量置きます(図2C)。
注:接着剤は広げられる必要がありますが、水っぽくはありません。 - ポイントの曲がった先端を腹部セグメント2-3の周りの右腹部の前方に当てます(図2C)。接着剤が乾く前に、フライの前後軸がポイントの曲がった先端に垂直になるようにフライをわずかに調整します。
- No.3の取り付けピンをカードポイントの広い方の端(図2D)に挿入し、昆虫ピン留めブロック(図2E)に固定します。各ピンまたはピンの行に対応する遺伝子型にラベルを付けます。

図2:サンプル調製 (A)成体の ショウジョウバエ は、表現型マーカーに基づいて選別され、氷上に70%エタノールを含む標識された微量遠心チューブに収集されます。ハエは4°Cで一晩保管されます。(B)紙のカードポイントは、#5鉗子のペアを使用して、狭い方の端をカードの残りの部分から90°曲げることによって準備されます。(C)ハエをチューブから回収し、短時間風乾させます。皮のりは、準備されたカードポイントの小さな折り畳まれた端に塗布され、腹部セグメント2-3で成虫のハエに接着されます。(D)標本は、識別ラベル付きで、#3ステンレス鋼の昆虫ピンに取り付けられます。(E)ピン留めされた標本は、画像取得の準備が整うまでサンプルボードに保存されます。 この図の拡大版を表示するには、ここをクリックしてください。
3. 高解像度フォーカススタッキングイメージング取得
- 組み立てられカスタマイズされたフォーカススタッキングイメージングシステムを使用して、フライアイの高解像度写真を取得します。
- 70-200 mm望遠レンズを77 mmレンズアダプターを介して20x Apo顕微鏡対物レンズに接続されたデジタル一眼レフカメラボディで写真を撮影します。
- 試料がディフューザーを介してフラッシュで照らされていることを確認します(図3)。
- スタックショットコントローラーとマクロレールを使用してZ位置を制御します。
- カメラ、フラッシュ、電動ステージを頑丈な陽極酸化アルミニウム三脚に接続します。
- ポイントマウントされた各サンプルをユニバーサルステージジンバルに配置し、目がレンズの方を向くようにヘッドを向けます。鉗子で頭をそっと動かして、頭の位置を調整します。
注意: 大規模で迅速な調整は、誤って斬首される可能性があります。 - カメラをラップトップコンピューターにテザリングした状態で、ソフトウェアで撮影設定を調整します。倍率20倍で試料を撮影するには、フラッシュパワー1/32、シャッタースピード1/200、絞りF2.8、ISO400を設定します。オートフォーカス機能と手ぶれ補正機能がオフになっていることを確認します。
メモ: これらの設定は、最適なフラッシュ照明、シャッタースピード、および被写界深度のバランスを取ります。他の倍率やレンズの組み合わせに合わせて調整する必要があります。 - 結果の画像スタック(10〜50枚の画像)を保存する場所を目的のファイルフォルダに設定します。イメージに十分なストレージ容量を確保します (イメージあたり ~8.5 MB)。
- 自動距離モードのスタックショットコントロールユニットのフォーカススタック設定を調整します。
- ステップサイズを5μmに設定し、フォーカススタックの開始位置と停止位置を設定してステップ数を計算します。
- ライブビューモードで試料を表示し、カメラをオートシュートモードで表示して、開始位置と停止位置を特定します。
- 試験片の最も近い部分にピントが合うようにレールを動かし( 開始 位置を設定)、次に最も関心のある特徴がピントが合っている場所 (終了位置 を設定)に移動します。
- カメラを マニュアル撮影 モードに戻し、スタックショットコントロールユニットから画像取得を開始します。
注:画像取得時間は、試料のサイズによって異なります。大きな試料に必要な被写界深度が深いほど、画像スタックに含まれるスライスが多くなり、全体の取得時間が長くなります。 - 参照されているフォーカススタッキングソフトウェアでファイルを開きます。スタック イメージを生成するには、[ スタック] |Align & Stack All (PMax) (すべて揃えてスタック)
- 最終イメージをコンピュータのハードドライブに.tifファイルとして保存するには、[ ファイル] |出力画像を保存します。
注:積み重ねられた画像ファイルの解像度と画像化する標本の数によっては、画像のバックアップに大容量の外付けハードドライブ(1 TB)が必要になる場合があります。このプロトコルでは、スタックされた画像は圧縮される前にそれぞれ約 100 MB です。

図3:画像取得(A)画像取得装置と、次のようにラベル付けされた部品:a)デジタル一眼レフカメラ本体;b)望遠レンズ;c)20x APO顕微鏡対物レンズとアダプター。d)フラッシュ;e)レンズとドームディフューザー。f)スタックショットコントローラー、マクロレール、ロータリーステージ。g)ユニバーサルステージジンバル。h)三脚。(B)光拡散器が設置されたイメージング装置。(C)イメージング用の位置にマウントされた標本のクローズアップ。この図の拡大版を表示するには、ここをクリックしてください。
4.成人の目の面積を計算するためのFIJI分析ワークフロー
- 画像分析のために、ImageJ.net のウェブサイトからFIJIソフトウェア15 を入手してください。
- 分析用の画像は、目が中央に配置され、適切な照明と最小限の周辺ぼかしで位置合わせされている画像を選択し、カメラとの適切な位置合わせを示しています。
- 画像の縮尺を調整します。
- 500 μmに対応する20倍の倍率のスケールバー画像をダウンロードしてください。あるいは、画像取得時に、同じ設定で定規を撮影することもできます。FIJIソフトウェアで画像を開きます。
- スケールバーの長さを測定します(図4A)。 直線ツールを使用して 、ラインを正確にトレースします。 [分析] |測定します。このピクセル距離は 500μm に相当します(図4B)。
- ミクロンあたりのピクセル数を計算します。これを使用して、ピクセル測定値をマイクロメートル測定値に変換します。
- FIJIでスタックイメージファイルを開きます(図4C)。
- ツールバーから「 虫眼鏡 」を選択して、フォーカス領域を拡大します。目とすぐ近くの頭のキューティクルで画面いっぱいにしてみてください(図4D)。
- ツールバーから 「フリーハンド選択ツール」 を選択します。網膜領域の輪郭をできるだけ密接に描き、最も外側の眼球列の輪郭に沿って描きます(図4E)。選択範囲の一部を削除するには、 オプション ボタンを押したまま、削除するピクセルを選択します。選択に追加するには、 option ボタンと shift ボタンを押したまま、追加するピクセルを選択します。
- 面積を計算するには、[ 分析] | トップメニュー(図4F)から測定します。新しいウィンドウが表示され 、面積、平均、最小 、 および最大 パラメーターが表示されます。これらのデータをコピーしてスプレッドシートに貼り付け、ピクセル単位からマイクロメートル単位への変換と文書化を行います。
- 適切な統計分析を実行します。
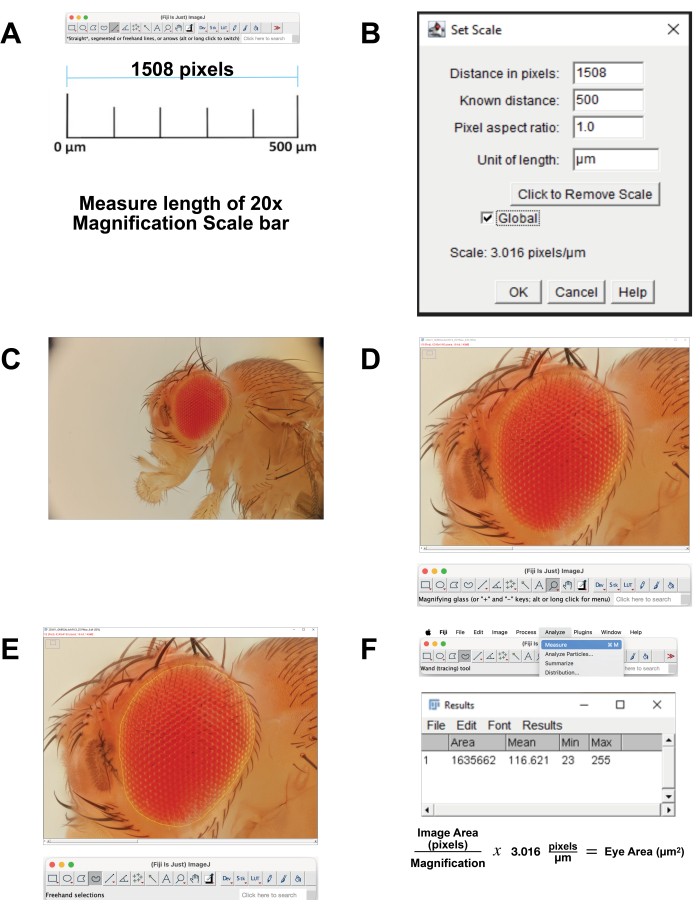
図4:FIJIでの画像分析 (A)元の画像をスケーリングします。キャリブレーション画像をダウンロードして、500 μmバーの長さを測定します。(B)スケールの設定機能を使用してスケーリングを調整します。(C)積み重ねた画像を開きます。(D)目が中央に集まり、ほぼ全画面表示になるように画像を拡大します。(E)フリーハンド選択ツールを使用して、オマチディアの最も外側の列と周囲のキューティクルとの間の境界で目の輪郭を描きます。(F)選択した領域内の面積を測定するには、[分析]をクリックします|メジャー |面積。この図の拡大版を表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
結果
ショウジョウバエの目は、組織のパターン形成、成長制御、および細胞死を研究するための優れたモデルシステムです。私たちは最近、細胞内pH(pHi)が組織の成長にどのように影響するかを調査した研究を発表しました。まず、発育中の眼におけるナトリウム-プロトン交換体DNhe2(哺乳類NHE1のオルソログ)の過剰発現がパターニング欠陥と増殖の増加を引?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここでは、成虫の ショウジョウバエ の構造のサンプル調製、高解像度イメージング、および分析の方法について説明します。 ショウジョウバエ の眼は、がん19、神経変性20 、代謝性疾患21などの疾患の根底にある分子メカニズムに重要な洞察をもたらした遺伝的に扱いやすいモデルシステムです。?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者には、開示すべき利益相反はありません。
謝辞
著者は、議論とサポートを提供してくれたGrillo-Hill pHlyラボのメンバーに感謝します。Tim Andriese、Randy Kirschner、Kitty (Ngoc-Huong) Nguyen、Marco Parent、Jonny Shaloub、Librado Veliz の優れた技術サポートに感謝します。この研究は、NIH SC3GM132049 and 1R16GM153640 awards (BKGH)、CSU Biotechnology Faculty-Student Research Award (LM and BKGH)、サンノゼ州立大学 (FJL) の理学部と生物科学部からのスタートアップ資金の支援を受けました。特に、このプロセスにおけるベルント・ベッカーの機知と支援には特筆すべき点があります。BioIcons(https://bioicons.com/)コミュニティに感謝します。特に、ピペットアイコンのServiere、図1と図2で使用されているショウジョウバエ、鉗子、卓上電子顕微鏡アイコンのDBCLSは、CC-BY 4.0 Unportedの下でライセンスされています。また、SciDraw(https://scidraw.io/)コミュニティ、特にクリエイティブ・コモンズ4.0ライセンス(CC-BY)の下でライセンスされているディオゴ・ロッシュ・デ・オリベイラ(doi.org/10.5281/zenodo.3925953)のフィギュアに高品質のアイコンを提供してくださったことに感謝します。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 1 mL serological pipette | ThermoFisher Scientific | 170353N | |
| 1.7 mL microcentrifuge tubes | Genesee Scientific | 24-282LR | |
| 20x Apo Microscope Objective | Mitutoyo Corp. | 378-804-3 | |
| Archival 65 lb cardstock | Neenah, Inc. | 91901 | |
| Canon EF 70-200 mm USM II telephoto lens | Canon | 3044C002 | |
| Canon EOS 6D Mark II DSLR Camera Body | Canon | 1897C002 | |
| Diffuser Dome | Macroscopic Solutions | PA-DIF-GIM-SM | |
| Diffuser for Mitutoyo M Plan APO Objectives | Macroscopic Solutions | mitutoyo-diffusers | |
| Drosophila vials and plugs | Genesee Scientific | 32-117BF | |
| Dumont #5 fine-tip forceps | Fisher Scientific | NC9889584 | |
| Goose feathers | Amazon | B01CMMJI6U | |
| Heavy-Duty Anodized Aluminum Tripod | Really Right Stuff, LLC | TFA-32G | |
| Kimwipes | Fisher Scientific | 06-666A | lint-free lab tissue |
| Levenhuk M1000 Plus Digital Camera | Levenhuk | 70358 | |
| No. 3 mounting pin | Indigo Instruments | 33414-3 | |
| Nutri-Fly Bloomington Drosophila media | Genesee Scientific | 66-113 | fly food |
| Point-Punch | M.C. Mieth Manufacturing, Inc. | 448Detail | |
| Screwknob Clamp | Really Right Stuff, LLC | SK-Clamp | For attaching the macro rail to the tripod |
| Stackshot Controller and Macro Rail | Cognisys Inc. | ST3X_100_BUNDLE | |
| Step-down Ring Adapter | RAF Camera | 763461174207 | Lens adapter to connect the microscope objective to the camera lens |
| Titebond Glue | Franklin International | 5013 | |
| Yongnuo YN-24-EX Macro Twin Lite Flash | Shenzhen Yongnuo Photographic Equipment Co. | YN-24EX | |
| Software | |||
| Canon EOS Utility (v. 3.16.1). | Canon | acquisition software | |
| FIJI | National Institutes of Health | Fiji is released as open source under the GNU General Public License. FIJI Version 2.14.0/1.54f | |
| GraphPad Prism | GraphPad Software, Boston, Massachusetts USA | Prism Version 10.3.1 | |
| Zerene Stacker (v.1.04) | Zerene Systems, LLC | Focus Stacking Software |
参考文献
- Rubin, G. M., et al. Comparative genomics of the eukaryotes. Science. 287 (5461), 2204-2215 (2000).
- Ugur, B., Chen, K., Bellen, H. J. Drosophila tools and assays for the study of human diseases. Dis Model Mech. 9 (3), 235-244 (2016).
- Hodgson, J. A., Parvy, J. -P., Yu, Y., Vidal, M., Cordero, J. B. Drosophila larval models of invasive tumorigenesis for in vivo studies on tumour/peripheral host tssue interactions during cancer cachexia. Int J Mol Sci. 22 (15), 8317(2021).
- Lam Wong, K. K., Verheyen, E. M. Metabolic reprogramming in cancer: mechanistic insights from Drosophila. Dis Model Mech. 14 (7), dmm048934(2021).
- Bonini, N. M. A perspective on Drosophila genetics and its insight into human neurodegenerative disease. Front Mol Biosci. 9, e1060796(2022).
- Drummond-Barbosa, D., Tennessen, J. M. Reclaiming Warburg: using developmental biology to gain insight into human metabolic diseases. Development. 147 (11), dev189340(2020).
- Ready, D. F., Hanson, T. E., Benzer, S. Development of the Drosophila retina, a neurocrystalline lattice. Dev Biol. 53 (2), 217-240 (1976).
- Johnson, R. I. Hexagonal patterning of the Drosophila eye. Dev Biol. 478, 173-182 (2021).
- Weasner, B. P., Kumar, J. P. The early history of the eye-antennal disc of Drosophila melanogaster. Genetics. 221 (1), iyac041(2022).
- Pichaud, F., Casares, F. Shaping an optical dome: The size and shape of the insect compound eye. Semin Cell Dev Biol. 130, 37-44 (2022).
- Oster, I. I., Crang, R. E. Scanning electron microscopy of Drosophila mutant and wild type eyes. Trans Am Microsc Soc. 91 (4), 600-602 (1972).
- Gibb, T. J., Oseto, C. Insect collection and identification: Techniques for the field and laboratory. , Academic Press. (2019).
- Mertens, J., Roie, M. V., Merckx, J., Dekoninck, W. The use of low cost compact cameras with focus stacking functionality in entomological digitization projects. ZooKeys. 712, 141-154 (2017).
- Brown, B. V. A further chemical alternative to critical-point-drying for preparing small (or large) flies. Fly Times. 11, 10(1993).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Method. 9 (7), 676-682 (2012).
- Grillo-Hill, B. K., Choi, C., Jimenez-Vidal, M., Barber, D. L. Increased H+ efflux is sufficient to induce dysplasia and necessary for viability with oncogene expression. eLife. 4, e03270(2015).
- White, K. A., Grillo-Hill, B. K., Barber, D. L. Cancer cell behaviors mediated by dysregulated pH dynamics at a glance. J Cell Sci. 130 (4), 663-669 (2017).
- Peralta, J., et al. Drosophila Nhe2 overexpression induces autophagic cell death. Mol Biol Cell. 35 (7), br13(2024).
- Munnik, C., Xaba, M. P., Malindisa, S. T., Russell, B. L., Sooklal, S. A. Drosophila melanogaster: A platform for anticancer drug discovery and personalized therapies. Front Genet. 13, 949241(2022).
- Nitta, Y., Sugie, A. Studies of neurodegenerative diseases using Drosophila and the development of novel approaches for their analysis. Fly. 16 (1), 275-298 (2022).
- Pletcher, R. C., et al. A genetic screen using the Drosophila melanogaster TRiP RNAi collection to identify metabolic enzymes required for eye development. G3: Genes|Genomes|Genetics. 9 (7), 2061-2070 (2019).
- Bangi, E., et al. A personalized platform identifies trametinib plus zoledronate for a patient with KRAS-mutant metastatic colorectal cancer. Sci Adv. 5 (5), eaav6528(2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved