Method Article
使用等离子体纳米镊子监测单个未修饰蛋白质的构象动力学
摘要
等离子体镊子利用金纳米结构中的局部表面等离子体共振,将单个纳米颗粒(包括蛋白质)捕获在纳米级光场中。散射信号的变化揭示了蛋白质的存在和构象动力学,无需荧光团修饰或表面栓系即可进行监测。
摘要
当前表征蛋白质的单分子技术通常需要标记、系绳或使用非天然溶液条件。这些变化会改变蛋白质生物物理学并降低所获取数据的有用性。等离子体镊子是一种在金纳米结构上使用局部表面等离子体共振 (LSPR) 来增强受限热点区域内的电场的技术。这种场增强允许使用低激光功率来捕获比传统光镊小得多的单个纳米颗粒,直径小至几纳米,例如单个蛋白质。在热点区域捕获单个蛋白质分子会引起局部折射率(n蛋白质 > n水)的变化,从而改变作为分子极化率乘积的光散射,而极化率受其体积、形状各向异性和折射率的影响。雪崩光电二极管 (APD) 收集光散射的后续变化。然后可以分析这些变化以确定捕获分子的变化,包括其大小、全局构象和构象随时间变化的动力学。在系统中加入微流体技术可以控制环境变化并实时监测它们对分子的后续影响。在该协议中,我们演示了使用等离子体 nanotweezers 系统捕获单个蛋白质分子、改变其环境溶液条件并监测其相应构象变化的步骤。
引言
目前用于研究蛋白质构象动力学的单分子技术包括基于标记的方法,如荧光共振能量转移 (FRET)1,2,基于束缚的方法,如光镊 3,4 和原子力显微镜 (AFM)5,基于干扰的技术,如干涉散射显微镜 (iSCAT)6,或基于纳米流体的技术,如纳米孔7,8,9.虽然这些方法有很多优点;一些关键限制使他们无法提供有关未修饰蛋白质构象动力学的数据。FRET 和光镊需要荧光团标记或拴在表面上,这可能会影响蛋白质的生物物理特性 10,11,12。iSCAT 虽然在技术上是无标记的,但也需要蛋白质和表面之间的相互作用,以观察两者之间产生的干扰,这可能会影响蛋白质的特性。此外,受信噪比的限制,由于设备噪声和类似斑点的背景波动,iSCAT 只能检测 >40 kDa 的蛋白质13。虽然可以通过机器学习来缓解这种大小限制,但缓冲区组件是有限的,因为它们会影响光学特性,从而导致数据噪声13,14。纳米孔呈现蛋白质通过孔的快速易位时间(通常在 5 μs 内),使它们无法检测较慢的构象动力学 15,16,尽管研究减轻了这些限制,例如在纳米孔电渗透阱中使用 DNA 折纸17 或掺入等离子体 18,19,20,21.此外,高盐浓度(通常在 1 M 左右)会降低数据对体内工作的适用性15,22。用于蛋白质表征的理想单分子技术应实时监测蛋白质并在较长时间(即毫秒)内捕获构象动力学,而无需修改蛋白质或非天然溶液条件。
等离子体镊子类似于传统的光镊,因为它们利用光来捕获物质。然而,等离子体镊子利用局部表面等离子体共振 (LSPR) 将电场增强几个数量级,以产生足够强的梯度力来捕获单个纳米颗粒23。此外,被捕获的颗粒在增强捕获的强度方面起着积极作用,称为纳米孔径结构的自诱导反作用 (SIBA) 捕获24。这种 SIBA 捕获允许低激光功率(即毫瓦)捕获直径只有几纳米的小颗粒,例如蛋白质 25,26,27。将单个蛋白质分子捕获在热点区域会导致局部折射率(n蛋白质> n水)发生变化,从而根据分子的极化率改变光散射,该偏振性受蛋白质体积、形状各向异性和折射率的影响28。然后,雪崩光电二极管 (APD) 检测到此信息,以监测光散射的后续变化。此外,等离子体镊子可以长时间(即几分钟到几小时)实时监测被捕获的蛋白质,而无需标记、系绳和恶劣的溶液条件29,满足理想的蛋白质单分子技术的标准。使用双纳米孔 (DNH) 结构,等离子体镊子已经证明它们能够捕获各种蛋白质并从中阐明关键信息,包括构象转变29、拆解动力学30、能量景观31、扩散跟踪32 和配体结合33,34。除了 DNH 结构外,替代结构几何形状已被证明可以捕获小粒径的颗粒 35,36。在该协议中,介绍了使用集成微流体系统设置和运行等离子体 nanotweezers 设置的基本步骤。我们希望该协议将有助于增加研究人员对等离子体镊子的可及性和理解,尤其是结构生物学和生物物理学领域的研究人员。
研究方案
注意: 请阅读所有所用化学品的所有相关安全数据表 (SDS),并遵守所有适当的安全实践,并根据需要佩戴个人防护设备(激光护目镜、实验室外套、手套)。
1. 构建等离子体镊子装置
注意:光学设置基于模块化光镊系统 (OTKB) 套件,使用不同的激光器和 APD(参见 材料表)。仅在合适的光学平台上使用光学设备,以减少外部振动对系统的影响。试剂盒中的激光为 976 nm,但由于 DNH 结构楔形共振的峰值共振波长约为 740-760 nm,因此 33。我们选择了 NIR 激光器 (852 nm),因为它接近共振峰,诱导 LSPR,并且硅基 APD 也具有更好的检测良率。具有较长20 或较短18 波长的激光器已被用于捕获生物分子。
- 在光电二极管激光支架中设置激光器并将其送入准直器,从而产生宽度为 1.7 mm 的准直激光束,用于我们的设置。
- 在光路上添加一个半波片以调整偏振。使用 Glan-Taylor 偏振器进行调整,以确保垂直偏振(S 偏振)具有最高的光强度。
注意:使用正确的极化对于确保 DNH 结构的最大电场增强至关重要,因为它们依赖于极化。 - 通过扩束器设置聚焦光线,该扩束器设置由一个平凹透镜 (f = -50 mm) 和一个平凸透镜 (f = 150 mm) 组成,以将光束宽度增加到 5 mm 左右,以填充底部物镜的整个后孔径。
- 使用二向色镜(805 nm 短通)将激光反射到所需位置。设置其后面的相机 (CCD)。
- 将光线重定向到底部物镜 (100x/1.25 NA),并将顶部物镜 (4x/0.1 NA) 定位到共焦距离以收集透射光。
- 添加另一个二向色镜(805 nm 短通),通过位于其焦距的平凸透镜将激光反射到 APD,以将光聚焦到 APD 中。在二向色镜后面添加白光源 (WLS),以便它可以穿过设置返回到达相机。 图 1 显示了完全组装的等离子体 nanotweezer 装置的示意图。
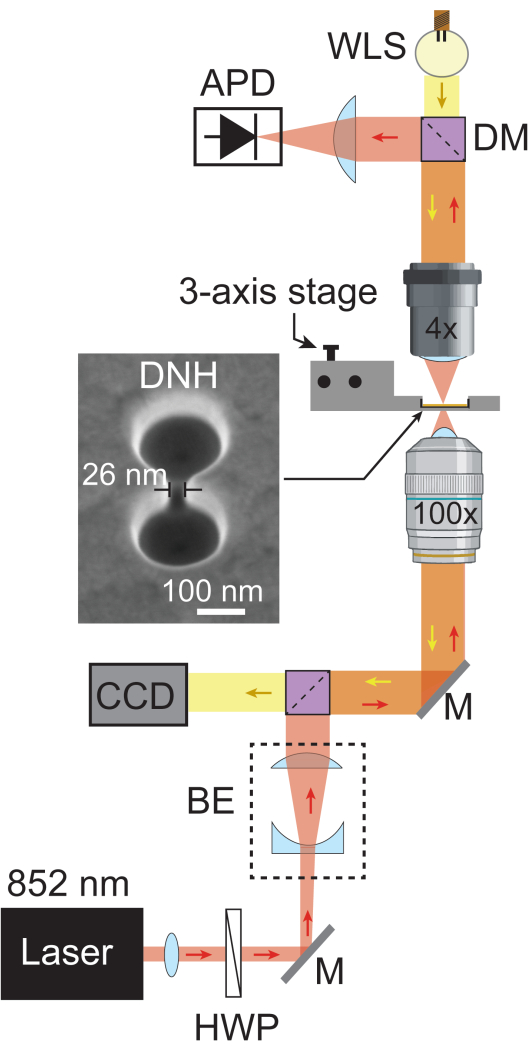
图 1:等离子体镊子设置。 该示意图显示了等离子体镊子装置的完整光路。852 nm 激光束(红色)穿过准直器和半波片,然后通过扩束器 (BE) 扩展到 ~5.1 mm。随后使用 100 倍物镜聚焦在样品上。透射的激光由 4 倍物镜收集,并由 APD 以 1 MHz 的采样率记录。来自 WLS(黄色)的白光在到达 CCD 之前穿过 4 倍物镜、样品和 100 倍物镜,CCD 提供样品的明场图像。两条光路相交的区域以橙色表示。以 20° 倾斜拍摄的 DNH 的 SEM 图像。缩写:BE = 扩束器,CCD = 电荷耦合器件,DM = 二向色镜,HWP = 半波片,M = 银镜,WLS = 白光源。 请单击此处查看此图的较大版本。
2. 制造 DNH 结构
- 如前所述,使用电子束蒸发在 550 μm 厚的熔融石英晶圆上沉积 5 nm Ti,然后沉积 100 nm Au29,33。将晶圆切成 1 cm x 1 cm 的样品,即可使用。
注:选择电子束蒸发是因为它会产生表面粗糙度低的金层,从而产生具有高捕获效率的高质量 DNH 结构37。 - 取空白金膜样品,在室温下使用镓离子源将其安装到扫描电子显微镜聚焦离子束 (SEM-FIB) 装置中。
- 确保 FIB 纳米孔径与 SEM 对齐,以在准确位置产生 DNH。
- 使用大电流离子束(例如 100 pA)创建标记框。该标记将用作参考指南,以定位光学设置下 DNH 结构的位置。
- 为了创建 DNH 结构,两个中心到中心距离为 200 nm、直径为 160 nm 的圆由矩形 (3 nm x 40 nm) 桥接。对于大约 20-30 nm 的间隙尺寸,使用 30 kV 的探针能量和 1 pA 的束流,以 0.09 的剂量因子蚀刻圆圈,而矩形剂量在 0.300 至 0.350 之间。
注意:在此探针能量下,使用的 SEM-FIB 具有 3 nm 的分辨率,可以可靠地制造间隙尺寸在 20-30 nm 之间的 DNH。我们不会使用较低的分辨率,因为这会产生更大的间隙尺寸,从而降低 DNH 的捕获效率。如果 FIB 不可行,则生产 DNH 结构的另一种方法是使用聚苯乙烯纳米球光刻38。由于制造 DNH 结构需要高精度,因此相同的配方在 SEM-FIB 使用之间可能无法产生相同的间隙尺寸。改变矩形的剂量和高度(0.280 至 0.350 剂量和 2 至 4 nm 高度)并测量间隙大小,目标是 20 nm 左右。
3. DNH 样品的涂层
- 使用耐溶剂容器,例如由硼硅酸盐玻璃 3.3 制成的结晶皿,并加入 20 mL 乙醇。
注意: 乙醇高度易燃且具有刺激性。仅在通风橱中使用。贮存于阴凉、通风良好的地方。戴手套和穿上实验室外套。 - 称取 32 mg 聚乙二醇)甲醚硫醇(PEG-硫醇;平均分子量 800 g/mol)并将其混合到乙醇中,确保所有 PEG-硫醇都已溶解,产生浓度约为 2 mM 的溶液,以最大限度地提高单层密度39。
注意:PEG-硫醇是一种刺激物;使用适当的个人防护装备处理。 - 使用直镊子将含有纳米结构的样品芯片加入混合物中,盖上盖子,并在室温下孵育过夜(~16 小时),使 PEG-硫醇在金表面形成自组装的单层。
- 孵育后,用直镊子将样品放在通风橱中合适的容器上冲洗样品。使用喷瓶将乙醇彻底喷洒在两侧。在随后储存或安装在等离子体镊子中之前,使用气枪充分干燥。
注:样品可以储存在 4 °C 下以提高使用寿命,因为它们会随着时间的推移而降解。
4. 将 PEG 涂层样品安装到流通池中
- 使用金层朝上的直镊子将涂层样品放入 3D 打印的流通池(由 Form 2 打印机使用 Clear V4 树脂打印)中(参见 补充图 1 ,了解流通池设计中的关键参数和值)。
- 使用直镊子撕下透明 PET 塑料双面胶带盖的一侧,并将其放在样品和流通池上,确保流通池中的纳米结构和进/出孔保持未覆盖状态。用圆镊轻轻按压胶带的边缘,以确保其正确粘附在流通池和样品上。
- 撕下胶带的另一面,然后用圆镊子轻轻地将玻璃盖玻片(厚度 0.17 mm)放在样品上。用圆镊子轻轻按压盖玻片的边缘,以确保其正确粘附。这会在流通池内形成一个液体通道(高度 = 50 μm,体积 = 3.5 μL)。
注意:请勿在纳米结构所在的位置或附近按压盖玻片,因为它们可能会损坏。如果盖玻片脏了,请用乙醇清洗并在安装前用气枪干燥。 - 使用小移液器吸头将等量的复制硅胶溶液 A 和 B 以 1:1 的比例(或根据制造商的说明)混合到显微镜载玻片上。
- 用混合复制的硅胶填充盖玻片和流通池之间的间隙,轻轻将其推到盖玻片下方。将流通池倒置,小心地将复制的硅胶放在孔的内壁周围,然后使用移液器吸头轻轻将其移动到样品底面熔融石英的可见边缘上(参见 补充图 2)。
- 根据制造商的说明,将金层朝上晾干,直到复制的硅胶完全凝固。
注意:当应用于样品的底面时,注意不要让硅胶覆盖纳米结构。注意不要将复制的硅胶强行置于盖玻片下,因为它可能会进入流通池的进/出孔或难以清洁的纳米结构(补充图 3)。
5. 连接微流体系统
注意:确保为系统使用干净的管路。这里将使用小内径 PTFE 管:内径 0.18 mm 和大内径 PTFE 管:0.8 mm,但其他内径值也适用。
- 设置注射泵并使用大型 PTFE 管将其连接到 3/2 通电磁阀。将阀的一个端口连接到缓冲容器,另一个端口连接到体积约为 1 mL 的固定线圈,该线圈由大 ID 管制成,以防止回流到注射器中。
注意:3/2 阀可以通过阀门控制器进行控制,该控制器确定溶液是从缓冲容器还是从保持线圈中注入/提取。 - 将保持线圈连接到 12/1 旋转双向微流体阀的中心阀。连接溶液容器,使用小内径管连接稀缺或有价值的样品,使用大内径管连接其余样品。使用一个阀仅用于进样到废液容器中,另一个阀仅用于进样到流通池中;使用小内径管路连接流通池输液阀。
- 将大内径管线从流通池的输出端连接到废液容器。 图 2 显示了全连接微流体系统的示意图。
注:小内径 PTFE 管路更适合用于流通池的进气口和用于蛋白质输注的阀,因为样品留在管路中的体积较小。大内径 PTFE 管更适用于更便宜或更丰富的材料,例如缓冲液。尝试使用最少的管路来进一步减少死体积。
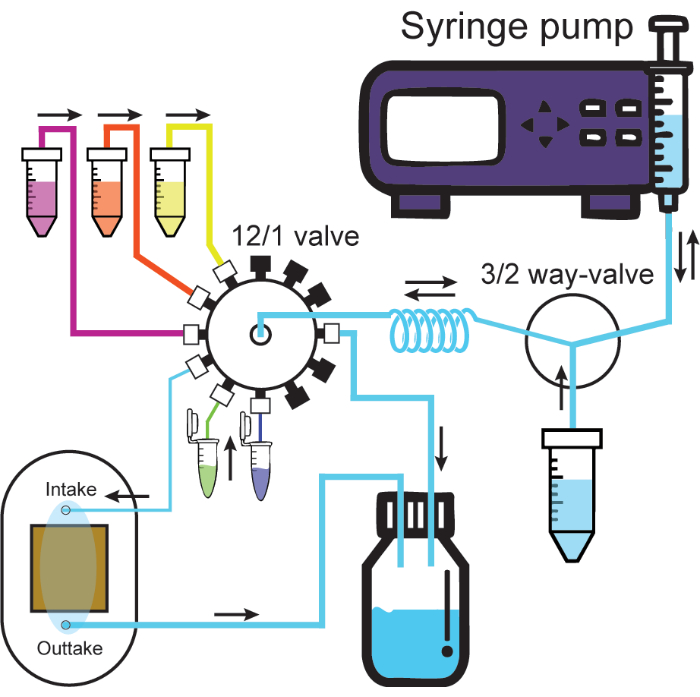
图 2:微流体系统。 示意图说明了微流体系统。注射泵通过两位三通阀的一个端口(溶液容器或保持线圈)注入或抽出溶液。连接到 12/1 阀的溶液在抽出时始终通过保持线圈,然后可以通过所需的通道注入。向连接到流通池的进样通道注入会将溶液从流通池推入废液容器中。粗管和细管代表 protocol 中大管和小 ID 管。12/1 阀上的黑色盖子代表密封通道。流向由黑色箭头标记。 请单击此处查看此图的较大版本。
6. 准备微流体系统
- 在 PC 上加载微流体系统 UI 并检查组件是否已正确连接。选择 wire 和 distributor 旁边的播放图标以打开它们各自的 UI。要绕过 3/2 通阀,请转动电线上的端口 1。
- 通过注入异丙醇 (IPA) 确保清洁系统内将使用的阀门,然后用所选缓冲液或蒸馏水多次清洗管路,以去除系统内的空气和 IPA。可以通过在分配器 UI 上选择所需的阀门并运行注射泵,选择注入/抽出,然后选择所需的体积和流速(例如,0.5 mL 体积和 0.2 mL/min 流速)来完成输注。
注:请勿将 IPA 注入流通池,因为这会降解流通池和胶水,从而导致泄漏。
7. 将流通池安装到等离子体镊中并检查是否有泄漏
- 将入口管和出口管连接到流通池的适当部分,然后将它们放在干净的组织上,金层朝上。
- 以高流速 (~0.3 mL/min) 将缓冲液注入流通池中,检查液体是否在流通池中的样品中移动,以及流通池外部或底部是否看不到液体。在不发生泄漏的情况下,可以使用更高的流速。如果样品渗漏,请卸载并重新挂载。
- 在 100 倍物镜上加入 1-2 滴浸油,然后将流通池放入等离子体 nanotweezers 载物台中,金层朝下。将金属夹放在流通池磁铁上并锁定载物台以帮助将其固定到位。
注意:客观油具有刺激性、健康危害性,并且对水生生物有毒,只能戴手套处理。
8. 在样品上定位纳米结构
- 打开白色光源。打开相机软件并增加增益和曝光时间,直到纳米结构可见。打开激光功率相对较高的激光器,手动调整 z 轴,直到看到激光光斑。优先增加曝光时间和增益,而不是增加激光功率以找到激光点。
注意: 激光对用户构成严重的潜在风险。确保使用适当的个人防护装备,例如在所需波长范围内具有足够光密度的激光护目镜。 - 打开压电控制器,并为 COM 端口和最大电压选择适当的设置。将 x、y 和 z 轴的值设置为最大电压的一半,以允许在各个方向上对齐载物台的良好范围。
注意: 压电控制器的 COM 端口可以在设备管理器的 ports 选项卡下找到和更改。 - 使用主载物台 x、y 和 z 轴控制旋钮移动激光光斑以与其中一个 DNH 叠加。确保 APD 已打开,然后轻轻地将外壳关闭到等离子体镊子上。
注意:使用相机软件中的标记工具(如果可用)将有助于在纳米结构上移动激光点。将标记设置为激光光斑的中心并关闭激光,但保持白色光源打开,以便更容易对准纳米结构。如果过多的激光到达 APD ,APD 可能会过度饱和并损坏。确保样品挡住激光路径,并逐渐增加激光功率以确保不会达到饱和。如有必要,可以在 APD 之前放置中性密度 (N.D) 滤光片,以减少到达它的光线。
9. 将激光器与所需的纳米结构最佳对准
- 打开与 APD 记录相关的软件,例如自制的 Labview UI,并将截止频率设置为 1 kHz。命名并设置所需的文件路径和将要保存的文件的命名格式。
- 关闭白色光源,然后重新打开激光。设置为适当的激光功率(例如,~20 mW),并使用压电控制调整 x、y 和 z 轴,直到 APD 信号尽可能高,避免 APD 饱和,并且迹线的标准偏差 (S.D.) 最小。
注意: APD 可能具有最佳的传输灵敏度范围(参见产品手册),非常适合保持,例如 1000-2000 mV。可以使用 N.D. 滤波器将透射率保持在该范围内。主要考虑因素是传输不要接近 APD 饱和点,因为捕获可能会将传输增加到饱和限制,从而导致数据丢失。
10. 将蛋白质注入流动槽
- 关闭激光器以保持纳米结构的使用寿命,并运行注射泵控制单元和分配器 UI 以设置阀门并提取所需量的蛋白质(例如,30 μL)。使用小内径管进行萃取时,较低的流速有助于确保所有溶液均被萃取 (~0.01 - 0.1 mL/min)。如果需要,可以使用更高的流速来减少等待时间。
注:最佳蛋白质体积取决于蛋白质的价值以及实验希望达到的目标。我们使用的典型蛋白质浓度为 1 μM,等分试样体积为 100 μL。如果蛋白质样品丰富,可以使用更高的浓度来缩短平均捕获时间。然而,如果浓度过高,在纳米结构中捕获两种相同蛋白质的风险将增加。如果实验在捕获初始蛋白质后需要输注不同的配体/蛋白质,则可能首选较低的等分试样体积,以最大程度地减少浪费和输注下一种溶液所需的时间。确保管路浸没在溶液中,以防止空气进入系统。如果管道未到达容器底部或溶液过度抽出,则应特别注意蛋白质等分试样。 - 使用微流体 UI,以与取出速率相似的流速 (~0.01 - 0.1 mL/min) 注入蛋白质溶液。以此速率进样,直到蛋白质溶液到达流通池,然后将流速降至 ≤0.001 mL/min。检查注射泵的体积和时间,以确保它们与预期体积匹配(例如,1 mL/min 的 1 mL 体积需要 1 分钟)。
注:所需的体积可以根据从微流体系统到流通池的管道体积来计算。如果知道管子的长度和内径,则可以使用圆柱体体积的在线计算器。输注速率可根据需要进行调整,例如,蛋白质输注的输注速度为 >0.001 mL/min,以减少捕集阱的平均等待时间。但是,建议谨慎,因为如果流速太高,这可能会阻止蛋白质进入纳米结构。
11. 收集数据
- 当蛋白质溶液注入流通池时,开始 APD 信号的数据记录。使用压电控制器 UI 根据需要调整 x、y 和 z 轴,因为系统可能会随着时间的推移而漂移。理想的陷印迹线具有与 图 3 中的迹线相同的一般模式。
- 在观察到传输和 S.D. 的巨大变化类似于示例捕获轨迹时,记下这种情况发生的时间以备将来的数据排序(例如,参见 图 4B )。
注意:系统中的漂移会导致传输发生变化和 SD 增加,有时非常突然,这可能会被误认为是蛋白质陷阱。确保信号跳变显示与示例陷阱相同的模式,并且在发生跳变时不存在外部干扰,例如对齐漂移或系统拾取的巨响。 - 如果需要在实验中释放蛋白质,请关闭激光器 ~5 秒,然后重新打开。迹线的传输变化应较大,S.D. 应显著降低,表明已返回到基线状态。
注:如果未观察到透射率和/或 SD 的较大变化,或者观察到与被捕获的蛋白质的类似痕迹,则蛋白质可能粘附在样品表面(例如,参见 图 5B)。
12. 卸载样品
- 执行所需的实验后,关闭激光器,将流通池从 3 轴载物台中取出,并断开微流体系统管道。
- 将流通池放在干净的纸巾上,样品的金层朝上。使用手术刀小心地切开玻璃盖玻片下的胶水,然后用圆镊子轻轻将其提起。将其丢弃在指定的碎玻璃箱中。
注意: 碎玻璃和使用尖锐物体可能会造成伤害。确保佩戴护目镜、手套和实验服。 - 以一定角度握住流通池,使金层仍然朝上,并使用圆镊子小心地去除流通池底部的胶水,以释放样品。复制硅胶可轻松去除,而不会损坏 DNH 结构。
- 使用直镊子吸取样品并用 IPA 彻底冲洗,然后加水,然后用气枪干燥。如果可重复使用,请将样品储存在合适的容器中,金层朝上。
注意:如果处理得当,样品的可用时间通常约为 1-2 周,尽管纳米结构的大小会随着时间的推移而变化。如果损坏严重,请改用新样品进行下一次实验。损坏可能以纳米结构附近金表面上存在的划痕或实验性能不佳的形式出现,表明纳米结构退化29。
13. 准备系统以备将来使用
- 使用折叠的物镜纸沿一个方向擦去物镜上的油,将其掀开并用纸巾的另一个干净部分重复此作。
注意:物镜容易出现污迹和划痕,这会严重影响性能。清洁时始终戴手套,避免接触有机材料。仅使用新的镜头清洁纸以防止污染,并只需一个动作即可沿单个方向清洁。避免摩擦镜头和来回清洁纸巾,以尽量减少损坏。如果干洗不充分,请在纸巾上涂抹少量乙醇,如前所述擦去油,最后用干纸巾完成。 - 如果接下来将使用相同的蛋白质/缓冲液,则可能不需要更换管路。如果使用不同的溶液,请更换管路,以降低先前溶液污染的风险。
注意:即使使用相同的溶液进行实验,也不要使用相同的管路超过 1 周,因为它会随着时间的推移而变脏。
结果
数据采集后,可以使用 MATLAB 代码对原始数据进行数据分析,以从 APD 收集的原始数据中生成跟踪。 图 3 描述了一个示例捕获轨迹,包括捕获前的基线,在激光器关闭约 5 秒后重新打开之前观察到透射率 (ΔT/T0) 和标准偏差发生较大变化的捕获事件。标准差的显着降低和传播恢复到与基线相似的水平表明蛋白质的释放。使用 MATLAB 函数 detrend.m 从迹线中删除线性漂移,然后将数据的平均值添加回去除趋势的迹线。有时,随着设置随时间漂移,我们需要对迹线进行去趋势处理,从而导致透射率线性下降(参见 图 3 中的灰色迹线)。捕获前后基线迹线的微小变化是由于阶段调整以最小的标准偏差优化基线,如图 4A 所示。有时,蛋白质分子在痕量中可见而未被捕获,称为路过蛋白。经过的蛋白质表现为传递的急剧变化,类似于典型的陷阱(图 4B),但持续时间明显更短,如图 4A 所示。功率谱密度 (PSD) 是另一种分析方法,通过提供不同频率的信号强度来确认蛋白质捕获。通过单分子光谱方法,蛋白质构象运动通常在 >1 μs 范围内出现40。 图 4C 表明,与基线相比,捕获蛋白质会导致更高的信号强度,至少在 10 kHz 范围内 (> 100 μs)。它还强调了将载物台与优化基线对齐的重要性,因为不良基线可能会增加 50-500 Hz 频率的噪声,该频率范围与蛋白质构象运动重叠。
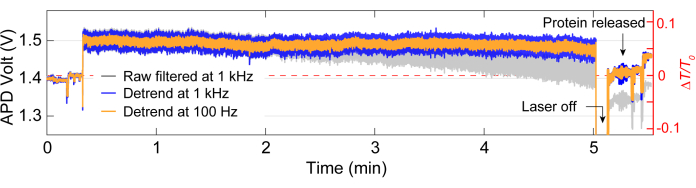
图 3:单个蛋白质的完整捕获轨迹。 完整捕获的代表性示踪,包括基线、捕获蛋白质和蛋白质释放。陷印前后的跟踪跳转是由于对齐造成的。 请单击此处查看此图的较大版本。
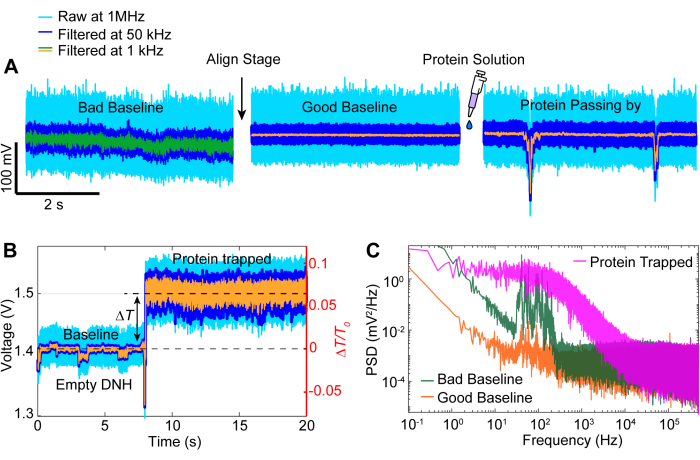
图 4:常见跟踪事件。(A) 从不良基线到良好基线的比对以及蛋白质靠近热点的示例。(B) 捕获曲线,显示了从 DNH 热点为空到蛋白质被捕获的基线过程。(C) (A) 中描述的良好和不良基线与 (B) 中捕获的蛋白质之间的功率谱密度 (PSD) 图。PSD 值越高,表示特定频率下的噪声越大。请单击此处查看此图的较大版本。
大多数捕获事件遵循与图 3 中的迹线相同的一般模式,但在实验过程中偶尔会出现问题。对于大多数实验,一旦所需的实验完成,应通过关闭激光器来手动释放蛋白质。然而,在某些情况下,蛋白质可以在不干预的情况下离开陷阱,如图 5A 所示。相反,有时即使在关闭激光后,蛋白质仍会留在捕获部位,这可能是由于蛋白质粘附在样品上。这种粘附会导致在关闭和打开激光器后产生嘈杂的痕迹(参见图 5B)。发生这种情况的可能性取决于蛋白质,因为某些蛋白质更容易被表面吸附41,42。使用 PEG-硫醇等涂层可以减少蛋白质粘附的机会39,43。除非有需要,例如研究蛋白质-蛋白质相互作用,否则另一个问题是双重捕获,即第二个蛋白质在第一个捕获之后被捕获。其特征是传播再次急剧增加,类似于第一个陷阱,并且标准偏差发生变化(参见图 5C)。
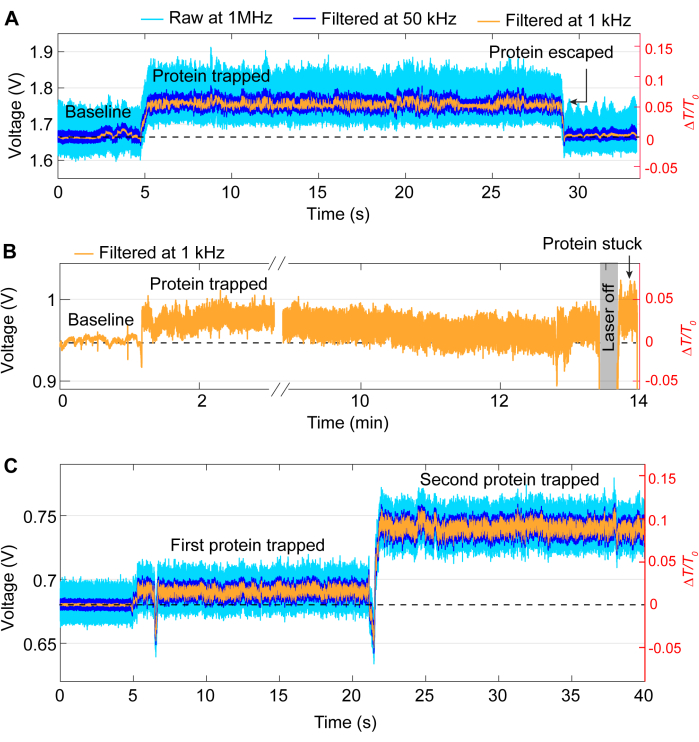
图 5:不良陷印事件的示例。(A) 蛋白质从 DNH 热点意外释放。 (B) 蛋白质在 DNH 热点处卡在样品表面的示例。 (C) 当第二种蛋白质被捕获,而第一种蛋白质仍保留在 DNH 热点中时,就会发生痕量跳跃。 请单击此处查看此图的较大版本。
对载脂铁蛋白分子的原位铁负载进行的一项代表性实验表明,使用等离子体镊子作为研究蛋白质构象动力学的工具29。铁蛋白是一种铁载体蛋白,以两种状态存在:不含铁的载脂铁蛋白和充满铁44,45 的全铁蛋白。亚铁通过 3 重通道进入蛋白质,在那里被氧化成三价铁并储存在蛋白质核心46 中。图 6A 描述了载脂铁蛋白的典型捕获痕迹,在蛋白质被捕获时,用亚铁溶液注入超过 20 分钟。沿整个轨迹在 b-e 点采集的 20 秒轨迹提供了对蛋白质随时间发生的变化的见解。在图 6B 中,载脂铁蛋白被捕获在标准 PBS 缓冲液中,在迹线中未观察到显着变化。图 6C、D 显示了迹线 S.D 的波动,这是由铁通过其 3 倍通道加载到蛋白质中引起的,导致通道打开时更动态的状态 (apo-),通道关闭时更紧凑的状态 (holo-)。铁蛋白分子充满铁后,它转变为全息形式,从而产生稳定的捕获痕迹,如图 6E 所示。图 6B-E 中的概率密度函数 (PDF) 进一步展示了蛋白质在暴露于不同溶液条件下随时间的变化。
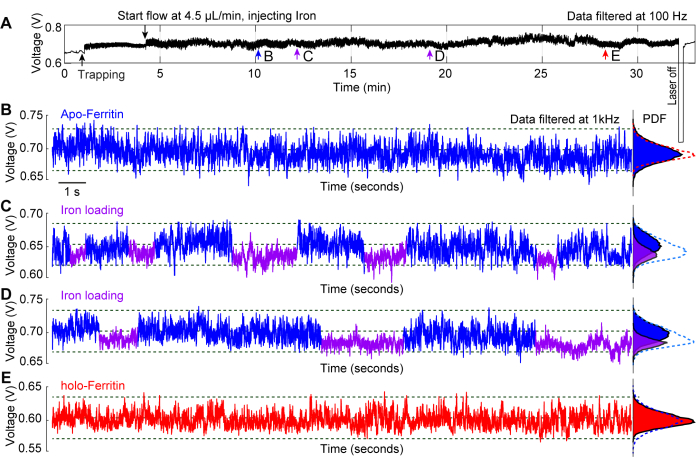
图 6:原 位 铁加载到捕获的脱铁蛋白中。 (A) 捕获了脱铁蛋白分子的 DNH 的完整传输轨迹,然后将亚铁溶液注入捕获位点,以观察铁蛋白与铁负载相关的构象变化。 (B) 在亚铁溶液到达热点之前被捕获的 apoferritin 的 20 秒捕获痕迹。 (C, D) 脱铁蛋白分子暴露于亚铁溶液后的 20 秒捕获痕迹。蓝色和紫色片段标记轨迹的较高和较低 SD,分别表示铁蛋白的柔性和刚性构象。 (E) 脱铁蛋白暴露于亚铁溶液 >20 分钟后的 20 秒捕获痕迹。右侧的概率密度函数 (PDF) 图显示了传输的分布,并用颜色编码为蓝色和紫色段。这个数字是从29 个修改而来的。 请单击此处查看此图的较大版本。
补充图 1:安装在 3D 打印流通池上的金 DNH 样品。 将样品放入特殊槽中,并使用双面 PET 胶带粘附在流通池上。我们流通池设计的关键参数和相关测量值已标记。 请点击此处下载此文件。
补充图 2:流通池背面,镶有金 DNH 样品并贴有内壁标签。 使用复制硅胶将样品密封在流通池中。 请点击此处下载此文件。
补充图 3:安装了金 DNH 的流通池示意图,并标有入口和出入口孔。请点击此处下载此文件。
讨论
该方案中的一个关键步骤是确保流通池在进入载物台之前不会泄漏,应事先在支架外以高流速进行测试。安装样品后的泄漏会损坏光学元件,尤其是底部物镜。
在实验过程中,对齐可能会随着时间的推移从其最佳位置漂移,由于等离子体镊子的灵敏度而导致信号变化。发生这种情况时,请使用压电控制重新调整基线的最大传输和最小标准差,因为嘈杂的基线会降低数据质量。应注意仔细记录何时进行对齐,以消除因陷印事件而混淆用户干扰的风险。如果发生重大漂移,请轻轻对齐载物台以最大限度地降低释放蛋白质的风险,并注意调整时间。
可以根据特定的实验需要对所提出的技术进行修改和更改。例如,温控载物台可以帮助根据需要冷却/加热样品,而不是使用激光加热来提高温度31,33。其他技术,例如干涉散射显微镜 (iSCAT),可以在 DNH 的散射场内提供蛋白质的干涉,获得与蛋白质极化率成比例的额外信号,这与粒径47,48 相关。
等离子体镊子纯粹是一种时间传感技术,因为数据由 APD(一种单像素检测器)记录。该技术无法提供有关蛋白质结构变化的直接信息,例如蛋白质的哪些区域参与构象变化,或者配体或其他蛋白质可能与蛋白质结合的位置。此外,由于数据采集卡的采样率 (1 MHz),该技术的时间范围限制为 >1 μs。考虑到奈奎斯特频率,其中最高可能的范围是采集速率的一半,在本例中,在理想条件下为 2 μs。
在该协议中,我们描述了设置等离子体 nanotweezers 实验以捕获单个蛋白质并监测其构象动力学随时间变化的过程。该技术可以在任何自制或商用显微镜上开发。与基于荧光或系留的方法(如 smFRET 和光镊)相反,该技术可以在没有标记或系绳的情况下捕获蛋白质,并且仍然实现单分子灵敏度。使用微流体系统捕获蛋白质时,可以改变溶液条件,从而实时监测不同溶液对蛋白质的影响。这些特性使等离子体镊子成为生物物理学和生物传感领域中很有前途的工具,特别是对于传统技术难以在其天然状态下研究的蛋白质。未来的应用将侧重于解码更多动态蛋白质的构象动力学,例如固有无序蛋白质和包含固有无序区域的蛋白质,以及膜蛋白,其动力学甚至结构都无法实现当前的技术。
披露声明
作者没有什么可披露的。
致谢
S.Z. 感谢生物技术和生物科学研究委员会博士生培训伙伴关系 (BBSRC DTP) (BB/T0083690/1) 的支持。作者感谢英国-印度教育和研究倡议 (UKIERI) 的资助。M.R. 感谢英国皇家学会和 Wolfson 基金会的支持。
材料
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
参考文献
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495(2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465(2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501(2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995(2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237(2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481(2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983(2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486(2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495(2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。