Method Article
Surveillance de la dynamique conformationnelle de protéines uniques non modifiées à l’aide de nanoweezers plasmoniques
Dans cet article
Résumé
Les nanopinces plasmoniques utilisent la résonance plasmonique de surface localisée dans les nanostructures d’or pour piéger des nanoparticules uniques, y compris des protéines, dans un champ optique à l’échelle nanométrique. Les modifications du signal diffusé révèlent la présence de protéines et la dynamique conformationnelle, ce qui permet une surveillance sans modifications de fluorophores ni attache de surface.
Résumé
Les techniques actuelles de caractérisation des protéines à molécule unique nécessitent généralement des marquages, des attaches ou l’utilisation de conditions de solution non natives. De tels changements peuvent modifier la biophysique des protéines et réduire l’utilité des données acquises. La nanopince plasmonique est une technique qui utilise la résonance plasmonique de surface localisée (LSPR) sur des nanostructures d’or pour améliorer le champ électrique dans une région de point chaud confinée. Cette amélioration du champ permet d’utiliser de faibles puissances laser pour piéger des nanoparticules uniques beaucoup plus petites que les pinces optiques conventionnelles, jusqu’à seulement quelques nanomètres de diamètre, comme les protéines uniques. Le piégeage de molécules protéiques uniques dans la région du point chaud induit un décalage de l’indice de réfraction local (protéine n > neau), modifiant la diffusion de la lumière en tant que produit de la polarisabilité de la molécule, qui est affectée par son volume, son anisotropie de forme et son indice de réfraction. Une photodiode d’avalanche (APD) recueille les modifications ultérieures de la diffusion de la lumière. Ces altérations peuvent ensuite être analysées pour déterminer les changements dans la molécule piégée, y compris sa taille, sa conformation globale et la dynamique du changement conformationnel au fil du temps. L’incorporation de la microfluidique dans le système permet de contrôler les changements environnementaux et de surveiller en temps réel leurs effets ultérieurs sur la molécule. Dans ce protocole, nous démontrons les étapes permettant de piéger des molécules de protéines uniques, de modifier leurs conditions de solution environnementale et de surveiller leurs changements conformationnels correspondants à l’aide d’un système de nanopinces plasmoniques.
Introduction
Les techniques actuelles à molécule unique pour interroger la dynamique conformationnelle des protéines comprennent des méthodes basées sur le marquage telles que le transfert d’énergie par résonance de fluorescence (FRET)1,2, des approches basées sur l’attache telles que les pinces optiques 3,4 et la microscopie à force atomique (AFM)5, des techniques basées sur les interférences telles que la microscopie à diffusion interférentielle (iSCAT)6, ou des techniques basées sur la nanofluidique telles que les nanopores7,8 et 9. Bien que ces méthodes présentent de nombreux avantages ; Quelques limitations clés les empêchent de fournir des données sur la dynamique conformationnelle des protéines non modifiées. Les pinces FRET et optiques nécessitent un marquage fluorophore ou une connexion à une surface, ce qui peut affecter les propriétés biophysiques des protéines 10,11,12. L’iSCAT, bien qu’il ne soit techniquement pas marqué, nécessite également une interaction entre la protéine et une surface pour observer les interférences générées entre les deux, ce qui affecte potentiellement les propriétés des protéines. De plus, limité par son rapport signal/bruit, iSCAT ne peut détecter que des protéines >40 kDa en raison du bruit de l’équipement et des fluctuations de fond semblables à des mouchetures13. Bien que cette limite de taille puisse être atténuée grâce à l’apprentissage automatique, les composants de la mémoire tampon sont limités car ils peuvent affecter les propriétés optiques, ce qui entraîne des données bruyantes13,14. Les nanopores présentent des temps de translocation rapides des protéines à travers le pore (généralement à moins de 5 μs), ce qui les rend incapables de détecter une dynamique conformationnelle plus lente15,16, bien que la recherche visant à atténuer ces limitations, comme l’utilisation d’origami d’ADN dans un piège électro-osmotique à nanopores17 ou l’incorporation de plasmoniques 18,19,20,21. De plus, des concentrations élevées de sel, généralement autour de 1 M, peuvent réduire l’applicabilité des données pour les travaux in vivo 15,22. La technique idéale pour la caractérisation des protéines à molécule unique devrait surveiller les protéines en temps réel et capturer la dynamique conformationnelle sur de plus longues durées (c’est-à-dire en millisecondes) sans qu’il soit nécessaire de modifier la protéine ou les conditions de solution non natives.
Les nanopinces plasmoniques sont similaires aux pinces optiques conventionnelles, en ce sens qu’elles utilisent la lumière pour piéger la matière. Les nanopinces plasmoniques, cependant, utilisent la résonance plasmonique de surface localisée (LSPR) pour augmenter le champ électrique de plusieurs ordres de grandeur afin de générer une force de gradient suffisamment forte pour piéger des nanoparticules uniques23. De plus, la particule piégée joue un rôle actif dans l’amélioration de la résistance du piège, connu sous le nom de piégeage à rétroaction auto-induite (SIBA) pour les structures à nanoouverture24. Ce piégeage SIBA permet à de faibles puissances laser (c’est-à-dire des milliwatts) de piéger de petites particules jusqu’à seulement quelques nanomètres de diamètre, telles que les protéines25, 26, 27. Le piégeage de molécules protéiques uniques dans la région du point chaud provoque un décalage de l’indice de réfraction local (protéine n > neau), modifiant la diffusion de la lumière en fonction de la polarisabilité de la molécule qui est influencée par le volume de la protéine, l’anisotropie de la forme et l’indice de réfraction28. Une photodiode d’avalanche (APD) détecte ensuite ces informations pour surveiller les changements ultérieurs de la diffusion de la lumière. De plus, les nanopinces plasmoniques permettent de surveiller les protéines piégées en temps réel sans marquage, attaches et conditions de solution difficiles pendant de longues périodes (c’est-à-dire de quelques minutes à quelques heures)29, répondant ainsi aux critères d’une technique idéale d’utilisation d’une seule molécule pour les protéines. À l’aide d’une structure à double nanotrou (DNH), les nanopinces plasmoniques ont démontré leur capacité à piéger diverses protéines et à élucider des informations clés à partir de celles-ci, notamment les transitions conformationnelles29, la cinétique de désassemblage30, les paysages énergétiques31, le suivi de diffusion32 et la liaison des ligands33,34. Outre les structures DNH, il a été démontré que des géométries de structure alternatives piègent les particules de petite taille35,36. Dans ce protocole, les étapes fondamentales de la mise en place et de l’exécution d’une configuration de nanopinces plasmoniques avec un système microfluidique intégré sont présentées. Nous espérons que ce protocole contribuera à accroître l’accessibilité et la compréhension des nanopinces plasmoniques pour les chercheurs, en particulier ceux dans les domaines de la biologie structurale et de la biophysique.
Protocole
ATTENTION : Veuillez lire toutes les fiches de données de sécurité (FDS) pertinentes pour tous les produits chimiques utilisés et respecter toutes les pratiques de sécurité appropriées, et porter un équipement de protection individuelle (lunettes de sécurité laser, blouses de laboratoire, gants) si nécessaire.
1. Construction de la configuration des nanopinces plasmoniques
REMARQUE : La configuration optique est basée sur le kit Modular Optical Tweezers System (OTKB) utilisant un laser et un APD différents (voir le tableau des matériaux). N’utilisez un équipement optique que sur une table optique appropriée pour réduire l’impact des vibrations externes sur le système. Le laser dans le kit était de 976 nm, mais comme la longueur d’onde de résonance maximale pour la résonance cunéiforme des structures DNH est d’environ 740-760 nm33. Nous avons choisi un laser NIR (852 nm) car il est proche du pic de résonance, induit un LSPR, et a également un meilleur taux de rendement de détection par l’APD à base de silicium. Des lasers avec des longueurs d’onde plus longuesde 20 ouplus courtes de 18 ont été utilisés pour piéger les biomolécules.
- Installez le laser dans le support laser à photodiode et introduisez-le dans un collimateur, ce qui conduit à un faisceau laser collimaté d’une largeur de 1,7 mm pour notre configuration.
- Ajoutez une plaque demi-onde au trajet de la lumière pour ajuster la polarisation. Ajustez à l’aide d’un polariseur Glan-Taylor pour vous assurer que la polarisation verticale (polarisation S) a l’intensité lumineuse la plus élevée.
REMARQUE : L’utilisation de la polarisation correcte est essentielle pour assurer une amélioration maximale du champ électrique des structures DNH car elles dépendent de la polarisation. - Focalisez la lumière à travers une configuration d’expansion de faisceau composée d’une lentille plan-concave (f = -50 mm) suivie d’une lentille plan-convexe (f = 150 mm) pour augmenter la largeur du faisceau à environ 5 mm afin de remplir toute l’ouverture arrière de l’objectif inférieur.
- Utilisez un miroir dichroïque (passe-court de 805 nm) pour réfléchir le laser à l’endroit souhaité. Installez la caméra (CCD) derrière elle.
- Redirigez la lumière vers l’objectif inférieur (100x/1,25 NA) et positionnez l’objectif supérieur (4x/0,1 NA) à la distance confocale pour collecter la lumière transmise.
- Ajoutez un autre miroir dichroïque (passe-court de 805 nm) pour réfléchir la lumière laser vers l’APD à travers une lentille plan-convexe positionnée à sa distance focale pour focaliser la lumière dans l’APD. Ajoutez la source de lumière blanche (WLS) derrière le miroir dichroïque afin qu’elle puisse revenir à travers la configuration pour atteindre la caméra. La figure 1 montre un schéma de la configuration entièrement assemblée des nanopinces plasmoniques.
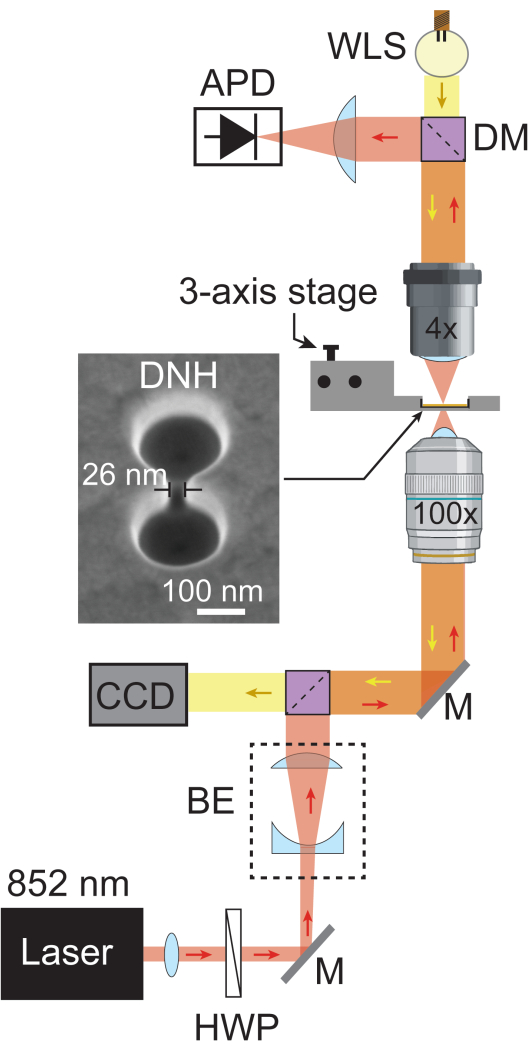
Figure 1 : Configuration des nanopinces plasmoniques. Le schéma montre le trajet optique complet de la configuration des nanopinces plasmoniques. Un faisceau laser de 852 nm (rouge) passe à travers un collimateur et une plaque demi-onde, puis est élargi par l’expanseur de faisceau (BE) à ~5,1 mm. Il est ensuite focalisé sur l’échantillon à l’aide d’un objectif 100x. La lumière laser transmise est collectée par un objectif 4x et enregistrée par un APD à une fréquence d’échantillonnage de 1 MHz. La lumière blanche du WLS (jaune) traverse l’objectif 4x, l’échantillon et l’objectif 100x avant d’atteindre le CCD, qui fournit une image en fond clair de l’échantillon. Les zones où les deux chemins lumineux se croisent sont représentées en orange. Image MEB de DNH prise à une inclinaison de 20°. Abréviations : BE = Expanseur de faisceau, CCD = Dispositif à couplage de charge, DM = Miroir dichroïque, HWP = Plaque demi-onde, M = Miroir argenté et WLS = Source de lumière blanche. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Fabrication des structures DNH
- Déposer 5 nm de Ti suivi de 100 nm d’Au sur une plaquette de silice fondue de 550 m d’épaisseur en utilisant l’évaporation par faisceau d’électrons comme décrit précédemment29,33. Coupez la plaquette en dés en échantillons de 1 cm x 1 cm prêts à l’emploi.
REMARQUE : L’évaporation par faisceau d’E a été choisie car elle produit une couche d’or à faible rugosité de surface, produisant des structures DNH de haute qualité avec une efficacité de piégeage élevée37. - Prenez l’échantillon de film d’or vierge et montez-le dans une configuration de faisceau d’ions focalisés au microscope électronique à balayage (SEM-FIB) à température ambiante à l’aide d’une source d’ions gallium.
- Assurez-vous que les nanoouvertures FIB sont alignées avec le MEB pour produire du DNH à des positions précises.
- Créez une boîte de repère à l’aide d’un faisceau d’ions à courant élevé (par exemple, 100 pA). Ce marqueur servira de guide de référence pour localiser la position des structures DNH sous le dispositif optique.
- Pour créer la structure DNH, deux cercles avec un entraxe de 200 nm et un diamètre de 160 nm sont reliés par un rectangle (3 nm x 40 nm). Pour une taille d’espace d’environ 20-30 nm, graver des cercles avec un facteur de dose de 0,09, tandis que la dose rectangulaire est comprise entre 0,300 et 0,350 en utilisant une énergie de sonde de 30 kV et un courant de faisceau de 1 pA.
REMARQUE : Le SEM-FIB utilisé a une résolution de 3 nm à cette énergie de sonde, ce qui permet une fabrication fiable de DNH avec des tailles d’écart comprises entre 20 et 30 nm. Nous n’utiliserions pas une résolution inférieure, car cela produirait des espaces plus grands, ce qui réduirait l’efficacité de piégeage du DNH. Si une FIB n’est pas réalisable, une autre méthode pour produire des structures DNH consiste à utiliser la lithographie sur nanosphères de polystyrène38. En raison de la haute précision requise pour fabriquer des structures DNH, la même recette peut ne pas produire la même taille d’espace entre les utilisations SEM-FIB. Modifiez la dose et la hauteur du rectangle (dose de 0,280 à 0,350 et hauteur de 2 à 4 nm) et mesurez la taille de l’écart, en visant environ 20 nm.
3. Revêtement des échantillons DNH
- Utilisez un récipient résistant aux solvants, tel qu’un plat de cristallisation en verre borosilicate 3.3, et ajoutez 20 ml d’éthanol.
ATTENTION : L’éthanol est hautement inflammable et irritant. À utiliser uniquement dans une hotte. Conserver dans un endroit frais et bien ventilé. Poignée avec des gants et une blouse de laboratoire. - Pesez 32 mg de poly(éthylène glycol) méthyléther thiol (PEG-thiol ; poids moléculaire moyen 800 g/mol) et mélangez-le dans l’éthanol, en vous assurant que tout le PEG-thiol s’est dissous pour produire une solution avec une concentration d’environ 2 mM pour maximiser la densité monocouche39.
ATTENTION : Le PEG-thiol est un irritant ; manipulez-le avec l’EPI approprié. - Ajoutez les puces d’échantillon contenant les nanostructures au mélange à l’aide d’une pince à épiler droite, couvrez et incubez pendant une nuit (~16 h) à température ambiante pour que le PEG-thiol forme une monocouche auto-assemblée sur la surface de l’or.
- Après l’incubation, rincer les échantillons en les tenant à l’aide d’une pince à épiler droite au-dessus d’un récipient approprié dans la hotte. Utilisez une bouteille à jet d’eau pour vaporiser soigneusement de l’éthanol de chaque côté. Sécher complètement à l’aide d’un pistolet à air comprimé avant de le ranger ou de le monter dans la nanopince plasmonique.
REMARQUE : Les échantillons peuvent être stockés à 4 °C pour améliorer la longévité car ils peuvent se dégrader avec le temps.
4. Montage de l’échantillon revêtu de PEG dans une cellule d’écoulement
- Placez l’échantillon revêtu dans la cellule d’écoulement imprimée en 3D (imprimée par une imprimante Form 2 avec de la résine Clear V4) à l’aide d’une pince à épiler droite avec la couche d’or vers le haut (voir la figure supplémentaire 1 pour les paramètres et valeurs clés de notre conception de cellule d’écoulement).
- Décollez un côté du couvercle du ruban adhésif double face en plastique PET transparent à l’aide d’une pince à épiler droite et placez-le sur l’échantillon et la cellule d’écoulement, en vous assurant que les nanostructures et les trous d’entrée/sortie de la cellule d’écoulement restent non couverts. Appuyez doucement sur les bords du ruban à l’aide d’une pince à épiler arrondie pour vous assurer qu’il a correctement adhéré à la cellule d’écoulement et à l’échantillon.
- Décollez l’autre côté du ruban et placez délicatement une lamelle en verre (épaisseur 0,17 mm) sur l’échantillon à l’aide d’une pince à épiler arrondie. Appuyez doucement sur les bords de la lamelle avec la pince à épiler arrondie pour vous assurer qu’elle a bien adhéré. Cela crée un canal liquide (hauteur = 50 μm, volume = 3,5 μL) à l’intérieur de la cellule d’écoulement.
REMARQUE : N’appuyez pas sur la lamelle de recouvrement à l’endroit ou à proximité de l’endroit où se trouvent les nanostructures, car elles pourraient être endommagées. Si la lamelle est sale, lavez-la à l’éthanol et séchez-la avec un pistolet à air comprimé avant de la monter. - Mélangez des parties égales de A et B des solutions de silicone dupliquées dans un rapport de 1:1 (ou selon les instructions du fabricant) sur une lame de microscope à l’aide d’une petite pointe de pipette.
- Remplissez les espaces entre la lamelle et la cellule d’écoulement avec le silicone à duplication mixte, en le poussant doucement sous la lamelle. Tenez la cellule d’écoulement à l’envers et placez soigneusement le silicone dupliqué autour de la paroi interne du trou avant d’utiliser la pointe de la pipette pour la déplacer doucement sur les bords visibles de la face inférieure de la silice fondue de l’échantillon (voir la figure supplémentaire 2).
- Laisser sécher avec la couche d’or vers le haut jusqu’à ce que le silicone dupliqué ait complètement pris, selon les instructions du fabricant.
REMARQUE : Veillez à ne pas laisser le silicone recouvrir les nanostructures lorsqu’il est appliqué sur la face inférieure de l’échantillon. Veillez à ne pas forcer la duplication du silicone sous la lamelle car il pourrait pénétrer dans les trous d’entrée/sortie de la cellule d’écoulement ou dans les nanostructures, qui peuvent être difficiles à nettoyer (Figure supplémentaire 3).
5. Connexion du système microfluidique
REMARQUE : Assurez-vous d’utiliser un tube propre pour le système. Ici, des tubes en PTFE de petit diamètre intérieur : 0,18 mm de diamètre intérieur et des tubes en PTFE de grand diamètre intérieur : 0,8 mm seront utilisés, mais d’autres valeurs de diamètre intérieur fonctionneront.
- Installez le pousse-seringue et connectez-le à l’électrovanne 3/2 voies à l’aide du grand tube en PTFE. Connectez un orifice de la vanne au récipient tampon et l’autre à une bobine de retenue, d’un volume d’environ 1 ml, fabriquée à partir de la grande tubulure ID pour éviter le reflux dans la seringue.
REMARQUE : La vanne 3/2 peut être contrôlée par un contrôleur de vanne, qui détermine si la solution est infusée/retirée du récipient tampon ou du serpentin de retenue. - Connectez la bobine de maintien à la vanne centrale de la vanne microfluidique bidirectionnelle rotative 12/1. Connectez les conteneurs pour trouver des solutions en utilisant des tubes de petit diamètre intérieur pour les échantillons rares ou précieux et des tubes de grand diamètre intérieur pour le reste. Utilisez une vanne uniquement pour l’injection dans un conteneur à déchets et une autre uniquement pour l’injection dans la cellule d’écoulement ; utilisez un petit tube ID pour la vanne de perfusion à cellules d’écoulement.
- Connectez les tubes de grand diamètre intérieur de la sortie de la cellule d’écoulement au conteneur à déchets. La figure 2 montre un schéma du système microfluidique entièrement connecté.
REMARQUE : La tubulure en PTFE de petit diamètre est préférable pour l’admission de la cellule d’écoulement et pour les vannes qui seront utilisées pour la perfusion de protéines en raison du plus petit volume où l’échantillon restera dans la tubulure. Les tubes en PTFE à grand ID sont préférables pour les matériaux moins coûteux ou plus abondants tels que les tampons. Essayez d’utiliser un tube minimal pour réduire davantage ce volume mort.
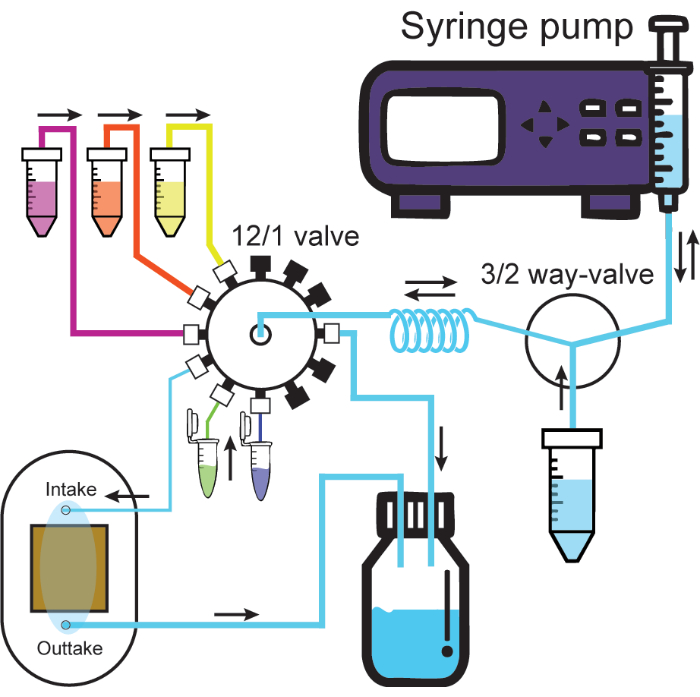
Figure 2 : Système microfluidique. Le schéma illustre le système microfluidique. Le pousse-seringue infuse ou aspire les solutions par un orifice de la vanne 3/2 voies, soit le récipient de solution, soit la bobine de retenue. Les solutions connectées à la vanne 12/1 passent toujours par le serpentin de maintien lorsqu’elles sont retirées et peuvent ensuite être infusées à travers le canal souhaité. L’infusion dans le canal d’admission connecté à la cellule d’écoulement poussera la solution de la cellule d’écoulement dans le récipient à déchets. Les tubes épais et minces représentent les tubes d’identification grands et petits du protocole. Les capuchons noirs sur la vanne 12/1 représentent des canaux étanches. Les directions d’écoulement sont indiquées par des flèches noires. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Préparation du système microfluidique
- Chargez l’interface utilisateur du système microfluidique sur le PC et vérifiez que les composants sont correctement connectés. Sélectionnez l’icône de lecture à côté du fil et du distributeur pour ouvrir leur interface utilisateur respective. Pour contourner la vanne 3/2 voies, tournez l’orifice 1 sur le fil.
- Assurez-vous que les vannes qui seront utilisées dans le système sont nettoyées en infusant de l’isopropanol (IPA), puis lavez les tubes plusieurs fois avec le ou les tampons de votre choix ou de l’eau distillée pour éliminer l’air et l’IPA dans le système. La perfusion peut être effectuée en sélectionnant la valve souhaitée sur l’interface utilisateur du distributeur et en faisant fonctionner le pousse-seringue, en sélectionnant infuser/retirer et en choisissant le volume et le débit souhaités (par exemple, un volume de 0,5 mL et un débit de 0,2 mL/min).
REMARQUE : N’infusez pas d’IPA dans les cellules d’écoulement, car cela dégraderait la cellule et la colle, ce qui pourrait entraîner des fuites.
7. Montage de la cellule d’écoulement dans des nanopinces plasmoniques et vérification des fuites
- Fixez les tubes d’entrée et de sortie à la partie appropriée de la cellule d’écoulement avant de les placer sur un tissu propre avec une couche d’or vers le haut.
- Injectez un tampon dans la cellule d’écoulement avec un débit élevé (~0,3 mL/min) et vérifiez que le fluide se déplace à travers l’échantillon dans la cellule d’écoulement et qu’aucun fluide n’est visible à l’extérieur ou en dessous de la cellule d’écoulement. Des débits plus élevés peuvent être utilisés, à condition qu’il n’y ait pas de fuite. En cas de fuite de l’échantillon, démontez et remontez.
- Ajoutez 1 à 2 gouttes d’huile d’immersion sur l’objectif 100x avant de placer la cellule d’écoulement dans l’étage de nanopince plasmonique avec la couche d’or vers le bas. Placez des clips métalliques sur les aimants de la cellule d’écoulement et verrouillez la platine pour aider à la maintenir en place.
ATTENTION : L’huile objective est irritante, dangereuse pour la santé et toxique pour la vie aquatique, manipulez uniquement avec des gants.
8. Localisation des nanostructures sur l’échantillon
- Allumez la source de lumière blanche. Ouvrez le logiciel de l’appareil photo et augmentez le gain et le temps d’exposition jusqu’à ce que les nanostructures soient visibles. Allumez le laser avec une puissance laser relativement élevée et ajustez manuellement l’axe z jusqu’à ce que le point laser soit visible. Privilégiez l’augmentation du temps d’exposition et du gain plutôt que l’augmentation de la puissance du laser pour trouver le point laser.
ATTENTION : Les lasers présentent un risque potentiel sérieux pour les utilisateurs. Assurez-vous d’utiliser des EPI appropriés, tels que des lunettes de sécurité laser avec une densité optique suffisante dans la gamme de longueurs d’onde requise. - Allumez le contrôleur piézoélectrique et sélectionnez les paramètres appropriés pour le port COM et la tension maximale. Réglez les valeurs des axes x, y et z sur la moitié de la tension maximale pour permettre une bonne plage d’alignement de la platine dans toutes les directions.
REMARQUE : Le port COM du contrôleur piézoélectrique peut être trouvé et modifié dans le gestionnaire de périphériques sous l’onglet ports. - Déplacez le spot laser pour qu’il se superpose à l’un des DNH à l’aide des boutons de commande des axes x, y et z de l’étage principal. Assurez-vous que l’APD est allumé, puis fermez doucement le boîtier à la nanopince plasmonique.
REMARQUE : L’utilisation d’un outil de marquage dans le logiciel de la caméra, s’il est disponible, aidera à déplacer le point laser sur une nanostructure. Placez le marqueur au centre du spot laser et éteignez le laser, mais laissez la source de lumière blanche allumée, pour permettre un alignement plus facile sur une nanostructure. L’APD peut être sursaturé et endommagé si trop de lumière laser l’atteint. Assurez-vous que l’échantillon se trouve dans le chemin du laser et augmentez progressivement la puissance du laser pour vous assurer que la saturation n’est pas atteinte. Des filtres à densité neutre (N.D) peuvent être placés avant l’APD pour réduire la lumière qui l’atteint si nécessaire.
9. Alignement optimal du laser avec la nanostructure souhaitée
- Ouvrez le logiciel associé à l’enregistrement APD, tel qu’une interface utilisateur Labview faite maison, et réglez la fréquence de coupure à 1 kHz. Nommez et définissez le chemin d’accès au fichier souhaité et le format de nommage des fichiers qui seront enregistrés.
- Éteignez la source de lumière blanche et rallumez le laser. Réglez sur une puissance laser appropriée (par exemple, ~20 mW) et utilisez les commandes piézoélectriques pour ajuster les axes x, y et z jusqu’à ce que le signal APD soit aussi élevé que possible, en évitant la saturation APD et avec un écart-type (S.D.) minimal de la trace.
REMARQUE : L’APD aura probablement une plage de sensibilité optimale pour la transmission (voir le manuel du produit), ce qui est idéal pour rester autour, par exemple, 1000-2000 mV. Des filtres N.D. peuvent être utilisés pour maintenir la transmission autour de cette plage. La principale considération est que la transmission ne soit pas proche du point de saturation APD, car le piégeage peut augmenter la transmission jusqu’à la limite de saturation, entraînant une perte de données.
10. Infuser des protéines dans la cellule de flux
- Éteignez le laser pour préserver la durée de vie des nanostructures et faites fonctionner l’unité de commande du pousse-seringue et l’interface utilisateur du distributeur pour régler la valve et prélever la quantité souhaitée de protéines (par exemple, 30 μL). Pour le retrait à l’aide de la tubulure à petit diamètre intérieur, un débit plus faible permet de s’assurer que toutes les solutions sont retirées (~0,01 - 0,1 mL/min). Des débits plus élevés peuvent être utilisés pour réduire le temps d’attente si vous le souhaitez.
REMARQUE : Le volume optimal de protéines à utiliser dépend de la valeur de la protéine et de ce que l’expérience espère accomplir. La concentration typique en protéines que nous utilisons est de 1 μM et un volume aliquote de 100 μL. Si l’échantillon de protéines est abondant, des concentrations plus élevées peuvent être utilisées pour réduire le temps moyen de piégeage. Cependant, si les concentrations sont trop élevées, le risque de piéger deux des mêmes protéines dans la nanostructure augmentera. Si l’expérience nécessite une perfusion d’un ligand/protéine différent après avoir piégé la protéine initiale, un volume d’aliquote plus faible peut être préféré pour minimiser les déchets et le temps nécessaire à la perfusion de la solution suivante. Assurez-vous que le tube reste immergé dans la solution pour empêcher l’air de pénétrer dans le système. Des précautions particulières doivent être prises pour les aliquotes de protéines si le tube n’atteint pas le fond du récipient ou si la solution est trop retirée. - À l’aide de l’interface utilisateur microfluidique, perfuser la solution protéique à un débit similaire au taux de retrait (~0,01 - 0,1 mL/min). Laisser infuser à ce rythme jusqu’à ce que la solution protéique atteigne la cellule d’écoulement, puis réduire le débit à ≤0,001 mL/min. Vérifiez le volume et le temps sur le pousse-seringue pour vous assurer qu’ils correspondent au volume prévu (p. ex., 1 min pour un volume de 1 mL à 1 mL/min).
REMARQUE : Le volume requis pour cela peut être calculé sur la base du volume du tube du système microfluidique à la cellule d’écoulement. Si la longueur et l’ID du tube sont connus, un calculateur en ligne du volume d’une bouteille peut être utilisé. Le débit de perfusion peut être ajusté à volonté, par exemple >0,001 mL/min pour l’infusion de protéines afin de réduire le temps d’attente moyen pour un piège. Cependant, la prudence est de mise car si le débit est trop élevé, cela peut empêcher les protéines de pénétrer dans la nanostructure.
11. Collecte des données
- Pendant que la solution protéique est infusée dans la cellule d’écoulement, commencez l’enregistrement des données du signal APD. Ajustez les axes x, y et z si nécessaire à l’aide de l’interface utilisateur du contrôleur piézoélectrique, car le système risque de dériver avec le temps. Les traces de piégeage idéales ont un modèle général cohérent suivant celui de la trace de la figure 3.
- Lorsque vous observez un changement important de transmission et d’écart-type similaire à la trace de piégeage exemplaire, notez le moment où cela se produit pour le tri futur des données (voir la figure 4B par exemple).
REMARQUE : La dérive dans le système peut entraîner des changements dans la transmission et une augmentation de S.D., parfois assez soudainement, qui pourrait être confondu avec un piège à protéines. Assurez-vous que le saut de signal affiche le même motif que le piège exemplaire et qu’aucune interférence externe n’était présente au moment du saut, telle qu’une dérive d’alignement ou des bruits forts captés par le système. - Si la protéine doit être libérée dans le cadre de l’expérience, éteignez le laser pendant ~5 secondes et rallumez-le. La trace doit présenter un changement important de transmission et un écart-type significativement plus faible, indiquant un retour à l’état de base.
REMARQUE : Si aucun changement important dans la transmission et/ou le S.D. n’est observé, ou si une trace similaire à la protéine piégée est observée, la protéine est probablement collée à la surface de l’échantillon (voir la figure 5B, par exemple).
12. Démontage de l’échantillon
- Après avoir effectué l’expérience souhaitée, éteignez le laser, retirez la cellule d’écoulement de l’étage à 3 axes et débranchez le tube du système microfluidique.
- Placez la cellule d’écoulement sur un tissu propre avec la couche d’or de l’échantillon vers le haut. À l’aide d’un scalpel, coupez soigneusement la colle sous la lamelle en verre et soulevez-la doucement à l’aide d’une pince à épiler arrondie. Jetez-le dans le bac en verre brisé désigné.
ATTENTION : Le verre brisé et l’utilisation d’objets pointus peuvent causer des blessures. Assurez-vous de porter des lunettes de sécurité, des gants et une blouse de laboratoire. - Tenez la cellule d’écoulement à un angle avec la couche d’or toujours tournée vers le haut et utilisez une pince à épiler arrondie pour retirer soigneusement la colle sur la face inférieure de la cellule d’écoulement afin de libérer l’échantillon. La duplication du silicone permet un retrait facile sans endommager les structures DNH.
- À l’aide d’une pince à épiler, prélevez l’échantillon et rincez-le abondamment avec de l’IPA, puis de l’eau avant de le sécher avec un pistolet à air comprimé. S’il est réutilisable, stockez l’échantillon dans un récipient approprié avec la couche d’or vers le haut.
REMARQUE : Les échantillons ont généralement une durée de vie d’environ 1 à 2 semaines s’ils sont manipulés correctement, bien que la taille des nanostructures change avec le temps. S’il est trop endommagé, passez à un échantillon frais pour l’expérience suivante. Les dommages peuvent prendre la forme de rayures présentes sur la surface de l’or près des nanostructures ou de mauvaises performances expérimentales, indiquant une dégradation des nanostructures29.
13. Préparation du système pour une utilisation future
- Utilisez un chiffon plié pour essuyer l’huile de l’objectif dans une direction, en le soulevant et en répétant l’opération avec une autre partie propre du tissu.
REMARQUE : Les objectifs sont sujets aux taches et aux rayures, ce qui peut nuire considérablement aux performances. Portez toujours des gants pendant le nettoyage et évitez de toucher les matières organiques. N’utilisez que du chiffon neuf pour éviter la contamination et nettoyez dans une seule direction en un seul mouvement. Évitez de frotter l’objectif et le chiffon de nettoyage d’avant en arrière pour minimiser les dommages. Si le nettoyage à sec ne suffit pas, appliquez une petite quantité d’éthanol sur le mouchoir, essuyez l’huile comme mentionné précédemment et terminez avec un chiffon sec. - Si les mêmes solutions protéiques/tampons sont utilisées ensuite, il se peut qu’il ne soit pas nécessaire de changer le tube. Si différentes solutions sont utilisées, remplacez le tube pour réduire le risque de contamination par les solutions précédentes.
REMARQUE : N’utilisez pas le même tube pendant plus de 1 semaine, même si vous effectuez des expériences avec les mêmes solutions, car il se salira avec le temps.
Résultats
Après l’acquisition des données, l’analyse des données brutes peut être effectuée à l’aide du code MATLAB pour générer des traces à partir des données brutes collectées par l’APD. La figure 3 représente un exemple de trace de piégeage comprenant la ligne de base avant le piégeage, l’événement de piégeage où un changement important de transmission (ΔT/T0) et d’écart-type est observé avant que le laser ne soit éteint pendant environ 5 secondes avant d’être rallumé. Une réduction significative de l’écart-type et un retour de la transmission à des niveaux similaires à ceux de référence indiquent la libération de protéines. La dérive linéaire est supprimée du tracé à l’aide de la fonction MATLAB detrend.m, puis la valeur moyenne des données est rajoutée au tracé supprimé. De temps en temps, nous devons démouler la trace au fur et à mesure que la configuration dérive au fil du temps, ce qui entraîne une diminution linéaire de la transmission (voir la trace grise dans la figure 3). De petits changements dans les traces de ligne de base avant et après le piégeage sont dus à l’ajustement de la platine pour optimiser la ligne de base avec un écart-type minimal, comme le montre la figure 4A. Parfois, des molécules de protéines sont visibles dans la trace sans être piégées, ce qu’on appelle des protéines de passage. Les protéines qui passent apparaissent comme un changement brutal de transmission, similaire à un piège typique (Figure 4B), mais avec une durée nettement plus courte, comme le montre la Figure 4A. La densité spectrale de puissance (PSD) présente une autre analyse pour confirmer le piégeage des protéines en fournissant une intensité de signal à différentes fréquences. Les mouvements conformationnels des protéines sont généralement observés dans la gamme >1 μs par des méthodes de spectroscopie à molécule unique40. La figure 4C montre que, par rapport à la ligne de base, le piégeage d’une protéine conduit à une intensité de signal plus élevée, au moins dans la gamme de 10 kHz (> 100 μs). Il souligne également l’importance d’aligner la platine sur une ligne de base optimisée, car une mauvaise ligne de base pourrait augmenter le bruit à des fréquences comprises entre 50 et 500 Hz, une gamme de fréquences superposée aux mouvements conformationnels des protéines.
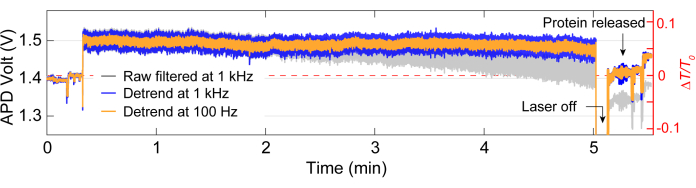
Figure 3 : Trace de piégeage complète pour une seule protéine. Trace représentative d’un piège complet, y compris la ligne de base, le piégeage d’une protéine et la libération de la protéine. Les sauts de trace avant et après le piégeage sont dus à l’alignement. Veuillez cliquer ici pour voir une version agrandie de cette figure.
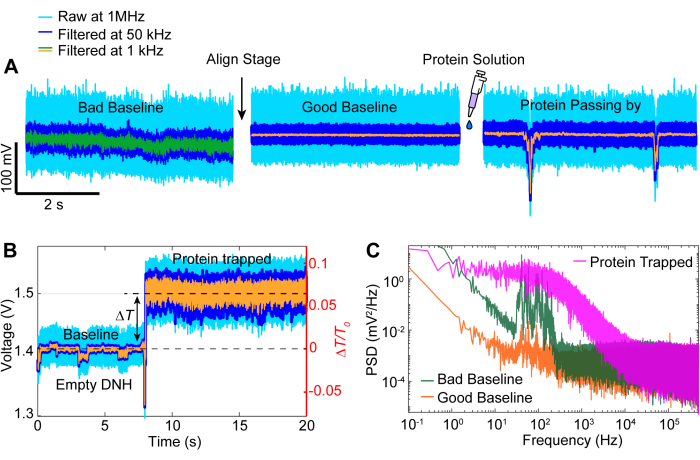
Figure 4 : Événements de trace courants. (A) Exemples d’un alignement d’une mauvaise à une bonne ligne de base et d’une protéine passant près du point chaud. (B) Trace de piégeage montrant le processus depuis la ligne de base lorsque le point chaud DNH est vide jusqu’au moment où la protéine est piégée. (C) Graphique de densité spectrale de puissance (PSD) entre les bonnes et les mauvaises lignes de base représentées en (A) et la protéine piégée en (B). Des valeurs PSD plus élevées indiquent un bruit plus important à des fréquences particulières. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La plupart des événements de piégeage suivent le même schéma général que la trace de la figure 3, bien que des problèmes occasionnels puissent survenir pendant les expériences. Pour la plupart des expériences, la protéine doit être libérée manuellement en éteignant le laser une fois l’expérience souhaitée terminée. Dans certains cas, cependant, la protéine peut quitter le piège sans intervention, comme le montre la figure 5A. À l’inverse, les protéines peuvent parfois rester sur le site de piégeage même après avoir éteint le laser, probablement en raison de la protéine qui adhère à l’échantillon. Ce collage entraîne une trace bruyante après l’extinction et la rallumage du laser (voir Figure 5B). La probabilité que cela se produise dépend de la protéine, car certaines protéines sont plus sujettes à l’adsorption de surface41,42. L’utilisation d’un enrobage tel que le PEG-thiol peut réduire les risques de collage des protéines39,43. À moins que vous ne le souhaitiez, comme l’étude des interactions protéine-protéine, un autre problème est le double piégeage, où une deuxième protéine est piégée après le premier piège. Celle-ci se caractérise par une autre forte augmentation de la transmission, similaire au premier piège, et une modification de l’écart-type (voir figure 5C).
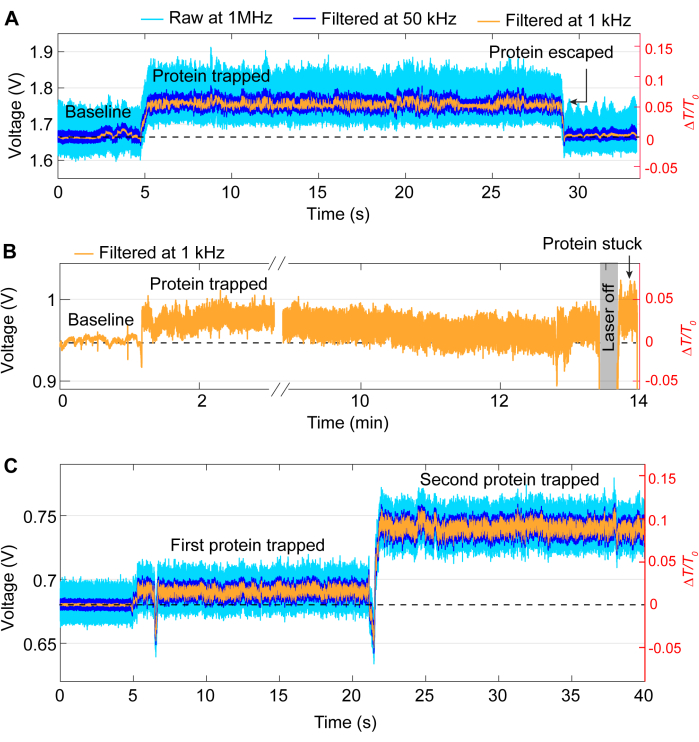
Figure 5 : Exemples d’événements de piégeage indésirables. (A) Libération involontaire d’une protéine à partir du point chaud de DNH. (B) Exemple de protéine qui se coince à la surface de l’échantillon dans le point chaud DNH. (C) Le saut de trace se produit lorsqu’une deuxième protéine est piégée alors que la première reste encore dans le point chaud DNH. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Une expérience représentative réalisée sur le chargement in situ du fer dans une molécule d’apo-ferritine démontre l’utilisation de nanopinces plasmoniques comme outil pour étudier la dynamique conformationnelledes protéines 29. La ferritine est une protéine porteuse de fer qui existe dans deux états : l’apo-ferritine, qui ne contient pas de fer, et l’holo-ferritine, qui est remplie de fer44,45. Le fer ferreux pénètre dans la protéine par des canaux 3 fois où il est oxydé en fer ferrique et stocké dans le noyau protéique46. La figure 6A représente une trace typique de piégeage de l’apo-ferritine avec une solution ferreuse perfusée pendant plus de 20 minutes pendant que la protéine est piégée. Les traces de 20 s prises le long de l’ensemble de la trace aux points b-e donnent un aperçu des changements qui se produisent sur la protéine au fil du temps. Dans la figure 6B, l’apo-ferritine est piégée dans un tampon PBS standard, et aucun changement significatif n’est observé dans la trace. Les figures 6C et D montrent les fluctuations du S.D des traces, qui sont causées par la charge de fer dans la protéine à travers ses canaux 3 fois, ce qui entraîne un état plus dynamique (apo-) où les canaux sont ouverts, et un état plus compact (holo-) avec les canaux fermés. Lorsque la molécule de ferritine a été remplie de fer, elle est passée à son holoforme, ce qui a donné une trace de piégeage stable, comme le montre la figure 6E. Les fonctions de densité de probabilité (PDF) des figures 6B-E mettent en évidence les changements que subit la protéine lors de l’exposition à différentes conditions de solution au fil du temps.
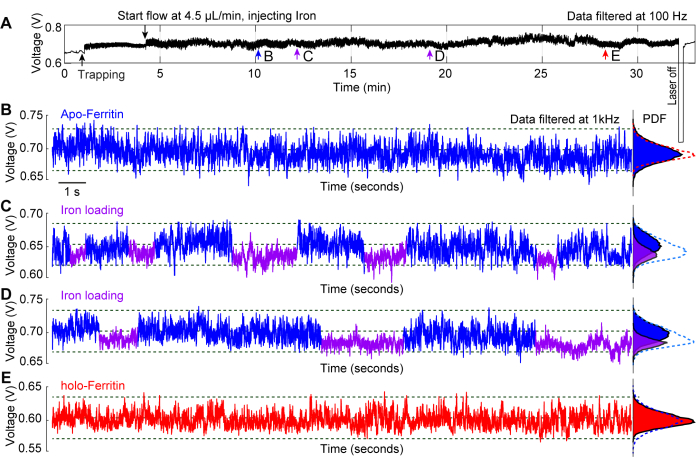
Figure 6 : Chargement in situ du fer dans une apoferritine piégée. (A) Trace de transmission complète d’un DNH avec une molécule d’apoferritine piégée, suivie de l’injection d’une solution ferreuse au site de piégeage pour observer les changements conformationnels de la ferritine associés à la charge en fer. (B) Trace de piégeage de 20 secondes d’une apoferritine piégée avant que la solution ferreuse n’atteigne le point chaud. (C, D) Traces de piégeage de 20 secondes après l’exposition de la molécule d’apoferritine à la solution ferreuse. Les segments bleus et violets marquent le S.D supérieur et inférieur de la trace, indiquant respectivement des conformations souples et rigides de la ferritine. (E) Trace de piégeage de 20 secondes après exposition de l’apoferritine à la solution ferreuse pendant >20 minutes. Les graphiques de la fonction de densité de probabilité (PDF) à droite montrent la distribution de la transmission et sont codés par couleur pour les segments bleu et violet. Ce chiffre a été modifié au lieu de29. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Échantillon d’or DNH monté sur la cellule d’écoulement imprimée en 3D. L’échantillon est placé dans une fente spéciale et collé à la cellule d’écoulement à l’aide d’un ruban adhésif PET double face. Les paramètres clés et les mesures associées pour la conception de nos cellules d’écoulement sont étiquetés. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Arrière de la cellule d’écoulement avec l’échantillon DNH doré monté et la paroi intérieure étiquetée. L’échantillon est scellé dans la cellule d’écoulement à l’aide de silicone dupliqué. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Schéma de la cellule d’écoulement avec DNH doré monté avec trous d’admission et de sortie étiquetés. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Une étape cruciale du protocole consiste à s’assurer que la cellule d’écoulement ne fuit pas avant d’être installée dans la scène, qui doit être testée à l’extérieur du support à un débit élevé au préalable. Une fuite après le montage de l’échantillon peut endommager les composants optiques, en particulier l’objectif inférieur.
L’alignement peut dériver de sa position optimale au fil du temps au cours d’une expérience, provoquant une variation du signal en raison de la sensibilité des nanopinces plasmoniques. Lorsque cela se produit, réalignez-vous à l’aide des commandes piézoélectriques jusqu’à la transmission maximale et l’écart-type minimal de la ligne de base, car une ligne de base bruyante réduit la qualité des données. Il faut prendre soin de prendre des notes méticuleuses sur le moment où les alignements sont effectués afin d’éliminer le risque de confondre l’interférence de l’utilisateur avec un événement de piégeage. En cas de dérive importante, alignez doucement la platine pour minimiser le risque de libération de la protéine et notez le temps d’ajustement.
Des modifications et des altérations de la technique présentée peuvent être apportées en fonction des besoins expérimentaux spécifiques. Par exemple, une platine à température contrôlée peut aider à refroidir/chauffer l’échantillon comme vous le souhaitez au lieu d’utiliser le chauffage laser pour augmenter la température31,33. D’autres techniques, par exemple la microscopie à diffusion interférométrique (iSCAT), peuvent fournir une interférence de la protéine dans le champ de diffusion du DNH, obtenant un signal supplémentaire proportionnel à la polarisabilité de la protéine, qui est associée à la taille des particules47,48.
Les nanopinces plasmoniques sont une technique purement de détection temporelle car les données sont enregistrées par l’APD (un détecteur à pixel unique). Cette technique ne fournit aucune information directe sur les changements structurels d’une protéine, tels que les régions de la protéine impliquées dans un changement de conformation ou l’endroit où un ligand ou une autre protéine peut se lier à la protéine. De plus, la technique est limitée à une plage temporelle de >1 μs en raison de la fréquence d’échantillonnage de la carte d’acquisition de données (1 MHz). Si l’on considère la fréquence de Nyquist, où la portée la plus élevée possible est la moitié du taux d’acquisition, dans ce cas, 2 μs dans des conditions parfaites.
Dans ce protocole, nous avons décrit le processus de mise en place d’une expérience de nanopinces plasmoniques pour piéger une seule protéine et surveiller les changements dans sa dynamique conformationnelle au fil du temps. La technique peut être développée sur n’importe quel microscope artisanal ou commercial. Contrairement aux approches basées sur la fluorescence ou le tethering, telles que le smFRET et les pinces optiques, la technique peut piéger des protéines sans marqueurs ni attaches tout en atteignant une sensibilité à une molécule unique. Les conditions de la solution peuvent être modifiées pendant que la protéine est piégée à l’aide d’un système microfluidique, ce qui permet de surveiller en temps réel l’effet des différentes solutions sur la protéine. Ces caractéristiques font des nanopinces plasmoniques un outil prometteur dans les domaines de la biophysique et de la biodétection, en particulier pour les protéines que les techniques conventionnelles peinent à interroger dans leur état natif. Les applications futures se concentreront sur le décodage de la dynamique conformationnelle de protéines plus dynamiques telles que les protéines intrinsèquement désordonnées et les protéines contenant des régions intrinsèquement désordonnées, et les protéines membranaires, dont la dynamique et même la structure échappent aux techniques actuelles.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
S.Z. remercie le Partenariat de formation doctorale du Conseil de recherches en biotechnologie et en sciences biologiques (BBSRC DTP) (BB/T0083690/1). Les auteurs remercient le financement de l’Initiative UK-India pour l’éducation et la recherche (UKIERI). M.R. apprécie le soutien de la Royal Society et de la Fondation Wolfson.
matériels
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
Références
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495(2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465(2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501(2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995(2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237(2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481(2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983(2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486(2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495(2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon