Method Article
Überwachung der Konformationsdynamik einzelner unmodifizierter Proteine mit einer plasmonischen Nanopinzette
In diesem Artikel
Zusammenfassung
Plasmonische Nanopinzetten nutzen die lokalisierte Oberflächenplasmonenresonanz in Goldnanostrukturen, um einzelne Nanopartikel, einschließlich Proteine, in einem optischen Feld im Nanometerbereich einzufangen. Veränderungen im Streusignal zeigen das Vorhandensein von Proteinen und die Konformationsdynamik und ermöglichen eine Überwachung ohne Fluorophormodifikationen oder Oberflächenbindung.
Zusammenfassung
Aktuelle Einzelmolekültechniken zur Charakterisierung von Proteinen erfordern in der Regel Markierungen, Tether oder die Verwendung nicht-nativer Lösungsbedingungen. Solche Veränderungen können die Biophysik der Proteine verändern und den Nutzen der gewonnenen Daten verringern. Die plasmonische Nanopinzette ist eine Technik, die die lokalisierte Oberflächenplasmonenresonanz (LSPR) auf Goldnanostrukturen verwendet, um das elektrische Feld innerhalb eines begrenzten Hotspot-Bereichs zu verstärken. Diese Feldverbesserung ermöglicht die Verwendung geringer Laserleistungen, um einzelne Nanopartikel einzufangen, die weitaus kleiner sind als herkömmliche optische Pinzetten, bis zu einem Durchmesser von nur wenigen Nanometern, wie z. B. einzelne Proteine. Das Einfangen einzelner Proteinmoleküle innerhalb der Hotspot-Region induziert eine Verschiebung des lokalen Brechungsindex (n-Protein >n-Wasser), wodurch sich die Lichtstreuung als Produkt der Polarisierbarkeit des Moleküls verändert, die durch sein Volumen, seine Formanisotropie und seinen Brechungsindex beeinflusst wird. Eine Avalanche-Photodiode (APD) sammelt die nachfolgenden Änderungen der Lichtstreuung. Diese Veränderungen können dann analysiert werden, um Veränderungen im gefangenen Molekül zu bestimmen, einschließlich seiner Größe, der globalen Konformation und der Dynamik der Konformationsänderung im Laufe der Zeit. Die Integration von Mikrofluidik in das System ermöglicht eine kontrollierte Umweltveränderung und eine Echtzeitüberwachung ihrer nachfolgenden Auswirkungen auf das Molekül. In diesem Protokoll demonstrieren wir die Schritte, um einzelne Proteinmoleküle einzufangen, ihre Umgebungsbedingungen zu verändern und ihre entsprechenden Konformationsänderungen mit einem plasmonischen Nanopinzettensystem zu überwachen.
Einleitung
Zu den derzeitigen Einzelmolekültechniken zur Untersuchung der Konformationsdynamik von Proteinen gehören markierungsbasierte Methoden wie der Fluoreszenzresonanz-Energietransfer (FRET)1,2, Tethering-basierte Ansätze wie die optische Pinzette 3,4 und die Rasterkraftmikroskopie (AFM)5, interferenzbasierte Techniken wie die Interferenzstreumikroskopie (iSCAT)6 oder nanofluidische Techniken wie Nanoporen7.8,9. Diese Methoden haben zwar viele Vorteile; Einige wichtige Einschränkungen hindern sie daran, Daten über die Konformationsdynamik unmodifizierter Proteine zu liefern. FRET- und optische Pinzetten erfordern eine Fluorophormarkierung oder eine Bindung an eine Oberfläche, was die biophysikalischen Eigenschaften der Proteine beeinträchtigen kann 10,11,12. iSCAT ist zwar technisch markierungsfrei, erfordert aber auch eine Wechselwirkung zwischen dem Protein und einer Oberfläche, um die zwischen den beiden erzeugte Interferenz zu beobachten, die möglicherweise die Eigenschaften der Proteine beeinflusst. Darüber hinaus kann iSCAT, begrenzt durch sein Signal-Rausch-Verhältnis, aufgrund von Geräterauschen und speckleartigen Hintergrundfluktuationen nur Proteine >40 kDa nachweisen13. Obwohl diese Größenbeschränkung durch maschinelles Lernen gemildert werden kann, sind die Pufferkomponenten begrenzt, da sie die optischen Eigenschaften beeinflussen können, was zu verrauschten Daten führt13,14. Nanoporen weisen schnelle Translokationszeiten von Proteinen durch die Pore auf (normalerweise innerhalb von 5 μs), wodurch sie nicht in der Lage sind, eine langsamere Konformationsdynamik zu erkennen15,16, obwohl Forschungen zur Linderung dieser Einschränkungen durchgeführt werden, wie z. B. die Verwendung von DNA-Origami in einer elektroosmotischen Nanoporenfalle17 oder der Einbau von Plasmonik 18,19,20,21. Darüber hinaus können hohe Salzkonzentrationen, typischerweise um 1 M, die Anwendbarkeit der Daten für In-vivo-Arbeiten verringern15,22. Die ideale Einzelmolekültechnik für die Proteincharakterisierung sollte Proteine in Echtzeit überwachen und die Konformationsdynamik über längere Zeiträume (d. h. Millisekunden) erfassen, ohne dass Änderungen am Protein oder nicht-native Lösungsbedingungen erforderlich sind.
Plasmonische Nanopinzetten ähneln herkömmlichen optischen Pinzetten in dem Sinne, dass sie Licht verwenden, um Materie einzufangen. Plasmonische Nanopinzetten verwenden jedoch die lokalisierte oberflächenplasmonische Resonanz (LSPR), um das elektrische Feld um mehrere Größenordnungen zu verstärken und eine Gradientenkraft zu erzeugen, die stark genug ist, um einzelne Nanopartikel einzufangen23. Darüber hinaus spielt das gefangene Partikel eine aktive Rolle bei der Erhöhung der Festigkeit der Falle, was als selbstinduziertes Rückwirkungsfallen (SIBA) für Nanoaperturstrukturen bekannt ist24. Diese SIBA-Falle ermöglicht es, kleine Partikel mit geringen Laserleistungen (d. h. Milliwatt) bis zu einem Durchmesser von nur wenigen Nanometern einzufangen, wie z. B. die Proteine 25,26,27. Das Einfangen einzelner Proteinmoleküle innerhalb der Hotspot-Region führt zu einer Verschiebung des lokalen Brechungsindex (n-Protein >n-Wasser), wodurch sich die Lichtstreuung basierend auf der Polarisierbarkeit des Moleküls verändert, die durch das Volumen, die Formanisotropie und den Brechungsindex des Proteins beeinflusst wird28. Eine Avalanche-Photodiode (APD) erkennt diese Informationen dann, um spätere Veränderungen der Lichtstreuung zu überwachen. Darüber hinaus ermöglichen plasmonische Nanopinzetten die Überwachung der gefangenen Proteine in Echtzeit ohne Markierung, Fesseln und raue Lösungsbedingungen über lange Zeiträume (d. h. Minuten bis Stunden)29 und erfüllen damit die Kriterien für eine ideale Einzelmolekültechnik für Proteine. Unter Verwendung einer Doppel-Nanoloch-Struktur (DNH) haben plasmonische Nanopinzetten ihre Fähigkeit demonstriert, verschiedene Proteine einzufangen und wichtige Informationen aus ihnen aufzuklären, einschließlich Konformationsübergänge29, Demontagekinetik30, Energielandschaften31, Diffusionsverfolgung32 und Ligandenbindung33,34. Neben DNH-Strukturen wurden alternative Strukturgeometrien gezeigt, um Partikel mit kleinen Partikelgrößeneinzufangen 35,36. In diesem Protokoll werden die grundlegenden Schritte zum Einrichten und Ausführen einer plasmonischen Nanopinzette mit einem integrierten Mikrofluidiksystem vorgestellt. Wir hoffen, dass dieses Protokoll dazu beitragen wird, die Zugänglichkeit und das Verständnis der plasmonischen Nanopinzette für Forscher zu verbessern, insbesondere für diejenigen in den Bereichen Strukturbiologie und Biophysik.
Protokoll
ACHTUNG: Bitte lesen Sie alle relevanten Sicherheitsdatenblätter (SDB) für alle verwendeten Chemikalien, halten Sie sich an alle entsprechenden Sicherheitspraktiken und tragen Sie bei Bedarf persönliche Schutzausrüstung (Laserschutzbrille, Laborkittel, Handschuhe).
1. Aufbau des Aufbaus der plasmonischen Nanopinzette
HINWEIS: Der optische Aufbau basiert auf dem Modular Optical Tweezers System (OTKB) Kit, das einen anderen Laser und APD verwendet (siehe Materialtabelle). Verwenden Sie optische Geräte nur auf einem geeigneten optischen Tisch, um die Auswirkungen externer Vibrationen auf das System zu reduzieren. Der Laser im Bausatz betrug 976 nm, aber da die maximale Resonanzwellenlänge für die Keilresonanz der DNH-Strukturen bei etwa 740-760 nmliegt, liegt 33. Wir haben uns für einen NIR-Laser (852 nm) entschieden, da er nahe am Resonanzpeak liegt, LSPR induziert und auch eine bessere Detektionsausbeute durch den siliziumbasierten APD aufweist. Laser mit längeren Wellenlängenvon 20 oderkürzeren 18 Wellenlängen wurden verwendet, um Biomoleküle einzufangen.
- Richten Sie den Laser in der Fotodioden-Laserhalterung ein und führen Sie ihn in einen Kollimator ein, der für unseren Aufbau zu einem kollimierten Laserstrahl mit einer Breite von 1,7 mm führt.
- Fügen Sie dem Strahlengang eine Halbwellenplatte hinzu, um die Polarisation anzupassen. Stellen Sie mit einem Glan-Taylor-Polarisator ein, um sicherzustellen, dass die vertikale Polarisation (S-Polarisation) die höchste Lichtintensität hat.
HINWEIS: Die Verwendung der richtigen Polarisation ist von entscheidender Bedeutung, um eine maximale Verstärkung des elektrischen Feldes durch die DNH-Strukturen zu gewährleisten, da diese polarisationsabhängig sind. - Fokussieren Sie das Licht durch einen Strahlaufweiter, der aus einer plankonkaven Linse (f = -50 mm) und einer plankonvexen Linse (f = 150 mm) besteht, um die Strahlbreite auf etwa 5 mm zu vergrößern und die gesamte hintere Öffnung des unteren Objektivs auszufüllen.
- Verwenden Sie einen dichroitischen Spiegel (805 nm Kurzpass), um den Laser an die gewünschte Stelle zu reflektieren. Stellen Sie die Kamera (CCD) dahinter auf.
- Lenken Sie das Licht in das untere Objektiv (100x/1,25 NA) und positionieren Sie das obere Objektiv (4x/0,1 NA) in der konfokalen Entfernung, um das übertragene Licht zu sammeln.
- Fügen Sie einen weiteren dichroitischen Spiegel (805 nm Kurzpass) hinzu, um das Laserlicht durch eine plankonvexe Linse, die in seiner Brennweite positioniert ist, in Richtung APD zu reflektieren und das Licht in den APD zu fokussieren. Fügen Sie die Weißlichtquelle (WLS) hinter dem dichroitischen Spiegel hinzu, damit sie durch das Setup zurückgeleitet werden kann, um die Kamera zu erreichen. Abbildung 1 zeigt ein Schema des vollständig zusammengesetzten Aufbaus der plasmonischen Nanopinzette.
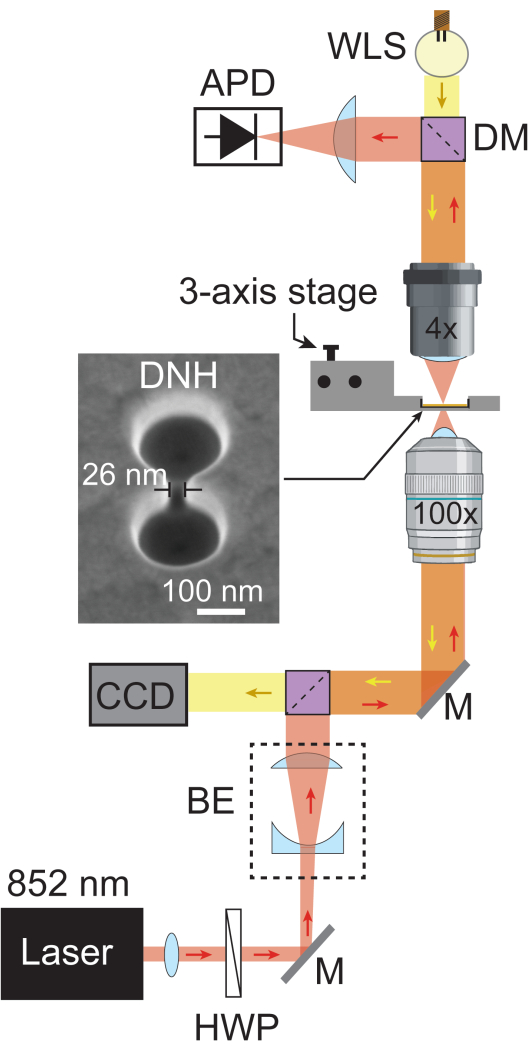
Abbildung 1: Aufbau der plasmonischen Nanopinzette. Das Schema zeigt den vollständigen optischen Weg des Aufbaus der plasmonischen Nanopinzette. Ein 852 nm Laserstrahl (rot) durchläuft einen Kollimator und eine Halbwellenplatte und wird dann durch den Strahlaufweiter (BE) auf ~5,1 mm erweitert. Anschließend wird es mit einem 100-fach-Objektiv auf die Probe fokussiert. Das transmittierte Laserlicht wird von einem 4x-Objektiv erfasst und von einem APD mit einer Abtastrate von 1 MHz aufgezeichnet. Weißes Licht von WLS (gelb) durchdringt das 4x-Objektiv, die Probe und das 100x-Objektiv, bevor es den CCD erreicht, der ein Hellfeldbild der Probe liefert. Bereiche, in denen sich beide Lichtwege kreuzen, werden orange dargestellt. REM-Aufnahme von DNH bei 20° Neigung. Abkürzungen: BE = Beam expander, CCD = Charge-coupled device, DM = Dichroitischer Spiegel, HWP = Halbwellenplatte, M = Silberspiegel und WLS = Weißlichtquelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Herstellung der DNH-Strukturen
- Abscheidung von 5 nm Ti gefolgt von 100 nm Au auf einem 550 μm dicken Quarzglaswafer mittels Elektronenstrahlverdampfung, wie zuvor beschrieben29,33. Würfeln Sie den Wafer auf 1 cm x 1 cm große Proben, die sofort verwendet werden können.
HINWEIS: Die E-Beam-Verdampfung wurde gewählt, da sie eine Goldschicht mit geringer Oberflächenrauheit erzeugt, wodurch hochwertige DNH-Strukturen mit hoher Einfangeffizienz erzeugtwerden 37. - Nehmen Sie die leere Goldfilmprobe und montieren Sie sie in einen Rasterelektronenmikroskop-fokussierten Ionenstrahl (REM-FIB), der bei Raumtemperatur mit einer Gallium-Ionenquelle aufgebaut ist.
- Stellen Sie sicher, dass die FIB-Nanoaperturen mit dem REM ausgerichtet sind, um DNH an genauen Positionen zu erzeugen.
- Erstellen Sie eine Markierungsbox mit einem Hochstrom-Ionenstrahl (z. B. 100 pA). Dieser Marker dient als Referenz, um die Position der DNH-Strukturen unter dem optischen Aufbau zu lokalisieren.
- Um die DNH-Struktur zu erzeugen, werden zwei Kreise mit einem Achsabstand von 200 nm und einem Durchmesser von 160 nm durch ein Rechteck (3 nm x 40 nm) überbrückt. Für eine Spaltgröße von etwa 20-30 nm werden Ätzkreise mit einem Dosisfaktor von 0,09 geätzt, während die Rechteckdosis zwischen 0,300 und 0,350 liegt, bei einer Sondenenergie von 30 kV und einem Strahlstrom von 1 pA.
HINWEIS: Das verwendete REM-FIB hat eine Auflösung von 3 nm bei dieser Sondenenergie, was eine zuverlässige Herstellung von DNH mit Spaltgrößen zwischen 20 und 30 nm ermöglicht. Wir würden keine niedrigere Auflösung verwenden, da dies zu größeren Lückengrößen führt, was die Einfangeffizienz von DNH verringert. Ist eine FIB nicht realisierbar, ist ein alternatives Verfahren zur Herstellung von DNH-Strukturen die Verwendung der Polystyrol-Nanosphären-Lithographie38. Aufgrund der hohen Präzision, die für die Herstellung von DNH-Strukturen erforderlich ist, kann es sein, dass das gleiche Rezept zwischen den SEM-FIB-Anwendungen nicht die gleiche Spaltgröße erzeugt. Ändern Sie die Dosis und Höhe des Rechtecks (0,280 bis 0,350 Dosis und 2 bis 4 nm Höhe) und messen Sie die Spaltgröße, wobei Sie auf etwa 20 nm abzielen.
3. Beschichtung von DNH-Proben
- Verwenden Sie ein lösungsmittelbeständiges Gefäß, z. B. eine Kristallisationsschale aus Borosilikatglas 3.3, und fügen Sie 20 ml Ethanol hinzu.
ACHTUNG: Ethanol ist leicht entzündlich und reizend. Nur in einem Abzug verwenden. An einem kühlen, gut belüfteten Ort lagern. Griff mit Handschuhen und einem Laborkittel. - Wiegen Sie 32 mg Poly(ethylenglykol)-methyletherthiol (PEG-Thiol; durchschnittliches Molekulargewicht 800 g/mol) und mischen Sie dieses in das Ethanol, wobei Sie sicherstellen, dass sich das gesamte PEG-Thiol aufgelöst hat, um eine Lösung mit einer Konzentration von etwa 2 mM zu erhalten, um die Monolagendichtezu maximieren 39.
ACHTUNG: PEG-Thiol ist ein Reizstoff; Behandeln Sie es mit geeigneter PSA. - Die Probenchips, die die Nanostrukturen enthalten, mit einer geraden Pinzette in die Mischung geben, abdecken und über Nacht (~16 h) bei Raumtemperatur inkubieren, damit das PEG-Thiol eine selbstorganisierte Monoschicht auf der Goldoberfläche bildet.
- Spülen Sie die Proben nach der Inkubation aus, indem Sie sie mit einer geraden Pinzette über einen geeigneten Behälter in der Abzugshaube halten. Sprühen Sie mit einer Spritzflasche gründlich Ethanol auf jede Seite. Trocknen Sie es vollständig mit einer Luftpistole, bevor Sie es anschließend aufbewahren oder in die plasmonische Nanopinzette einbauen.
HINWEIS: Die Proben können bei 4 °C gelagert werden, um die Langlebigkeit zu verbessern, da sie sich im Laufe der Zeit zersetzen können.
4. Einbau der PEG-beschichteten Probe in eine Durchflusszelle
- Legen Sie die beschichtete Probe mit einer geraden Pinzette in die 3D-gedruckte Durchflusszelle (gedruckt von einem Form-2-Drucker mit Clear V4-Harz), wobei die Goldschicht nach oben zeigt (siehe Ergänzende Abbildung 1 für wichtige Parameter und Werte in unserem Durchflusszellendesign).
- Ziehen Sie eine Seite der doppelseitigen Klebebandabdeckung aus durchsichtigem PET-Kunststoff mit einer geraden Pinzette ab und legen Sie sie auf die Probe und die Durchflusszelle, wobei Sie darauf achten, dass die Nanostrukturen und Ein- und Auslasslöcher in der Durchflusszelle unbedeckt bleiben. Drücken Sie vorsichtig mit einer abgerundeten Pinzette um die Ränder des Bandes, um sicherzustellen, dass es richtig an der Durchflusszelle und der Probe haftet.
- Ziehen Sie die andere Seite des Klebebandes ab und legen Sie mit einer abgerundeten Pinzette vorsichtig ein Deckglas (Dicke 0,17 mm) über die Probe. Drücken Sie mit der abgerundeten Pinzette vorsichtig um die Ränder des Deckglases, um sicherzustellen, dass es richtig haftet. Dadurch entsteht ein Flüssigkeitskanal (Höhe = 50 μm, Volumen = 3,5 μL) innerhalb der Durchflusszelle.
HINWEIS: Drücken Sie das Deckglas nicht an oder in der Nähe der Stelle, an der sich die Nanostrukturen befinden, da diese beschädigt werden können. Wenn das Deckglas verschmutzt ist, waschen Sie es mit Ethanol und trocknen Sie es mit einer Luftpistole, bevor Sie es montieren. - Mischen Sie gleiche Teile von A und B der Dupliziersilikonlösungen im Verhältnis 1:1 (oder gemäß den Anweisungen des Herstellers) mit einer kleinen Pipettenspitze auf einen Objektträger.
- Füllen Sie die Lücken zwischen Deckglas und Durchflusszelle mit dem gemischten Dupliziersilikon und schieben Sie es vorsichtig unter das Deckglas. Halten Sie die Durchflusszelle auf den Kopf und platzieren Sie vorsichtig Dupliziersilikon um die Innenwand des Lochs, bevor Sie es mit der Pipettenspitze vorsichtig auf die sichtbaren Ränder der Quarzglasunterseite der Probe schieben (siehe ergänzende Abbildung 2).
- Mit der Goldschicht nach oben trocknen lassen, bis das Dupliziersilikon gemäß den Anweisungen des Herstellers vollständig ausgehärtet ist.
HINWEIS: Achten Sie darauf, dass Silikon die Nanostrukturen nicht bedeckt, wenn es auf die Unterseite der Probe aufgetragen wird. Achten Sie darauf, dass das Dupliziersilikon nicht unter das Deckglas gedrückt wird, da es in die Ein- und Auslassöffnungen der Durchflusszelle oder in die Nanostrukturen eindringen kann, die schwer zu reinigen sein können (Ergänzende Abbildung 3).
5. Anschließen des Mikrofluidiksystems
HINWEIS: Stellen Sie sicher, dass das System saubere Schläuche verwendet. Hier werden PTFE-Schläuche mit kleinem Innendurchmesser von 0,18 mm und PTFE-Schläuchen mit großem Innendurchmesser von 0,8 mm verwendet, aber auch andere Innendurchmesser funktionieren.
- Stellen Sie die Spritzenpumpe auf und verbinden Sie sie mit dem großen PTFE-Schlauch mit dem 3/2-Wege-Magnetventil. Verbinden Sie einen Anschluss des Ventils mit dem Pufferbehälter und den anderen mit einer Halteschlange mit einem Volumen von etwa 1 mL, die aus dem großen Innenschlauch besteht, um einen Rückfluss in die Spritze zu verhindern.
HINWEIS: Das 3/2-Ventil kann über eine Ventilsteuerung gesteuert werden, die bestimmt, ob die Lösung aus dem Pufferbehälter oder aus der Halteschlange infundiert/entnommen wird. - Verbinden Sie die Haltespule mit dem Mittelventil des bidirektionalen Mikrofluidik-Drehventils 12/1. Verbinden Sie Behälter für Lösungen mit kleinen ID-Schläuchen für seltene oder wertvolle Proben und großen ID-Schläuchen für den Rest. Verwenden Sie ein Ventil nur zum Aufgießen in einen Abfallbehälter und ein anderes nur zum Aufgießen in die Durchflusszelle. Verwenden Sie einen kleinen Innenschlauch für das Durchflusszellen-Infusionsventil.
- Schließen Sie große ID-Schläuche vom Ausgang der Durchflusszelle an den Abfallbehälter an. Abbildung 2 zeigt ein Schema des vollständig vernetzten mikrofluidischen Systems.
HINWEIS: PTFE-Schläuche mit kleinem Innendurchmesser sind für die Aufnahme der Durchflusszelle und für Ventile, die für die Proteininfusion verwendet werden, aufgrund des kleineren Volumens, in dem die Probe im Schlauch verbleibt, vorzuziehen. PTFE-Schläuche mit großem Innendurchmesser sind für weniger teure oder reichlichere Materialien wie Puffer vorzuziehen. Versuchen Sie, minimale Schläuche zu verwenden, um dieses Totvolumen weiter zu reduzieren.
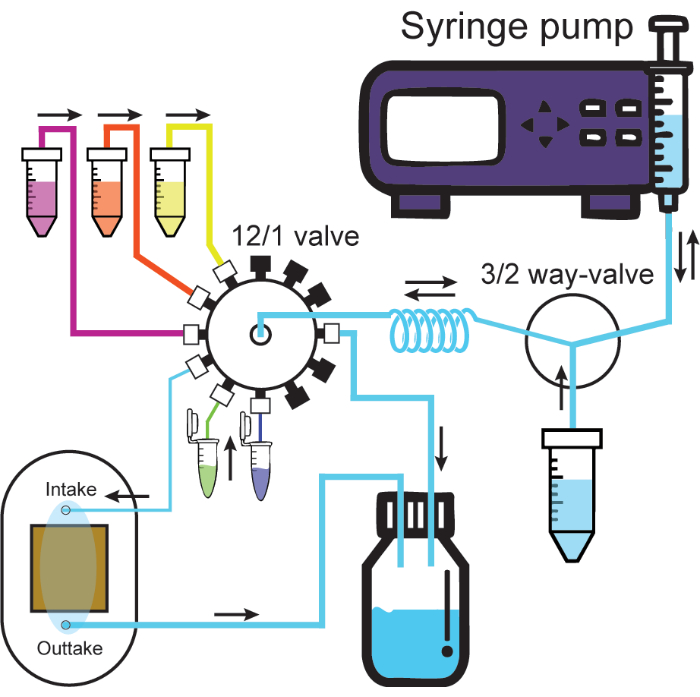
Abbildung 2: Mikrofluidisches System. Das Schema veranschaulicht das mikrofluidische System. Die Spritzenpumpe infundiert oder entzieht Lösungen durch einen Anschluss des 3/2-Wege-Ventils, entweder den Lösungsbehälter oder die Halteschlange. Lösungen, die mit dem Ventil 12/1 verbunden sind, durchlaufen beim Herausziehen immer die Halteschlange und können dann durch den gewünschten Kanal infundiert werden. Durch die Infusion in den Ansaugkanal, der mit der Durchflusszelle verbunden ist, wird die Lösung aus der Durchflusszelle in den Abfallbehälter gedrückt. Dicke und dünne Schläuche stellen große und kleine ID-Schläuche aus dem Protokoll dar. Schwarze Kappen am 12/1-Ventil stellen versiegelte Kanäle dar. Fließrichtungen sind durch schwarze Pfeile gekennzeichnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Vorbereitung des Mikrofluidiksystems
- Laden Sie die Benutzeroberfläche des Mikrofluidiksystems auf den PC und überprüfen Sie, ob die Komponenten ordnungsgemäß angeschlossen sind. Wählen Sie das Wiedergabesymbol neben dem Draht und dem Verteiler aus, um die jeweilige Benutzeroberfläche zu öffnen. Um das 3/2-Wege-Ventil zu umgehen, drehen Sie Anschluss 1 am Kabel.
- Stellen Sie sicher, dass die Ventile, die im System verwendet werden, durch Aufgießen von Isopropanol (IPA) gereinigt werden, und waschen Sie die Schläuche dann mehrmals mit Puffern Ihrer Wahl oder destilliertem Wasser, um Luft und IPA aus dem System zu entfernen. Die Infusion kann durch Auswahl des gewünschten Ventils auf der Benutzeroberfläche des Verteilers und Betreiben der Spritzenpumpe, Auswahl von Infusion/Entnahme und Auswahl des gewünschten Volumens und der gewünschten Durchflussrate (z. B. 0,5 mL Volumen und 0,2 mL/min Durchflussrate) erfolgen.
HINWEIS: Infundieren Sie IPA nicht in Durchflusszellen, da dies die Zelle und den Klebstoff zersetzt, was zu Undichtigkeiten führen kann.
7. Einbau der Durchflusszelle in eine plasmonische Nanopinzette und Prüfung auf Dichtheit
- Befestigen Sie die Einlass- und Auslassschläuche an der entsprechenden Stelle der Durchflusszelle, bevor Sie sie mit einer goldenen Schicht nach oben auf sauberes Gewebe legen.
- Infundieren Sie Puffer mit einer hohen Flussrate (~0,3 ml/min) in die Durchflusszelle und prüfen Sie, ob sich die Flüssigkeit über die Probe in der Durchflusszelle bewegt und dass keine Flüssigkeit an der Außen- oder Unterseite der Durchflusszelle sichtbar ist. Höhere Durchflussmengen können verwendet werden, sofern keine Leckagen auftreten. Wenn die Probe undicht wird, wird die Halterung getrennt und wieder bereitgestellt.
- Geben Sie 1-2 Tropfen Immersionsöl auf das 100x-Objektiv, bevor Sie die Durchflusszelle mit der Goldschicht nach unten in den plasmonischen Nanopinzettentisch einsetzen. Platzieren Sie Metallklammern über den Magneten der Durchflusszelle und verriegeln Sie den Tisch, um ihn an Ort und Stelle zu halten.
ACHTUNG: Objektives Öl ist reizend, gesundheitsschädlich und giftig für Wasserlebewesen, nur mit Handschuhen anfassen.
8. Lokalisierung von Nanostrukturen auf der Probe
- Schalten Sie die weiße Lichtquelle ein. Öffnen Sie die Kamerasoftware und erhöhen Sie die Verstärkung und Belichtungszeit, bis die Nanostrukturen sichtbar sind. Schalten Sie den Laser mit relativ hoher Laserleistung ein und stellen Sie die Z-Achse manuell ein, bis der Laserpunkt sichtbar ist. Priorisieren Sie die Erhöhung der Belichtungszeit und des Belichtungsgewinns gegenüber der Erhöhung der Laserleistung, um den Laserpunkt zu finden.
VORSICHT: Laser stellen ein ernsthaftes potenzielles Risiko für die Benutzer dar. Stellen Sie sicher, dass geeignete PSA wie z. B. Laserschutzbrillen mit ausreichender optischer Dichte innerhalb des erforderlichen Wellenlängenbereichs verwendet werden. - Schalten Sie den piezoelektrischen Controller ein und wählen Sie die entsprechenden Einstellungen für den COM-Port und die maximale Spannung. Stellen Sie die Werte für die x-, y- und z-Achsen auf die Hälfte der maximalen Spannung ein, um einen guten Bereich zum Ausrichten des Tisches in alle Richtungen zu ermöglichen.
HINWEIS: Der COM-Port des piezoelektrischen Controllers kann im Geräte-Manager unter der Registerkarte Ports gefunden und geändert werden. - Bewegen Sie den Laserspot so, dass er mit einem der DNH überlagert wird, indem Sie die Drehregler für die X-, Y- und Z-Achsen der Hauptbühne verwenden. Stellen Sie sicher, dass die APD eingeschaltet ist, und schließen Sie dann vorsichtig das Gehäuse mit der plasmonischen Nanopinzette.
HINWEIS: Die Verwendung eines Markierungswerkzeugs in der Kamerasoftware, falls verfügbar, hilft dabei, den Laserpunkt über eine Nanostruktur zu bewegen. Setzen Sie den Marker in die Mitte des Laserspots und schalten Sie den Laser aus, aber lassen Sie die weiße Lichtquelle eingeschaltet, um eine einfachere Ausrichtung an einer Nanostruktur zu ermöglichen. Der APD kann übersättigt und beschädigt werden, wenn zu viel Laserlicht ihn erreicht. Stellen Sie sicher, dass sich die Probe im Weg des Laserwegs befindet, und erhöhen Sie die Laserleistung allmählich, um sicherzustellen, dass die Sättigung nicht erreicht wird. Neutraldichtefilter (N.D.) können vor dem APD platziert werden, um das Licht, das ihn erreicht, bei Bedarf zu reduzieren.
9. Den Laser optimal auf die gewünschte Nanostruktur ausrichten
- Öffnen Sie die Software, die mit der APD-Aufzeichnung verknüpft ist, z. B. eine selbstgemachte Labview-Benutzeroberfläche, und stellen Sie die Grenzfrequenz auf 1 kHz ein. Benennen und legen Sie den gewünschten Dateipfad und das Benennungsformat der Dateien fest, die gespeichert werden sollen.
- Schalten Sie die weiße Lichtquelle aus und den Laser wieder ein. Stellen Sie die Laserleistung auf eine geeignete Laserleistung ein (z. B. ~20 mW) und stellen Sie mit den piezoelektrischen Reglern die x-, y- und z-Achsen so lange ein, bis das APD-Signal so hoch wie möglich ist, wodurch eine APD-Sättigung vermieden wird und die Standardabweichung (S.D.) der Leiterbahn minimal ist.
HINWEIS: Der APD verfügt wahrscheinlich über einen optimalen Empfindlichkeitsbereich für die Übertragung (siehe Produkthandbuch), der ideal ist, um z. B. 1000-2000 mV zu bleiben. N.D.-Filter können verwendet werden, um die Übertragung in diesem Bereich zu halten. Die Hauptüberlegung ist, dass die Übertragung nicht in der Nähe des APD-Sättigungspunkts liegt, da Trapping die Übertragung bis zur Sättigungsgrenze erhöhen kann, was zu Datenverlust führt.
10. Infusion von Proteinen in die Durchflusszelle
- Schalten Sie den Laser aus, um die Lebensdauer der Nanostrukturen zu erhalten, und lassen Sie die Steuereinheit der Spritzenpumpe und die Benutzeroberfläche des Verteilers laufen, um das Ventil einzustellen und die gewünschte Proteinmenge (z. B. 30 μl) zu entnehmen. Bei der Entnahme mit dem kleinen Innenschlauch hilft eine niedrigere Durchflussrate, sicherzustellen, dass alle Lösungen entnommen werden (~0,01 - 0,1 mL/min). Höhere Durchflussraten können auf Wunsch verwendet werden, um die Wartezeit zu reduzieren.
HINWEIS: Das optimale Proteinvolumen hängt davon ab, wie wertvoll das Protein ist und was mit dem Experiment erreicht werden soll. Die typische Proteinkonzentration, die wir verwenden, beträgt 1 μM und ein aliquotes Volumen von 100 μL. Wenn die Proteinprobe reichlich vorhanden ist, können höhere Konzentrationen verwendet werden, um die durchschnittliche Einfangzeit zu verkürzen. Sind die Konzentrationen jedoch zu hoch, steigt das Risiko, zwei gleiche Proteine in der Nanostruktur einzufangen. Wenn das Experiment eine Infusion eines anderen Liganden/Proteins nach dem Einfangen des Ausgangsproteins erfordert, kann ein niedrigeres aliquotes Volumen bevorzugt werden, um den Abfall und die Zeit für die Infusion der nächsten Lösung zu minimieren. Stellen Sie sicher, dass der Schlauch in Lösung eingetaucht bleibt, um zu verhindern, dass Luft in das System eindringt. Besondere Vorsicht ist bei Proteinaliquoten geboten, wenn der Schlauch nicht bis zum Boden des Behälters reicht oder die Lösung zu stark zurückgezogen wird. - Infizieren Sie die Proteinlösung mit der Mikrofluidik-Benutzeroberfläche mit einer ähnlichen Flussrate wie die Entnahmerate (~0,01 - 0,1 ml/min). Lassen Sie die Proteinlösung mit dieser Geschwindigkeit ziehen, bis die Proteinlösung die Durchflusszelle erreicht, und reduzieren Sie dann die Flussrate auf ≤0,001 ml/min. Überprüfen Sie das Volumen und die Zeit an der Spritzenpumpe, um sicherzustellen, dass sie mit dem erwarteten Volumen übereinstimmen (z. B. 1 min für 1 mL Volumen bei 1 mL/min).
HINWEIS: Das dafür erforderliche Volumen kann anhand des Volumens des Schlauchs vom mikrofluidischen System zur Durchflusszelle berechnet werden. Sind die Länge und ID des Schlauches bekannt, kann ein Online-Rechner für das Volumen einer Flasche verwendet werden. Die Infusionsrate kann nach Belieben angepasst werden, z. B. >0,001 mL/min für die Proteininfusion, um die durchschnittliche Wartezeit auf eine Falle zu verkürzen. Es ist jedoch Vorsicht geboten, denn wenn die Flussrate zu hoch ist, kann dies verhindern, dass Proteine in die Nanostruktur gelangen.
11. Erhebung von Daten
- Während die Proteinlösung in die Durchflusszelle infundiert wird, starten Sie die Datenaufzeichnung des APD-Signals. Passen Sie die x-, y- und z-Achsen nach Bedarf über die Benutzeroberfläche des piezoelektrischen Controllers an, da das System im Laufe der Zeit wahrscheinlich driftet. Ideale Überfüllungsspuren weisen ein konsistentes allgemeines Muster auf, das dem der Spur in Abbildung 3 folgt.
- Wenn Sie eine große Änderung der Transmission und S.D. beobachten, die der exemplarischen Trapping-Spur ähnelt, notieren Sie sich den Zeitpunkt, zu dem dies geschieht, für die zukünftige Datensortierung (siehe z. B. Abbildung 4B ).
HINWEIS: Eine Drift im System kann zu Veränderungen in der Übertragung und einem Anstieg des SD führen, manchmal ziemlich plötzlich, was mit einer Proteinfalle verwechselt werden könnte. Stellen Sie sicher, dass der Signalsprung das gleiche Muster wie die Exemplarfalle aufweist und dass beim Sprung keine externen Interferenzen vorhanden waren, wie z. B. Ausrichtungsdrift oder laute Geräusche, die vom System aufgenommen werden. - Wenn das Protein im Rahmen des Experiments freigesetzt werden muss, schalten Sie den Laser für ~5 s aus und wieder ein. Die Kurve sollte eine starke Änderung der Transmission und einen signifikant niedrigeren S.D. aufweisen, was auf eine Rückkehr zum Ausgangszustand hinweist.
HINWEIS: Wenn keine große Änderung der Transmission und/oder S.D. beobachtet wird oder eine ähnliche Spur des einzufangenden Proteins beobachtet wird, haftet das Protein wahrscheinlich an der Probenoberfläche fest (siehe z. B. Abbildung 5B).
12. Demontage der Probe
- Schalten Sie nach Durchführung des gewünschten Experiments den Laser aus, nehmen Sie die Durchflusszelle aus dem 3-Achsen-Tisch und trennen Sie die Schläuche des mikrofluidischen Systems.
- Platzieren Sie die Durchflusszelle mit der Goldschicht der Probe nach oben auf sauberem Gewebe. Schneiden Sie mit einem Skalpell vorsichtig den Kleber unter dem Glasdeckglas ab und heben Sie ihn vorsichtig mit einer abgerundeten Pinzette ab. Entsorgen Sie es in der dafür vorgesehenen Glasscherbentonne.
ACHTUNG: Glasscherben und die Verwendung von scharfen Gegenständen können zu Verletzungen führen. Stellen Sie sicher, dass eine Schutzbrille, Handschuhe und ein Laborkittel getragen werden. - Halten Sie die Durchflusszelle schräg, wobei die Goldschicht immer noch nach oben zeigt, und entfernen Sie mit einer abgerundeten Pinzette vorsichtig den Kleber auf der Unterseite der Durchflusszelle, um die Probe zu befreien. Das Duplizieren von Silikon ermöglicht eine einfache Entfernung, ohne die DNH-Strukturen zu beschädigen.
- Nehmen Sie die Probe mit einer geraden Pinzette auf und spülen Sie sie gründlich mit IPA und dann mit Wasser ab, bevor Sie sie mit einer Luftpistole trocknen. Wenn die Probe wiederverwendbar ist, lagern Sie sie in einem geeigneten Behälter mit der Goldschicht nach oben.
HINWEIS: Proben haben bei sachgemäßer Handhabung in der Regel eine nutzbare Spanne von etwa 1-2 Wochen, obwohl sich die Größe der Nanostrukturen im Laufe der Zeit ändert. Wenn sie übermäßig beschädigt ist, wechseln Sie für das nächste Experiment zu einer frischen Probe. Die Beschädigung kann in Form von Kratzern auf der Goldoberfläche in der Nähe der Nanostrukturen oder durch eine schlechte experimentelle Leistung erfolgen, was auf eine Verschlechterung der Nanostrukturenhinweist 29.
13. Vorbereiten des Systems für die zukünftige Verwendung
- Wischen Sie das Öl mit gefaltetem Linsentuch in eine Richtung vom Objektiv ab, heben Sie es ab und wiederholen Sie es mit einem anderen sauberen Teil des Tuchs.
HINWEIS: Objektive sind anfällig für Schmieren und Kratzer, was die Leistung erheblich beeinträchtigen kann. Tragen Sie während der Reinigung immer Handschuhe und vermeiden Sie es, organisches Material zu berühren. Verwenden Sie nur ein neues Reinigungstuch für die Linsen, um eine Kontamination zu vermeiden, und reinigen Sie es mit einer einzigen Bewegung in eine Richtung. Vermeiden Sie es, die Linse und das Reinigungstuch hin und her zu reiben, um Schäden zu minimieren. Wenn die chemische Reinigung nicht ausreicht, tragen Sie eine kleine Menge Ethanol auf das Tuch auf, wischen Sie das Öl wie zuvor erwähnt ab und beenden Sie es mit einem trockenen Tuch. - Wenn als nächstes die gleichen Protein-/Pufferlösungen verwendet werden, muss der Schlauch möglicherweise nicht gewechselt werden. Wenn andere Lösungen verwendet werden, ersetzen Sie die Schläuche, um das Risiko einer Kontamination durch frühere Lösungen zu verringern.
HINWEIS: Verwenden Sie denselben Schlauch nicht länger als 1 Woche, auch wenn Sie Experimente mit denselben Lösungen durchführen, da er mit der Zeit schmutzig wird.
Ergebnisse
Nach der Datenerfassung kann eine Datenanalyse der Rohdaten mithilfe von MATLAB-Code durchgeführt werden, um Ablaufverfolgungen aus den vom APD gesammelten Rohdaten zu generieren. Abbildung 3 zeigt ein Beispiel für eine Trapping-Spur einschließlich der Baseline vor dem Trapping, dem Trapping-Ereignis, bei dem eine große Änderung der Transmission (ΔT/T0) und der Standardabweichung beobachtet wird, bevor der Laser für etwa 5 Sekunden ausgeschaltet wird, bevor er wieder eingeschaltet wird. Eine signifikante Verringerung der Standardabweichung und die Rückkehr der Übertragung auf ein ähnliches Niveau wie der Ausgangswert deuten auf die Freisetzung von Protein hin. Die lineare Drift wird mit der MATLAB-Funktion detrend.m aus der Kurve entfernt, und dann wird der Mittelwert der Daten wieder zur trendbereinigten Kurve addiert. Gelegentlich müssen wir den Trend der Kurve verringern, da der Aufbau im Laufe der Zeit driftet, was zu einer linearen Abnahme der Transmission führt (siehe graue Kurve in Abbildung 3). Geringe Änderungen in den Baseline-Traces vor und nach dem Trapping sind auf die Stufenanpassung zurückzuführen, um die Baseline mit minimaler Standardabweichung zu optimieren, wie in Abbildung 4A dargestellt. Manchmal sind Proteinmoleküle in der Spur sichtbar, ohne eingeschlossen zu sein, was als vorbeigehende Proteine bezeichnet wird. Vorbeigehende Proteine erscheinen als eine starke Änderung der Transmission, ähnlich einer typischen Falle (Abbildung 4B), jedoch mit einer deutlich kürzeren Dauer, wie in Abbildung 4A gezeigt. Die spektrale Leistungsdichte (PSD) stellt eine weitere Analyse dar, um das Einfangen von Proteinen zu bestätigen, indem die Signalstärke bei verschiedenen Frequenzen bereitgestellt wird. Konformationsbewegungen von Proteinen werden typischerweise im Bereich von >1 μs mit Einzelmolekülspektroskopiemethoden40 beobachtet. Abbildung 4C zeigt, dass das Einfangen eines Proteins im Vergleich zum Ausgangswert zu einer höheren Signalstärke führt, zumindest im Bereich von 10 kHz (> 100 μs). Es unterstreicht auch, wie wichtig es ist, den Tisch auf eine optimierte Grundlinie auszurichten, da eine schlechte Grundlinie das Rauschen bei Frequenzen zwischen 50 und 500 Hz erhöhen kann, einem Frequenzbereich, der von Proteinkonformationsbewegungen überlagert wird.
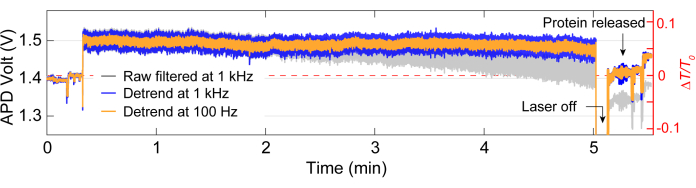
Abbildung 3: Vollständige Fangspur für ein einzelnes Protein. Repräsentative Spur für eine vollständige Falle, einschließlich der Baseline, des Einfangens eines Proteins und der Freisetzung des Proteins. Sprünge in der Leiterbahn vor und nach dem Überfüllen sind auf die Ausrichtung zurückzuführen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
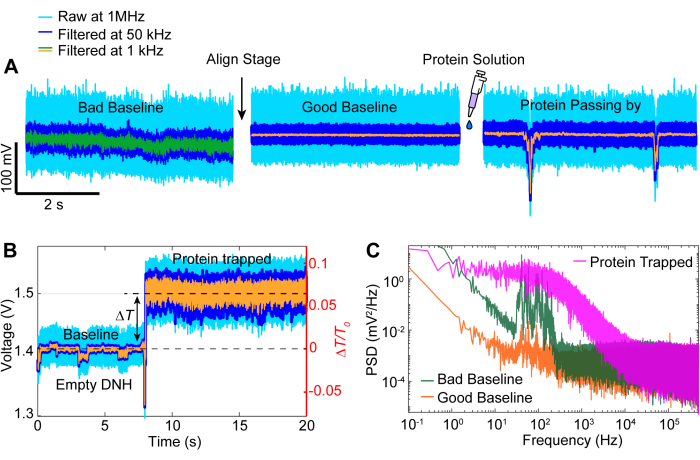
Abbildung 4: Häufige Ablaufverfolgungsereignisse. (A) Beispiele für eine Ausrichtung von einer schlechten zu einer guten Basislinie und ein Protein, das nahe am Hotspot vorbeigeht. (B) Trapping-Trace, die den Prozess von der Baseline, wenn der DNH-Hotspot leer ist, bis zum Zeitpunkt, an dem das Protein gefangen ist, zeigt. (C) Diagramm der spektralen Leistungsdichte (PSD) zwischen den guten und schlechten Ausgangslinien, die in (A) dargestellt sind, und dem in (B) gefangenen Protein. Höhere PSD-Werte weisen auf ein stärkeres Rauschen bei bestimmten Frequenzen hin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die meisten Überfüllungsereignisse folgen dem gleichen allgemeinen Muster wie die Spur in Abbildung 3, obwohl während der Experimente gelegentlich Probleme auftreten können. Bei den meisten Experimenten sollte das Protein manuell freigesetzt werden, indem der Laser ausgeschaltet wird, sobald das gewünschte Experiment abgeschlossen ist. In einigen Fällen kann das Protein die Falle jedoch ohne Eingreifen verlassen, wie in Abbildung 5A gezeigt. Umgekehrt können Proteine manchmal auch nach dem Ausschalten des Lasers an der Fangstelle verbleiben, wahrscheinlich weil das Protein an der Probe haftet. Dieses Verkleben führt zu einer verrauschten Spur nach dem Aus- und Einschalten des Lasers (siehe Abbildung 5B). Die Wahrscheinlichkeit, dass dies geschieht, hängt vom Protein ab, da einige Proteine anfälliger für Oberflächenadsorption sind41,42. Die Verwendung einer Beschichtung wie PEG-Thiol kann die Wahrscheinlichkeit eines Protein-Haftens verringern39,43. Wenn dies nicht gewünscht ist, wie z. B. bei der Untersuchung von Protein-Protein-Wechselwirkungen, ist ein weiteres Problem die doppelte Falle, bei der ein zweites Protein nach der ersten Falle gefangen wird. Diese ist gekennzeichnet durch einen weiteren starken Anstieg der Transmission, ähnlich wie bei der ersten Falle, und eine Änderung der Standardabweichung (siehe Abbildung 5C).
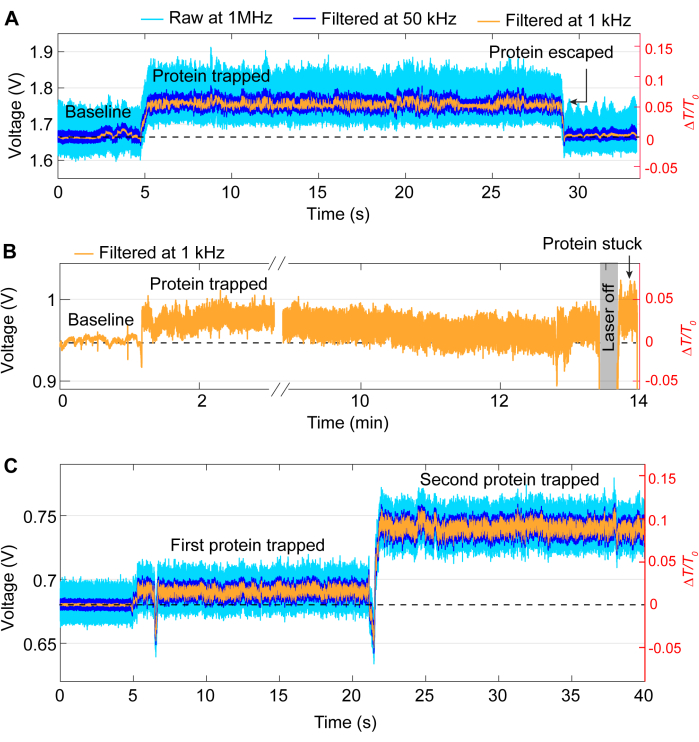
Abbildung 5: Beispiele für unerwünschte Trapping-Ereignisse. (A) Unbeabsichtigte Freisetzung eines Proteins aus dem DNH-Hotspot. (B) Beispiel für ein Protein, das an der Probenoberfläche im DNH-Hotspot hängen bleibt. (C) Ein Spurensprung tritt auf, wenn ein zweites Protein gefangen wird, während das erste noch im DNH-Hotspot verbleibt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ein repräsentatives Experiment, das an der in situ Eisenbeladung eines Apo-Ferritin-Moleküls durchgeführt wurde, zeigt die Verwendung einer plasmonischen Nanopinzette als Werkzeug zur Untersuchung der Konformationsdynamik von Proteinen29. Ferritin ist ein Eisenträgerprotein, das in zwei Zuständen vorliegt: Apo-Ferritin, das kein Eisen enthält, und Holo-Ferritin, das mit Eisengefüllt ist 44,45. Eisen gelangt durch 3-fache Kanäle in das Protein, wo es zu Eisen oxidiert und im Proteinkerngespeichert wird 46. Abbildung 6A zeigt eine typische Einfangspur von Apo-Ferritin mit einer eisenhaltigen Lösung, die über 20 Minuten lang infundiert wird, während das Protein eingeschlossen ist. Die 20-s-Spuren, die entlang der gesamten Spur an den Punkten b-e genommen wurden, geben Aufschluss über die Veränderungen, die am Protein im Laufe der Zeit auftreten. In Abbildung 6B ist Apo-Ferritin in einem Standard-PBS-Puffer gefangen, und es werden keine signifikanten Veränderungen in der Spur beobachtet. Abbildung 6C, D zeigt Fluktuationen in der S.D der Spuren, die durch die Eisenbeladung des Proteins durch seine 3-fachen Kanäle verursacht werden, was zu einem dynamischeren Zustand (Apo-) führt, in dem die Kanäle offen sind, und einem kompakteren Zustand (Holo-) mit geschlossenen Kanälen. Nachdem das Ferritinmolekül mit Eisen gefüllt wurde, ging es in seine Holoform über, was zu einer stabilen Einklemmspur führte, wie in Abbildung 6E gezeigt. Wahrscheinlichkeitsdichtefunktionen (PDF) in Abbildung 6B-E zeigen außerdem die Veränderungen, die das Protein erfährt, wenn es im Laufe der Zeit verschiedenen Lösungsbedingungen ausgesetzt wird.
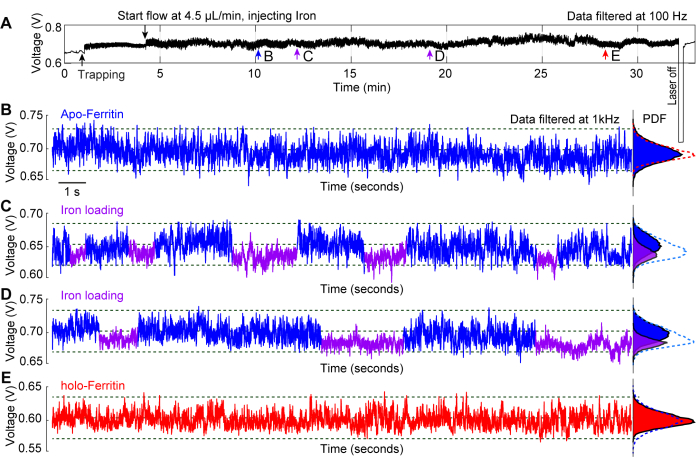
Abbildung 6: In situ Eisenbeladung in ein eingeschlossenes Apoferritin. (A) Vollständige Transmissionsspur eines DNH mit eingeschlossenem Apoferritin-Molekül, gefolgt von der Injektion einer eisenhaltigen Lösung an die Einfangstelle, um die Konformationsänderungen des Ferritins in Verbindung mit der Eisenbeladung zu beobachten. (B) 20-s-Einschlussspur eines Apoferritins, das eingeschlossen wurde, bevor die eisenhaltige Lösung den Hotspot erreichte. (C, D) 20-s-Fangspuren, nachdem das Apoferritinmolekül der eisenhaltigen Lösung ausgesetzt wurde. Blaue und violette Segmente markieren den oberen und unteren S.D. der Spur und weisen auf flexible bzw. starre Ferritinkonformationen hin. (E) 20-s-Fangspur, nachdem Apoferritin >20 Minuten lang der eisenhaltigen Lösung ausgesetzt wurde. Die Diagramme der Wahrscheinlichkeitsdichtefunktion (PDF) auf der rechten Seite zeigen die Verteilung der Übertragung und sind farbcodiert in den blauen und violetten Segmenten. Diese Zahl wurde von29 geändert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Gold-DNH-Probe, die auf der 3D-gedruckten Durchflusszelle montiert ist. Die Probe wird in einen speziellen Schlitz gelegt und mit doppelseitigem PET-Klebeband auf die Durchflusszelle geklebt. Wichtige Parameter und zugehörige Messungen für unser Durchflusszellendesign sind gekennzeichnet. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Rückseite der Durchflusszelle mit goldener DNH-Probe montiert und Innenwand beschriftet. Die Probe wird in der Durchflusszelle mit Dupliziersilikon versiegelt. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3: Diagramm der Durchflusszelle mit goldenem DNH, montiert mit beschrifteten Ein- und Auslasslöchern. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Ein entscheidender Schritt im Protokoll besteht darin, sicherzustellen, dass die Durchflusszelle nicht undicht wird, bevor sie in den Tisch eingesetzt wird, der zuvor außerhalb der Montierung bei einer hohen Durchflussrate getestet werden sollte. Leckagen nach dem Einbetten der Probe können optische Komponenten, insbesondere das untere Objektiv, beschädigen.
Die Ausrichtung kann im Laufe eines Experiments im Laufe eines Experiments von ihrer optimalen Position abweichen, was aufgrund der Empfindlichkeit der plasmonischen Nanopinzette zu Signalvariationen führt. Wenn dies der Fall ist, richten Sie die piezoelektrischen Steuerungen auf maximale Transmission und minimale Standardabweichung der Basislinie aus, da eine verrauschte Basislinie die Datenqualität verringert. Es sollte darauf geachtet werden, dass sorgfältig notiert wird, wann Ausrichtungen vorgenommen werden, um das Risiko einer verwirrenden Benutzerinterferenz durch ein Überfüllungsereignis auszuschließen. Wenn eine größere Drift auftritt, richten Sie den Tisch vorsichtig aus, um das Risiko einer Freisetzung des Proteins zu minimieren, und notieren Sie den Zeitpunkt der Anpassung.
Modifikationen und Modifikationen an der vorgestellten Technik können auf der Grundlage spezifischer experimenteller Anforderungen vorgenommen werden. Zum Beispiel kann ein temperaturgesteuerter Tisch dazu beitragen, die Probe wie gewünscht zu kühlen/zu erwärmen, anstatt die Temperatur durch Laserheizung zu erhöhen31,33. Andere Techniken, beispielsweise die interferometrische Streumikroskopie (iSCAT), können eine Interferenz des Proteins innerhalb des Streufeldes des DNH bereitstellen und so ein zusätzliches Signal erhalten, das proportional zur Polarisierbarkeit des Proteins ist, was mit der Partikelgröße47,48 assoziiert ist.
Bei der plasmonischen Nanopinzette handelt es sich um eine rein zeitliche Sensortechnik, da die Daten vom APD (einem Einzelpixel-Detektor) aufgezeichnet werden. Diese Technik liefert keine direkten Informationen über die strukturellen Veränderungen eines Proteins, z. B. welche Regionen des Proteins an einer Konformationsänderung beteiligt sind oder wo ein Ligand oder ein anderes Protein an das Protein binden kann. Darüber hinaus ist die Technik aufgrund der Abtastrate der Datenerfassungskarte (1 MHz) auf einen zeitlichen Bereich von >1 μs beschränkt. Unter Berücksichtigung der Nyquist-Frequenz, bei der der höchstmögliche Bereich die Hälfte der Erfassungsrate beträgt, in diesem Fall 2 μs unter perfekten Bedingungen.
In diesem Protokoll haben wir den Prozess des Aufbaus eines plasmonischen Nanopinzetten-Experiments beschrieben, um ein einzelnes Protein einzufangen und Veränderungen in seiner Konformationsdynamik im Laufe der Zeit zu überwachen. Die Technik kann auf jedem selbstgebauten oder kommerziellen Mikroskop entwickelt werden. Im Gegensatz zu Fluoreszenz- oder Tethering-basierten Ansätzen, wie z. B. smFRET und optischen Pinzetten, kann die Technik Proteine ohne Markierungen oder Fesseln einfangen und dennoch eine Einzelmolekül-Empfindlichkeit erreichen. Die Lösungsbedingungen können mit Hilfe eines Mikrofluidiksystems geändert werden, während das Protein gefangen ist, was eine Echtzeitüberwachung der Wirkung verschiedener Lösungen auf das Protein ermöglicht. Diese Eigenschaften machen plasmonische Nanopinzetten zu einem vielversprechenden Werkzeug in den Bereichen Biophysik und Biosensorik, insbesondere für Proteine, die mit herkömmlichen Techniken nur schwer in ihrem ursprünglichen Zustand abgefragt werden können. Zukünftige Anwendungen werden sich auf die Entschlüsselung der Konformationsdynamik dynamischerer Proteine konzentrieren, wie z. B. intrinsisch ungeordnete Proteine und Proteine, die intrinsisch ungeordnete Regionen enthalten, sowie Membranproteine, deren Dynamik und sogar Struktur sich den derzeitigen Techniken entziehen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die S.Z. bedankt sich für die Unterstützung durch die Biotechnology and Biological Sciences Research Council Doctoral Training Partnership (BBSRC DTP) (BB/T0083690/1). Die Autoren danken der Finanzierung durch die UK-India Education and Research Initiative (UKIERI). M.R. bedankt sich für die Unterstützung durch die Royal Society und die Wolfson Foundation.
Materialien
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
Referenzen
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495(2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465(2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501(2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995(2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237(2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481(2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983(2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486(2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495(2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten