Method Article
プラズモニックナノピンセットを用いた単一非修飾タンパク質のコンフォメーションダイナミクスのモニタリング
要約
プラズモニックナノピンセットは、金ナノ構造の局在化表面プラズモン共鳴を利用して、ナノメートルスケールの光学場内にタンパク質を含む単一のナノ粒子をトラップします。散乱シグナルの変化により、タンパク質の存在とコンフォメーションダイナミクスが明らかになり、フルオロフォアの修飾や表面テザリングなしでのモニタリングが可能になります。
要約
タンパク質を特徴付けるための現在の単一分子技術では、通常、標識、テザー、または非天然溶液条件の使用が必要です。このような変化は、タンパク質の生物物理学を変化させ、取得したデータの有用性を低下させる可能性があります。プラズモニックナノピンセットは、金ナノ構造上の局在表面プラズモン共鳴(LSPR)を使用して、限られたホットスポット領域内の電場を強化する技術です。この磁場強化により、従来の光ピンセットよりもはるかに小さい、直径わずか数ナノメートルの単一タンパク質など、単一のナノ粒子を低レーザー出力で捕捉することができます。ホットスポット領域内に単一のタンパク質分子をトラップすると、局所的な屈折率(n個のタンパク質 >n個の水)がシフトし、分子の体積、形状異方性、および屈折率の影響を受ける分子の偏光性の積として光散乱が変化します。アバランシェフォトダイオード(APD)は、その後の光散乱の変化を収集します。次に、これらの変化を解析して、トラップされた分子のサイズ、全体的なコンフォメーション、経時的なコンフォメーション変化のダイナミクスなど、トラップされた分子の変化を特定できます。マイクロ流体工学をシステムに組み込むことで、制御された環境変化と、その後の分子への影響のリアルタイムモニタリングが可能になります。このプロトコルでは、プラズモンナノピンセットシステムを使用して、単一のタンパク質分子をトラップし、それらの環境溶液条件を変更し、対応するコンフォメーション変化を監視する手順を示します。
概要
タンパク質のコンフォメーションダイナミクスを調査するための現在の1分子技術には、蛍光共鳴エネルギー移動(FRET)1,2などの標識ベースの方法、光ピンセット3,4や原子間力顕微鏡(AFM)5などのテザリングベースのアプローチ、干渉散乱顕微鏡(iSCAT)6などの干渉ベースの技術、またはナノポア7などのナノ流体ベースの技術が含まれます。8,9.これらの方法には多くの利点がありますが。いくつかの重要な制限により、未修飾のタンパク質コンフォメーションダイナミクスに関するデータを提供することができなくなります。FRETおよび光ピンセットは、蛍光色素の標識または表面へのテザリングを必要とし、これはタンパク質の生物物理学的特性に影響を与える可能性がある10,11,12。iSCATは、技術的にはラベルフリーですが、タンパク質の特性に影響を与える可能性のある2つの間で生成される干渉を観察するために、タンパク質と表面との間の相互作用も必要です。さらに、iSCATは、そのS/N比によって制限され、機器のノイズとスペックルのようなバックグラウンド変動13のために、>40 kDaのタンパク質しか検出できない。このサイズ制限は機械学習によって緩和できるが、バッファ部品は光学特性に影響を与える可能性があるため、データにノイズが多くなる可能性があるため、制限されている13,14。ナノ細孔は、細孔を通るタンパク質の高速な転座時間(通常は5μs以内)を示すため、より遅いコンフォメーションダイナミクスを検出することができない15,16が、ナノ細孔電気浸透トラップ17におけるDNAオリガミの使用やプラズモニクスの組み込みなど、これらの制限を緩和する研究が行われている18,19,20,21 .さらに、高塩濃度(通常は約1 M)は、in vivo研究15,22に対するデータの適用性を低下させる可能性がある。タンパク質の特性評価に理想的な単一分子技術は、タンパク質をリアルタイムでモニタリングし、タンパク質や非天然溶液条件を変更することなく、より長い期間(つまり、ミリ秒)にわたってコンフォメーションダイナミクスを捕捉する必要があります。
プラズモニックナノピンセットは、光を使用して物質を捕捉するという意味で、従来の光ピンセットと似ています。しかし、プラズモニックナノピンセットは、局在表面プラズモン共鳴(LSPR)を利用して電場を数桁増強し、単一のナノ粒子を捕捉するのに十分な強度の勾配力を生成する23。さらに、トラップされた粒子は、ナノアパーチャ構造24の自己誘起逆作用(SIBA)トラップとして知られるトラップの強度を高める上で積極的な役割を果たす。このSIBAトラップにより、低レーザー出力(ミリワット)で、タンパク質25,26,27などの直径わずか数ナノメートルまでの小さな粒子をトラップできます。ホットスポット領域内に単一のタンパク質分子を捕捉すると、局所屈折率(nタンパク質>n水)がシフトし、タンパク質の体積、形状異方性、および屈折率28に影響を受ける分子の分極性に基づいて光散乱が変化する。次に、アバランシェフォトダイオード(APD)がこの情報を検出し、その後の光散乱の変化を監視します。さらに、プラズモニックナノピンセットは、標識、テザー、および過酷な溶液条件なしで、長期間(すなわち、数分から数時間)29にトラップされたタンパク質をリアルタイムでモニタリングすることができ、タンパク質の理想的な単一分子技術の基準を満たしています。プラズモニックナノピンセットは、ダブルナノホール(DNH)構造を用いて、様々なタンパク質を捕捉し、それらからコンフォメーション遷移29、分解速度論30、エネルギーランドスケープ31、拡散追跡32、リガンド結合33,34などの重要な情報を解明する能力を実証してきた。DNH構造の他に、別の構造形状が小さな粒子サイズの粒子を捕捉することが実証されています35,36。このプロトコルでは、統合されたマイクロ流体システムを備えたプラズモニックナノピンセットのセットアップと実行のための基本的な手順が提示されます。このプロトコールが、研究者、特に構造生物学および生物物理学分野の研究者にとって、プラズモンナノピンセットのアクセシビリティと理解を高めるのに役立つことを願っています。
プロトコル
注意: 使用するすべての化学物質に関連するすべての安全データシート(SDS)を読み、すべての適切な安全慣行を順守し、必要に応じて個人用保護具(レーザー安全ゴーグル、実験コート、手袋)を着用してください。
1. プラズモニックナノピンセットのセットアップの構築
注意: 光学セットアップは、異なるレーザーとAPDを利用したモジュラー光ピンセットシステム(OTKB)キットに基づいています(材料の表を参照)。システムへの外部振動の影響を減らすために、適切な光学テーブル上の光学機器のみを使用してください。キットのレーザーは976nmでしたが、DNH構造のウェッジ共振のピーク共振波長は約740〜760nm33です。NIRレーザー(852nm)は、共鳴ピークに近く、LSPRを誘導し、シリコンベースのAPDによる検出歩留まりも優れているため、NIRレーザー(852nm)を選択しました。生体分子を捕捉するために、より長い20波長またはより短い18波長のレーザーが使用されています。
- フォトダイオードレーザーマウントにレーザーをセットし、コリメータに送り込むと、幅1.7mmのコリメートレーザービームが得られます。
- 光路に半波長板を付加し、偏光を調整します。Glan-Taylor偏光子を使用して調整し、垂直偏光(S偏光)の光強度が最も高いことを確認します。
注:DNH構造は分極に依存するため、DNH構造からの電界を最大限に強化するには、正しい分極を使用することが重要です。 - 平凹レンズ(f = -50 mm)と平凸レンズ(f = 150 mm)で構成されるビームエキスパンダーセットアップを介して光を集束させ、ビーム幅を約5 mmに拡大して、下部対物レンズの背面開口部全体を埋めます。
- ダイクロイックミラー(805 nmショートパス)を使用して、レーザーを目的の場所に反射します。その後ろにカメラ(CCD)をセットアップします。
- 光を下部の対物レンズ(100x / 1.25 NA)にリダイレクトし、上部の対物レンズ(4x / 0.1 NA)を共焦点距離に配置して透過光を収集します。
- 別のダイクロイックミラー(805 nmショートパス)を追加して、焦点距離に配置された平凸レンズを介してレーザー光をAPDに向けて反射し、光をAPDに集束させます。ダイクロイックミラーの後ろに白色光源(WLS)を追加して、セットアップを通って戻ってカメラに到達できるようにします。 図1 は、完全に組み立てられたプラズモニックナノピンセットのセットアップの概略図を示しています。
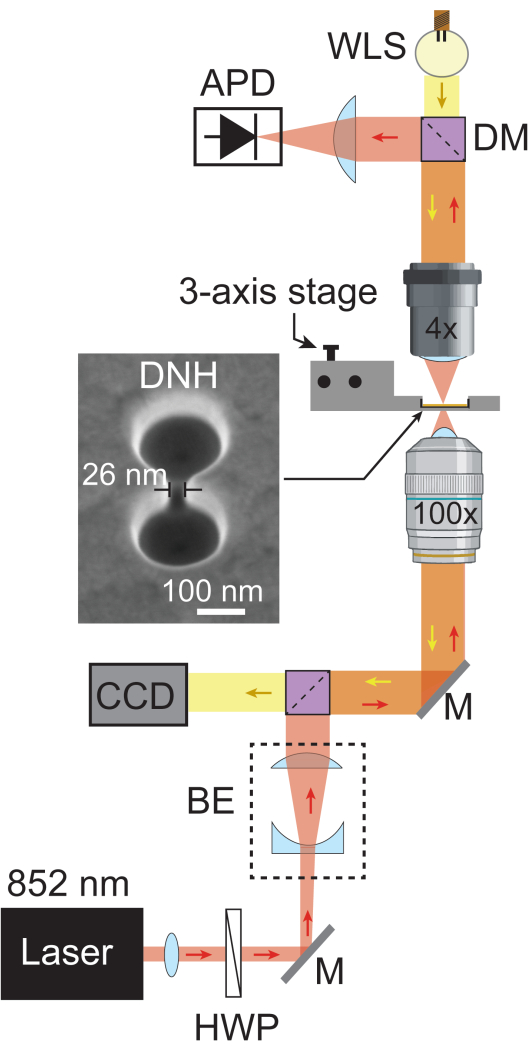
図1:プラズモニックナノピンセットのセットアップ。 この概略図は、プラズモニックナノピンセットのセットアップの完全な光路を示しています。852 nm のレーザー ビーム (赤) は、コリメータと半波長板を通過し、ビーム エキスパンダー (BE) によって ~5.1 mm まで拡大されます。その後、100倍の対物レンズを使用してサンプルに焦点を合わせます。透過したレーザー光は、4倍対物レンズによって収集され、1MHzのサンプリングレートでAPDによって記録されます。WLS(黄色)からの白色光は、4倍対物レンズ、サンプル、および100倍対物レンズを通過してからCCDに到達し、サンプルの明視野画像を提供します。両方の光路が交差する領域はオレンジ色で示されます。20°の傾斜で撮影されたDNHのSEM画像。略語:BE =ビームエキスパンダー、CCD =電荷結合デバイス、DM =ダイクロイックミラー、HWP =半波長板、M =シルバーミラー、WLS =白色光源。 この図の拡大版を表示するには、ここをクリックしてください。
2. DNH構造の作製
- 前述したように、電子ビーム蒸着を使用して、厚さ550μmの石英ウエハーに5 nm Tiを堆積させ、続いて100 nmのAuを堆積させます29,33。ウェーハを1 cm x 1 cmのサンプルにさいの目に切って、すぐに使用できます。
注:電子ビーム蒸着は、表面粗さの低い金層を生成し、高いトラップ効率37で高品質のDNH構造を生成するため、選択されました。 - ブランクの金膜サンプルを取り出し、ガリウムイオン源を使用して室温で走査型電子顕微鏡集束イオンビーム(SEM-FIB)セットアップにマウントします。
- FIBナノアパーチャがSEMと整列し、正確な位置でDNHを生成することを確認します。
- 大電流イオンビーム(例:100pA)を用いてマーカーボックスを作成します。このマーカーは、光学セットアップの下でDNH構造の位置を特定するための参照ガイドとして機能します。
- DNH構造を作成するには、中心間距離が200 nmと直径160 nmの2つの円を長方形(3 nm x 40 nm)で橋渡しします。ギャップサイズが約20〜30nmの場合、線量係数0.09のエッチング円、長方形の線量は0.300〜0.350の間であり、プローブエネルギーは30kV、ビーム電流は1pAです。
注:使用されるSEM-FIBは、このプローブエネルギーで3 nmの分解能を持ち、20〜30 nmのギャップサイズでDNHを信頼性の高い製造できます。低解像度は、ギャップサイズが大きくなり、DNHのトラップ効率が低下するため、使用しません。FIBが実現不可能な場合、DNH構造を製造するための代替方法は、ポリスチレンナノスフィアリソグラフィー38を使用することである。DNH構造の製造には高い精度が求められるため、同じレシピではSEM-FIBの使用間で同じギャップサイズが生成されない場合があります。長方形の線量と高さ(0.280〜0.350線量、高さ2〜4nm)を変更し、ギャップサイズを測定し、約20nmを目指します。
3. DNHサンプルのコーティング
- ホウケイ酸ガラス3.3で作られた結晶化皿などの耐溶剤性容器を使用し、エタノール20mLを追加します。
注意:エタノールは非常に可燃性で刺激性があります。ドラフトでのみ使用してください。涼しく換気の良い場所に保管してください。手袋と白衣を着用して取り扱ってください。 - ポリ(エチレングリコール)メチルエーテルチオール(PEG-チオール;平均分子量800g / mol)の32mgを秤量し、これをエタノールに混合し、すべてのPEG-チオールが溶解して約2mMの濃度の溶液を生成し、単層密度を最大化する39。
注意:PEG-チオールは刺激物です。適切なPPEで取り扱ってください。 - ナノ構造を含むサンプルチップをストレートピンセットを使用して混合物に加え、蓋をして、PEG-チオールが金表面に自己組織化単分子膜を形成するために室温で一晩(~16時間)インキュベートします。
- インキュベーション後、サンプルをドラフト内の適切な容器にまっすぐなピンセットで保持して、サンプルをすすぎます。噴出ボトルを使用して、両側にエタノールをしっかりとスプレーします。その後の保管またはプラズモニックナノピンセットへの取り付け前に、エアガンを使用して完全に乾かしてください。
注:サンプルは時間の経過とともに劣化する可能性があるため、寿命を延ばすために4°Cで保存できます。
4. PEGコーティングされたサンプルをフローセルにマウントする
- コーティングされたサンプルを、金の層を上に向けてストレートピンセットを使用して、3Dプリントされたフローセル(Form 2プリンターでClear V4樹脂でプリント)に入れます(フローセル設計の主要なパラメータと値については 、補足図1 を参照)。
- 透明なPETプラスチック両面テープカバーの片面をストレートピンセットで剥がし、サンプルとフローセルに置き、フローセルのナノ構造とインテーク/アウトテイクホールが覆われないようにします。丸みを帯びたピンセットでテープの端をそっと押して、テープがフローセルとサンプルに適切に接着されていることを確認します。
- テープの反対側をはがし、丸みを帯びたピンセットを使用して、ガラスカバースリップ(厚さ0.17 mm)をサンプルにそっと置きます。丸みを帯びたピンセットでカバーガラスの端をそっと押して、カバーガラスが適切に接着されていることを確認します。これにより、フローセル内に液体チャネル(高さ = 50 μm、体積 = 3.5 μL)が作成されます。
注意: ナノ構造が位置する場所またはその近くでカバースリップを押さないでください。カバーガラスが汚れている場合は、エタノールで洗浄し、エアガンで乾かしてから取り付けてください。 - 複製するシリコーン溶液のAとBの等量を1:1の比率で(または製造元の指示に従って)小さなピペットチップを使用して顕微鏡スライドに混合します。
- カバースリップとフローセルの間の隙間を混合デュプリケーティングシリコンで埋め、カバースリップの下にそっと押し込みます。フローセルを逆さまにし、穴の内壁の周りに複写シリコンを慎重に配置してから、ピペットチップを使用して、サンプルの溶融シリカ下側の可視エッジにフローセルをそっと動かします( 補足図2を参照)。
- 製造元の指示に従って、複製するシリコーンが完全に固まるまで、金の層を上に向けて乾かします。
注:サンプルの下側に塗布する際に、シリコーンがナノ構造を覆わないように注意してください。複製するシリコーンは、フローセルの吸気/排出穴やナノ構造に入る可能性があり、洗浄が困難になる可能性があるため、カバーガラスの下に無理に押し込まないように注意してください(補足図3)。
5. マイクロ流体システムの接続
注意: システムには清潔なチューブを使用していることを確認してください。ここでは、内径小PTFEチューブ:内径0.18mm、内径大PTFEチューブ:0.8mmを使用しますが、それ以外の内径値でも機能します。
- シリンジポンプをセットし、大型のPTFEチューブで3/2ウェイソレノイドバルブに接続します。バルブの一方のポートをバッファー容器に接続し、もう一方のポートを大きなIDチューブから作られた容量約1 mLの保持コイルに接続して、シリンジへの逆流を防ぎます。
注:3/2バルブは、溶液がバッファーコンテナから注入/回収されるか、保持コイルから注入/回収されるかを決定するバルブコントローラーを介して制御できます。 - 保持コイルを12/1回転双方向マイクロ流体バルブの中央バルブに接続します。溶液用の容器は、希少なサンプルや貴重なサンプルには小さなIDチューブを使用し、残りのサンプルには大きなIDチューブを使用して接続します。1つのバルブは廃棄物容器への注入にのみ使用し、もう1つのバルブはフローセルへの注入にのみ使用します。フローセル輸液バルブには小さなIDチューブを使用してください。
- フローセルの出力から廃棄物容器に大型IDチューブを接続します。 図2 は、全結合型マイクロ流体システムの概略図を示しています。
注:スモールIDのPTFEチューブは、フローセルの摂取や、サンプルがチューブ内に留まる体積が小さいため、タンパク質注入に使用されるバルブに適しています。ラージIDのPTFEチューブは、バッファーなどの安価または豊富な材料に適しています。このデッドボリュームをさらに減らすために、最小限のチューブを使用してみてください。
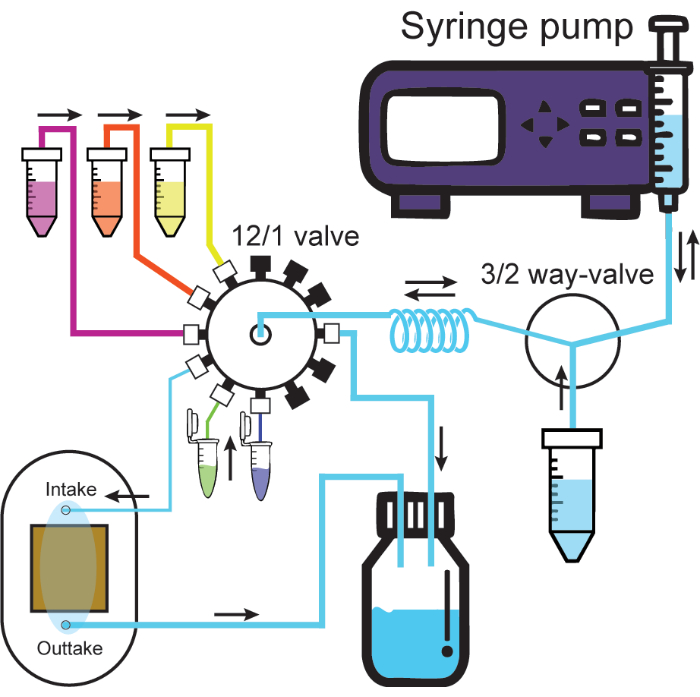
図2:マイクロ流体システム。 回路図は、マイクロ流体システムを示しています。シリンジポンプは、3/2ウェイバルブの1つのポート(溶液容器または保持コイル)を介して溶液を注入または引き出します。12/1バルブに接続された溶液は、引き抜くときに常に保持コイルを通過し、その後、目的のチャネルを介して注入することができます。フローセルに接続されたインテークチャネルに注入すると、フローセルから溶液が廃棄物容器に押し出されます。太いチューブと細いチューブは、プロトコルの大小のIDチューブを表しています。12/1バルブの黒いキャップは、密閉されたチャネルを表しています。フロー方向は黒の矢印でラベル付けされています。 この図の拡大版を表示するには、ここをクリックしてください。
6. マイクロ流体システムの準備
- マイクロ流体システムのUIをPCに読み込み、コンポーネントが正しく接続されていることを確認します。ワイヤーとディストリビューターの横にある再生アイコンを選択して、それぞれのUIを開きます。3/2ウェイバルブをバイパスするには、ワイヤのポート1を回します。
- システム内で使用されるバルブは、イソプロパノール(IPA)を注入して洗浄し、選択したバッファーまたは蒸留水でチューブを複数回洗浄して、システム内の空気とIPAを除去します。輸液は、ディストリビューターUIで目的のバルブを選択し、シリンジポンプを運転し、注入/引き出しを選択し、目的の容量と流量(例:0.5 mLの容量と0.2 mL/分の流量)を選択することで行うことができます。
注:IPAをフローセルに注入すると、セルと接着剤が劣化し、漏れの原因となる可能性があるため、注入しないでください。
7. プラズモンナノピンセットにフローセルを装着し、漏れがないか確認
- インレットチューブとアウトレットチューブをフローセルの適切な部分に取り付けてから、金の層を上に向けてきれいな組織に配置します。
- 高流速(~0.3 mL/分)でフローセルにバッファーを注入し、液体がフローセル内のサンプルを横切って移動すること、およびフローセルの外側または下側に液体が見えないことを確認します。漏れが発生しない限り、より高い流量を使用することができます。サンプルが漏れた場合は、マウントを解除して再マウントします。
- 100倍対物レンズに1〜2滴の浸漬油を加えてから、金層を下に向けてフローセルをプラズモニックナノピンセットステージに配置します。フローセルの磁石に金属製のクリップを取り付け、ステージをロックして所定の位置に保持します。
注意: 客観的なオイルは、刺激性があり、健康に害を及ぼし、水生生物に有毒ですので、手袋を着用してのみ取り扱ってください。
8. サンプル上のナノ構造の位置特定
- 白色光源をオンにします。カメラソフトウェアを開き、ナノ構造が見えるまでゲインと露光時間を増やします。比較的高いレーザー出力でレーザーをオンにし、レーザースポットが見えるまでz軸を手動で調整します。レーザースポットを見つけるためにレーザー出力を上げることよりも、露光時間とゲインを増やすことを優先します。
注意: レーザーは、ユーザーに深刻な潜在的リスクをもたらします。必要な波長範囲内で十分な光学密度を持つレーザー安全ゴーグルなどの適切なPPEが使用されていることを確認してください。 - 圧電コントローラーの電源を入れ、COMポートと最大容量の適切な設定を選択しますtage。X軸、Y軸、Z軸の値を最大電圧の半分に設定して、ステージを全方向に整列させるのに適した範囲を確保します。
注意: 圧電コントローラーのCOMポートは、デバイスマネージャーの[ポート]タブで見つけて変更できます。 - メインステージのx軸、y軸、z軸のコントロールノブを使用して、レーザースポットをDNHの1つと重ね合わせます。APDがオンになっていることを確認してから、筐体をプラズモニックナノピンセットにそっと閉じます。
注:カメラソフトウェアのマーキングツール(利用可能な場合)を使用すると、レーザースポットをナノ構造上に移動するのに役立ちます。マーカーをレーザースポットの中心にセットし、レーザーをオフにしますが、白色光源はオンのままにして、ナノ構造への位置合わせを容易にします。APDは、レーザー光が到達しすぎると、過飽和になり、損傷する可能性があります。サンプルがレーザー経路の邪魔になっていることを確認し、レーザー出力を徐々に増やして飽和に達しないようにします。必要に応じて、APDの前にND(ND)フィルターを配置して、APDに到達する光を減らすことができます。
9. レーザーを目的のナノ構造に最適に整列させる
- APD録音に関連付けられているソフトウェア(自作のLabView UIなど)を開き、カットオフ周波数を1 kHzに設定します。目的のファイルパスと、保存するファイルの命名形式に名前を付けて設定します。
- 白色光源をオフにしてから、レーザーをオンに戻します。適切なレーザー出力(~20 mWなど)に設定し、圧電制御を使用して、APD信号ができるだけ高くなるまでx軸、y軸、z軸を調整します。これにより、APDの飽和を回避し、トレースの標準偏差(SD)が最小限に抑えられます。
注意: APDは、伝送に最適な感度範囲(製品マニュアルを参照)を持っている可能性が高く、これは1000〜2000 mVなどの周囲に留まるのに理想的です。NDフィルターを使用して、透過率をこの範囲に保つことができます。主な考慮事項は、トラッピングによって伝送が飽和限界まで増加し、データの損失につながる可能性があるため、伝送がAPD飽和点に近づかないようにすることです。
10. フローセルへのタンパク質の注入
- ナノ構造の寿命を維持するためにレーザーをオフにし、シリンジポンプ制御ユニットとディストリビューターUIを実行してバルブを設定し、必要な量のタンパク質(30μLなど)を引き出します。スモールIDチューブを使用して回収する場合、低流量ですべての溶液を確実に回収できます(~0.01 - 0.1 mL/分)。必要に応じて、より高い流量を使用して、待ち時間を短縮できます。
注:使用するタンパク質の最適な量は、タンパク質の価値と実験で何を達成したいかによって異なります。私たちが使用する一般的なタンパク質濃度は1μMで、アリコート容量は100μLです。タンパク質サンプルが豊富な場合は、高濃度を使用して平均トラップ時間を短縮できます。しかし、濃度が高すぎると、ナノ構造内に同じタンパク質が2つ閉じ込められるリスクが高まります。実験で最初のタンパク質をトラップした後に異なるリガンド/タンパク質を注入する必要がある場合は、廃棄物と次の溶液の注入にかかる時間を最小限に抑えるために、より少ないアリコート量が好ましい場合があります。空気がシステムに入るのを防ぐために、チューブが溶液に沈んでいることを確認してください。チューブが容器の底に達しない場合、または溶液が過剰に引き出された場合は、タンパク質アリコートに特別な注意を払う必要があります。 - マイクロフルイディクスUIを使用して、離脱速度と同様の流速(~0.01 - 0.1 mL/分)でタンパク質溶液を注入します。タンパク質溶液がフローセルに到達するまでこの速度で注入し、その後、流速を≤0.001mL/minに減らします。シリンジポンプの容量と時間をチェックして、予想される容量と一致していることを確認します(たとえば、1 mL の容量を 1 mL/分で 1 分)。
注:これに必要な容量は、マイクロ流体システムからフローセルまでのチューブの体積に基づいて計算できます。チューブの長さとIDがわかっている場合は、シリンダーの体積のオンライン計算機を使用できます。注入速度は、必要に応じて調整でき、例えば、タンパク質注入の場合は>0.001 mL/minとすることで、トラップの平均待ち時間を短縮することができます。ただし、流量が高すぎると、タンパク質がナノ構造に侵入しなくなる可能性があるため、注意が必要です。
11. データの収集
- タンパク質溶液をフローセルに注入している間に、APDシグナルのデータ記録を開始します。圧電コントローラーUIを使用して、必要に応じてx軸、y軸、z軸を調整します。これは、システムが時間の経過とともにドリフトする可能性があるためです。理想的なトラッピング・トレースは、 図 3 のトレースに続いて一貫した一般的なパターンを持ちます。
- 模範的なトラッピングトレースと同様に、透過率とS.D.の大きな変化を観察した場合は、将来のデータソートのためにこれが発生する時間を書き留めておきます(たとえば、 図4B を参照)。
注:システム内のドリフトは、伝達の変化とSDの増加につながる可能性があり、時には突然、タンパク質トラップと間違われる可能性があります。信号ジャンプが模範トラップと同じパターンを示し、ジャンプが発生したときにドリフトの位置合わせやシステムによる大きなノイズの拾い上げなど、外部からの干渉がなかったことを確認します。 - 実験の一環としてタンパク質を放出する必要がある場合は、レーザーを~5秒間オフにしてから、再度オンにします。トレースは、伝送に大きな変化があり、S.D.が大幅に低下し、ベースライン状態に戻ることを示しています。
注:透過性および/またはS.D.の大きな変化が観察されない場合、またはトラップされるタンパク質と同様の微量が観察される場合、タンパク質はサンプル表面に付着している可能性があります(たとえば、 図5Bを参照)。
12. サンプルのアンマウント
- 目的の実験を行った後、レーザーをオフにし、フローセルを3軸ステージから取り出し、マイクロ流体システムのチューブを外します。
- サンプルの金層を上に向けて、フローセルをきれいな組織の上に置きます。メスを使って、ガラスカバースリップの下の接着剤を慎重に切り、丸みを帯びたピンセットでそっと持ち上げます。指定された割れたガラスのゴミ箱に捨ててください。
注意: ガラスの破片や鋭利なものの使用は、怪我をする可能性があります。安全ゴーグル、手袋、白衣を着用していることを確認してください。 - 金の層が上を向いたままフローセルを斜めに保持し、丸みを帯びたピンセットを使用してフローセルの下側の接着剤を慎重に取り除き、サンプルを解放します。シリコーンを複製することで、DNH構造を損傷することなく簡単に取り外すことができます。
- ストレートピンセットを使用してサンプルを手に取り、IPAで十分にすすぎ、次に水をまいてからエアガンで乾燥させます。再利用可能な場合は、金の層が上を向くようにサンプルを適切な容器に保管してください。
注:サンプルは、適切に処理された場合、通常、約1〜2週間の使用可能なスパンがありますが、ナノ構造のサイズは時間とともに変化します。過度に損傷している場合は、次の実験のために新しいサンプルに切り替えてください。損傷は、ナノ構造体の近くの金表面に存在する引っかき傷の形で、または実験性能の低下によるものであり、ナノ構造体の劣化を示している29。
13. 将来の使用に備えたシステムの準備
- 折りたたんだレンズティッシュを使用して、対物レンズからオイルを一方向に拭き取り、持ち上げて、ティッシュの別のきれいな部分で繰り返します。
注意: 対物レンズはにじみや引っかき傷がつきやすく、パフォーマンスを著しく損なう可能性があります。清掃中は常に手袋を着用し、有機物に触れないようにしてください。汚染を防ぐために新しいレンズクリーニングティッシュのみを使用し、1回の動きで一方向にクリーニングします。損傷を最小限に抑えるために、レンズをこすったり、ティッシュを前後にクリーニングしたりしないでください。ドライクリーニングが不十分な場合は、ティッシュに少量のエタノールを塗布し、前述のように油を拭き取り、ドライティッシュで仕上げます。 - 次に同じタンパク質/バッファー溶液を使用する場合は、チューブを交換する必要がない場合があります。異なる溶液を使用する場合は、チューブを交換して、以前の溶液からの汚染のリスクを減らしてください。
注:同じ溶液で実験を行う場合でも、時間の経過とともに汚れるため、同じチューブを1週間以上使用しないでください。
結果
データ取得後、MATLABコードを使用して生データに対してデータ解析を実行し、APDによって収集された生データからトレースを生成できます。 図3 は、トラップ前のベースライン、レーザーが再びオンになる前に約5秒間オフになる前に透過率(ΔT/T0)と標準偏差の大きな変化が観察されるトラッピングイベントを含む、例示的なトラッピングトレースを示しています。標準偏差の大幅な減少とベースラインと同様のレベルへの感染の回復は、タンパク質の放出を示しています。線形ドリフトは、MATLAB 関数 detrend.m を使用してトレースから削除され、データの平均値がトレンド除去されたトレースに再度追加されます。場合によっては、セットアップが時間の経過とともにドリフトするため、トレースのトレンドを下げる必要があり、伝送が直線的に減少します( 図3の灰色のトレースを参照)。トラップ前後のベースライントレースの小さな変化は、 図4Aに示すように、最小の標準偏差でベースラインを最適化するためのステージ調整によるものです。時々、タンパク質分子はトラップされずにトレースに見える、パスバイタンパク質と呼ばれます。通過するタンパク質は、典型的なトラップ(図4B)と同様に、伝達の急激な変化として現れますが、 図4Aに示すように、持続時間が大幅に短くなります。パワースペクトル密度(PSD)は、さまざまな周波数で信号強度を提供することにより、タンパク質の捕捉を確認する別の分析を提供します。タンパク質の立体配座運動は、典型的には、単一分子分光学法40によって>1μsの範囲で見られる。 図4C は、ベースラインと比較して、タンパク質をトラップすると、少なくとも10kHzの範囲(>100μs)内でシグナル強度が高くなることを示しています。また、ベースラインが悪いと、タンパク質のコンフォメーション運動が重なる周波数範囲である50〜500Hzの周波数でノイズが増加する可能性があるため、ステージを最適化されたベースラインに合わせることの重要性も強調しています。
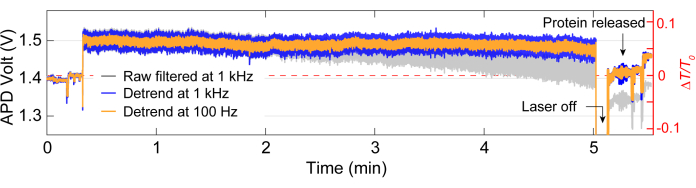
図3:単一タンパク質の完全なトラッピングトレース。 ベースライン、タンパク質のトラップ、タンパク質の放出など、完全なトラップの代表的なトレース。トラッピングの前後のトレースのジャンプは、アライメントによるものです。 この図の拡大版を表示するには、ここをクリックしてください。
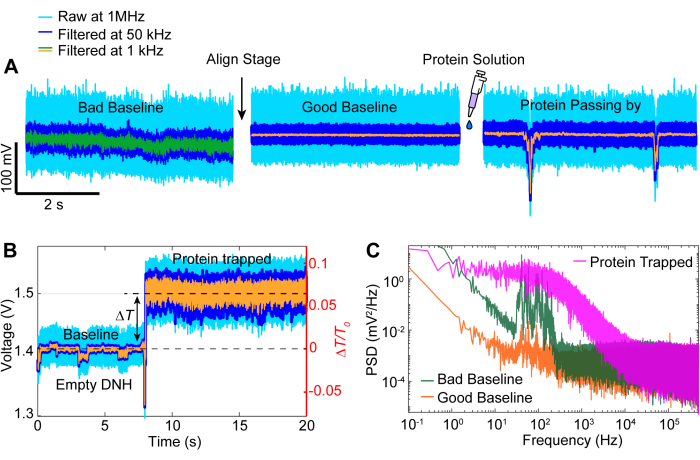
図 4: 一般的なトレース イベント。(A)ベースラインが悪いものから良いものへのアラインメントと、ホットスポットの近くを通過するタンパク質の例。(B)DNHホットスポットが空のベースラインからタンパク質がトラップされるまでのプロセスを示すトラッピングトレース。(C)(A)に示されている良好なベースラインと悪いベースラインと(B)にトラップされたタンパク質との間のパワースペクトル密度(PSD)プロット。PSD値が高いほど、特定の周波数でのノイズが大きいことを示します。この図の拡大版を表示するには、ここをクリックしてください。
ほとんどのトラッピングイベントは、図3のトレースと同じ一般的なパターンに従いますが、実験中に問題が発生することがあります。ほとんどの実験では、目的の実験が完了したら、レーザーをオフにしてタンパク質を手動で放出する必要があります。ただし、場合によっては、図5Aに示すように、タンパク質が介入せずにトラップを離れることがあります。逆に、レーザーをオフにした後でもタンパク質がトラップ部位に残ることがありますが、これはタンパク質がサンプルに付着している可能性があります。この固着により、レーザーをオフにしてからオンにした後、ノイズの多いトレースが発生します (図 5B を参照)。これが起こる可能性はタンパク質に依存し、一部のタンパク質は表面吸着を受けやすい41,42。PEG-チオールなどのコーティングを使用すると、タンパク質が付着する可能性を減らすことができます39,43。タンパク質間相互作用の研究など、望ましくない場合を除き、別の問題は、最初のトラップの後に2番目のタンパク質がトラップされるダブルトラップです。これは、最初のトラップと同様に、透過率がさらに急激に増加し、標準偏差が変化することを特徴としています (図 5C を参照)。
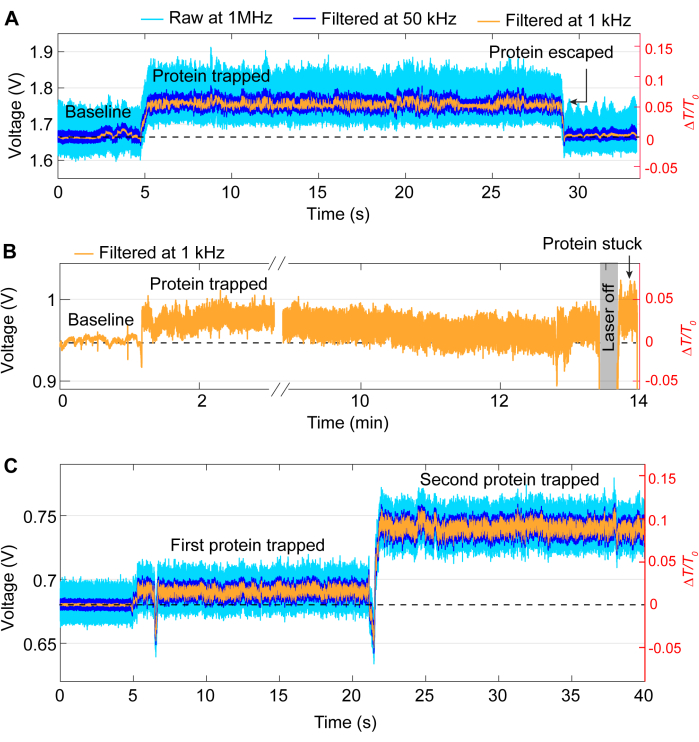
図5:望ましくないトラップイベントの例。(A) DNHホットスポットからのタンパク質の意図しない放出。 (B) DNHホットスポットのサンプル表面にタンパク質が付着した例。 (C) トレースジャンプは、2番目のタンパク質がトラップされ、最初のタンパク質がまだDNHホットスポットに残っている場合に発生します。 この図の拡大版を表示するには、ここをクリックしてください。
アポフェリチン分子へのin situ鉄負荷で行われた代表的な実験は、タンパク質の立体配座ダイナミクスを調査するためのツールとしてのプラズモニックナノピンセットの使用を実証している29。フェリチンは、鉄を含まないアポフェリチンと鉄を充填したホロフェリチンの2つの状態に存在する鉄キャリアタンパク質です44,45。第一鉄は、3倍チャネルを通じてタンパク質に入り込み、そこで第二鉄に酸化され、タンパク質コア46に貯蔵される。図6Aは、タンパク質がトラップされている間に20分以上注入された鉄溶液によるアポフェリチンの典型的なトラップトレースを示しています。ポイントb-eで全体のトレースに沿って取得された20秒のトレースは、時間の経過とともにタンパク質に発生する変化についての洞察を提供します。図6Bでは、アポフェリチンは標準的なPBSバッファーにトラップされており、トレースに大きな変化は観察されません。図6C、Dは、トレースのS.Dの変動を示しており、これは、その3フォールドチャネルを介してタンパク質に鉄がロードされることによって引き起こされ、チャネルが開いているより動的な状態(apo-)と、チャネルが閉じている状態のよりコンパクトな状態(holo-)をもたらします。フェリチン分子が鉄で満たされると、フェリチン分子はホロフォームに移行し、図6Eに示すように安定したトラップトレースが得られます。図6B-Eの確率密度関数(PDF)は、タンパク質がさまざまな溶液条件に経時的にさらされたときに受ける変化をさらに示しています。
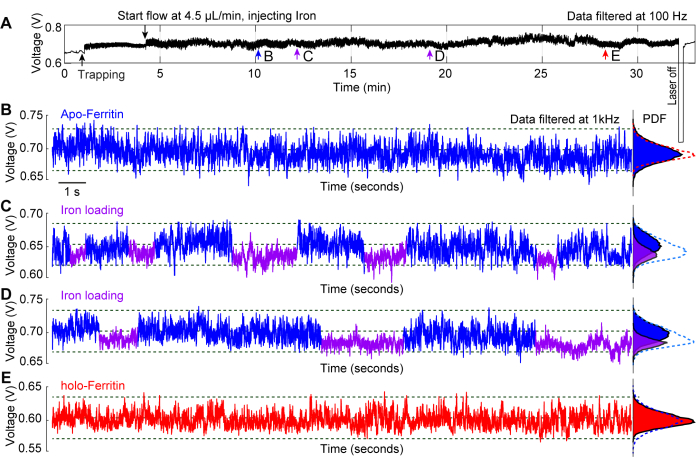
図6:トラップされたアポフェリチンへのin situ鉄のローディング。 (A)アポフェリチン分子をトラップしたDNHの全透過トレースに続いて、そのトラップ部位に鉄溶液を注入して、鉄の負荷に伴うフェリチンのコンフォメーション変化を観察します。(B)鉄溶液がホットスポットに到達する前にトラップされたアポフェリチンの20秒トラップトレース。(C、D)アポフェリチン分子が鉄溶液に曝露された後の20秒のトレースを捕捉します。青と紫のセグメントは、トレースのS.Dの高低を示し、それぞれフェリチンの柔軟なコンフォメーションとリジッドなコンフォメーションを示しています。(E)アポフェリチンを鉄溶液に>20分間曝露した後の20秒間の捕捉痕跡。右側の確率密度関数(PDF)プロットは、透過の分布を示しており、青と紫のセグメントに色分けされています。この数値は平成29年から修正されています。この図の拡大版を表示するには、ここをクリックしてください。
補足図1:3Dプリントされたフローセルに取り付けられた金DNHサンプル。 サンプルを専用のスロットに入れ、両面PET粘着テープを使用してフローセルに接着します。当社のフローセル設計の主要なパラメータと関連する測定値にはラベルが付けられています。 このファイルをダウンロードするには、ここをクリックしてください。
補足図2:金のDNHサンプルが取り付けられ、内壁にラベルが貼られたフローセルの背面。 サンプルは、複写シリコーンを使用してフローセルに密封されます。 このファイルをダウンロードするには、ここをクリックしてください。
補足図3:金のDNHが取り付けられ、吸気孔と排気孔がラベル付けされたフローセルの図。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
このプロトコルの重要なステップは、ステージにセットする前にフローセルが漏れないようにすることです。これは、事前にマウントの外部で高流量でテストする必要があります。サンプルをマウントした後の漏れは、光学部品、特に下部対物レンズに損傷を与える可能性があります。
アライメントは、実験中に時間の経過とともに最適な位置からドリフトする可能性があり、プラズモンナノピンセットの感度による信号の変動を引き起こします。これが発生した場合は、ノイズの多いベースラインがデータの品質を低下させるため、圧電制御を使用してベースラインの最大伝送と最小標準偏差に再調整します。トラッピング イベントに対するユーザーの干渉を混乱させるリスクを排除するために、アラインメントがいつ行われるかについて細心の注意を払ってメモを取るように注意する必要があります。大きなドリフトが発生した場合は、タンパク質が放出されるリスクを最小限に抑えるためにステージをゆっくりと位置合わせし、調整の時間に注意してください。
提示された技術に対する修正および変更は、特定の実験的ニーズに基づいて行うことができる。例えば、温度制御されたステージは、レーザー加熱を使用して温度を上昇させる代わりに、必要に応じてサンプルを冷却/加熱するのに役立つ31,33。他の技術、例えば、干渉散乱顕微鏡(iSCAT)は、DNHの散乱場内でタンパク質の干渉を提供し、粒子サイズ47,48に関連するタンパク質の分極率に比例した追加のシグナルを得ることができる。
プラズモニックナノピンセットは、APD(シングルピクセル検出器)によってデータが記録されるため、純粋に時間センシング技術です。この手法では、タンパク質のどの領域がコンフォメーション変化に関与しているか、リガンドや他のタンパク質がタンパク質に結合する可能性がある場所など、タンパク質の構造変化に関する直接的な情報は提供されません。さらに、この手法は、データ・アクイジション・カードのサンプリング・レート(1MHz)により、>1μsの時間範囲に制限されます。ナイキスト周波数を考慮すると、可能な最大範囲はアクイジションレートの半分であり、この場合は完全な条件下で2μsです。
このプロトコルでは、単一のタンパク質をトラップし、そのコンフォメーションダイナミクスの経時的な変化を監視するためのプラズモンナノピンセット実験を設定するプロセスについて説明しました。この技術は、自作または市販の顕微鏡で開発できます。smFRETや光ピンセットなどの蛍光やテザリングベースのアプローチとは対照的に、この技術は、標識やテザーなしでタンパク質をトラップし、それでも単一分子の感度を達成できます。マイクロ流体システムを使用してタンパク質をトラップしている間に溶液条件を変更できるため、タンパク質に対するさまざまな溶液の影響をリアルタイムで監視できます。これらの特徴により、プラズモニックナノピンセットは、生物物理学およびバイオセンシング分野、特に従来の技術では本来の状態で調査するのが難しいタンパク質にとって有望なツールとなっています。将来のアプリケーションは、天然変性タンパク質や天然変性領域を含むタンパク質、さらには現在の技術ではダイナミクスや構造さえも解明できない膜タンパク質など、よりダイナミックなタンパク質のコンフォメーションダイナミクスの解読に焦点を当てるでしょう。
開示事項
著者は何も開示していません。
謝辞
S.Z.は、バイオテクノロジーおよび生物科学研究評議会博士課程研修パートナーシップ(BBSRC DTP)(BB/T0083690/1)からの支援を認めています。著者らは、UK-India Education and Research Initiative(UKIERI)からの資金提供を認めています。M.R.は、王立協会とウォルフソン財団からの支援に感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
参考文献
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495 (2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465 (2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501 (2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995 (2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237 (2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481 (2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983 (2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486 (2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved