Method Article
플라즈모닉 나노핀셋을 사용한 단일 비변형 단백질의 구조적 역학 모니터링
요약
플라즈몬 나노핀셋은 금 나노 구조에서 국부적인 표면 플라즈몬 공명을 사용하여 나노미터 규모의 광학 필드 내에서 단백질을 포함한 단일 나노 입자를 가둡니다. 산란 신호의 변화는 단백질의 존재와 구조적 역학을 드러내므로 형광단 변형이나 표면 테더링 없이 모니터링할 수 있습니다.
초록
단백질을 특성화하기 위한 현재의 단일 분자 기술은 일반적으로 표지, 테더 또는 비네이티브 용액 조건의 사용을 필요로 합니다. 이러한 변화는 단백질 생물 물리학을 변경하고 수집된 데이터의 유용성을 감소시킬 수 있습니다. 플라즈몬 나노핀셋은 금 나노 구조에 국소 표면 플라즈몬 공명(LSPR)을 사용하여 제한된 핫스팟 영역 내의 전기장을 향상시키는 기술입니다. 이 자기장 향상을 통해 낮은 레이저 출력을 사용하여 기존 광학 핀셋보다 훨씬 작은 단일 나노 입자를 단일 단백질과 같이 직경이 몇 나노미터에 불과한 단일 나노 입자까지 포획할 수 있습니다. 핫스팟 영역 내에서 단일 단백질 분자를 포획하면 국소 굴절률(n단백질 >n 물)의 이동을 유도하여 분자의 부피, 형상 이방성 및 굴절률의 영향을 받는 분자의 분극성의 산물로 광 산란을 변경합니다. APD(avalanche photodiode)는 광 산란의 후속 변화를 수집합니다. 그런 다음 이러한 변화를 분석하여 갇힌 분자의 크기, 전체 형태 및 시간 경과에 따른 구조적 변화의 역학을 포함한 분자의 변화를 결정할 수 있습니다. 시스템 내에 microfluidics를 통합하면 제어된 환경 변화와 분자에 대한 후속 효과의 실시간 모니터링이 가능합니다. 이 프로토콜에서는 플라즈몬 나노핀셋 시스템을 사용하여 단일 단백질 분자를 포획하고, 환경 용액 조건을 변경하고, 해당 구조적 변화를 모니터링하는 단계를 보여줍니다.
서문
단백질 구조적 역학을 조사하기 위한 현재 단일 분자 기술에는 형광 공명 에너지 전달(FRET)1,2과 같은 라벨링 기반 방법, 광학 핀셋 3,4 및 원자력 현미경(AFM)5과 같은 테더링 기반 방법, 간섭 산란 현미경(iSCAT)6과 같은 간섭 기반 기술 또는 나노포어7와 같은 나노유체 기반 기술이 포함됩니다.8,9. 이러한 방법에는 많은 장점이 있습니다. 몇 가지 주요 제한 사항으로 인해 변형되지 않은 단백질 구조 역학에 대한 데이터를 제공할 수 없습니다. FRET 및 광학 핀셋은 형광단 표지 또는 표면에 테더링이 필요하며, 이는 단백질의 생물물리학적 특성에 영향을 미칠 수 있습니다 10,11,12. iSCAT은 기술적으로 표지가 없지만 단백질의 특성에 잠재적으로 영향을 미칠 수 있는 둘 사이에서 생성된 간섭을 관찰하기 위해 단백질과 표면 간의 상호 작용이 필요합니다. 더욱이, 신호 대 잡음비로 인해 제한되는 iSCAT은 장비 잡음 및 반점과 같은 배경 변동으로 인해 단백질 >40 kDa만 검출할 수 있습니다13. 이 크기 제한은 기계 학습을 통해 완화할 수 있지만 버퍼 구성 요소는 광학 속성에 영향을 줄 수 있으므로 제한되어 노이즈가 있는 데이터13,14를 초래할 수 있습니다. Nanopores는 숨구멍을 통해서 단백질의 빠른 전좌 시간을 선물해, nanopore 전기 삼투압 함정17에 있는 DNA 종이접기의 사용 또는 플라스몬ics 18,19,20,21의 통합과 같은 이 제한을 완화하는 것에 대한 연구가 더 느린 구조상 역동성 15,16를 검출할 수 없게 하더라도, 그(것)들을 더 느린 구조상 역동성15,16를 검출할 수 없게 한다. 또한, 일반적으로 약 1M의 높은 염 농도는 생체 내 작업에 대한 데이터의 적용 가능성을 감소시킬 수 있습니다15,22. 단백질 특성 분석을 위한 이상적인 단일 분자 기법은 단백질을 실시간으로 모니터링하고 단백질 또는 비네이티브 용액 조건을 수정할 필요 없이 더 긴 기간(즉, 밀리초)에 걸쳐 구조적 역학을 캡처해야 합니다.
플라즈몬 나노 핀셋은 빛을 사용하여 물질을 가둔다는 점에서 기존의 광학 핀셋과 유사합니다. 그러나 플라즈몬 나노 핀셋은 국부 표면 플라즈몬 공명 (LSPR)을 사용하여 전기장을 몇 배로 향상시켜 단일 나노 입자를 가둘 수있을만큼 강한 구배력을 생성합니다(23). 또한, 포획된 입자는 나노조리개 구조에 대한 SIBA(Self-Induced Back-Action) 포획으로 알려진 트랩의 강도를 향상시키는 데 적극적인 역할을 합니다24. 이 SIBA 트래핑을 사용하면 낮은 레이저 출력(즉, 밀리와트)으로 단백질25,26,27과 같은 직경이 몇 나노미터에 불과한 작은 입자를 포획할 수 있습니다. 핫스팟 영역 내에 단일 단백질 분자를 가두면 국소 굴절률(n단백질 >n 물)이 이동하여 단백질의 부피, 형상 이방성 및 굴절률의 영향을 받는 분자의 분극성에 따라 광 산란이 변경됩니다28. 그런 다음 APD(avalanche photodiode)가 이 정보를 감지하여 광 산란의 후속 변화를 모니터링합니다. 또한, 플라즈몬 나노핀셋을 사용하면 장기간(즉, 몇 분에서 몇 시간) 동안 라벨링, 테더 및 가혹한 용액 조건 없이 포획된 단백질을 실시간으로 모니터링할 수 있으며29 단백질에 대한 이상적인 단일 분자 기술의 기준을 충족합니다. 이중 나노홀(DNH) 구조를 사용하여 플라즈몬 나노핀셋은 구조적 전이29, 분해 역학30, 에너지 경관31, 확산 추적32 및 리간드 결합33,34를 포함하여 다양한 단백질을 가두고 주요 정보를 해명하는 능력을 입증했습니다. DNH 구조 외에도 작은 입자 크기35,36을 가진 입자를 가두는 대체 구조 형상이 입증되었습니다. 이 프로토콜에서는 통합 미세유체역학 시스템으로 플라즈몬 나노핀셋 설정을 설정하고 실행하는 기본 단계가 제시됩니다. 우리는 이 프로토콜이 연구자, 특히 구조 생물학 및 생물 물리학 분야의 연구자들에게 플라즈몬 나노 핀셋에 대한 접근성과 이해를 높이는 데 도움이 되기를 바랍니다.
프로토콜
주의: 사용된 모든 화학 물질에 대한 모든 관련 안전 데이터 시트(SDS)를 읽고 모든 적절한 안전 관행을 준수하고 필요에 따라 개인 보호 장비(레이저 보안경, 실험실 코트, 장갑)를 착용하십시오.
1. 플라즈모닉 나노핀셋 설정 구축
참고: 광학 설정은 다른 레이저와 APD를 사용하는 OTKB(Modular Optical Tweezers System) 키트를 기반으로 합니다(재료 표 참조). 시스템에 대한 외부 진동의 영향을 줄이기 위해 적절한 광학 테이블에서만 광학 장비를 사용하십시오. 키트의 레이저는 976nm였지만 DNH 구조의 쐐기 공진에 대한 최대 공진 파장은 약 740-760nm이므로33. NIR 레이저(852nm)를 선택한 이유는 공진 피크에 가깝고 LSP를 유도하며 실리콘 기반 APD에 의한 검출 수율이 더 우수하기 때문입니다. 20 파장이 더 길거나18 파장이 더 짧은 레이저가 생체 분자를 가두는 데 사용되었습니다.
- 포토다이오드 레이저 마운트에 레이저를 설정하고 콜리메이터에 공급하면 설정에서 너비가 1.7mm인 시준된 레이저 빔이 생성됩니다.
- 빛의 경로에 반파장판을 추가하여 편광을 조정합니다. Glan-Taylor 편광자를 사용하여 수직 편광(S-편광)이 가장 높은 광도를 갖도록 조정합니다.
참고: DNH 구조는 분극에 의존하므로 DNH 구조에서 최대한의 전기장 향상을 보장하기 위해 올바른 분극을 사용하는 것이 중요합니다. - 평면 오목 렌즈(f = -50mm)와 평면 볼록 렌즈(f = 150mm)로 구성된 빔 확장기 설정을 통해 빛의 초점을 맞추어 빔 너비를 약 5mm로 늘려 하단 대물렌즈의 전체 후면 조리개를 채웁니다.
- 이색성 미러(805nm 숏 패스)를 사용하여 레이저를 원하는 위치로 반사합니다. 그 뒤에 카메라(CCD)를 설정합니다.
- 빛을 하단 대물렌즈(100x/1.25 NA)로 리디렉션하고 상단 대물렌즈(4x/0.1 NA)를 공초점 거리로 배치하여 투과된 빛을 수집합니다.
- 또 다른 이색성 미러(805nm 숏 패스)를 추가하여 초점 거리에 위치한 plano-convex lens를 통해 APD를 향해 레이저 광을 반사하여 APD로 빛의 초점을 맞춥니다. 이색성 미러 뒤에 백색 광원(WLS)을 추가하여 설정을 다시 통과하여 카메라에 도달할 수 있도록 합니다. 그림 1 은 완전히 조립된 플라즈몬 나노핀셋 설정의 개략도를 보여줍니다.
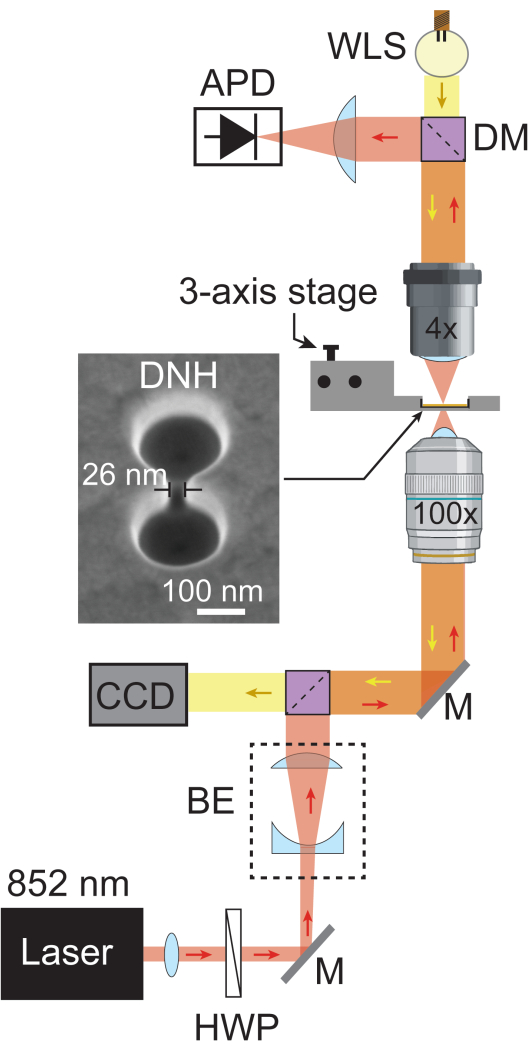
그림 1: 플라즈모닉 나노핀셋 설정. 이 회로도는 플라즈몬 나노핀셋 설정의 전체 광학 경로를 보여줍니다. 852nm 레이저 빔(빨간색)은 시준기와 반파장 플레이트를 통과한 다음 빔 확장기(BE)에 의해 ~5.1mm로 확장됩니다. 그런 다음 100x 대물렌즈를 사용하여 샘플에 초점을 맞춥니다. 투과된 레이저 광은 4x 대물렌즈에 의해 수집되고 1MHz의 샘플링 속도로 APD에 의해 기록됩니다. WLS(노란색)의 백색광은 CCD에 도달하기 전에 4x 대물렌즈, 샘플 및 100x 대물렌즈를 통과하여 샘플의 명시야 이미지를 제공합니다. 두 빛의 경로가 교차하는 영역은 주황색으로 표시됩니다. 20° 기울기에서 촬영한 DNH의 SEM 이미지. 약어: BE = 빔 익스팬더, CCD = 전하 결합 소자, DM = 이색성 미러, HWP = 반파 플레이트, M = 실버 미러, WLS = 백색 광원. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. DNH 구조 제작
- 앞서 설명한 바와 같이 전자빔 증착을 사용하여 550μm 두께의 용융 실리카 웨이퍼에 5nm Ti 후 100nm Au를 증착합니다29,33. 웨이퍼를 1cm x 1cm 샘플로 깍둑썰기하여 사용할 준비가 되었습니다.
참고: 전자빔 증발은 표면 거칠기가 낮은 금층을 생성하여 높은 트래핑 효율을 가진 고품질 DNH 구조를 생성하기 때문에선택되었습니다 37. - 빈 금 필름 샘플을 채취하여 갈륨 이온 소스를 사용하여 실온에서 주사 전자 현미경 집속 이온 빔(SEM-FIB) 설정에 장착합니다.
- FIB 나노 조리개가 SEM과 정렬되어 정확한 위치에서 DNH를 생성하는지 확인합니다.
- 고전류 이온 빔(예: 100pA)을 사용하여 마커 상자를 만듭니다. 이 마커는 광학 설정에서 DNH 구조의 위치를 찾기 위한 참조 가이드 역할을 합니다.
- DNH 구조를 만들기 위해 중심 간 거리가 200nm 및 160nm 인 두 개의 원을 직사각형(3nm x 40nm)으로 연결합니다. 약 20-30nm의 갭 크기의 경우 선량 계수가 0.09인 에칭 원을 그리는 반면, 직사각형 선량은 30kV의 프로브 에너지와 1pA의 빔 전류를 사용하여 0.300 - 0.350입니다.
참고: 사용된 SEM-FIB는 이 프로브 에너지에서 3nm의 분해능을 가지므로 20-30nm 사이의 갭 크기로 DNH를 안정적으로 제작할 수 있습니다. 더 낮은 해상도를 사용하면 더 큰 갭 크기가 생성되어 DNH의 포획 효율이 감소하므로 더 낮은 해상도를 사용하지 않습니다. FIB가 실현 불가능한 경우 DNH 구조를 생성하기 위한 대체 방법은 폴리스티렌 나노스피어 리소그래피38을 사용하는 것입니다. DNH 구조를 제작하는 데 필요한 높은 정밀도로 인해 동일한 레시피가 SEM-FIB 사용 간에 동일한 갭 크기를 생성하지 않을 수 있습니다. 직사각형의 선량과 높이(0.280-0.350 선량 및 2-4nm 높이)를 변경하고 약 20nm를 목표로 갭 크기를 측정합니다.
3. DNH 샘플의 코팅
- 붕규산 유리 3.3으로 만든 결정화 접시와 같은 내용제성 용기를 사용하고 에탄올 20mL를 추가합니다.
주의 : 에탄올은 가연성이 높고 자극성입니다. 흄 후드에서만 사용하십시오. 서늘하고 통풍이 잘 되는 곳에 보관하십시오. 장갑과 실험복으로 다루십시오. - 폴리(에틸렌 글리콜) 메틸 에테르 티올(PEG-티올, 평균 분자량 800g/mol) 32mg의 무게를 달아 에탄올에 혼합하여 모든 PEG-티올이 용해되어 약 2mM 농도의 용액을 생성하여 단층 밀도39를 최대화합니다.
주의: PEG-티올은 자극제입니다. 적절한 PPE로 처리하십시오. - 나노 구조를 포함하는 샘플 칩을 직선 핀셋을 사용하여 혼합물에 추가하고, 뚜껑을 덮고, PEG-thiol이 금 표면에 자체 조립 단층을 형성할 수 있도록 실온에서 하룻밤(~16시간) 배양합니다.
- 배양 후, 흄 후드의 적절한 용기 위에 스트레이트 핀셋으로 샘플을 잡고 샘플을 헹굽니다. 물병을 사용하여 양쪽에 에탄올을 철저히 분사합니다. 후속 보관 또는 플라즈모닉 나노핀셋에 장착하기 전에 에어건을 사용하여 완전히 건조하십시오.
참고: 샘플은 시간이 지남에 따라 저하될 수 있으므로 수명을 향상시키기 위해 4°C에서 보관할 수 있습니다.
4. PEG 코팅된 시료를 플로우 셀(flow cell)에 장착
- 금층이 위를 향하도록 직선 핀셋을 사용하여 코팅된 샘플을 3D 프린팅된 플로우 셀(Clear V4 레진으로 Form 2 프린터로 인쇄)에 넣습니다(플로우 셀 설계의 주요 매개변수와 값은 보충 그림 1 참조).
- 직선 핀셋을 사용하여 투명 PET 플라스틱 양면 테이프 커버의 한쪽 면을 벗겨내고 샘플과 플로우 셀에 올려 놓고 플로우 셀의 나노 구조와 흡입구/배출 구멍이 덮이지 않았는지 확인합니다. 둥근 핀셋으로 테이프의 가장자리 주위를 부드럽게 눌러 플로우 셀과 샘플에 제대로 부착되었는지 확인합니다.
- 테이프의 다른 면을 떼어내고 둥근 핀셋을 사용하여 샘플 위에 유리 커버슬립(두께 0.17mm)을 부드럽게 놓습니다. 둥근 핀셋으로 커버슬립의 가장자리 주위를 부드럽게 눌러 제대로 부착되었는지 확인합니다. 이렇게 하면 플로우 셀 내에 액체 채널(높이 = 50μm, 부피 = 3.5μL)이 생성됩니다.
알림: 나노 구조가 있는 곳이나 근처에서 커버슬립이 손상될 수 있으므로 커버슬립을 누르지 마십시오. 커버슬립이 더러우면 에탄올로 씻고 에어건으로 건조시킨 후 장착하십시오. - 복제 실리콘 용액의 A와 B를 1:1 비율로(또는 제조업체 지침에 따라) 작은 피펫 팁을 사용하여 현미경 슬라이드에 동일한 부분으로 혼합합니다.
- 커버슬립과 플로우 셀 사이의 틈을 혼합 복제 실리콘으로 채우고 커버슬립 아래로 부드럽게 밀어 넣습니다. 플로우 셀(flow cell)을 거꾸로 잡고 구멍 내벽 주위에 복제 실리콘을 조심스럽게 배치한 후 피펫 팁을 사용하여 시료 아래쪽의 용융 실리카의 보이는 가장자리로 부드럽게 이동합니다( 보충 그림 2 참조).
- 제조업체의 지침에 따라 복제 실리콘이 완전히 굳을 때까지 금층이 위쪽을 향하도록 건조시킵니다.
참고: 샘플의 아래쪽에 적용할 때 실리콘이 나노 구조를 덮지 않도록 주의하십시오. 복제 실리콘이 플로우 셀의 흡입구/배출구 또는 나노 구조로 들어갈 수 있으므로 커버슬립 아래로 억지로 밀어 넣지 않도록 주의해야 하며, 이는 세척하기 어려울 수 있습니다(보충 그림 3).
5. 미세유체 시스템 연결
참고: 시스템에 깨끗한 튜브를 사용하는지 확인하십시오. 여기서 작은 ID PTFE 튜브: 0.18mm ID 및 큰 ID PTFE 튜브: 0.8mm가 사용되지만 다른 ID 값도 작동합니다.
- 시린지 펌프를 설정하고 대형 PTFE 튜브를 사용하여 3/2방향 솔레노이드 밸브에 연결합니다. 밸브의 한 포트를 버퍼 용기에 연결하고 다른 포트를 대형 ID 튜브로 만든 약 1mL 용량의 홀딩 코일에 연결하여 주사기로의 역류를 방지합니다.
알림: 3/2 밸브는 밸브 컨트롤러를 통해 제어할 수 있으며, 이는 용액이 버퍼 용기 또는 유지 코일에서 주입/회수되는지 여부를 결정합니다. - 홀딩 코일을 12/1 로터리 양방향 미세유체 밸브의 중앙 밸브에 연결합니다. 용액을 위한 용기를 연결하고, 부족하거나 가치 있는 시료에는 작은 ID 튜빙을 사용하고 나머지는 대형 ID 튜빙을 사용합니다. 하나의 밸브는 폐기물 용기에 주입하기 위해서만 사용하고 다른 밸브는 플로우 셀에 주입하기 위해서만 사용하십시오. 플로우 셀 주입 밸브에 작은 ID 튜브를 사용합니다.
- flow cell의 output에서 waste container까지 대형 ID 튜빙을 연결합니다. 그림 2 는 완전히 연결된 미세유체 시스템의 개략도를 보여줍니다.
참고: 작은 ID PTFE 튜빙은 플로우 셀의 흡입과 샘플이 튜빙에 남아 있는 부피가 작기 때문에 단백질 주입에 사용되는 밸브에 선호됩니다. 대형 ID PTFE 튜빙은 완충액과 같이 저렴하거나 풍부한 재료에 선호됩니다. 이 데드 볼륨을 더 줄이기 위해 최소한의 튜브를 사용하십시오.
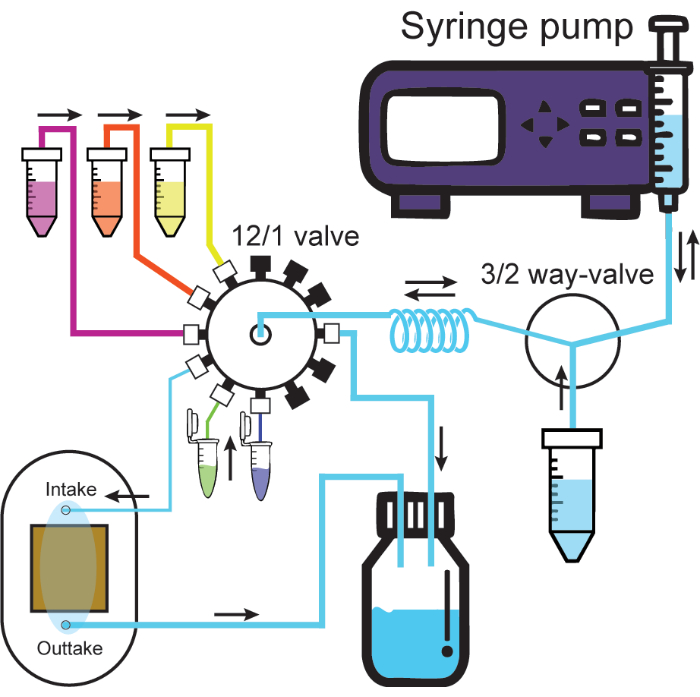
그림 2: 미세유체 시스템. 개략도는 미세유체 시스템을 보여줍니다. 시린지 펌프는 3/2방향 밸브의 한 포트(용액 용기 또는 홀딩 코일)를 통해 용액을 주입하거나 회수합니다. 12/1 밸브에 연결된 용액은 인출될 때 항상 홀딩 코일을 통과한 다음 원하는 채널을 통해 주입될 수 있습니다. flow cell에 연결된 흡입 채널로의 주입은 flow cell에서 폐기물 용기로 용액을 밀어냅니다. 두껍고 얇은 튜브는 프로토콜의 크고 작은 ID 튜브를 나타냅니다. 12/1 밸브의 검은색 캡은 밀폐된 채널을 나타냅니다. 흐름 방향은 검은색 화살표로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
6. 미세유체 시스템 준비
- PC에서 미세유체 시스템 UI를 로드하고 구성 요소가 제대로 연결되어 있는지 확인합니다. 전선 및 분배기 옆에 있는 재생 아이콘을 선택하여 해당 UI를 엽니다. 3/2방향 밸브를 우회하려면 와이어의 포트 1을 돌립니다.
- 시스템 내에서 사용할 밸브를 이소프로판올(IPA)을 주입하여 세척한 다음 선택한 완충액 또는 증류수로 튜브를 여러 번 세척하여 시스템 내의 공기와 IPA를 제거합니다. 주입은 분배기 UI에서 원하는 밸브를 선택하고 주사기 펌프를 실행하고, 주입/회수를 선택하고, 원하는 부피와 유속(예: 0.5mL 부피 및 0.2mL/분 유속)을 선택하여 수행할 수 있습니다.
참고: 플로우 셀(flow cells)에 IPA를 주입하면 셀과 접착제가 저하되어 누출이 발생할 수 있으므로 주입하지 마십시오.
7. 플로우 셀을 플라즈모닉 나노핀셋에 장착하고 누출 확인
- 주입구 및 배출 튜브를 플로우 셀의 해당 부분에 부착한 후 금색 층이 위를 향하도록 하여 깨끗한 조직에 놓습니다.
- 높은 유속(~0.3mL/분)으로 플로우 셀에 완충액을 주입하고 플로우 셀의 시료를 가로질러 유체가 이동하는지, 플로우 셀의 외부 또는 아래쪽에 유체가 보이지 않는지 확인합니다. 누출이 발생하지 않는 한 더 높은 유량을 사용할 수 있습니다. 샘플이 누출되면 마운트 해제했다가 다시 마운트합니다.
- 100x 대물렌즈에 이멀젼 오일 1-2방울을 떨어뜨린 후 골드 층이 아래를 향하도록 하여 플로우 셀을 플라즈몬 나노핀셋 스테이지에 넣습니다. 플로우 셀 자석 위에 금속 클립을 놓고 스테이지를 잠궈 제자리에 고정할 수 있습니다.
주의 : 대물렌즈 오일은 자극적이고 건강에 해롭으며 수생 생물에 유독하며 장갑으로만 취급하십시오.
8. 샘플에서 나노 구조 찾기
- 백색 광원을 켭니다. 카메라 소프트웨어를 열고 나노 구조가 보일 때까지 게인과 노출 시간을 늘립니다. 상대적으로 높은 레이저 출력으로 레이저를 켜고 레이저 스폿이 보일 때까지 z축을 수동으로 조정합니다. 레이저 스폿을 찾기 위해 레이저 출력을 높이는 것보다 노출 시간과 이득을 늘리는 것을 우선시합니다.
주의 : 레이저는 사용자에게 심각한 잠재적 위험을 초래합니다. 필요한 파장 범위 내에서 충분한 광학 밀도를 가진 레이저 보안경과 같은 적절한 PPE가 사용되는지 확인합니다. - 압전 컨트롤러를 켜고 COM 포트 및 최대 볼륨에 대한 적절한 설정을 선택합니다.tage. x축, y축 및 z축의 값을 최대 볼륨의 절반으로 설정합니다.tage를 모든 방향으로 정렬할 수 있는 좋은 범위를 허용합니다.
참고: 압전 컨트롤러의 COM 포트는 포트 탭 아래의 장치 관리자에서 찾아 변경할 수 있습니다. - 메인 스테이지 x축, y축 및 z축 컨트롤 노브를 사용하여 레이저 스폿을 DNH 중 하나로 오버레이합니다. APD가 켜져 있는지 확인한 다음 인클로저를 플라즈모닉 나노핀셋으로 부드럽게 닫습니다.
참고: 사용 가능한 경우 카메라 소프트웨어의 마킹 도구를 사용하면 나노 구조 위로 레이저 스폿을 이동하는 데 도움이 됩니다. 마커를 레이저 스폿의 중앙으로 설정하고 레이저를 끄되 백색 광원은 켜둔 상태로 두어 나노 구조에 더 쉽게 정렬할 수 있습니다. APD는 너무 많은 레이저 광선이 도달하면 과포화되어 손상될 수 있습니다. 샘플이 레이저 경로를 방해하는지 확인하고 채도에 도달하지 않도록 레이저 출력을 점차적으로 높입니다. 중성 농도(N.D) 필터는 APD 앞에 배치하여 필요한 경우 APD에 도달하는 빛을 줄일 수 있습니다.
9. 레이저를 원하는 나노 구조와 최적으로 정렬
- 집에서 만든 Labview UI와 같은 APD 녹음과 관련된 소프트웨어를 열고 차단 주파수를 1kHz로 설정합니다. 원하는 파일 경로와 저장할 파일의 명명 형식을 지정하고 설정합니다.
- 백색 광원을 껐다가 레이저를 다시 켭니다. 적절한 레이저 출력(예: ~20mW)으로 설정하고 압전 컨트롤을 사용하여 APD 신호가 가능한 한 높아질 때까지 x, y 및 z축을 조정하여 APD 포화를 피하고 트레이스의 표준 편차(SD)를 최소화합니다.
알림: APD는 전송을 위한 최적의 감도 범위(제품 설명서 참조)를 가질 가능성이 높으며, 이는 1000-2000mV와 같이 유지하기에 이상적입니다. ND 필터를 사용하여 이 범위 주변의 전송을 유지할 수 있습니다. 주요 고려 사항은 트래핑으로 인해 포화 한계까지 전송이 증가하여 데이터가 손실될 수 있으므로 전송이 APD 포화 지점에 가깝지 않도록 하는 것입니다.
10. 플로우 셀(flow cell)에 단백질 주입
- 나노 구조의 수명을 보존하기 위해 레이저를 끄고 주사기 펌프 제어 장치와 분배기 UI를 실행하여 밸브를 설정하고 원하는 양의 단백질(예: 30μL)을 회수합니다. 소형 ID 튜빙을 사용하여 회수하는 경우 유속이 낮을수록 모든 용액이 회수되도록 하는 데 도움이 됩니다(~0.01 - 0.1 mL/분). 원하는 경우 더 높은 유량을 사용하여 대기 시간을 줄일 수 있습니다.
참고: 사용할 단백질의 최적 부피는 단백질의 가치와 실험이 달성하고자 하는 바에 따라 다릅니다. 우리가 사용하는 일반적인 단백질 농도는 1μM이고 분취 부피는 100μL입니다. 단백질 샘플이 풍부하면 더 높은 농도를 사용하여 평균 포획 시간을 줄일 수 있습니다. 그러나 농도가 너무 높으면 나노 구조 내에서 동일한 단백질 중 두 개를 가두는 위험이 증가합니다. 실험에서 초기 단백질을 포획한 후 다른 리간드/단백질을 주입해야 하는 경우, 낭비를 최소화하고 다음 용액을 주입하는 데 걸리는 시간을 최소화하기 위해 더 낮은 부분 표본 부피를 사용하는 것이 선호될 수 있습니다. 공기가 시스템에 유입되는 것을 방지하기 위해 튜브가 용액에 잠겨 있는지 확인하십시오. 단백질 분취액이 튜브가 용기 바닥에 닿지 않거나 용액이 과도하게 회수된 경우 특별한 주의를 기울여야 합니다. - 미세유체역학(microfluidics) UI를 사용하여 회수 속도(~0.01 - 0.1 mL/분)와 유사한 유속으로 단백질 용액을 주입합니다. 단백질 용액이 플로우 셀(flow cell)에 도달할 때까지 이 속도로 주입한 다음 유속을 ≤0.001mL/분으로 줄입니다. 주사기 펌프의 부피와 시간을 확인하여 예상 부피와 일치하는지 확인합니다(예: 1mL/분에서 1mL 부피에 대해 1분).
참고: 이에 필요한 부피는 미세유체 시스템에서 플로우 셀까지의 튜빙 부피를 기준으로 계산할 수 있습니다. 튜브의 길이와 ID를 알고 있으면 실린더의 부피에 대한 온라인 계산기를 사용할 수 있습니다. 주입 속도는 원하는대로 조정할 수 있습니다 (예 : 단백질 주입의 경우 >0.001 mL / 분)를 통해 트랩의 평균 대기 시간을 줄일 수 있습니다. 그러나 유속이 너무 높으면 단백질이 나노 구조에 들어가는 것을 방지할 수 있으므로 주의하는 것이 좋습니다.
11. 데이터 수집
- 단백질 용액이 플로우 셀(flow cell)에 주입되는 동안 APD 신호의 데이터 기록을 시작합니다. 시간이 지남에 따라 시스템이 드리프트될 수 있으므로 압전 컨트롤러 UI를 사용하여 필요에 따라 x, y 및 z축을 조정합니다. 이상적인 트래핑 트레이스는 그림 3의 트레이스를 따르는 일관된 일반 패턴을 가지고 있습니다.
- 예시적인 트래핑 추적과 유사한 전송 및 S.D.의 큰 변화를 관찰하면 향후 데이터 정렬을 위해 이것이 발생하는 시간을 기록해 두십시오(예: 그림 4B 참조).
참고: 시스템에서 표류하면 전달의 변화와 S.D.의 증가로 이어질 수 있으며, 때로는 매우 갑작스럽게 단백질 트랩으로 오인될 수 있습니다. 신호 점프가 예시 트랩과 동일한 패턴을 표시하고 점프가 발생했을 때 드리프트 정렬 또는 시스템에서 포착되는 큰 소음과 같은 외부 간섭이 없었는지 확인합니다. - 실험의 일환으로 단백질을 방출해야 하는 경우 레이저를 ~5초 동안 껐다가 다시 켜십시오. 추적은 전송의 변화가 크고 S.D.가 현저히 낮아야 하며, 이는 기준선 상태로 복귀했음을 나타냅니다.
참고: 투과 및/또는 S.D.의 큰 변화가 관찰되지 않거나 포획되는 단백질과 유사한 흔적이 관찰되는 경우 단백질이 샘플 표면에 달라붙었을 가능성이 있습니다(예: 그림 5B 참조).
12. 샘플 마운트 해제
- 원하는 실험을 수행한 후 레이저를 끄고 플로우 셀을 3축 스테이지에서 꺼내 미세유체 시스템 튜브를 분리합니다.
- 플로우 셀(flow cell)을 샘플의 금색 층이 위를 향하도록 깨끗한 조직에 놓습니다. 메스를 사용하여 유리 커버슬립 아래의 접착제를 조심스럽게 자르고 둥근 핀셋으로 부드럽게 들어 올립니다. 지정된 깨진 유리 쓰레기통에 버리십시오.
주의 : 깨진 유리와 날카로운 물체를 사용하면 부상을 입을 수 있습니다. 보안경, 장갑 및 실험복을 착용하십시오. - 금층이 여전히 위쪽을 향하도록 flow cell을 비스듬히 잡고 둥근 핀셋을 사용하여 flow cell 아래쪽의 접착제를 조심스럽게 제거하여 샘플을 빼냅니다. 실리콘을 복제하면 DNH 구조를 손상시키지 않고 쉽게 제거할 수 있습니다.
- 스트레이트 핀셋을 사용하여 샘플을 집어 올리고 IPA로 철저히 헹군 다음 물을 뿌린 다음 에어건으로 말리십시오. 재사용할 수 있는 경우 금층이 위를 향하도록 샘플을 적절한 용기에 보관하십시오.
참고: 샘플은 적절하게 취급하면 일반적으로 약 1-2주의 사용 가능한 기간을 갖지만 나노 구조의 크기는 시간이 지남에 따라 변합니다. 과도하게 손상된 경우 다음 실험을 위해 새 샘플로 전환하십시오. 손상은 나노 구조 근처의 금 표면에 존재하는 긁힘의 형태 또는 열악한 실험 성능으로 인해 발생할 수 있으며, 이는 나노 구조의 열화를 나타냅니다(29).
13. 향후 사용을 위한 시스템 준비
- 접힌 렌즈 티슈를 사용하여 대물렌즈의 오일을 한 방향으로 닦아내고 들어 올린 다음 조직의 다른 깨끗한 부분으로 반복합니다.
참고: 대물렌즈는 번지거나 긁히기 쉬워 성능이 크게 저하될 수 있습니다. 청소하는 동안 항상 장갑을 착용하고 유기물을 만지지 마십시오. 새 렌즈 청소용 티슈만 사용하여 오염을 방지하고 한 번의 동작으로 한 방향으로 청소하십시오. 손상을 최소화하기 위해 렌즈를 문지르거나 티슈를 앞뒤로 닦지 마십시오. 드라이 클리닝이 충분하지 않은 경우 티슈에 소량의 에탄올을 바르고 앞서 언급한대로 오일을 닦아내고 마른 티슈로 마무리합니다. - 다음에 동일한 단백질/완충 용액을 사용하는 경우 튜브를 변경할 필요가 없을 수 있습니다. 다른 용액을 사용하는 경우 튜브를 교체하여 이전 용액으로 인한 오염 위험을 줄이십시오.
알림: 동일한 용액으로 실험을 수행하더라도 시간이 지남에 따라 더러워지므로 동일한 튜브를 1주일 이상 사용하지 마십시오.
결과
데이터 수집 후에는 MATLAB 코드를 사용하여 원시 데이터에 대한 데이터 분석을 수행하여 APD에서 수집한 원시 데이터에서 트레이스를 생성할 수 있습니다. 그림 3 은 트래핑 전의 기준선, 레이저가 약 5초 동안 꺼지기 전에 다시 켜지기 전에 투과율(ΔT/T0) 및 표준 편차의 큰 변화가 관찰되는 트래핑 이벤트를 포함한 예시적인 트래핑 추적을 보여줍니다. 표준 편차가 크게 감소하고 기준선과 유사한 수준으로 전파가 돌아오는 것은 단백질의 방출을 나타냅니다. 선형 드리프트는 MATLAB 함수 detrend.m을 사용하여 트레이스에서 제거된 다음, 데이터의 평균값이 추세 제거된 트레이스에 다시 추가됩니다. 때때로, 시간이 지남에 따라 설정이 드리프트됨에 따라 추적의 추세를 제거해야 하며, 이로 인해 전송이 선형적으로 감소합니다( 그림 3의 회색 추적 참조). 트래핑 전후의 베이스라인 트레이스의 작은 변화는 그림 4A에서 볼 수 있듯이 최소 표준 편차로 베이스라인을 최적화하기 위한 스테이지 조정 때문입니다. 때때로, 단백질 분자는 갇히지 않고 흔적에서 볼 수 있으며, 이를 통과 단백질(passing-by protein)이라고 합니다. 지나가는 단백질은 일반적인 트랩(그림 4B)과 유사하게 전달의 급격한 변화로 나타나지만, 그림 4A에서 볼 수 있듯이 지속 시간이 상당히 짧습니다. 파워 스펙트럼 밀도(PSD)는 다양한 주파수에서 신호 강도를 제공하여 단백질 포획을 확인하기 위한 또 다른 분석을 제공합니다. 단백질 구조적 운동은 일반적으로 단일 분자 분광법 방법에 의해 >1μs 범위에서 볼 수 있습니다40. 그림 4C 는 베이스라인과 비교하여 단백질을 포획하면 적어도 10kHz 범위(> 100μs) 내에서 신호 강도가 높아진다는 것을 보여줍니다. 또한 잘못된 베이스라인으로 인해 단백질 구조적 운동이 중첩된 주파수 범위인 50-500Hz 사이의 주파수에서 잡음이 증가할 수 있으므로 스테이지를 최적화된 베이스라인에 정렬하는 것이 중요하다는 점을 강조합니다.
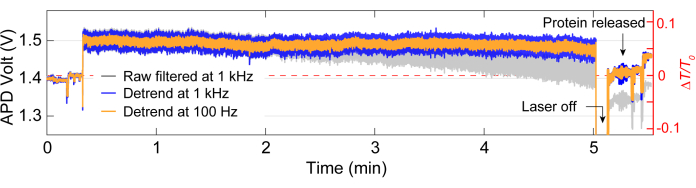
그림 3: 단일 단백질에 대한 전체 포획 추적. baseline, traping a protein, releasing of the protein을 포함한 full trap에 대한 대표적인 추적입니다. 트랩핑 전후의 트레이스 점프는 정렬로 인한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
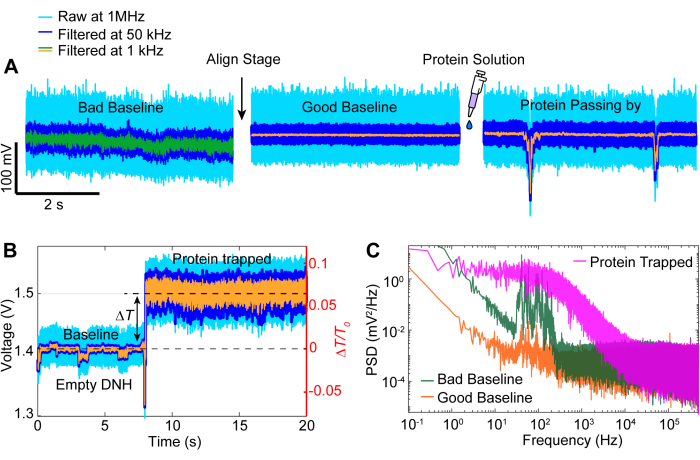
그림 4: 일반적인 추적 이벤트. (A) 나쁜 기준선에서 양호한 기준선으로의 정렬과 단백질이 핫스팟에 가깝게 통과하는 예. (B) DNH 핫스팟이 비어 있을 때 기준선에서 단백질이 포획될 때까지의 과정을 보여주는 트래핑 트레이스. (C) (A)에 묘사된 양호한 기준선과 나쁜 기준선과 (B)에 갇힌 단백질 사이의 전력 스펙트럼 밀도(PSD) 플롯. PSD 값이 높을수록 특정 주파수에서 잡음이 더 크다는 것을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
대부분의 트랩 이벤트는 그림 3의 추적과 동일한 일반적인 패턴을 따르지만 실험 중에 가끔 문제가 발생할 수 있습니다. 대부분의 실험에서 원하는 실험이 완료되면 레이저를 꺼서 단백질을 수동으로 방출해야 합니다. 그러나 일부 경우에는 그림 5A와 같이 단백질이 개입 없이 트랩을 떠날 수 있습니다. 반대로, 때때로 단백질은 레이저를 끈 후에도 포획 부위에 남아 있을 수 있는데, 이는 단백질이 샘플에 달라붙기 때문일 수 있습니다. 이로 인해 레이저를 껐다가 켠 후 잡음이 나는 흔적이 발생합니다(그림 5B 참조). 이러한 현상이 발생할 가능성은 단백질에 따라 달라지는데, 일부 단백질은 표면 흡착에 더 취약하기 때문입니다41,42. PEG-티올과 같은 코팅을 사용하면 단백질이 고착될 가능성을 줄일 수 있습니다39,43. 단백질-단백질 상호 작용 연구와 같이 원하지 않는 한, 또 다른 문제는 첫 번째 트랩 후에 두 번째 단백질이 포획되는 이중 트래핑입니다. 이는 첫 번째 트랩과 유사한 또 다른 급격한 전파 증가와 표준 편차의 변화를 특징으로 합니다(그림 5C 참조).
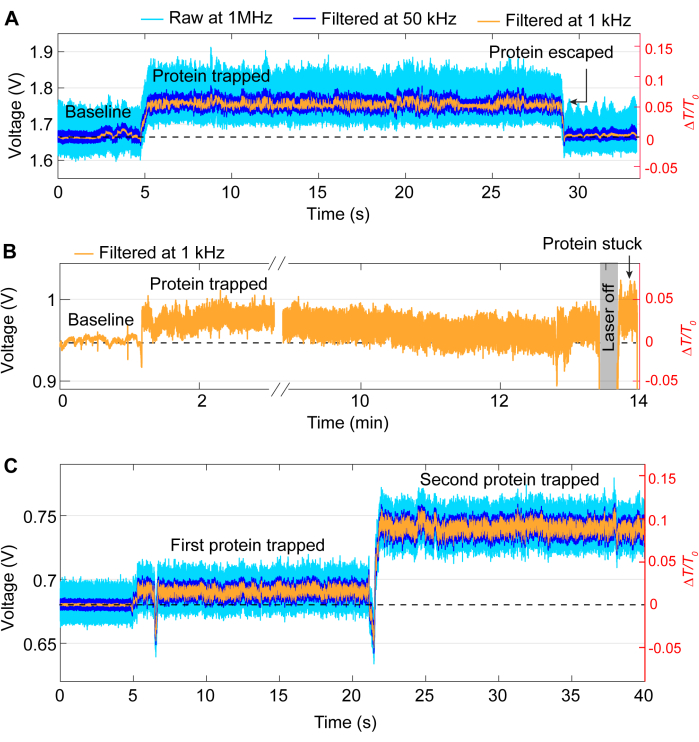
그림 5: 바람직하지 않은 트래핑 이벤트의 예. (A) DNH 핫스팟에서 단백질의 의도하지 않은 방출. (B) DNH 핫스팟의 샘플 표면에 단백질이 달라붙는 예. (C) 트레이스 점프(trace jump)는 첫 번째 단백질이 DNH 핫스팟에 남아 있는 동안 두 번째 단백질이 포획될 때 발생합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
아포-페리틴 분자에 대한 현장 철 로딩으로 수행된 대표적인 실험은 단백질 구조적 역학을 조사하기 위한 도구로 플라즈몬 나노핀셋을 사용하는 것을 보여줍니다29. 페리틴(Ferritin)은 철을 함유하지 않은 아포-페리틴(apo-ferritin)과 철로 채워진 홀로-페리틴(holo-ferritin)의 두 가지 상태로 존재하는 철 운반 단백질입니다44,45. 철은 3중 채널을 통해 단백질로 들어가 철로 산화되어 단백질 코어(46)에 저장됩니다. 그림 6A는 단백질이 포획되는 동안 20분 이상 주입된 철 용액과 함께 아포-페리틴의 일반적인 포획 흔적을 보여줍니다. b-e 지점에서 전체 추적을 따라 취한 20초 추적은 시간이 지남에 따라 단백질에 발생하는 변화에 대한 통찰력을 제공합니다. 그림 6B에서 아포-페리틴은 표준 PBS 버퍼에 갇혀 있으며 추적에서 중요한 변화가 관찰되지 않습니다. 그림 6C, D는 3중 채널을 통해 단백질로 철이 로딩되어 발생하여 채널이 열린 상태에서 보다 동적인 상태(apo-)와 채널이 닫힌 상태에서 더 조밀한 상태(holo-)를 초래하는 트레이스의 S.D 변동을 보여줍니다. 페리틴 분자가 철로 채워지면 홀로폼으로 전이되어 그림 6E와 같이 안정적인 포획 흔적이 생성되었습니다. 그림 6B-E의 확률 밀도 함수(PDF)는 시간이 지남에 따라 단백질이 다양한 용액 조건에 노출될 때 겪는 변화를 보여줍니다.
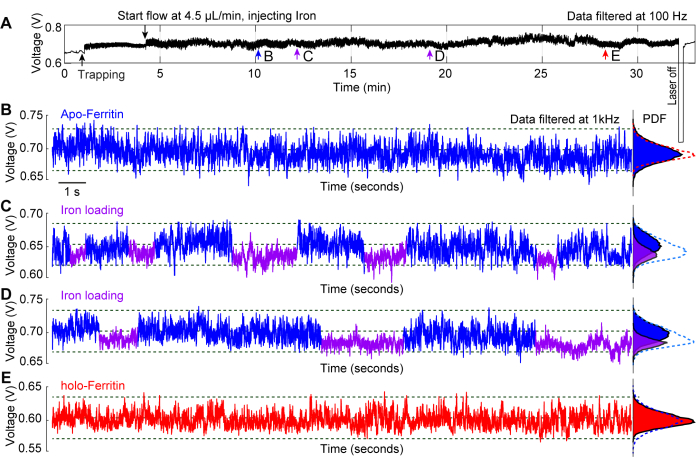
그림 6: 갇힌 아포페리틴에 주입되는 현장 철. (A) 아포페리틴 분자가 포획된 DNH의 전체 투과 추적, 그 후 포획 부위에 철 용액을 주입하여 철 로딩과 관련된 페리틴의 구조적 변화를 관찰합니다. (B) 철 용액이 핫스팟에 도달하기 전에 갇힌 아포페리틴의 20초 포획 흔적. (C, D) 아포페리틴 분자가 철 용액에 노출된 후 20초 트래핑 흔적. 파란색과 자주색 세그먼트는 트레이스의 더 높은 S.D와 더 낮은 S.D를 표시하며, 이는 각각 페리틴의 유연하고 단단한 형태를 나타냅니다. (E) 아포페리틴이 철 용액에 >20분 동안 노출된 후 20초 포획 흔적. 오른쪽의 확률 밀도 함수(PDF) 플롯은 투과 분포를 보여주며 파란색 및 자주색 세그먼트에 색상으로 구분되어 있습니다. 이 수치는29에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 3D 프린팅 플로우 셀에 장착된 금 DNH 샘플. 샘플을 특수 슬롯에 넣고 양면 PET 접착 테이프를 사용하여 플로우 셀에 부착합니다. flow cell 설계를 위한 주요 파라미터 및 관련 측정값이 표시되어 있습니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: 골드 DNH 샘플이 장착되고 내벽에 라벨이 부착된 플로우 셀 뒷면. 샘플은 복제 실리콘을 사용하여 플로우 셀에서 밀봉됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 3: 흡입구 및 배출구 구멍이라고 표시된 금색 DNH가 장착된 플로우 셀의 다이어그램. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
프로토콜의 중요한 단계는 스테이지로 설정되기 전에 플로우 셀이 누출되지 않도록 하는 것이며, 이는 사전에 높은 유속으로 마운트 외부에서 테스트해야 합니다. 샘플이 장착된 후 누출이 발생하면 광학 부품, 특히 하단 대물렌즈가 손상될 수 있습니다.
정렬은 실험 중에 시간이 지남에 따라 최적 위치에서 드리프트되어 플라즈몬 나노핀셋의 감도로 인해 신호 변동을 일으킬 수 있습니다. 이 경우 노이즈가 있는 베이스라인으로 인해 데이터 품질이 저하되므로 압전 제어를 사용하여 베이스라인의 최대 전송 및 최소 표준 편차로 재정렬합니다. 트래핑 이벤트에 대한 사용자 간섭을 혼동할 위험을 제거하기 위해 정렬이 이루어지는 시기를 세심하게 기록해야 합니다. 큰 표류가 발생하는 경우 단백질이 방출될 위험을 최소화하기 위해 스테이지를 부드럽게 정렬하고 조정 시간을 기록하십시오.
제시된 기술에 대한 수정 및 변경은 특정 실험 요구 사항에 따라 이루어질 수 있습니다. 예를 들어, 온도 제어 스테이지는 레이저 가열을 사용하여 온도를 높이는 대신 원하는 대로 샘플을 냉각/가열하는 데 도움이 될 수 있습니다 31,33. 다른 기술, 예를 들어, 간섭계 산란 현미경 (iSCAT)은 DNH의 산란 필드 내에서 단백질의 간섭을 제공하여 입자 크기47,48과 관련된 단백질의 분극성에 비례하는 추가 신호를 얻을 수 있습니다.
플라즈몬 나노핀셋은 데이터가 APD(단일 픽셀 검출기)에 의해 기록되기 때문에 순전히 시간 감지 기술입니다. 이 기술은 단백질의 어느 영역이 구조적 변화에 관여하는지 또는 리간드 또는 다른 단백질이 단백질에 결합할 수 있는 위치와 같은 단백질의 구조적 변화에 대한 직접적인 정보를 제공하지 않습니다. 또한 이 기술은 데이터 수집 카드의 샘플링 속도(1MHz)로 인해 >1μs의 시간 범위로 제한됩니다. 가능한 가장 높은 범위가 획득 속도의 절반인 Nyquist 주파수를 고려할 때, 이 경우 완벽한 조건에서 2μs입니다.
이 프로토콜에서는 단일 단백질을 포획하고 시간 경과에 따른 구조적 역학의 변화를 모니터링하기 위해 플라즈몬 나노핀셋 실험을 설정하는 과정을 설명했습니다. 이 기술은 모든 가정용 또는 상업용 현미경에서 개발할 수 있습니다. smFRET 및 광학 핀셋과 같은 형광 또는 테더링 기반 접근 방식과 달리 이 기술은 표지나 테더 없이 단백질을 포획하면서도 단일 분자 감도를 달성할 수 있습니다. 단백질이 미세유체역학 시스템을 사용하여 포획되는 동안 용액 조건을 변경할 수 있으므로 단백질에 대한 다양한 용액의 영향을 실시간으로 모니터링할 수 있습니다. 이러한 특징으로 인해 플라즈몬 나노핀셋은 생물물리학 및 생물감지 분야에서 유망한 도구가 되었으며, 특히 기존 기술이 원래 상태에서 조사하기 어려운 단백질의 경우 더욱 그렇습니다. 향후 응용 분야는 본질적으로 무질서한 단백질, 본질적으로 무질서한 영역을 포함하는 단백질, 그리고 역학과 구조가 현재 기술을 피하는 막 단백질과 같은 보다 역동적인 단백질의 구조적 역학을 해독하는 데 중점을 둘 것입니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
S.Z.는 생명공학 및 생명 과학 연구 위원회 박사 교육 파트너십(BBSRC DTP)(BB/T0083690/1)의 지원을 인정합니다. 저자는 UKIERI(UKIERI)의 자금 지원을 인정합니다. M.R.은 왕립학회(Royal Society)와 울프슨 재단(Wolfson Foundation)의 지원에 감사하고 있습니다.
자료
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
참고문헌
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495(2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465(2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501(2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995(2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237(2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481(2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983(2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486(2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495(2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유