Method Article
Monitoraggio della dinamica conformazionale di singole proteine non modificate mediante Nanotweezers plasmonici
In questo articolo
Riepilogo
Le nanopinzette plasmoniche utilizzano la risonanza plasmonica di superficie localizzata nelle nanostrutture d'oro per intrappolare singole nanoparticelle, comprese le proteine, all'interno di un campo ottico su scala nanometrica. I cambiamenti nel segnale diffuso rivelano la presenza di proteine e la dinamica conformazionale, consentendo il monitoraggio senza modifiche dei fluorofori o tethering di superficie.
Abstract
Le attuali tecniche a singola molecola per caratterizzare le proteine richiedono tipicamente etichette, cavi o l'uso di condizioni di soluzione non native. Tali cambiamenti possono alterare la biofisica delle proteine e ridurre l'utilità dei dati acquisiti. Le nanotweezers plasmoniche sono una tecnica che utilizza la risonanza plasmonica di superficie localizzata (LSPR) su nanostrutture d'oro per migliorare il campo elettrico all'interno di una regione hotspot confinata. Questo miglioramento del campo consente l'uso di basse potenze laser per intrappolare singole nanoparticelle molto più piccole delle pinzette ottiche convenzionali, fino a pochi nanometri di diametro, come le singole proteine. L'intrappolamento di singole molecole proteiche all'interno della regione hotspot induce uno spostamento dell'indice di rifrazione locale (nproteina >n acqua), alterando la diffusione della luce come prodotto della polarizzabilità della molecola, che è influenzata dal suo volume, forma anisotropia e indice di rifrazione. Un fotodiodo a valanga (APD) raccoglie le successive variazioni nella diffusione della luce. Queste alterazioni possono quindi essere analizzate per determinare i cambiamenti nella molecola intrappolata, comprese le sue dimensioni, la conformazione globale e la dinamica del cambiamento conformazionale nel tempo. L'incorporazione della microfluidica all'interno del sistema consente di controllare i cambiamenti ambientali e di monitorare in tempo reale i loro successivi effetti sulla molecola. In questo protocollo, dimostriamo i passaggi per intrappolare singole molecole proteiche, alterare le loro condizioni di soluzione ambientale e monitorare i loro corrispondenti cambiamenti conformazionali utilizzando un sistema di pinzette plasmoniche.
Introduzione
Le attuali tecniche a singola molecola per interrogare la dinamica conformazionale delle proteine includono metodi basati sull'etichettatura come il trasferimento di energia per risonanza a fluorescenza (FRET)1,2, approcci basati sul tethering come le pinzette ottiche 3,4 e la microscopia a forza atomica (AFM)5, tecniche basate sull'interferenza come la microscopia a dispersione di interferenza (iSCAT)6 o tecniche basate sulla nanofluidica come i nanopori7,8,9. Sebbene questi metodi abbiano molti vantaggi; Alcune limitazioni chiave impediscono loro di fornire dati sulle dinamiche conformazionali delle proteine non modificate. Le pinzette FRET e ottiche richiedono l'etichettatura con fluorofori o il fissaggio a una superficie, che può influire sulle proprietà biofisiche delle proteine 10,11,12. iSCAT, sebbene tecnicamente privo di marcature, richiede anche l'interazione tra la proteina e una superficie per osservare l'interferenza generata tra le due che potenzialmente influisce sulle proprietà delle proteine. Inoltre, limitato dal suo rapporto segnale/rumore, iSCAT è in grado di rilevare solo proteine >40 kDa a causa del rumore dell'apparecchiatura e delle fluttuazioni di fondo simili a macchie13. Sebbene questo limite di dimensioni possa essere alleviato attraverso l'apprendimento automatico, i componenti del buffer sono limitati in quanto possono influire sulle proprietà ottiche, causando dati rumorosi13,14. I nanopori presentano tempi di traslocazione rapidi delle proteine attraverso il poro (di solito entro 5 μs), rendendoli incapaci di rilevare dinamiche conformazionali più lente15,16, sebbene la ricerca per alleviare queste limitazioni, come l'uso di origami di DNA in una trappola elettro-osmotica a nanopori17 o l'incorporazione di plasmonica 18,19,20,21. Inoltre, alte concentrazioni di sale, tipicamente intorno a 1 M, possono ridurre l'applicabilità dei dati per il lavoro in vivo 15,22. La tecnica ideale a singola molecola per la caratterizzazione delle proteine dovrebbe monitorare le proteine in tempo reale e catturare le dinamiche conformazionali per periodi più lunghi (ad esempio, millisecondi) senza la necessità di modifiche alle condizioni della proteina o della soluzione non nativa.
Le nanotweezer plasmoniche sono simili alle pinzette ottiche convenzionali, nel senso che usano la luce per intrappolare la materia. Le nanotweezer plasmoniche, tuttavia, utilizzano la risonanza plasmonica di superficie localizzata (LSPR) per migliorare il campo elettrico di diversi ordini di grandezza per generare una forza di gradiente abbastanza forte da intrappolare singole nanoparticelle23. Inoltre, la particella intrappolata svolge un ruolo attivo nel migliorare la forza della trappola, nota come intrappolamento autoindotto all'indietro (SIBA) per strutture di nanoapertura24. Questo intrappolamento SIBA consente a basse potenze laser (cioè milliwatt) di intrappolare piccole particelle fino a pochi nanometri di diametro, come le proteine 25,26,27. L'intrappolamento di singole molecole proteiche all'interno della regione hotspot provoca uno spostamento dell'indice di rifrazione locale (nproteina > nacqua), alterando la diffusione della luce in base alla polarizzabilità della molecola che è influenzata dal volume della proteina, dalla forma, dall'anisotropia e dall'indice di rifrazione28. Un fotodiodo a valanga (APD) rileva quindi queste informazioni per monitorare i successivi cambiamenti nella diffusione della luce. Inoltre, le nanotweezer plasmoniche consentono il monitoraggio delle proteine intrappolate in tempo reale senza marcatura, cavi e condizioni di soluzione difficili per lunghi periodi di tempo (ad esempio, da minuti a ore)29, soddisfacendo i criteri per una tecnica ideale a singola molecola per le proteine. Utilizzando una struttura a doppio nanoforo (DNH), le nanotweezer plasmoniche hanno dimostrato la loro capacità di intrappolare varie proteine e di chiarire informazioni chiave da esse, tra cui le transizioni conformazionali29, la cinetica di disassemblaggio30, i paesaggi energetici31, il tracciamento della diffusione32 e il legame del ligando33,34. Oltre alle strutture DNH, è stato dimostrato che geometrie di struttura alternative intrappolano particelle con particelle di piccole dimensioni35,36. In questo protocollo vengono presentati i passaggi fondamentali per impostare ed eseguire una configurazione di nanotweezers plasmonici con un sistema microfluidico integrato. Ci auguriamo che questo protocollo contribuisca ad aumentare l'accessibilità e la comprensione delle nanopinzette plasmoniche per i ricercatori, in particolare quelli nel campo della biologia strutturale e della biofisica.
Protocollo
ATTENZIONE: Si prega di leggere tutte le schede di sicurezza (SDS) pertinenti per tutte le sostanze chimiche utilizzate e di attenersi a tutte le pratiche di sicurezza appropriate e di indossare dispositivi di protezione individuale (occhiali di sicurezza laser, camici da laboratorio, guanti) come richiesto.
1. Costruire la configurazione delle nanotweezers plasmoniche
NOTA: La configurazione ottica si basa sul kit Modular Optical Tweezers System (OTKB) che utilizza un laser e un APD diversi (vedere la tabella dei materiali). Utilizzare l'apparecchiatura ottica solo su un tavolo ottico adatto per ridurre l'impatto delle vibrazioni esterne sul sistema. Il laser nel kit era a 976 nm, ma come lunghezza d'onda di risonanza di picco per la risonanza a cuneo delle strutture DNH è di circa 740-760 nm33. Abbiamo scelto un laser NIR (852 nm) perché è vicino al picco di risonanza, induce LSPR e ha anche un migliore tasso di resa di rilevamento grazie all'APD a base di silicio. Laser con lunghezze d'onda più lunghedi 20 opiù corte di 18 sono stati utilizzati per intrappolare le biomolecole.
- Posiziona il laser nella montatura laser a fotodiodo e inseriscilo in un collimatore, che porta a un raggio laser collimato con larghezza di 1,7 mm per la nostra configurazione.
- Aggiungi una piastra a semionda al percorso della luce per regolare la polarizzazione. Regolare utilizzando un polarizzatore Glan-Taylor per garantire che la polarizzazione verticale (polarizzazione S) abbia la massima intensità luminosa.
NOTA: L'uso della polarizzazione corretta è fondamentale per garantire il massimo miglioramento del campo elettrico dalle strutture DNH poiché dipendono dalla polarizzazione. - Focalizzare la luce attraverso una configurazione di espansione del fascio composta da una lente piano-concava (f = -50 mm) seguita da una lente piano-convessa (f = 150 mm) per aumentare la larghezza del fascio a circa 5 mm per riempire l'intera apertura posteriore dell'obiettivo inferiore.
- Utilizzare uno specchio dicroico (805 nm a passaggio corto) per riflettere il laser nella posizione desiderata. Installa la telecamera (CCD) dietro di essa.
- Reindirizzare la luce verso l'obiettivo inferiore (100x/1,25 NA) e posizionare l'obiettivo superiore (4x/0,1 NA) alla distanza confocale per raccogliere la luce trasmessa.
- Aggiungere un altro specchio dicroico (805 nm a passaggio corto) per riflettere la luce laser verso l'APD attraverso una lente piano-convessa posizionata alla sua lunghezza focale per focalizzare la luce nell'APD. Aggiungi la sorgente di luce bianca (WLS) dietro lo specchio dicroico in modo che possa passare attraverso la configurazione per raggiungere la fotocamera. La Figura 1 mostra uno schema della configurazione delle nanotweezers plasmoniche completamente assemblate.
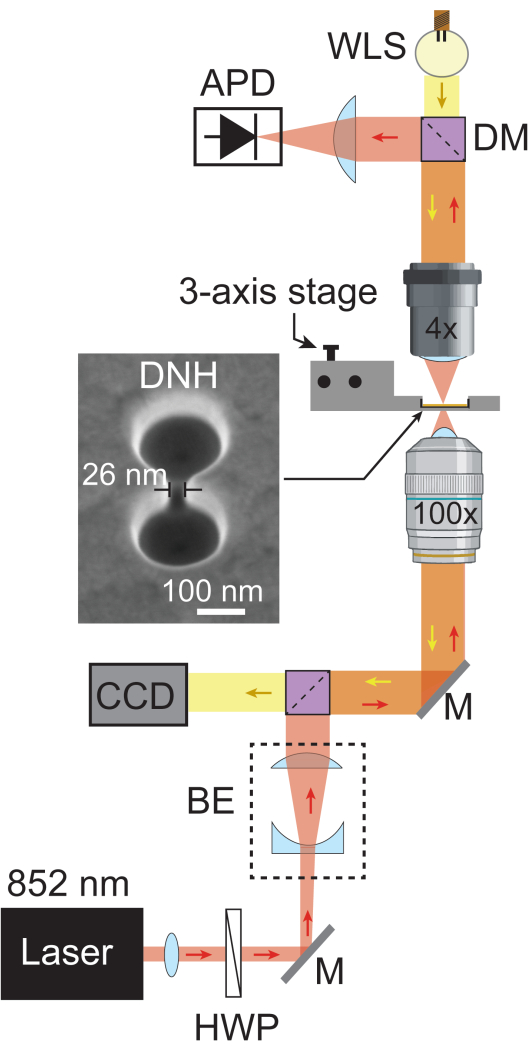
Figura 1: Configurazione delle pinzette plasmoniche. Lo schema mostra l'intero percorso ottico della configurazione plasmonica delle nanotweezer. Un raggio laser da 852 nm (rosso) passa attraverso un collimatore e una piastra a semionda, quindi viene espanso dall'espansore del raggio (BE) a ~5,1 mm. Successivamente viene focalizzato sul campione utilizzando un obiettivo 100x. La luce laser trasmessa viene raccolta da un obiettivo 4x e registrata da un APD a una frequenza di campionamento di 1 MHz. La luce bianca del WLS (gialla) passa attraverso l'obiettivo 4x, il campione e l'obiettivo 100x prima di raggiungere il CCD, che fornisce un'immagine in campo chiaro del campione. Le aree in cui entrambi i percorsi di luce si incrociano sono rappresentate in arancione. Immagine SEM di DNH scattata con un'inclinazione di 20°. Abbreviazioni: BE = Espansore di fascio, CCD = Dispositivo ad accoppiamento di carica, DM = Specchio dicroico, HWP = Piastra a semionda, M = Specchio d'argento e WLS = Sorgente di luce bianca. Clicca qui per visualizzare una versione più grande di questa figura.
2. Fabbricazione delle strutture DNH
- Depositare 5 nm di Ti seguito da 100 nm di Au su un wafer di silice fusa di 550 μm di spessore utilizzando l'evaporazione e-beam come descritto in precedenza 29,33. Tagliare a dadini il wafer in campioni di 1 cm x 1 cm pronti per l'uso.
NOTA: L'evaporazione E-beam è stata scelta in quanto produce uno strato d'oro con bassa rugosità superficiale, producendo strutture DNH di alta qualità con un'elevata efficienza di intrappolamento37. - Prelevare il campione di pellicola d'oro bianco e montarlo in una configurazione a fascio ionico focalizzato su microscopio elettronico a scansione (SEM-FIB) a temperatura ambiente utilizzando una sorgente di ioni gallio.
- Assicurarsi che le nanoaperture FIB siano allineate con il SEM per produrre DNH in posizioni accurate.
- Creare una scatola di marcatura utilizzando un fascio di ioni ad alta corrente (ad esempio, 100 pA). Questo marcatore servirà come guida di riferimento per individuare la posizione delle strutture DNH sotto la configurazione ottica.
- Per creare la struttura DNH, due cerchi con una distanza da centro a centro di 200 nm e un diametro di 160 nm sono collegati da un rettangolo (3 nm x 40 nm). Per una dimensione del gap di circa 20-30 nm, incidere i cerchi con un fattore di dose di 0,09, mentre la dose del rettangolo è compresa tra 0,300 e 0,350 utilizzando un'energia della sonda di 30 kV e una corrente del fascio di 1 pA.
NOTA: Il SEM-FIB utilizzato ha una risoluzione di 3 nm a questa energia della sonda, consentendo una fabbricazione affidabile di DNH con dimensioni del gap comprese tra 20 e 30 nm. Non utilizzeremmo una risoluzione più bassa in quanto ciò produrrebbe spazi di dimensioni maggiori, riducendo l'efficienza di intrappolamento del DNH. Se un FIB non è fattibile, un metodo alternativo per produrre strutture DNH è l'utilizzo della litografia a nanosfere di polistirene38. A causa dell'elevata precisione richiesta per fabbricare strutture DNH, la stessa ricetta potrebbe non produrre la stessa dimensione di spazio tra gli usi SEM-FIB. Modificare la dose e l'altezza del rettangolo (da 0,280 a 0,350 dosi e da 2 a 4 nm di altezza) e misurare la dimensione dello spazio, puntando a circa 20 nm.
3. Rivestimento di campioni DNH
- Utilizzare un contenitore resistente ai solventi, come una capsula di cristallizzazione in vetro borosilicato 3.3, e aggiungere 20 ml di etanolo.
ATTENZIONE: L'etanolo è altamente infiammabile e irritante. Utilizzare solo in cappa aspirante. Conservare in un luogo fresco e ben ventilato. Maneggiare con guanti e camice da laboratorio. - Pesare 32 mg di poli(etilenglicole) metil etere tiolo (PEG-tiolo; peso molecolare medio 800 g/mol) e mescolarlo all'etanolo, assicurandosi che tutto il PEG-tiolo si sia disciolto per produrre una soluzione con una concentrazione di circa 2 mM per massimizzare la densità del monostrato39.
ATTENZIONE: Il PEG-tiolo è un irritante; maneggiarlo con DPI adeguati. - Aggiungere i frammenti di campione contenenti le nanostrutture alla miscela utilizzando una pinzetta diritta, coprire e incubare per una notte (~16 ore) a temperatura ambiente affinché il PEG-tiolo formi un monostrato autoassemblato sulla superficie dell'oro.
- Dopo l'incubazione, sciacquare i campioni tenendoli con una pinzetta diritta su un contenitore adatto nella cappa aspirante. Usa un flacone a spruzzo per spruzzare accuratamente l'etanolo su ciascun lato. Asciugare completamente con una pistola ad aria compressa prima di riporlo o montarlo nelle pinzette plasmoniche.
NOTA: I campioni possono essere conservati a 4 °C per migliorare la longevità in quanto possono degradarsi nel tempo.
4. Montaggio del campione rivestito in PEG in una cella di flusso
- Posiziona il campione rivestito nella cella a flusso stampata in 3D (stampata da una stampante Form 2 con resina Clear V4) utilizzando una pinzetta diritta con lo strato d'oro rivolto verso l'alto (vedi la Figura 1 supplementare per i parametri e i valori chiave nel nostro design della cella a flusso).
- Staccare un lato del coperchio del nastro biadesivo in plastica PET trasparente utilizzando una pinzetta diritta e posizionarlo sul campione e sulla cella di flusso, assicurandosi che le nanostrutture e i fori di aspirazione/uscita nella cella di flusso rimangano scoperti. Premere delicatamente intorno ai bordi del nastro con una pinzetta arrotondata per assicurarsi che aderisca correttamente alla cella di flusso e al campione.
- Staccare l'altro lato del nastro e posizionare delicatamente un vetrino coprioggetti di vetro (spessore 0,17 mm) sul campione utilizzando una pinzetta arrotondata. Premere delicatamente intorno ai bordi del vetrino coprioggetti con le pinzette arrotondate per assicurarsi che aderisca correttamente. Questo crea un canale del liquido (altezza = 50 μm, volume = 3,5 μL) all'interno della cella di flusso.
NOTA: Non premere il vetrino coprioggetti in corrispondenza o vicino al punto in cui si trovano le nanostrutture poiché potrebbero danneggiarsi. Se il vetrino coprioggetti è sporco, lavarlo con etanolo e asciugarlo con una pistola ad aria compressa prima di montarlo. - Mescolare parti uguali di A e B delle soluzioni di silicone per duplicazione in un rapporto 1:1 (o secondo le istruzioni del produttore) su un vetrino da microscopio utilizzando una piccola punta per pipetta.
- Riempire gli spazi tra il vetrino coprioggetti e la cella di flusso con il silicone di duplicazione misto, spingendolo delicatamente sotto il vetrino. Tenere la cella di flusso capovolta e posizionare con cura il silicone per duplicazione attorno alla parete interna del foro prima di utilizzare la punta della pipetta per spostarla delicatamente sui bordi visibili della parte inferiore in silice fusa del campione (vedere la Figura 2 supplementare).
- Lasciare asciugare con lo strato d'oro rivolto verso l'alto fino a quando il silicone da duplicazione non si sarà completamente solidificato, secondo le istruzioni del produttore.
NOTA: Fare attenzione a non permettere al silicone di coprire le nanostrutture quando viene applicato sul lato inferiore del campione. Fare attenzione a non forzare il silicone di duplicazione sotto il vetrino coprioggetti in quanto potrebbe entrare nei fori di aspirazione/uscita della cella a flusso o nelle nanostrutture, che possono essere difficili da pulire (Figura 3 supplementare).
5. Collegamento del sistema microfluidico
NOTA: Assicurarsi che il sistema sia dotato di tubi puliti. In questo caso, verranno utilizzati tubi in PTFE con ID piccolo: 0,18 mm di diametro interno e tubi in PTFE con ID grande: 0,8 mm, ma funzioneranno altri valori ID.
- Installare la pompa a siringa e collegarla all'elettrovalvola a 3/2 vie utilizzando il grande tubo in PTFE. Collegare una porta della valvola al contenitore del tampone e l'altra a una bobina di contenimento, di circa 1 mL di volume, ricavata dal tubo ID grande per evitare il riflusso nella siringa.
NOTA: La valvola 3/2 può essere controllata tramite un controller della valvola, che determina se la soluzione viene infusa/prelevata dal contenitore tampone o dalla bobina di mantenimento. - Collegare la bobina di tenuta alla valvola centrale della valvola microfluidica bidirezionale rotativa 12/1. Collegare i contenitori per le soluzioni utilizzando tubi ID piccoli per campioni scarsi o preziosi e tubi ID grandi per il resto. Utilizzare una valvola solo per l'infusione in un contenitore di scarico e un'altra solo per l'infusione nella cella di flusso; utilizzare un tubo con ID piccolo per la valvola di infusione della cella a flusso.
- Collegare un tubo ID di grandi dimensioni dall'uscita della cella di flusso al contenitore dei rifiuti. La Figura 2 mostra uno schema del sistema microfluidico completamente connesso.
NOTA: Il tubo in PTFE con diametro interno piccolo è preferibile per l'aspirazione della cella a flusso e per le valvole che verranno utilizzate per l'infusione di proteine a causa del volume inferiore in cui il campione rimarrà nel tubo. I tubi in PTFE con diametro interno grande sono preferibili per materiali meno costosi o più abbondanti come i tamponi. Cerca di utilizzare un tubo minimo per ridurre ulteriormente questo volume morto.
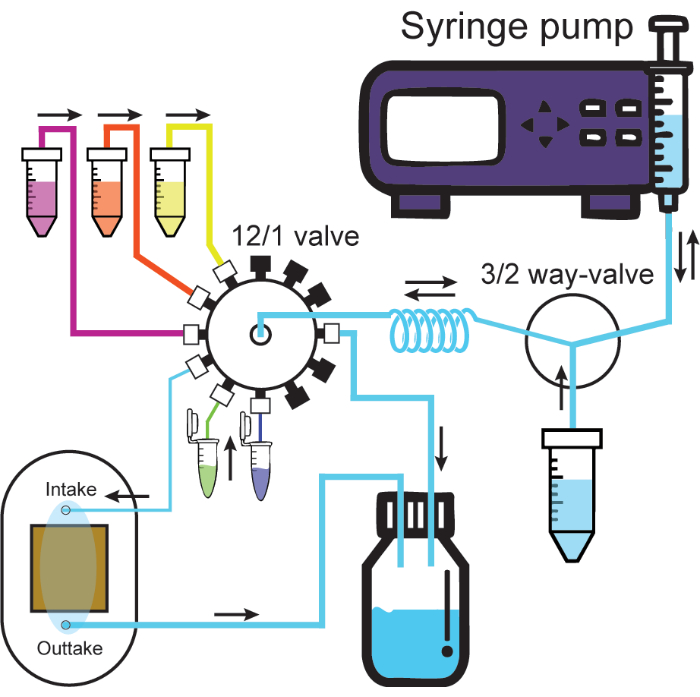
Figura 2: Sistema microfluidico. Lo schema illustra il sistema microfluidico. La pompa a siringa infonde o preleva le soluzioni attraverso una porta della valvola a 3/2 vie, il contenitore della soluzione o la bobina di tenuta. Le soluzioni collegate alla valvola 12/1 passano sempre attraverso la bobina di tenuta quando vengono estratte e possono quindi essere infuse attraverso il canale desiderato. L'infusione nel canale di aspirazione collegato alla cella a flusso spingerà la soluzione dalla cella a flusso nel contenitore dei rifiuti. I tubi spessi e sottili rappresentano tubi ID grandi e piccoli dal protocollo. I cappucci neri sulla valvola 12/1 rappresentano i canali sigillati. Le direzioni del flusso sono contrassegnate da frecce nere. Clicca qui per visualizzare una versione più grande di questa figura.
6. Preparazione del sistema microfluidico
- Caricare l'interfaccia utente del sistema microfluidico sul PC e verificare che i componenti siano collegati correttamente. Seleziona l'icona di riproduzione accanto al cavo e al distributore per aprire la rispettiva interfaccia utente. Per bypassare la valvola a 3/2 vie, ruotare la porta 1 sul filo.
- Assicurarsi che le valvole che verranno utilizzate all'interno del sistema siano pulite infondendo isopropanolo (IPA) e quindi lavare i tubi più volte con tampone a scelta o acqua distillata per rimuovere l'aria e l'IPA all'interno del sistema. L'infusione può essere effettuata selezionando la valvola desiderata sull'interfaccia utente del distributore e facendo funzionare la pompa a siringa, selezionando infondere/prelevare e scegliendo il volume e la portata desiderati (ad esempio, 0,5 mL di volume e 0,2 mL/min di portata).
NOTA: Non infondere l'alcool isopropilico nelle celle a flusso, poiché ciò degraderebbe la cella e la colla, causando perdite.
7. Montaggio della cella di flusso in nanotweezer plasmonici e controllo delle perdite
- Collegare i tubi di ingresso e di uscita alla parte appropriata della cella a flusso prima di posizionarli su un fazzoletto pulito con uno strato d'oro rivolto verso l'alto.
- Infuse il tampone nella cella a flusso con una portata elevata (~0,3 mL/min) e verificate che il fluido si muova attraverso il campione nella cella a flusso e che non sia visibile alcun fluido all'esterno o sul lato inferiore della cella a flusso. È possibile utilizzare portate più elevate, a condizione che non si verifichino perdite. Se il campione perde, smontare e rimontare.
- Aggiungere 1-2 gocce di olio da immersione sull'obiettivo 100x prima di posizionare la cella di flusso nello stadio di pinzetta plasmonica con lo strato d'oro rivolto verso il basso. Posiziona le clip metalliche sopra i magneti delle celle di flusso e blocca il tavolino per mantenerlo in posizione.
ATTENZIONE: L'olio obiettivo è irritante, pericoloso per la salute e tossico per la vita acquatica, maneggiare solo con i guanti.
8. Localizzazione di nanostrutture su campione
- Accendi la sorgente di luce bianca. Apri il software della fotocamera e aumenta il guadagno e il tempo di esposizione fino a quando le nanostrutture non sono visibili. Accendere il laser con una potenza laser relativamente elevata e regolare manualmente l'asse z fino a quando il punto laser non è visibile. Dai la priorità all'aumento del tempo di esposizione e del guadagno rispetto all'aumento della potenza del laser per trovare il punto laser.
ATTENZIONE: I laser rappresentano un serio rischio potenziale per gli utenti. Assicurarsi che vengano utilizzati DPI appropriati, come occhiali di sicurezza laser, con densità ottica sufficiente entro l'intervallo di lunghezze d'onda richiesto. - Accendere il controller piezoelettrico e selezionare le impostazioni appropriate per la porta COM e la tensione massima. Impostare i valori per gli assi x, y e z a metà della tensione massima per consentire un buon intervallo per allineare il tavolino in tutte le direzioni.
NOTA: La porta COM del controller piezoelettrico può essere trovata e modificata in Gestione dispositivi nella scheda delle porte. - Spostare lo spot laser in sovrapposizione con uno dei DNH utilizzando le manopole di controllo degli assi x, y e z dello stadio principale. Assicurarsi che l'APD sia acceso, quindi chiudere delicatamente l'involucro alle pinzette plasmoniche.
NOTA: L'utilizzo di uno strumento di marcatura nel software della fotocamera, se disponibile, aiuterà a spostare il punto laser su una nanostruttura. Posiziona il marcatore al centro del punto laser e spegni il laser, ma lascia accesa la sorgente di luce bianca per consentire un più facile allineamento a una nanostruttura. L'APD può essere sovrasaturo e danneggiato se viene raggiunto da troppa luce laser. Assicurarsi che il campione sia d'intralcio al percorso del laser e aumentare gradualmente la potenza del laser per garantire che la saturazione non venga raggiunta. I filtri a densità neutra (N.D) possono essere posizionati prima dell'APD per ridurre la luce che lo raggiunge, se necessario.
9. Allineamento ottimale del laser con la nanostruttura desiderata
- Aprire il software associato alla registrazione APD, come ad esempio un'interfaccia utente Labview fatta in casa, e impostare la frequenza di taglio a 1 kHz. Assegna un nome e imposta il percorso del file desiderato e il formato di denominazione dei file che verranno salvati.
- Spegnere la sorgente di luce bianca e riaccendere il laser. Impostare su una potenza laser appropriata (ad esempio, ~20 mW) e utilizzare i controlli piezoelettrici per regolare gli assi x, y e z fino a quando il segnale APD non è il più alto possibile, evitando la saturazione APD e con una deviazione standard minima (S.D.) della traccia.
NOTA: L'APD avrà probabilmente un intervallo di sensibilità ottimale per la trasmissione (vedere il manuale del prodotto) che è l'ideale per rimanere intorno, ad esempio 1000-2000 mV. I filtri N.D. possono essere utilizzati per mantenere la trasmissione intorno a questo intervallo. La considerazione principale è che la trasmissione non sia vicina al punto di saturazione APD, poiché l'abbondanza può aumentare la trasmissione fino al limite di saturazione, con conseguente perdita di dati.
10. Infondere proteine nella cella di flusso
- Spegnere il laser per preservare la durata delle nanostrutture ed eseguire l'unità di controllo della pompa a siringa e l'interfaccia utente del distributore per impostare la valvola e prelevare la quantità desiderata di proteine (ad esempio, 30 μL). Per il prelievo con il tubo con diametro interno, una velocità di flusso inferiore aiuta a garantire che tutte le soluzioni vengano prelevate (~0,01 - 0,1 mL/min). Se lo si desidera, è possibile utilizzare portate più elevate per ridurre il tempo di attesa.
NOTA: Il volume ottimale di proteine da utilizzare dipende dal valore della proteina e da ciò che l'esperimento spera di ottenere. La concentrazione tipica di proteine che utilizziamo è di 1 μM e un volume aliquota di 100 μL. Se il campione di proteine è abbondante, è possibile utilizzare concentrazioni più elevate per ridurre il tempo medio di intrappolamento. Tuttavia, se le concentrazioni sono troppo elevate, il rischio di intrappolare due delle stesse proteine all'interno della nanostruttura aumenterà. Se l'esperimento richiede un'infusione di un ligando/proteina diverso dopo aver intrappolato la proteina iniziale, si può preferire un volume aliquota inferiore per ridurre al minimo gli sprechi e il tempo necessario per infondere la soluzione successiva. Assicurarsi che il tubo rimanga immerso nella soluzione per evitare che l'aria entri nel sistema. Prestare particolare attenzione alle aliquote proteiche se il tubo non raggiunge il fondo del contenitore o se la soluzione viene prelevata eccessivamente. - Utilizzando l'interfaccia utente microfluidica, infondere la soluzione proteica a una velocità di flusso simile alla velocità di prelievo (~0,01 - 0,1 mL/min). Lasciare in infusione a questa velocità fino a quando la soluzione proteica raggiunge la cella di flusso, quindi ridurre la velocità di flusso a ≤0,001 mL/min. Controllare il volume e il tempo sulla pompa a siringa per assicurarsi che corrispondano al volume previsto (ad esempio, 1 min per un volume di 1 ml a 1 ml/min).
NOTA: Il volume richiesto per questo può essere calcolato in base al volume del tubo dal sistema microfluidico alla cella di flusso. Se la lunghezza e l'ID del tubo sono noti, è possibile utilizzare un calcolatore online per il volume di un cilindro. La velocità di infusione può essere regolata a piacere, ad esempio >0,001 ml/min per l'infusione di proteine, per ridurre il tempo medio di attesa per una trappola. Tuttavia, si consiglia cautela in quanto se la velocità di flusso è troppo elevata, ciò potrebbe impedire alle proteine di entrare nella nanostruttura.
11. Raccolta dei dati
- Mentre la soluzione proteica viene infusa nella cella di flusso, avviare la registrazione dei dati del segnale APD. Regolare gli assi x, y e z in base alle esigenze utilizzando l'interfaccia utente del controller piezoelettrico, poiché è probabile che il sistema si sposti nel tempo. Le tracce di trapping ideali hanno un modello generale coerente che segue quello della traccia nella Figura 3.
- Dopo aver osservato un grande cambiamento nella trasmissione e nella S.D. simile alla traccia di intrappolamento esemplare, annotare il momento in cui ciò si verifica per il futuro ordinamento dei dati (vedere la Figura 4B per esempio).
NOTA: La deriva nel sistema può portare a cambiamenti nella trasmissione e ad un aumento della S.D., a volte abbastanza improvvisamente, che potrebbe essere scambiata per una trappola proteica. Assicurarsi che il salto del segnale visualizzi lo stesso schema della trappola esemplare e che non fossero presenti interferenze esterne quando si è verificato il salto, come deriva di allineamento o rumori forti rilevati dal sistema. - Se la proteina deve essere rilasciata come parte dell'esperimento, spegnere il laser per ~5 s e riaccenderlo. Il tracciato dovrebbe avere un grande cambiamento nella trasmissione e un S.D. significativamente più basso, indicando un ritorno allo stato basale.
NOTA: Se non si osserva un grande cambiamento nella trasmissione e/o nella S.D., o si osserva una traccia simile alla proteina intrappolata, è probabile che la proteina sia attaccata alla superficie del campione (vedere la Figura 5B, ad esempio).
12. Smontaggio del campione
- Dopo aver eseguito l'esperimento desiderato, spegnere il laser, estrarre la cella di flusso dallo stadio a 3 assi e scollegare il tubo del sistema microfluidico.
- Posizionare la cella di flusso su un tessuto pulito con lo strato d'oro del campione rivolto verso l'alto. Usando un bisturi, taglia con cura la colla sotto il vetrino coprioggetti e sollevala delicatamente con una pinzetta arrotondata. Smaltirlo nell'apposito cestino per vetri rotti.
ATTENZIONE: I vetri rotti e l'uso di oggetti appuntiti possono causare lesioni. Assicurarsi che siano indossati occhiali di sicurezza, guanti e un camice da laboratorio. - Tenere la cella di flusso inclinata con lo strato d'oro ancora rivolto verso l'alto e utilizzare una pinzetta arrotondata per rimuovere con cura la colla sul lato inferiore della cella di flusso per liberare il campione. Il silicone per duplicazione consente una facile rimozione senza danneggiare le strutture DNH.
- Usando una pinzetta dritta, prelevare il campione e sciacquare abbondantemente con alcool isopropilico e innaffiare prima di asciugarlo con una pistola ad aria compressa. Se riutilizzabile, conservare il campione in un contenitore adatto con lo strato d'oro rivolto verso l'alto.
NOTA: I campioni hanno in genere un intervallo utilizzabile di circa 1-2 settimane se maneggiati correttamente, sebbene le dimensioni delle nanostrutture cambino nel tempo. Se eccessivamente danneggiato, passa a un campione nuovo per l'esperimento successivo. Il danno può essere sotto forma di graffi presenti sulla superficie dell'oro vicino alle nanostrutture o da scarse prestazioni sperimentali, indicando il degrado delle nanostrutture29.
13. Preparazione del sistema per un uso futuro
- Usa un panno per lenti piegato per rimuovere l'olio dall'obiettivo in una direzione, sollevandolo e ripetendolo con un'altra parte pulita del tessuto.
NOTA: Gli obiettivi sono soggetti a sbavature e graffi, che possono compromettere significativamente le prestazioni. Indossare sempre i guanti durante la pulizia ed evitare di toccare il materiale organico. Utilizzare solo un nuovo panno per la pulizia delle lenti per evitare la contaminazione e pulire in un'unica direzione con un solo movimento. Evitare di strofinare la lente e il panno di pulizia avanti e indietro per ridurre al minimo i danni. Se il lavaggio a secco non è sufficiente, applicare una piccola quantità di etanolo sul fazzoletto, rimuovere l'olio come accennato in precedenza e terminare con un fazzoletto asciutto. - Se successivamente verranno utilizzate le stesse soluzioni proteiche/tampone, potrebbe non essere necessario sostituire il tubo. Se vengono utilizzate soluzioni diverse, sostituire il tubo per ridurre il rischio di contaminazione da soluzioni precedenti.
NOTA: Non utilizzare lo stesso tubo per più di 1 settimana, anche se si eseguono esperimenti con le stesse soluzioni, poiché si sporcherà nel tempo.
Risultati
Dopo l'acquisizione dei dati, l'analisi dei dati può essere eseguita sui dati grezzi utilizzando il codice MATLAB per generare tracce dai dati grezzi raccolti dall'APD. La Figura 3 illustra una traccia di intrappolamento esemplare che include la linea di base prima dell'intrappolamento, l'evento di intrappolamento in cui si osserva un grande cambiamento nella trasmissione (ΔT/T0) e la deviazione standard prima che il laser venga spento per circa 5 secondi prima di essere riacceso. Una riduzione significativa della deviazione standard e il ritorno della trasmissione a livelli simili a quelli del basale indica il rilascio di proteine. La deriva lineare viene rimossa dalla traccia utilizzando la funzione MATLAB detrend.m, quindi il valore medio dei dati viene aggiunto nuovamente alla traccia detrendizzata. Occasionalmente, abbiamo bisogno di detrendare la traccia man mano che la configurazione si sposta nel tempo, causando una diminuzione lineare della trasmissione (vedi la traccia grigia nella Figura 3). Piccole variazioni nelle tracce di base prima e dopo l'intrappolamento sono dovute alla regolazione dello stadio per ottimizzare la linea di base con una deviazione standard minima, come dimostrato nella Figura 4A. A volte, le molecole proteiche sono visibili nella traccia senza essere intrappolate, chiamate proteine passanti. Le proteine che passano appaiono come un brusco cambiamento nella trasmissione, simile a una tipica trappola (Figura 4B), ma con una durata significativamente più breve, come mostrato nella Figura 4A. La densità spettrale di potenza (PSD) presenta un'altra analisi per confermare l'intrappolamento delle proteine fornendo la potenza del segnale a varie frequenze. I movimenti conformazionali delle proteine sono tipicamente osservati nell'intervallo >1 μs con metodi di spettroscopia a singola molecola40. La Figura 4C dimostra che, rispetto alla linea di base, l'intrappolamento di una proteina porta a una maggiore potenza del segnale, almeno nell'intervallo di 10 kHz (> 100 μs). Evidenzia anche l'importanza di allineare lo stadio a una linea di base ottimizzata, poiché una cattiva linea di base potrebbe aumentare il rumore a frequenze comprese tra 50 e 500 Hz, una gamma di frequenze sovrapposta a movimenti conformazionali delle proteine.
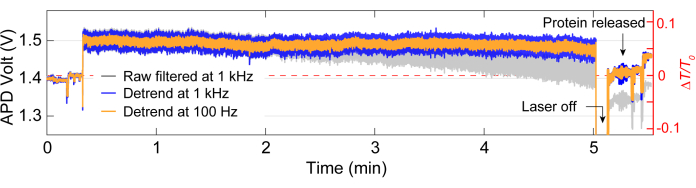
Figura 3: Traccia completa di intrappolamento per una singola proteina. Traccia rappresentativa di una trappola completa, compresa la linea di base, l'intrappolamento di una proteina e il rilascio della proteina. I salti in traccia prima e dopo l'intrappolamento sono dovuti all'allineamento. Clicca qui per visualizzare una versione più grande di questa figura.
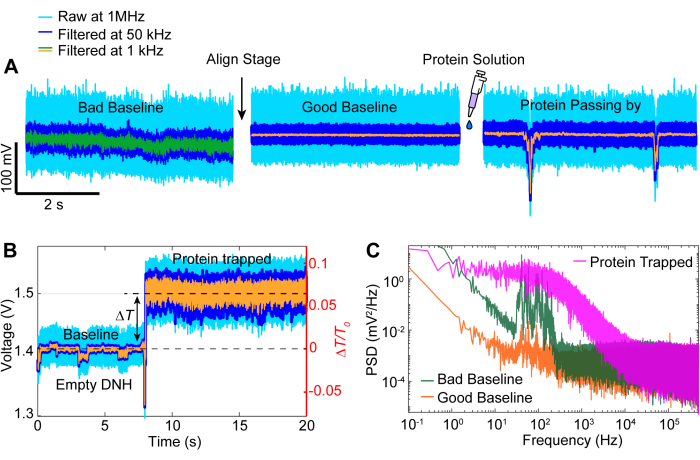
Figura 4: eventi di traccia comuni. (A) Esempi di un allineamento da una linea di base cattiva a una buona e di una proteina che passa vicino all'hotspot. (B) Traccia di intrappolamento che mostra il processo dalla linea di base quando l'hotspot DNH è vuoto a quando la proteina è intrappolata. (C) Grafico della densità spettrale di potenza (PSD) tra le linee di base buone e cattive rappresentate in (A) e la proteina intrappolata in (B). Valori PSD più elevati indicano un rumore maggiore a frequenze particolari. Clicca qui per visualizzare una versione più grande di questa figura.
La maggior parte degli eventi di intercettazione segue lo stesso schema generale della traccia nella Figura 3, anche se durante gli esperimenti possono sorgere problemi occasionali. Per la maggior parte degli esperimenti, la proteina deve essere rilasciata manualmente spegnendo il laser una volta completato l'esperimento desiderato. In alcuni casi, tuttavia, la proteina può lasciare la trappola senza intervento, come mostrato nella Figura 5A. Al contrario, a volte le proteine possono rimanere nel sito di intrappolamento anche dopo aver spento il laser, probabilmente a causa del fatto che la proteina si attacca al campione. Questo incollaggio provoca una traccia rumorosa dopo lo spegnimento e l'accensione del laser (vedere la Figura 5B). La probabilità che ciò accada dipende dalla proteina, poiché alcune proteine sono più inclini all'adsorbimento superficiale41,42. L'uso di un rivestimento come il PEG-tiolo può ridurre le possibilità di adesione delle proteine 39,43. A meno che non lo si desideri, come lo studio delle interazioni proteina-proteina, un altro problema è il double trapping, in cui una seconda proteina viene intrappolata dopo la prima trappola. Questo è caratterizzato da un altro forte aumento della trasmissione, simile alla prima trappola, e da un cambiamento nella deviazione standard (vedi Figura 5C).
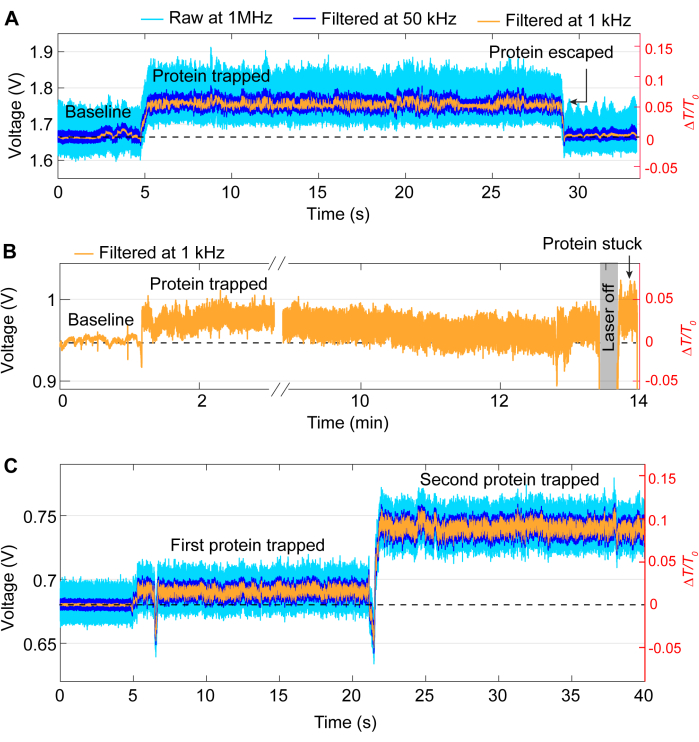
Figura 5: Esempi di eventi di intercettazione indesiderati. (A) Rilascio involontario di una proteina dal punto caldo di DNH. (B) Esempio di proteina che si blocca sulla superficie del campione nell'hotspot DNH. (C) Il salto di traccia si verifica quando una seconda proteina è intrappolata mentre la prima rimane ancora nell'hotspot DNH. Clicca qui per visualizzare una versione più grande di questa figura.
Un esperimento rappresentativo condotto sul carico di ferro in situ su una molecola di apo-ferritina dimostra l'uso di nanotweezers plasmonici come strumento per studiare la dinamica conformazionale delle proteine29. La ferritina è una proteina trasportatrice di ferro che esiste in due stati: apo-ferritina, che non contiene ferro, e olo-ferritina, che è piena di ferro44,45. Il ferro ferroso entra nella proteina attraverso 3 canali dove viene ossidato a ferro ferrico e immagazzinato nel nucleo proteico46. La Figura 6A illustra una tipica traccia di intrappolamento di apo-ferritina con una soluzione ferrosa infusa per oltre 20 minuti mentre la proteina è intrappolata. Le tracce di 20 s prese lungo l'intera traccia nei punti b-e forniscono informazioni sui cambiamenti che si verificano alla proteina nel tempo. Nella Figura 6B, l'apo-ferritina è intrappolata in un tampone PBS standard e non si osservano cambiamenti significativi nella traccia. La Figura 6C, D mostra le fluttuazioni nella S.D delle tracce, che sono causate dal carico di ferro nella proteina attraverso i suoi 3 canali multipli, risultando in uno stato più dinamico (apo-) in cui i canali sono aperti, e uno stato più compatto (olo-) con i canali chiusi. Dopo che la molecola di ferritina è stata riempita di ferro, è passata alla sua oloforma, risultando in una traccia di intrappolamento stabile, come mostrato nella Figura 6E. Le funzioni di densità di probabilità (PDF) nelle Figure 6B-E mostrano ulteriormente i cambiamenti che la proteina subisce in seguito all'esposizione a diverse condizioni di soluzione nel tempo.
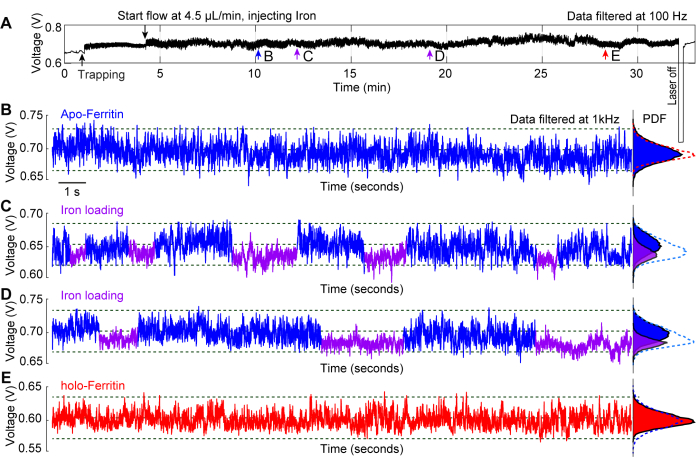
Figura 6: Caricamento in situ del ferro in un'apoferritina intrappolata. (A) Traccia di trasmissione completa di un DNH con una molecola di apoferritina intrappolata, seguita dall'iniezione di una soluzione ferrosa nel sito di intrappolamento per osservare i cambiamenti conformazionali della ferritina associati al carico di ferro. (B) Traccia di intrappolamento di 20 secondi di un'apoferritina intrappolata prima che la soluzione ferrosa raggiungesse il punto caldo. (C, D) Tracce di intrappolamento di 20 secondi dopo che la molecola di apoferritina è stata esposta alla soluzione ferrosa. I segmenti blu e viola segnano la S.D superiore e inferiore della traccia, indicando rispettivamente conformazioni flessibili e rigide della ferritina. (E) Traccia di intrappolamento di 20 secondi dopo che l'apoferritina è stata esposta alla soluzione ferrosa per >20 minuti. I grafici della funzione di densità di probabilità (PDF) a destra mostrano la distribuzione della trasmissione e sono codificati a colori per i segmenti blu e viola. Questa cifra è stata modificata da29. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 1 supplementare: Campione di DNH in oro montato sulla cella di flusso stampata in 3D. Il campione viene inserito in un'apposita fessura e fatto aderire alla cella di flusso mediante nastro biadesivo in PET. I parametri chiave e le misure associate per il design delle nostre celle a flusso sono etichettati. Clicca qui per scaricare questo file.
Figura 2 supplementare: Retro della cella di flusso con campione DNH dorato montato ed etichettata sulla parete interna. Il campione viene sigillato nella cella di flusso mediante silicone per duplicazione. Clicca qui per scaricare questo file.
Figura 3 supplementare: Schema della cella di flusso con DNH dorato montata con i fori di aspirazione e di uscita etichettati. Clicca qui per scaricare questo file.
Discussione
Un passaggio cruciale del protocollo consiste nel garantire che la cella di flusso non perda prima di essere inserita nello stadio, che deve essere testato in anticipo all'esterno della montatura a una portata elevata. Le perdite dopo il montaggio del campione possono danneggiare i componenti ottici, in particolare l'obiettivo inferiore.
L'allineamento può deviare dalla sua posizione ottimale nel tempo durante un esperimento, causando variazioni di segnale dovute alla sensibilità delle nanotweezer plasmoniche. In questo caso, riallineare utilizzando i controlli piezoelettrici alla massima trasmissione e alla minima deviazione standard della linea di base, poiché una linea di base rumorosa riduce la qualità dei dati. È necessario prestare attenzione a prendere nota meticolosamente di quando vengono effettuati gli allineamenti per eliminare il rischio di confondere l'interferenza dell'utente per un evento di trapping. Se si verifica una deriva maggiore, allineare delicatamente il tavolino per ridurre al minimo il rischio di rilascio della proteina e annotare il tempo di regolazione.
Modifiche e alterazioni alla tecnica presentata possono essere apportate in base a specifiche esigenze sperimentali. Ad esempio, un tavolino a temperatura controllata può aiutare a raffreddare/riscaldare il campione come desiderato invece di utilizzare il riscaldamento laser per aumentare la temperatura31,33. Altre tecniche, ad esempio la microscopia a scattering interferometrico (iSCAT), possono fornire l'interferenza della proteina all'interno del campo di scattering del DNH, ottenendo un segnale aggiuntivo proporzionale alla polarizzabilità della proteina, che è associata alla dimensione delle particelle47,48.
Le nanotweezer plasmoniche sono puramente una tecnica di rilevamento temporale in quanto i dati vengono registrati dall'APD (un rivelatore a pixel singolo). Questa tecnica non fornisce informazioni dirette sui cambiamenti strutturali di una proteina, come ad esempio quali regioni della proteina sono coinvolte in un cambiamento conformazionale o dove un ligando o un'altra proteina può legarsi alla proteina. Inoltre, la tecnica è limitata a un intervallo temporale di >1 μs a causa della frequenza di campionamento della scheda di acquisizione dati (1 MHz). Considerando la frequenza di Nyquist, dove l'intervallo più alto possibile è la metà della velocità di acquisizione, in questo caso, 2 μs in condizioni perfette.
In questo protocollo, abbiamo descritto il processo di messa a punto di un esperimento plasmonico con nanotweezers per intrappolare una singola proteina e monitorare i cambiamenti nelle sue dinamiche conformazionali nel tempo. La tecnica può essere sviluppata su qualsiasi microscopio costruito in casa o in commercio. Contrariamente agli approcci basati sulla fluorescenza o sul tethering, come smFRET e pinzette ottiche, la tecnica può intrappolare le proteine senza etichette o cavi e raggiungere comunque la sensibilità di una singola molecola. Le condizioni della soluzione possono essere modificate mentre la proteina è intrappolata utilizzando un sistema microfluidico, consentendo il monitoraggio in tempo reale dell'effetto di diverse soluzioni sulla proteina. Queste caratteristiche rendono le nanotweezer plasmoniche uno strumento promettente nel campo della biofisica e del biorilevamento, in particolare per le proteine che le tecniche convenzionali faticano a interrogare nel loro stato nativo. Le applicazioni future si concentreranno sulla decodifica delle dinamiche conformazionali di proteine più dinamiche, come le proteine intrinsecamente disordinate e le proteine contenenti regioni intrinsecamente disordinate, e le proteine di membrana, le cui dinamiche e persino la cui struttura sfuggono alle tecniche attuali.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
S.Z. riconosce il sostegno del Biotechnology and Biological Sciences Research Council Doctoral Training Partnership (BBSRC DTP) (BB/T0083690/1). Gli autori riconoscono il finanziamento dell'Iniziativa per l'istruzione e la ricerca Regno Unito-India (UKIERI). M.R. apprezza il sostegno della Royal Society e della Wolfson Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| 100x Objective (NA = 1.25) | Olympus | PLN100XO | Clean oil only with lens cleaning tissue in one motion |
| 12-1 rotary bidirectional microfluidic valve | Elveflow | ||
| 3/2 way solenoid valve | Elveflow | ||
| 4x Objective (NA = 0.1) | Olympus | PLN4XP | |
| Airgun | RS Components | 666-6772 | |
| Avalanche photodiode (APD) | Thorlabs | APD120A/M | Do not oversaturate, 50 MHz bandwidth, static sensitive |
| Butterfly laser diode | Thorlabs | FPL852S | |
| Charge-coupled device (camera) | Hikrobot | MV-CE200-10UC | |
| Crystallising dish | VWR | 216-0065 | |
| Data acquisition card | National instruments | USB-6361 | 2 MHz sampling rate |
| Double-sided tape | Adhesive Research Inc | ARcare92712 | Cut appropriate shape for flow cell with a laser cutter |
| Ethanol | Fume hood only, use gloves | ||
| Fibreport collimator | Thorlabs | PAF2-A7B | |
| Glass microscope coverslips (thickness 0.17 mm) | |||
| Half wave plate | Thorlabs | AHWP10M-980 | |
| Ideal-tek 120 mm, stainless steel, straight tweezers | RS Components | 282-7472 | For holding sample and peeling double sided tape |
| Isopropanol (IPA) | Fume hood only, use gloves | ||
| Lens cleaning tissue | Thorlabs | MC-5 | |
| Metrosil rapid duplicating silicone parts A and B | Metrodent | MSILR/1 | |
| Microscope slides (75 mm x 26 mm) | |||
| Modular Optical Tweezers System | Thorlabs | OTKB/M-CUSTOM | |
| Objective oil | Olympus | Use gloves, store between 2oC-8oC | |
| Photodiode laser mount | Thorlabs | CLD1015 | |
| Piezoelectric controller | Thorlabs | MDT693B | |
| Piezoelectric stage | Thorlabs | Nanomax 300 | |
| Planoconcave lens (f = -50 mm) | Thorlabs | LC1715-B | |
| Planoconvex lens (f = 150 mm) | Thorlabs | LA1433-B | |
| Planoconvex lens (f = 60 mm) | Thorlabs | LA1134-B | |
| Poly(ethylene glycol) methyl ether thiol (PEG-thiol) 800 MW | Sigma Aldrich | 729108 | Store between 2oC-8oC |
| PTFE tubing (ID: 0.18 mm) | Vici Jour | JR-T-6805-M25 | |
| PTFE tubing (ID: 0.8 mm) | Cole-Parmer | WZ-21942-72 | |
| SEM-FIB | Zeiss | Crossbeam 550 | Gallium ion source |
| Shortpass dichroic mirror 805 nm | Thorlabs | DMSP805 | |
| Silver mirror | Thorlabs | PF-10-03-P01 | |
| Syringe pump | Harvard Apparatus | 70-4511 | |
| Weller Erem 120 mm stainless steel, flat, rounded tweezers | RS Components | 176-1150 | For holding glass coverslip and peeling glue from flow cell |
Riferimenti
- Peter Lu, H. P. Sizing up single-molecule enzymatic conformational dynamics. Chem Soc Rev. 43 (4), 1118-1143 (2014).
- Mazal, H., Haran, G. Single-molecule FRET methods to study the dynamics of proteins at work. Curr Opin Biomed Eng. 12, 8-17 (2019).
- Ritchie, D. B., Woodside, M. T. Probing the structural dynamics of proteins and nucleic acids with optical tweezers. Curr Opin Str Biol. 34, 43-51 (2015).
- Neupane, K., Solanki, A., Sosova, I., Belov, M., Woodside, M. T. Diverse metastable structures formed by small oligomers of α-Synuclein probed by force spectroscopy. PLoS One. 9 (1), e86495(2014).
- Hughes, M. L., Dougan, L. The physics of pulling polyproteins: a review of single molecule force spectroscopy using the AFM to study protein unfolding. Rep Prog Phys. 79 (7), 076601-076601 (2016).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Schmid, S., Dekker, C. Nanopores: a versatile tool to study protein dynamics. Essays Biochem. 65 (1), 93-107 (2021).
- Van Meervelt, V., et al. Real-time conformational changes and controlled orientation of native proteins inside a protein nanoreactor. J Am Chem Soc. 139 (51), 18640-18646 (2017).
- Awasthi, S., Ying, C., Li, J., Mayer, M. Simultaneous determination of the size and shape of single α-Synuclein oligomers in solution. ACS Nano. 17 (13), 12325-12335 (2023).
- Sµnchez-Rico, C., Von Vithenberg, L., Warner, L., Lamb, D. C., Sattler, M. Effects of fluorophore attachment on protein conformation and dynamics studied by spFRET and NMR spectroscopy. Chemistry. 23 (57), 14267-14277 (2017).
- Yin, L., et al. How does fluorescent labeling affect the binding kinetics of proteins with intact cells. Biosens Bioelectr. 66, 412-416 (2015).
- Berkovich, R., et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 109 (36), 14416-14421 (2012).
- Dahmardeh, M., Mirzaalian Dastjerdi, H., Mazal, H., Köstler, H., Sandoghdar, V. Self-supervised machine learning pushes the sensitivity limit in label-free detection of single proteins below 10 kDa. Nat Meth. 20 (3), 442-447 (2023).
- Becker, J., et al. A quantitative description for optical mass measurement of single biomolecules. ACS Photon. 10 (8), 2699-2710 (2023).
- Houghtaling, J., et al. Estimation of shape, volume, and dipole moment of individual proteins freely transiting a synthetic nanopore. ACS Nano. 13 (5), 5231-5242 (2019).
- Plesa, C., et al. Fast translocation of proteins through solid state nanopores. Nano Lett. 13 (2), 658-663 (2013).
- Schmid, S., Stömmer, P., Dietz, H., Dekker, C. Nanopore electro-osmotic trap for the label-free study of single proteins and their conformations. Nat Nanotechnol. 16 (11), 1244-1250 (2021).
- Shi, X., Verschueren, D. V., Dekker, C. Active delivery of single DNA molecules into a plasmonic nanopore for label-free optical sensing. Nano Lett. 18 (12), 8003-8010 (2018).
- Verschueren, D. V., et al. Label-free optical detection of DNA translocations through plasmonic nanopores. ACS Nano. 13 (1), 61-70 (2019).
- Verschueren, D., Shi, X., Dekker, C. Nano-optical tweezing of single proteins in plasmonic nanopores. Small Meth. 3 (5), 1800465(2019).
- Peri, S. S. S., et al. Quantification of low affinity binding interactions between natural killer cell inhibitory receptors and targeting ligands with a self-induced back-action actuated nanopore electrophoresis (SANE) sensor. Nanotechnology. 32 (4), 045501(2020).
- Leong, I. W., Tsutsui, M., Yokota, K., Taniguchi, M. Salt gradient control of translocation dynamics in a solid-state nanopore. Anal Chem. 93 (49), 16700-16708 (2021).
- Juan, M. L., Righini, M., Quidant, R. Plasmon nano-optical tweezers. Nat Photon. 5 (6), 349-356 (2011).
- Juan, M. L., Gordon, R., Pang, Y., Eftekhari, F., Quidant, R. Self-induced back-action optical trapping of dielectric nanoparticles. Nat Phys. 5 (12), 915-919 (2009).
- Pang, Y., Gordon, R. Optical trapping of 12 nm dielectric spheres using double-nanoholes in a gold film. Nano Lett. 11 (9), 3763-3767 (2011).
- Pang, Y., Gordon, R. Optical trapping of a single protein. Nano Lett. 12 (1), 402-406 (2012).
- Kotnala, A., Gordon, R. Quantification of high-efficiency trapping of nanoparticles in a double nanohole optical tweezer. Nano Lett. 14 (2), 853-856 (2014).
- Booth, L. S., et al. Modelling of the dynamic polarizability of macromolecules for single-molecule optical biosensing. Sci Rep. 12 (1), 1995(2022).
- Yousefi, A., et al. Optical monitoring of in situ Iron loading into single, native ferritin proteins. Nano Lett. 23 (8), 3251-3258 (2023).
- Yousefi, A., et al. Structural Flexibility and Disassembly Kinetics of Single Ferritin Molecules Using Optical Nanotweezers. ACS Nano. 18 (24), 15617-15626 (2024).
- Peters, M., et al. Energy landscape of conformational changes for a single unmodified protein. NPJ Biosens. 1 (1), 14-14 (2024).
- Peters, M., McIntosh, D., Branzan Albu, A., Ying, C., Gordon, R. Label-free tracking of proteins through plasmon-enhanced interference. ACS Nanosci Au. 4 (1), 69-75 (2024).
- Ying, C., et al. Watching single unmodified enzymes at work. arXiv. , (2021).
- Yang-Schulz, A., et al. Direct observation of small molecule activator binding to single PR65 protein. NPJ Biosensing. 2 (1), 1-10 (2025).
- Yang, W., van Dijk, M., Primavera, C., Dekker, C. FIB-milled plasmonic nanoapertures allow for long trapping times of individual proteins. iScience. 24 (11), 103237(2021).
- Koya, A. N., et al. Novel plasmonic nanocavities for optical trapping-assisted biosensing applications. Adv Optical Mater. 8 (7), 1901481(2020).
- Goswami, A., Umashankar, R., Gupta, A. K., Aravindan, S., Rao, P. V. Development of a microstructured surface using the FIB. J Micromanufact. 1 (1), 53-61 (2018).
- Onuta, T. D., Waegele, M., DuFort, C. C., Schaich, W. L., Dragnea, B. Optical field enhancement at cusps between adjacent nanoapertures. Nano Lett. 7 (3), 557-564 (2007).
- Al-Ani, A., et al. The influence of PEG-thiol derivatives on controlling cellular and bacterial interactions with gold surfaces. Appl Surf Sci. 462, 980-990 (2018).
- Schuler, B., Hofmann, H. Single-molecule spectroscopy of protein folding dynamics-expanding scope and timescales. Curr Opin Str Biol. 23 (1), 36-47 (2013).
- Kopac, T. Protein corona, understanding the nanoparticle-protein interactions and future perspectives: A critical review. Int J Biol Macromol. 169, 290-301 (2021).
- Li, X., Guo, W., Xu, R., Song, Z., Ni, T. The interaction mechanism between gold nanoparticles and proteins: Lysozyme, trypsin, pepsin, γ-globulin, and hemoglobin. Spectrochim Acta A Mol Biomol Spectrosc. 272, 120983(2022).
- Emilsson, G., et al. Strongly stretched protein resistant poly(ethylene glycol) brushes prepared by grafting-To. ACS Appl Mater Inter. 7 (14), 7505-7515 (2015).
- Theil, E. C., Liu, X. S., Tosha, T. Gated pores in the ferritin protein nanocage. Inorg Chim Acta. 361 (4), 868-874 (2008).
- Theil, E. C., et al. The ferritin iron entry and exit problem. Inorg Chim Acta. 297 (1), 242-251 (2000).
- Takahashi, T., Kuyucak, S. Functional properties of threefold and fourfold channels in Ferritin D=deduced from electrostatic calculations. Biophys J. 84 (4), 2256-2263 (2003).
- Gemeinhardt, A., et al. Label-free imaging of single proteins secreted from living cells via iSCAT microscopy. J Vis Exp. (141), e58486(2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nat Comm. 5 (1), 4495(2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon