Method Article
Isolierung von Fidelity Varianten von RNA-Viren und Charakterisierung von Virus-Mutation Frequency
In diesem Artikel
Zusammenfassung
Der vorliegende Artikel beschreibt die notwendigen Schritte zu isolieren und zu charakterisieren RNA-Polymerase Treue Varianten von RNA-Viren und wie man Mutationsfrequenz Daten verwenden, um Treue Veränderungen in der Gewebekultur zu bestätigen.
Zusammenfassung
RNA-Viren verwenden RNA abhängigen RNA-Polymerasen auf ihre Genome zu replizieren. Die eigensicheren hohen Fehlerquote dieser Enzyme ist ein großer Faktor für die Generation der extremen Vielfalt der Population, die auf Viren Anpassung und Entwicklung erleichtert. Zunehmende Anzeichen zeigt, dass die intrinsische Fehlerrate und die daraus resultierenden Mutation Frequenzen von RNA-Viren können moduliert werden durch subtile Aminosäure Änderungen an der viralen Polymerase. Obwohl biochemische Assays für einige virale RNA-Polymerasen, die quantitative Messung der Inkorporation Treue erlauben existieren, hier beschreiben wir eine einfache Methode zur Messung der Mutation Frequenzen von RNA-Viren, die sich als so genau wie biochemische Ansätze bei der Identifizierung Treue zu verändern Mutationen hat. Der Ansatz nutzt konventionellen virologischen und Sequenzierung Techniken, die in den meisten biologischen Laboren durchgeführt werden können. Basierend auf unseren Erfahrungen mit einer Reihe von verschiedenen Viren, haben wir die wichtigsten Schritte, die optimiert, um die Wahrscheinlichkeit zu isolieren Treue Varianten und die Erzeugung der Daten der statistischen Signifikanz erhöhen müssen identifiziert werden. Die Isolierung und Charakterisierung der Treue zu verändern Mutationen können neue Einblicke in die Polymerase-Struktur und Funktion 3.1 bieten. Darüber hinaus können diese Treue Varianten nützliche Werkzeuge werden bei der Charakterisierung von Mechanismen der Virus-Anpassung und Weiterentwicklung 4-7.
Protokoll
1. Bestimmen Sie den Wertebereich der Mutagen-Konzentrationen, die nur minimal toxisch für Zellen ist
Der Zweck dieser Übung ist es, festzustellen, welcher Bereich von mutagen Konzentrationen während einer Infektion ohne Selbstbeteiligung Zelltoxizität verwendet werden kann. Im Grunde wollen Sie die Bedingungen, die für die Virus-Infektion benötigt werden zu reproduzieren. Für die meisten Viren, Infektionen dauern zwischen 2 und 7 Tagen. Bereiten Sie genug Platten zu Probe-Zellen an jedem Tag. Wenn nicht-adhärente Zellen verwendet werden, ändern Sie die Protokoll entsprechend.
- Am Tag vor dem Experiment, Saatgut 7 x 10 5 HeLa Zellen / Well in einer 6-Well-Platte, die eine sub-konfluenten (75%) Monolage erreichen den Tag des Experiments. Jede Vertiefung der Platte wird mit einer anderen Konzentration von Mutagen behandelt werden, wodurch eine Reichweite von 6-Konzentrationen.
- Am Tag des Experiments, bereiten mutagen Verdünnungen in Zellkulturmedium. Für HeLa-Zellen, nutzen eine Reihe von 0 bis 1000 nM für die Basis-Analoga (Ribavirin, 5-Fluorouracil, 5-Azacytidin), 0 bis 50 mM für MgCl 2 und 0 bis 5 mM für MnCl 2.
- Saugen Sie das Medium aus den Vertiefungen und ersetzen mit 2 ml mutagen-ergänzt Medium und zum Inkubator.
- Alle 24 Stunden, verwenden Sie eine Platte von Zellen, um die Lebensfähigkeit der Zellen zu überprüfen. Dies kann durch die Durchführung Trypanblaufärbung oder über den Handel Fluoreszenz / Lumineszenz-Assays (zB Promega ist CellTiter-Glo ® Luminescent Zellviabilitätsmessungen Assay) durchgeführt werden.
- Für Trypanblau-Färbung, lösen Zellen aus einer Platte (behandelt mit unterschiedlichen Konzentrationen mutagen) und sanft Pellet Zellen durch Zentrifugation.
- Überstand verwerfen und die Zellen in PBS (Serum kann mit Färbung stören).
- Mix 1 Volumen Zellsuspension in PBS mit 1 Volumen von 0,4% Trypanblau, für 2 Minuten bei Raumtemperatur inkubieren.
- In einem Hämacytometer, zählen die lebensfähigen (ungefärbten) und nicht lebensfähigen (blau gefärbt)-Zellen. Errechnen Sie den Prozentsatz der lebenden Zellen für jede Konzentration der mutagen, einschließlich der unbehandelten Kontrolle. Wir finden, dass Bedingungen, die sich in weniger als 50% Zelltod durch die Infektion Endpunkt (wenn maximale Titer erreicht werden) ideal für die Isolierung von mutagen Widerstand.
2. Bestimmen Sie die optimale ungiftig mutagen Konzentration, mäßig reduziert Virustiter (ca. 0,5-2 log-Reduktion)
Diese Übung dient dazu, die Konzentration von mutagen, dass ein starker Selektionsdruck ausüben will, ohne über-Mutagenese der Bevölkerung zu bestimmen. Für die RNA-Mutagene, finden wir, dass dies zu 10,5-2 logs Reduzierung der Virustiter entspricht. Bei diesen Konzentrationen ist jedes Genom in mindestens ein oder zwei Positionen mutiert. Das Mutagen kann in der Generation der Resistenz-Mutation, die dann über Passage ausgewählt Hilfe. Wenn zu viele Mutationen eingeführt werden (bei sehr hohen Konzentrationen mutagen), werden die Mutanten mit dem Resistenz-Mutation selbst sein letal mutiert, behindern ihre Isolation.
- Seed-Platten mit Zellen mit den gleichen Bedingungen wie für Schritt 1
- Am Tag des Experiments, bereiten mutagen Verdünnungen in Zellkulturmedium. Verwenden Sie den gleichen Bereich von Konzentrationen wie oben bestimmt, sondern schließt Konzentrationen, die in mehr als 50% Zelltod führte. Bereiten Sie genügend Medium in jede Vertiefung zweimal Abdeckung (4 ml pro Well), so dass für eine Vorbehandlung der Zellen vor der Infektion, wobei jede Konzentration mutagen.
- Saugen Sie das Medium und Vorbehandlung Zellen mit Mutagen durch Inkubation in mutagen-ergänzt Medium für 2 Stunden. Für die meisten Zelltypen ist das ausreichend Zeit für die Aufnahme von mutagen.
- Nehmen Sie mittel-und infizieren mit dem Virus bei niedrigen Multiplizität der Infektion MOI (0,1 oder 0,01) in einem minimalen Volumen (200 ul für 6-Well-Platten). Inkubieren 15-60 Minuten, damit Viren, Zellen zu infizieren. Rock the Platte in regelmäßigen Abständen, um sicherzustellen, dass das Inokulum der Zellmonoschicht abdeckt.
- Saugen Sie den inokulierten Virus und waschen zweimal mit 2 ml PBS, um so viel des Inokulums wie möglich zu entfernen.
- Add-Medium mit den entsprechenden Konzentrationen mutagen in jede Vertiefung und inkubieren Zellen für den Gegenwert von 3-6 Replikationszyklen.
- Ernte-Virus aus jeder Vertiefung und bestimmen die antivirale Wirkung auf Virustiter. Dies kann durch Standard-Plaque-Assay oder Grenzverdünnung (TCID 50) durchgeführt werden. Hinweis: Die Quantifizierung der Virus durch Techniken, die Maßnahme nur die RNA-Synthese nicht geeignet, weil die mutagene Wirkung nicht nachgewiesen werden können. Mutagenisierten Genomen mit letalen Mutationen möglicherweise noch durch qRT-PCR zum Beispiel erkannt werden, aber nicht in Viren Lebensfähigkeit Assays beobachtet werden.
- Aus den berechneten Titer, Ermittlung der Konzentration des mutagen, dass Virustiter reduziert (im Vergleich zu den unbehandelten Kontrolltieren Infektion) durch 10,5-2 Protokolle, die auch nicht sehr toxisch für Zellen (im Idealfall weniger als 50% Toxizität).
3. Isolation und Identifikationfication des Mutagen resistenten Varianten
Führen großer Bevölkerungszahl Passagen in der optimalen Konzentration mutagen oben definiert und überprüfen Virustiter über den Flur Serie. Als Kontrolle Passage-Virus in einem Wachstumsmedium ohne mutagen. Als weitere Kontrolle das potenzielle Auftreten von defekten störende Partikel (DI)-Monitor, führen Sie frische Infektionen in Abwesenheit von Mutagen bei jeder Passage Schritt (unpassaged-Steuerung).
- Am Tag vor der Infektion, Saatgut 25 cm 2-Kolben mit 1,5 x 10 6 HeLa-Zellen (andere Kolben Größen verwendet werden kann) zu erhalten sub Monolayers am folgenden Tag.
- Am Tag der Infektion, Vorbehandlung Zellen für 2 Stunden mit Medium, welches die optimale Konzentration der einzelnen mutagen, in Abschnitt 2 bestimmt.
- Nehmen Sie mittel-und infizieren Zellen mit einem Mindestvolumen bei einer MOI von 1 oder die größte MOI, die nicht in defekten Einmischung (DI) Partikelbildung ist Ergebnis für das Virus untersucht.
- Nach 30-60 Minuten der Infektion, absaugen Inokulum zwei Waschgänge mit PBS, dann fügen Sie frisches Medium mit Mutagen bei den entsprechenden Konzentrationen ergänzt.
- Inkubieren für die Zeit in den Abschnitten 1 und 2, die maximale Virustiter in diesen Bedingungen entspricht bestimmt. Ernten Sie die Virennachkommen.
- Titer-Virus bei jedem Durchgang und wiederholen Sie den 3 vorangegangenen Schritte.
- Während der ersten paar Passagen, sollte Virustiter von Mutagen behandelten Proben entsprechend fallen, im Vergleich zu den ursprünglichen Virustiter und der Kontrolle (unbehandelte und unpassaged) Virustiter. Wenn Virustiter von mutagen passagiert Proben wieder klettern, um das gleiche Niveau wie bei der unbehandelten Kontrolle, die Bevölkerung wahrscheinlich enthält ein Mutagen resistenten Variante. Bis zu 20 oder 30 Durchgänge erforderlich sein, obwohl wir die meisten unserer Treue Varianten isoliert zwischen 5 und 15 Passagen.
- Sobald Virustiter für eine bestimmte Passage Serie erreichen die gleiche Größenordnung wie die unbehandelten Kontrolle Titer, extrahieren RNA aus allen Proben, einschließlich der unbehandelten Kontrolle von derselben Stelle an. RNA Extraktion Kits oder Trizol-Extraktion verwendet werden kann.
- Führen RT-PCR mit Primern, dass die Polymerase oder Replikase Gene des Virus von Interesse zu verstärken. In einem zweiten Schritt soll das gesamte Genom (mindestens kodierenden Regionen) sequenziert, um zu prüfen, ob Resistenzphänotypen auf andere Virus-Gene Karte als gut. Dies ist besonders wichtig für die Basis-Analog-Mutagene, wie Ribavirin, die andere Aspekte der Virus-und Zell-Funktion beeinträchtigt. In diesem Fall wird die Variante kann als resistent gegenüber einer dieser anderen antiviralen Aktivitäten und nicht eine Treue-Variante sein.
- Purify der RT-PCR-Produkten mit einem PCR-Purification-Kit und die Reihenfolge der Mutagen-resistente Population Konsensussequenz erhalten. Include Hintergrund steuert für die Sequenzierung Fehler (siehe Diskussion).
- Mit einem Sequenz-Alignment-Software und das Virus Konsensussequenz als Referenz, richten Sie die Sequenzen. Identifizieren Sie die neue Punktmutationen, mit besonderem Augenmerk auf alle, die sich ausschließlich erscheinen in der Mutagen behandelt Bevölkerung an der Stelle, wo Virustiter erreichen normales Niveau. Wenn diese Mutation nicht vorhanden in früheren Passagen und nicht in unbehandelten Kontrollen aus der gleichen Passage-Nummer (Angabe Anpassung an Zellkulturpassage), dann ist diese Mutation ist wahrscheinlich verantwortlich, zumindest teilweise, für mutagen Widerstand. Verlassen Sie sich nicht auf Basis genannte Sequenz (die Text-Version der Sequenz) und die Ausrichtung Software allein. Überprüfen Sie die Chromatogramme Minderheit Spitzen, die durch die Ausrichtung Software verpasst haben. Eine Mutante entspricht 20-30% der Gesamtbevölkerung wird immer noch als Peak zeigen, aber zu klein, um als 'N' von Standard-Sequenzanalyse identifiziert werden.
4. Sobald eine Mutation identifiziert wurde, zu isolieren oder zu generieren die Variante ein und bestätigen den Widerstand Phänotyp, mehrere RNA Mutagene
Als nächstes wird die Variante präsentiert der identifizierten Mutation isoliert, um die Anbindung an den Widerstand Phänotyp zu bestätigen. Es ist wichtig, dass die Mutation zu verändern Treue Verdacht in eine genetisch sauberen Hintergrund untersucht (das heißt, nicht präsentieren zusätzliche Mutationen an anderer Stelle in das Genom). In der besten Lage, existiert eine infektiöse cDNA-Klon, würde die Erzeugung einer Bestandsaufnahme der Mutagen-resistente Variante von Mutagenese auf eine saubere genetischen Hintergrund zu ermöglichen. In diesem Fall ist § 4 nicht erforderlich. Wenn jedoch ein cDNA-Klon nicht verfügbar ist, kann die Isolierung durch Plaque-Aufreinigung von Viren, wie unten beschrieben durchgeführt werden. Mehr als eine Runde Plaquereinigung erforderlich, um die Variante auf eine saubere, genetischen Hintergrund zu isolieren.
- Die Isolierung des Mutagen resistente Mutante von Plaque-Assay.
- Zur Isolierung der Mutanten identifiziert, führen Sie eine Standard-Plaque-Assay unter Agarose (0,5 bis 1% letzte wt / vol) Overlay in 6-well Platten. Bereiten Sie serielle Verdünnungen des Virus, an der Börse Titer basiert auf dilutions, dass zwischen 10 und 50 gut getrennt Plaques produzieren wird.
- Wenn Plaques deutlich sichtbar (in der Regel 2 bis 5 Tage nach der Infektion, je nach Virus) sind, markieren Sie die Lage der Plaques auf den Platten und mit einem p200 Pipette mit Filter Spitze, sanft stürzen die Spitze durch die Agarose-Overlay man aufpassen, nicht lösen und verschieben Sie die Overlay-Position (das wäre in Kreuzkontamination von einzelnen Plaques führen).
- Heben Sie die Spitze aus der Überlagerung und Übertragung der Agarose-Stecker in die Spitze in ein Eppendorf mit 250 ul Medium und Wirbel enthalten. Mach dir keine Sorgen, wenn die Spitze, entfernt nicht enthält Agarose, für viele RNA-Viren, enthält durchschnittlich Plaque für viele RNA-Viren 10 5 Viren und einer ausreichenden Menge wird durch einfaches Berühren der Spitze an die Oberfläche der Platte übertragen werden.
- Wählen Sie bis zu 10 Plaques pro Mutagenbehandlung. Abhängig von der Chromatogramme der Sequenzierung, die identifizierten die Mutation, ungefähr abschätzen, wie viel Prozent der Bevölkerung enthält die gewünschte Mutation. Ziel ist es, drei oder vier Tafeln mit der Mutation zu isolieren. Einige dieser Mutanten führen auch zusätzliche, unerwünschte Mutationen, die später durch Sequenzierung identifiziert werden.
- Entpacken Sie die RNA aus diesen Proben (aber sparen die Hälfte der Probe in einen größeren Bestand an Viren zu machen), und führen Sie RT-PCR, dass die Sequenzierung des gesamten Genoms ermöglichen wird. Im Durchschnitt werden RNA-Viren bis zu zwei Mutations Unterschiede in Bezug auf die Konsensus-Sequenz, also Abfolge 3 oder 4 plaquegereinigt Viren zu einem Zeitpunkt, zu dem Isolat, das enthält die gewünschte Mutation ohne zusätzliche Mutationen zu identifizieren.
- Einmal identifiziert, einen größeren Bestand an dieses Virus für alle nachfolgenden Untersuchungen mit Hilfe der Plaque-gereinigt Probe erhalten über einen größeren Kolben von Zellen, zB 8x10 6 HeLa-Zellen in einer T75-Kolben infizieren ..
- Bestätigen Sie die mutagen Empfindlichkeit / Resistenz durch die identifizierten Mutationen verliehen.
- Mit dem isolierten oder neu generierte Klon und ein Wildtyp-Kontrolle Virus unter ähnlichen Bedingungen hergestellt, wiederholen Sie den Experimenten in Abschnitt 2 entweder über eine vollständige Palette von mutagen Konzentrationen, oder die Konzentration, bei der die Resistenz-Mutation wurde erzeugt.
- Verwenden Sie verschiedene RNA mutagenen Bedingungen (Ribavirin, 5-Fluorouracil, 5-Azacytidin erhöhte Mg 2 +, Mn 2 +). Wenn die Polymerase-Variante ist resistent gegen mehr als eine Art von mutagen, dann ist es wahrscheinlicher, dass diese Variante High Fidelity ist. Alternativ ist es möglich, dass eine Resistenz-Mutation spezifisch für eine einzelne mutagene Bedingung ist, zumal einige dieser Verbindungen RNA-Viren beeinträchtigt durch eine Reihe von Mechanismen 8.
5. Überprüfen Sie die Replikation Preise
Seit Treue zu verändern Mutationen am häufigsten auf die Polymerase-Karte ist es möglich, dass die gleiche Mutation Polymerase bedeutend verändern wird Replikation Kinetik und es ist wichtig, um Gemeinsamkeiten und Unterschiede in der Replikation ermöglichen einen besseren Vergleich der Unterschiede in der Mutation Frequenzen durchgeführt unten zu bestimmen. Dazu untersuchen die Replikation von mindestens zwei kostenlosen Ansätze - eine, die Virus-Produktion und andere, die RNA-Synthese untersucht untersucht.
- Ein-Schritt-Virus Wachstumskinetik
- Am Tag vor dem Experiment, Saatgut 6-well Platten nach Bedarf, eine Platte pro Zeitpunkt getestet werden. Erwägen Sie die Verwendung dreifachen Wells für jede Mutante und dem Wildtyp-Virus.
- Am Tag des Experiments, entfernen Sie das Medium und infizieren Brunnen mit jedem Virus bei einer MOI von 10, um sicherzustellen, dass jede Zelle gleichzeitig infiziert ist. Inkubieren 30-60 Minuten bei 37 ° C.
- Rock-Platten alle 10 Minuten, um zu vermeiden Trocknung der Zellmonolayer. Entfernen Sie Virus und waschen zweimal mit 2 ml PBS. Es ist wichtig, so viel des Inokulums wie möglich zu entfernen. Ersetzen Sie mit Wachstumsmedium.
- Nach der Infektion zum Zeitpunkt = 0, Ernte des Virus von einer Platte. Zurück Platten Inkubator und Ernte der Viren in regelmäßigen Abständen, dass eine einzige Replikationszyklus (zB für 3h, 5h, 7h, 9h, 12h, 24h) erstrecken.
- Titer-Virus, zu jedem Zeitpunkt (z. B. Plaque-Assay, TCID50, FFU-Assay) geerntet und Grafik die Wachstumskurven der Titer gegen die Zeit.
- Kinetik der RNA-Synthese
Die Kinetik der RNA-Synthese kann überwacht mit einer der Ansätze unten angegeben werden. Wenn möglich, sollten die gleichen Proben verwendet werden, um in einem Schritt Wachstumskinetik bestimmen verwendet werden, um RNA-Spiegel gemessen werden.- qRT-PCR. Dieses Verfahren liefert sehr quantitative Maßnahmen der Replikation über einen großen Bereich von wenigen Genomkopien bis> 10 10, in Abhängigkeit von der Empfindlichkeit des Tests. Design-Primer und Sonden, die ein kleines Fragment (<200 bp) eine hoch konservierte genomische Region zu decken.
- Northern Blot-Analyse. Obwohl weniger quantitative als qRT-PCR, ermöglicht diese Technik visuelle Bestätigung, dass die Replikation Ergebnisse in voller Länge Genome und dass keine signifikanten Kettenabbruch als Ergebnis der Polymerase-Mutation auftritt.
- Expression eines Reportergens. Wenn ein cDNA-Klon Ausdruck eines Reportergens (zB Luciferase) verfügbar ist, dann kann dies als Ersatz für replikativen Kapazität untersucht werden. Allerdings sollte das rekombinante Virus nicht für andere Anwendungen (z. B. Bestimmung der Mutationsrate) verwendet werden, da die selektive wirkenden Drücke auf dieses Virus wird nicht die gleiche sein, insbesondere da diese Viren eine Tendenz, die eingefügt Reportergen zu löschen haben.
6. Messen Mutationshäufigkeiten
Dies ist ein kritischer Schritt in der Bestätigung, dass die identifizierten Polymerase-Mutation Resistenz verleihen, zum Mutagen verändert Replikationsgenauigkeit. Es ist wichtig zu beachten, dass die Mutation Frequenzen gemessen hier nicht Mutationsraten. Um zu bestimmen, Preise, eine sehr sorgfältige Messung der Replikation Kinetik (RNA-Menge synthetisiert und Länge der Replikationszyklus) müssen berücksichtigt werden in. Messung Mutation Frequenzen jedoch, solange Passage Geschichte und Replikation Kinetik überwacht werden, liefert reproduzierbare, quantitative Maßnahmen der Replikation Treue. Mutation Frequenzen können entweder in die lebensfähige Viren Bevölkerung (Plaque Klone oder Grenzverdünnung) oder in der gesamten Viruspopulation (Virus Lager oder Überstand) bestimmt werden. Um festzustellen, Mutationshäufigkeiten bereiten Virus Aktien aus einer späteren Stelle (z. B. Kanal 2 oder darüber hinaus). Es ist wichtig, dass das Virus Bevölkerung hat Zeit, um ihre genetische Vielfalt näher zu einer Mutation-Auswahl Gleichgewicht zu erweitern musste.
- Mutation Frequenzen von lebensfähigen Viruspopulation
Dieser Ansatz, obwohl mehr mühsam, gibt Auskunft darüber, wie viele Mutationen vorhanden sind, im Durchschnitt Genoms, dass die Replikation Kompetenz behält. Es sollte jedoch beachtet werden, dass eine Tendenz für höhere Fitness-Varianten auftreten und unteren Fitness, praktikable Varianten, die nicht leicht Plaque, zum Beispiel, kann nicht erkannt werden. Als solches ist es auch erlaubt eine bessere Maßnahmen Synonym (DS) und nicht-synonym (dN) Nukleotidsubstitutionen, die verwendet werden, um zu erkunden, ob die positive Selektion ist auf die Bevölkerung wirken können. Da jedoch weniger Mutationen quantifiziert werden, wird eine größere Anzahl von Sequenzen für die statistische Analyse benötigt werden. Dieses Verfahren beruht auf Isolierung einzelner Viren von beiden Plaquereinigung oder Grenzverdünnung, wie oben beschrieben. Als Ausgangspunkt empfehlen wir Isolation von 48 einzelnen "Klone" von Wildtyp-Virus und der Mutagen-resistenten Variante. Jeder Klonpopulation auf diese Weise isoliert wird voraussichtlich zu tragen, was Mutation des Gründers Genoms vorgestellt. Die Menge an RNA, die in der isolierten Plaque oder Grenzverdünnung gut ist in der Regel ausreichend für die Amplifikation mittels RT-PCR. Falls erforderlich, kann ein kurzer Verstärkung (weniger als ein Replikationszyklus) auf eine minimale Anzahl von Zellen (z. B. 24-Well-Platte-Format) verwendet werden, um mehr RNA erhalten werden, jedoch mindestens Verstärkung durchgeführt werden, um die Ansammlung von neuen Mutationen zu vermeiden. Beachten Sie, dass jeder Klon und der Bevölkerung, die verglichen werden müssen die gleiche Anzahl von Replikationszyklen unterziehen.- Isolate von 24 bis 48 Virusklone durch Plaque-Reinigung oder Grenzverdünnung.
- Auszug RNA aus den isolierten klonalen Populationen
- RT-PCR zu amplifizieren ein Fragment Höhe von bis zu 3kb für jede Probe. Am besten ist es, die strukturellen Protein Region, die mehr lebensfähige Mutationen als weitere konservierte Bereiche von Nicht-Strukturgene tolerieren neigt zu decken.
- Purify der PCR-Produkte, die Reihenfolge und führen Mutationsanalyse (Abschnitt 7).
- Mutation Frequenzen von insgesamt Viruspopulation
Obwohl ein Vorteil dieser zweiten Methode ist, dass selbst geringe Fitness-Varianten werden in der Reihenfolge aufgenommen werden, ermöglicht ein umfassenderes Bild des Spektrums von Mutationen. Es kann jedoch nicht ideal für phylogenetische Analysen, dass lebensfähige Viren Populationen (z. B. dN / dS-Werte) und die Identifizierung der wichtigsten Mutationen übernehmen, da tödliche Änderungen (veränderte RNA-Struktur, Stoppcodons, dramatische Aminosäureaustausche) nicht vollständig identifiziert werden und würde in der Analyse beibehalten werden. Dennoch ist es diese Technik erlaubt dem Forscher, die statistisch signifikante Daten zu erhalten, um Änderungen der Treue zu bestätigen, wenn es einen Mangel an einer in-vitro-biochemischen Assays. Diese Technik beruht auf RT-PCR-Amplifikation von insgesamt Virion RNA, einschließlich Genomen mit Low-Fitness oder letalen Mutationen, die nicht produzieren wird Plaques. Die Mutation Frequenzen, die durch dieses Verfahren erhalten werden kann, 10-fach höher als von Plaque oder Grenzverdünnung Klonen.- Auszug RNA aus der gesamten Bevölkerung Virion
- RT-PCR Amplify ein 800 bis 1200 Nukleotidbereich aus einem Teil der genomischen codierenden Sequenz, die bekanntermaßen Mutationen zu tolerieren und haben genetische Varianz (zB Strukturproteine) ist. Größere Fragmente werden nicht leicht insert into Klonierungsvektoren wie TopoTA und der unzureichenden Anzahl von Transformanten zu produzieren. Obwohl kleinere Fragmente noch besser werden, kann die Abdeckung des Genoms sequenziert werden zu wenig, um statistische Signifikanz zu erreichen. Ein Fragment von mindestens 800 bp erlaubt Sequenzabdeckung mit zwei Primern und ist ein guter Kompromiss zwischen einer möglichst Sequenzabdeckung und Minimierung Sequenzierung Kosten. Wir finden, dass zwischen 70 und 100 Sequenzen für ein 800 Nukleotid-Region reproduzierbar bestätigt die veränderten Treue der Varianten haben wir im Labor untersucht wurden. Beachten Sie, dass andere Vektor / Klonierungsverfahren mit gleicher Effizienz genutzt werden kann.
- Purify der RT-PCR-Produkt mit einem kommerziellen Kit oder durch Standard-DNA-Extraktion / Fällung.
- Wenn der RT-PCR Enzyme verwenden Sie nicht produzieren A Überhänge, führen Sie eine 10 Minuten Verlängerung durch Zugabe von 1 uM ATP und Taq-Polymerase
- TopoTA Klon folgenden Anweisungen des Herstellers
- Für jedes Virus Population, die untersucht werden, wählen Sie 96 Kolonien, mit einer positiven einfügen blau / weiß-Screening auf XGal beschichteten Platten identifiziert. Zunächst testen die Anwesenheit von Einsätzen für jeden einzelnen Genomregion geklont, um die Gültigkeit der blau / weiß-Screening von Plasmid-size-Screening auf Agarosegelen oder Single-Kolonie-PCR bestätigen. Mit der Fragmentgröße und der oben genannten Bedingungen erreichen wir 90% Positive
- Wachsen jede Kolonie in flüssiger Brühe über Nacht in 1 ml LB-Medium in 96-Well-Platten Bakterienkultur
- Am nächsten Tag vorzubereiten Minipreps in 96-well Format.
- Sequence jede Platte mit genügend Primer (Primer für die RT-PCR zum Beispiel verwendet werden, oder TopoTA m13-Primer) auf eine maximale Abdeckung des geklonten Segment zu erhalten. Führen Mutationsanalyse (Abschnitt 7).
7. Die Sequenzanalyse
Führen Sie Sequenz-Analysen mit einem Verweis oder Konsensus-Sequenz für jede Bevölkerungsgruppe und entsprechende Ausrichtung Software. Wir empfehlen Lasergene oder Sequencher, die sich leicht identifizieren können SNPs in Bezug auf Konsens.
- Richten Sie die Sequenzen mit Hilfe geeigneter Software (zB Lasergene oder Sequencher).
- Discard schlechter Qualität Sequenzen (schlechte Basis Berufung, zu viele 'N ist oder zu kurz in der Länge). Identifizieren Sie die Nukleotid-Bereich, die von allen Sequenzen abgedeckt ist. Da die verschiedenen Regionen des Genoms mehr oder weniger toleriert werden Mutationen, zum Vergleich Gründen ist es unerlässlich, dass die gleiche Region in vollem Umfang für jeden sequenziert Klon für die Analyse erhalten abgedeckt. Wenn also mehrere Sequenzreaktionen pro Klon durchgeführt werden, und eine Sequenz nicht für einen bestimmten Klon, entsorgen Sie die clone (alle Sequenzen) aus der Analyse.
- Identifizieren und zählen die SNPs, die sich von der Referenz-Stamms sind.
- Berechnen Sie die Mutationsrate durch Division der Gesamtzahl der SNPs durch die Gesamtzahl der Nukleotide sequenziert (Anzahl der Klone x Länge der Region sequenziert) identifiziert. Die Darstellung dieser Zahl als durchschnittliche Anzahl der Mutationen pro 10K nt sequenziert macht die Zahl nutzerfreundlicher dann verlassen sie als pro Nukleotid. Zum Beispiel für den Wildtyp Bevölkerung in Tabelle 1, 55 mutations/121, 978 insgesamt Nukleotide X 10.000 = 4.51 Mutationen pro 10.000 Nukleotide sequenziert.
- Wenn die gleiche SNP erscheint auf einer großen Anzahl von Klonen, präsentieren zwei Werte, oder auch ausschließen, diese wiederholten Mutationen. Im Allgemeinen sind für ein Virus Bevölkerung aus einem homogenen Eltern (durch Plaque-Reinigung oder die an einer infektiösen Klon) produziert und passagiert nur ein paar Mal in der Zellkultur, hat eine positive Selektion nicht die Auswirkungen genug, um die Anhäufung von gleichen SNP und Mutation verursachen ausgeübt Frequenz gemessenen Werte auf diese Weise besser auf die Fehlerhäufigkeit der Polymerase mit minimalen Auswirkungen von positiven oder Reinigen Auswahl.
- Bestimmen Sie die Mutation Verteilung. Machen Sie eine Rangliste der Anzahl der Klone in jeder Population, die derzeit 0, 1, 2, 3, etc. .. Mutationen in der Region sequenziert.
- Berechnen viralen Vielfalt der Population durch paarweise Distanz gegenüber. Von den gereinigten und manuell bearbeitet Sequenzen vorbereiten eine Ausrichtung mit der entsprechenden Referenz Region. Es gibt verschiedene Alignment-Programme zur Verfügung: ClustalW / X ( http://www.clustal.org/ ), Muskel ( http://www.drive5.com/muscle/ ), Ebiox für Mac-Nutzer ( http://www. ebioinformatics.org / Ebiox / ), Make etc. sicher, dass alle Sequenzen die gleiche Länge, Ernte, wenn notwendig sind. Es wird empfohlen, dass der Start der Sequenzen eine Codierung Codon nach posterior Analyse zu erleichtern. Wir schlagen vor, dass man sämtliche Ausrichtungen in FASTA-Format, die sich leicht von den meisten Software ausgelesen wird.
- Für die Durchführung der paarweisen Abstand Analyse, Berechnung aller möglichen paarweisen Vergleiche zwischen den Sequenzen, die derselben Bevölkerung. Durchschnittliche Mutationen im Vergleich finden dann berechnet, um der Bevölkerung Heterogenität anzugeben.
- Die Werte für synonym (DS) und nicht-synonym (dN) in der Bevölkerung kann leicht erhalten werden. Wir finden die MEGA-Software (http://www.megasoftware.net/) nützlich und benutzerfreundlich für diese Art der Analyse.
- Um Auswahl Richtung geben wir zwei Möglichkeiten, obwohl sie nicht die einzigen, die möglich sind. 1) Finden Sie das Verhältnis zwischen dN und DS. Werte größer als 1 bedeuten eine positive Selektion. Werte kleiner als 1 bedeuten, Reinigung Auswahl. 2) Verwenden Sie die Datamonkey Webserver ( http://www.datamonkey.org/ ). Laden Sie Ihre Ausrichtungen in FASTA-Format und analysieren sie mit der SLAC-Modul. Dadurch erhalten Sie eine Schätzung der dN / dS.
- Statistische Analysen durchführen. Abhängig von der Datenmenge und der Anzahl der Sequenzen, können eine Vielzahl von Tests verwendet werden. Einige Studien haben auf Chi-Quadrat-Tests an der Gesamtzahl der Mutationen gegenüber Gesamtzahl der Konsens Nukleotiden bestehen, wenn alle Klone 9 sind kombiniert verlassen. Andere Studien haben exakten Fisher-Tests durchgeführt, berechnet auf die Zahl der Sequenzen präsentiert Mutationen im Vergleich zu der Zahl der Sequenzen ohne Mutationen 10. Wenn genug Mutationsdaten erzeugt wird, empfiehlt sich ein Platz Summe Test wie Mann-Whitney-U Um dies zu tun, zählen die Anzahl von Klonen in jedem Virus Bevölkerung nach der Anzahl der Mutationen auf jeder RNA sequenziert. Der Mann Whitney wird dann für Unterschiede in der Mutation Verteilung der Bevölkerung zu testen. Aus diesem Grund empfehlen wir die Sequenzierung von mindestens 800 Basenpaaren, um die Wahrscheinlichkeit des Findens Klone mit mehreren Mutationen zu erhöhen. Dieser Test ist robust, erfordert aber größeren Stichproben. Auf der anderen Seite, es bedarf nicht der gleichen Stichprobe für die beiden Populationen, die verglichen werden (z. B. werden die Proben aus Tabelle 1 n 1 = 148 und n 2 = 84).
8. Repräsentative Ergebnisse:
Die dosisabhängige Wirkung von mutagen Konzentration auf die Lebensfähigkeit der Zellen und Viren Lebensfähigkeit ist in Abbildung 1 dargestellt. In diesem Beispiel haben wir festgestellt, dass Viruspassage in 100 uM AZC wurde in Virustiter durch die gezielte 10,5-2 log reduziert, aber HeLa Lebensfähigkeit der Zellen wurde nicht negativ während der 2 Tage für Virus-Infektion erforderlich beeinflusst. Dieser Pilotversuch führte zur Wahl von 100 uM AZC Konzentration für die serielle Passage des Virus, um mutagen Widerstand zu wählen. Abbildung 2 zeigt die erste Reduktion in Titer von Entstehen einer mutagen Widerstand Phänotyp gefolgt. Während der ersten paar Passagen in mutagen, als tödliche Mutationen anhäufen, kommt es zu einem deutlichen Rückgang der Virustiter. Allmählich entsteht ein Mutagen-resistente Variante eine ihrer Entstehung fällt mit einer Rückkehr zu Virustiter unterscheidet sich nicht von unbehandelten Kontrollen. In dieser Phase stellt einen großen Anteil des Virus Bevölkerung der Resistenz-Mutation. Die Sequenzierung des Virus Bevölkerung zeigt die Aminosäure-Änderung (en) verantwortlich. Einmal identifiziert und isoliert oder neu generiert, kann der Mutagen-resistente Viren werden weniger empfindlich als der Wildtyp auf verschiedene RNA Mutagene (Basenanaloga unterschiedlicher Struktur, zum Beispiel). Abbildung 3 zeigt eine RNA mutagen resistent Coxsackie-Virus B3, die höher als Titer Wildtyp in Gegenwart von Ribavirin, 5-Fluorouracil und 5-Azacytidin und hohe MgCl 2 und MnCl 2. Breite Resistenz gegen RNA Mutagene ist ein starker Indikator für ein erhöhtes Replikationsgenauigkeit. Prüfen, ob die Replikation Kinetik der Treue Variante ist ähnlich zu Wildtyp-Virus im Vergleich der Mutation Frequenzen helfen werden. Abbildung 4 zeigt einen Schritt Wachstumskinetik einer High-Fidelity-Variante im Vergleich zum Wildtyp. Wenn die Replikation Preise und letzte Titer nicht ähnlich sind, dann sollten Maßnahmen ergriffen werden, um Viren Populationen von ähnlicher Größe, die die gleiche Anzahl von Runden-Replikation durchlaufen haben zu vergleichen. Die Verbindung zwischen RNA-Synthese-Rate und die Replikation Treue ist nicht gut charakterisiert, besonders in vivo. Eine langsamere Replikation Rate kann in einer verminderten Mutationsfrequenz (höhere Genauigkeit) führen, obwohl dies nicht eine absolute Regel, wie in Abbildung 4 dargestellt. Mit den oben genannten Parametern hergestellt wurde, kann die Mutation Frequenzen der Treue-Variante und Wildtyp-Populationen im Vergleich zu genetischen Bestätigung verändert Replikationsgenauigkeit erhalten. Abbildung 5 zeigt eine Sequenz-Alignment von einem Wildtyp-und High-Fidelity-Variante, mit Punktmutationen identifiziert. Die Mutationen gezählt werden, gestaffelt nach Anzahl der Mutationen pro Klon (Tabelle 1) und dargestellt als eine durchschnittliche Mutationsrate pro Einwohner, pro 10.000 Nukleotide sequenziert, Abbildung 6.

Abbildung 1. Die Bestimmung der optimalen Bedingungen für die RNA-mutagen Widerstand wählen:. Beibehaltung der hohen Zellvitalität mit einer moderaten (1-2 log drop in Virustiter) HeLa-Zellen wurden mit den angegebenen Konzentrationen von Ribavirin behandelten und infizierten mit Wildtyp-Virus Coxsackie B3 bei einer MOI von 0,01. 48 Stunden nach der Infektion wurde die Nachkommen Virus geerntet und Titer wurden durch TCID 50 bestimmt. Der prozentuale Anteil der überlebenden Zellen die Behandlung bei 48 Stunden, durch Trypanblau-Färbung bestimmt, ist unterhalb der x-Achse dargestellt. Die Ergebnisse zeigen, dass Konzentrationen von 100 und 200 uM Virustiter zu reduzieren um 1-2 log, ohne die Lebensfähigkeit der Zellen.
Abbildung 2. Serielle Passage in Anwesenheit von moderaten Konzentrationen von RNA Mutagene wählt für mutagen resistenten Populationen. In dieser Zahl, Chikungunya-Virus in HeLa-Zellen wurde in Gegenwart von 50 uM Ribavirin (graue Balken) passagiert. Control-Passagen wurden in Abwesenheit von Ribavirin (schwarze Balken) durchgeführt. Nach jedem Durchgang wurde das Virus Nachkommen von klassischen Plaque-Assay auf BHK-Zellen quantifiziert. Die mutagene Wirkung ist in den ersten Passagen deutlich (P1 und P2 im Vergleich zu p0 ab Bevölkerung) bei denen das behandelte Virustiter Drop von 2 log. Allmählich Titer normal (unbehandelt) Werte zurück. Keine signifikanten Unterschiede sind in Passage 5 Mutagen behandelt Bevölkerung im Vergleich zu unbehandelten beobachtet, was darauf hindeutet, dass resistente Varianten ausgewählt wurden. In der Tat identifiziert Konsens Sequenzierung der Bevölkerung einzigartige Mutationen im Virus Bevölkerung unterziehen Ribavirin Behandlung.
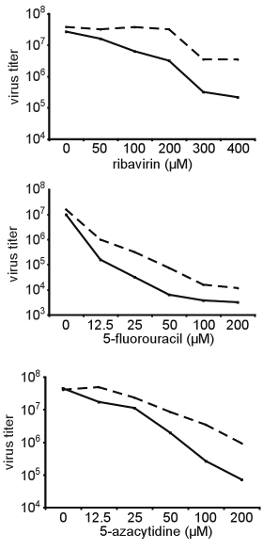
Abbildung 3. Bestätigung der breiten Widerstand auf Mutagene unterschiedlicher Struktur RNA. Sehen Sie hier wurde die High-Fidelity-A372V Variante des Coxsackie-Virus B3, die zunächst auf dem Bildschirm in Kapitel 3 beschrieben wurde isoliert aus einem infektiösen Klon erzeugt und getestet für seine relative Empfindlichkeit gegenüber verschiedenen Konzentrationen von verschiedenen RNA Mutagene (Ribavirin, 5-Fluorouracil, 5-Azacytidin). HeLa-Zellen wurden mit den angegebenen Konzentrationen von Ribavirin behandelten und infizierten mit Wildtyp-Virus Coxsackie B3 bei einer MOI von 0,01. 48 Stunden nach der Infektion wurde die Nachkommen Virus geerntet und Titer wurden durch TCID 50 bestimmt. Dargestellt sind die Titer von Wildtyp (durchgezogene Linien) und A372V Variante (gestrichelte Linien) als Funktion der Konzentration mutagen. A372V konsequent Titer höher als Wildtyp unter allen denkbaren Bedingungen getestet.
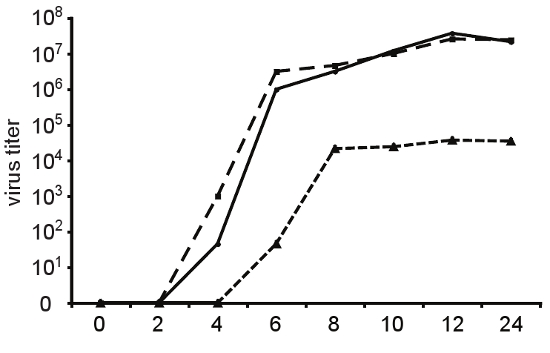
Abbildung 4. Replication Preise und Treue Varianten. Um die Ein-Schritt-Wachstumskinetik von Virus-Produktion zu bestimmen, wurden HeLa-Zellen bei einer MOI = 10 entweder mit Wildtyp (durchgezogene Linie), High-Fidelity-Variante A372V (lange Striche) oder replikationsdefizient Variante Cx64 (kurz infiziert Striche) von Coxsackie-Virus B3. Zu den Zeitpunkten angegeben, Virus Nachkommen von Zellen und Überstände durch Frost-Tau wurde geerntet und titriert durch TCID 50. Die Treue Erhöhung der A372V nicht mit einer beobachtbaren Replikation Fehler in Gewebekultur zusammenfallen. Die Variante Cx64 stellt eine erhebliche Verzögerung bei der Replikation Kinetik und erreicht maximale Titer, die 1000-fache niedriger als Wildtyp-Virus sind.
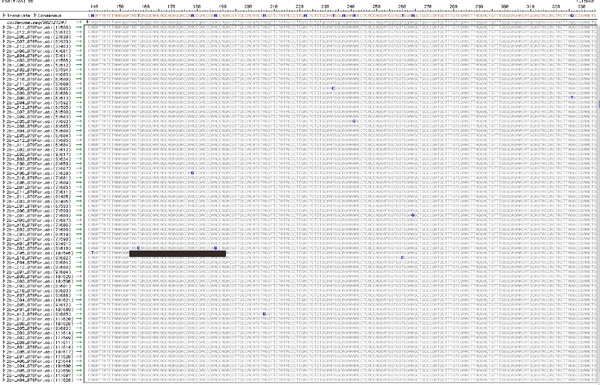
Abbildung 5. Die Angleichung der TopoTA klonierten Sequenzen von jedem Virus Bevölkerung. Mit dem Ansatz in § 7, jede Sequenz aus geklonten RT-PCR-Produkt erhalten beschriebenen stammt vermutlich aus einer einzigen, einzigartigen Genom innerhalb des gesamten Virus Bevölkerung und wäre somit, tragen einzigartige Mutationen. Die Abbildung zeigt einen typischen Ausrichtung, nach Bereinigung von schlechter Qualität Sequenzen und Visualisierung von SNPs. Die gesamte SNPs (10 in dieser Abbildung) innerhalb einer Population sind gezählt und die Anzahl der SNPs, die auf jedem Klon werden zur Kenntnis genommen. Zum Beispiel enthält der Klon von einem bar unterstrichen, 2 einzigartige Mutationen während 8 weitere Klone einer einzigen, einzigartigen Mutation enthalten. Diese Daten können verwendet werden, um Tabelle 1 zu kompilieren. Um eine größere Version dieses Bild bitte hier klicken .
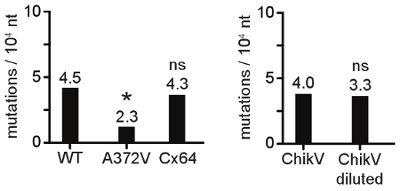
Abbildung 6. Grafische Darstellung der Mutation Frequenzen Viruspopulationen. Zur einfacheren Interpretation kann die numerische Daten aus Reihenfolge und statistische Analysen erhalten entweder als Diagramm oder Histogramm (hier abgebildet) vertreten sein. A372V Virus erzeugt weniger Mutationen als der Wildtyp und präsentiert eine signifikant niedrigere Mutationsrate (*, p <0,01). Die Cx64-Variante, die Titer repliziert 1000-fach niedriger als der Wildtyp, DruckEltern die gleiche Mutation Frequenz (ns, nicht signifikant) darauf hinweist, dass die Replikation Geschwindigkeit und Genauigkeit sind nicht notwendigerweise miteinander verbunden. Das gleiche Chikungunya-Virus (CHIKV) Bevölkerung ergibt ähnliche Mutation Frequenzen, ob das Virus Lager, oder ein 10 5-facher Verdünnung ist für die RNA-Extraktion verwendet.
Mutation Verteilung Zusammenfassung der statistischen Auswertung.
Hinweis: Für jeden Klon, ist es unerlässlich, dass die gleichen genomischen Region (und die Länge der Sequenz) abgedeckt ist. In diesem Fall, 859 Nukleotiden pro Klon. Dies ist entscheidend für statistische Auswertungen. Auf der anderen Seite, Rangsummen Tests für die statistische Analyse verwendet erfordern nicht die Stichprobengröße auf die gleiche ist, ist der Forscher frei Bevölkerung von unterschiedlichen Stichprobengröße zu vergleichen. Daher kann der 142 Klone der Wildtyp auf 84 Klone von A372V verglichen werden.
| # Klone mit n-Mutationen | Wildtyp | A372V |
| 7 Mutationen | 0 | 0 |
| 6 Mutationen | 0 | 0 |
| 5 Mutationen | 0 | 0 |
| 4 Mutationen | 0 | 0 |
| 3 Mutationen | 1 | 0 |
| 2 Mutationen | 6 | 2 |
| 1-Mutationen | 40 | 14 |
| 0-Mutationen | 95 | 68 |
| Insgesamt Mutationen | 55 | 18 |
| Insgesamt Klone sequenziert | 142 | 84 |
| Insgesamt Nukleotide sequenziert | 121.978 | 72.156 |
| Mutations/10 4 nt | 4,51 | 2,49 |
Tabelle 1. . Mutation Verteilung Zusammenfassung der statistischen Auswertung Hinweis: Für jeden Klon, ist es unerlässlich, dass die gleichen genomischen Region (und die Länge der Sequenz) abgedeckt ist. In diesem Fall, 859 Nukleotiden pro Klon. Dies ist entscheidend für statistische Auswertungen. Auf der anderen Seite, Rangsummen Tests für die statistische Analyse verwendet erfordern nicht die Stichprobengröße auf die gleiche ist, ist der Forscher frei Bevölkerung von unterschiedlichen Stichprobengröße zu vergleichen. Daher kann der 142 Klone der Wildtyp auf 84 Klone von A372V verglichen werden.
Diskussion
Wahl der Zell-Linie. Die Wirksamkeit von Basis-Analoga als RNA Mutagene korreliert mit ihrer relativen Aufnahme durch verschiedene Zelltypen 11. Wenn der Zelllinie, die normalerweise für Viruspassage dient erweist sich als refraktär Aufnahme oder zu empfindlich (hohe Zelltoxizität) mutagen, kann es notwendig sein, um eine andere Zelllinie, die diese Anforderungen erfüllt und ist immer noch permissive die virale Replikation verwenden. Sobald die mutagen Widerstand Variante isoliert ist, kann der Rest der Charakterisierung in der ursprünglichen, bevorzugte Zelllinie durchgeführt werden. In unserer Erfahrung, HeLa Zellen leicht nehmen mutagen; BHK-Zellen benötigen bis zu 10-fach höhere Konzentrationen und Vero-Zellen sind refraktär gegenüber mutagen Aufnahme.
Wahl des mutagen. Bei dem Versuch, Treue-Varianten durch Mutagenbehandlung, die Wahrscheinlichkeit eines Erfolgs zu isolieren wird erhöht, wenn mehr als eine Art von Mutagen verwendet wird. Basis analoger Mutagene unterschiedlicher Struktur, die fälschlicherweise in Genome während der Replikation eingebaut wird überwiegend induzieren Ergebnis in eine bestimmte Untergruppe von Mutationen in den nachfolgenden Replikationszyklen: Ribavirin behandelt Gefälligkeiten GtoA und CtoU Übergang Mutationen 12; 5-Azacytidin hat eine ähnliche Tendenz, mit der Neben der CtoG und GtoC Transversionen 13; 5-Fluorouracil bevorzugt induziert Atog und UTOC Übergänge 14. Alternativ höhere Konzentrationen von Mg 2 + oder Mn 2 + können dem Medium ergänzt werden, um die gesamte Mutationsfrequenz von RNA-Viren, ohne die Vorspannung über 12 beschrieben zu erhöhen. Je nach Virus "Codon-Sequenzen und die Codon-Änderungen erforderlich, um eine Treue-Variante zu erzeugen, einige von diesen Bedingungen wird die Entstehung dieser Variante gegenüber anderen zu bevorzugen. Für die höheren Treue Poliovirus G64S und Coxsackie Virus A372V, Ribavirin Behandlung am ehesten für die Varianten ausgewählt, weil die erforderliche Atog Übergang an die Codon-Website entsprach der Mutationen hauptsächlich durch dieses Ribavirin generiert.
MOI vs Populationsgröße. In der Virologie, Pay-Protokolle für Gewebekultur Infektion besonderes Augenmerk auf die Multiplizität der Infektion (MOI), um die Ansammlung von defekten störende Partikel (low MOI) zu vermeiden oder Rekombination zwischen Viren zu fördern (hohes MOI), für Beispiel. So wählen Sie für die Entstehung Veranstaltungen über serielle Passage ist es auch wichtig, Viruspopulation Größe zu betrachten. Da die resistente Mutante existiert zunächst bei einer niedrigen Frequenz ist es am besten, einen möglichst großen Bevölkerungszahl wie möglich von einem Kanal übertragen auf die nächste (10 5 -10 6 Viren, z. B.) nicht zu verlieren diese neuen Varianten bei jeder Passage. Intensivierung der Größe von gut oder Kolben (Anzahl der Zellen infiziert) kann dazu beitragen, den Anstieg der MOI zu minimieren, wenn dies von Belang ist. Auf der anderen Seite, für Experimente, in denen die Empfindlichkeit eines Virus mutagen getestet wird, ist gering MOI Infektion durchgeführt, um die Anzahl der Replikationszyklen vorkommende in das Experiment zu erhöhen und die Rettung von mutagenisierten Genomen zu vermeiden durch eine höhere Fitness Genome durch Komplementation bei co-infizierten Zellen. Dies ist wichtig, da die Mutationen für die Nachkommenschaft Genome in der ersten Runde der Replikation generiert wird nicht sofort erkannt werden. Die meisten dieser mutagenisierten RNAs noch in Virion verpackt werden. Es ist in die nächste Runde der Infektion, die letalen Mutationen in diesen Genomen wird in einer abgebrochenen Replikationszyklus führen, und Reduzierung der Virustiter. Es kann notwendig sein, um für mehrere Runden der Akkumulation von Mutationen erlauben, bevor eine signifikante Wirkung von tödlichen Mutagenese zu beobachten ist. Schließlich, wenn mehr als die Passage-Serie in der Gegenwart von mutagen, die Virustiter bis Aussterben weiter abfällt, dann sollte der Forscher versuchen Passagierung Virus in allmählich steigenden Mengen von mutagen (ausgehend von einem sehr niedrigen Konzentrationen).
Isolation und Generierung der RNA mutagen resistenten Klon aus der RNA mutagen-resistenten Population. RNA Mutagene einführen mehrere zufällige Mutationen zu jedem Genom, sondern Selektion auf Resistenz wird nur bereichern (und zu beheben, um Consensus-Sequenz) die Resistenz-Mutation. Zur Identifizierung dieser Mutation, wir Abfolge der Mutagen resistente Population (Konsens der Bevölkerung) und nicht die einzelnen Viren. Daher sind die einzigen, zufällige Mutationen durch die mutagen erstellt wurden, nicht in der Reihenfolge erkannt, nur die Mutationen, die im Konsens Veränderungen nach Auswahl, zu finden sind. In unserer Erfahrung sind wir nur zu identifizieren ein oder zwei solcher Konsensus-Sequenz ändert. Sobald die mutagen resistenten Population erhalten wird und die Resistenz-Mutation identifiziert ist, ist es notwendig, eine reine Bestandsaufnahme dieser Variante zu erzeugen. Oben beschrieben, haben wir eine Gedenktafel Reinigungsverfahren. Alternativ, wenn das Virus von Interesse führt nicht zu leicht identifizierbaren Plaques, können die gewünschte Variante purif werdenIED durch limitierende Verdünnung. Dieser Ansatz ist im Wesentlichen ein TCID 50 in 96-Well-Format, in denen das Virus Lager so verdünnt, dass weniger als 50% der Brunnen infiziert sind. Mit dieser Verdünnung wird der gleiche Ansatz wie oben genommen, bei der Isolierung bis zu 10 individuelle Varianten und bestätigt ihre Sequenzen. Wie bereits erwähnt, in den besten Fällen ist eine infektiöse cDNA Klon des Virus-Stamm zur Verfügung. Isolation der Variante wäre also nicht notwendig sein. Nach unserer Erfahrung sind Treue Varianten das Ergebnis der einzelnen Aminosäure-Substitutionen und kann somit unter Verwendung einfacher, kommerzialisiert Mutagenese-Kits wie Quikchange (Agilent) werden. Eine zweite Möglichkeit ist, einen cDNA-Klon von einem eng verwandten Stamm verwenden. Allerdings, wenn ein verwandter Stamm verwendet wird, empfiehlt sich die Verwendung sowohl diesen Ansatz und die Virusisolierung (zB Plaque-Reinigung), weil wir festgestellt, dass die gleiche Treue zu verändern Mutation auf beiden eng verwandten Viren werden nicht unbedingt die gleiche Wirkung haben.
Fidelity und Replikation. Selektion von RNA mutagen resistente Varianten haben in der Isolation sowohl höhere als auch niedrigere Treue Varianten mit Wachstum, die ähnliche Merkmale auf ihre Wildtyp-Gegenstücke 4,12,15 sind geführt. Derzeit ist das Bindeglied zwischen Polymerase Erwerbsquoten und Treue nicht vollständig verstanden. In-vitro-biochemische Untersuchungen unter Verwendung von gereinigten RNA-Polymerase haben gezeigt, dass eine höhere Klangtreue Varianten langsamer Verarbeitungsgeschwindigkeit haben, während niedrigere Treue Varianten schnellere Verarbeitung 1-3,12 neigen. In Gewebekulturen, sind diese Unterschiede in der Regel nicht erkennbar, was darauf hindeutet, dass die Verfügbarkeit von Ressourcen, anstatt intrinsische Polymerase-Aktivität Kinetik ist der geschwindigkeitsbestimmende Schritt. Wenn die Treue Variante repliziert mit Kinetik, die sich nicht signifikant von Wildtyp-, dann einen Vergleich ihrer Mutation Frequenzen können direkt vorgenommen werden. Wenn eine sehr signifikante Veränderung bei der Replikation Kinetik vorhanden ist, dann sollten die Daten normalisiert, um zur kinetischen Unterschiede, zum Beispiel durch den Vergleich Viren, die die gleiche Anzahl von Replikationszyklen unterzogen haben Konto. In unserer Erfahrung, auch wenn keine signifikanten Unterschiede in einem Schritt Wachstumskinetik zwischen Wildtyp und High-Fidelity-Varianten beobachtet wurden, beobachteten wir, dass eine höhere Klangtreue Varianten durchweg höhere Titer (innerhalb von 1 log) im Vergleich zum Wildtyp, aber sie machen etwas weniger RNA (innerhalb der gleichen Größenordnung), weiter darauf hindeutet, dass die Genome sie produzieren weniger Mutationen enthalten und somit mehr infektiös.
Die Probenaufbereitung und Sequenzierung. Für alle Schritte in dieser Protokolle ist es unerlässlich, dass High-Fidelity-, Korrektur-Enzymen für die PCR und RT-PCR dienen zur Begrenzung der Einführung zusätzlicher Mutationen, da sie nicht von biologisch relevanten Mutationen unterschieden werden. Es ist wichtig, dass das Virus Populationen verglichen werden unter den gleichen Bedingungen (Passage Geschichte, Gewebekulturmedium, Temperatur, RNA-Extraktions-Verfahren, RT-PCR-Protokolle, etc.) erstellt worden Es ist auch wichtig, um sicherzustellen, dass genügend Ausgangsmaterial wurde erhalten aus der RNA-Extraktion, so dass eine starke Bande von RT-PCR generiert. A 1 / 100 Verdünnung der RNA-Probe sollte auch eine nachweisbare RT-PCR-Bande, was darauf hinweist, dass die Probe eine ausreichende Zahl von RNA-Molekülen auf Vertretung Bias zu vermeiden (Verstärkung der gleiche Genom mehrfach) enthält. Da Mutationsfrequenz ist eine Distribution, würde man erwarten, dass ähnliche Werte unabhängig von der Populationsgröße wird erreicht werden, sofern die genannten Bias nicht auftritt. Wie Abbildung 6 zeigt, gibt ein 10 5-fachen Verdünnung von einem Virus Lager eine Mutation Frequenz nicht wesentlich verschieden von den Elterntieren.
Bis in die optimale Bedingungen für TopoTA Klonen gefunden werden, bestätigen das Vorhandensein von Einsätzen nach blau / weiß-Screening, durch Kolonie-PCR-Sequenzierung vor. Als Kontrolle für Mutationsanalysen Rauschen (Mutationen durch RT-PCR und Sequenzierung eingeführt), Klon eines PCR-Produkts aus einem Plasmid mit dem gleichen Virus-Sequenz und / oder geklont und sequenziert RT-PCR-Produkte von in vitro transkribierten RNA entsprechend Virusgenom (werden bewusst, dass verschiedene in-vitro-Transkription Enzyme haben unterschiedliche Fehlerraten und dürfen nicht geben wertvolle Informationen über die wahren Hintergründe Fehler in der Prozedur). Einige Viren-Sequenzen können giftig für Bakterien, so ist es wichtig, diese vor einer Entscheidung über die Region des viralen Genoms für Mutationsfrequenz sequenziert werden zu überprüfen. Bei der Analyse der Sequenzen durch TopoTA erhalten, beachten Sie, dass jeder Klon sollte nur ein insert / Sequenz enthalten. Wenn ein Doppel-Peak beobachtet, was auf eine gemischte Bevölkerung, ist es möglich, dass zwei benachbarte Bakterienkolonien ausgewählt wurden. Es ist auch möglich, obwohl sehr unwahrscheinlich, die niedrigen Frequenzen Mutation in bakteriellen Replikation gegeben, dass die Mutation während der Amplifikation von Plasmid wurde in der Bakterienkultur eingeführt. In Plaque purified Populationen kann eine Doppel-Spitze repräsentieren überlappende Plaques, oder ein Virus, dass der Erwerb einer neuen Mutation oder Zurücksetzen eine Mutation während der Plaque-Entwicklung. Seien Sie konsequent und entscheiden, ob zu zählen oder nicht zählen diese Mutationen.
Schließlich, im Auge behalten, dass die Mutation verwendeten Frequenzen hier relative Werte. Sie gelten nur in den Vergleich Viruspopulationen unter den gleichen Bedingungen aufgewachsen, und sequenziert über den gleichen Bereich! Sie sollten nicht als absolute Werte der Mutationsrate, oder die Mutation Häufigkeit des Genoms als Ganzes betrachtet werden. Allerdings, wenn die Bedingungen geregelt sind, erlauben sie eine reproduzierbare, quantitative Vergleiche von Unterschieden in Mutation Verbreitung und Häufigkeit.
Offenlegungen
Danksagungen
Diese Arbeit wurde durch Mittel aus dem Medizin-und Gesundheitsforschung Zuschuss von der Stadt Paris, die Französisch Nationalen gewähren ANR-09-JCJC-0118-1, und der ERC Starting Grant RNAvirusPopDivNVax Projekt nicht unterstützt. 242719.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|
| Ribavirin | Sigma | R9644-10MG | |
| 5-Fluorouracil | Sigma | F6627-1G | |
| 5-Azacytidin | Sigma | A2385-100MG | |
| MgCl 2 | Sigma | M1028-100ML | |
| MnCl 2 | Sigma | M1787 | |
| Trypanblau | Sigma | T8154-20ML | |
| TopoTA Cloning Kit | Invitrogen | 10351021 | |
| Quikchange Mutagenese-Kit | Agilent | 200516 | Wenn ein cDNA infektiösen Klon ist verfügbar |
| 96-Well-Miniprep Kit | Macherey-Nagel | 740625 | |
| Lasergene, Sequencher | DNAstar, Gene Codes Corporation | www.dnastar.com www.genecodes.com | Oder andere Ausrichtung Software |
Referenzen
- Arias, A. Determinants of RNA-dependent RNA polymerase (in)fidelity revealed by kinetic analysis of the polymerase encoded by a foot-and-mouth disease virus mutant with reduced sensitivity to ribavirin. J Virol. 82, 12346-12355 (2008).
- Arnold, J. J., Vignuzzi, M., Stone, J. K., Andino, R., Cameron, C. E. Remote site control of an active site fidelity checkpoint in a viral RNA-dependent RNA polymerase. J Biol Chem. 280, 25706-25716 (2005).
- Korneeva, V. S., Cameron, C. E. Structure-function relationships of the viral RNA-dependent RNA polymerase: fidelity, replication speed, and initiation mechanism determined by a residue in the ribose-binding pocket. J Biol Chem. 282, 16135-16145 (2007).
- Pfeiffer, J. K., Kirkegaard, K. A single mutation in poliovirus RNA-dependent RNA polymerase confers resistance to mutagenic nucleotide analogs via increased fidelity. Proc Natl Acad Sci U S A. 100, 7289-7294 (2003).
- Pfeiffer, J. K., Kirkegaard, K. Increased fidelity reduces poliovirus fitness and virulence under selective pressure in mice. PLoS Pathog. 1, e11-e11 (2005).
- Vignuzzi, M., Stone, J. K., Arnold, J. J., Cameron, C. E., Andino, R. Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population. Nature. 439, 344-348 (2006).
- Vignuzzi, M., Wendt, E., Andino, R. Engineering attenuated virus vaccines by controlling replication fidelity. Nat Med. 14, 154-161 (2008).
- Crotty, S., Cameron, C., Andino, R. Ribavirin's antiviral mechanism of action: lethal mutagenesis. J Mol Med. 80, 86-95 (2002).
- Coffey, L. L., Vignuzzi, M. Host alternation of chikungunya virus increases fitness while restricting population diversity and adaptability to novel selective pressures. J Virol. 85, 1025-1035 (2011).
- Ciota, A. T. Role of the mutant spectrum in adaptation and replication of West Nile virus. J Gen Virol. 88, 865-874 (2007).
- Ibarra, K. D., Pfeiffer, J. K. Reduced ribavirin antiviral efficacy via nucleoside transporter-mediated drug resistance. J Virol. 83, 4538-4547 (2009).
- Levi, L. I. Fidelity variants of RNA dependent RNA polymerases uncover an indirect, mutagenic activity of amiloride compounds. PLoS Pathog. 6, e1001163-e1001163 (2010).
- Sierra, S., Dávila, M., Lowenstein, P. R., Domingo, E. Response of foot-and-mouth disease virus to increased mutagenesis: influence of viral load and fitness in loss of infectivity. J Virol. 74, 8316-8323 (2000).
- Ruiz-Jarabo, C. M., Ly, C., Domingo, E., de la Torre, J. C. Lethal mutagenesis of the prototypic arenavirus lymphocytic choriomeningitis virus (LCMV). Virology. 308, 37-47 (2003).
- Sierra, M. Foot-and-mouth disease virus mutant with decreased sensitivity to ribavirin: implications for error catastrophe. J Virol. 81, 2012-2024 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten