Method Article
Isolement des variantes des virus à ARN de Fidelity et caractérisation de la fréquence de mutation de virus
Dans cet article
Résumé
Le présent article décrit les étapes nécessaires pour isoler et caractériser l'ARN polymérase variantes fidélité des virus à ARN et la façon d'utiliser les données de fréquence de mutation pour confirmer les modifications fidélité dans la culture de tissus.
Résumé
Les virus à ARN utilisent l'ARN dépendante ARN polymérases à répliquer leurs génomes. Le taux d'erreur intrinsèque élevée de ces enzymes est un contributeur important à la génération de la diversité de la population extrêmes qui facilite l'adaptation du virus et son évolution. L'augmentation de preuve montre que le taux d'erreur intrinsèque, et les fréquences de mutation résultant, des virus à ARN peuvent être modulés par de subtiles changements d'acides aminés de la polymérase virale. Bien que des dosages biochimiques existent pour certains ARN polymérases virales qui permettent une mesure quantitative de l'incorporation de fidélité, ici nous décrire une méthode simple pour mesurer les fréquences de mutation des virus à ARN qui s'est avéré être aussi précis que des approches biochimiques dans l'identification des mutations altérant la fidélité. L'approche utilise les techniques classiques virologiques et séquençage qui peuvent être effectuées dans les laboratoires les plus biologie. Basé sur notre expérience avec un certain nombre de virus différents, nous avons identifié les étapes clés qui doivent être optimisés pour augmenter la probabilité d'isoler variantes fidélité et générer des données de la signification statistique. L'isolement et la caractérisation de mutations altérant la fidélité peuvent fournir de nouveaux aperçus sur la structure et la fonction polymérase 1-3. En outre, ces variantes de fidélité peuvent être des outils utiles pour caractériser les mécanismes d'adaptation du virus et l'évolution 4-7.
Protocole
1. Déterminer la gamme de concentrations mutagène qui est très peu toxique pour les cellules
Le but de cet exercice est de déterminer quelle gamme de concentrations mutagène peut être utilisé pendant une infection sans excès de toxicité cellulaire. Essentiellement, vous voulez reproduire les conditions qui seront nécessaires pour l'infection de virus. Pour la plupart des virus, des infections durent entre 2 et 7 jours. Préparer assez d'assiettes pour les cellules de l'échantillon à chaque jour. Si des cellules non adhérentes sont utilisés, de modifier le protocole en conséquence.
- Le jour avant l'expérience, les graines de 7 x 10 5 cellules HeLa / puits dans une plaque à 6 puits qui permettent d'atteindre un sous-confluentes (75%) monocouche du jour de l'expérience. Chaque puits de la plaque sera traité avec une concentration différente de mutagène, permettant une gamme de 6 concentrations.
- Le jour de l'expérience, préparer des dilutions mutagène en milieu de culture tissulaire. Pour les cellules HeLa, utilisez une fourchette de 0 à 1000 um pour les analogues de base (ribavirine, le 5-fluorouracile, 5-azacytidine), 0 à 50 mM pour MgCl 2, et de 0 à 5 mm pour MnCl2.
- Aspirer le milieu à partir des puits et la remplacer par 2 ml mutagène-complété moyen et retour à la couveuse.
- Toutes les 24 heures, utilisez une plaque de cellules pour vérifier la viabilité cellulaire. Ceci peut être accompli en effectuant coloration bleu trypan ou en utilisant la fluorescence commercialisé / luminescence tests (par exemple Promega CellTiter-Glo ® dosage luminescent viabilité cellulaire).
- Pour la coloration exclusion du bleu trypan, détacher les cellules d'une plaque (traitées avec différentes concentrations d'agent mutagène) et en douceur les cellules par centrifugation.
- Jeter les cellules surnageant et remettre en PBS (sérum peut interférer avec la coloration).
- Mélanger 1 volume de suspension cellulaire dans du PBS avec 1 volume de 0,4% de bleu trypan, incuber pendant 2 minutes à température ambiante.
- Dans un hématimètre, compter le viables (non colorées) et non viables (bleu teinté) des cellules. Calculer le pourcentage de cellules viables pour chaque concentration de substance mutagène, y compris le témoin non traité. Nous constatons que les conditions de ce résultat en moins de 50% de mort cellulaire par le critère d'infection (lorsque les titres sont atteintes) sont idéales pour l'isolation de résistance mutagène.
2. Déterminer la concentration optimale non mutagène toxique qui réduit modérément les titres de virus (environ 0,5 à 2 log de réduction)
Cet exercice sert à déterminer la concentration de substance mutagène qui exercent une forte pression sélective, sans trop mutagenèse la population. Pour l'ARN mutagènes, nous constatons que cela correspond à 10,5 à 2 bûches réductions du titre du virus. A ces concentrations, chaque génome est muté dans au moins une ou deux positions. Le mutagène peut aider à la génération de la mutation de résistance, qui seront ensuite sélectionnés sur le passage. Si un trop grand nombre de mutations sont introduites (à des concentrations très élevées mutagène), les mutants porteurs de la mutation de résistance seront eux-mêmes mortellement mutagenèse, entravant leur isolement.
- Plaques semences avec des cellules en utilisant les mêmes conditions que pour l'étape 1
- Le jour de l'expérience, préparer des dilutions mutagène en milieu de culture tissulaire. Utilisez la même gamme de concentrations aussi déterminé ci-dessus, mais excluent les concentrations qui ont entraîné la mort cellulaire dans plus de 50%. Préparez assez moyenne pour couvrir chaque puits deux fois (4 ml par puits), permettant un prétraitement des cellules avant l'infection, avec chaque concentration d'agent mutagène.
- Aspirer les cellules moyennes et prétraiter avec mutagène par incubation dans mutagène-complété moyen pendant 2 heures. Pour la plupart des types cellulaires, ce n'est le temps nécessaire pour l'absorption du mutagène.
- Retirer moyennes et infecter avec le virus à faible multiplicité d'infection MOI (0,1 ou 0,01) dans un volume minimum (200 ul pour plaques de 6 puits). Incuber 15-60 minutes pour permettre aux virus d'infecter les cellules. Rocher de la plaque à intervalles réguliers pour s'assurer que l'inoculum couvre la monocouche cellulaire.
- Aspirer le virus inoculé et laver deux fois avec 2 ml de PBS pour enlever autant de l'inoculum que possible.
- Ajouter moyenne avec les concentrations appropriées du mutagène à chaque puits et incuber les cellules pour l'équivalent de 3-6 cycles de réplication.
- Le virus de la récolte de chaque puits et de déterminer l'effet antiviral sur le titre du virus. Cela peut être fait par dosage de plaque standard ou limitant la dilution (DICT 50). Note: La quantification des virus par des techniques qui mesurent la synthèse d'ARN ne peut pas être approprié en raison des effets mutagènes peuvent pas être détectées. Génomes mutagénisées contenant des mutations létales peuvent encore être détectés par qRT PCR par exemple, mais ne serait pas observée dans les essais de viabilité du virus.
- Du titres calculé, mesurer la concentration de substance mutagène qui réduit les titres de virus (par rapport à la lutte contre l'infection non traitée) par 10,5 à 2 billes qui n'est également pas très toxique pour les cellules (idéalement, moins de 50% la toxicité).
3. Isolement et identificationcation de variantes résistantes mutagène
Effectuer grands passages taille de la population de la concentration optimale mutagène défini ci-dessus et vérifier les titres de virus dans la série passage. Comme contrôle, le virus de passage dans le milieu de croissance sans aucun agent mutagène. Comme un autre contrôle pour surveiller l'émergence potentielle de particules défectives interférentes (DI), effectuer des infections nouvelles en l'absence du mutagène à chaque étape de passage (contrôle unpassaged).
- Le jour avant l'infection, les graines de 25 cm 2 flacons avec 1,5 x 10 6 cellules HeLa (tailles autre flacon peut être utilisé) pour obtenir des sous-confluentes monocouches le jour suivant.
- Le jour de l'infection, prétraiter les cellules pendant 2 heures avec un milieu contenant la concentration optimale de chaque agent mutagène, déterminé à l'article 2.
- Retirer moyen et infecter les cellules avec un volume minimal à une MOI de 1 ou le plus grand MOI qui ne résulte pas de la formation de particules défectueuses (DI) pour le virus interférant étudié.
- Après 30-60 minutes de l'infection, aspirer inoculum et laver deux fois avec du PBS, puis ajoutez un milieu frais complété avec mutagène sur les concentrations appropriées.
- Incuber pendant la période de temps déterminée dans les sections 1 et 2 qui correspond au titre du virus au maximum dans ces conditions. Récolte du virus progéniture.
- Virus de titre à chaque passage et de répéter les 3 étapes précédentes.
- Pendant les passages des premières années, les titres de virus mutagène des échantillons traités devrait baisser en conséquence, par rapport à l'original et titre du virus de la commande (non traitées et unpassaged) les titres de virus. Si les titres de virus mutagène des passages échantillons remonter aux mêmes niveaux que le témoin non traité, la population contient probablement une variante résistante mutagène. Jusqu'à 20 ou 30 passages peuvent être nécessaires, mais nous avons isolé la plupart de nos variantes fidélité entre 5 et 15 passages.
- Une fois les titres de virus pour une série passage donné atteindre la même ampleur que les titres témoin non traité, extrait d'ARN de tous les échantillons, y compris les témoins non traités par le nombre même passage. Des kits d'extraction d'ARN ou d'extraction Trizol peuvent être utilisés.
- Effectuer la RT-PCR utilisant des amorces qui amplifient les gènes polymérase ou réplicase du virus de l'intérêt. Dans une deuxième étape, l'ensemble du génome (au moins les régions de codage) doivent être séquencés pour déterminer si phénotypes de résistance carte pour d'autres gènes du virus aussi bien. Ceci est particulièrement important pour les agents mutagènes de base analogique, comme la ribavirine, qui affecte d'autres aspects du virus et la fonction des cellules. Dans ce cas, la variante peut être résistante à l'un de ces autres activités antivirales et ne sera pas une variante fidélité.
- Purifier les produits de RT-PCR en utilisant un kit de purification de PCR et la séquence d'obtenir la séquence mutagène-population résistante consensus. Inclure les contrôles de fond pour l'erreur de séquençage (voir discussion).
- En utilisant un logiciel d'alignement de séquences et la séquence consensus du virus comme une référence, d'aligner les séquences. Identifier les mutations ponctuelles de nouveaux, avec une attention particulière à celles qui apparaissent exclusivement dans la population mutagène traités au passage où les titres de virus d'atteindre des niveaux normaux. Si cette mutation n'est pas présente dans les passages plus tôt et pas présente dans les témoins non traités par le nombre même passage (indiquant l'adaptation au passage de culture cellulaire), alors cette mutation est probablement responsable, au moins en partie, pour la résistance mutagène. Ne comptez pas sur la base dite séquence (la version texte de la séquence) et le logiciel d'alignement seul. Vérifiez les chromatogrammes des pics minoritaires qui peuvent avoir été oubliées par les logiciels d'alignement. Un mutant qui représente 20-30% de la population totale devrait toujours afficher comme un pic, mais trop petits pour être identifiés comme un «N» par analyse de séquence standard.
4. Une fois qu'une mutation a été identifiée, d'isoler ou de générer la variante et de confirmer le phénotype de résistance à plusieurs agents mutagènes ARN
Ensuite, la variante présentant la mutation identifiée est isolé afin de confirmer son lien avec le phénotype de résistance. Il est essentiel que la mutation soupçonnée de changer la fidélité est étudiée dans un contexte génétique propre (qui est, ne présentant pas de mutations supplémentaires ailleurs dans le génome). Dans le meilleur des cas, un clone d'ADNc infectieux existe qui permettrait à la génération d'un stock de la variante mutagène résistant par mutagenèse dirigée sur un fond propre génétique. Dans ce cas, l'article 4 n'est pas nécessaire. Cependant, si un clone d'ADNc n'est pas disponible, l'isolement peut être fait par purification sur plaque de virus, décrit ci-dessous. Plus d'un tour de purification de plaque peut être nécessaire d'isoler la variante sur une surface propre, le patrimoine génétique.
- L'isolement du mutant résistant mutagène par dosage de plaque.
- Pour isoler le mutant identifié, effectuez un essai de plaque standard sous agarose (0,5 à 1% final poids / volume) dans la superposition des plaques 6 puits. Préparer des dilutions en série du virus, sur la base des titres d'actions, au DILutions qui produisent entre 10 et 50 ainsi séparés des plaques.
- Lorsque les plaques sont clairement visibles (généralement 2 à 5 jours après l'infection, selon le virus), marquer l'emplacement des plaques sur les assiettes et les aide d'une pipette P200 avec le bout du filtre, doucement plonger la pointe grâce à la superposition d'agarose en faisant attention de ne pas déloger et changer de position de l'overlay (qui entraînerait la contamination croisée des plaques individuelles).
- Soulevez délicatement la pointe de la superposition et le transfert de la prise d'agarose contenu dans l'embout d'une eppendorf contenant 250 ul de milieu et de vortex. Ne vous inquiétez pas si la pointe qui est supprimé ne contient pas d'agarose, pour de nombreux virus à ARN, une plaque en moyenne pour les virus à ARN de nombreux contient 10 5 virus et une quantité suffisante sera transféré par simple toucher de la pointe à la surface de la plaque.
- Pick up à 10 plaques par traitement mutagène. Selon les chromatogrammes de séquençage qui a identifié la mutation, d'estimer quel pourcentage environ de la population contient la mutation désirée. L'objectif est d'isoler trois ou quatre plaques avec la mutation. Certains de ces mutants seront également effectuer d'autres, des mutations non désirées, qui sera plus tard identifiée par séquençage.
- Extrait de l'ARN à partir de ces échantillons (mais économiser la moitié de l'échantillon pour faire un plus grand stock de virus), et effectuer des RT-PCR qui permettra le séquençage du génome entier. En moyenne, les virus à ARN contient jusqu'à deux différences par rapport aux mutations de la séquence consensus: ainsi, la séquence 3 ou 4 purifié sur plaque virus à un moment d'identifier l'isolat qui contient la mutation désirée sans aucune mutations supplémentaires.
- Une fois identifiés, faire un plus grand stock de ce virus pour toutes les études en aval, en utilisant l'échantillon purifié sur plaque obtenu ci-dessus pour infecter un grand flacon de cellules, par exemple 8x10 6 cellules HeLa dans un flacon T75 ..
- Confirmer la sensibilité mutagène / résistance conférée par la mutation identifiée.
- Utilisation du clone isolé ou nouvellement générés, et un virus de type sauvage témoin préparé dans des conditions similaires, répéter les expériences dans la section 2 en utilisant soit une gamme complète de concentrations mutagène, ou la concentration à laquelle la mutation de résistance a été généré.
- Utiliser plusieurs conditions différentes ARN mutagène (ribavirine, le 5-fluorouracile, 5-azacytidine, l'augmentation de Mg 2 +, Mn 2 +). Si la variante polymérase est résistant à plus d'une sorte de substance mutagène, alors il est plus probable que cette variante est de haute fidélité. Alternativement, il est possible qu'une mutation de résistance est spécifique à une seule condition mutagènes, d'autant que certains de ces composés affectent les virus à ARN à travers un certain nombre de mécanismes 8.
5. Vérifier les tarifs réplication
Depuis la fidélité qui altèrent le plus souvent des mutations carte pour la polymérase, il est possible que la mutation polymérase même modifier considérablement la cinétique de réplication et il est important de déterminer les similarités et les différences dans la réplication qui va permettre une meilleure comparaison des différences de fréquences de mutation effectuée ci-dessous. Pour ce faire, examiner la réplication par au moins deux approches complémentaires - l'une qui examine la production de virus et un autre qui examine la synthèse d'ARN.
- Une étape cinétique de croissance du virus
- Le jour avant l'expérience, les graines de plaques de 6 puits, au besoin, une plaque par point le temps d'être testés. Pensez à utiliser des puits en triple pour chaque mutant et le virus de type sauvage.
- Le jour de l'expérience, éliminer le milieu et d'infecter les puits avec chaque virus à une MOI de 10 pour s'assurer que chaque cellule est infectée simultanément. Incuber 30-60 minutes à 37 ° C.
- Rocher plaques toutes les 10 minutes pour éviter le dessèchement de la monocouche cellulaire. Enlever virus et laver deux fois avec 2 ml de PBS. Il est important d'enlever autant de l'inoculum que possible. Remplacez-la par le milieu de croissance.
- Suite à l'infection au temps = 0, la récolte du virus d'une assiette. Retour à l'incubateur et plaques de récolte du virus à intervalles réguliers qui couvrent un cycle de réplication unique (par exemple pour 3h, 5h, 7h, 9h, 12h, 24h).
- Virus de titre, récolté à chaque point du temps (essai de plaque, par exemple, DICT50, dosage FFU), et le graphique des courbes de croissance de titre en fonction du temps.
- Cinétique de synthèse de l'ARN
La cinétique de la synthèse de l'ARN peut être contrôlé en utilisant l'une des approches ci-dessous. Si possible, les mêmes échantillons utilisés pour déterminer la cinétique de croissance en une seule étape devrait être utilisée pour mesurer les niveaux d'ARN.- qRT-PCR. Cette procédure donne des mesures hautement quantitative de la réplication sur une grande plage, à partir d'un génome quelques exemplaires> 10 10, en fonction de la sensibilité du dosage. Amorces et les sondes de conception qui couvrent un petit fragment (<200 pb) d'une région très conservée du génome.
- Analyse par Northern blot. Bien que moins quantitative que qRT-PCR, cette technique permet une confirmation visuelle que les résultats de réplication dans génomes entiers et que nul de terminaison de chaîne significative survient à la suite de la mutation polymérase.
- Expression d'un gène rapporteur. Si un clone d'ADNc exprimant un gène rapporteur (luciférase, par exemple) est disponible, alors cela peut être utilisé comme substitut d'examiner la capacité réplicative. Cependant, le virus recombinant ne doit pas être utilisé pour d'autres applications (telles que la détermination de la fréquence de mutation), car les pressions sélectives agissant sur ce virus ne sera pas la même chose, en particulier depuis ces virus ont tendance à supprimer le gène rapporteur inséré.
6. Mesure fréquences de mutation
Ceci est une étape critique dans confirmant que la mutation identifiée polymérase conférant une résistance aux agents mutagènes réplication modifie la fidélité. Il est important de noter que les fréquences de mutation mesuré ici ne sont pas les taux de mutation. Pour déterminer les taux, une mesure très prudent de la cinétique de la réplication (quantité d'ARN synthétisée et la longueur du cycle de réplication) doivent être pris po Mesure fréquences de mutation Cependant, aussi longtemps que l'histoire le passage et la cinétique de réplication sont surveillés, fournit reproductibles, des mesures quantitatives de la réplication fidélité. Fréquences de mutation peuvent être déterminés soit dans la population des virus viables (clones plaque ou de dilution limite) ou dans la population totale du virus (stock de virus ou de surnageant). Afin de déterminer les fréquences de mutation, de préparer des stocks de virus à partir d'un autre passage (passage par exemple 2 ou au-delà). Il est important que la population virale a eu le temps d'élargir sa diversité génétique plus proche de l'équilibre mutation-sélection.
- Fréquences de mutation de la population des virus viables
Cette approche, bien plus laborieuse, donne des informations sur le nombre de mutations sont présents sur le génome moyenne qui conserve la compétence de la réplication. Il faut noter, cependant, qu'un biais pour des variantes de fitness supérieure et inférieure se fera de fitness, des variantes viables qui ne sont pas facilement la plaque, par exemple, peut ne pas être détecté. Comme tel, il permet également de meilleures mesures de synonymes (DS) et non synonymes (DN) des substitutions nucléotidiques qui peuvent être utilisées pour déterminer si la sélection positive agit sur la population. Cependant, parce que moins de mutations seront quantifiés, un plus grand nombre de séquences seront nécessaires pour l'analyse statistique. Cette technique repose sur l'isolement du virus individuels soit par purification sur plaque ou de dilution limite, comme décrit ci-dessus. Comme point de départ, nous recommandons l'isolement de 48 individuels des «clones» du virus de type sauvage et la variante mutagène résistant. Chaque population clonale isolées de cette manière est prévu de réaliser ce que la mutation du génome fondateur présentés. La quantité d'ARN présentes dans la plaque isolés ou en limitant la dilution est bien généralement suffisante pour l'amplification par RT-PCR. Si nécessaire, une amplification de courte durée (moins d'un cycle de réplication) sur un nombre minimum de cellules (par exemple 24 format de plaque bien) peut être utilisé pour obtenir plus d'ARN, mais l'amplification minimale doit être effectuée afin d'éviter l'accumulation de nouvelles mutations. Notez que chaque clone et la population à comparer doivent subir le même nombre de cycles de réplication.- Isoler de 24 à 48 clones de virus par purification sur plaque ou de dilution limite.
- Extrait d'ARN à partir des populations isolées clonale
- RT-PCR d'amplifier un fragment couvrant jusqu'à 3kb pour chaque échantillon. Il est préférable de couvrir la région de la protéine de structure, ce qui tend à tolérer des mutations plus viable que les régions les plus conservées des gènes non structuraux.
- Purifier la PCR produits, la séquence et effectuez une analyse de mutation (article 7).
- Fréquences de mutation du virus de la population totale
Bien que l'avantage de cette seconde méthode est que même des variantes de fitness faibles seront inclus dans le séquençage, permettant une vision plus large du spectre de mutations. Cependant, il peut ne pas être idéal pour des analyses phylogénétiques qui assument les populations des virus viables (par exemple, dN / dS valeurs) et l'identification des mutations les plus pertinentes, puisque les changements létale (modifié la structure des ARN, des codons stop, dramatiques changements d'acides aminés) ne peut pas être entièrement identifié et seraient retenues dans l'analyse. Néanmoins, il cette technique permet au chercheur d'obtenir les données les plus statistiquement significatif pour confirmer l'altération de la fidélité quand il ya une absence d'un test in vitro biochimiques. Cette technique repose sur la RT-PCR de l'ARN du virion total, y compris les génomes avec une faible condition physique ou de mutations létales qui ne seront pas des plages. La fréquence des mutations obtenues par cette méthode peut être 10 fois plus élevé que par plaque ou clonage par dilution limitante.- Extraire l'ARN de la population totale du virion
- RT-PCR amplify une région de 800 à 1200 nucléotides d'une partie de la séquence génomique codant qui est connu à tolérer les mutations et ont une variance génétique (par exemple des protéines de structure). Des fragments plus grands seront pas insérer facilement dans des vecteurs de clonage, comme TopoTA et produira un nombre insuffisant de transformants. Bien que plus petits fragments sont encore mieux, la couverture du génome séquencé peut être trop peu pour obtenir une signification statistique. Un fragment d'au moins 800 pb permet la couverture de séquence avec deux amorces et est un bon compromis entre maximiser la couverture séquence et en minimisant les coûts de séquençage. Nous constatons que entre 70 et 100 séquences couvrant une région 800 nucléotides confirme la fidélité reproductible modifié les variantes que nous avons étudiés dans le laboratoire. Notez que d'autres vecteurs / clonage méthodes peuvent être utilisées avec une efficacité égale.
- Purifier le produit de RT-PCR en utilisant un kit commercial, ou par extraction d'ADN standard / précipitation.
- Si les enzymes de RT-PCR que vous utilisez ne produisent pas de surplombs, effectuer une extension de 10 minutes en ajoutant 1 uM et ATP Taq polymérase
- Clone TopoTA suivant les instructions du fabricant
- Pour chaque population virale à étudier, sélectionner 96 colonies, identifiés comme ayant un insert positif en bleu / blanc de dépistage sur Xgal plaques revêtues. Initialement, tester la présence d'inserts pour chaque région du génome cloné étant différentes, afin de confirmer la validité de bleu / blanc de dépistage par le dépistage taille du plasmide sur gels d'agarose ou d'une seule colonie de PCR. Utilisation de la taille des fragments et des conditions ci-dessus, nous obtenons 90% positifs
- Cultivez chaque colonie dans un bouillon de liquides durant la nuit dans 1 ml de milieu LB à 96 plaques de culture bactérienne ainsi
- Le lendemain de préparer minipreps au format 96 puits.
- Séquence chaque assiette avec des amorces assez (les amorces utilisées pour la RT-PCR par exemple, ou TopoTA amorces M13) pour obtenir une couverture maximale du segment cloné. Effectuer une analyse de mutation (article 7).
7. L'analyse des séquences
Effectuer des analyses de séquence en utilisant une référence ou une séquence consensus pour chaque population et des logiciels d'alignement approprié. Nous recommandons Lasergene ou Sequencher qui peuvent facilement identifier les SNPs à l'égard de consensus.
- Aligner les séquences en utilisant un logiciel approprié (par exemple, ou Lasergene Sequencher).
- Jeter des séquences de mauvaise qualité (en appelant de base mauvaise, trop de 'N ou trop court en longueur). Identifier la gamme de nucléotides qui est couvert par toutes les séquences. Depuis les différentes régions du génome sera plus ou moins de tolérer des mutations, pour des raisons de comparaison, il est essentiel que la même région est entièrement couverte pour chaque clone séquencé retenus pour analyse. Ainsi, si les réactions de plusieurs séquences sont réalisées par clone, et une séquence échoue pour un clone donné, jeter le clone (toutes les séquences) de l'analyse.
- Identifier et compter les SNP qui sont différentes de la souche de référence.
- Calculer la fréquence des mutations en divisant le nombre total de SNP identifiés par le nombre total de nucléotides séquencés (nombre de clones x longueur de la région séquencée). Présentant ce nombre que le nombre moyen de mutations par séquençage 10K nt rend le nombre plus convivial, puis le quitter que par nucléotide. Par exemple, pour la population de type sauvage dans le tableau 1, 55 mutations/121, 978 nucléotides totale X 10 000 = 4,51 pour 10.000 mutations nucléotides séquencés.
- Si les mêmes SNP apparaît sur un grand nombre de clones, présente deux valeurs qui incluent ou excluent ces mutations répétées. Généralement, pour une population de virus produit à partir d'un parent homogène (par purification sur plaque ou d'un clone infectieux) et passages seulement quelques fois dans la culture cellulaire, la sélection positive n'a pas exercé ses effets assez pour causer l'accumulation de la même SNP et la mutation valeurs de la fréquence mesurée de cette façon de mieux refléter la fréquence d'erreur de la polymérase avec des effets minimes de la sélection positive ou l'épuration.
- Déterminer la répartition de mutation. Faire une liste de classement du nombre de clones dans chaque population qui présentent 0, 1, 2, 3, etc .. mutations dans la région séquencée.
- Calculer la diversité des populations virales par comparaison à distance par paire. De la nettoyer et préparer les séquences modifiées manuellement un alignement, y compris la région de référence. Il ya plusieurs programmes d'alignement disponibles: ClustalW / X ( http://www.clustal.org/ ), le muscle ( http://www.drive5.com/muscle/ ), EbioX pour les utilisateurs Mac ( http://www. ebioinformatics.org / ebiox / ), etc Assurez-vous que toutes vos séquences sont de même longueur, de cultures, si nécessaire. Il est recommandé que le début de la séquence est un codon codant pour faciliter l'analyse postérieure. Nous vous suggérons de garder tous les alignements au format fasta, qui est facilement lu par la plupart des logiciels disponibles.
- Pour effectuer l'analyse à distance par paires, calculer toutes les comparaisons possibles entre les paires de séquences appartenant à la même population. Moyenne des mutations trouvées dans les comparaisons peuvent alors être calculée pour indiquer l'hétérogénéité de la population.
- Les valeurs des synonymes (DS) et non synonymes (DN) au sein de la population peuvent être facilement obtenues. Nous trouvons le logiciel MEGA (http://www.megasoftware.net/) utile et convivial pour ce genre d'analyse.
- Pour obtenir des directives de sélection nous donne deux possibilités même si elles ne sont pas les seules possibles. 1) Obtenir le rapport entre dN et dS. Des valeurs supérieures à 1 signifie une sélection positive. Des valeurs inférieures à 1 signifie la sélection purifiante. 2) Utiliser le serveur web Datamonkey ( http://www.datamonkey.org/ ). Téléchargez vos alignements au format fasta et les analyser avec le module SLAC. Cela vous donnera une estimation de dN / dS.
- Effectuer des analyses statistiques. Selon la quantité de données et le nombre de séquences, une variété de tests peuvent être utilisés. Certaines études se sont appuyés sur les tests du chi carré du nombre total de mutations par rapport au nombre total de nucléotides consensus où tous les clones sont combinés 9. D'autres études ont effectué des tests de Fisher, calculée sur le nombre de séquences présentant des mutations par rapport au nombre de séquences sans mutations 10. Si un nombre suffisant de données mutationnel est généré, nous recommandons d'effectuer un test de la somme classé comme Mann Whitney U. Pour ce faire, le rang le nombre de clones dans chaque population de virus par le nombre de mutations présentes sur chaque séquencé l'ARN. Le Whitney Mann sera ensuite tester les différences dans la distribution des populations d'une mutation. Pour cette raison, nous recommandons séquençage d'au moins 800 paires de base, pour augmenter la probabilité de trouver des clones avec des mutations multiples. Ce test est robuste, mais nécessite des échantillons plus importants. D'autre part, il ne nécessite pas la même taille d'échantillon pour les deux populations comparées (par exemple, les échantillons du tableau 1 sont n 1 = 148 et n 2 = 84).
8. Les résultats représentatifs:
L'effet dose-dépendant de la concentration mutagène sur la viabilité cellulaire et la viabilité du virus est montré dans la figure 1. Dans cet exemple, nous avons constaté que le passage du virus dans 100 AZC uM a été réduite du titre du virus par la cible de 10,5 à 2 log, mais la viabilité des cellules HeLa pas négativement impacté au cours des deux jours nécessaires à l'infection virale. Cette expérience pilote a conduit au choix d'une concentration de 100 pM pour AZC passages successifs de virus, de sélectionner pour la résistance mutagène. La figure 2 illustre la réduction initiale du titre, suivie par l'émergence d'un phénotype de résistance mutagène. Pendant les premiers passages peu mutagène, comme les mutations létales d'accumuler, une baisse significative des titres de virus se produit. Progressivement, une variante mutagène résistant émerge une émergence coïncide avec son retour à titre de virus ne diffèrent pas de témoins non traités. A ce stade, un grand pourcentage de la population virale présente la mutation de résistance. Le séquençage du virus de cette population révèle le changement d'acide aminé (s) responsable. Une fois identifié et isolé ou nouvellement générés, le virus mutagène résistant peut être moins sensible que de type sauvage à différents ARN mutagènes (analogues de base de la structure différente, par exemple). La figure 3 montre un ARN mutagène résistants virus Coxsackie B3 que les titres plus élevés que de type sauvage en présence de ribavirine, le 5-fluorouracile, et la 5-azacytidine, et de haute MgCl2 et MnCl2. Vaste résistance aux agents mutagènes ARN est un indicateur fort de la fidélité de la réplication accrue. Vérifier que la cinétique de réplication de la variante de la fidélité est similaire au virus de type sauvage aidera à la comparaison des fréquences de mutation. La figure 4 montre la cinétique de croissance, étape d'une variante de haute fidélité par rapport au type sauvage. Si les taux de réplication et de titres définitifs ne sont pas similaires, alors des mesures doivent être prises pour comparer les populations du virus de taille similaire, qui ont subi le même nombre de tours de la réplication. Le lien entre le taux de synthèse de l'ARN et de fidélité de la réplication n'est pas bien caractérisée, en particulier in vivo. Un taux plus lent de réplication peut entraîner une diminution de la fréquence de mutation (haute fidélité), mais ce n'est pas une règle absolue, comme le montre la figure 4. Avec les paramètres ci-dessus établie, la fréquence des mutations de la variante de la fidélité et les populations de type sauvage peut être comparé à obtenir la confirmation génétique de fidélité de la réplication modifiée. La figure 5 montre un alignement de séquences de type sauvage et haute variante de la fidélité, à des mutations ponctuelles identifiées. Les mutations sont comptés, classés selon le nombre de mutations par clone (tableau 1), et présentée comme une fréquence de mutation moyenne par la population, pour 10.000 nucléotides séquencés, la figure 6.

Figure 1. Déterminer les conditions optimales pour sélectionner des ARN mutagène résistance:. Rétention de la viabilité cellulaire élevée avec une moyenne des cellules HeLa (chute de log 1-2 dans le titre viral) ont été traitées avec des concentrations indiquées de la ribavirine et infectés par le virus Coxsackie B3 de type sauvage du virus à une MOI de 0,01. 48 heures après l'infection, le virus a été récolté et la descendance des titres ont été déterminées par DICT 50. Le pourcentage de cellules survivantes de traitement à 48 heures, déterminé par coloration au bleu trypan, est indiqué ci-dessous l'axe des x. Les résultats montrent que les concentrations de 100 et 200 uM de réduire les titres de virus par le journal de 1-2, sans affecter la viabilité cellulaire.
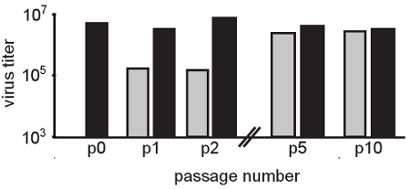
Figure 2. Passage en série, en présence de concentrations modérées de l'ARN mutagènes sélectionne pour les populations résistantes mutagène. Dans cette figure, le virus Chikungunya a été repiquées dans les cellules HeLa en présence de 50 uM (barres grises) ribavirine. Passages de contrôle ont été effectuées en l'absence de la ribavirine (barres noires). Après chaque passage, la progéniture du virus a été quantifiée par dosage de plaque classique sur des cellules BHK. L'effet mutagène est évident au cours des premiers passages (P1 et P2 par rapport à la population de départ p0) où le virus traitée goutte titres par 2 log. Progressivement, les titres reviennent à la normale (non traitée) niveaux. Aucune différence significative n'est observée dans les populations passage mutagène 5 traités par rapport aux traités, suggérant que des variants résistants ont été sélectionnés. En effet, le séquençage de consensus de la population identifié des mutations uniques dans la population virale sous traitement ribavirine.
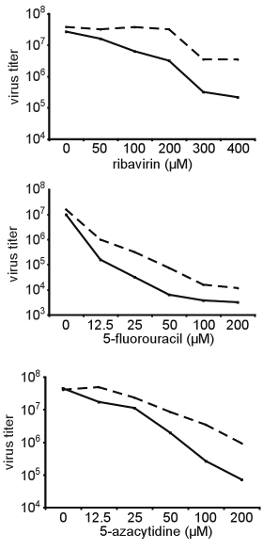
Figure 3. Confirmation de la résistance large à l'ARN mutagènes de structure différente. Montré ici, la haute fidélité A372V variante du virus Coxsackie B3 qui a été initialement isolé dans l'écran décrit à la section 3 a été généré à partir d'un clone infectieux et testé pour sa sensibilité relative à différentes concentrations de différents ARN mutagènes (ribavirine, le 5-fluorouracile, 5-azacytidine). Des cellules HeLa ont été traitées avec des concentrations indiquées de la ribavirine et infectés par le virus Coxsackie B3 de type sauvage du virus à une MOI de 0,01. 48 heures après l'infection, le virus a été récolté et la descendance des titres ont été déterminées par DICT 50. On voit ici les titres de type sauvage (lignes continues) et A372V variante (lignes en pointillés) en fonction de la concentration mutagène. A372V constamment plus élevés que les titres de type sauvage dans toutes les conditions testées.
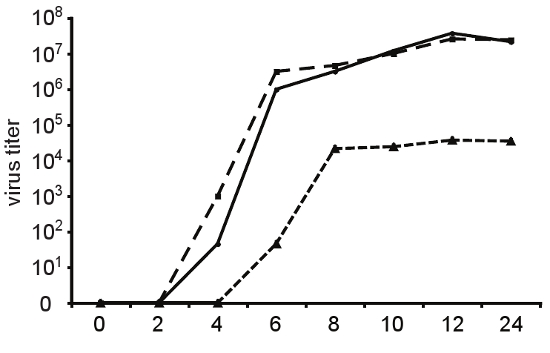
Figure 4. Taux de réplication et de variantes fidélité. Afin de déterminer la cinétique de croissance en une étape de la production de virus, les cellules HeLa ont été infectées à une MOI = 10 avec soit de type sauvage (ligne continue), de haute fidélité variante A372V (tirets longs) ou d'une variante de réplication déficiente Cx64 (court tirets) du virus Coxsackie B3. Aux points de temps indiqué, la progéniture du virus a été récolté à partir des cellules et les surnageants par gel-dégel et titrés par DICT 50. L'augmentation de la fidélité de A372V ne coïncide pas avec un défaut de réplication observables dans une culture tissulaire. La Cx64 variante présente un retard important dans la cinétique de la réplication et atteint au maximum des titres qui sont mille fois plus faible que le virus de type sauvage.
Figure 5. L'alignement des séquences clonées TopoTA de chaque population de virus. Utilisant l'approche décrite à la section 7, chaque séquence clonée provenant de RT-PCR produit provient vraisemblablement d'un seul génome unique au sein de la population totale du virus et serait donc, porteurs de mutations uniques. La figure montre un alignement typique, après le nettoyage des séquences de mauvaise qualité et la visualisation des SNP. Le SNP total (10 dans ce chiffre) dans une population sont comptés, et le nombre de SNP apparaissant sur chaque clone sont notées. Par exemple, le clone soulignée par une barre, contient deux mutations uniques tandis que 8 autres clones contiennent une seule, unique mutation. Ces données sont utilisées pour compiler le tableau 1. Pour voir une version agrandie de cette figure s'il vous plaît cliquez ici .
Figure 6. Représentation graphique des fréquences de mutation du virus de la population. Pour faciliter l'interprétation, les données numériques obtenus à partir de la séquence et les analyses statistiques peuvent être représentées soit par un graphique, ou l'histogramme (illustré ici). Le virus A372V génère moins de mutations que le type sauvage et présente une fréquence de mutation significativement plus bas (*, p <0,01). La variante Cx64, qui se réplique à des titres de 1000 fois plus faible que celui du type sauvage, presents de la fréquence même mutation (ns, non significatif) indique que la vitesse de réplication et de la fidélité ne sont pas nécessairement liés. Le même virus Chikungunya (CHIKV) de la population donne des fréquences mutation similaire si le stock de virus, ou une dilution de 10 5 fois, est utilisé pour l'extraction de l'ARN.
Résumé de distribution de mutation pour l'analyse statistique.
Remarque: Pour chaque clone, il est essentiel que la région génomique mêmes (et la longueur de la séquence) est couvert. Dans ce cas, 859 nucléotides par clone. Cela est essentiel pour les analyses statistiques. D'autre part, les tests de la somme des rangs utilisés pour l'analyse statistique ne nécessitent pas la taille des échantillons soit la même, le chercheur est libre de comparer la population des différentes taille de l'échantillon. Ainsi, les 142 clones de type sauvage peut être comparé aux 84 clones de A372V.
| # Clones avec des mutations n | de type sauvage | A372V |
| 7 mutations | 0 | 0 |
| 6 mutations | 0 | 0 |
| 5 mutations | 0 | 0 |
| 4 mutations | 0 | 0 |
| 3 mutations | 1 | 0 |
| 2 mutations | 6 | 2 |
| Une mutation | 40 | 14 |
| 0 mutations | 95 | 68 |
| Total des mutations | 55 | 18 |
| Total des clones séquencés | 142 | 84 |
| Nucléotides totale séquencé | 121978 | 72156 |
| Mutations/10 4 NT | 4,51 | 2,49 |
Tableau 1. . Synthèse de distribution de mutation pour l'analyse statistique Note: Pour chaque clone, il est essentiel que la même région génomique (et la longueur de la séquence) est couvert. Dans ce cas, 859 nucléotides par clone. Cela est essentiel pour les analyses statistiques. D'autre part, les tests de la somme des rangs utilisés pour l'analyse statistique ne nécessitent pas la taille des échantillons soit la même, le chercheur est libre de comparer la population des différentes taille de l'échantillon. Ainsi, les 142 clones de type sauvage peut être comparé aux 84 clones de A372V.
Discussion
Choix de la lignée cellulaire. L'efficacité des analogues de base que l'ARN mutagènes en corrélation avec leur absorption relative par différents types de cellules 11. Si la lignée cellulaire qui est normalement utilisé pour le passage du virus s'avère être réfractaire à l'absorption mutagène ou trop sensibles (toxicité cellulaire élevée), il peut être nécessaire d'utiliser une autre lignée cellulaire qui répond à ces exigences et est toujours permissives à la réplication virale. Une fois que la variante la résistance mutagène est isolé, le reste de la caractérisation peut être réalisée dans l'original, lignée cellulaire de choix. Dans notre expérience, les cellules HeLa facilement prendre jusqu'à mutagène; cellules BHK nécessitent jusqu'à 10 fois plus élevé et les concentrations de cellules Vero sont réfractaires à l'absorption mutagène.
Choix du mutagène. En essayant d'isoler variantes fidélité par traitement mutagène, la probabilité de succès augmente si plus d'un type d'agent mutagène est utilisé. Base de mutagènes analogique de structure différente, qui sont incorporés dans les génomes tort lors de la réplication sera principalement induisent entraîner un sous-ensemble spécifique de mutations dans les cycles de réplication ultérieure: traitement ribavirine favorise GTOA de transition et de mutations CTOU 12; 5-azacytidine a un biais similaire, avec le Outre des CtoG et transversions GTOC 13; 5-fluorouracile préférentiellement induit des transitions et des ATOG UtoC 14. Alternativement, des concentrations plus élevées de Mg 2 + ou Mn 2 + peut être complétée au milieu pour augmenter la fréquence de mutation globale des virus à ARN, sans le biais décrits ci-dessus 12. Selon le codon séquences du virus, et les changements de codons nécessaires pour générer une variante fidélité, certaines de ces conditions seront favorables à l'émergence de cette variante par rapport aux autres. Pour plus de fidélité le poliovirus et le virus Coxsackie G64S A372V, ribavirine plus facilement sélectionnés pour les variantes, car la transition ATOG requis sur le site codon correspondait à la prédominance des mutations générées par cette ribavirine.
La taille de la population vs MOI. En virologie, les protocoles d'infection culture de tissus accorder une attention particulière à la multiplicité d'infection (MOI), pour éviter l'accumulation de particules défectives interférentes (faible MOI) ou pour favoriser la recombinaison entre les virus (MOI élevé), pour par exemple. Pour sélectionner des événements de l'émergence au cours de série repiquage, il est également important de considérer la taille des populations de virus. Depuis le mutant résistant existe d'abord à basse fréquence, il est préférable de transférer une aussi grande taille de la population que possible d'un passage à la suivante (10 5 -10 6 virus, par exemple) pour éviter de perdre ces variantes émergentes à chaque passage. Amplifier l'importance de bien ou flacon (nombre de cellules infectées) peut aider à minimiser l'augmentation de la MOI, si ce n'est de préoccupation. D'autre part, pour des expériences dans lesquelles la sensibilité d'un virus à mutagène est actuellement testé, faible d'infection MOI est effectuée afin d'augmenter le nombre de cycles de réplication se produisent dans l'expérience et d'éviter le sauvetage des génomes mutagenèse par les génomes de fitness supérieur à travers complémentation en collaboration cellules infectées. Ceci est important car les mutations générées sur les génomes descendance durant le premier tour de la réplication ne sera pas immédiatement détecté. La plupart de ces ARNs mutagénisées sera toujours emballés dans des virions. C'est dans le prochain cycle d'infection qui mutations létales présents dans ces génomes se traduira par un cycle de réplication avorté, et la réduction du titre du virus. Il peut être nécessaire pour permettre plusieurs cycles d'accumulation de mutations avant un effet significatif de la mutagenèse létale est observée. Enfin, si le passage sur la série, en présence du mutagène, les titres de virus continuent à baisser jusqu'à l'extinction, alors le chercheur devrait essayer de repiquage du virus dans progressivement des quantités croissantes d'agent mutagène (à partir d'une concentration très faible).
L'isolement et la génération du clone résistant à mutagène ARN à partir de l'ARN mutagène résistant population. Mutagènes ARN d'introduire de multiples mutations aléatoires à chaque génome, mais sélection pour la résistance ne enrichir (et fixer à la séquence consensus) la mutation de résistance. Pour identifier cette mutation, nous avons la séquence de la population résistante mutagène (consensus de la population) et non des virus individuels. Par conséquent, la seule, des mutations aléatoires créés par le mutagène ne sont pas détectés dans la séquence; que les mutations qui entraînent des changements consensus suivant la sélection, sont trouvés. Dans notre expérience, nous avons seulement identifier un ou deux changements tels séquence consensus. Une fois que la population résistante mutagène est obtenue et la mutation de résistance est identifié, il est nécessaire de générer un stock plus pure de cette variante. Ci-dessus, nous avons décrit une procédure de purification sur plaque. Alternativement, si le virus d'intérêt ne produit pas de plaques facilement identifiables, la variante souhaitée peut être PurifIED en limitant la dilution. Cette approche est essentiellement un 50 DICT au format 96 puits, où le stock de virus est dilué de telle sorte que moins de 50% des puits sont contaminés. En utilisant cette dilution, la même approche que ci-dessus est prise, dans l'isolement jusqu'à 10 variantes individuelles et confirmant leurs séquences. Comme mentionné précédemment, dans le meilleur des cas, un clone d'ADNc infectieux de la souche du virus est disponible. Isolement de la variante ne serait donc pas nécessaire. Dans notre expérience, la fidélité variantes sont le résultat de simples substitutions d'acides aminés et peuvent ainsi être générés en utilisant de simples kits commercialisés tels que la mutagenèse QuikChange (Agilent). Une option secondaire est d'utiliser un clone d'ADNc d'une souche étroitement liés. Cependant, si une souche apparentée est utilisé, nous recommandons fortement d'utiliser à la fois cette approche et l'isolement du virus (purification sur plaque, par exemple) parce que nous avons constaté que la même mutation altérant la fidélité de deux virus étroitement apparentés ne sera pas nécessairement le même effet.
Fidelity et de réplication. Sélection de variants résistants mutagène ARN ont abouti à l'isolement des deux variantes de fidélité supérieures et inférieures avec des caractéristiques de croissance qui sont semblables à leurs homologues de type sauvage 4,12,15. Actuellement, le lien entre les taux d'activité de la polymérase et la fidélité n'est pas entièrement comprise. In vitro, des études biochimiques utilisant l'ARN polymérase purifiée ont montré que plus la fidélité variantes ont des taux de traitement plus lent, tandis que la baisse des variantes de fidélité ont tendance à avoir un traitement plus rapide 1-3,12. En culture de tissu, ces différences ne sont généralement pas évidente, ce qui suggère que la disponibilité des ressources, plutôt que de la cinétique intrinsèque de l'activité polymérase, est l'étape limitante. Si la variante la fidélité réplique avec une cinétique qui ne sont pas significativement différents de type sauvage, puis une comparaison de leurs fréquences de mutation peut être faite directement. Si un changement très important dans la cinétique de réplication existe, alors les données doivent être normalisées pour tenir compte des différences cinétiques, par exemple en comparant les virus qui ont subi le même nombre de cycles de réplication. Dans notre expérience, même si aucune différence significative dans une seule étape cinétique de croissance ont été observées entre le type sauvage et de haute fidélité de variantes, nous avons observé que plus la fidélité constamment variantes titre plus élevé (moins de 1 log) par rapport au type sauvage, mais ils font un peu moins d'ARN (dans les du même ordre de grandeur), plus loin en suggérant que les génomes contiennent elles produisent moins de mutations et sont donc plus infectieux.
La préparation des échantillons et le séquençage. Pour toutes les étapes de ces protocoles, il est impératif que haute-fidélité, la relecture des enzymes sont utilisées pour la PCR et RT-PCR afin de limiter l'introduction de mutations supplémentaires, car ils ne peuvent pas être distingués des mutations biologiquement pertinentes. Il est essentiel que les populations de virus à comparer ont été préparés dans les mêmes conditions (historique des passages, milieu de culture tissulaire, la température, la méthode d'extraction d'ARN, RT-PCR des protocoles, etc) Il est également important de s'assurer que suffisamment de matériel de départ a été obtenu à partir de l'extraction d'ARN tel que une forte bande est généré par RT-PCR. Une dilution 1 / 100 de l'échantillon d'ARN devrait également donner une détectable par RT-PCR bande, ce qui indique que l'échantillon contient un nombre suffisant de molécules d'ARN pour éviter les biais de représentation (amplification du génome mêmes à plusieurs reprises). Depuis la fréquence de mutation est une distribution, on pourrait s'attendre à ce que des valeurs similaires seront obtenus indépendamment de la taille de la population, à condition que le biais susmentionnés ne se produit pas. Comme la figure 6 montre, à 10 5 fois la dilution d'un stock de virus donne une fréquence de mutation qui n'est pas significativement différente de la souche parentale.
Jusque dans les conditions optimales pour le clonage TopoTA sont trouvés, de confirmer la présence d'inserts après criblage bleu / blanc, par PCR sur colonie avant le séquençage. Comme un contrôle de bruit mutationnelle (mutations introduites par RT-PCR et séquençage), clone d'un produit de PCR à partir d'un plasmide portant les mêmes séquences virales et / ou le clone et la séquence de RT-PCR des produits ARN transcrit in vitro correspondant au génome du virus (soit conscients que différents dans les enzymes de transcription in vitro ont un taux d'erreur différent et ne peuvent donner des informations utiles à l'erreur de fond réel dans votre procédure). Certaines séquences de virus peuvent être toxiques pour les bactéries, il est donc important de vérifier cela avant de décider sur la région du génome viral à être séquencé pour la fréquence de mutation. En analysant les séquences obtenues par TopoTA, notez que chaque clone doit contenir qu'une seule insertion / séquence. Si un double pic est observé, suggérant une population mixte, il est possible que les deux voisins colonies bactériennes ont été sélectionnés. Il est également possible, bien que hautement improbable étant donné les fréquences de mutation faible réplication bactérienne, que la mutation a été introduite lors de l'amplification du plasmide dans la culture bactérienne. Dans la plaque purified populations, un double pic peut représenter des plaques qui se chevauchent, ou un virus qui est l'acquisition d'une nouvelle mutation ou de revenir d'une mutation au cours du développement de plaque. Soyez cohérent et décider sur l'opportunité de compter ou ne comptent pas ces mutations.
Enfin, gardez à l'esprit que les fréquences de mutation utilisés ici sont des valeurs relatives. Ils ne sont valables que dans la comparaison des populations de virus cultivés dans les mêmes conditions, et séquencé au cours de la même région! Ils ne devraient pas être prises comme des valeurs absolues des taux de mutation, ou la fréquence des mutations du génome dans son ensemble. Toutefois, lorsque les conditions sont contrôlées, elles ne permettent reproductibles, des comparaisons quantitatives des différences dans la répartition et la fréquence de mutation.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par un financement de la recherche médicale et sanitaire subvention de la Ville de Paris, les Français nationaux ANR-09-JCJC-0118-1, et l'ERC Starting Grant RNAvirusPopDivNVax Projet aucune. 242719.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|
| ribavirine | Sigma | R9644-10MG | |
| 5-fluorouracile | Sigma | F6627-1G | |
| 5-azacytidine | Sigma | A2385-100MG | |
| MgCl 2 | Sigma | M1028-100ML | |
| MnCl2 | Sigma | M1787 | |
| Le bleu trypan | Sigma | T8154-20ML | |
| TopoTA kit de clonage | Invitrogen | 10351021 | |
| QuikChange mutagenèse kit | Agilent | 200516 | Si un clone d'ADNc infectieux est disponible |
| 96-ainsi Miniprep Kit | Macherey-Nagel | 740625 | |
| Lasergene, Sequencher | DNAstar, Gene Codes Corporation | www.dnastar.com www.genecodes.com | Ou d'autres logiciels d'alignement |
Références
- Arias, A. Determinants of RNA-dependent RNA polymerase (in)fidelity revealed by kinetic analysis of the polymerase encoded by a foot-and-mouth disease virus mutant with reduced sensitivity to ribavirin. J Virol. 82, 12346-12355 (2008).
- Arnold, J. J., Vignuzzi, M., Stone, J. K., Andino, R., Cameron, C. E. Remote site control of an active site fidelity checkpoint in a viral RNA-dependent RNA polymerase. J Biol Chem. 280, 25706-25716 (2005).
- Korneeva, V. S., Cameron, C. E. Structure-function relationships of the viral RNA-dependent RNA polymerase: fidelity, replication speed, and initiation mechanism determined by a residue in the ribose-binding pocket. J Biol Chem. 282, 16135-16145 (2007).
- Pfeiffer, J. K., Kirkegaard, K. A single mutation in poliovirus RNA-dependent RNA polymerase confers resistance to mutagenic nucleotide analogs via increased fidelity. Proc Natl Acad Sci U S A. 100, 7289-7294 (2003).
- Pfeiffer, J. K., Kirkegaard, K. Increased fidelity reduces poliovirus fitness and virulence under selective pressure in mice. PLoS Pathog. 1, e11-e11 (2005).
- Vignuzzi, M., Stone, J. K., Arnold, J. J., Cameron, C. E., Andino, R. Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population. Nature. 439, 344-348 (2006).
- Vignuzzi, M., Wendt, E., Andino, R. Engineering attenuated virus vaccines by controlling replication fidelity. Nat Med. 14, 154-161 (2008).
- Crotty, S., Cameron, C., Andino, R. Ribavirin's antiviral mechanism of action: lethal mutagenesis. J Mol Med. 80, 86-95 (2002).
- Coffey, L. L., Vignuzzi, M. Host alternation of chikungunya virus increases fitness while restricting population diversity and adaptability to novel selective pressures. J Virol. 85, 1025-1035 (2011).
- Ciota, A. T. Role of the mutant spectrum in adaptation and replication of West Nile virus. J Gen Virol. 88, 865-874 (2007).
- Ibarra, K. D., Pfeiffer, J. K. Reduced ribavirin antiviral efficacy via nucleoside transporter-mediated drug resistance. J Virol. 83, 4538-4547 (2009).
- Levi, L. I. Fidelity variants of RNA dependent RNA polymerases uncover an indirect, mutagenic activity of amiloride compounds. PLoS Pathog. 6, e1001163-e1001163 (2010).
- Sierra, S., Dávila, M., Lowenstein, P. R., Domingo, E. Response of foot-and-mouth disease virus to increased mutagenesis: influence of viral load and fitness in loss of infectivity. J Virol. 74, 8316-8323 (2000).
- Ruiz-Jarabo, C. M., Ly, C., Domingo, E., de la Torre, J. C. Lethal mutagenesis of the prototypic arenavirus lymphocytic choriomeningitis virus (LCMV). Virology. 308, 37-47 (2003).
- Sierra, M. Foot-and-mouth disease virus mutant with decreased sensitivity to ribavirin: implications for error catastrophe. J Virol. 81, 2012-2024 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon