Method Article
Isolamento de variantes de vírus de RNA Fidelity e Caracterização de frequência de mutação de vírus
Neste Artigo
Resumo
O presente artigo descreve os passos necessários para isolar e caracterizar variantes RNA polimerase fidelidade de vírus RNA e como usar dados de freqüência de mutação para confirmar as alterações fidelidade em cultura de tecidos.
Resumo
Vírus RNA polimerase RNA dependente utilizar RNA para replicar os seus genomas. A taxa de erro intrinsecamente alta destas enzimas é um grande contribuinte para a geração de diversidade populacional extrema que facilita a adaptação e evolução do vírus. Crescente evidência mostra que as taxas de erro intrínseco, e as freqüências de mutação resultante, de vírus de RNA pode ser modulada por sutis mudanças de aminoácidos para a polimerase viral. Embora ensaios bioquímicos existem por algum polimerases RNA viral que permitem a medida quantitativa da incorporação fidelidade, aqui nós descrevemos um método simples de medir freqüências de mutação de vírus de RNA, que provou ser tão precisos como abordagens bioquímicas na identificação de mutações alterando a fidelidade. A abordagem utiliza técnicas convencionais de virológica e seqüenciamento que podem ser executadas na maioria dos laboratórios de biologia. Com base em nossa experiência com uma série de vírus diferentes, identificamos os principais passos que devem ser otimizado para aumentar a probabilidade de isolar variantes fidelidade e gerar dados de significância estatística. O isolamento e caracterização de mutações alterando a fidelidade pode fornecer novos insights sobre a estrutura e função da polimerase 1-3. Além disso, essas variantes fidelidade podem ser ferramentas úteis na caracterização de mecanismos de adaptação do vírus ea evolução 4-7.
Protocolo
1. Determinar a gama de concentrações mutagênico que é minimamente tóxico para as células
O objetivo deste exercício é determinar o que gama de concentrações mutagênico pode ser usada durante uma infecção sem toxicidade celular em excesso. Basicamente, você vai querer reproduzir as condições que serão necessárias para a infecção pelo vírus. Para a maioria dos vírus, infecções duram entre 2 e 7 dias. Prepare pratos suficiente para as células da amostra em cada dia. Se as células não aderentes são usados, modificar o protocolo de acordo.
- No dia antes do experimento, sementes de 7 x 10 5 células HeLa / bem em uma placa de 6 poços que alcançam uma sub-monocamada confluente (75%) do dia do experimento. Cada poço da placa vai ser tratada com uma concentração diferente de mutagênico, permitindo uma série de seis concentrações.
- No dia do experimento, preparar diluições mutagênico em meio de cultura de tecidos. Para as células HeLa, use um intervalo de 0 a 1000 M para análogos de base (ribavirina, 5-fluorouracil, 5 azacitidina), de 0 a 50 mM para MgCl 2 e 0-5 mM para MnCl 2.
- Aspirar o meio dos poços e substituir com 2 ml de meio mutagéneo suplementada e retornar à incubadora.
- A cada 24 horas, use uma placa de células para verificar a viabilidade celular. Isso pode ser feito através da realização de coloração azul de tripano ou usando comercializados fluorescência / luminescência ensaios (CellTiter-Glo, por exemplo Promega ® da Assay Viabilidade Luminescent Cell).
- Para trypan coloração azul exclusão, retire as células de uma placa (tratados com diferentes concentrações de mutagênico) e células suavemente pellet por centrifugação.
- Descartar sobrenadante e ressuspender as células em PBS (soro pode interferir com a coloração).
- Misture 1 volume de suspensão celular em PBS com 1 volume de 0,4% azul de tripano, incubar por 2 minutos em temperatura ambiente.
- Em um hemocitômetro, contar o viáveis (sem manchas) e não viáveis (azul manchado) células. Calcular a porcentagem de células viáveis para cada concentração do mutagéneo, incluindo o controle sem tratamento. Nós achamos que as condições que resultam em menos de 50% a morte das células pelo ponto de extremidade infecção (quando os títulos máximos são alcançados) são ideais para o isolamento de resistência mutagênico.
2. Determinar a concentração ideal mutagênico não tóxico que moderadamente reduz títulos de vírus (aproximadamente 0,5-2 redução de log)
Este exercício serve para determinar a concentração de agentes mutagénicos que irá exercer uma forte pressão seletiva, sem excesso de mutagenizing da população. Para RNA mutagénicos, vemos que isso corresponde a 10,5-2 logs reduções no título de vírus. Nessas concentrações, cada genoma é mutante em pelo menos uma ou duas posições. O agente mutagênico pode ajudar na geração de mutação de resistência, que depois serão selecionados mais de passagem. Se as mutações demais são introduzidos (em concentrações muito elevadas mutagênico), os mutantes levando a mutação de resistência serão eles próprios letalmente mutagenizados, impedindo o seu isolamento.
- Placas de semente com células usando as mesmas condições que para o passo 1
- No dia do experimento, preparar diluições mutagênico em meio de cultura de tecidos. Use a mesma gama de concentrações, conforme determinado acima, mas exclui as concentrações que resultou em mais de 50% a morte das células. Prepare o suficiente para cobrir média cada poço duas vezes (4 ml por poço), permitindo um pré-tratamento das células antes da infecção, com cada concentração do mutagéneo.
- Aspirar as células médio e pré-tratamento com agente mutagênico, incubando em mutagéneo suplementada médio por 2 horas. Para a maioria dos tipos de células, isso é tempo suficiente para a absorção do mutagéneo.
- Remova o meio e infectar com o vírus em multiplicidade de infecção baixa MOI (0,1 ou 0,01) em um volume mínimo (200 mL por 6 bem-chapas). Incubar 15-60 minutos para permitir que vírus para infectar as células. Rocha a placa em intervalos regulares para assegurar que o inóculo cobre a monocamada de células.
- Aspirar o vírus inoculado e lavar duas vezes com 2 ml de PBS para remover o máximo do inóculo possível.
- Adicionar médio com as concentrações adequadas de mutagênico para células de cada poço e incubar para o equivalente a 3-6 ciclos de replicação.
- Vírus da colheita de cada bem e determinar o efeito antiviral em título vírus. Isto pode ser feito através do teste de placa padrão ou limitação de diluição (TCID 50). Nota: Quantificação do vírus por técnicas que medem a síntese de RNA só pode não ser adequado, porque os efeitos mutagênicos podem não ser detectados. Genomas mutagenizados contendo mutações letais ainda pode ser detectado por qRT-PCR, por exemplo, mas não seria observada em ensaios de viabilidade vírus.
- De os títulos calculados, identificar a concentração de agente mutagénico que reduz os títulos de vírus (em comparação com o controle de infecção não tratada) por 10,5-2 registros que também não é altamente tóxico para as células (idealmente, a toxicidade inferior a 50%).
3. Isolamento e identificaçãoficação de variantes resistentes mutagênico
Executar passagens tamanho grande população na concentração mutagênico ideal definido acima e confira os títulos de vírus em toda a série passagem. Como controle, o vírus passagem em meio de crescimento, sem qualquer mutagênico. Como um outro controle para monitorar o surgimento potencial de interferir com defeito partículas (DI), execute infecções fresco na ausência de mutagênico em cada etapa passagem (controle unpassaged).
- No dia anterior semente infecção, cm 25 2 frascos com 1,5 x 10 6 células HeLa (tamanhos frasco outro pode ser usado) para obter sub-monocamadas confluentes no dia seguinte.
- No dia da infecção, as células pré-tratamento por 2 horas com meio contendo a concentração ideal de cada mutagênico, determinado no ponto 2.
- Remover médio e infectar as células com um volume mínimo em um MOI de 1 ou o maior MOI que não resulte em defeito formação de partículas interferentes (DI) para o vírus em estudo.
- Após 30-60 minutos de infecção, aspirado de inóculo e lavar duas vezes com PBS, em seguida, adicione meio fresco suplementado com mutagênico nas concentrações adequadas.
- Incubar durante o período de tempo determinado nos pontos 1 e 2, que corresponde ao título máximo do vírus nessas condições. Recolher o vírus progênie.
- Título de vírus em cada passagem e repita o 3 etapas anteriores.
- Durante o primeiro poucas passagens, os títulos de vírus mutagênico de amostras tratadas deve cair em conformidade, em comparação com o título de vírus original eo controle (sem tratamento e unpassaged) títulos de vírus. Se os títulos de vírus mutagênico de amostras de várias passagens subir de volta para os mesmos níveis como o controle sem tratamento, a população provavelmente contém uma variante resistente mutagênico. Até 20 ou 30 passagens podem ser necessárias, embora a maioria dos isolados variantes nossa fidelidade entre 5 e 15 passagens.
- Uma vez que os títulos de vírus para uma série determinada passagem atingir a mesma magnitude que os títulos de controle sem tratamento, extração de RNA a partir de todas as amostras, incluindo o controle sem tratamento a partir do número mesma passagem. Kits de extração de RNA ou extração Trizol pode ser usado.
- Executar RT-PCR usando primers que amplificam os genes da polimerase ou replicase do vírus de interesse. Em uma segunda etapa, o genoma inteiro (pelo menos as regiões de codificação) deverá ser seqüenciados para examinar se fenótipos de resistência mapa para genes de vírus de outros também. Isto é particularmente importante para mutagénicos de base analógica, como a ribavirina, que afeta outros aspectos de vírus e de função celular. Neste caso, a variante pode ser resistente a uma dessas outras atividades antiviral e não será uma variante fidelidade.
- Purificar os produtos RT-PCR usando um kit de purificação de PCR ea seqüência para obter o mutagênico resistente à seqüência consenso população. Incluem controles de fundo para o seqüenciamento de erro (ver Discussão).
- Usando um software de alinhamento de seqüência ea seqüência consenso vírus como uma referência, alinhe as seqüências. Identificar as mutações novo ponto, com particular atenção a qualquer que aparecem exclusivamente na população mutagênico tratados na passagem em que os títulos de vírus atingir níveis normais. Se essa mutação não está presente em passagens anteriores e não presentes em controles não tratados a partir do número mesma passagem (indicando adaptação a passagem de cultura de células), então essa mutação é provável responsável, pelo menos em parte, para a resistência mutagênico. Não confie na base de chamada seqüência (a versão texto da seqüência) e software de alinhamento sozinho. Verifique os cromatogramas com picos de minoria que pode ter sido perdida por software de alinhamento. Um mutante que representa 20-30% do total da população ainda vai mostrar como um pico, mas pequena demais para ser identificado como um 'N' pela análise da seqüência padrão.
4. Uma vez que uma mutação foi identificada, isolar ou gerar a variante e confirmar o fenótipo de resistência aos agentes mutagénicos vários RNA
Em seguida, a variante apresentar a mutação identificada é isolada para confirmar sua ligação com o fenótipo de resistência. É essencial que a mutação suspeita de mudar a fidelidade é estudada em um fundo geneticamente limpa (isto é, não apresentando mutações adicionais em outras partes do genoma). Na melhor situação, um clone de cDNA infeccioso existe que permita a geração de um estoque da variante resistente mutagênico por mutagênese sítio dirigida sobre um fundo limpo genética. Neste caso, a secção 4 não é necessário. No entanto, se um clone de cDNA não está disponível, o isolamento pode ser feito através de purificação de placa de vírus, descritos a seguir. Mais de uma rodada de purificação de placa pode ser necessário para isolar a variante em um plano de fundo genético.
- Isolamento dos mutantes resistentes mutagênico por ensaio de placa.
- Para isolar o mutante identificados, realizar um ensaio de placa padrão sob agarose (0,5 a 1% finais wt / vol) overlay em 6-lamelas. Preparar diluições de série do vírus, com base no estoque dos títulos, para dildoras que irá produzir entre 10 e 50 bem separados placas.
- Quando as placas são claramente visíveis (normalmente 2 a 5 dias após a infecção, dependendo do vírus), marcar a localização das placas nas placas e usando uma pipeta p200 com ponta de filtro, delicadamente mergulhe a ponta através da sobreposição de agarose com cuidado para não desalojar e mudar de posição a sobreposição (que resultaria na contaminação cruzada de placas individuais).
- Levante cuidadosamente a ponta para fora da sobreposição e transferir o plug-agarose contidas na ponta, para um eppendorf contendo 250 mL de médio e vortex. Não se preocupe se a ponta que é removido não contém agarose, para muitos vírus RNA, uma placa média para muitos vírus RNA contém 10 5 vírus e uma quantidade suficiente será transferido simplesmente por tocar a ponta para a superfície da placa.
- Pick up a 10 placas por tratamento mutagênico. Dependendo da cromatogramas de seqüenciamento, que identificou a mutação, a estimativa de aproximadamente qual a percentagem da população contém a mutação desejada. O objetivo é isolar três ou quatro placas com a mutação. Alguns destes mutantes também carregam adicionais, mutações indesejáveis, que será posteriormente identificado por seqüenciamento.
- Extrair o RNA a partir dessas amostras (mas salvar metade da amostra para fazer um estoque maior de vírus), e realizar RT-PCRs que permitam o seqüenciamento do genoma inteiro. Em média, os vírus RNA vai conter até duas diferenças mutacionais no que diz respeito à seqüência consenso, assim, seqüência de 3 ou 4 placas purificada vírus de cada vez para identificar o isolado que contém a mutação desejada, sem quaisquer mutações adicionais.
- Uma vez identificados, faça um estoque maior do vírus para todos os estudos a jusante, com a amostra de placa purificada obtida acima para infectar um maior frasco de células, por exemplo, 8x10 6 células HeLa em um frasco T75 ..
- Confirmar a sensibilidade mutagênico / resistência conferida pela mutação identificada.
- Usando o clone isolado ou recém-gerado, e um vírus de controle tipo selvagem produzida em condições semelhantes, repetir os experimentos na seção 2 usando uma gama completa de concentrações, mutagénica ou a concentração na qual a mutação de resistência foi gerado.
- Use várias condições diferentes RNA mutagênico (ribavirina, 5-fluorouracil, 5 azacitidina, aumentou Mg 2 +, Mn 2 +). Se a variante da polimerase é resistente a mais de um tipo de mutagênico, então é mais provável que esta variante é de alta fidelidade. Alternativamente, é possível que uma mutação de resistência é específico para uma única condição mutagênicos, especialmente uma vez que alguns destes compostos afetam vírus RNA através de uma série de mecanismos de 8.
5. Veja os preços de replicação
Desde a fidelidade de alteração mutações mais freqüentemente mapa para a polimerase, é possível que a mutação da polimerase mesmo vai alterar significativamente a cinética de replicação e é importante para determinar semelhanças e diferenças na replicação que irá permitir uma melhor comparação das diferenças nas freqüências de mutação realizada a seguir. Para fazer isso, examine a replicação em pelo menos duas abordagens complementares - que examina a produção de vírus e outro que examina a síntese de RNA.
- Uma etapa de crescimento da cinética do vírus
- No dia antes do experimento, sementes de seis bem-placas conforme necessário, uma placa por ponto de tempo para ser testada. Considere o uso de poços em triplicado para cada mutante eo vírus do tipo selvagem.
- No dia do experimento, remova a mídia e infectar poços com cada vírus em um MOI de 10 para garantir que cada célula é simultaneamente infectados. Incubar 30-60 minutos a 37 ° C.
- Placas de rocha a cada 10 minutos para evitar a secagem da monocamada de células. Remover vírus e lavar duas vezes com 2 ml de PBS. É importante remover o máximo do inóculo possível. Substituir por meio de crescimento.
- Após a infecção no momento da colheita = 0, o vírus de um prato. Retorno placas para incubadora e colher os vírus em intervalos regulares que abrangem um ciclo de replicação simples (por exemplo, para 3h, 5h, 7h, 9h, 12h, 24h).
- Título de vírus, colhidas em cada momento (ensaio de placa, por exemplo, TCID50, ensaio UFF), e gráfico as curvas de crescimento de título contra o tempo.
- Cinética de síntese de RNA
A cinética da síntese de RNA pode ser monitorado usando uma das abordagens indicado abaixo. Se possível, as mesmas amostras usadas para determinar a cinética de uma etapa de crescimento deve ser usado para medir os níveis de RNA.- qRT-PCR. Este procedimento dá medidas altamente quantitativa de replicação sobre uma grande faixa, a partir de um genoma cópias poucos a> 10 10, dependendo da sensibilidade do ensaio. Primers design e sondas que cobrem um pequeno fragmento (<200 pb) de uma região altamente conservada do genoma.
- Northern blot. Embora menos do que quantitativa qRT-PCR, esta técnica permite a confirmação visual de que os resultados de replicação em todo o comprimento do genoma e que nenhuma terminação da cadeia significativa ocorre como resultado da mutação da polimerase.
- Expressão de um gene repórter. Se um clone de cDNA expressar um gene repórter (luciferase, por exemplo) está disponível, então esta pode ser usada como um substituto para examinar capacidade de replicação. No entanto, o vírus recombinante não deve ser usado para outras aplicações (como a determinação da frequência de mutação), pois as pressões seletivas atuando sobre esse vírus não será o mesmo, em particular, uma vez que esses vírus têm uma tendência para excluir o gene repórter inserido.
6. Mutação freqüências medida
Esta é uma etapa crítica em que confirma que a mutação identificada polimerase confere resistência à mutagênico replicação altera fidelidade. É importante notar que as freqüências de mutação medido aqui não são as taxas de mutação. Para determinar as taxas, uma medida muito cuidado da cinética de replicação (quantidade de RNA sintetizado e duração do ciclo de replicação) deve ser tomada dentro de medição freqüências de mutação no entanto, enquanto a história de passagem e cinética de replicação são monitorados, fornece reprodutível, medidas quantitativas de replicação fidelidade. Freqüências de mutação pode ser determinada tanto na população de vírus viáveis (clones placa ou diluição limitante) ou na população de vírus total (ações vírus ou sobrenadante). Para determinar as freqüências de mutação, prepare stocks de vírus a partir de uma passagem posterior (por exemplo, passagem 2 ou mais). É importante que a população de vírus teve tempo para ampliar sua diversidade genética mais perto de um equilíbrio mutação-seleção.
- Freqüências de mutação do vírus da população viável
Essa abordagem, embora mais trabalhoso, dá informações sobre como muitas mutações estão presentes no genoma média que mantém a competência de replicação. Deve-se notar, entretanto, que um viés para as variantes maior aptidão ocorrerá e menor aptidão, variantes viável que não são facilmente placa, por exemplo, pode não ser detectado. Como tal, ela também permite melhores medidas de sinônimos (dS) e não-sinônimos (dN) substituições de nucleotídeos que podem ser usados para explorar se a seleção positiva está agindo sobre a população. No entanto, porque as mutações menos será quantificado, um número maior de seqüências serão necessários para a análise estatística. Esta técnica se baseia no isolamento de vírus individuais por qualquer placa ou de purificação de diluição limitante, como descrito acima. Como ponto de partida, recomendamos o isolamento de 48 individual "clones" do vírus de tipo selvagem ea variante mutagênico-resistente. Cada população clonal isolado desta forma espera-se realizar qualquer mutação no genoma fundador apresentados. A quantidade de RNA presente na placa isolada ou limitar a diluição também é geralmente suficiente para amplificação por RT-PCR. Se necessário, uma amplificação de curta duração (menos de um ciclo de replicação) de um número mínimo de células (por exemplo, formato de 24 placa também) pode ser usado para obter mais RNA, no entanto, a amplificação mínimo deve ser realizada para evitar o acúmulo de novas mutações. Note que cada clone e população a ser comparado deve submeter-se ao mesmo número de ciclos de replicação.- Isolar 24-48 clones de vírus de purificação de placa ou de diluição limitante.
- Extrato de RNA das populações isoladas clonal
- RT-PCR amplificar um fragmento cobrindo até 3kb para cada amostra. É melhor cobrir a região proteína estrutural, que tende a tolerar mutações mais viável do que regiões mais conservadas de genes não-estruturais.
- Purificar o PCR produtos seqüência e executar a análise de mutação (seção 7).
- Freqüências de mutação do vírus da população total de
Embora uma vantagem deste segundo método é que, mesmo variantes de fitness baixos serão incluídos no seqüenciamento, permitindo uma visão mais global do espectro de mutações. No entanto, não pode ser ideal para análises filogenéticas que assumem populações vírus viável (por exemplo, dN / dS valores) e identificação das mutações mais relevantes, uma vez que mudanças letal (alterado RNA estrutura, códons de parada, dramáticas mudanças de aminoácidos) não pode ser inteiramente identificado e seriam retidos na análise. No entanto, esta técnica permite ao pesquisador obter os dados mais estatisticamente significativa para confirmar a alteração de fidelidade quando há uma falta de um ensaio in vitro em bioquímica. Esta técnica se baseia em RT-PCR de amplificação de RNA virion total, incluindo genomas com aptidão baixa ou mutações letais que não vai produzir placas. As freqüências de mutação obtidos por este método pode ser 10 vezes maior do que por placa ou limitar a clonagem de diluição.- Extrato de RNA a partir da população total de virion
- RT-PCR amplify uma região 800-1200 nucleotídeo de uma parte da seqüência do genoma de codificação que é conhecido por tolerar mutações e têm variância genética (por exemplo, as proteínas estruturais). Fragmentos maiores não irá inserir prontamente em vetores de clonagem, como TopoTA e vai produzir um número insuficiente de transformantes. Apesar de fragmentos menores são ainda melhores, a cobertura do genoma seqüenciado pode ser muito pouco para obter significância estatística. Um fragmento de pelo menos 800 bp permite cobertura seqüência com dois primers e é um bom compromisso entre a maximização da cobertura seqüência e minimizando os custos de seqüenciamento. Descobrimos que entre 70 e 100 seqüências cobrindo uma região 800 nucleotídeos reproducibly confirma a fidelidade alteradas das variantes que temos estudado no laboratório. Note-se que outras vector / clonagem métodos podem ser usados com a mesma eficiência.
- Purificar o produto RT-PCR usando um kit comercial ou por extracção de DNA padrão / precipitação.
- Se as enzimas RT-PCR que você usa não produzem A saliências, executar uma extensão de 10 minutos, acrescentando 1 mM de ATP e Taq polimerase
- TopoTA clone seguir as instruções do fabricante
- Para cada população de vírus a serem estudados, selecione 96 colônias, identificados como tendo uma inserção positiva azul / branco triagem em XGal placas revestidas. Inicialmente, testar a presença de inserções para cada região do genoma diferente, sendo clonado, para confirmar a validade do azul / branco triagem por triagem tamanho do plasmídeo em gel de agarose ou de colônia única PCR. Usando o tamanho do fragmento e condições acima, conseguimos positivos 90%
- Crescer cada colônia em caldo de líquido durante a noite em 1 ml de meio LB em 96 placas de cultura de bactérias bem
- No dia seguinte, prepare minipreps em formato de 96 poços.
- Placa seqüência, cada um com primers suficiente (os primers utilizados para RT-PCR por exemplo, ou primers TopoTA M13) para obter o máximo de cobertura do segmento clonado. Realizar a análise de mutação (seção 7).
7. Análise da seqüência
Realizar análises seqüência usando uma referência ou uma seqüência consenso para cada população e software de alinhamento apropriado. Recomendamos Lasergene ou Sequencher que pode facilmente identificar SNPs em relação ao consenso.
- Alinhar as seqüências usando o software apropriado (por exemplo Lasergene ou Sequencher).
- Descartar seqüências de baixa qualidade (chamado de base ruim, muitos 'N ou muito curto de comprimento). Identificar o intervalo de nucleotídeos que é coberta por todas as sequências. Desde diferentes regiões do genoma vai mais ou menos toleram mutações, por motivos de comparação, é essencial que a mesma região é totalmente coberta para cada clone seqüenciado encaminhado para análise. Assim, se as reações diversas seqüência são realizadas por clone, e uma seqüência de falhar por um determinado clone, descarte o clone (todas as seqüências) da análise.
- Identificar e contar os SNPs que são diferentes da cepa de referência.
- Calcular a freqüência de mutação, dividindo o número total de SNPs identificados pelo número total de nucleotídeos seqüenciados (número de clones x comprimento da região seqüenciados). Apresentar este número como número médio de mutações por 10K nt seqüenciados torna o número mais user-friendly, em seguida, deixá-lo como por nucleotídeos. Por exemplo, para a população do tipo selvagem na Tabela 1, 55 mutations/121, 978 nucleotídeos total X 10.000 = 4,51 mutações por 10.000 nucleotídeos seqüenciados.
- Se o SNP mesma aparece em um grande número de clones, apresentamos dois valores que incluir ou excluir estas mutações repetidas. Geralmente, para uma população de vírus produzido a partir de um pai homogênea (por placa ou purificação de um clone infeccioso) e várias passagens apenas algumas vezes em cultura de células, a seleção positiva, não tem exercido os seus efeitos o suficiente para provocar o acúmulo do SNP mesma ea mutação valores de freqüência medidos dessa forma refletir melhor a freqüência de erro da polimerase com efeitos mínimos de seleção positiva ou depurar.
- Determinar a distribuição de mutação. Faça uma lista de classificação do número de clones de cada população que apresentam 0, 1, 2, 3, etc .. mutações na região seqüenciada.
- Calcular a diversidade da população viral, por comparação distância pairwise. Da limpeza e seqüências editado manualmente preparar um alinhamento, incluindo a região de referência. Existem programas de alinhamento vários disponíveis: ClustalW / X ( http://www.clustal.org/ ), músculo ( http://www.drive5.com/muscle/ ), EbioX para usuários de Mac ( http://www. ebioinformatics.org / ebiox / ), etc Certifique-se de todas as suas sequências têm o mesmo comprimento, corte se necessário. Recomenda-se que o início das sequências é um códon de codificação para facilitar a análise posterior. Sugerimos manter todos os alinhamentos em formato fasta, que é facilmente lido pela maioria dos softwares disponíveis.
- Para realizar a análise de distância pairwise, calcular todas as possíveis comparações entre as seqüências de pares pertencentes à mesma população. Mutações média encontrada nas comparações pode então ser calculada para indicar heterogeneidade da população.
- Valores de sinônimos (dS) e não-sinônimos (dN) dentro da população pode ser facilmente obtida. Encontramos o software MEGA (http://www.megasoftware.net/) útil e amigável para este tipo de análise.
- Para obter direção seleção damos duas possibilidades, embora não sejam os únicos possíveis. 1) Obter a relação entre dN e dS. Valores superiores a uma média de seleção positiva. Valores inferiores a 1 significam seleção purificadora. 2) Use o servidor Datamonkey ( http://www.datamonkey.org/ ). Upload de suas alinhamentos em formato fasta e analisá-los com o módulo SLAC. Isto lhe dará uma estimativa de dN / dS.
- Realizar análises estatísticas. Dependendo da quantidade de dados e número de seqüências, uma variedade de testes pode ser usado. Alguns estudos têm contado com testes de qui quadrado do número total de mutações versus o número total de nucleotídeos consenso onde todos os clones são combinados 9. Outros estudos realizados testes exato de Fisher, calculado sobre o número de seqüências que apresentam mutações em relação ao número de seqüências sem mutações 10. Se um número suficiente de dados de mutação é gerado, recomendamos realizar um teste de soma classificado como Mann Whitney U. Para isso, a pontuação do número de clones em cada população de vírus em número de mutações presentes em cada seqüência de RNA. A Whitney Mann, então, testar as diferenças na distribuição de mutação das populações. Por este motivo, recomendamos o seqüenciamento de pelo menos 800 pares de base, para aumentar a probabilidade de encontrar clones com múltiplas mutações. Este teste é robusto, mas requer maiores tamanhos de amostra. Por outro lado, ele não exige que o tamanho da amostra igual para as duas populações sendo comparado (por exemplo, as amostras da Tabela 1 são n 1 = 148 e n 2 = 84).
8. Resultados representativos:
O efeito dose-dependente do mutagéneo concentração na viabilidade celular e da viabilidade do vírus é mostrado na Figura 1. Neste exemplo, descobrimos que a passagem do vírus em 100 AZC mM foi reduzida no título do vírus pela log 10,5-2 alvo, mas a viabilidade das células HeLa foi não afetou negativamente durante os 2 dias necessários para a infecção pelo vírus. Esta experiência-piloto levou à escolha da concentração de 100 mM AZC para a passagem serial de vírus, à selecção de resistência mutagênico. Figura 2 ilustra a redução inicial no título, seguido pelo aparecimento de um fenótipo de resistência mutagênico. Durante o primeiro poucas passagens em mutagênico, como mutações letais acumular, uma queda significativa nos títulos de vírus ocorre. Gradualmente, uma variante resistente mutagênico surge uma emergência suas coincide com o regresso aos títulos de vírus não é diferente de controles não tratados. Nesta fase, uma grande porcentagem da população de vírus apresenta a mutação de resistência. Seqüenciamento do vírus nesta população revela a mudança de aminoácido (s) responsável. Uma vez identificados e isolados ou recém-gerado, o vírus mutagênico resistente pode ser menos sensível do que a do tipo selvagem para diferentes RNA mutagénicos (análogos de base de estrutura diferente, por exemplo). Figura 3 mostra um RNA vírus mutagênico resistentes Coxsackie B3 que os títulos superiores tipo selvagem, na presença de ribavirina, 5-fluorouracil e 5-azacitidina e MgCl 2 e alta MnCl 2. Resistência ampla para RNA mutagénicos é um forte indicador de fidelidade de replicação aumentada. Verificando que a cinética de replicação da variante fidelidade é semelhante ao vírus de tipo selvagem ajudará na comparação das frequências de mutação. Figura 4 mostra a um passo do crescimento da cinética de uma variante de alta fidelidade em relação ao tipo selvagem. Se as taxas de replicação e títulos de final não são semelhantes, então devem ser tomadas medidas para comparar populações de vírus de tamanho similar, que sofreram o mesmo número de rodadas de replicação. A ligação entre a taxa de síntese de RNA e de fidelidade de replicação não está bem caracterizada, especialmente in vivo. A taxa mais lenta replicação pode resultar em uma diminuição da frequência de mutação (maior fidelidade), embora esta não seja uma regra absoluta, como mostrado na Figura 4. Com os parâmetros acima estabelecidos, as freqüências de mutação da variante fidelidade e populações de tipo selvagem pode ser comparado a obter a confirmação genética de fidelidade de replicação alterada. Figura 5 mostra um alinhamento de sequências de um tipo selvagem e variante de alta fidelidade, com mutações pontuais identificadas. As mutações são contados, classificados de acordo com número de mutações por clone (Tabela 1), e representada como uma freqüência de mutação médio por população, por 10.000 nucleotídeos seqüenciados, Figura 6.

Figura 1. Determinar as condições ideais para selecionar para RNA resistência mutagênico:. Retenção de viabilidade celular com uma alta moderada células (drop log 1-2 no título de vírus) HeLa foram tratadas com concentrações indicadas de ribavirina e infectados com o vírus tipo selvagem Coxsackie B3 em uma MOI de 0,01. 48 horas após a infecção, o vírus progênie foi colhida e os títulos foram determinados por TCID 50. A porcentagem de células sobreviventes ao tratamento às 48 horas, determinado pelo Trypan coloração azul, é indicado abaixo do eixo-x. Os resultados mostram que as concentrações de 100 e 200 mM reduzir títulos vírus por log 1-2, sem afetar a viabilidade celular.
Figura 2. Passagem de série na presença de concentrações moderadas de RNA mutagénicos seleciona para as populações resistentes mutagênico. Nesta figura, Chikungunya vírus foi passado em células HeLa, na presença de 50 mM ribavirina (barras cinza). Passagens de controle foram realizados na ausência de ribavirina (barras pretas). Após cada passagem, progênie do vírus foi quantificada por ensaio de placa clássico sobre células BHK. O efeito mutagênico é evidente durante as passagens de primeira (P1 e P2 em relação ao p0 população de partida), onde o vírus tratados queda títulos por 2 log. Gradualmente, os títulos voltam ao normal (sem tratamento) níveis. Não existem diferenças significativas são observadas em populações passagem 5 mutagênico tratados em comparação com não tratado, sugerindo que as variantes resistentes foram selecionados. Na verdade, o seqüenciamento de consenso da população identificaram mutações únicas na população de vírus em tratamento ribavirina.
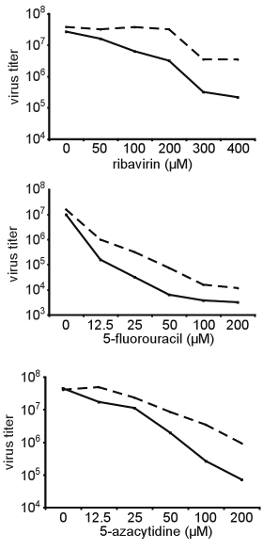
Figura 3. Confirmação de resistência ampla para RNA mutagénicos de estrutura diferente. Mostrado aqui, a alta fidelidade A372V variante de vírus Coxsackie B3 que foi inicialmente isolada na tela descrito na Seção 3 foi gerada a partir de um clone infeccioso e testado para a sua sensibilidade em relação a diferentes concentrações de diferentes RNA mutagénicos (ribavirina, 5-fluorouracil, 5 azacitidina). Células HeLa foram tratadas com concentrações indicadas de ribavirina e infectados com o vírus tipo selvagem Coxsackie B3 em um MOI de 0,01. 48 horas após a infecção, o vírus progênie foi colhida e os títulos foram determinados por TCID 50. Mostrados aqui estão os títulos de tipo selvagem (linhas sólidas) e variante A372V (linhas tracejadas) em função da concentração mutagênico. A372V consistentemente mais elevados do que os títulos de tipo selvagem em todas as condições testadas.
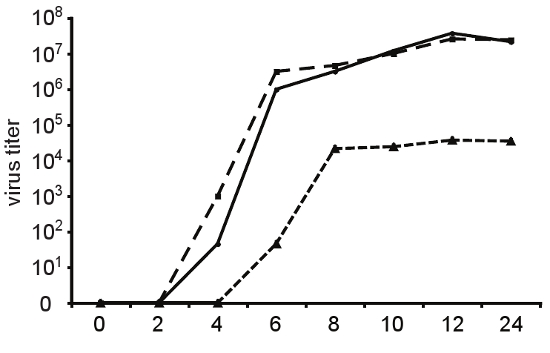
Figura 4. Taxas de replicação e variantes de fidelidade. Para determinar a cinética de uma etapa de crescimento da produção do vírus, as células HeLa foram infectadas no MOI = 10 com qualquer tipo selvagem (linha sólida), de alta fidelidade variante A372V (traços longos), ou variante de replicação deficiente Cx64 (curto traços) de vírus Coxsackie B3. Em pontos de tempo indicado, progênie do vírus foi colhida a partir de células e sobrenadantes de congelamento e descongelamento e tituladas por TCID 50. O aumento da fidelidade A372V não coincide com um defeito de replicação observáveis em cultura de tecidos. A Cx64 variante apresenta um atraso significativo na cinética de replicação e atinge máximo de títulos que são mil vezes menor do que o vírus de tipo selvagem.
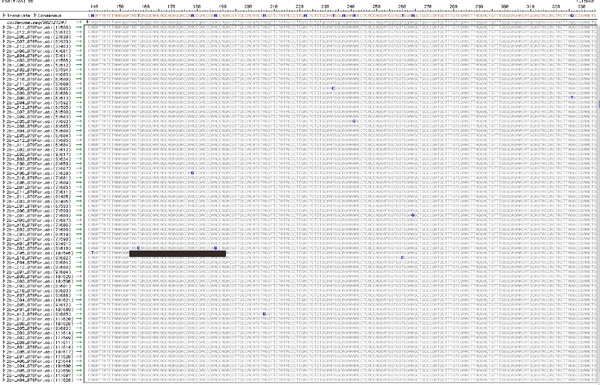
Figura 5. Alinhamento das seqüências TopoTA clonada a partir de cada população de vírus. Usando a abordagem descrita na Seção 7, cada seqüência obtida a partir de produtos clonados RT-PCR, presumivelmente, origina a partir de um genoma único e exclusivo na população de vírus total e seria, portanto, carregam mutações únicas. A figura mostra um alinhamento típico, após a limpeza de seqüências de baixa qualidade e visualização de SNPs. Os SNPs total (10 nesta figura) dentro de uma população são contados, eo número de SNPs que aparecem em cada clone são anotados. Por exemplo, o clone sublinhada por uma barra, contém duas mutações únicas enquanto 8 clones outros contêm uma mutação única, exclusiva. Esta informação é usada para compilar Tabela 1. Para ver uma versão maior desta figura, por favor clique aqui .
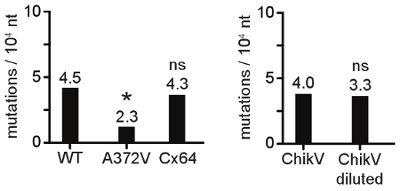
Figura 6. Representação gráfica das frequências de mutação das populações de vírus. Para facilitar a interpretação, os dados numéricos obtidos a partir de seqüência e análise estatística pode ser representado tanto como um gráfico ou histograma (mostrado aqui). Vírus A372V gera mutações menos de tipo selvagem e apresenta uma freqüência de mutação significativamente menor (*, p <0,01). A variante Cx64, que replica a títulos de 1.000 vezes menor do que o tipo selvagem, presents a freqüência mesma mutação (ns, não significativo), indicando que a velocidade de replicação e fidelidade não são necessariamente ligados. O mesmo vírus Chikungunya população (CHIKV) dá freqüências mutação semelhante se o estoque de vírus, ou uma diluição de 10 5 vezes, é usado para a extração de RNA.
Resumo de mutação de distribuição para análise estatística.
Nota: Para cada clone, é essencial que a região genômica mesmo (e comprimento de seqüência) é coberto. Neste caso, 859 nucleotídeos por clone. Isso é fundamental para análises estatísticas. Por outro lado, os testes de classificação soma usado para análise estatística não exigem que o tamanho da amostra a ser o mesmo, o pesquisador é livre para comparar população de diferentes tamanho da amostra. Assim, os 142 clones de tipo selvagem pode ser comparado aos 84 clones de A372V.
| # Clones com mutações n | tipo selvagem | A372V |
| 7 mutações | 0 | 0 |
| 6 mutações | 0 | 0 |
| 5 mutações | 0 | 0 |
| 4 mutações | 0 | 0 |
| 3 mutações | 1 | 0 |
| 2 mutações | 6 | 2 |
| 1 mutações | 40 | 14 |
| 0 mutações | 95 | 68 |
| Total de mutações | 55 | 18 |
| Total de clones seqüenciado | 142 | 84 |
| Total de nucleotídeos seqüenciados | 121978 | 72156 |
| Mutations/10 4 nt | 4,51 | 2,49 |
Tabela 1. . Síntese mutação de distribuição para análise estatística Nota: Para cada clone, é essencial que a mesma região genômica (e comprimento de seqüência) é coberto. Neste caso, 859 nucleotídeos por clone. Isso é fundamental para análises estatísticas. Por outro lado, os testes de classificação soma usado para análise estatística não exigem que o tamanho da amostra a ser o mesmo, o pesquisador é livre para comparar população de diferentes tamanho da amostra. Assim, os 142 clones de tipo selvagem pode ser comparado aos 84 clones de A372V.
Discussão
Escolha da linha celular. A eficácia de análogos de base como RNA mutagénicos correlaciona-se com a sua aceitação em relação a diferentes tipos de células 11. Se a linha de células que é normalmente utilizado para a passagem do vírus se mostra refratária à absorção mutagênico ou muito sensíveis (alta toxicidade celular), pode ser necessário usar uma outra linha de células que atenda a esses requisitos e ainda é permissivo para a replicação viral. Uma vez que a variante resistência mutagênico é isolado, o restante da caracterização pode ser realizada no original linha celular, de preferência. Em nossa experiência, as células HeLa absorver a mutagênico; células BHK requerer até 10 vezes maiores concentrações e células Vero são refratários à captação mutagênico.
Escolha de mutagênico. Na tentativa de isolar variantes fidelidade por mutagênico tratamento, a probabilidade de sucesso é maior se houver mais de um tipo de mutagênico é usado. Mutagénicos de base analógica de estrutura diferente, que são erroneamente incorporados genomas durante a replicação será predominantemente induzir resultar em um subconjunto específico de mutações nos ciclos de replicação subsequentes: ribavirina favorece GtoA e mutações de transição CtoU 12; 5 azacitidina tem um viés similar, com a Além de CtoG e transversões GtoC 13; 5-fluorouracil, preferencialmente induz transições Atogue e UtoC 14. Alternativamente, maiores concentrações de Mg 2 + ou Mn 2 + pode ser suplementada ao meio para aumentar a freqüência de mutação dos vírus RNA total, sem o viés descrito acima de 12. Dependendo seqüências do vírus códon, e as mudanças códon necessária para gerar uma variante fidelidade, algumas destas condições irá favorecer o surgimento desta variante em detrimento de outros. Para a maior fidelidade G64S poliovírus e vírus Coxsackie A372V, tratamento ribavirina mais prontamente selecionados para as variantes porque a transição Atogue necessários no local códon correspondeu à mutações predominantemente gerado por este ribavirina.
MOI tamanho da população vs. Em virologia, protocolos para a infecção por cultura de tecidos prestar especial atenção à multiplicidade de infecção (MOI), para evitar o acúmulo de partículas defeituoso interferente (baixa MOI) ou para promover a recombinação entre os vírus (alta MOI), para exemplo. Selecionar para eventos emergência sobre serial passaging, também é importante considerar o tamanho da população de vírus. Desde o mutante resistente inicialmente existe em baixa freqüência, é melhor transferir tão grande tamanho de uma população possível de uma passagem para a próxima (10 5 -10 6 vírus, por exemplo) para evitar a perda destas variantes emergentes em cada passagem. Ampliação do tamanho do bem ou frasco (número de células infectadas) podem ajudar a minimizar o aumento do MOI se isso é preocupante. Por outro lado, para experimentos em que a sensibilidade de um vírus para mutagênico está sendo testado, infecção MOI baixa é realizada, a fim de aumentar o número de ciclos de replicação que ocorre no experimento e para evitar resgate de genomas mutagenizados por genomas maior aptidão através de complementação em co-células infectadas. Isso é importante porque as mutações no genoma da progênie gerada durante a primeira rodada de replicação não será imediatamente detectada. A maioria destes RNAs mutagenizados ainda serão embalados em virion. É na próxima rodada de infecção que as mutações letais presentes nestes genomas resultará em um ciclo de replicação abortada, e redução do título de vírus. Pode ser necessário para permitir várias rodadas de acumulação de mutações antes de um efeito significativo da mutagênese letal é observado. Finalmente, se ao longo da série passagem na presença de agentes mutagénicos, os títulos de vírus continue a cair até a extinção, em seguida, o pesquisador deve tentar passaging vírus gradualmente crescentes quantidades de mutagênico (a partir de uma concentração muito baixa).
Mutagénicos isolamento e geração do clone mutagênico RNA resistentes da população RNA mutagênico-resistente. RNA introduzir múltiplas mutações aleatórias para cada genoma, mas a seleção para resistência só irá enriquecer (e corrigir a seqüência de consenso) a mutação de resistência. Para identificar esta mutação, a sequência de população resistente mutagênico (consenso da população) e não vírus individual. Assim, o único, mutações aleatórias criadas pelo mutagênico não são detectados na seqüência, apenas as mutações que resultam em mudanças consenso após a seleção, são encontrados. Em nossa experiência, identificam apenas uma ou duas alterações na seqüência de tal consenso. Uma vez que a população resistente mutagênico é obtida e a mutação de resistência é identificada, é necessário para gerar um estoque de mais puro desta variante. Acima, descrevemos um procedimento de purificação de placa. Alternativamente, se o vírus de interesse não produz placas facilmente identificáveis, a variante desejada pode ser Purified limitando diluição. Esta abordagem é, essencialmente, a 50 TCID em formato de 96 poços, onde o estoque de vírus é diluído de tal forma que menos de 50% dos poços estão infectados. Usando esta diluição, a mesma abordagem que acima foi tirado, de isolar até 10 variantes individuais e confirmando suas seqüências. Como mencionado, no melhor dos casos, um clone infeccioso de cDNA da cepa do vírus está disponível. Isolamento da variante seria, portanto, não ser necessário. Em nossa experiência, fidelidade variantes são o resultado de um único substituições de aminoácidos e pode assim ser gerados usando simples, kits mutagênese comercializado como Quikchange (Agilent). A opção secundária é a utilização de um clone de cDNA de uma cepa intimamente relacionados. No entanto, se uma cepa relacionada é usado, nós sugerimos usando tanto a abordagem deste e isolamento do vírus (purificação de placa, por exemplo) porque descobriram que a mutação mesma fidelidade alterando em dois vírus estreitamente relacionados não vai necessariamente ter o mesmo efeito.
Fidelidade e Seleção de replicação. Variantes de RNA mutagênico resistentes resultaram no isolamento de ambas as variantes de maior e menor fidelidade com características de crescimento que são semelhantes aos seus homólogos do tipo selvagem 4,12,15. Atualmente, a ligação entre as taxas de atividade da polimerase e fidelidade não é totalmente compreendido. In vitro estudos bioquímicos utilizando purificada RNA polimerase têm demonstrado que as variantes com maior fidelidade têm taxas de processamento mais lento, enquanto variantes menor fidelidade tendem a ter um processamento mais rápido 1-3,12. Em cultura de tecidos, essas diferenças geralmente não são evidentes, sugerindo que a disponibilidade de recursos, ao invés de cinética de atividade intrínseca da polimerase, é o passo limitante. Se a variante fidelidade repetições, com cinética de que não são significativamente diferentes de tipo selvagem, em seguida, uma comparação de suas freqüências de mutação pode ser feita diretamente. Se uma mudança muito significativa na cinética de replicação existe, então os dados devem ser normalizados para explicar as diferenças cinética, por exemplo, comparando vírus que sofreram o mesmo número de ciclos de replicação. Em nossa experiência, embora nenhuma diferença significativa em uma etapa cinética de crescimento foram observadas entre o tipo selvagem e variantes de alta fidelidade, observou-se que as variantes com maior fidelidade consistentemente mais elevada titulação (dentro de um log) em relação ao tipo selvagem, mas eles fazem um pouco menos RNA (dentro da mesma ordem de magnitude), sugerindo ainda que os genomas que contêm produzir mutações menos e são, portanto, mais infeccioso.
Preparação de amostras e seqüenciamento. Para todos os passos nestes protocolos, é imperativo que a alta-fidelidade, uma prova de leitura de enzimas são utilizadas para PCR e RT-PCR para limitar a introdução de mutações adicionais, uma vez que não pode ser distinguido de mutações biologicamente relevantes. É fundamental que as populações de vírus a serem comparados foram preparadas nas mesmas condições (história passagem, meio de cultura de tecidos, a temperatura, método de extração de RNA, RT-PCR protocolos, etc) Também é importante assegurar que o material de partida foi o suficiente obtidos a partir da extração de RNA de modo que uma banda forte é gerada por RT-PCR. A diluição de 1 / 100 da amostra de RNA também deve dar uma banda de RT-PCR detectável, indicando que a amostra contém um número suficiente de moléculas de RNA para evitar viés de representação (amplificação do genoma do mesmo repetidamente). Desde frequência de mutação é uma distribuição, seria de esperar que os valores semelhantes poderão ser obtidas independentemente do tamanho da população, desde o viés referido não está ocorrendo. Como a Figura 6 mostra, a 10 de diluição de 5 vezes de um estoque de vírus dá uma frequência de mutação que não é significativamente diferente do estoque parental.
Até que as condições ideais para TopoTA clonagem são encontradas, confirmar a presença de inserções após triagem azul / branco, por colônia PCR antes de seqüenciamento. Como um controle para o ruído de mutação (mutações introduzidas por RT-PCR e sequenciamento), clone de um produto PCR de um rolamento plasmídeo a sequência viral mesmo e / ou clone e seqüência RT-PCR em produtos de RNA transcrito vitro correspondente ao genoma do vírus (seja ciente de que diferentes enzimas de transcrição in vitro têm taxas de erro diferentes e não podem dar informações úteis como para o erro de fundo de verdade em seu procedimento). Algumas seqüências de vírus podem ser tóxica para bactérias, por isso, é importante verificar isso antes de decidir sobre a região do genoma viral a ser sequenciado para a freqüência de mutação. Na análise de seqüências obtidas pelos TopoTA, note que cada clone deve conter apenas inserir um / seqüência. Se um pico duplo é observada, sugerindo uma população mista, é possível que dois vizinhos colônias bacterianas foram selecionados. Também é possível, embora improvável, dada a baixa freqüência de mutação na replicação bacteriana, que a mutação foi introduzida durante a amplificação do plasmídeo na cultura bacteriana. Na placa purified populações, um pico duplo pode representar placas sobrepostas, ou um vírus que está adquirindo uma nova mutação ou reverter uma mutação durante o desenvolvimento da placa. Ser consistente e decidir sobre a contar ou não contar estas mutações.
Finalmente, tenha em mente que as freqüências de mutação usadas aqui são valores relativos. Eles são válidos apenas para comparar populações de vírus cultivadas sob a mesma condição, e seqüenciado sobre a mesma região! Eles não devem ser tomados como valores absolutos da taxa de mutação, ou a freqüência de mutação do genoma como um todo. No entanto, quando as condições são controladas, permitem realmente reprodutível, comparações quantitativas de diferenças na distribuição de mutação e de freqüência.
Divulgações
Agradecimentos
Este trabalho foi financiado por fundos da Medicina e Pesquisa em Saúde doação da Cidade de Paris, os franceses Nacional conceder ANR-09-JCJC-0118-1, ea ERC Começando Grant Projeto RNAvirusPopDivNVax não. 242.719.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
|---|---|---|---|
| ribavirina | Sigma | R9644-10mg | |
| 5-fluorouracil | Sigma | F6627-1G | |
| 5-azacitidina | Sigma | A2385-100mg | |
| MgCl 2 | Sigma | M1028-100ML | |
| MnCl 2 | Sigma | M1787 | |
| Azul de tripano | Sigma | T8154-20ML | |
| TopoTA clonagem kit | Invitrogen | 10351021 | |
| Quikchange mutagênese kit | Agilent | 200516 | Se um clone de cDNA infeccioso está disponível |
| De 96 poços miniprep kit | Macherey-Nagel | 740625 | |
| Lasergene, Sequencher | DNAstar, Gene Codes Corporação | www.dnastar.com www.genecodes.com | Ou software de alinhamento outras |
Referências
- Arias, A. Determinants of RNA-dependent RNA polymerase (in)fidelity revealed by kinetic analysis of the polymerase encoded by a foot-and-mouth disease virus mutant with reduced sensitivity to ribavirin. J Virol. 82, 12346-12355 (2008).
- Arnold, J. J., Vignuzzi, M., Stone, J. K., Andino, R., Cameron, C. E. Remote site control of an active site fidelity checkpoint in a viral RNA-dependent RNA polymerase. J Biol Chem. 280, 25706-25716 (2005).
- Korneeva, V. S., Cameron, C. E. Structure-function relationships of the viral RNA-dependent RNA polymerase: fidelity, replication speed, and initiation mechanism determined by a residue in the ribose-binding pocket. J Biol Chem. 282, 16135-16145 (2007).
- Pfeiffer, J. K., Kirkegaard, K. A single mutation in poliovirus RNA-dependent RNA polymerase confers resistance to mutagenic nucleotide analogs via increased fidelity. Proc Natl Acad Sci U S A. 100, 7289-7294 (2003).
- Pfeiffer, J. K., Kirkegaard, K. Increased fidelity reduces poliovirus fitness and virulence under selective pressure in mice. PLoS Pathog. 1, e11-e11 (2005).
- Vignuzzi, M., Stone, J. K., Arnold, J. J., Cameron, C. E., Andino, R. Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population. Nature. 439, 344-348 (2006).
- Vignuzzi, M., Wendt, E., Andino, R. Engineering attenuated virus vaccines by controlling replication fidelity. Nat Med. 14, 154-161 (2008).
- Crotty, S., Cameron, C., Andino, R. Ribavirin's antiviral mechanism of action: lethal mutagenesis. J Mol Med. 80, 86-95 (2002).
- Coffey, L. L., Vignuzzi, M. Host alternation of chikungunya virus increases fitness while restricting population diversity and adaptability to novel selective pressures. J Virol. 85, 1025-1035 (2011).
- Ciota, A. T. Role of the mutant spectrum in adaptation and replication of West Nile virus. J Gen Virol. 88, 865-874 (2007).
- Ibarra, K. D., Pfeiffer, J. K. Reduced ribavirin antiviral efficacy via nucleoside transporter-mediated drug resistance. J Virol. 83, 4538-4547 (2009).
- Levi, L. I. Fidelity variants of RNA dependent RNA polymerases uncover an indirect, mutagenic activity of amiloride compounds. PLoS Pathog. 6, e1001163-e1001163 (2010).
- Sierra, S., Dávila, M., Lowenstein, P. R., Domingo, E. Response of foot-and-mouth disease virus to increased mutagenesis: influence of viral load and fitness in loss of infectivity. J Virol. 74, 8316-8323 (2000).
- Ruiz-Jarabo, C. M., Ly, C., Domingo, E., de la Torre, J. C. Lethal mutagenesis of the prototypic arenavirus lymphocytic choriomeningitis virus (LCMV). Virology. 308, 37-47 (2003).
- Sierra, M. Foot-and-mouth disease virus mutant with decreased sensitivity to ribavirin: implications for error catastrophe. J Virol. 81, 2012-2024 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados