Method Article
Oral Biofilm Analyse Palatal Expander durch Fluoreszenz in situ Hybridisierung und konfokale Laser Scanning Mikroskopie
In diesem Artikel
Zusammenfassung
Wir präsentieren ein Protokoll für die strukturelle und Analyse der Zusammensetzung der natürlichen oralen Biofilm aus kieferorthopädischen Apparaturen mit In situ Hybridisierung (FISH) und konfokale Laser Scanning Mikroskopie (CLSM). Oral Biofilm-Proben wurden von palatinal Expander gesammelt, Schaben Acrylharz-abplatzt ihrer Oberfläche und deren Weiterleitung für die molekulare Verarbeitung.
Zusammenfassung
Konfokale Laser Scanning Mikroskopie (CLSM) der natürlichen heterogenen Biofilm ist heute durch ein umfassendes Angebot an Färbetechniken, von denen einer Fluoreszenz in situ Hybridisierung (FISH) erleichtert. 1,2 Wir haben eine Pilotstudie, in der mündlichen Biofilm Proben aus festen gesammelt wurden, kieferorthopädischen Apparaturen (palatinale Expander) wurden durch FISH mit dem Ziel, die dreidimensionale Organisation der natürlichen Biofilm und Plaque zu beurteilen gefärbt. 3,4 FISH schafft eine Gelegenheit, um Zellen in ihrer Muttersprache Biofilm-Umgebung Fleck durch die Verwendung von fluoreszenzmarkierten 16S rRNA-Targeting-Sonden. 4-7,19 Im Vergleich zu alternativen Techniken wie Immunfluoreszenz Kennzeichnung, ist dies eine kostengünstige, präzise und einfache Kennzeichnung Technik, um verschiedene bakterielle Gruppen in gemischten Biofilm Konsortien zu untersuchen. 18,20 Allgemeine Sonden verwendet wurden, die Bindung an Eubakterien ( EUB338 + + EUB338II EUB338III; nachfolgend EUBmix), 8-10 Firmicutes (LGC354 AC;. Wurden nachfolgend LGCmix), 9,10 und Bacteroidetes (Bac303) 11 Darüber hinaus spezifische Sonden Bindung an Streptococcus mutans (MUT590) 12,13 und Porphyromonas gingivalis (POGI) 13,14 verwendet . Die extreme Härte der Oberfläche beteiligten Materialien (Edelstahl und Acryl) zwang uns, neue Wege zur Herstellung des Biofilms zu finden. Da diese Oberflächen-Materialien könnten nicht ohne weiteres mit einem Kryotom geschnitten werden, wurden verschiedene Methoden zur Probenahme untersucht werden, um intakte orale Biofilm zu erhalten. Die meisten praktikable dieser Ansätze ist in dieser Mitteilung vorgestellt. Kleine Flocken des Biofilms tragenden Acrylharz wurden mit einem sterilen Skalpell abgekratzt, kümmert sich nicht um den Biofilm-Struktur beschädigen. Pinzetten wurden verwendet, um Biofilm aus dem Stahl-Oberflächen zu sammeln. Einmal gesammelt, wurden die Proben fixiert und direkt auf Polysine beschichteten Glasplättchen gelegt. FISH wurde direkt auf diese Folien mit den Sonden m durchgeführtentioned oben. Verschiedene FISH-Protokolle wurden kombiniert und modifiziert werden, um ein neues Protokoll, das einfach zu handhaben war zu schaffen. 5,10,14,15 Anschließend werden die Proben mit einem konfokalen Laser-Scanning-Mikroskopie analysiert. Namhafte Konfigurationen 3,4,16,17 visualisiert werden, einschließlich Pilz-style-Formationen und Gruppen von Kokken durch Kanäle durchzogen. Darüber hinaus wurden die bakterielle Zusammensetzung dieser typischen Biofilm Strukturen analysiert und 2D-und 3D-Bilder erstellt.
Protokoll
1. Probengewinnung
- Entfernen Sie feste palatinale Expander nach 4 Monaten von intraoralen Service. Verwenden Sie NOLA Dry Field System die Geräte, bevor Sie sie (Abbildungen 1 und 2) zu isolieren.
- Gebrauch sterilisiert Zangen, Handschuhen und Trays zu Expander ohne Zugabe von Verunreinigungen (Abb. 3 und 4) zu entfernen.
- Shop-Objekte in Sarstedt 120 ml Fläschchen bei -20 ° C. Nehmen Sie zum Labor auf Eis und Verfahren innerhalb von 24 Stunden.
2. Biofilm Fixierung
- Abkratzen Biofilm Flocken mit einem sterilen Skalpell oder sammeln Stücke mit einer sterilen Pinzette.
- Add eiskalte 4% PFA-Lösung bis die Probe gut bedeckt ist.
- Inkubieren Sie die Mischung bei +4 ° C (nicht einfrieren!) Für 3 bis 12 Stunden. Längere Fixierung Zeiten oder höher Fixierung Temperaturen machen die Zellhüllen der gram-negativen Zellen weniger durchlässig für Oligonukleotid-Sonden.
- Entfernen PFA-Lösung und Waschen mit eiskaltem 1X PBS. Wiederholen Sie diesen Schritt 2-3 mal zu remove restliche PFA.
- Resuspendieren der Probe in 1 vol. eiskaltes 1X PBS geben und 1 vol. eiskaltem 96% (v / v) Ethanol.
- Lagern Sie die Proben bei -20 ° C. Proben fixiert nach diesem Protokoll kann für mehrere Monate bis Jahre gespeichert werden.
3. Die Dehydratisierung von festen Proben
- Übernehmen 5-30 ul der PFA-fixierten Probenmaterials auf einen Objektträger.
- Trocken bei 46 ° C für ca. 15 min oder bei Raumtemperatur länger. Tauwetter das Lysozym und das Formamid.
- Add 250 ul Lysozym (1mg/ml) bei Raumtemperatur für 10 min und erleichtert so das Eindringen der Sonden in den Zellen durch die teilweise Zerstörung der Zellwände.
- Dip Abgleiten in 50%, 80% und 96% (v / v) Ethanol für jeweils 3 min. Die entwässernde Wirkung der Ethanol-Konzentration Serie wird zerfallen die Zellmembranen.
- Trocknen Sie die Objektträger bei 46 ° C für 10 min.
4. In-situ-Hybridisierung
- Bereiten Sie 1 mlfrischen Hybridisierungspuffer (siehe Tabelle 1 für Konzentrationen). Formamid-Konzentrationen in dieser Studie verwendet: 10% (EUBmix), 20% (Bac303, POGI, MUT590) bzw. 45% (LGCmix).
- Auftauen Oligonukleotidsonde Lösungen. Aufgetaute Sonden sollten auf Eis gehalten werden und vor Licht geschützt.
- Add 2 ul jeder Sonde zu 200 ul Hybridisierungspuffer, gut mischen und wenden Sie die Mischung auf die dehydrierte Probe auf einen Objektträger.
- Legen Sie ein Stück Seidenpapier in eine quadratische Petrischale und gießen Sie die verbleibenden Hybridisierungspuffer auf die Tissue-Papier.
- Anschließend wird der Objektträger horizontal in die Schüssel, und schließen Sie die Schüssel. Inkubieren in einem Ofen bei 46 ° C für 1-5 Stunden (90 min wird in den meisten Fällen genügen). Das Gericht fungiert als einer feuchten Kammer verhindert Verdunstung Hybridisierungslösung aus der Folie. Insbesondere kann die Verdunstung von Formamid verursachen unspezifische Sonde Bindung an Nicht-Zielzellen.
- Bereiten Sie 50 ml Waschpuffer (siehe Tabelle 2 für concentrations). Bereiten Sie den Waschpuffer in einem 50 ml-Tube und Vorwärmung bis 48 ° C in einem Wasserbad. Der Waschschritt wird bei 48 ° C durchgeführt
- Entfernen Sie die Schale mit der Folie aus der Hybridisierungsofen. Sofort abwaschen Hybridisierungspuffer mit einem kleinen Volumen von vorgewärmten Waschpuffer, und übertragen Sie die Folie in den verbleibenden Waschpuffer.
- Das Röhrchen mit dem Waschpuffer und dem Schlitten zurück ins Wasserbad und Inkubieren bei 48 ° C für 10-15 min.
- Entfernen Sie Folie aus der Tube und tauchen Sie ein in eiskaltem ddH 2 O für 2-3 Sekunden, um restliche Waschpuffer zu beseitigen.
- Der Luft trocknen die Folie so schnell wie möglich (die Verwendung von Druckluft wird empfohlen). Schnelle Trocknung reduziert Sonde Dissoziation.
- Getrocknete Folien können im Dunkeln bei -20 ° C für mehrere Wochen gelagert, ohne signifikanten Verlust der Sonde-verliehenen Fluoreszenzsignal.
5. Mikroskopie
- Nach FISH und Waschen apply zwei Tropfen antifadent in der Nähe der linken und rechten Ende eines Schiebers (gefroren Scheiben sollten auf Raumtemperatur erwärmt vor diesem Schritt werden).
- Legen Sie ein Mikroskop Abdeckung auf der Oberseite gleiten und warten, bis die antifadent über die gesamte Folie ausgebreitet hat. Beachten Sie, dass eine übermäßige Menge an antifadent kann das mikroskopische Bild verwischen.
- Beachten Sie die Proben unter einem konfokalen Laser-Scanning-Mikroskop mit geeigneten Filtern oder Lasern ausgestattet. Wir verwendeten eine Leica TCS-Einheit (HCX PL APO/63x; NA 1,2). Die Daten können mit einer Software wie Imaris oder AMIRA analysiert werden.
- Slides in antifadent eingebettet werden bei +4 ° C gelagert werden (nicht einfrieren) für bis zu 7 Tage vor der Sonde-verliehenen Fluoreszenz beginnt zu sinken. Alternativ kann die antifadent mit ddH 2 O entfernt werden, und die getrockneten Objektträger können bei -20 ° C über einen längeren Zeitraum gelagert werden.
6. Repräsentative Ergebnisse:
Scraping Biofilm aus festsitzenden kieferorthopädischen Apparaturen (Abbildung 5) ergibt geeignet Flocken (Abb. 6), die direkt auf beschichtete Objektträger für die Mikroskopie hybridisiert werden können. Auf diese Weise können verschiedene Gruppen von orobiome Bakterien in ihrer natürlichen dreidimensionalen Umgebung von Tagging bakterielle rRNA mit unterschiedlich markierten Sonden (Abb. 7 und 8) identifiziert werden. In Abbildung 7 wurde Biofilm mit EUBmix (grün, alle Bakterien) und LGCmix (gelb, Firmicutes) gefärbt. Firmicutes erscheinen in grün, wie sie mit gelben und blauen Flecken waren. In Abbildung 8 wurde Biofilm mit EUBmix (rot, alle Bakterien), Bac303 (blau, Bacteroidetes) und POGI (gelb, Porphyromonas gingivalis) gefärbt. Porphyromonas gingivalis in gelblich-weiß gezeigt wird, wie alle drei Sonden an die DNA binden und die Überlappung der Farben ergibt sich ein weißes Signal. Morphologischen Unterschiede zwischen Gruppen von Bakterien können auch identifiziert werden (Abb. 9 und 10). Große Cluster von Kokken sind in Abbildung 9, wo Färbung mit EUBmix durchgeführt wurde gezeigt (grün, alle bacteria). Verschiedene Formen von oralen Bakterien wurden visualisiert in Abbildung 10, wo coccoid und fadenförmige Bakterien durch Färbung mit EUBmix (rot) unterschieden werden können. Auch eine typische pilzförmige Struktur des Biofilms können über 3D-Modellierung der CLSM-Daten (Abbildungen 11 und 12) bearbeitet werden Klicken Sie hier, um einen Film von der 3D-Modellierung zu sehen.
Abbildung 1. Fester palatinale Expander in situ.
Abbildung 2. Nola Dry Field System.

Abbildung 3. Sterilisierte Zangen, Handschuhen und Fach.
Abbildung 4. Removed Expander.

Abbildung 5. Abschaben Biofilm mit einem sterilen Skalpell.

Abbildung 6. Resin Flocken direkt auf beschichtete Objektträger hybridisiert.

Abbildung 7 CLSM Bild:. Ausdifferenzierung eines bestimmten bakteriellen Gruppe. 2D-Überlagerung von 3D-CLSM-Stack-Daten. Biofilm gefärbt mit EUBmix (grün, alle Bakterien) und LGCmix (gelb, Firmicutes).

Abbildung 8 CLSM Bild:. Ausdifferenzierung eines bestimmten bakteriellen Gruppe. 2D-Überlagerung von 3D-CLSM-Stack-Daten. Biofilm gefärbt mit EUBmix (rot, alle Bakterien), Bac303 (blau, Bacteroidetes) und POGI (gelb, Porphyromonas gingivalis).
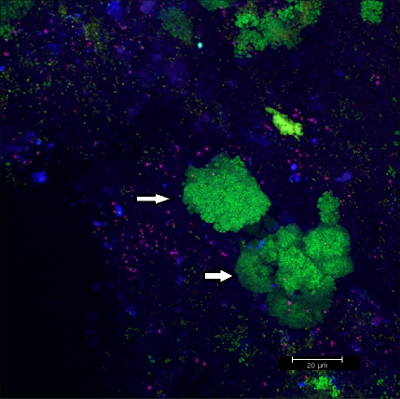
Abbildung 9 CLSM Bild:. Differenzierung von spezifischen Morphologien. 2D-Überlagerung von 3D-CLSM-Stack-Daten. Biofilm gefärbt mit EUBmix (grün, alle Bakterien). Große Cluster von Kokken (Pfeile).
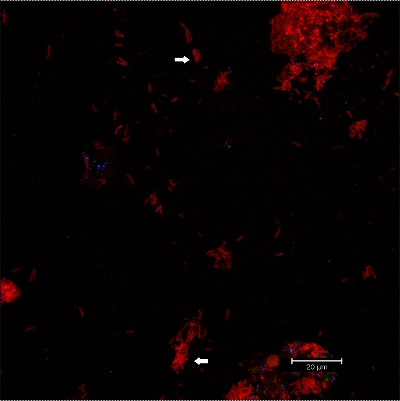
. Abbildung 10 CLSM-Bilder: Differenzierung von spezifischen Morphologien. 2D-Überlagerung von 3D-CLSM-Stack-Daten. Biofilm gefärbt mit EUBmix (rot, alle Bakterien). Kokkoiden (Pfeil unten) und fadenförmige Bakterien (Pfeil oben) unterschieden werden kann.
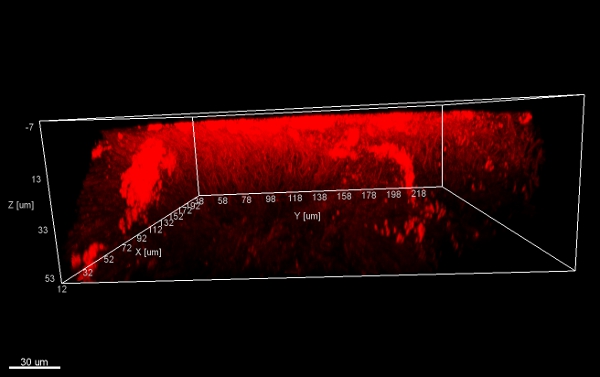
Abbildung 11. Mushroom Struktur, 3D-Ansichten von unten. Stacks von Biofilm-Flocken (abgeschabt der Oberfläche eines kieferorthopädischen Gerätes), mit EUBmix und Verfahren angefärbtssed mit Imaris (CLSM-Bild).

Abbildung 12. Mushroom, 3D Seitenansicht. Stacks von Biofilm-Flocken (abgeschabt der Oberfläche eines kieferorthopädischen Gerätes) mit Imaris (CLSM Bild) verarbeitet.
| Hybridisierungspuffer (200 ul) | |||
| Formamid-Konzentration | 10% | 20% | 45% |
| 5 M NaCl | 36 | 36 | 36 |
| 1 M Tris-HCl | 4 | 4 | 4 |
| 2% SDS | 1 | 1 | 1 |
| FA | 20 | 40 | 90 |
| DdH 2 O | 138 | 118 | 68 |
| Jede FISH-Sonde | 2 | 2 | 2 |
Tabelle 1. Bestandteile des Hybridisierungspuffer (Konzentrationen in ul).
| Waschpuffer (50 ml) | |||
| Formamid-Konzentration | 10% | 20% | 45% |
| 5 M NaCl | 4500 | 2150 | 300 |
| 1 M Tris-HCl | 1000 | 1000 | 1000 |
| 0,5 M EDTA | 0 | 500 | 500 |
| ddH 2 O | 44 500 | 46 350 | 48 200 |
Tabelle 2. Inhaltsstoffe von Wasch-Puffer (Konzentrationen in ul).
Diskussion
Das Protokoll beschrieben ist eine sehr praktikable Ansatz zur Färbung Biofilme aus harten Materialien gesammelt. Sampling und Hybridisierung sind die wichtigsten Schritte. Bei der Probenahme ist darauf zu achten, dass Scheiben von ausreichender Dicke gesammelt werden, um sicherzustellen, dass der Biofilm-Struktur intakt bleibt. Während der Hybridisierung ist es wichtig, starken Temperaturschwankungen zu vermeiden, wodurch die unspezifische Bindung oder Verlust der Bindung, der fluoreszenzmarkierten Sonden. Diese FISH-Protokoll ist eine elegante Methode zur normalen heterogenen Biofilm direkt auf Glas-Objektträger Fleck ohne Unterbrechung seiner Zusammensetzung. Die Folien können sofort für die Mikroskopie, ohne die Notwendigkeit, die Probe erneut verschieben werden. Auf diese Weise ist eine Verbesserung gegenüber Färbung in Röhren von weniger Querkraft wird, um den Biofilm aufgebracht werden. Slices> 50 um dicke vorbereitet und auf diese Weise untersucht werden.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde von der Hygiene Fonds der Medizinischen Universität Graz unterstützt. Alle Probanden gaben ihre Einwilligung. Institutionelle Zustimmung des Studienprotokoll wurde von der Medizinischen Universität Graz erhalten.
Materialien
| Name | Company | Catalog Number | Comments |
| Oligonucleotides | Target organism | Fluorochrome | |
|---|---|---|---|
| EUB338 - 338III | Eubacteria | Cy3 | |
| LGC354 A-C | Firmicutes | FITC | |
| Bac303 | Bacteroidetes | Cy5 | |
| Mut590 | Streptococcus mutans | FITC | |
| POGI | Porphyromonas gingivalis | FITC |
Table 3. Oligonucleotides (refer to probeBase13 for details).
| Component | Company | Comments |
|---|---|---|
| 4% paraformaldehyde | 4% solution in PBS | |
| 1 x PBS | 3 x solution prepared | |
| ddH2O | Millipore | |
| Ethanol (100%) | Merck | |
| Lysozyme | Roth | 100 mg/ml stock solution |
| Formamide | Roth | 99.5% solution |
| 5 M NaCl | Roth | |
| 1 M Tris-HCl | Roth | |
| 2% SDS | Roth | |
| FISH probes (oligonucleotides) | Invitrogen, Sigma | |
| ProLong Gold antifadent | Invitrogen | |
| Nail polish | ||
| Equipment | ||
| Incubator | Thermo Scientific | |
| Water bath | IKA Tez | |
| 50 ml tubes | Sarstedt | |
| Polysine-coated slides | Thermo Scientific |
Table 4. Materials and equipment.
Referenzen
- Sunde, P. T. Fluorescence in situ hybridization (FISH) for direct visualization of bacteria in periapical lesions of asymptomatic root-filled teeth. Microbiology. 149, 1095-1102 (2003).
- Sussman, M., Loya, Y., Fine, M., Rosenberg, E. The marine fireworm Hermodice carunculata is a winter reservoir and spring-summer vector for the coral-bleaching pathogen Vibrio shiloi. Environ. Microbiol. 5, 250-255 (2003).
- Kolenbrander, P. E. Communication among oral bacteria. Microbiol. Mol. Biol. Rev. 66, 486-505 (2002).
- Zijnge, V. Oral biofilm architecture on natural teeth. PLoS One. 5, e9321-e9321 (2010).
- Thurnheer, T., Gmur, R., Giertsen, E., Guggenheim, B. Automated fluorescent in situ hybridization for the specific detection and quantification of oral streptococci in dental plaque. J. Microbiol. Methods. 44, 39-47 (2001).
- Hojo, K., Nagaoka, S., Ohshima, T., Maeda, N. Bacterial interactions in dental biofilm development. J. Dent. Res. 88, 982-990 (2009).
- Jung, D. J. Visualization of initial bacterial colonization on dentine and enamel in situ. J. Microbiol. Methods. 81, 166-174 (2010).
- Al-Ahmad, A. Bacterial colonization of enamel in situ investigated using fluorescence in situ hybridization. J. Med. Microbiol. 58, 1359-1366 (2009).
- Bryers, J. D. Medical biofilms. Biotechnol. Bioeng. 100, 1-18 (2008).
- Cardinale, M., Vieira de Castro, J., Muller, H., Berg, G., Grube, M. In situ analysis of the bacterial community associated with the reindeer lichen Cladonia arbuscula reveals predominance of Alphaproteobacteria. FEMS Microbiol. Ecol. 66, 63-71 (2008).
- Aas, J. A., Paster, B. J., Stokes, L. N., Olsen, I., Dewhirst, F. E. Defining the normal bacterial flora of the oral cavity. J. Clin. Microbiol. 43, 5721-5732 (2005).
- Chin, M. Y., Busscher, H. J., Evans, R., Noar, J., Pratten, J. Early biofilm formation and the effects of antimicrobial agents on orthodontic bonding materials in a parallel plate flow chamber. Eur. J. Orthod. 28, 1-7 (2006).
- Loy, A., Maixner, F., Wagner, M., Horn, M. probeBase--an online resource for rRNA-targeted oligonucleotide probes: new features 2007. Nucleic. Acids. Res. 35, (2007).
- Diaz, P. I. Molecular characterization of subject-specific oral microflora during initial colonization of enamel. Appl. Environ. Microbiol. 72, 2837-2848 (2006).
- Dige, I., Nilsson, H., Kilian, M., Nyvad, B. In situ identification of streptococci and other bacteria in initial dental biofilm by confocal laser scanning microscopy and fluorescence in situ hybridization. Eur. J. Oral Sci. 115, 459-467 (2007).
- Kolenbrander, P. E. Bacterial interactions and successions during plaque development. Periodontology. 42, 47-79 (2000).
- Kolenbrander, P. E., Palmer, R. J., Periasamy, S., Jakubovics, N. S. Oral multispecies biofilm development and the key role of cell-cell distance. Nat. Rev. Microbiol. 8, 471-480 (2010).
- Chalmers, N. I. Use of quantum dot luminescent probes to achieve single-cell resolution of human oral bacteria in biofilms. Appl. Environ. Microbiol. 73, 630-636 (2007).
- Wecke, J. A novel technique for monitoring the development of bacterial biofilms in human periodontal pockets. FEMS Microbiol. Lett. 191, 95-101 (2000).
- Moter, A., Gobel, U. B. Fluorescence in situ hybridization (FISH) for direct visualization of microorganisms. J. Microbiol. Methods. 41, 85-112 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten