Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantitative Magnetresonanztomographie von Skelettmuskelerkrankung
In diesem Artikel
Zusammenfassung
Neuromuskuläre Erkrankungen weisen oft ein zeitlich variierenden, räumlich heterogen und vielschichtige Pathologie. Das Ziel des Protokolls ist es, diese Pathologie unter Verwendung von nicht-invasive Magnetresonanzbildgebungsverfahren zu charakterisieren.
Zusammenfassung
Quantitative magnetic resonance imaging (qMRI) describes the development and use of MRI to quantify physical, chemical, and/or biological properties of living systems. Neuromuscular diseases often exhibit a temporally varying, spatially heterogeneous, and multi-faceted pathology. The goal of this protocol is to characterize this pathology using qMRI methods. The MRI acquisition protocol begins with localizer images (used to locate the position of the body and tissue of interest within the MRI system), quality control measurements of relevant magnetic field distributions, and structural imaging for general anatomical characterization. The qMRI portion of the protocol includes measurements of the longitudinal and transverse relaxation time constants (T1 and T2, respectively). Also acquired are diffusion-tensor MRI data, in which water diffusivity is measured and used to infer pathological processes such as edema. Quantitative magnetization transfer imaging is used to characterize the relative tissue content of macromolecular and free water protons. Lastly, fat-water MRI methods are used to characterize fibro-adipose tissue replacement of muscle. In addition to describing the data acquisition and analysis procedures, this paper also discusses the potential problems associated with these methods, the analysis and interpretation of the data, MRI safety, and strategies for artifact reduction and protocol optimization.
Einleitung
Quantitative Magnetresonanztomographie (qMRI) beschreibt die Entwicklung und Verwendung von MRI zu quantifizieren physikalischen, chemischen und / oder biologischen Eigenschaften von lebenden Systemen. QMRI erfordert, dass man ein biophysikalischen Modell für das System zu übernehmen, von dem Gewebe von Interesse zusammengesetzt und einer MRI-Impulsfolge. Die Impulsfolge ist so konzipiert, um die Bilder "Signalintensitäten auf den Parameter von Interesse in dem Modell zu sensibilisieren. MRI-Signaleigenschaften (Signalgröße, Frequenz und / oder Phase) werden gemessen und analysiert nach dem Modell. Das Ziel ist, eine unvoreingenommene, quantitative Abschätzung eines physikalischen oder biologischen Parameter zu produzieren kontinuierlich verteilt, physikalische Maßeinheiten mit. Oft sind die Gleichungen, die das System beschreiben, werden auf einer Pixel-für-Pixel-Basis analysiert und eingebaut, Erzeugen eines Bildes, dessen Pixelwerte direkt die Werte der Variablen widerspiegeln. Ein solches Bild wird als ein parametrisches Karte.
Eine gemeinsame Nutzung von qMRI ist die development und Anwendung von Biomarkern. Biomarkers kann eine Krankheit Mechanismus zu untersuchen, stellen Sie eine Diagnose verwendet werden, eine Prognose zu bestimmen, und / oder eine therapeutische Reaktion zu beurteilen. Sie können die Form der Konzentrationen oder Aktivitäten von endogener oder exogener Moleküle, eine histologische Probe, eine physikalische Größe, oder ein internes Bild. Einige allgemeine Anforderungen von Biomarkern sind, dass sie objektiv eine kontinuierlich verteilte Variable unter Verwendung physikalischer Maßeinheiten messen; haben eine klare, gut verstanden Beziehung mit der Pathologie von Interesse; sind empfindlich gegenüber Verbesserung und klinischer Zustand verschlechtert; und können mit geeigneten Genauigkeit und Präzision gemessen werden. Nicht-invasive oder minimal-invasive Biomarker sind besonders wünschenswert, da sie den Komfort des Patienten zu fördern und minimal die Pathologie von Interesse stören.
Ein Ziel für die Entwicklung von bildbasierten Biomarker für Muskelerkrankung ist Muskelerkrankung in einer Weise zu reflektieren, die complementar sindy, spezifischer als mehr räumlich selektiver als, und / oder weniger invasiv als die existierenden Ansätze. Ein besonderer Vorteil der qMRI in dieser Hinsicht ist, dass sie das Potential hat, um mehrere Arten von Informationen zu integrieren und somit potentiell viele Aspekte des Krankheitsprozesses charakterisieren. Diese Fähigkeit ist sehr wichtig bei Muskelerkrankungen, die häufig eine räumlich variable komplexe Pathologie aufweisen, die Entzündung, Nekrose und / oder Atrophie mit Fettersatz, Fibrose, Störung des myofilament Gitter ( "Z-Disk-Streaming") und Membranschädigung beinhaltet . Ein weiterer Vorteil der qMRI Methoden ist, dass qualitative oder semi-quantitative Beschreibungen von Kontrast-basierten MR-Bilder spiegeln nicht nur Pathologie, sondern auch Unterschiede in der Bildaufnahmeparameter, die Hardware und die menschliche Wahrnehmung. Ein Beispiel für diese letzte Ausgabe wurde von Wokke et al., Die zeigten , dass semi-quantitative Bewertung der Fettinfiltration sind sehr variabel und häufig falsch, wHenne im Vergleich zu quantitativen Fett / Wasser - MRI (FWMRI) 1.
Das hier beschriebene Protokoll umfasst Pulssequenzen zum Messen der Längs (T 1) und Querrichtung (T 2) Relaxationszeitkonstanten, quantitative Magnetisierungstransfer (QMT) Parameter, Wasserdiffusionskoeffizienten Diffusionstensors MRI (DT-MRI) verwendet und Muskelstruktur unter Verwendung von Strukturbilder und FWMRI. T 1 wird unter Verwendung einer Inversion - Recovery - Sequenz gemessen wird , in dem der Netto - Magnetisierungsvektor invertiert und seine Größe abgetastet wird, da das System zum Gleichgewicht zurückkehrt. T 2 wird durch wiederholtes Refokussierung transversale Magnetisierung unter Verwendung einer Folge von Refokussierungspulsen wie die Carr-Purcell-Meiboom Gill (CPMG) -Methode, und Abtasten der resultierenden Spin-Echos gemessen. T 1 und T 2 können Daten unter Verwendung von nicht-linearen Kurvenanpassungsverfahren analysiert werden , die entweder eine Anzahl von Expone nehmenntial Komponenten a priori (typischerweise zwischen einem und drei) oder durch eine lineare inverse Ansatz , die die beobachteten Daten zu der Summe aus einer großen Anzahl von abklingenden Exponentialfunktionen passt, in einem Spektrum von Signalamplituden führt. Dieser Ansatz erfordert eine nicht negative kleinsten Quadrate (NNLS) Lösung 3 und umfasst in der Regel zusätzliche Regularisierung stabile Ergebnisse zu erzielen. T 1 und T 2 Messungen wurden umfassend zu untersuchen Muskelerkrankungen und Verletzungen 4-9 verwendet. T 1 - Werte werden in der Regel in fett infiltriert Regionen Muskel verringert und erhöht in entzündeten Bereichen 4-6; T 2 -Werte werden in beiden fett infiltriert und entzündete Regionen erhöht 10.
QMT-MRI charakterisiert das freie Wasser und feststoffartigen hochmolekularen Protonen Pools in Gewebe, indem das Verhältnis von hochmolekularen zu freien Wasserprotonen Schätzung (die Pool-Größe-Verhältnis, PSR); die intrinsische entspannenation Raten dieser Pools; und die Wechselkurse zwischen ihnen. Gemeinsame QMT Ansätze umfassen gepulste Sättigung 11 und selektive Inversion - Recovery 12,13 Methoden. Das Protokoll unten beschreibt die Verwendung des gepulsten Sättigungs Ansatz, der die breite Linienbreite des hochmolekularen Protonensignals, bezogen auf die schmale Linienbreite des Wasserprotonensignals ausnutzt. Durch die Sättigung des hochmolekularen Signal bei Resonanzfrequenzen ausreichend verschieden von dem Wassersignal, wird das Wassersignal als Folge der Magnetisierungstransfer zwischen den festen und freien Wasserprotonen Pools reduziert. Die Daten werden mit Hilfe eines quantitativen biophysikalischen Modells analysiert. QMT wurde bei gesunden Muskeln 14,15, und eine kürzlich abstrakt erschien beschreibt seine Umsetzung in Muskelerkrankung 16 entwickelt und angewendet. QMT wurde verwendet , um kleine Tiermodelle von Muskelentzündung zu untersuchen, wobei es , dass eine Entzündung des PSR gezeigt wurde , 17 verringert. Da MTsowohl makromolekularen und Wassergehalte reflektiert, kann MT - Daten auch 18,19 Fibrose reflektieren.
DT-MRI verwendet, um das anisotrope Diffusionsverhalten von Wassermolekülen in Geweben mit geordneten, länglichen Zellen zu quantifizieren. In DT-MRT, Wasserdiffusion in sechs oder mehr unterschiedlichen Richtungen gemessen; Diese Signale werden dann einem Tensor Modell 20 ausgestattet. Der Diffusions - Tensor - D wird diagonalisiert drei Eigenwerte zu erhalten (das sind die drei Haupt Diffusivitäten) und drei Eigenvektoren (die die Richtungen entsprechend den drei Diffusionskoeffizienten angeben). Diese und andere quantitative Indizes abgeleitet von D liefern Informationen über Gewebestruktur und die Orientierung auf einer mikroskopischen Ebene. Die Diffusionseigenschaften der Muskeln, insbesondere der dritte Eigenwert von D und der Grad der Diffusions Anisotropie reflektieren Muskelentzündung 17 und Muskelschäden aufgrund experimenteller Schädigung 21, Strain Injury 22 und Krankheit 23,24. Andere mögliche Einflüsse auf die Diffusionseigenschaften des Muskels gehören unter anderem Veränderungen in der Zelldurchmesser 25 und Membran - Permeabilität ändert.
Schließlich Muskelatrophie, ohne oder ohne makroskopische Fettinfiltration, ist eine pathologische Komponente vieler Muskelerkrankungen. Muskelschwund kann mit Strukturbilder ausgewertet werden Muskelquerschnittsfläche oder das Volumen und FW-MRI zu messen Fettinfiltration zu bewerten. Fettinfiltration kann qualitativ in T 1 beschrieben - und T 2 -gewichteten Bilder 26, aber Fett und Wasser Signale werden am besten gemessen durch Erzeugen von Bildern, das die verschiedenen Resonanzfrequenzen von Fett und Wasserprotonen 27-29 auszunutzen. Quantitative Fett / Wasser - Bildgebungsverfahren wurden in Muskelerkrankungen wie Muskeldystrophie 1,30,31, angewendet und der Verlust der Gehfähigkeit in diesen 31 Patienten vorhersagen kann.
Die hier beschriebene Protokoll verwendet qMRI all dieser Messungen Muskelzustand in den Autoimmunentzündungs Myopathien Dermatomyositis (DM) und Polymyositis (PM) zu charakterisieren. Weitere Einzelheiten des Protokolls, einschließlich der Reproduzierbarkeit wurden zuvor 32 veröffentlicht. Das Protokoll umfasst Standard-Pulssequenzen sowie Radiofrequenz (RF) und Magnetfeldgradienten Objekte speziell auf unseren Systemen programmiert. Die Autoren gehen davon aus, dass das Protokoll in anderen neuromuskulären Erkrankungen, die durch Muskelschwund, Entzündungen gekennzeichnet, anwendbar ist, und Fettinfiltration (wie die Muskeldystrophien).Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Der Leser wird darauf hingewiesen, dass alle Forschung am Menschen muß von der lokalen für die Nutzung von Human Subjects in Forschung Institutional Review Board (IRB) genehmigt werden. Forschung Teilnehmer müssen der Zweck, Verfahren, Risiken informiert werden und Nutzen der vorgeschlagenen Forschung; die Verfügbarkeit von alternativen Behandlungen oder Verfahren; die Verfügbarkeit der Vergütung; und ihre Rechte auf Privatsphäre und ihre Zustimmung zu entziehen und beenden ihre Teilnahme. Vor der MRI-Test-Session, ein Ermittler muss eine potenzielle Studienteilnehmer mit einem IRB-genehmigten informierte Zustimmung Dokument (ICD) präsentieren, erklären deren Inhalt, und fragen Sie den potenziellen Forschungsteilnehmer, wenn er / sie an der Studie teilnehmen möchte. Wenn ja, wird der Teilnehmer müssen unterschreiben und datieren die ICD vor hier einer der Schritte des Protokolls zum Abschluss.
1. Maßnahmen vor dem Testtag
- Beschränken Lebensgewohnheiten , die die D Confound Könnteata
- Weisen Sie den Teilnehmer nicht moderate oder schwere Übung während der 48 Stunden vor dem Test durchzuführen. Weisen Sie den Teilnehmer zu enthalten Over-the-counter Medikamente und Alkoholkonsum während der 24 h vor dem Test. Weisen Sie den Teilnehmer auf, keine Tabakkonsum oder Koffeinkonsum während der 6 Stunden vor dem Test.
- Vor der Prüfung bestätigen, dass der Teilnehmer mit diesen Anweisungen konform war.
- Bereiten Sie das MRT - System
- Sicherstellen , dass die Verfügbarkeit aller notwendigen Geräte, wie sie in der Tabelle der Materialien und Geräte aufgeführt.
- Definieren Sie ein MRI-Protokoll; 5 - vorgeschlagenen Parameter sind in den Tabellen 1 gefunden.
2. Testtag: Bereiten Sie sich für MRI Datenerfassung
- Conduct Sicherheit Screening
- Bildschirm für mögliche Gefahren in der MRT-Umgebung durch eine MRI-Sicherheit-t mitgeregnet Arbeiter die Forschungsteilnehmer mit einem geeigneten MRI-Sicherheit Form, wie sie bei www.mrisafety.com gefunden präsentieren Gesundheitswesen.
- Wenn es irgendwelche implantierten magnetischen oder magnetisch empfindlichen Gegenständen, sicherzustellen, dass sie für die MRI-Scan-sicher sind.
- Bereiten Sie das MRT - System
- Stellen Sie sicher, dass das gesamte Personal entfernt alle magnetischen und magnetisch empfindlichen Objekte vor Betreten des Raumes, die das MRT-System untergebracht ist. Führen Sie jedes Mal, dies zu überprüfen, dass jemand das MRI Raum betritt.
- Bereiten Sie die MRI-System durch die Empfangsspule auf der Patientenliege des MRI-Systems setzt. Auch legen Sie eine Matratze mit Laken und Kissen mit Kissenbezug auf dem Bett. Haben Riemen rund um die Oberschenkel und Polstern oder Kissen unter dem Knie zu legen.
- Starten Sie die Software-Schnittstelle, geben Sie Patientendaten, und öffnen Sie das Bildgebungsprotokoll.
- Positionieren Sie den Forschungsteilnehmer auf dem MRI - Scanner Tabelle
- Beachten Sie die Forschungsteilnehmer, wie er / sie für magnetisch empfindliche Objekte seiner / ihrer Person und Kleidung überprüft. Sichern Sie diese Objekte außerhalb des MRT-Raums in einem verschließbaren Behälter. Geben Sie den MRT-Raum mit dem Forschungsteilnehmer unmittelbar nach Abschluss dieses Schritts.
- Positionieren Sie den Teilnehmer auf der Patientenliege in Rücken, Füße-erste Position. Platzieren Sie den Körperteil zu werden, so nahe an der Mittellinie des Tisches als praktisch abgebildet. Platz Kissen oder Kissen unter den Knien zurück Zugentlastung für den unteren zu bieten und ein Kissen unter den Kopf legen. Zur Begrenzung der Bewegung, sanft, aber effektiv sichern Sie die Oberschenkel, Bein und Fuß und sorgen dafür, dass die Teilnehmer bequem ist.
- Legen Sie die HF-Empfängerspule um den Teilnehmer Oberschenkel und verbinden Sie es mit dem MRI-System.
- Weisen Sie den Teilnehmer und die komplette Finale Pre-Prüfschritte
- Geben Sie Anweisungen, wie Sie mit den Ermittlern zu kommunizieren. Geben Sie die participant mit Gehörschutz und eine Signaleinrichtung, die verwendet werden können, um darauf aufmerksam zu machen, wenn nötig. Weisen Sie den Teilnehmer von der Notwendigkeit zu bleiben noch während und zwischen allen Bildgebungssequenzen.
- Vorzurücken die Patientenliege in den MRI-Scanner, so dass der Körperteil abgebildet werden, um die Mitte des MRI-Scanner ausgerichtet ist.
- die MRT-Raum Nach dem Verlassen, bestätigen, dass der Patient Kommunikationssystem arbeitet und sehen, dass die Teilnehmer bequem ist. Während des gesamten Protokoll kommunizieren regelmäßig mit dem Teilnehmer seine / ihre Komfort und die Einhaltung der Anweisungen zu gewährleisten.
3. Tag der Prüfung: Erwerben Sie die MRT-Daten
- Vorbereitende Schritte
- Da die MRI - System ermittelt die Instrumentaleinstellungen und Kalibrierungen vor jeder Bildgebungssequenz (Mittenfrequenz, Empfängerverstärkungskalibrierung, etc.), diese Prozesse zu überwachen und sicherzustellen , dass jeder Schritt durchgeführt wird correctly.
- eine geeignete Software-Schnittstelle, eine Reihe von Lokalisierungsbildern (auch als Pilot oder Scout-Bilder bekannt ist) zu erwerben; Verwendung vorgeschlagenen Parameter in Tabelle 2.
- Bestimmen Sie, wo die zentrale Scheibe zu platzieren für qMRI Datenerfassungen durch Bereiche mit Schäden zu identifizieren und / oder durch die Schichtposition in Bezug auf reproduzierbare anatomische Orientierungspunkte zu verweisen.
- Sende- und Empfangsspule Kalibrierung Schritte
- Für diese Schritte sowie alle nachfolgenden Abbildungsstufen definieren Bereich der Anatomie , in denen die Homogenität des statischen Magnetfelds (B 0) zu optimieren, ein Verfahren , bekannt als "Shim". Siehe Abbildung 1A für die typische Anordnung der Shim - Volumen von Interesse (VOI) in den vorliegenden Studien verwendet.
- Wenn der MRI-Scanner ein Multi-Element-Übertragungsspule hat, ein RF-Kalibrierungsdatensatzes zu erwerben.
- Wenn der MRI-Scanner ein Multi-Element-Empfangsspule hat, zu erwerbeneine räumliche Empfindlichkeit Karte der Spulen.
- Erwerben Strukturelle MRT - Daten
- Erwerben Sie hohe Auflösung, Multi-Slice, T 1 -gewichtete Bilder , die eine schnelle Spin-Echo (FSE) Sequenz verwendet wird ; die Abbildungsparameter in den vorliegenden Untersuchungen sind in Tabelle 1 bereitgestellt.
- Erwerben Sie hohe Auflösung, Multi-Slice, T 2 -gewichteten Bilder eine FSE - Sequenz verwendet wird ; die Abbildungsparameter in den vorliegenden Untersuchungen sind in Tabelle 2 bereitgestellt.
- Erfassung von Daten für die Qualitätskontrolle in Echtzeit und machen Nachbearbeitung Korrekturen
- Erwerben dreidimensionalen (3D) mehrere Gradienten-Echo - Daten für die Berechnung des B 0 -Feldes Karten. Die Abbildungsparameter in den vorliegenden Untersuchungen sind in Tabelle 3 bereitgestellt.
- Untersuchen Sie die Feldkarten, um sicherzustellen, dass es keine Abweichungen von mehr als ± 60 Hz (ca. 0,5Teile pro Million bei 3 Tesla) über das Bild. Wenn es, nehmen einen alternativen Ansatz zur Shim (verschiedene Verfahren, unterschiedliche Platzierung von VOI, etc.).
- Erwerben 3D-Daten für die Berechnung der Nutationswinkel Karten. Die Abbildungsparameter in den vorliegenden Untersuchungen sind in Tabelle 2 bereitgestellt.
- Prüfen die Feldkarten, um sicherzustellen, dass es keine Bereiche gibt, die stark von der Nenn Nutationswinkel abweichen. Für die HF-Impulse, die in diesem Protokoll verwendet werden, Abweichungen von mehr als ± 30% der Nenn Nutationswinkel betrachtet übertrieben.
- Erwerben Sie die qMRI Daten
- Erwerben 3D - Bilder für die Berechnung der T 1, eine Inversion - Recovery - Sequenz verwendet wird . Die Abbildungsparameter in den vorliegenden Untersuchungen sind in Tabelle 3 dargestellt.
- Wiederholen Sie den T 1 Messung in Gegenwart von Fettsignalunterdrückung (FS; dieser Parameter abbreviated T 1, FS).
- Erwerben Einzelschichtbilder für die Berechnung der T 2, eine Mehrfach - Spin-Echosequenz verwendet wird . Verwenden Sie die Bildparameter , die in Tabelle 3.
- Wiederholen Sie den T 2 Messung in Gegenwart von FS (T 2, FS).
- Erwerben 3D - Bilder für die Berechnung der Parameter QMT, eine gepulste Sättigungssequenz mit FS und die Abbildungsparameter in Tabelle 4 verwendet wird .
- Erwerben Mehrschichtdaten zur Berechnung von Diffusionsparametern tensor, eine Reihe von diffusionsgewichteten Bildern verwenden. Die Abbildungsparameter in dieser Untersuchungen sind in Tabelle 4 angegeben.
- Erwerben Sie 3D-Daten für die Berechnung von Fett / Wasser-Bilder, eine Serie von sechs GRE-Bilder verwenden. Die Abbildungsparameter in dieser Untersuchungen sind in Tabelle 5 angegeben.
- Nach dem qMRI Protokoll abschließen
- Sicher gehen, dassAlle Bilder sind von geeigneter Qualität, indem sie auf potentiell korrigierbaren Artefakte und durch Messen des ausreichenden Signal-zu-Rausch-Verhältnis zu untersuchen.
- Für jeden qMRI Datensatz mehrere Regionen von Interesse (ROIs) in der Bildserie definieren und das Signal in Abhängigkeit von dem jeweiligen Parameter prüfen (zum Beispiel für die T 1 -abhängigen erfassten Daten in den Schritten 3.5.1 und 3.5.2, plot des Signals in Abhängigkeit von TI und sicherzustellen, dass die Daten, die die Inversion-Recovery-Funktion unten aufgeführten in Schritt 4.1.2) folgen.
- eine persönliche Screening für magnetisch empfindliche Objekte Nach Abschluss geben Sie den MRT-Raum. Entfernen Sie den Teilnehmer aus dem Magneten, entfernen Sie alle Gurte und Polster, und die Teilnehmer unterstützen, die MRI-Scanner austritt und die MRT-Raum.
- Übertragen Sie die Daten unter Verwendung von Methoden konform mit den lokalen Gesundheitsdatenschutzgesetze, zu einer lokalen Arbeitsstation für die Verarbeitung; Daten können als Digital Imaging Kommunikation in der Medizin (DICOM-Dateien) oder im Kreditoren exportiert werden'S proprietäres Format (das Verfahren in diesem Protokoll verwendet).
4. Analysieren Sie den qMRI Daten
- Berechnen Sie die Parametric Karten
- Verwenden Sie ein Computerprogramm für das wissenschaftliche Rechnen und Bildanalyse. Durch die Untersuchung eines Histogramms der Signalintensitäten in dem Bild, bilden einen Signalschwellenbasierten Bildmaske, die Bereiche des Signals von Bereichen Rausch abgrenzt. Führen Sie die folgenden Schritte für jedes Pixel in den Signalanteile der Bilder.
- Analysieren Sie den T - 1 - Daten durch die Signalintensität S für jede Inversionszeit (TI) zu messen. Dann passen Sie die Werte für S zu einer Inversion-Recovery mit reduzierter Pre-Delay - Modell:
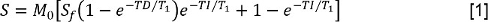
wobei M 0 eine Signalintensität , die die Magnetisierung in dem Gleichgewichtszustand ist, ist S f das Inversionsverhältnis,und TD ist die Pre-Delay - Zeit. Dann passen die Daten mit FS auf dem gleichen Modell, so dass die Bestimmung der longitudinalen Relaxationszeit mit FS, T 1, FS. - Analysieren Sie den T - 2 - Daten von S an jedem TE zu messen. Dann passen die Daten an einen monoexponentiellen Zerfall Modell:

wobei N das Signal an der Basislinie versetzt sind . Der Leser kann auch entscheiden, die Daten an einen multiexponentiellen Modells, wie es unten zu passen:
wobei J die Anzahl der exponentiellen Komponenten und f und T 2, j sind die Signalanteil und T & sub2 ; -Werte , die mit der j - ten Komponente ist. Oder kann das Lesegerät verwenden , um einen nichtnegativen kleinsten Quadrate (NNLS) Methode 3. In der lAtter Fall der Multi-exponentielle Relaxation Analysis (MERA) Toolbox 33 ist frei verfügbar; andere Programme sind ebenfalls verfügbar. Wiederholen Sie diese Analysen für die Daten mit und ohne FS. - Um die QMT Daten zu analysieren, messen S für jede Bestrahlungsleistung und Frequenz - Offset. Korrigieren Sie die nominellen Bestrahlungsleistungen (dargestellt durch ω 1 in der Gleichung unten) mit den Nutationswinkels Karten. Korrektur der Frequenz - Offsets (Δ f in der Gleichung unten) durch den B - 0 Karten unter Verwendung der aufgebrachten Offset - Frequenzen einzustellen. Dann passen die Daten an das folgende Modell 34,35
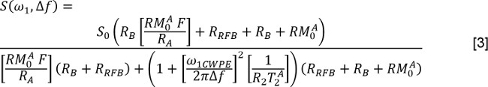
wo aus dem hochmolekularen Pool zur freien Wasserpool der Wechselkurs, der Längsrelaxation Rate des freien Wasserpool ist, ist die Längsrelaxation Rate des hochmolekularen Pool (angenommen 1 s -1) ist das PSR wird die T 2 des freien Wasserpool, und ω 1CWPE ist die mittlere Leistung des Sättigungsimpulses. Die Sättigungsrate der longitudinalen Magnetisierung des hochmolekularen Pool wird von einem Super-Lorentz - Modell beschrieben, wie 34,35 in der Arbeit von Henkelman und Kollegen beschrieben. - Auf die entsprechenden nicht-diffusionsgewichtete Bild Um die DTI - Daten zu analysieren, verwenden Sie zuerst eine affine Transformationsalgorithmus 36 , um jedes diffusionsgewichtete Bild zu registrieren. Dann wird für jedes Pixel messen , um die Werte für S in dem nicht-diffusionsgewichtete Bild und in jeder diffusionsgewichtete Richtung. Bilden eine Matrix der Diffusionskodierung Richtungen zusammen. Mit multivariaten, gewichtet Regression der kleinsten Quadrate, regredieren die Signaldaten auf der Diffusionskodierung Matrix und Form D. Diagonalisieren D und einer Größe Sortierung der Eigenwerte durchführen und ihre Eigenvektoren. Berechnen Sie dann den Mittelwert Diffusität (MD) als:

wobei λ 1, λ 2 und λ 3 die Eigenwerte des Diffusionstensors. Auch die Berechnung der fraktionierte Anisotropie (FA) als:
- Analysieren Sie die FWMRI Daten einen quantitativen Ansatz, der Signale von Wasser und Fett auf Basis der chemischen Verschiebung trennt (wie der FattyRiot Algorithmus, zum kostenlosen Download von https://github.com/welcheb/FattyRiot).
- Definieren Sie Regions of Interest für Analyse
- Geben Sie ROIs auf die anatomischen Bilder (durch die Grenzen der einzelnen Muskeln von Interesse definiert wurden). Ein Beispiel ist in Abbildung 1 dargestellt.
- Ändern Sie die Größe der ROIs der Matrixgröße der qMRI Bilder anzupassen. Bei Bedarf stellen Sie die Ausrichtung der ROIs die qMRI Karte passen (zum Beispiel, wenn der Teilnehmer bewegtzwischen Akquisitionen, könnte eine Übersetzung der ROI Position, um die Muskel Grenzen überlappend) zu vermeiden, erforderlich.
- Untersuchen Sie jeden ROI. Falls notwendig, sicherzustellen, dass keine Pixel enthalten sind, die Teilvolumenartefakte enthalten, nicht-Kontraktions Gewebe und Flussartefakte; bitte Abbildung 1 für Beispiele.
- Berechne den Mittelwert und die Standardabweichung der Werte in qMRI alle Pixel innerhalb der ausgewählten ROIs.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Abbildung 1 zeigt repräsentative axiale anatomische Bilder an der Mitte des Oberschenkels eines Patienten mit Polymyositis erworben. Ebenfalls gezeigt ist die Lage des in-plane Vorsprung des Shimvolumen. Repräsentative Parameterkarten für jede qMRI Methode, die alle aus dem gleichen Patienten erhalten werden , werden aus den Figuren bereitgestellt 2 - 7.
Die 2A
Access restricted. Please log in or start a trial to view this content.
Diskussion
Muskelerkrankungen wie die Muskeldystrophien und idiopathische entzündliche Myopathien bilden der Gruppe von Krankheiten, die in Ätiologie und als individuelle Einheiten, selten in ihrer Häufigkeit heterogen sind. Zum Beispiel, Muskeldystrophie Duchenne - die häufigste Form der Muskeldystrophie - hat eine Inzidenz von 1 zu 3'500 bei männlichen Neugeborenen 37,38; Dermatomyositis, zu dem dieses Protokoll angewendet wurde, hat eine Inzidenz von 1 in 100.000 39. Je höher kollektive Auftreten...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
None of the authors has a financial conflict of interest to report.
Danksagungen
We acknowledge grant support from the National Institutes of Health: NIH/NIAMS R01 AR050101 (BMD), NIH/NIAMS R01 AR057091 (BMD/JHP), NIH/NIBEB K25 EB013659 (RDD), and the Vanderbilt CTSA award RR024975. We also thank the reviewers for the comments and the subject for participating in these studies.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 3T human MRI system | Philips Medical Systems (Best, the Netherlands) | Achieva/Intera | |
| Cardiac phased array receive coil | Philips Medical Systems | ||
| Pillows, straps, bolsters, and other positioning devices | |||
| Computer with MATLAB software | The Mathworks, Inc (Natick, MA) | r. 2014 |
Referenzen
- Wokke, B. H., et al. Comparison of Dixon and T1-weighted MR methods to assess the degree of fat infiltration in duchenne muscular dystrophy patients. J Magn Reson Imaging. 38 (3), 619-624 (2013).
- Carr, H., Purcell, E. Effects of diffusion on free precession in NMR experiments. Phys Rev. 94, 630-638 (1954).
- Whittall, K. P., MacKay, A. L. Quantitative interpretation of NMR relaxation data. Journal of Magnetic Resonance. 84 (1), 134-152 (1989).
- Park, J. H., et al. Dermatomyositis: correlative MR imaging and P-31 MR spectroscopy for quantitative characterization of inflammatory disease. Radiology. 177 (2), 473-479 (1990).
- Park, J. H., et al. Magnetic resonance imaging and p-31 magnetic resonance spectroscopy provide unique quantitative data useful in the longitudinal management of patients with dermatomyositis. Arthritis & Rheumatism. 37 (5), 736-746 (1994).
- Park, J. H., et al. Use of magnetic resonance imaging and p-31 magnetic resonance spectroscopy to detect and quantify muscle dysfunction in the amyopathic and myopathic variants of dermatomyositis. Arthritis & Rheumatism. 38 (1), 68-77 (1995).
- Huang, Y., et al. Quantitative MR relaxometry study of muscle composition and function in Duchenne muscular dystrophy. J Magn Reson Imaging. 4 (1), 59-64 (1994).
- Kim, H. K., et al. T2 mapping in Duchenne muscular dystrophy: distribution of disease activity and correlation with clinical assessments. Radiology. 255 (3), 899-908 (2010).
- Arpan, I., et al. T2 mapping provides multiple approaches for the characterization of muscle involvement in neuromuscular diseases: a cross-sectional study of lower leg muscles in 5-15-year-old boys with Duchenne muscular dystrophy. NMR in Biomedicine. 26 (3), 320-328 (2013).
- Fan, R. H., Does, M. D. Compartmental relaxation and diffusion tensor imaging measurements in vivo in λ-carrageenan-induced edema in rat skeletal muscle. NMR in Biomedicine. 21 (6), 566-573 (2008).
- Sled, J. G., Pike, G. B. Quantitative interpretation of magnetization transfer in spoiled gradient echo MRI sequences. J Magn Reson. 145 (1), 24-36 (2000).
- Gochberg, D. F., Gore, J. C. Quantitative magnetization transfer imaging via selective inversion recovery with short repetition times. Magn Reson Med. 57 (2), 437-441 (2007).
- Li, K., et al. Optimized inversion recovery sequences for quantitative T1 and magnetization transfer imaging. Magn Reson Med. 64 (2), 491-500 (2010).
- Louie, E. A., Gochberg, D. F., Does, M. D., Damon, B. M. Magnetization transfer and T2 measurements of isolated muscle: effect of pH. Magn Reson Med. 61 (3), 560-569 (2009).
- Sinclair, C. D. J., et al. Quantitative magnetization transfer in in vivo healthy human skeletal muscle at 3 T. Magn Reson Med. 64 (6), 1739-1748 (2010).
- Sinclair, C., et al. Multi-parameter quantitation of coincident fat and water skeletal muscle pathology. Proc 21st Ann Meeting ISMRM. , (2013).
- Bryant, N., et al. Multi-parametric MRI characterization of inflammation in murine skeletal muscle. NMR Biomed. 27 (6), 716-725 (2014).
- Aisen, A. M., Doi, K., Swanson, S. D. Detection of liver fibrosis with magnetic cross-relaxation. Magn Reson Med. 31 (5), 551-556 (1994).
- Kim, H., et al. Induced hepatic fibrosis in rats: hepatic steatosis, macromolecule content, perfusion parameters, and their correlations-preliminary MR imaging in rats. Radiology. 247 (3), 696-705 (2008).
- Basser, P. J., Mattiello, J., LeBihan, D. MR diffusion tensor spectroscopy and imaging. Biophys J. 66 (1), 259-267 (1994).
- Heemskerk, A., Strijkers, G., Drost, M., van Bochove, G., Nicolay, K. Skeletal muscle degeneration and regeneration following femoral artery ligation in the mouse: diffusion tensor imaging monitoring. Radiology. 243 (2), 413-421 (2007).
- Zaraiskaya, T., Kumbhare, D., Noseworthy, M. D. Diffusion tensor imaging in evaluation of human skeletal muscle injury. J Magn Reson Imaging. 24 (2), 402-408 (2006).
- Qi, J., Olsen, N. J., Price, R. R., Winston, J. A., Park, J. H. Diffusion-weighted imaging of inflammatory myopathies: polymyositis and dermatomyositis. J Magn Reson Imaging. 27 (1), 212-217 (2008).
- McMillan, A. B., Shi, D., Pratt, S. J., Lovering, R. M. Diffusion tensor MRI to assess damage in healthy and dystrophic skeletal muscle after lengthening contractions. J Biomed Biotech. , (2011).
- Scheel, M., et al. Fiber type characterization in skeletal muscle by diffusion tensor imaging. NMR Biomed. 26 (10), 1220-1224 (2013).
- Kaufman, L. D., Gruber, B. L., Gerstman, D. P., Kaell, A. T. Preliminary observations on the role of magnetic resonance imaging for polymyositis and dermatomyositis. Annalsrheumatic Dis. 46 (8), 569-572 (1987).
- Dixon, W. T. Simple proton spectroscopic imaging. Radiology. 153 (1), 189-194 (1984).
- Glover, G. H. Multipoint Dixon technique for water and fat proton and susceptibility imaging. J Magn Reson Imaging. 1 (5), 521-530 (1991).
- Berglund, J., Kullberg, J. Three-dimensional water/fat separation and T2* estimation based on whole-image optimization--application in breathhold liver imaging at 1.5 T. Magn Reson Med. 67 (6), 1684-1693 (2012).
- Gloor, M., et al. Quantification of fat infiltration in oculopharyngeal muscular dystrophy: Comparison of three MR imaging methods. J Magn Reson Imaging. 33 (1), 203-210 (2011).
- Fischmann, A., et al. Quantitative MRI and loss of free ambulation in Duchenne muscular dystrophy. J Neurol. 260 (4), 969-974 (2013).
- Li, K., et al. Multi-parametric MRI characterization of healthy human thigh muscles at 3.0 T - relaxation, magnetization transfer, fat/water, and diffusion tensor imaging. NMR Biomed. 27 (9), 1070-1084 (2014).
- Does, M. Multi-Exponential Relaxation Analysis (MERA) Toolbox, Version 2. , Available from: http://www.vuiis.vanderbilt.edu/~doesmd/MERA/MERA_Toolbox.html (2014).
- Morrison, C., Stanisz, G., Henkelman, R. M. Modeling magnetization transfer for biological-like systems using a semi-solid pool with a super-Lorentzian lineshape and dipolar reservoir. J Magn Reson Series B. 108 (2), 103-113 (1995).
- Li, J. G., Graham, S. J., Henkelman, R. M. A flexible magnetization transfer line shape derived from tissue experimental data. Magn Reson Med. 37 (6), 866-871 (1997).
- Mangin, J. F., Poupon, C., Clark, C., Le Bihan, D., Bloch, I. Distortion correction and robust tensor estimation for MR diffusion imaging. Med Image Anal. 6 (3), 191-198 (2002).
- Moser, H. Duchenne muscular dystrophy: pathogenetic aspects and genetic prevention. Hum Genet. 66 (1), 17-40 (1984).
- van Essen, A. J., Busch, H. F., te Meerman, G. J., ten Kate, L. P. Birth and population prevalence of Duchenne muscular dystrophy in The Netherlands. Hum Genet. 88 (3), 258-266 (1992).
- Bendewald, M. J., Wetter, D. A., Li, X., Davis, M. P. Incidence of dermatomyositis and clinically amyopathic dermatomyositis: A population-based study in olmsted county, minnesota. Arch Dermatol. 146 (1), 26-30 (2010).
- Carlier, P. G. Global T2 versus water T2 in NMR imaging of fatty infiltrated muscles: different methodology, different information and different implications. Neuromuscul Disord. 24 (5), 390-392 (2014).
- Foley, J. M., Jayaraman, R. C., Prior, B. M., Pivarnik, J. M., Meyer, R. A. MR measurements of muscle damage and adaptation after eccentric exercise. J Appl Physiol. 87 (6), 2311-2318 (1999).
- Garrood, P., et al. MR imaging in Duchenne muscular dystrophy: quantification of T1-weighted signal, contrast uptake, and the effects of exercise. J Magn Reson Imaging. 30 (5), 1130-1138 (2009).
- Bratton, C. B., Hopkins, A. L., Weinberg, J. W. Nuclear magnetic resonance studies of living muscle. Science. 147, 738-739 (1965).
- Fleckenstein, J. L., Canby, R. C., Parkey, R. W., Peshock, R. M. Acute effects of exercise on MR imaging of skeletal muscle in normal volunteers. AJR Am J Roentgenol. 151 (2), 231-237 (1988).
- Williams, S., Heemskerk, A., Welch, E., Damon, B., Park, J. The quantitative effects of inclusion of fat on muscle diffusion tensor MRI measurements. J Magn Reson Imaging. 38 (5), 1292-1297 (2013).
- Hernando, D., et al. Removal of olefinic fat chemical shift artifact in diffusion MRI. Magn Reson Med. 65 (3), 692-701 (2011).
- Willcocks, R. J., et al. Longitudinal measurements of MRI-T2 in boys with Duchenne muscular dystrophy: effects of age and disease progression. Neuromuscul Disord. 24 (5), 393-401 (2014).
- Poon, C. S., Henkelman, R. M. Practical T2 quantitation for clinical applications. J Magn Reson Imaging. 2 (5), 541-553 (1992).
- Does, M. D., Gore, J. C. Complications of nonlinear echo time spacing for measurement of T2. NMR Biomed. 13 (1), 1-7 (2000).
- Poon, C. S., Henkelman, R. M. 180° refocusing pulses which are insensitive to static and radiofrequency field inhomogeneity. J Magn Reson. 99 (1), 45-55 (1992).
- Hollingsworth, K. G., de Sousa, P. L., Straub, V., Carlier, P. G. Towards harmonization of protocols for MRI outcome measures in skeletal muscle studies: consensus recommendations from two TREAT-NMD NMR workshops, 2 May 2010, Stockholm, Sweden, 1-2 October 2009, Paris, France. Neuromuscul Disord. 22, Suppl 2. S54-S67 (2010).
- Underhill, H. R., Rostomily, R. C., Mikheev, A. M., Yuan, C., Yarnykh, V. L. Fast bound pool fraction imaging of the in vivo rat brain: Association with myelin content and validation in the C6 glioma model. Neuroimage. 54 (3), 2052-2065 (2011).
- Smith, S. A., et al. Quantitative magnetization transfer characteristics of the human cervical spinal cord in vivo: application to adrenomyeloneuropathy. Magn Reson Med. 61 (1), 22-27 (2009).
- Li, K. D. R., Dortch, R. D., Gochberg, D. F., Smith, S. A., Damon, B. M., Park, J. H. Quantitative magnetization transfer with fat component in human muscles. Proc. 20th Ann Meeting ISMRM. , (2012).
- Damon, B. M. Effects of image noise in muscle diffusion tensor (DT)-MRI assessed using numerical simulations. Magn Reson Med. 60 (4), 934-944 (2008).
- Damon, B. M., Buck, A. K. W., Ding, Z. Diffusion-tensor MRI-based skeletal muscle fiber tracking. Imaging Med. 3 (6), 675-687 (2011).
- Froeling, M., Nederveen, A. J., Nicolay, K., Strijkers, G. J. DTI of human skeletal muscle: the effects of diffusion encoding parameters, signal-to-noise ratio and T2 on tensor indices and fiber tracts. NMR in Biomedicine. 26 (11), 1339-1352 (2013).
- Basser, P. J., Pajevic, S. Statistical artifacts in diffusion tensor MRI (DT-MRI) caused by background noise. Magn Reson Med. 44 (1), 41-50 (2000).
- Anderson, A. W. Theoretical analysis of the effects of noise on diffusion tensor imaging. Magn Reson Med. 46 (6), 1174-1188 (2001).
- Saupe, N., White, L. M., Stainsby, J., Tomlinson, G., Sussman, M. S. Diffusion tensor imaging and fiber tractography of skeletal muscle: optimization of B value for imaging at 1.5 T. AJR Am J Roentgenol. 192 (6), W282-W290 (2009).
- Levin, D. I., Gilles, B., Madler, B., Pai, D. K. Extracting skeletal muscle fiber fields from noisy diffusion tensor data. Med Image Anal. 15 (3), 340-353 (2011).
- Sinha, U., Sinha, S., Hodgson, J. A., Edgerton, R. V. Human soleus muscle architecture at different ankle joint angles from magnetic resonance diffusion tensor imaging. J Appl Physiol. 110 (3), 807-819 (2011).
- Jones, D. K., Cercignani, M. Twenty-five pitfalls in the analysis of diffusion MRI data. NMR Biomed. 23 (7), 803-820 (2010).
- Hamilton, G., et al. In vivo characterization of the liver fat 1H MR spectrum. NMR Biomed. 24 (7), 784-790 (2011).
- Hernando, D., Kellman, P., Haldar, J. P., Liang, Z. P. Robust water/fat separation in the presence of large field inhomogeneities using a graph cut algorithm. Magn Reson Med. 63 (1), 79-90 (2010).
- Hernando, D., Hines, C. D., Yu, H., Reeder, S. B. Addressing phase errors in fat-water imaging using a mixed magnitude/complex fitting method. Magn Reson Med. 67 (3), 638-644 (2012).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten