Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Planarian Scrunching als quantitatives Behavioral Readout für Noxious Stimuli Sensing
In diesem Artikel
Zusammenfassung
Süßwasserplanarier weisen drei Gangarten (Gleiten, Peristaltik und Scrunching) auf, die sich durch quantitative Verhaltensanalysen unterscheiden. Wir beschreiben eine Methode, um Scrunching mit verschiedenen schädlichen Reizen, Quantifizierung davon und Unterscheidung von Peristaltik und Gleiten zu induzieren. Anhand von Gen Knockdown zeigen wir die Spezifität des Scrunchings als quantitative phänotypische Auslesung.
Zusammenfassung
Süßwasserplanarier gleiten normalerweise sanft durch Ziliarantrieb auf ihrer ventralen Seite. Bestimmte Umgebungsbedingungen können jedoch muskulierungsgetriebene Formen der Fortbewegung induzieren: Peristalsis oder Scrunching. Während peristaltik auf einen Ziliardefekt zurückgeht, ist das Zerkleinern unabhängig von der Zilienfunktion und eine spezifische Reaktion auf bestimmte Reize, einschließlich Amputation, schädliche Temperatur, extremer pH-Wert und Ethanol. So sind diese beiden muskulentgetriebenen Gangarten mechanistisch verschieden. Sie können jedoch qualitativ schwer zu unterscheiden sein. Hier stellen wir ein Protokoll zur Induktion von Scrunching mit verschiedenen physikalischen und chemischen Reizen zur Verfügung. Wir beschreiben die quantitative Charakterisierung des Scrunchings, mit dem man es mit frei verfügbarer Software von Peristaltik und Gleiten unterscheidet. Da Scrunching ein universeller planarischer Gang ist, wenn auch mit charakteristischen artspezifischen Unterschieden, kann dieses Protokoll bei geeigneten Überlegungen allgemein auf alle Arten von Planariern angewendet werden. Um dies zu demonstrieren, vergleichen wir die Reaktion der beiden beliebtesten planarianArten, die in der Verhaltensforschung verwendet werden, Dugesia japonica und Schmidtea mediterranea, mit dem gleichen Satz physikalischer und chemischer Reize. Darüber hinaus ermöglicht die Scrunching-Spezifität die Verwendung dieses Protokolls in Verbindung mit RNA-Interferenzen und/oder pharmakologischer Exposition zur Sezieren der molekularen Ziele und neuronalen Schaltkreise, was potenziell mechanistische Einblicke in wichtige Aspekte der Nozieption und neuromuskulären Kommunikation bietet.
Einleitung
Zusätzlich zu ihrer Popularität für Die Stammzell- und Regenerationsforschung1,2,3, Süßwasserplanarier sind seit langem in Verhaltensstudien verwendet4,5, unter Ausnutzung ihrer vergleichsweise großen (ein paar Millimeter In-Länge), Leichtigkeit und niedrige Kosten der Laborwartung, und breites Spektrum von beobachtbaren Verhaltensweisen. Die Einführung von Computer Vision und automatisiertes Tracking in planarische Verhaltensstudien6,7,8,9,10,11 haben eine quantitative Differenzierung von Verhaltensphänotypen ermöglicht. Tierverhalten ist eine direkte Auslesung der neuronalen Funktion. Da das planarische Nervensystem von mittlerer Größe und Komplexität ist, aber konservierte Schlüsselelemente mit dem Wirbeltier gehirn12,13,14teilen, kann das Studium des planarischen Verhaltens Einblicke in konservierte Mechanismen neuronaler Wirkung geben, die in komplexeren Organismen schwer direkt zu erforschen sind. So sind Planarier ein wertvolles Modell für vergleichende neurobiologische Studien8,12,15,16,17,18,19,20,21. Darüber hinaus ermöglicht die aquatische Umwelt eine schnelle und einfache Exposition gegenüber Chemikalien, um ihre Wirkung auf die Gehirnfunktion bei regenerierenden und erwachsenen Planariern zu untersuchen, was sie zu einem beliebten System für Neurotoxikologie22,23,24,25,26macht.
Die Planarier besitzen drei verschiedene Gangarten, die als Gleiten, Peristalismus und Zerkleinern bezeichnet werden. Jeder Gang wird unter bestimmten Umständen ausgestellt: Gleiten ist der Standard-Gang, Peristaltik tritt auf, wenn Die Ziliarfunktion kompromittiert wird7,27, und Scrunching ist ein Escape-Gang – unabhängig von der Zilienfunktion – als Reaktion auf bestimmte schädliche Reize7. Wir haben gezeigt, dass Scrunching eine spezifische Reaktion ist, die durch das Gefühl bestimmter chemischer oder physikalischer Hinweise, einschließlich extremer Temperaturen oder pH-Wert, mechanischer Verletzungen oder spezifischer chemischer Induktoren, ausgelöst wird, und daher keine allgemeine Stressreaktionist 7,28,29.
Aufgrund seiner Spezifität und stereotypen Parameter, die leicht mit diesem Protokoll quantifiziert werden können, ist Scrunching ein leistungsstarker Verhaltens-Phänotyp, der es Forschern ermöglicht, mechanistische Studien durchzuführen, die sensorische Bahnen und neuronale Kontrolle des Verhaltenssezieren 25,28. Zusätzlich, Scrunching hat sich gezeigt, dass ein empfindlicher Endpunkt zu assay nachteilige chemische Auswirkungen auf die Entwicklung des Nervensystems und Funktion in neurotoxikologischen Studien22,24,25,30. Da mehrere verschiedene Sinneswege zu konvergieren scheinen, um Durchschnaufen durch verschiedene Mechanismen zu induzieren28, scrunching unterscheidet sich von anderen planarischen Verhaltensweisen, weil verschiedene, aber spezifische, Reize verwendet werden können, um verschiedene neuronale Schaltkreise zu sezieren und zu studieren, wie verschiedene Signale integriert sind, um den scrunching Phänotyp zu produzieren.
Wichtig ist, dass es Artenunterschiede gibt, wobei eine Chemikalie bei einer planarienden Art zerkleinern kann, aber eine andere Verhaltensreaktion in einer anderen. Zum Beispiel haben wir festgestellt, dass Anandamid bei der planarischen Art Dugesia japonica Zerkleinerung induziert, aber Peristalsis in Schmidtea mediterranea28induziert. Dieses Beispiel unterstreicht die Bedeutung der zuverlässigen Unterscheidung zwischen den verschiedenen Gangarten, da sie die phänotypischen Manifestationen unterschiedlicher molekularer Mechanismen sind. Die Unterscheidung von Derperistaltik ist jedoch schwierig, wenn man qualitative Beobachtungsdaten verwendet, da beide Gangarten muskulierungsgetrieben sind und qualitative Ähnlichkeiten aufweisen7,28. Daher ist es notwendig, zilienbildende Oder eine quantitative Verhaltensstudie durchzuführen, die eine Unterscheidung anhand der charakteristischen Parameter7,28ermöglicht. Da die Zilien-Bildgebung experimentell anspruchsvoll ist und spezielle Geräte wie ein Hochvergrößerungsmikroskop und eine Hochgeschwindigkeitskamera7,28erfordert, ist sie für Forscher nicht so breit zugänglich wie quantitative Verhaltensanalysen.
Hier stellen wir ein Protokoll für (1) die Induktion von Scrunching unter Verwendung verschiedener physikalischer (noxious temperature, amputation, near-UV light) und chemical (allyl isothiocyanate (AITC), cinnamaldehyd) Reize und (2) die quantitative Analyse des planarischen Verhaltens mit frei verfügbarer Software vor. Durch die Quantifizierung von vier Parametern (Frequenz der Körperlängenschwingungen, relative Geschwindigkeit, maximale Amplitude und Asymmetrie der Körperdehnung und Kontraktion)7kann das Schrumpfen von Gleiten, Peristalismus und anderen in der Literatur berichteten Verhaltenszuständen unterschieden werden, wie schlangenartige Fortbewegung15 oder Epilepsien15. Darüber hinaus, während Scrunching zwischen verschiedenen planrischen Artenkonserviertwird 7 , jede Art hat ihre eigene charakteristische Häufigkeit und Geschwindigkeit; Sobald also die Gleit- und Schimmergeschwindigkeiten einer Art bestimmt sind, kann die Geschwindigkeit allein als Mittel verwendet werden, um das Schrumpfen von Gleiten und Peristalismus29zu unterscheiden. Das Protokoll geht davon aus, dass es keine vorherige Ausbildung in computergestützter Bildanalyse oder Verhaltensstudien gibt und somit auch für planare Verhaltensexperimente in einem Lehrlaborkontext auf Bachelor-Ebene angewendet werden kann. Beispieldaten zur Erleichterung der Protokollanpassung finden Sie im Ergänzungsmaterial.
Protokoll
1. Quantitative planarische Verhaltenstests
- Experimentelle Einrichtung
- Legen Sie ein dimmbares LED-Panel auf eine flache Oberfläche. Das LED-Panel dient zwei Zwecken: (1) einen einheitlichen weißen Hintergrund zu bieten und (2) als einstellbare Lichtquelle zu verwenden, um einen angemessenen Kontrast zu erhalten. Legen Sie eine 100 mm Petrischale Arena auf die LED-Platte.
HINWEIS: Um den Durchsatz zu erhöhen, kann eine Multi-Well-Platte als Arena23,24verwendet werden, aber größere Arenen erleichtern die automatisierte Bildanalyse. - Montieren Sie eine Kamera auf einem Ringständer über der Arena (Abbildung 1A). Passen Sie die Kameraposition, -höhe und -fokussierung nach Bedarf so an, dass die gesamte Arena innerhalb des Sichtfeldes zentriert ist und im Fokus steht (Abbildung 1B).
HINWEIS: Die Auflösung der Kamera muss hoch genug sein, um einen Planarier deutlich von dem homogenen Hintergrund des LED-Panels zu unterscheiden. - Füllen Sie die Arena mit den entsprechenden Belichtungsmedien (planares Wasser oder chemische Lösung) auf ein halbmaximales Volumen (dies wird als Bad bezeichnet). Dies entspricht ca. 25 ml für eine 100 mm Petrischale. Schalten Sie das LED-Panel ein und schalten Sie alle anderen Lichtquellen aus, die sich negativ auf die Aufnahmequalität auswirken können (d. h. Lichtquellen in der Nähe, die eine Blendung auf die Arena erzeugen).
VORSICHT: Verwalten Sie gefährliche chemische Lösungen angemessen, indem Sie die volle persönliche Schutzausrüstung (PSA) tragen und den Versuchsaufbau bei Bedarf auf eine Dunstabzugshaube verschieben. Befolgen Sie die Bundes- und Landesvorschriften zur Abfallentsorgung. - Lassen Sie einen Planarier in Richtung der Mitte der Arena mit einer Transferpipette fallen. Beginnen Sie mit der Aufnahme. Zeichnen Sie Daten als Bildsequenzen im nativenFidschi-31-Format auf (TIFF, GIF, JPEG, PNG, DICOM, BMP, PGM oder FITS; siehe BildanalyseAbschnitt 1.2).
HINWEIS: Da das Verhalten und die Empfindlichkeit gegenüber externen Reizen zwischen den einzelnen Planarien variieren, ist es wichtig, Daten über eine ausreichend große Anzahl biologischer Replikationen zu sammeln, zusätzlich zu technischen Replikationen. Wir haben mit bis zu 10 mittelgroßen (4-7 mm) Planariern in einer 100 mm Petrischale auf einmal gearbeitet. Während die Zeit effizient ist, erschweren mehrere Planarier in der Petrischale gleichzeitig die Datenanalyse, da Planarier Wege kreuzen können.- Zeichnen Sie für Gleitversuche mindestens 1 Frame pro Sekunde (FPS) auf. Für Scrunching/Peristalsis-Experimente, aufzeichnung mit einem FPS, die mindestens doppelt so viel Scrunching / Peristalsis Frequenz der planarian Arten ist. Wenn die planare Art eine unbekannte Scrunching/Peristalistis-Häufigkeit hat, verwenden Sie 10 FPS als Ausgangspunkt und erhöhen/verringern Sie es entsprechend.
- Übertragen Sie bei Verwendung einer chemischen Lösung den Planarier mit möglichst wenigen Tropfen planarem Wasser, damit sich die Konzentration der chemischen Lösung nicht wesentlich ändert.
- Zeichnen Sie für Gleitversuche 1-2 Minuten Gleitverhalten auf. Für Scrunching/Peristalsis-Experimente, aufzeichnung lang genug, um mindestens 3 aufeinander folgende Schwingungen in einer geraden Linie auftreten zu erfassen. Beenden Sie die Aufzeichnung, sobald das Experiment abgeschlossen ist.
ANMERKUNG: Wenn ein Planarier das Beendigungskriterium nicht innerhalb eines festgelegten Zeitraums erfüllt, der über Replikationen hinweg konsistent sein muss und empirisch auf der Grundlage des Stimulus bestimmt wird, beenden Sie die Aufzeichnung und testen Sie einen anderen Planarier.- Wenn der Planarier die Grenze der Arena erreicht, ohne das Beendigungskriterium zu erfüllen, pipette den Planarier zurück in die Mitte der Arena.
HINWEIS: Vermeiden Sie wiederholtePipettieren einer Person für die Aufnahme, da dies ihr Verhalten ändern kann.
- Wenn der Planarier die Grenze der Arena erreicht, ohne das Beendigungskriterium zu erfüllen, pipette den Planarier zurück in die Mitte der Arena.
- Entfernen Sie die Planarier aus der Arena und entsorgen Sie das planare Wasser oder die chemische Lösung in geeigneten Abfallbehältern. Planarier, die sich im planaren Wasser befanden, können in ihren Heimatcontainer zurückgebracht werden.
HINWEIS: Vermeiden Sie Kreuzkontaminationen, indem Sie verschiedene Arenen für verschiedene Medien verwenden (d. h. das Gleiten in planaren Wasserexperimenten sollte nicht in einer Arena durchgeführt werden, die zuvor für Scrunching-/Peristalismusexperimente mit chemischer Exposition verwendet wurde).- Seriell spülen Planarier einer chemischen Lösung in 3 sauberen 100 mm Petrischalen ausgesetzt, die mit 25 ml planarem Wasser gefüllt sind, um chemikalienverdünnend zu verdünnen. Wenn Scrunching oder Peristalsis induziert wurde, legen Sie diese Planarier in einen separaten Behälter. Planarier können nach einem Monat in ihren Heimatcontainer zurückgebracht werden, da die meisten Zellen zu diesem Zeitpunkt umgedreht wären1.
HINWEIS: Wenn mehrere verschiedene Experimente für dieselbe Population von Planariern erforderlich sind, z. B. für eine RNAi-Population, erlauben Sie Planariern, sich 24 Stunden lang zu erholen, bevor sie das nächste Experiment ausführen. Ordnen Sie die Experimente so an, dass das am wenigsten invasive Experiment zuerst und das invasiveexperiment (z.B. Amputation) zuletzt durchgeführt wird. - Wenn Sie mehrere Experimente in der gleichen Arena durchführen, entsorgen Sie die Badlösung ordnungsgemäß und entfernen Sie alle Schleimpfade, indem Sie die Arena mit einem Papiertuch zwischen den Läufen abwischen.
HINWEIS: Das Protokoll kann hier angehalten werden.
- Seriell spülen Planarier einer chemischen Lösung in 3 sauberen 100 mm Petrischalen ausgesetzt, die mit 25 ml planarem Wasser gefüllt sind, um chemikalienverdünnend zu verdünnen. Wenn Scrunching oder Peristalsis induziert wurde, legen Sie diese Planarier in einen separaten Behälter. Planarier können nach einem Monat in ihren Heimatcontainer zurückgebracht werden, da die meisten Zellen zu diesem Zeitpunkt umgedreht wären1.
- Legen Sie ein dimmbares LED-Panel auf eine flache Oberfläche. Das LED-Panel dient zwei Zwecken: (1) einen einheitlichen weißen Hintergrund zu bieten und (2) als einstellbare Lichtquelle zu verwenden, um einen angemessenen Kontrast zu erhalten. Legen Sie eine 100 mm Petrischale Arena auf die LED-Platte.
- Quantitative Analyse des planarischen Verhaltens
- Führen Sie planarische Verhaltenstests durch, wie in Abschnitt 1.1 beschrieben.
- Öffnen Sie die Rohbildsequenz für ein Experiment in Fidschi (Datei > Importieren > Bildsequenz) und wählen Sie das erste Bild in der Bildsequenz aus. Aktivieren Sie im Fenster Sequenzoptionen das Kontrollkästchen "Namen numerisch sortieren" und klicken Sie auf "OK". Nachdem die Bildsequenz geladen wurde, konvertieren Sie die Bildsequenz in 8-Bit(Bild > Typ > 8-Bit) und verwenden Sie das Pfeilwerkzeug oder den Schieberegler am unteren Rand des Bildstapels, um die Bildsequenz zu beobachten oder durchzuschwenken.
HINWEIS: Für Gleitversuche können alle Daten verwendet werden, solange der Planarier während der gesamten Aufzeichnung deutlich zu sehen ist. In der Regel reicht es jedoch aus, die freie Bewegung in der Mitte der Arena zu analysieren, indem die relevanten Teile, wie unten beschrieben, extrahiert werden. - Um einen Zeitraum und eine Interessenregion zu extrahieren, zeichnen Sie einen Interessenbereich, der den vollständigen Pfad eines Planariers mit dem Rechteckwerkzeug umfasst (Abbildung 2A, 2B). Klicken Sie mit der rechten Maustaste auf den Bildstapel und wählen Sie Duplizieren..., aktivieren Sie das Kontrollkästchen für Duplicate Stack, geben Sie den ersten und letzten Frame der Interessenssequenz ein, und klicken Sie auf OK. Wenn mehrere Planarier gleichzeitig abgebildet wurden, wiederholen Sie diese Regionsauswahl und den Duplizierungsschritt für jeden Planarier in der Arena, so dass es so viele offene Bildstapel gibt, wie es Planarier in der Arena gibt. Die folgenden Schritte (Schritte 1.2.4-1.2.10) sollten für jeden Bildstapel nacheinander ausgeführt werden.
- Für Gleitversuche extrahieren Sie eine Zeit des Gleitens, in der sich der Planarier mindestens das Doppelte seiner Körperlänge bewegt.
ANMERKUNG: Je mehr Gleitdaten pro Planarier extrahiert werden, desto zuverlässiger werden die Daten sein. Der Planarier muss sich für die Gleitanalyse nicht in einer geraden Linie bewegen. - Für Scrunching/Peristalistis-Experimente, extrahieren Sie eine Instanz, wenn der Planarier mindestens drei aufeinander folgende (idealerweise mehr) Körperschwingungen in einer geraden Linie durchläuft, um sicherzustellen, dass jede Schwingung ein vollständiger Dehnungs-Kontraktionszyklus ist, da volle Schwingungen notwendig sind, um die Frequenz genau zu bestimmen.
HINWEIS: Je mehr Schwingungen extrahiert werden können, desto zuverlässiger werden die Daten sein. Verwenden Sie keine Sequenzen, in denen sich der Planarier dreht, da diese zu ungenauen Längenmessungen führen.
- Für Gleitversuche extrahieren Sie eine Zeit des Gleitens, in der sich der Planarier mindestens das Doppelte seiner Körperlänge bewegt.
- Wenden Sie einen Schwellenwert auf den duplizierten Bildstapel (Bild > Anpassen > Schwellenwert) an, um das Bild zu binarisieren und den Planarier aus dem Hintergrund zu extrahieren. Passen Sie die Schiebestangen nach Bedarf so an, dass der gesamte Planarier rot hervorgehoben ist. Die genauen Werte hängen von der Bildqualität ab. Lassen Sie die Felder für Dunklen Hintergrund, Stapelhistogrammund Nicht zurücksetzen Bereich unaktiviert. Blättern Sie durch den Bildstapel, um einen guten Schwellenwertbereich sicherzustellen (d. h., der Planarier ist gut vom Hintergrund im gesamten Stapel getrennt), und klicken Sie dann auf Anwenden.
- Legen Sie im Fenster Stapel in Binär konvertieren die Methode auf Standard und den Hintergrund auf Lightfest. Deaktivieren Sie alle Kontrollkästchen in diesem Fenster, und klicken Sie dann auf OK. Es wird ein binarisiertes Bild mit einem schwarzen Planarier auf weißem Hintergrund angezeigt (Abbildung 2C). Stellen Sie sicher, dass der gesamte Planarier in allen Frames der Bildsequenz sichtbar ist.
HINWEIS: Unerwünschte Objekte in der binarisierten Bildsequenz, die kleiner oder größer als der Planarier sind, können in der nachfolgenden Analyse mithilfe eines Größenfilters herausgefiltert werden (Abbildung 2Ciii). - Legen Sie Messungen fest, indem Sie auf Analysieren > Messungen festlegenklicken. Aktivieren Sie die Kontrollkästchen für Bereich, Massenmittelpunkt, Stapelpositionund Ellipse anpassen , und klicken Sie auf OK.
HINWEIS: Diese Parameter müssen nur einmal pro Fidschi-Sitzung festgelegt werden. - Wählen Sie den offenen Bildstapel aus, und wählen Sie Analysieren > Partikel analysierenaus.
- Wählen Sie im Fenster Partikel analysieren die Option Anzeigen > Masken aus, um einen neuen Stapel zu öffnen, der alle Objekte anzeigt, die mit den ausgewählten Parametern erkannt wurden. Dies kann verwendet werden, um visuell zu überprüfen, ob nur Messungen des Planariers durchgeführt werden. In diesem Schritt kann ein Größenfilter eingestellt werden, um unerwünschtes Rauschen zu entfernen, indem der ungefähre Bereich des Planariers (in Pixel2 Einheiten) in den bereitgestellten Raum eingegeben wird. Aktivieren Sie die Kontrollkästchen für Anzeigeergebnisse und Ergebnisse löschen, und klicken Sie auf OK.
HINWEIS: Wenn der Index (erste Spalte) im Ergebnisfenster nicht der Slice-Nummer für alle Zeilen entspricht, bedeutet dies, dass entweder zu viele oder zu wenige Objekte nachverfolgt wurden. Eine Möglichkeit für diese Diskrepanz ist das Vorhandensein anderer Objekte neben dem Planarier oder dass der Planarier nicht in bestimmten Rahmen verfolgt wurde. - Schwenken Sie durch den Maskenbildstapel mit dem Schieberegler am unteren Rand des Bedienfelds. Wenn es Einstellgeräusche gibt oder Frames vorhanden sind, denen ein Planar fehlt, schließen Sie das Ergebnisfenster und den Maskenbildstapel. Wiederholen Sie die Schritte 1.2.7-1.2.8, indem Sie den Flächenfilter so anpassen, dass nur andere Objekte als der Planarier entfernt werden.
HINWEIS: Wenn der Planarier im Rahmen in der Maske fehlt, deutet dies darauf hin, dass die untere Grenze des Flächenfilters zu hoch eingestellt wurde. - Speichern Sie die Daten im Fenster Ergebnisse mit Datei>Speichern unter. Fügen Sie dem Dateinamen die Erweiterung .csv hinzu, um Daten als durch Kommas getrennte Werte zu speichern. Sobald Daten für den Image-Stack gespeichert sind, schließen Sie den entsprechenden Bildstapel und die Fenster Ergebnisse und Masken.
- Importieren Sie Daten und analysieren Sie sie mit einer beliebigen Tabellenkalkulationssoftware oder Freeware. Um die Gleitgeschwindigkeit zu berechnen, lesen Sie Abschnitt 1.3. Um den vollständigen Parametersatz scrunching/peristalsis zu berechnen, lesen Sie Abschnitt 1.4.
HINWEIS: Das Protokoll kann hier angehalten werden. - Um die Pixel- in die tatsächliche Längenkonvertierung zu bestimmen, öffnen Sie ein Bild in Fidschi mit einer Referenzlänge (z. B. dem Durchmesser der Arena). Wählen Sie das Linienwerkzeug aus, und zeichnen Sie eine Linie über die bekannte Länge.
- Konvertieren Sie Pixeleinheiten in eine Standardlängeneinheit, indem Sie auf Analysieren > Skalierung festlegenklicken. Geben Sie die Länge ein, die der auf dem Bild gezeichneten Linie im Feld "Bekannter Abstand" entspricht, und ändern Sie die Längeneinheit von Pixel zu der gewählten Standardlängeneinheit. Der Umrechnungsfaktor wird neben Scalegeschrieben.
HINWEIS: Für Gleit- oder Scrunching/Peristalsis-Analysen in den Abschnitten 1.3 und 1.4 ist kein Pixelkonvertierungswert erforderlich.
- Berechnung der Gleitgeschwindigkeit
- Laden Sie mit der in Abschnitt 1.2 gespeicherten Datendatei die X- und Y-Koordinaten (C000) und die Hauptachsendaten. Wenn die Daten als durch Kommas getrennte Wertedatei gespeichert werden, entsprechen diese Listen den Spalten "XM", "YM" und "Major".
- Berechnen Sie die Verschiebung (d) des planaren Massenmittelpunkts in Pixeln für jeden Frame in Bezug auf den nächsten Frame mit den Datenspalten "XM" und "YM". Die Verdrängung (d) wird gegeben durch:
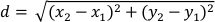
wobei x1 und y1 auf die COM-Koordinaten (XM, YM) eines Frames und x2 und y2 auf die COM-Koordinaten (XM, YM) des nachfolgenden Frames verweisen. - Legen Sie die planare Körperlänge als95. Perzentil der Spalte "Major" fest. Da Planarier ein Wandpräferenzverhalten32aufweisen, stellt dies sicher, dass die berechnete planare Körperlänge repräsentativ dafür ist, wann der Planarier verlängert ist24.
- Normalisieren Sie die Verschiebung durch planare Körperlänge, indem Sie die Pixelverschiebungen pro Frame durch die planare Körperlänge (l)dividieren. Normalisierte Verschiebung (dn) wird gegeben durch:
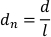
- Generieren Sie eine Liste normalisierter Geschwindigkeiten, indem Sie die normalisierten Verschiebungen durch die zeitweise pro Frame dividieren (inverse des aufgezeichneten FPS). Die normalisierte Gleitgeschwindigkeit (sn) wird gegeben durch:
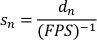
- Berechnen Sie die normalisierte Gleitgeschwindigkeit des Planariers, indem Sie den Durchschnitt der Normalisierungsgeschwindigkeitsliste (sn)verwenden. Die Standardabweichung kann als Unsicherheitsmessung für den Planarier verwendet werden.
- Wiederholen Sie die Schritte 1.3.1-1.3.6 für jeden zu analysierenden Planarier. Durchschnitt und nehmen Sie die Standardabweichung der Gleitgeschwindigkeiten für alle Planarier, um die Gleitgeschwindigkeit und die damit verbundene Unsicherheit für eine planare Bevölkerung zu erhalten.
- Unterscheidung von Scrunching- und Peristalsis-Gangs mit dem vollständigen Parametersatz
- Laden Sie die Hauptachsendatenliste aus der in Abschnitt 1.2 gespeicherten Datendatei. Wenn die Daten als durch Kommas getrennte Wertedatei gespeichert werden, entspricht dies der Spalte Major.
- Erstellen Sie eine Liste, die jeden Datenpunkt in der Spalte "Major" nummert, beginnend mit 0. Konvertieren Sie diese Liste in die pro Frame verstrichene Zeit, indem Sie sie durch das aufgezeichnete FPS dividieren.
- Zeichnen Sie die Hauptspaltendaten in Bezug auf die verstrichene Zeit, um ein Scrunching/Peristalsis-Oszillationsdiagramm zu erzeugen (Abbildung 3A). Mit dem Schwingungsdiagramm schneiden Sie die Daten auf mindestens drei aufeinander folgende, gerade Schwingungen ab (Abbildung 3Bi). Trimmen Sie die Daten, um bei lokalen Spitzen (maximale Dehnung der Schwingung) oder Trog (minimale Dehnung der Schwingung) zu beginnen und zu enden.
ANMERKUNG: Wenn lokale Extrema nicht annähernd gleich sind (Spitzen/Tröge unterscheiden sich dramatisch in den Höhen), deutet dies darauf hin, dass die Schwingungen nicht gerade sind (Abbildung 3Bii). Extrahieren Sie eine weitere Sequenz von mindestens drei aufeinanderfolgenden, geraden Schwingungen. Siehe Abschnitt 1.2. - Bestätigen Sie, dass die Schwingungssequenz des Interesses ordnungsgemäß extrahiert und getrimmt wurde, indem Sie die getrimmten Major-Daten in Bezug auf die Zeit neu zeichnen. Verwenden Sie diese zugeschnittene Datenliste für alle nachfolgenden Berechnungen.
- Um die Schwingungsfrequenz zu berechnen (m )m, dividieren Sie die Anzahl der Schwingungen (On) durch die Gesamtzahl der Datenpunkte in der getrimmten Hauptachsendatenliste (N). Multiplizieren Sie FPS mit diesem Wert, um Frequenz in Schwingungen pro Sekunde zu erhalten.
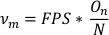
- So berechnen Sie die maximale Dehnung (|| max), subtrahieren sie die absolute minimale Körperlänge (lmin) von der absoluten maximalen Körperlänge (lmax). Normalisieren Sie sich auf eine verlängerte Körperlänge, indem Sie sie durch die absolute maximale Körperlänge dividieren.
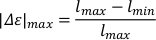
- Um die Geschwindigkeit pro Körperlänge (v*m)zu berechnen, multiplizieren Sie die berechnete maximale Dehnung mit der Schwingungsfrequenz.
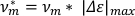
HINWEIS: Geschwindigkeit allein kann verwendet werden, um zwischen Scrunching und Peristalsis Gangarten7zu unterscheiden. - Um den Bruchteil der Zeit zu berechnen, die für die Dehnung aufgewendet wurde (flang), nehmen Sie die Ableitung der getrimmten Hauptachsendatenliste in Bezug auf die Zeit. Teilen Sie die Anzahl der positiven Datenpunkte (d. h. wenn das Derivat >0 (np)ist, durch die Gesamtzahl der Datenpunkte in der Hauptachsendatenliste (nt)).
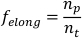
ANMERKUNG: Scrunching Planarians zeigen einen asymmetrischen Bruchteil der Zeit, die er langnzügig aufgewendet hat, während Planarier, die Peristalistis durchführen, gleiche Zeit damit verbringen,7zu verkürzen und zu kontrahieren. - Wiederholen Sie die Schritte 1.4.1-1.4.8 für jeden zu analysierenden Planarier. Berechnen Sie einen planarianpopulation parameter set, indem Sie die durchschnittliche und Standardabweichung jedes Parameters verwenden.
HINWEIS: Der Parametersatz kann verwendet werden, um zu bestimmen, ob das Schwingungsverhalten schrumpft, Peristalsis oder eine andere Form der Fortbewegung mit periodischen Körperformänderungen. Sowohl Scrunching als auch Peristaltik haben feste Parameter für eine bestimmte Art7, wobei die Scrunching-Parameter im Allgemeinen größer sind als die Peristallistenparameter7. Während es möglich ist, dass einer der Parameter außerhalb des artspezifischen Bereichs liegen kann, wie wir zuvor mit chemischer Induktion28beobachtet haben, muss das beobachtete Verhalten mit mindestens 3 von 4 veröffentlichten Parametern übereinstimmen, die entweder als Peristalse oder Scrunching kategorisiert werden müssen.
2. Scrunching Induktion
- Physikalische Reize (schädliche Temperatur, UV-Licht, Amputation)
- Alle physikalischen Reizexperimente finden Sie in Abschnitt 1.1 für den Versuchsaufbau.
HINWEIS: Es ist am besten, eine große Arena, wie eine 100 mm Petrischale, für physikalische Reizexperimente zu verwenden, um mehr Freiraum für das Manövrieren einer Pipette und/oder Rasierklinge zu ermöglichen. - Um das Scrunching über die schädliche Temperatur zu induzieren, wärmen Sie planares Wasser in einem Glasbecher (mindestens 100 l pro zu prüfender Planar) bis 65 °C auf einer Kochplatte.
- Platzieren Sie einen Planarier in der Mitte der Arena. Warten Sie, bis sich der Planarier aufrecht orientiert und zu gleiten beginnt. Beginnen Sie mit der Aufnahme.
- Mit einer P-200 Pipette, langsam Pipette 100 l des 65 °C planaren Wasser spostoral auf das Ende des Planariers zu induzieren.
HINWEIS: Stellen Sie sicher, dass das beheizte planare Wasser bei 65°C bleibt. Erhitzen Sie das Wasser bei Bedarf auf 65 °C, bevor Sie ein weiteres Experiment starten. Da Druck auch Schimmer ncrunching induzieren kann, ist langsames Pipettieren notwendig. Das Pipettier von Raumtemperaturwasser in der gleichen Weise wie im Experiment kann als Steuerungs- und Übungsoption dienen. - Beenden Sie die Aufnahme, sobald das Scrunching beendet wurde. Legen Sie den Planarier in einen Bergungsbehälter und tauschen Sie die Medien in der Petrischale mit frischem, Raumtemperatur planarian Wasser, wenn mehr Experimente laufen.
- Um das Scrunching durch Amputation zu induzieren, überträgt einen Planarier in die Mitte der Arena und wartet, bis sich der Planarier aufrecht orientiert und beginnt zu gleiten. Beginnen Sie mit der Aufnahme.
- Amputieren Sie den Planarier mit einer sauberen Rasierklinge. Amputationen können überall entlang des Planariers durchgeführt werden, solange die Schnittstelle experimenteübergreifend konsistent ist.
HINWEIS: Aus dem vorderen Teil werden Scrunching-Parameter extrahiert. Vermeiden Sie also, die Sicht der Kamera auf diesen Teil des Planariers zu behindern, wenn Sie den Schnitt anwenden, indem Sie sich vom hinteren Ende nähern. Kunststoff-Abdeckungsscheine eignen sich auch gut zum Schneiden und sind eine sicherere Option, insbesondere in einem Unterrichtsumfeld. - Beenden Sie die Aufnahme, sobald das vordere Stück aufgehört hat zu zerkleinern. Entfernen Sie beide Teile, legen Sie sie in einen separaten Behälter und lassen Sie sie für 7 Tage regenerieren. Amputierte Planarier können nach der Regenerierten wieder in den Heimcontainer eingearbeitet werden.
- Amputieren Sie den Planarier mit einer sauberen Rasierklinge. Amputationen können überall entlang des Planariers durchgeführt werden, solange die Schnittstelle experimenteübergreifend konsistent ist.
- Um das Scrunching mit UV-Nahlicht zu induzieren, befestigen Sie geeignete Filter (z. B. Roscolux-Filter) an der Kameralinse, um die Menge an reflektiertem Nah-UV-Licht zu reduzieren, das von der Kamera erfasst wird, und kann die Bildgebung der Reaktion des Planariers beeinträchtigen. Anstatt das LED-Panel zu verwenden, um die Arena von unten zu beleuchten, verwenden Sie die rote Umgebungsbeleuchtung, auf die Planarier unempfindlich sind33.
- Füllen Sie eine 100 mm Petrischale Arena mit planarem Wasser und legen Sie einen einzigen Planarier (5-9 mm) in der Mitte der Arena. Beginnen Sie mit der Aufnahme bei 10 FPS.
- Halten Sie einen Class II UV-Laserpointer (405 ± 10 nm, Ausgangsleistung <5 mW) ca. 30 cm von der Arena entfernt. Positionieren Sie den Laserpointer in einem 45°-Winkel vom gleitenden Planarier und leuchten Sie den Laserpointer dann 5-10 Sekunden lang auf halbem Weg zwischen dem hinteren Ende des Rachens und der Schwanzspitze, um das Schrumpfen zu induzieren.
HINWEIS: Die Leistung des Laserpointers kann mit einem nahezu UV-empfindlichen Leistungsmesser gemessen werden. - Warten Sie, bis der Planarier wieder mit dem Gleiten beginnt, bevor er zwei weitere Stimulationen an derselben Person versucht, um die Reproduzierbarkeit der Reaktion zu testen. Wenn der Planarier weiterhin das gleiche Verhalten zeigt, beenden Sie die Aufnahme und legen Sie den Planarier wieder in seinen Container. Wenn sich das Verhalten zwischen den Stimulationen ändert, zeigen zusätzliche Tests, welche Reaktion am deutlichsten ist.
HINWEIS: Planarier können sich auf nahezu UV-Licht desensibilisieren und nicht mehr reagieren. Konsekutive Stimulationen erfordern eine Ruhezeit von 8-10 Sekunden.
- Alle physikalischen Reizexperimente finden Sie in Abschnitt 1.1 für den Versuchsaufbau.
- Chemischer Stimulus (AITC)
- Um das Scrunching mit einer Chemikalie, z.B. dem TRPA1-Agonisten AITC28,zu induzieren, werden Dieparaten idealerweise in ein Bad der Chemikalie eingetaucht. Gegebenenfalls kann die Pipettenz wie in Abschnitt 2.1.2.3 beschrieben angewendet werden.
VORSICHT: AITC ist entzündlich, akut giftig, kann Haut- und Augenreizungen, Atem- und Hautsensibilisierung verursachen und ist gefährlich für das Wasserleben. AITC-Öl sollte in einer Dunstabzugshaube behandelt werden. Vor der Herstellung von Lagerlösungen von AITC, ziehen Sie entsprechende PSA (Nitrilhandschuhe und eine Laborschicht) und stellen Sie geeignete feste und flüssige Sondermüllentsorgungsbehälter auf. - In einer Dunstabzugshaube, machen Sie eine 10 mM Stammlösung von AITC in planarem Wasser in einem 50 ml Zentrifugenrohr. Diese Lagerlösung ist bis zu einem Monat bei 4°C einsetzbar.
- Aus diesem Bestand eine 25 ml Arbeitslösung von 100 M AITC in planarem Wasser in einem 50 ml Zentrifugenrohr vorbereiten. Diese 100-M-AITC-Lösung wird verwendet, um Scrunching bei Planariern zu induzieren.
ANMERKUNG: 100 M AITC induziert konsistentes Schrumpfen in D. japonica und S. mediterranea planarians28. Für andere Aqua-Planarier können 100 m als Ausgangskonzentration dienen und entsprechend angepasst werden. - Richten Sie den Versuchsaufbau ein (siehe Abschnitt 1.1). Füllen Sie die Arena mit der AITC-Arbeitslösung und legen Sie sie in einen sekundären Behälter. Der sekundäre Behälter sollte mindestens das Doppelte des Volumens der Arena aufnehmen.
HINWEIS: Experimente können in einer Dunstabzugshaube durchgeführt werden, um zusätzliche Sicherheit zu gewährleisten. - Übertragen Sie bis zu 10 Planarier in die Mitte der Arena und beginnen Sie mit der Aufnahme.
- Sobald die Planarier desensibilisiert werden und aufhören zu zerschwirnen, hören Sie auf zu zeichnen. Entfernen Sie die Planarien aus der AITC-Lösung und spülen Sie (siehe Abschnitt 1.1). Entsorgen Sie feste und flüssige AITC-Abfälle in geeigneten Abfallbehältern.
- Überprüfen Sie die Spezifität der Antwort auf AITC mithilfe von RNAi auf TRPA128 gemäß Standardprotokollen.
- Aus diesem Bestand eine 25 ml Arbeitslösung von 100 M AITC in planarem Wasser in einem 50 ml Zentrifugenrohr vorbereiten. Diese 100-M-AITC-Lösung wird verwendet, um Scrunching bei Planariern zu induzieren.
- Um das Scrunching mit einer Chemikalie, z.B. dem TRPA1-Agonisten AITC28,zu induzieren, werden Dieparaten idealerweise in ein Bad der Chemikalie eingetaucht. Gegebenenfalls kann die Pipettenz wie in Abschnitt 2.1.2.3 beschrieben angewendet werden.
Ergebnisse
Die extraokulare Nah-UV-Wahrnehmung bei S. mediterranea planarians ist TRPA1-abhängig und wurde vorgeschlagen, mit H2O2 Release17in Verbindung zu stehen. Da die H2O2-Exposition BEI S. mediterranea und D. japonica planarians28TRPA1-abhängiges Scrunching induziert, können die Schritte in Abschnitt 2.1.4 verwendet werden, um zu testen, ob die UV-nahe Lichtexposition bei beiden Arten zu Einem Scrunching f...
Diskussion
Mit diesem Protokoll kann man die Auswirkungen physikalischer und chemischer Reize7,28,29 oder genetische Manipulation (RNAi)28,29 auf planare Fortbewegung quantitativ untersuchen. Um die räumliche Auflösung zu maximieren, ist es am besten, die Kamera so nah wie möglich an die Arena zu bewegen und gleichzeitig sicherzustellen, dass sich die gesamte Arena im Sichtfeld ...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Die Autoren danken Herrn Tapan Goel für die Kommentare zum Manuskript. Diese Arbeit wurde durch den NSF CAREER Grant 1555109 finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Allyl isothiocyanate, 95% (AITC) | Sigma-Aldrich | 377430-5G | CAUTION: Flammable and acutely toxic; handle in a fume hood with appropriate PPE. |
| Camera lens, 2/3 25mm F/1.4 | Tamron | 23FM25SP | |
| Cell culture plates, 6 well, tissue culture treated | Genesee Scientific | 25-105 | |
| Centrifuge tubes, 50 mL polypropylene, sterile | MedSupply Partners | 62-1019-2 | |
| Cinnamaldehyde, >95% | Sigma-Aldrich | W228613-100G-K | |
| Dimmable A4 LED Tracer Light Box | Amazon | B07HD631RP | |
| Flea3 USB3 camera | FLIR | FL3-U3-13E4M | |
| Heat resistant gloves | Fisher Scientific | 11-394-298 | |
| Hot plate | Fisher Scientific | HP88854200 | |
| Instant Ocean Sea Salt, prepared in deionized water | Instant Ocean | SS15-10 | Prepare in deionized water at 0.5 g/L. |
| Montjüic salts, prepared in Milli-Q water | Sigma-Aldrich | various | Prepare in milli-Q water at 1.6 mM NaCl, 1.0 mM CaCl2, 1.0 mM MgSO4, 0.1 mM MgCl2, 0.1 mM KCl, 1.2 mM NaHCO3; adjust pH to 7.0 with HCl. |
| Petri dishes, 100 mm x 20 mm, sterile polystyrene | Simport | D210-7 | |
| Pipette, 20-200 μL range | Rainin | 17008652 | |
| PYREX 150 mL beaker | Sigma-Aldrich | CLS1000150 | |
| Razor blade, 0.22 mm | VWR | 55411-050 | |
| Roscolux color filter: Golden Amber | Rosco | R21 | Alternatively purchase the Roscolux Designer Color Selector (Musson Theatrical product #SBLUX0306) which includes all 3 color filters together. |
| Roscolux color filter: Medium Red | Rosco | R27 | |
| Roscolux color filter: Storaro Red | Rosco | R2001 | |
| Samco transfer pipette, 62 µL large aperture | Thermo Fisher | 691TS | |
| Support stand | Fisher Scientific | 12-947-976 | |
| Thermometer | VWR | 89095-600 | |
| UV laser pointer | Amazon | B082DGS86R | This is a Class II laser (405nm ±10nm) with output power <5 mW. |
Referenzen
- Rink, J. C. Stem cell systems and regeneration in planaria. Development Genes and Evolution. 223, 67-84 (2013).
- Reddien, P. W., Alvarado, A. S. Fundamentals of Planarian Regeneration. Annual Review of Cell and Developmental Biology. 20, 725-757 (2004).
- Cebrià, F. Regenerating the central nervous system: how easy for planarians. Development Genes and Evolution. 217, 733-748 (2007).
- Pearl, R. Memoirs: The Movements and Reactions of Fresh-Water Planarians: A Study in Animal Behaviour. Journal of Cell Science. , 2-46 (1903).
- Mc Connell, J. . A Manual of Psychological Experimentation on Planarians. , (1967).
- Talbot, J., Schötz, E. M. Quantitative characterization of planarian wild-type behavior as a platform for screening locomotion phenotypes. Journal of Experimental Biology. 214, 1063-1067 (2011).
- Cochet-Escartin, O., Mickolajczk, K. J., Collins, E. M. S. Scrunching: a novel escape gait in planarians. Physical Biology. 12, 055001 (2015).
- Inoue, T., et al. Planarian shows decision-making behavior in response to multiple stimuli by integrative brain function. Zoological Letters. 1, 1-15 (2015).
- Arenas, O. M., et al. Activation of planarian TRPA1 by reactive oxygen species reveals a conserved mechanism for animal nociception. Nature Neuroscience. 20, 1686-1693 (2017).
- Shomrat, T., Levin, M. An automated training paradigm reveals long-term memory in planarians and its persistence through head regeneration. Journal of Experimental Biology. 216, 3799-3810 (2013).
- Blackiston, D., Shomrat, T., Nicolas, C. L., Granata, C., Levin, M. A Second-Generation device for automated training and quantitative behavior analyses of Molecularly-Tractable model organisms. PLoS One. 5, 1-20 (2010).
- Ross, K. G., Currie, K. W., Pearson, B. J., Zayas, R. M. Nervous system development and regeneration in freshwater planarians. Wiley Interdisciplinary Reviews-Developmental Biology. 6, 266 (2017).
- Cebrià, F., et al. The expression of neural-specific genes reveals the structural and molecular complexity of the planarian central nervous system. Mechanisms of Development. , 116-204 (2002).
- Mineta, K., et al. Origin and evolutionary process of the CNS elucidated by comparative genomics analysis of planarian ESTs. Proceedings of the National Academy of Sciences of the United States of America. 100, 7666-7671 (2003).
- Ross, K. G., et al. SoxB1 Activity Regulates Sensory Neuron Regeneration, Maintenance, and Function in Planarians. Developmental Cell. 47, 331-347 (2018).
- Nishimura, K., et al. Reconstruction of Dopaminergic Neural Network and Locomotion Function in Planarian Regenerates. Developmental Neurobiology. 67, 1059-1078 (2007).
- Birkholz, T. R., Beane, W. S. The planarian TRPA1 homolog mediates extraocular behavioral responses to near-ultraviolet light. Journal of Experimental Biology. 220, 2616-2625 (2017).
- Currie, K. W., Molinaro, A. M., Pearson, B. J. Neuronal sources of hedgehog modulate neurogenesis in the adult planarian brain. Elife. 5, (2016).
- Talbot, J. A., Currie, K. W., Pearson, B. J., Collins, E. M. S. Smed-dynA-1 is a planarian nervous system specific dynamin 1 homolog required for normal locomotion. Biology Open. , 1-8 (2014).
- Currie, K. W., Pearson, B. J. Transcription factors lhx1/5-1 and pitx are required for the maintenance and regeneration of serotonergic neurons in planarians. Development. 140, 3577-3588 (2013).
- Hagstrom, D., et al. Planarian cholinesterase: molecular and functional characterization of an evolutionarily ancient enzyme to study organophosphorus pesticide toxicity. Archives of Toxicology. 92, 1161-1176 (2018).
- Hagstrom, D., Cochet-Escartin, O., Collins, E. M. S. Planarian brain regeneration as a model system for developmental neurotoxicology. Regeneration. 3, 65-77 (2016).
- Hagstrom, D., Cochet-Escartin, O., Zhang, S., Khuu, C., Collins, E. M. S. Freshwater planarians as an alternative animal model for neurotoxicology. Toxicological Sciences. 147, 270-285 (2015).
- Zhang, S., Hagstrom, D., Hayes, P., Graham, A., Collins, E. M. S. Multi-behavioral endpoint testing of an 87-chemical compound library in freshwater planarians. Toxicological Sciences. , 26-44 (2019).
- Zhang, S., Hagstrom, D., Siper, N., Behl, M., Collins, E. M. S. Screening for neurotoxic potential of 15 flame retardants using freshwater planarians. Neurotoxicology and Teratology. 73, 54-66 (2019).
- Wu, J. P., Li, M. H. The use of freshwater planarians in environmental toxicology studies: Advantages and potential. Ecotoxicology and Environmental Safety. 161, 45-56 (2018).
- Rompolas, P., Azimzadeh, J., Marshall, W. F., King, S. M. Analysis of ciliary assembly and function in planaria. Methods in Enzymology. 525, 245-264 (2013).
- Sabry, Z., et al. Pharmacological or genetic targeting of Transient Receptor Potential (TRP) channels can disrupt the planarian escape response. PLoS One. , 753244 (2019).
- Cochet-Escartin, O., Carter, J. A., Chakraverti-Wuerthwein, M., Sinha, J., Collins, E. M. S. Slo1 regulates ethanol-induced scrunching in freshwater planarians. Physical Biology. 13, 1-12 (2016).
- Hagstrom, D., Truong, L., Zhang, S., Tanguay, R. L., Collins, E. M. S., et al. Comparative analysis of zebrafish and planarian model systems for developmental neurotoxicity screens using an 87-compound library. Toxicological Sciences. , (2019).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9, 676-682 (2012).
- Akiyama, Y., Agata, K., Inoue, T. Spontaneous Behaviors and Wall-Curvature Lead to Apparent Wall Preference in Planarian. PLoS One. 10, 0142214 (2015).
- Paskin, T. R., Jellies, J., Bacher, J., Beane, W. S. Planarian Phototactic Assay Reveals Differential Behavioral Responses Based on Wavelength. PLoS One. 9, 114708 (2014).
- Petrus, M., et al. A role of TRPA1 in mechanical hyperalgesia is revealed by pharmacological inhibition. Molecular Pain. 3, 40 (2007).
- Takano, T., et al. Regeneration-dependent conditional gene knockdown (Readyknock) in planarian: Demonstration of requirement for Djsnap-25 expression in the brain for negative phototactic behavior. Development, Growth & Differentiation. 49, 383-394 (2007).
- Nishimura, K., et al. Identification of glutamic acid decarboxylase gene and distribution of GABAergic nervous system in the planarian Dugesia japonica. Neuroscience. 153, 1103-1114 (2008).
- Inoue, T., Yamashita, T., Agata, K. Thermosensory signaling by TRPM is processed by brain serotonergic neurons to produce planarian thermotaxis. Journal of Neuroscience. 34, 15701-15714 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten