Method Article
4D-Mikroskopie: Entschlüsselung der Caenorhabditis elegans Embryonalentwicklung mittels Nomarski-Mikroskopie
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Vorbereitung und Befestigung von Caenorhabditis elegans-Embryonen , zur Aufzeichnung der Entwicklung unter einem 4D-Mikroskop und zur Verfolgung der Zelllinie vor.
Zusammenfassung
Die 4D-Mikroskopie ist ein unschätzbares Werkzeug zur Entschlüsselung des embryonalen Entwicklungsprozesses bei verschiedenen Tieren. In den letzten Jahrzehnten hat sich Caenorhabditis elegans zu einem der besten Modelle für das Studium der Entwicklung entwickelt. Aus optischer Sicht machen seine Größe und sein transparenter Körper diesen Fadenwurm zu einem idealen Exemplar für die DIC-Mikroskopie (Differential Interference Contrast oder Nomarski). Dieser Artikel veranschaulicht ein Protokoll für die Züchtung von C. elegans-Nematoden, die Vorbereitung und Montage ihrer Embryonen, die Durchführung von 4D-Mikroskopie und die Verfolgung von Zelllinien . Die Methode basiert auf multifokalen Zeitrafferaufzeichnungen von Nomarski-Bildern und Analysen mit spezifischer Software. Diese Technik zeigt die embryonale Entwicklungsdynamik auf zellulärer Ebene. Jeder embryonale Defekt bei Mutanten, wie Probleme in der Spindelorientierung, Zellmigration, Apoptose oder Zellschicksalsspezifikation, kann effizient erkannt und bewertet werden. Praktisch jede einzelne Zelle des Embryos kann bis zu dem Moment verfolgt werden, in dem sich der Embryo zu bewegen beginnt. Die Verfolgung der vollständigen Zelllinie eines C. elegans-Embryos durch 4D-DIC-Mikroskopie ist mühsam, aber die Verwendung spezifischer Software erleichtert diese Aufgabe erheblich. Darüber hinaus ist diese Technik im Labor einfach zu implementieren. Die 4D-Mikroskopie ist ein vielseitiges Werkzeug und eröffnet die Möglichkeit, eine beispiellose Analyse der Embryonalentwicklung durchzuführen.
Einleitung
Die 4D-Mikroskopie ist ein multifokales Zeitraffer-Aufzeichnungssystem, mit dem Forscher die Zelldynamik einer biologischen Probe sowohl räumlich als auch im Zeitverlauf registrieren und quantifizieren können. Zellkulturen, Hefen oder lebendes Gewebe können einer 4D-Analyse unterzogen werden, aber diese Technik eignet sich besonders für die Analyse der Entwicklung lebender Embryonen. Die Auflösung dieser Analyse erreicht das Niveau jeder einzelnen Zelle des Embryos. Jede Zellteilung kann nachgewiesen werden, und Zellbewegungen können im Laufe der Zeit verfolgt werden. Zellschicksale werden nach der Position und Form bewertet, die Zellen einnehmen. Die Verwendung von Nomarski-Optiken verstärkt den Kontrast von unbefleckten transparenten Proben durch orthogonal polarisierte Lichtstrahlen, die in der Fokusebene interferieren. Die resultierenden Bilder erscheinen dreidimensional, auf einer Seite beleuchtet.
Andere Methoden, die auf der Verwendung von konfokaler Mikroskopie und transgenen GFP-Tieren zum automatischen Nachweis von Kernen und zur Erzeugung von Zelllinien basieren, wurden entwickelt 1,2. Der Vorteil dieser Systeme liegt auf der Hand: Die Software überschreibt die Notwendigkeit, jeden Kern über einen bestimmten Zeitraum manuell zu markieren (obwohl in den späten Phasen eine gewisse manuelle Überwachung erforderlich ist). Zelluläre Prozesse mit Veränderungen der Zellform oder Membrandynamik, wie sie bei Zelldifferenzierung, Migration, Apoptose oder Leichenverschlingung auftreten, bleiben jedoch als schwarzer Hintergrund in den fluoreszierend markierten Kernbildern verborgen.
Im Gegensatz dazu zeigt die 4D-Nomarski-Mikroskopie (auch DIC-Mikroskopie genannt, Differentielle Interferenzkontrastmikroskopie) sowohl Kern- als auch Zellformänderungen, die während der Entwicklung von Wildtyp- oder Mutantentieren auftreten. Dies ermöglicht die Verfolgung von Zelllinien mit Standardmikroskopen, bei denen nur Durchlicht verwendet wird. Es besteht keine allgemeine Notwendigkeit, transgene Tiere zu verwenden, außer um spezifische Expressionsmuster zu zeigen, in diesem Fall können fluoreszierende Scans interkaliert werden. Daher könnte dies der optimale Ansatz für viele Labore sein, die an dynamischen Zellprozessen wie Embryogenese oder Apoptose arbeiten, die unter DIC-Mikroskopie 3,4,5,6,7 hervorgehoben werden können.
Für die Aufnahme mikroskopischer Bilder und die Rekonstruktion von Zelllinien, 3D-Modellen, Zellmigrationspfaden usw. in der aufgezeichneten Probe stehen mehrere flexible und benutzerfreundliche Programme zur Verfügung. In einem Standardexperiment werden Bilder in einer Reihe von Fokusebenen in einem konstanten Abstand aufgenommen, deren Anzahl von der Probendicke abhängt. Die zeitliche Auflösung der Analyse kann durch Erhöhung der Scanfrequenz optimiert werden. Es gibt praktisch keine andere Begrenzung für die Dauer der Aufzeichnung als die Computerspeicherkapazität. Zum Beispiel erfassen wir für eine C. elegans-Embryoentwicklungsanalyse routinemäßig Bilder auf 30 Fokusebenen (jeweils 1 Mikrometer-Schritt), alle 30 Sekunden für 12 Stunden.
Diese Systeme wurden auf die Analyse mehrerer tierischer Embryonen wie Caenorhabditis elegans8,9,10, Drosophila melanogaster11, anderer Nematodenembryonen12,13, Bärtierchen 14,15 und sogar früher Mausembryonen 16 angewendet. Die einzige Voraussetzung ist, einen transparenten Embryo zu haben, der sich auf dem Objektträger unter dem Mikroskop entwickeln kann.
Zusammenfassend ist die DIC-basierte 4D-Mikroskopie besonders nützlich für 1) die Analyse der Embryonalentwicklung von kleinen, transparenten Tieren: Verfolgung der Zelllinie, Zellmigrationspfade, Erstellung von 3D-Modellen usw.; 2) Definition von Genexpressionsmustern; 3) Untersuchung der Zellkulturdynamik, von Hefe bis zu menschlichen Zellen; 4) Analyse der Gewebedynamik oder der Embryonenfragmente; 5) Quantifizierung der Zelltodkinetik und des Leichenverschlings; und 6) Durchführung einer vergleichenden Phylogenieanalyse auf der Grundlage der embryonalen Entwicklungsmerkmale. Wenn Interesse an einem dieser Themen (oder ähnlichen) besteht, kann die 4D-Mikroskopie verwendet werden.
Protokoll
1. C. elegans auf Petrischalen anbauen
- Bereiten Sie NGM-Platten vor und säen Sie sie mit E. coli OP50 als Nahrungsquelle aus (Abbildung 1). Züchten und pflegen Sie C. elegans wie beschrieben17. Die ausgesäten Teller bis zu einem Monat bei 4 °C lagern.
- Stellen Sie die Platten auf die gewünschte Temperatur ein, bevor Sie die Würmer hinzufügen.
- Um die Würmer zu übertragen, entfernen Sie ein Stück Agar von einem alten Teller und legen Sie es auf einen frischen Teller.
- Alternativ können Sie einzelne Tiere mit einem sterilen Wurmpicker (ein 1-Zoll-Stück 32-Gauge-Platindraht mit abgeflachter Spitze, montiert an der Spitze einer Pasteur-Pipette) fangen und auf die neue Platte legen.
- Züchten Sie die Würmer bei der gewünschten Temperatur.
- Züchten Sie C. elegans Würmer bei 20 ° C, der Standardtemperatur. Um jedoch die Entwicklung thermosensitiver Mutanten zu analysieren, führen Sie eine Inkubation über Nacht durch, normalerweise bei 25 ° C. Passen Sie die Dauer und Temperatur dieser Inkubation nach Bedarf an, abhängig von der spezifischen Mutante.
2. Bereiten Sie die 4D-Mikroskopieaufzeichnung vor, bevor Sie die Embryonen montieren (Abbildung 2)
- Richten Sie das Mikroskop und die Temperaturkontrolle ein, bevor Sie den Embryo vorbereiten. C. elegans Embryonen teilen sich sehr schnell. Bereiten Sie sich darauf vor, sofort nach dem Einsetzen der Embryonen mit der Aufzeichnung zu beginnen.
- Stellen Sie die Aufnahmetemperatur entweder auf 15 °C, 20 °C oder 25 °C ein.
- Zeichnen Sie die Embryonen routinemäßig bei 25 ° C auf. Zeichnen Sie thermoempfindliche Mutanten bei der restriktiven Temperatur auf, um ihre Phänotypen zu zeigen. Zeichnen Sie eine WT-Steuerung (wenn sie in einer anderen Vorbereitung durchgeführt wird) bei der gleichen Temperatur wie die Mutanten auf.
HINWEIS: WT-Embryonen entwickeln sich schneller bei 25 °C und langsamer bei 15 °C ohne zusätzliche Unterschiede in der Zelllinie. Stellen Sie Mikroskope in einen temperaturkontrollierten Raum. Eine zusätzliche Steuerung durch Kühlen oder Erwärmen des Objektträgers ist sehr wünschenswert. Dies kann erreicht werden, indem Wasser bei einer bestimmten Temperatur durch einen Metallring um das Mikroskopobjektiv und den Kondensator zirkuliert. Das Objektiv und die Aufbereitung stehen in direktem Kontakt durch das Tauchöl und die Temperaturübertragung ist effizient. Dieses System ermöglicht eine präzise Steuerung der Aufzeichnungstemperatur und die Durchführung von Temperaturverschiebungen während der Embryonalentwicklung.
- Zeichnen Sie die Embryonen routinemäßig bei 25 ° C auf. Zeichnen Sie thermoempfindliche Mutanten bei der restriktiven Temperatur auf, um ihre Phänotypen zu zeigen. Zeichnen Sie eine WT-Steuerung (wenn sie in einer anderen Vorbereitung durchgeführt wird) bei der gleichen Temperatur wie die Mutanten auf.
- Definieren Sie die Aufzeichnungsparameter in der Mikroskopie-Software.
- Für eine Standardaufzeichnung von C. elegans (ohne fluoreszierende Scans) wählen Sie:
Z-Stacks von 30 Fokusebenen in einem Abstand von jeweils 1 Mikrometer.
30-Sekunden-Intervalle zwischen dem Beginn jedes Z-Stacks.
1500 Z-Stacks (12,5 Stunden Aufzeichnung).
HINWEIS: Sowohl kommerzielle als auch Open-Source-Mikroskopsteuerungsprogramme können verwendet werden, um diesen Workflow für die Aufnahme von Bildern zu definieren. Jetzt ist das Mikroskop bereit für die Aufnahme.
- Für eine Standardaufzeichnung von C. elegans (ohne fluoreszierende Scans) wählen Sie:
3. Bereiten Sie die Embryonen vor und montieren Sie sie
- Bereiten Sie ein dünnes, homogenes Agar-Pad als ersten Schritt vor, um ein schönes Bild zu erhalten (Abbildung 3).
- Bereiten Sie 50 ml einer 4,5% igen Agarlösung in entionisiertem Wasser vor. In der Mikrowelle zum Kochen erhitzen und 0,5 – 1 ml in 3 ml Glasröhrchen gießen.
- Verschließen Sie die Tuben vorsichtig mit Wachsfolie, um eine Austrocknung zu vermeiden. Versiegelte Agarröhrchen können bei Raumtemperatur bis zu zwei Monate gelagert werden.
- Auf dem Labortisch befindet sich ein Heizblock bei 80 °C mit:
ein Reagenzglas aus reinem Vaseline (geschmolzen), mit einem feinen Pinsel im Inneren.
ein Reagenzglas mit destilliertem Wasser, das eine Pasteur-Pipette enthält.
HINWEIS: Dies stellt sicher, dass alle erforderlichen Materialien heiß sind und das Agar bei der Herstellung des Pads nicht erstarrt. - Entfernen Sie die Wachsfolie von der Oberseite einer der Agarröhrchen und erhitzen Sie sie vorsichtig über einem Alkoholbrenner, um den Agar zu schmelzen. Seien Sie vorsichtig, da heißer Agar, der aus der Glasröhre ausgestoßen wird, Verbrennungen verursachen kann.
- Sobald das Agar geschmolzen ist, legen Sie das Rohr in den Hitzeblock, um das Agar in flüssiger Form zu halten.
- Alternativ können Sie die Agarröhrchen 1 Stunde vor dem Experiment in den Wärmeblock legen, um sie ohne Brenner zu schmelzen. Das geschmolzene Agar sollte nach einem Tag entsorgt werden.
- Legen Sie einen Objektträger (Objektträger A) zwischen zwei andere auf ein Stück Plastik.
- Nehmen Sie eine weitere Folie (Folie B) und halten Sie sie mit den Fingern in einer Hand.
- Legen Sie dagegen einen kleinen Tropfen geschmolzenen Agar mit der warmen Pasteur-Pipette in die Mitte von Folie A.
- Drücken Sie sofort Folie B auf den Agartropfen, um ein sehr dünnes Pad zwischen den Folien A und B zu erzeugen. Halten Sie diese Folien bis Schritt 3.2.3 zusammen.
- Montiere die Embryonen.
- Sammeln Sie 5-10 gravide Hermaphroditen mit dem Pflücker und legen Sie sie in ein mit Wasser gefülltes Uhrmacherglas.
- Verwenden Sie ein Skalpell, um die Hermaphroditennematoden aufzuschneiden und frühe Eier (1-4 Zellen) aus der Gebärmutter unter dem Stereomikroskop zu extrahieren.
- Nehmen Sie die Folie aus Schritt 3.1.10 und schieben Sie Folie B vorsichtig ab, um das Agar-Pad auf Folie A freizulegen.
- Legen Sie ein frühes Ei in die Mitte des Agar-Pads, indem Sie es mit einem Kapillarrohr pipettieren.
- Alternativ können Sie einen Tropfen mit einer Reihe von Embryonen auf das Agar-Pad pipettieren und dann nach Embryonen im Frühstadium suchen. Führen Sie diesen Schritt unter dem Stereomikroskop durch.
- Wenn nötig, bewegen Sie das Ei, indem Sie es mit einer Wimpern anstoßen, die an das Ende eines Zahnstochers geklebt ist.
- Überschüssiges Wasser mit der Kapillarpipette entfernen.
HINWEIS: Eine Kapillarpipette kann leicht zubereitet werden, indem eine Pasteur-Pipette über einem Alkoholbrenner erhitzt und von beiden Enden gezogen wird. - Decken Sie die Zubereitung vorsichtig mit einem Deckglas ab. Um Luftblasen zu vermeiden, legen Sie eine Kante des Deckglases auf den Objektträger und schieben Sie vorsichtig ein Skalpell entlang der angrenzenden Kante, um das Deckglas langsam und schräg auf die Zubereitung zu drapieren.
- Verwenden Sie eine Pipette, um 3/4 des Raumes, der das Agar-Pad umgibt, mit Wasser zu füllen. Lassen Sie 1/4 des Raumes mit Luft.
- Verschließen Sie das Deckglas mit Vaseline, um eine Austrocknung während langer Aufnahmezeiten zu vermeiden.
- Verwenden Sie die feine Bürste, um eine dünne Schicht geschmolzenes Vaseline um den Rand des Deckglases zu ziehen.
HINWEIS: Jetzt ist die Vorbereitung bereit für die Aufnahme.
4. Stellen Sie den DIC ein und starten Sie die 4D-Mikroskopieaufzeichnung
- Legen Sie den Objektträger auf den Mikroskoptisch. Fokussieren Sie den Embryo mit dem Objektiv mit niedriger Vergrößerung (5x oder 10x).
- Wechseln Sie zum 100-fachen Tauchobjektiv.
- Stellen Sie die optischen Komponenten des Mikroskops ein, um ein Nomarski-Bild zu erhalten.
- Fokussieren Sie den Kondensator.
- Öffnen Sie die Öffnung des Kondensators vollständig und schließen Sie die Feldmembran (dies führt zu einer höheren numerischen Apertur und damit zu einer höheren Auflösung).
- Überprüfen Sie, ob beide Polarisatoren auf dem Mikroskop so ausgerichtet sind, dass sie die maximale Lichtextinktion verursachen.
- Drehen Sie das Wollaston-Prisma, um ein schönes dreidimensionales Bild des Embryos zu erhalten, das auf einer Seite beleuchtet ist. Drehen Sie das Prisma in die andere Richtung, um den Effekt zu erzielen, dass der Embryo auf der anderen Seite beleuchtet wird.
HINWEIS: Diese Schritte können vor der Aufnahme an einem Prüfmuster durchgeführt werden, so dass nur eine Feinabstimmung an der zu analysierenden Probe erforderlich ist.
- Starten Sie die Bildaufnahme im Mikroskop.
5. Analysieren Sie den 4D-Film (Abbildung 4).
HINWEIS: Sobald die Aufzeichnung abgeschlossen ist, verwenden Sie eine Software zur Verfolgung der Zelllinie, um die Zelllinie zu rekonstruieren und zu analysieren.
Cell Lineage Tracing Software ist ein leistungsfähiges Werkzeug für detaillierte Analysen der Embryonalentwicklung oder Dynamik in Zellkulturen oder Gewebefragmenten. Das Programm extrahiert und quantifiziert mehrere Datensätze über die Zelldynamik der Probe, die die Generierung der vollständigen Zelllinie jeder einzelnen aufgezeichneten Zelle umfassen, einschließlich Zellteilungen, Zellzykluslänge, Migration oder Apoptose sowie ihrer Kinetik. Darüber hinaus kann die Zelldifferenzierung durch die morphologischen Veränderungen der Zelle oder durch die Expression spezifischer Marker erreicht werden. Grundsätzlich zeigt der Softwarebildschirm zwei Fenster an: Im linken Fenster kann der 4D-Film vorwärts und rückwärts oder auf und ab bis zur oberen oder unteren Ebene abgespielt werden, so dass jede Zelle während der gesamten Aufnahme in Zeit und Raum verfolgt werden kann. Auf der rechten Witwe wird die Zelllinie erzeugt. Ein Klick auf einen Zellkern im 4D-Film erzeugt einen Punkt im Linienfenster, der die Informationen des Zellnamens, des Schicksals und der räumlichen Koordinaten speichert. Die Zelllinie einer bestimmten Zelle wird erzeugt, indem der 4D-Film vorwärts abgespielt und regelmäßig auf den Kern geklickt wird, um die Mitose dieser bestimmten Zelle im Laufe der Zeit zu markieren. Die Wiederholung dieses Prozesses für jede der aufgezeichneten Zellen erzeugt die vollständige Zelllinie des Embryos oder der Probe. Die gespeicherten Informationen für die räumlichen Koordinaten jeder Zelle werden später verwendet, um 3D-Embryonenmodelle und Zellmigrationspfade zu rekonstruieren.
- Öffnen Sie die Linienverfolgungssoftware und erstellen Sie ein neues Projekt, indem Sie zum oberen Balkenmenü gehen und Folgendes auswählen:
Datei | Neues Projekt. - Wählen Sie je nach Aufzeichnungstemperatur die Zelllinienvorlage aus: DB08 für die Aufzeichnung bei 25 °C, DB10 für die Aufzeichnung bei 20 °C und DB12 für die Aufzeichnung bei 15 °C.
- Stellen Sie die Aufzeichnungsparameter im sich abzeichnenden Fenster ein: Scananzahl (normalerweise 1500), Zeit zwischen den Scans (30 Sekunden), Pegelanzahl (30) und Abstand zwischen den Pegeln (1 Mikrometer).
- Wählen Sie die Bilddatei und das Format aus.
- Wählen Sie das Bildverzeichnis aus, in dem die Bilder gespeichert wurden.
- Wählen Sie, ob Bilder als Einzelbilder (ein Bild pro Level und Zeit) oder als Multi-Image-Z-Stacks gespeichert werden sollen.
- Bestimmen Sie die Dateibenennung und das Bildformat. Routinemäßig werden einzelne Bilder unter den folgenden Namen gespeichert:
X0000L00C1 (für Scan 0, Level 0, Kanal 1)
X0000L01C1 (für Scan 0, Level 1, Kanal 1)
X0000L02C1 (für Scan 0, Level 2, Kanal 1)
...
X0001L00C1 (für Scan 1, Level 0, Kanal 1)
X0001L01C1 (für Scan 1, Level 1, Kanal 1)
...
X0300L04C1 (für Scan 300, Level 4, Kanal 1)
...
HINWEIS: Das Speichern der Bilder in einem komprimierten Format spart Platz auf Ihrer Festplatte.
- Definieren Sie die Lichtkanäle: 1 für DIC-Optiken, 2 für GFP, 3 für RFP usw. Fügen Sie diejenigen hinzu, die in der 4D-Aufnahme verwendet wurden. Klicken Sie auf "Kanalverarbeitung aktiviert", um sie zu erkennen.
- Beginnen Sie, die Zelllinie des Embryos zu verfolgen. Der Bildschirm enthält jetzt zwei Hauptfenster: das Videofenster und das Zellenlinienfenster.
- Wählen Sie im Linienfenster einen Linienzweig aus und klicken Sie mit der Maus auf den Zellkern, der dieser Zelle im Videofenster entspricht.
- Folgen Sie der Zelle räumlich und im Laufe der Zeit, indem Sie den 4D-Film vorwärts, rückwärts oder eine Ebene höher oder unten mit den Cursortasten abspielen.
- Klicken Sie regelmäßig auf den Zellkern. Dadurch wird ein Punkt im Linienzweig erzeugt und die räumlichen Koordinaten der Zelle zu diesem Zeitpunkt registriert. Infolgedessen schreitet die Zelllinie voran und 3D-Rekonstruktionen des Embryos sind möglich.
- Markieren Sie die Mitose, indem Sie auf die Eingabetaste klicken. Wählen Sie dann eine der Tochterzellen aus und folgen Sie ihr wie zuvor.
- Wiederholen Sie den Vorgang (Schritte 5.6.1 bis 5.6.4) für den Rest der embryonalen Zellen, um die vollständige Zelllinie zu verfolgen oder bestimmten Zellen von Interesse zu folgen, z. B. denen, die sich einer Apoptose unterziehen.
- Vergleichen Sie die Mutantenlinie mit der stereotypen WT C. elegans-Zelllinie.
Ergebnisse
Um die embryonale Entwicklung einer C. elegans-Mutante für das Gen gsr-1 zu charakterisieren, das für das Enzym Glutathionreduktase kodiert, das zur Regeneration von reduziertem Glutathion (GSH) erforderlich ist und an der Aufrechterhaltung der Redoxhomöostase im Nematoden beteiligt ist, führten wir eine 4D-Mikroskopie einer gsr-1 (tm3574) Deletionsmutante durch, bei der es sich um einen Funktionsverlust des Allels handelt, der einen frühen embryonalen Arrestphänotyp18 verursacht. Sowohl WT als auch ausgewogene GSR-1 (tm3574) mutierte C. elegans Nematoden wurden auf NGM-Platten gezüchtet, die mit E. coli OP50 als Nahrungsquelle17 ausgesät waren. GSR-1 (TM3574) Würmer wurden zwei Generationen lang bei 20 °C als heterozygot gezüchtet und dann wurden segregierende homozygote Würmer (die dank der mütterlichen Belastung bis ins Erwachsenenalter heranwachsen können) für eine Inkubation über Nacht vor der Embryonenanalyse auf 25 °C verschoben. Wurmplatten wurden in Kartons inkubiert, um Kondensation zu vermeiden (Abbildung 1). Gravide Nematoden wurden aufgeschnitten, um junge Embryonen zu extrahieren.
Um die embryonale Entwicklung der Mutante mit der stereotypen WT unter identischen Bedingungen zu vergleichen, wurden ein WT (als Kontrolle) und ein gsr-1 (tm3574) Embryo nebeneinander auf das gleiche Präparat gelegt. Der 4D-Mikroskopie-Workflow wurde auf einem standardmäßigen motorisierten aufrechten Mikroskop ausgeführt, das mit DIC-Optik ausgestattet war. Die ausgewählten Aufzeichnungsparameter im Mikroskopsteuerungsprogramm waren: Z-Stacks von 30 Fokusebenen in jeweils 1 Mikron Abstand, 30-Sekunden-Intervalle zwischen dem Beginn jedes Z-Stacks und 1500 Z-Stacks (12,5 Stunden Aufnahme). Die Aufnahmetemperatur wurde auf 25 °C eingestellt (sowohl im Raum als auch auf der Mikroskopstufe) (Abbildung 2).
Sobald die Aufzeichnung abgeschlossen war, wurde die Bilddatei geöffnet, und die Zelllinie wurde mit einer Linienverfolgungssoftware rekonstruiert, indem auf die im Videofenster gezeigten Zellkerne geklickt wurde (Abbildung 4). Die nachgezeichnete gsr-1 (tm3574) mutierte embryonale Zelllinie wurde mit der im Hintergrund dargestellten C. elegans WT-Linie verglichen. Ein wesentliches Ergebnis war der Nachweis einer fortschreitenden Verzögerung des Zellzyklus während der Embryonalentwicklung. Infolgedessen blockierten mutierte Embryonen in Zwischenstadien, während WT-Embryonen fortschritten und schließlich als Larven schlüpften.
Die Vorbereitung und direkte Beobachtung von Embryonen unter dem Mikroskop oder die Immunfärbung mit Antikörpern gegen spätembryonale Marker könnte das Vorhandensein eines hohen Prozentsatzes junger Embryonen in der Mutante im Vergleich zum WT zeigen. Der Embryonenarrest könnte dann als die plausibelste Erklärung abgeleitet werden. Der direkte Nachweis und die genaue Quantifizierung der Zellzyklusverzögerung können jedoch nur durch ein 4D-Mikroskopie-Experiment elegant und einfach gezeigt und quantifiziert werden. Andere wichtige Merkmale der Embryonalentwicklung wie Zelldifferenzierung oder Apoptose (Abbildung 5) können ebenfalls dynamisch mit Hilfe der 4D-Mikroskopie visualisiert werden, die eine detaillierte Analyse mehrerer Aspekte der Entwicklung in einem einzigen Experiment ermöglicht.

Abbildung 1: C. elegans Nematoden, die unter Laborbedingungen wachsen. Nematoden werden auf mit E. coli ausgesäten NGM-Platten gezüchtet, in Kartons gelagert und entweder bei 15 °C, 20 °C oder 25 °C inkubiert . Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
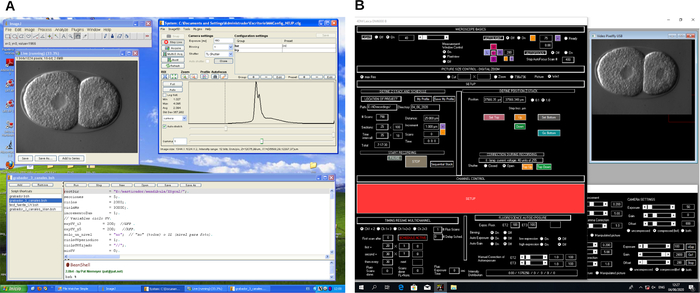
Abbildung 2: Screenshot der 4D-Mikroskopie-Aufnahmesoftware. Beispiel für zwei verschiedene Mikroskopsteuerungssoftwareprogramme (A und B). Diese Programme erstellen Workflows zur Steuerung der Mikroskop- und Bilderfassung während der 4D-Mikroskopieaufnahme. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Serienfotos der Agar-Pad-Vorbereitung und -Montage des C. elegans-Embryos zeigen. A . zubereitete Agarröhrchen. B-C. Vorbereitung des Agar-Pads. D. Rutsche teilweise mit Wasser gefüllt. E. Versiegeln der Rutsche mit Vaseline. F. Letzte Vorbereitung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
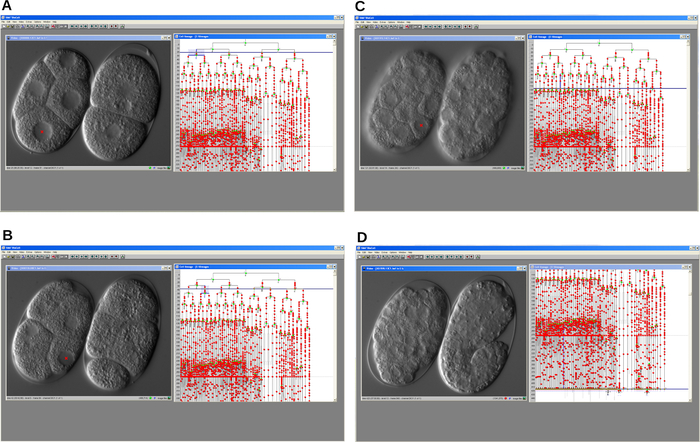
Abbildung 4: Serielle Screenshots von Cell Lineage Tracer-Software. Das Programm ermöglicht die Rekonstruktion der embryonalen Zelllinie einer Zellzyklusverzögerungsmutante (links) und eines WT (rechts) C. elegans-Embryos. Ein. Ein früher Schritt der Entwicklung. B-C. Die Entwicklung beider Embryonen schreitet im Laufe der Zeit voran. D. WT-Embryo entwickelt sich richtig und beginnt mit der Dehnung, während die Mutante anhält. In allen Fällen zeigt das Programm das Videofenster und das Linienfenster an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
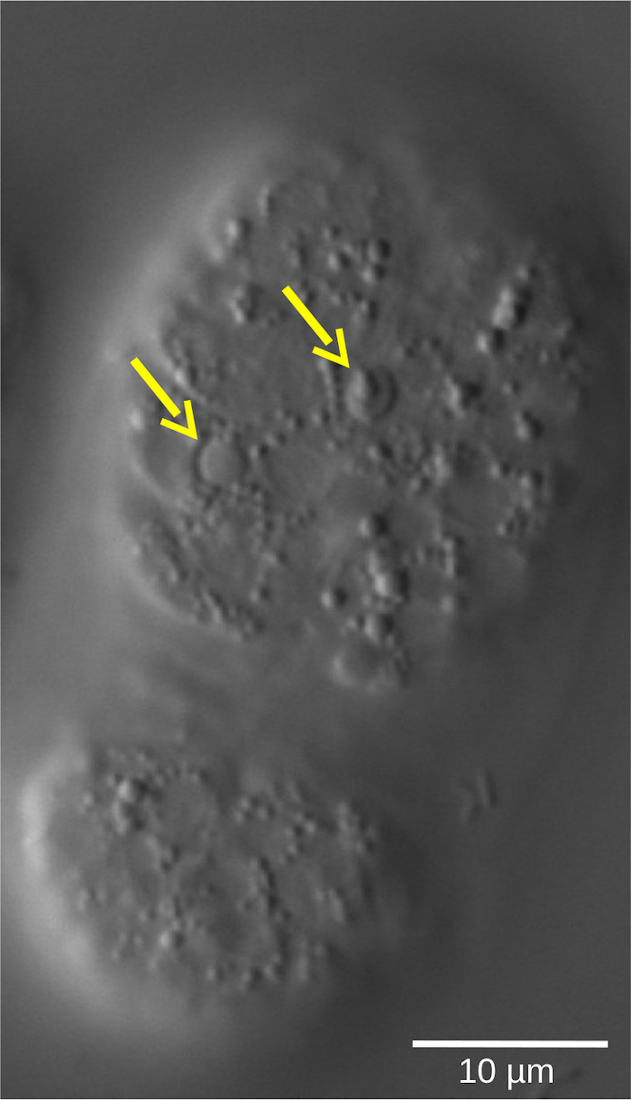
Abbildung 5: Linsenrefraktile Form apoptotischer Zellen in einem WT-Embryo von C. elegans . Das durch morphologische Merkmale definierte Zellschicksal kann mittels 4D-Mikroskopie beurteilt werden. Das Bild zeigt einen C. elegans-Embryo im Bohnenstadium. Lebende Zellen zeigen glattgeformte Kerne, die von einem körnigen Zytoplasma umgeben sind. Im Gegensatz dazu kondensieren apoptotische Zellen (gelbe Pfeile) und nehmen eine linsenartige, feuerfeste Form an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Eine der größten Herausforderungen in der modernen Biologie ist das Verständnis der Entwicklung vielzelliger Organismen. C. elegans hat sich als eines der am besten geeigneten Modelle für die Untersuchung der feinen Koordination zwischen Zellproliferation und Zelldifferenzierung im sich entwickelnden Embryo herausgestellt. Aus optischer Sicht machen sein transparenter Körper und seine geringe Größe diesen Fadenwurm zu einem idealen Exemplar für die DIC-Mikroskopie. Andere Organismen mit ähnlichen Eigenschaften wurden ebenfalls einer 4D-Mikroskopie-Analyseunterzogen 11,12,13,14,15,16.
Für diese Entwicklungsstudien liefert die Geninaktivierung durch Vorwärts- oder Rückwärtsgenetik einen Hinweis auf ihre Beteiligung an der Embryogenese. Sobald nachgewiesen wurde, dass ein Gen eine Rolle bei der Entwicklung spielt, besteht der nächste Schritt darin, seine genaue Rolle bei der Festlegung des richtigen Körperplans zu definieren. Die Immunfärbung ist der gewählte Ansatz für die meisten Modelle. Diese Technik klärt Probleme bei der Zelldifferenzierung oder Expression spezifischer Marker auf. Eine wesentliche Einschränkung dieses Ansatzes besteht jedoch darin, dass er nur eine statische Ansicht des Ausdrucks eines einzelnen oder mehrerer Marker an einem festen Punkt in der Entwicklung bietet. Eine dynamische Sicht auf diese Marker während der gesamten Entwicklung kann nur durch die Färbung verschiedener Embryonen zu unterschiedlichen Zeitpunkten erhalten werden. Zudem ist eine Zelllinienrekonstruktion in solchen fixierten Proben nicht möglich.
Die 4D-Mikroskopie ist ein komplementärer Ansatz zur Untersuchung der Embryonalentwicklung. Diese Technik zeigt die Entwicklungsdynamik bei einer Auflösung auf Zellebene. Jeder Defekt im Embryo wie Probleme in der Spindelorientierung, Zellmigration, Apoptose, Zellschicksalsspezifikation usw. werden in einem 4D-Film angezeigt, der vom Forscher vorwärts und rückwärts visualisiert, quantifiziert und bewertet werden kann. Mit dieser Technik kann praktisch jede einzelne Zelle im Embryo bis zu dem Moment verfolgt werden, in dem sich der Embryo zu bewegen beginnt. Embryonen, die einer 4D-Mikroskopie mit nur sichtbarem Licht und Nomarski-Optik unterzogen werden, erleiden keine Lichtschäden. Fluoreszierende Scans können auch innerhalb der Aufzeichnung interkaliert werden, um zu erkennen, wann und wo ein Gen exprimiert wird. Embryonen, die erhebliche Lichtschäden erleiden, werden durch die Verlängerung des Zellzyklus identifiziert, die im Vergleich zu einem Standard-WT-Linienembryo starke UV-Bestrahlung verursacht. In diesem Fall können Lichtschäden reduziert werden, indem die Intensität der UV-Lampe verringert und die Kameraempfindlichkeit oder die Belichtungszeit erhöht werden. Morphologische Merkmale und molekulare Marker können helfen, die embryonale Entwicklung einer Mutante zu klären.
Die Einrichtung eines 4D-Mikroskopiesystems ist im Labor einfach zu implementieren und ermöglicht nach einiger Übung eine unübertroffene Analyse der Zelldynamik und Linienverfolgung von Zellkulturen und lebenden transparenten Proben auf einer Auflösungsebene jeder einzelnen Zelle im Mikroskopfeld. Die Zelllinienverfolgung auf DIC-Bildern wird immer noch von Hand verarbeitet. Es ist zeitaufwendig und obwohl die Software Abstammungsfehler erkennt, wie z.B. verschiedene Abstammungszweige, die dieselbe Zelle markieren, sind Fehler möglich. Während die automatische Erkennung von GFP-markierten Zellen gut entwickelt ist2, befindet sich die ergänzende Linienverfolgungssoftware, die auf unmarkierten Zellen und Bildern des sichtbaren Lichts basiert, noch im Frühstadium und ist für eine vollständige Embryonenanalyse nicht wirklich nützlich. Ohne Zweifel wird die Anwendung von Bilderkennungssystemen auf dem Gebiet der Mikroskopie des sichtbaren Lichts einen großen Fortschritt auf diesem Gebiet bringen.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren danken der Rioja Salud Foundation (Fondos FEDER) und dem spanischen Ministerio de Ciencia, Innovación y Universidades (MCIU) (Grant PGC2018-094276-B-I00). Cristina Romero Aranda wird durch ein Stipendium der AECC (Asociación Española Contra el Cáncer) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Caenorhabditis elegans (N2) | GCG (Caenorhabditis Genetics Center) | N2 | WT C. elegans strain. Can be requested at GCG (Caenorhabditis Genetics Center): https://cgc.umn.edu/ |
| Caenorhabditis elegans (VZ454) | GCG (Caenorhabditis Genetics Center) | VZ454 | gsr-1(tm3574) C. elegans mutant strain. Can be requested at GCG (Caenorhabditis Genetics Center): https://cgc.umn.edu/ |
| Cell Lineage Tracing software | SIMI | Simi BioCell | This is the software to reconstruct the embryo cell lineage. For a detailed explanation check at: http://www.simi.com/en/products/cell-research/simi-biocell.html |
| Microscope camera | Hamamatsu | Orca-R2 | Miscroscope camera for both transmitted and UV light |
| Microscope control software | Caenotec | Time to Live | This software controls the microscope to perform the 4D image capture. Can be requested at: Caenotec Prof. Ralf Schnabel Kleine Dorfstr. 9 38312 Börßum, Germany, Ph: ++49 151 11653356 r.schnabel(at)tu-bs.de |
| Microscope control software | Micro-manager | Micro-manager | This software controls the microscope to perform the 4D image capture. Can be downloaded at: https://micro-manager.org/ |
| Motorized microscope | Leica | Leica DM6000 | Motorized upright microscope to perform 4D microscopy |
| Standard equipment in a Molecular Biology lab. | |||
| Stereomicroscope | Leica | MZ16FA | Steromicroscope to manipulate nematodes and prepare embryos. |
Referenzen
- Hardin, J. Imaging embryonic morphogenesis in C. elegans. Methods in Cell Biology. 106, 377-412 (2011).
- Mace, D. L., Weisdepp, P., Gevirtzman, L., Boyle, T., Waterston, R. H. A high-fidelity cell lineage tracing method for obtaining systematic spatiotemporal gene expression patterns in Caenorhabditis elegans. G3: Genes, Genomes, Genetics (Bethesda). 3 (5), 851-863 (2013).
- Nieto, C., et al. ccz-1 mediates the digestion of apoptotic corpses in C. elegans. Journal of Cell Science. 123 (12), 2001-2007 (2010).
- Cabello, J., et al. PDR-1/hParkin negatively regulates the phagocytosis of apoptotic cell corpses in Caenorhabditis elegans. Cell Death & Disease. 5, 1120 (2014).
- Pinto, S. M., Almendinger, J., Cabello, J., Hengartner, M. O. Loss of Acetylcholine Signaling Reduces Cell Clearance Deficiencies in Caenorhabditis elegans. PLOS One. 11 (2), 0149274 (2016).
- Sáenz-Narciso, B., Gómez-Orte, E., Zheleva, A., Gastaca, I., Cabello, J. Control of developmental networks by Rac/Rho small GTPases: How cytoskeletal changes during embryogenesis are orchestrated. Bioessays. 38 (12), 1246-1254 (2016).
- Zheleva, A. Reduction of mRNA export unmasks different tissue sensitivities to low mRNA levels during Caenorhabditis elegans development. PLOS Genetics. 15 (9), 1008338 (2019).
- Schnabel, R., Hutter, H., Moerman, D., Schnabel, H. Assessing normal embryogenesis in Caenorhabditis elegans using a 4D microscope: variability of development and regional specification. Developmental Biology. 184 (2), 234-265 (1997).
- Verbrugghe, K. J. C., Chan, R. C. Imaging C. elegans Embryos using an Epifluorescent Microscope and Open Source Software. Journal of Visualized Experiments. (49), e2625 (2011).
- Boyd, L., Hajjar, C., O'Connell, K. Time-lapse Microscopy of Early Embryogenesis in Caenorhabditis elegans. Journal of Visualized Experiments. (54), e2852 (2011).
- Urbach, R., Schnabel, R., Technau, G. M. The pattern of neuroblast formation, mitotic domains and proneural gene expression during early brain development in Drosophila. Development. 130 (16), 3589-3606 (2003).
- Dolinski, C., Borgonie, G., Schnabel, R., Baldwin, J. G. Buccal capsule development as a consideration for phylogenetic analysis of Rhabditida (Nemata). Development Genes and Evolution. 208 (9), 495-503 (1998).
- Houthoofd, W., Jacobsen, K., Mertens, C., Vangestel, S., Coomans, A., Borgonie, G. Embryonic cell lineage of the marine nematode Pellioditis marina. Developmental Biology. 258 (1), 57-69 (2003).
- Hejnol, A., Schnabel, R. The eutardigrade Thulinia stephaniae has an indeterminate development and the potential to regulate early blastomere ablations. Development. 132 (6), 1349-1361 (2005).
- Hejnol, A., Schnabel, R. What a couple of dimensions can do for you: Comparative developmental studies using 4D microscopy-examples from tardigrade development. Integrative and Comparative Biology. 46 (2), 151-161 (2006).
- Bischoff, M., Parfitt, D. E., Zernicka-Goetz, M. Formation of the embryonic-abembryonic axis of the mouse blastocyst: relationships between orientation of early cleavage divisions and pattern of symmetric/asymmetric divisions. Development. 135 (5), 953-962 (2008).
- Mora-Lorca, J. A. Glutathione reductase gsr-1 is an essential gene required for Caenorhabditis elegans early embryonic development. Free Radical Biology and Medicine. 96, 446-461 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten