Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Biplanare Videoradiographie zur Untersuchung des Handgelenks und der distalen Radioulnargelenke
In diesem Artikel
Zusammenfassung
Die biplanare Videoradiographie (BVR) ist ein fortschrittliches bildgebendes Verfahren zum Verständnis der dreidimensionalen Bewegung von Skelettknochen und Implantaten. Durch die Kombination von dichtebasierten Bildvolumen und Röntgenbildern der distalen oberen Extremität wird der BVR zur Untersuchung der In-vivo-Bewegung des Handgelenks und des distalen Radioulnargelenks sowie für Gelenkendoprothesen verwendet.
Zusammenfassung
Die genaue Messung der Skelettkinematik in vivo ist unerlässlich für das Verständnis der normalen Gelenkfunktion, des Einflusses der Pathologie, des Krankheitsverlaufs und der Auswirkungen von Behandlungen. Messsysteme, die Marker für die Hautoberfläche verwenden, um auf die Skelettbewegung zu schließen, haben wichtige Einblicke in die normale und pathologische Kinematik geliefert, jedoch kann mit diesen Systemen keine genaue Arthrokinematik erreicht werden, insbesondere bei dynamischen Aktivitäten. In den letzten zwei Jahrzehnten haben biplanare Videoradiographie-Systeme (BVR) es vielen Forschern ermöglicht, die Skelettkinematik der Gelenke bei Aktivitäten des täglichen Lebens direkt zu untersuchen. Um BVR-Systeme für die distale obere Extremität zu implementieren, werden Videoröntgenaufnahmen des distalen Radius und der Hand von zwei kalibrierten Röntgenquellen aufgenommen, während ein Proband eine bestimmte Aufgabe ausführt. Dreidimensionale (3D) Starrkörperpositionen werden aus den Videoröntgenbildern über eine Best-Fit-Registrierung von 3D-Modellprojektionen auf jede BVR-Ansicht berechnet. Bei den 3D-Modellen handelt es sich um dichtebasierte Bildvolumina des spezifischen Knochens, die aus unabhängig gewonnenen Computertomographie-Daten abgeleitet werden. Durch die Verwendung von Grafikprozessoren und Hochleistungsrechensystemen hat sich dieser modellbasierte Tracking-Ansatz als schnell und genau bei der Bewertung der Biomechanik des Handgelenks und des distalen Radioulnargelenks erwiesen. In dieser Studie fassten wir zunächst die vorangegangenen Studien zusammen, die die Submillimeter- und Subgrad-Übereinstimmung von BVR mit einem in vitro optischen Motion-Capture-System bei der Bewertung der Kinematik des Handgelenks und des distalen Radioulnargelenks festgestellt haben. Darüber hinaus haben wir die BVR verwendet, um das Rotationsverhalten des Handgelenks zu berechnen, das Gelenkmuster der Komponenten des Implantats zueinander zu bewerten und die dynamische Veränderung der ulnaren Varianz während der Pronosupination des Unterarms zu beurteilen. In Zukunft könnten Handwurzelknochen durch die Hinzufügung von flachen Röntgendetektoren, mehr Röntgenquellen (z. B. multiplanare Videoradiographie) oder fortschrittlichen Computer-Vision-Algorithmen detaillierter erfasst werden.
Einleitung
Die genaue Messung der Skelettkinematik in vivo ist unerlässlich für das Verständnis einer gesunden und ersetzten Gelenkfunktion, des Einflusses der Pathologie, des Krankheitsverlaufs und der Auswirkungen von Behandlungen. Die nicht-invasive Quantifizierung der Skelettkinematik an der Gelenkoberfläche (Arthrokinematik) ist entscheidend für das Verständnis von Gelenkpathologien und -erkrankungen, wie z. B. Arthrose, aber sie ist auch technisch anspruchsvoll. Früher haben Techniken, die Marker auf der Hautoberfläche verwenden, um auf die Skelettbewegung zu schließen, wichtige Einblicke in die gesunde und pathologische Kinematik geliefert. Eine genaue Arthrokinematik kann mit diesen Techniken jedoch nicht erreicht werden, insbesondere bei dynamischen Aktivitäten wie Aktivitäten des täglichen Lebens. Diese optischen Systeme sind von Natur aus in ihrer Genauigkeit eingeschränkt, da die Hautbewegung relativ zu den darunter liegenden Knochen die Hauptfehlerquelle bei der Analyse menschlicher Bewegungen darstellt 1,2.
Die derzeit neuesten Methoden zur Quantifizierung der dreidimensionalen (3D) Skelettkinematik sind das bildbasierte Tracking, nämlich die Biplane Videoradiographie (BVR)3 und die serielle Computertomographie (CT) Band4 sowie die Magnetresonanztomographie (MRT)5. Obwohl reguläre 3D-CT- und MRT-basierte Technologien sehr genau und in vielen Krankenhäusern auf der ganzen Welt zugänglich sind, sind sie nicht in der Lage, die dynamische Bewegung der Gelenke zu messen. Bildgebende Verfahren wie die 4D-CT6 und die dynamische MRT7 wurden in den letzten Jahren entwickelt, um dieses Manko zu beheben. Diese Methoden setzen die Patienten jedoch entweder einer hohen Strahlendosis aus oder leiden unter einer geringen zeitlichen Auflösung.
Durch die Kombination neuartiger Computer-Vision-Algorithmen und traditioneller Röntgensysteme hat sich BVR als genau für mehrere Gelenke bei Tieren und Menschen erwiesen. Gelöst entweder mit markerbasierten oder modellbasierten Tracking-Algorithmen. Markerbasierte Ansätze verfolgen Tantalkügelchen, die in Knochen oder Weichgewebe eingebracht werden, und sind optimal für Tier- und In-vitro-Tests. Für die In-vivo-Forschung am Menschen sind sie jedoch unerschwinglich invasiv. Glücklicherweise bieten Verbesserungen bei modellbasierten Tracking-Algorithmen eine praktikable Alternative. Modellbasierte BVR-Tracking-Ansätze beim Menschen beinhalten die Aufbereitung der durch CT oder MRT aufgenommenen volumetrischen Bildsätze in einer statischen Haltung und die Erfassung der Bewegungen von Interessenten im Sichtfeld von zwei Röntgenbildern. Die meisten modellbasierten Tracking-Anwendungen generieren dann digital rekonstruierte Röntgenbilder (DRR) des Knochens oder Implantats aus den statischen CT- oder MRT-Bildern und gleichen sie mit merkmalserweiterten Videoröntgenbildern ab, wobei Metriken verwendet werden, die die Ähnlichkeit zwischen DRRs und Videoröntgenaufnahmen belegen8. Dieser Vorgang wird als "Tracking" des Knochens oder Implantats bezeichnet.
Die primären Ausgangsvariablen der Verfolgung von Knochen oder Implantaten sind die Starrkörperkinematik, aus der Gelenkkinematik, Banddehnungen 9,10, Gelenkabstände als Surrogat für die Knorpeldicke11, Gelenkkontakt12,13 und andere Biomarker berechnet werden können. Kürzlich haben wir die Genauigkeit des modellbasierten Trackings der BVR bei der Berechnung der Biomechanik des Handgelenks, der Handgelenkstotalendoprothetik (TWA) und des distalen Radioulnargelenks (DRUJ) dokumentiert14,15. Im folgenden Abschnitt wird ein detailliertes Protokoll dieser validierten Methode zur Untersuchung der Bewegung des Skeletthandgelenks, der Totalendoprothetik des Handgelenks und des distalen Radioulnargelenks bei verschiedenen Aufgaben vorgestellt. Wir segmentieren die dichtebasierten Bildvolumina der Knochen und Implantate aus den CT-Bildvolumen, verfolgen diese partiellen Bildvolumina innerhalb der Videoröntgenbilder und bestimmen Ergebnisse wie Rotationszentrum, Kontaktmuster und ulnare Varianz, um die Stärken und Grenzen dieser Methode zu demonstrieren.
Protokoll
Diese Studie wurde vom Institutional Review Board (IRB) des Lifespan - Rhode Island Hospital, einem AAHRPP-akkreditierten IRB, genehmigt. Insgesamt 16 Patienten gaben eine unterschriebene Einverständniserklärung gemäß den institutionellen Richtlinien ab.
1. Datenerfassung
- Computertomographie (CT)
- Bereiten Sie die Proben oder Probanden für das CT vor.
HINWEIS: Für die Genauigkeitsbewertungwurden 14,15 Jahre lang 6 intakte Unterarme von vier intakten Leichenproben (70,5 ± 12,3 Jahre, 4 rechte, 2 weiblich) und 1 Unterarm (49 Jahre, rechts, weiblich) mit einer Handgelenkstotalendoprothetik verwendet. Für die in vivo-Studie 16 wurden 10 Probanden mit gesunden Handgelenken (57,0 ± 5,2 Jahre, acht Frauen, neun rechtshändige dominant) und 6 Patienten (74,7 ± 5,6 Jahre, zwei Frauen, zwei rechte, vier dominante Handgelenke) aufgenommen, die sich einer Handgelenkstotalendoprothetik unterzogen hatten. - Um jedes Subjekt4 abzubilden, nehmen Sie einen CT-Scan des distalen Unterarms mit einem Erfassungsdurchmesser von 25 cm auf und rekonstruieren Sie mit einem Sichtfeld von 20 cm unter Verwendung des Bone Plus-Faltungsalgorithmus unter Verwendung eines klinischen 16-Zeilen-CT-Scanners. Verwenden Sie Röhreneinstellungen von 80 kVp und 80 mA. Begrenzen Sie die Exposition des Unterarms auf ca. 7 cm proximal der distalen Radiusfläche zu den distalen Phalangen.
HINWEIS: In dieser Studie betrugen die Voxelabmessungen des Bildes 0,39 × 0,39 mm2 in der Querebene des Unterarms und 0,625 mm entlang der Längsachse des Unterarms. - Übertragen Sie die aufgenommenen CT-Bilder mit einem HIPPA-zugelassenen Protokoll im DICOM-Format (Digital Imaging and Communications in Medicine) auf den Computer des Labors.
- Bereiten Sie die Proben oder Probanden für das CT vor.
- Biplanare Videoradiographie (BVR)
- Versuchsaufbau (Abbildung 1). Um einen Gesichtsfelddurchmesser zu ermöglichen, der in der Lage ist, den distalen Radius, die distale Ulna, die Handwurzelknochen und den Mittelhandknochen zu erfassen, sowie eine Perspektive zur Minimierung knöcherner Okklusionen in verschiedenen Handgelenkshaltungen, positionieren Sie die Bildverstärker (IIs) unterhalb der Unterarme und stellen Sie den Winkel zwischen ihnen auf 110° ein (Röntgenquellen richten sich nach unten auf die entsprechenden IIs in einem Winkel von 55° relativ zum Boden).
- Erzeugen Sie die Röntgenstrahlen im kontinuierlichen Modus mit einer Belichtungseinstellung zwischen 65 und 75 kV, abhängig von der Sichtbarkeit des Knochens/Implantats, und 80 mA. Stellen Sie den Abstand zwischen Quelle und Bild beider Röntgen-II-Paare auf ca. 130 cm und den Abstand zwischen Quelle und Hand auf 90 cm ein.
HINWEIS: In dieser Studie trugen die Probanden im Labor eine Röntgenschutzschürze, eine Halsabdeckung für den Schilddrüsenkragenschild und ein digitales Dosimeter. Alle Forscher standen während des Experiments hinter einem Bleischutzglas. - Platzieren Sie einen Bock oder eine ähnliche Struktur zwischen den IIs und verwenden Sie ihn zum Platzieren von Gegenständen (z. B. Türklinke, Hammer, Krug) für die Motive.
- Erzeugen Sie die Röntgenstrahlen im kontinuierlichen Modus mit einer Belichtungseinstellung zwischen 65 und 75 kV, abhängig von der Sichtbarkeit des Knochens/Implantats, und 80 mA. Stellen Sie den Abstand zwischen Quelle und Bild beider Röntgen-II-Paare auf ca. 130 cm und den Abstand zwischen Quelle und Hand auf 90 cm ein.
- Versuchsaufbau (Abbildung 1). Um einen Gesichtsfelddurchmesser zu ermöglichen, der in der Lage ist, den distalen Radius, die distale Ulna, die Handwurzelknochen und den Mittelhandknochen zu erfassen, sowie eine Perspektive zur Minimierung knöcherner Okklusionen in verschiedenen Handgelenkshaltungen, positionieren Sie die Bildverstärker (IIs) unterhalb der Unterarme und stellen Sie den Winkel zwischen ihnen auf 110° ein (Röntgenquellen richten sich nach unten auf die entsprechenden IIs in einem Winkel von 55° relativ zum Boden).
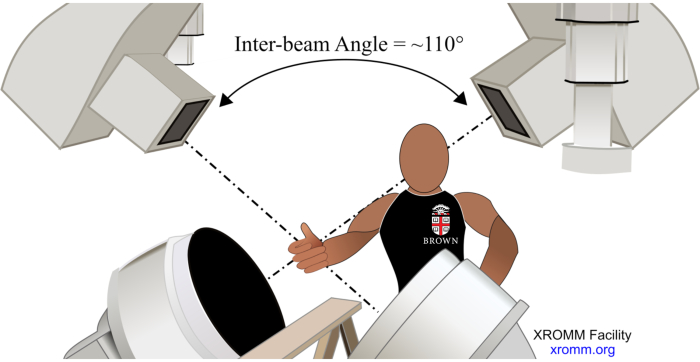
Abbildung 1. Versuchsaufbau. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Unverzerrtes Gitter (Abbildung 2A). Da die externen elektromagnetischen Felder das Verhalten der Röntgenstrahlen (d. h. die Elektronenwege) beeinflussen, leiden die Röntgenbilder unter nichtlinearen nadelkissenförmigen und S-förmigen Verzerrungen17. Um diese Verzerrungen zu entfernen, verwenden Sie das Bild eines flachen Stücks Präzisionsperforiertes Blech (d. h. ein unverzerrtes Gitter)18,19. Platzieren Sie verzerrungsfreie Gitter auf den IIs und erfassen Sie ihre Bilder sowohl vor als auch nach den Videoradiographiedaten. Speichern Sie diese Bilder als 8-Bit-TIFF-Bilder für beide Röntgenquellen.
- Kalibrierung (Abbildung 2B). Eine Kalibrierung der Röntgenquellen ist notwendig, um die 3D-Koordinaten der Objekte (Knochen oder Implantate) zu berechnen20. Verwenden Sie Bilder eines 3D-Objekts mit bekannter Geometrie, das regelmäßig röntgenundurchlässige Kugeln beabstandet hat, um eine Beziehung zwischen der Position und Ausrichtung der Röntgenquelle im 3D-Raum und dem Bildraum (d. h. Röntgenbildern) herzustellen. Ein kubisches Objekt (d. h. ein Kalibrierwürfel) aus Acrylplatten und Abstandshaltern mit Stahlkugeln sind für BVR-Einstellungen19 effektiv und genau.
- Erfassen Sie Röntgenbilder des Kalibrierwürfels18 in verschiedenen Ausrichtungen 4 mal vor der Durchführung des Experiments und 4 mal nach dem Experiment. Die Kalibrierungen nach dem Experiment dienen dazu, zu bestätigen, dass der Versuchsaufbau während des gesamten Experiments unverändert bleibt. Speichern Sie alle Bilder als 8-Bit-TIFF-Bilder für beide Röntgenquellen.
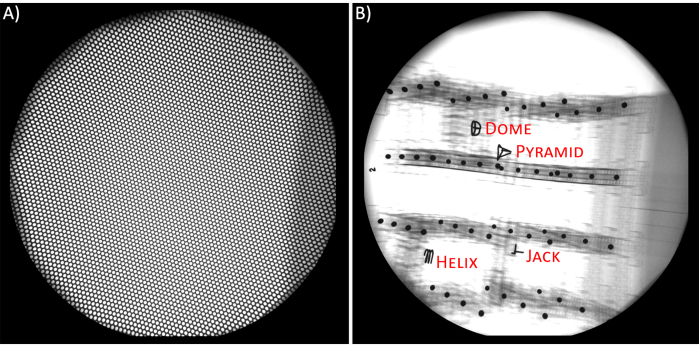
Abbildung 2. A) Unverzerrtes Gitter. B) Kalibrierwürfel und seine Referenzelemente. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Beschreibung der Aufgaben. Definieren Sie verschiedene Aufgaben, um die Bewegung des Handgelenks während der Flexion-Streckung, der radial-ulnaren Abweichung und der Zirkumduktion16 zu erfassen. Beschreiben Sie DRUJ-Bewegungsaufgaben als Unterarmdrehung von einer Haltehaltung zur vollen Pronation oder zur vollständigen Supination, definiert als Drehung eines Türknaufs mit T-Griff im Uhrzeigersinn und gegen den Uhrzeigersinn21. Nehmen Sie Videoröntgenaufnahmen des Handgelenks in seiner neutralen Haltung (d. h. neutrale statische Aufgabe) auf. Erfassen Sie die neutrale Pose mit dem Handrücken koplanar mit der Rückseite des Unterarms.
HINWEIS: In der Ausgangshaltung für alle Aufgaben befand sich die Schulter in Adduktion, der Ellenbogen wurde auf Gelenkhöhe abgestützt und der Unterarm war frei und in seiner neutralen Rotation. Die Aufgaben der Flexion-Extension und der radial-ulnaren Abweichung wurden als die Rotation der Handfläche relativ zur volar-dorsalen bzw. radial-ulnaren Seite der Hand beschrieben. Die Zirkumduktion wurde als die Bewegung des Handgelenks beschrieben, die darauf abzielte, einen maximalen aktiven Bewegungsumfang in alle Richtungen zu erreichen. - Aufnahme von Videoröntgenaufnahmen. Erfassen Sie Röntgenbilder mit einer Rate von 200 Hz und einer Verschlusszeit von 500 μs. Nehmen Sie für jede Aufgabe zwei Sekunden Bildgebung auf, um 2 Wiederholungen zu erhalten. Speichern Sie Röntgenbilder, die von jeder Röntgenquelle aufgenommen wurden, als CINE-Video in einem 8-Bit-Format.
HINWEIS: In dieser Studie hatten die Röntgenbilder eine Bildgröße von 1760×1760 Pixeln und eine Auflösung von ca. 0,22 mm×0,22 mm pro Pixel.
HINWEIS: Aktuelle Röntgenquellen verfügen über eine Option zur Vergrößerung des Sichtfelds (FOV). In dieser Studie wurde jedoch der von der Hardware angebotene vergrößerte Modus nicht verwendet. Basierend auf der Aufgabenbeschreibung können Sie mit dieser Funktion möglicherweise Ausgaben mit höherer Auflösung abrufen. Wenn diese Funktion verwendet wird, müssen die Schritte "Unverzerrtes Raster" und "Kalibrierung" in diesem Modus erfasst werden.
- Optical Motion Capture (OMC) - Genauigkeitsstudie
- Verwenden Sie das in früheren Studien beschriebene optische Motion-Capture-Datenerfassungsprotokoll (OMC)14,15. Befestigen Sie zwei Marker-Cluster mit jeweils vier retroreflektierenden Markerkugeln mit einem Durchmesser von 9,5 mm direkt an den dritten Mittelhand- und Radiusknochen. Erfassen Sie Markierungsbewegungen mit 8 optischen Motion-Capture-Kameras. Synchronisieren Sie den Start der OMC-Datenerfassung mit dem BVR durch einen aktiven externen Low-Trigger.
2. Datenverarbeitung
- Computertomographie (CT)
- DICOM-Anonymisierung. Anonymisieren Sie die DICOMs und importieren Sie sie in eine Bildverarbeitungssoftware für die 3D-Modellierung (z. B. Mimics Materialise). Öffnen Sie in der Mimics Materialise-Software in der Symbolleiste "Datei " das Fenster "Anonymisieren ". Wenn das neue Fenster geöffnet wird, wählen Sie auf der linken Registerkarte Alle Bilder anonymisieren und klicken Sie auf die Schaltfläche Anonymisieren unten rechts.
- Segmentierung (Abbildung 3). Segmentieren Sie die Knochen oder Implantate von Interesse nach einer zuvor beschriebenen Methodik 16,22,23.
- Wählen Sie in Kürze in der Symbolleiste "Segment" die Option "Schwellenwert " aus, und wählen Sie beim Öffnen des Fensters die vordefinierten Schwellenwerte für "Knochen " aus. Nachdem die Maske mit Schwellenwert erstellt wurde, verwenden Sie die Option "Geteilte Maske" und malen Sie manuell die Bereiche von Bones oder Implantatkomponenten.
- Wenn alle Bereiche farbig sind, klicken Sie auf OK und warten Sie, bis die Software die Masken für jeden Knochen und die Implantatkomponente automatisch generiert. Überprüfen Sie alle Masken, um sicherzustellen, dass sie die vorgesehenen Bereiche vollständig abdecken. Wenn nicht, verwenden Sie die Option Masken bearbeiten , um die Regionen manuell und einzeln zu ändern.
HINWEIS: Um das Handgelenk zu untersuchen, sollten der 2. und 3. Mittelhandknochen und die Radiusknochen segmentiert und vom Rest der Knochen getrennt werden. Optional können der Kopf und andere Knochen für die Nachbearbeitung segmentiert werden.
HINWEIS: Um die DRUJ zu untersuchen, sollten der Radius und die Elle segmentiert und vom Rest der Knochen getrennt werden.
HINWEIS: Bei den Implantaten sollten die radiale Komponente und die Karpalkomponente mit ihren Schrauben segmentiert werden.
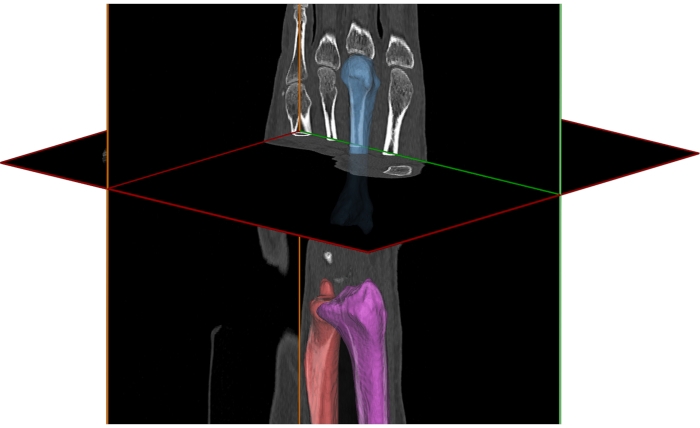
Abbildung 3. Computertomographie-Aufnahme des Handgelenks und rekonstruierte Modelle des Radius, des dritten Mittelhandknochens und der Elle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Segmentierung - nur radiale Komponente. Aufgrund von dichtebasierten Streifenartefakten der Oberfläche der radialen Komponente in den CT-Bildern kann das radiale Komponentenmodell nicht aus den Bildern segmentiert werden. Verwenden Sie daher ein CAD-Modell oder einen 3D-Oberflächenscanner, um das 3D-Modell des explantierten Radialbauteils zu generieren. Registrieren Sie dieses Modell für die grobe Segmentierung der radialen Komponente mit der Standardmethode von Mimics Materialise.
HINWEIS: In dieser Studie haben wir einen Oberflächenscanner mit einer Auflösung von 0,1 mm verwendet. Das Implantat wurde auf einen Tisch gelegt, und der 3D-Scanner wurde manuell um das Implantat herum bewegt, um alle äußeren Oberflächen zu erfassen. - Partielle Bildvolumen. Die Tracking-Software erfordert, dass ein segmentierter Body of Interest als dichtebasiertes Bildvolumen eingegeben wird. Um dieses Teilvolumen zu erstellen, generieren Sie eine Vollmaske des Bildvolumens, die alle Grauwerte enthält, indem Sie in der Symbolleiste "Segment" die Option "Schwellenwert" auswählen. Passen Sie die Schwellenwerte von den minimalen bis zu den maximalen Grauwerten an.
- Verwenden Sie dann die Option Boolesche Operatoren und subtrahieren Sie das segmentierte Volumen des interessierenden Knochens oder Implantats von der generierten Vollmaske. Ändern Sie die Farbe der Maske dieses Teilvolumens in Schwarz (RGB: 0,0/0,0/0,0), da die Registrierungssoftware nur Graustufen-Bildvolumes akzeptiert. Exportieren Sie die Maske als DICOM, indem Sie DICOM exportieren... in der Software. Die Ausgabe ist ein Bildvolumen des isolierten Knochens oder Implantats.
- Dreidimensionale Modelle. Konvertieren Sie die segmentierten Masken in triangulierte, vernetzte 3D-Oberflächenmodelle, indem Sie die Option 3D berechnen mit der optimalen Auflösung auswählen. Exportieren Sie diese Modelle im VRML-Format, das die 3D-Position der Scheitelpunkte (Punkte) und die Reihenfolge ihrer Verbindungen enthält. Die Modelle werden für die Konstruktion des Koordinatensystems und die weitere Visualisierung generiert.
- Vorverarbeitung der Videoradiographie
- XMALab18 Import. Erstellen Sie für jeden Probanden eine neue Testversion in der XMALab-Software. Weisen Sie die Ordnerpfade den Undistortion Grid-Bildern und 8 Sätzen von Kalibrierungsbildern im Dialogfeld "Neue Testversion " zu.
HINWEIS: Wenn die Röntgenquellen während der Aufnahme umgedreht werden, muss die Option "Spiegeln" in der XMALab-Software aktiviert werden. - Entzerrung. Um Röntgenbilder zu entzerren, klicken Sie, nachdem die Testversion erstellt wurde und die Software die Rasterbilder im Arbeitsbereich "Verzerrung" geöffnet hat, auf die Schaltfläche "Verzerrung berechnen ". XMALab ermittelt und zeigt automatisch die verzerrten Bilder der Raster an. Sobald die Mittelpunkte aller Bohrungen von der Software erkannt wurden, ändern Sie den Arbeitsbereich im Menü oben links in den Arbeitsbereich Kalibrierung . Weitere Informationen und Unterstützung beim Debuggen potenzieller Probleme finden Sie in der Referenz24.
- Kalibrierung. In dieser Phase bestimmt die XMALab-Software die Kalibriermatrizen. In XMALab werden die Röntgenquellen als Lochkameras mit intrinsischen und extrinsischen Parametern modelliert, wie z. B. Brennweite, Mittelpunkt der Quelle und Abstände von Quelle zu Bild, und die Kalibrierungsphase ist ein Prozess, der diese Metriken bestimmt18. Wählen Sie zum Kalibrieren jedes der 4 Referenzelemente aus, die sich im Kalibrierungswürfel für alle Bildsätze und für jede Ansicht befinden. In der Konfiguration waren diese Referenzen Pyramid, Jack, Dome und Helix (Abbildung 2B). Wenn die Software die Punkte nicht automatisch findet, drücken Sie auf Kalibrierung berechnen. Nachdem alle Kalibrierungsbilder kalibriert wurden, wählen Sie Kamera-Setup optimieren aus.
- Exportieren von Kalibrierungsmatrizen. Sortieren Sie nach der Optimierung alle 8 Kalibrierungssätze (d. h. die 4 Sätze, die vor dem Experiment erfasst wurden, und die 4 Sätze, die nach dem Experiment erfasst wurden) und wählen Sie den Bildsatz mit dem geringsten Fehler unverzerrt aus. Suchen Sie diesen Wert, indem Sie auf die Informationsoption in der oberen rechten Ecke jedes Kalibrierungsfensters klicken. Exportieren Sie das Bild mit dem geringsten Fehler, indem Sie die Option MayaCams 2.0 aus dem Datei-Export-Menü auswählen. Weitere Informationen und zum Debuggen möglicher Probleme finden Sie in der Referenz20.
- Videoradiographie Verzerrung. Ändern Sie den Arbeitsbereich auf Marker-Tracking und laden Sie die Videoröntgenbilder, die im CINE-Format aufgenommen wurden, indem Sie das +-Symbol drücken. Nachdem der Ladevorgang abgeschlossen ist, exportieren Sie die unverzerrten Testbilder, indem Sie im Menü Datei-Export die Option Unverzerrte Testbilder auswählen.
- XMALab18 Import. Erstellen Sie für jeden Probanden eine neue Testversion in der XMALab-Software. Weisen Sie die Ordnerpfade den Undistortion Grid-Bildern und 8 Sätzen von Kalibrierungsbildern im Dialogfeld "Neue Testversion " zu.
- Modellbasiertes Tracking (Abbildung 4)
- Verbesserte Funktionen von Röntgenbildern/DRRs (Abbildung 4A-B). Das Verbessern der Bildmerkmale mithilfe von Filtern ist ein notwendiger Schritt, um eine genaue Übereinstimmung zu erzielen. Definieren Sie die Parameter der vier Filter, einschließlich Kontrast (Intensitätserkennung), Sobel (Kantenerkennung), Gauß (Unschärfe/Glättung) und Schärfen (Fettdruck der Kanten) in der Software. Diese Filter können ausgewählt werden, indem Sie mit der rechten Maustaste auf die Rad-Renderer- oder DRR-Renderer-Objekte klicken, die im Widget oben links angezeigt werden.
HINWEIS: In dieser Studie wurden die Ränder der Knochenbilder auf den Röntgenbildern mit einem Sobel-Filter mit einem Skalenfaktor von 3 und einem Mischwert von 0,4 verbessert. Zusätzlich wurde ein Kontrastfilter mit einem Alpha (für Bildkontrast) von 2,5 und Beta (für die Bildhelligkeit) von 0,9 verwendet, um die Qualität der Röntgenbilder weiter zu verbessern. Um die DRRs mit dem Röntgenbild abzugleichen, wurde ein Strahlenintensitätswert von 0,35 mit einem Sobel-Filter mit einem Mischwert von 0,1 und einem Skalenfaktor von 1,7 verwendet. - Manuelle Initialisierung. Verschieben Sie die DRR jedes Knochens und jeder Komponente mithilfe des Drehpunkts , um die bestmögliche Übereinstimmung zwischen DRR und Röntgenbildern zu erzielen. Der Drehpunkt hilft Benutzern, den ausgewählten DRR aus dem unteren linken Widget zu drehen und zu verschieben. Dieser Schritt ist notwendig, da die Position des Knochens oder Implantats mindestens einmal und mindestens für den ersten Frame initialisiert werden muss, bevor die Optimierung durchgeführt wird. Drücken Sie W , um den DRR in den Translationsmodus zu verschieben, und drücken Sie E , um den DRR zu drehen.
HINWEIS: Das Ändern der Position des Drehpunkts ist in der Regel nicht erforderlich, aber es ist mit der Option Drehpunkt verschieben in der Symbolleiste möglich. - Optimierung. Drücken Sie C oder klicken Sie in der Symbolleiste auf die Schaltfläche Strom verfolgen, um die beste Übereinstimmung zwischen dem DRR und den Röntgenbildern zu finden. Das Standardverfahren der Software verwendet die Kostenfunktion der normalisierten Kreuzkorrelation (NCC) und sowohl die Methode zur Optimierung des Partikelschwarms als auch die Downhill-Simplex-Verfahren26,27. Dieser Vorgang wird auch als Matching- oder Tracking-Schritt bezeichnet. Das Optimierungsverfahren kann auch über den Tracking-Dialog ausgeführt werden. Drücken Sie dazu die Option Tracking-Dialog in der Symbolleiste. Wenn das neue Fenster geöffnet wird, ändern Sie die Werte "Von Frame" und "Bis Frame" in 0 und drücken Sie "OK".
HINWEIS: NCC ist eine Funktion, die die Ähnlichkeit zwischen Röntgenbildern und DRRs misst, und ein NCC-Wert, der näher an 0 liegt, zeigte eine bessere Übereinstimmung. Das Erreichen dieses Wertes hängt jedoch vollständig von den Bildfiltern und der Aufnahmequalität ab. Der Wert von NCC kann im "Terminalfenster" beobachtet werden. - Matching-Prozess - Erster Durchgang. Nachdem der erste Frame automatisch optimiert wurde, öffnen Sie das Dialogfeld "Tracking", ändern Sie den Wert "Von Frame " auf 0 und den Wert von "Bis Frame" auf die letzte Framenummer (diese befindet sich rechts unten). Der standardmäßige Abgleichsprozess der Software verwendet den aktuellen Frame als Anfangsposition des DRR für den Abgleichsprozess. Der Optimierungsprozess verwendet die Methode der Partikelschwarmoptimierung (PSO) in einem Bereich von 3 mm und 3° der Ausgangsposition.
- Abgleichsprozess - Letzter Durchlauf (Abbildung 4C). Da der automatische Abgleichsprozess aufgrund einer fehlerhaften Initialisierung oder von Filtern fehlschlagen kann, überprüfen Sie alle Frames manuell, um sicherzustellen, dass die Optimierung korrekt ausgeführt wurde. Wenn die DRR und die Röntgenbilder nicht korrekt übereinstimmen, wiederholen Sie die in den Abschnitten 2.3.2 und 2.3.3 beschriebenen Schritte.
HINWEIS: Die Verfolgung des Handgelenks wurde zunächst mit einem Modell durchgeführt, das den zweiten und dritten Mittelhandknochen (MC2-MC3) umfasste, um die Auswirkungen der Knochenüberlappung zu reduzieren und die Übereinstimmungsgenauigkeit zu verbessern. Nach der Lokalisierung der kombinierten DRR-Position und -Rotation in den Röntgenbildern wurden die abgestimmten und optimierten MC2-MC3-Positionen in das MC3-Teilbildvolumen transformiert und die Anfangspositionen des MC3 geseedet. Anhand dieser gesetzten Positionen wurde der MC3 nachgeführt. Ähnlich wie bei MC3 wurden auch der Radius, die Ulna, die radiale Komponente und die Karpalkomponente in den Videoröntgenaufnahmen während aller Aufgaben verfolgt.
- Verbesserte Funktionen von Röntgenbildern/DRRs (Abbildung 4A-B). Das Verbessern der Bildmerkmale mithilfe von Filtern ist ein notwendiger Schritt, um eine genaue Übereinstimmung zu erzielen. Definieren Sie die Parameter der vier Filter, einschließlich Kontrast (Intensitätserkennung), Sobel (Kantenerkennung), Gauß (Unschärfe/Glättung) und Schärfen (Fettdruck der Kanten) in der Software. Diese Filter können ausgewählt werden, indem Sie mit der rechten Maustaste auf die Rad-Renderer- oder DRR-Renderer-Objekte klicken, die im Widget oben links angezeigt werden.
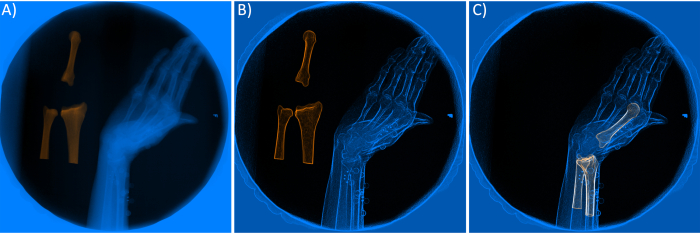
Abbildung 4. A) Aufgenommenes Röntgenbild einer Röntgenquelle mit digital rekonstruierten Röntgenbildern (DRRs) der Knochen. B) Verbesserte (gefilterte) Röntgenaufnahmen und DRRs. C) Angepasste DRRs nach dem Optimierungsprozess. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Datenreduzierung. Basierend auf der Bildrate und der Qualität der Röntgenbilder können Filteransätze wie die Methode des gleitenden Durchschnitts (mit einer Spanne von 5 Bildern) verwendet werden. Wählen Sie dazu in der Symbolleiste die Option Tracking-Erweiterter Dialog und klicken Sie auf die Schaltfläche Glätten . Dieser Ansatz wurde für alle Aufnahmen in dieser Studie verwendet.
- Exportieren von Knochen-/Implantatpositionen. Nachdem jeder Knochen mit den Röntgenbildern abgeglichen und alle Frames optimiert wurden, exportieren Sie die 4x4-Transformationsmatrizen der DRRs in die Röntgenwelt, indem Sie auf die Schaltfläche Save Tracking klicken und die Ausgabe im Matrixformat auswählen. Diese Transformationsmatrix definiert die Rotation und Translation des Teilbildvolumens des Knochens/Implantats im kalibrierten Röntgenweltkoordinatensystem und kann für zusätzliche Verarbeitungs- und Bewegungsberechnungen verwendet werden. Das Koordinatensystem des Teilbildvolumens muss für eine aussagekräftige biomechanische Analyse in das anatomische Koordinatensystem übertragen werden.
3. Datenanalyse
- Koordinatensysteme (Abbildung 5). Um die Kinematik zu erfassen, konstruieren Sie Koordinatensysteme (CS) für die Knochen und implantierten Komponenten auf der Grundlage ihrer anatomischen und geometrischen Orientierungspunkte. Nachdem diese CS erstellt wurden, registrieren Sie die Modelle, die sich in der neuen CS befinden, in den Oberflächenmodellen, die in Abschnitt 2.1.4 mit dem iterativen Algorithmus für den nächsten Punkt generiert wurden. Diese Eins-zu-Eins-Registrierung berechnet eine neue 4x4-Transformationsmatrix, die für Kinematikberechnungen verwendet wird.
HINWEIS: In dieser Studie wurden die CS für den Radius, die Elle, den dritten Mittelhandknochen, die Karpalkomponente, die radiale Komponente, die Polyethylenkappe und den Kopf mit Methoden erstellt, die zuvor beschrieben wurden 16,22,23.
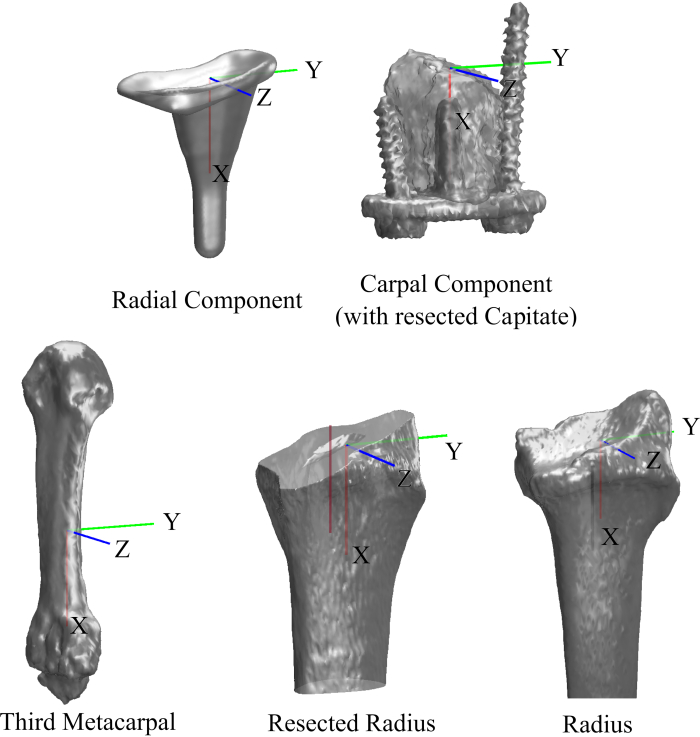
Abbildung 5. Koordinatensysteme der Knochen und der Komponenten des Implantats. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Kinematik. Definieren Sie in dieser Studie die Bewegung des Handgelenks als Bewegung des MC3 relativ zum Radius, und die kinematischen Daten wurden relativ zur neutralen Handgelenksposition berichtet.
- Berechnen Sie für die DRUJ-Analyse die helixalen Bewegungsachsenparameter (HAM)28 , um die Kinematik als die Relativbewegung des Radius CS in der ulnaren CS in Bezug auf die neutrale Pose zu beschreiben.
- Verwenden Sie die HAM-Analyse, um das Rotationszentrum (COR) des Handgelenks bei Beuge-Streckung und radial-ulnarer Abweichung zu bestimmen.
- Um die eindimensionale proximal-distale Verschiebung des COR zu beschreiben, definiert man den projizierten COR für die gesunden Handgelenke als einen Punkt auf der Mittelachse des Kopfknochens, der den kürzesten Abstand von der Rotationsachse der Schraube16 aufwies.
- Zerlegen Sie die HAM-Rotation im ulnaren Koordinatensystem mit Hilfe der HAM-Schraubenachse und berechnen Sie die Pronation (positiv) und Supination (negativ) des Unterarms. Die neutrale Pose wurde definiert, wenn die Pronations-Supination (PS) Null war.
- Definieren Sie abschließend die ulnare Varianz (UV) als den Abstand zwischen der ulnaren Fovea und der Sigmakerbe auf dem Radius.
- Artikulationsmuster
- Berechnen Sie die Kontaktmuster zwischen der Polyethylenkappe und dem radialen Bauteil unter Verwendung der Handgelenkskinematik und der komponentenspezifischen Abstandsfelder29.
- Berechnen Sie Entfernungsfelder für jedes 3D-Oberflächenmodell als volumetrisches Array von vorzeichenbehafteten Entfernungen von der Oberfläche.
- Berechnen Sie die Näherungswerte der Entfernungsfelder auf der Oberfläche der Polyethylenkappe und der radialen Komponente für jede Haltung.
- Um die Kontaktfläche zwischen den Komponenten zu erhalten, passen Sie diese Näherungswerte an die Auflösung des Erfassungssystems an (die Abstandsausschlussschwelle wurde in dieser Studie mit 0,70 mm gewählt).
- Statistische Analyse
- Verwenden Sie die Bland-Altman-Analyse, um die Genauigkeit zu bewerten, indem Sie die Verzerrung (mittlere Differenzen) und die 95%-Übereinstimmung (Verzerrung ± 1,96SD) zwischen BVR und OMCdemonstrieren 14, 30.
- Verwenden Sie verallgemeinerte lineare Modelle (GLM), um das Rotationszentrumsverhalten des gesunden Handgelenks zu beschreiben und eine Beziehung zwischen der Änderung von UV und PS (UV = p 1×PS2 + p2×PS) zu finden, wobei p1 und p2 Koeffizienten waren, die mit Hilfe der linearen Regression optimiert wurden. Verwenden Sie die RMSE-Metrik (Root-Mean-Squared-Error), um die Genauigkeit des Modells zu bewerten.
Ergebnisse
Die Auswahl der 2D-zu-3D-Bildregistrierungssoftware für das modellbasierte Tracking hängt zum Teil vom Zugriff auf Grafikprozessoreinheiten (GPU) und High-Performance-Computing-Systeme (HPC) ab. Diese Programme haben unterschiedliche Pipelines, und bis jetzt gibt es keine gemeinsame Methodik für die Programme. In dieser Studie verwenden wir Autoscoper, ein Open-Source-Programm zur 2D-zu-3D-Bildregistrierung, das an der Brown Universityentwickelt wurde 25. Die W...
Diskussion
Die biplanare Videoradiographie (BVR) ist eine bildbasierte Methode, mit der Knochen- und Implantatbewegungen im Handgelenk und im distalen Radioulnargelenk submillimeter- und subgradgenau gemessen werden können. In den Studien, die wir hier beschrieben haben, wurde die BVR verwendet, um ein genaues Muster der projizierten COR für ein gesundes Handgelenk sowie TWA-Kontaktmuster zu identifizieren. Solche Erkenntnisse können in das Design von Handgelenk-Totalprothesen der nächsten Gene...
Offenlegungen
Wir haben keinen Interessenkonflikt zu deklarieren.
Danksagungen
Die Autoren danken Josephine Kalshoven und Lauren Parola für die Überarbeitung des Protokolls. Die Autoren danken auch Erika Tavares und Rohit Badida für ihre Hilfe bei der Datenerfassung sowie Kalpit Shah, Arnold-Peter Weiss und Scott Wolfe für ihre Hilfe bei der Dateninterpretation. Diese Studie war möglich mit Unterstützung der National Institutes of Health P30GM122732 (COBRE Bio-engineering Core) und einem Zuschuss der American Foundation for Surgery of the Hand (AFSH).
Materialien
| Name | Company | Catalog Number | Comments |
| 3D Surface Scanner | Artec 3D | Artec Space SpiderTM | Luxembourg |
| Autoscoper | Brown University | https://simtk.org/projects/autoscoper | https://doi.org/10.1016/j.jbiomech.2019.05.040 |
| CT Scanner | General Electric (GE) | Lightspeed 16 | Milwaukee, WI, USA |
| Geomagic Wrap 3D | 3DSystems | Version 2017 | Rock Hill, SC, USA |
| Graphics Processing Unit (GPU) | Nvidia | GeForce GTX 1080 | CUDA-enabled GPU |
| High-speed Video Cameras | Phantom | Version 10 | Vision Research, Wayne, NJ, USA |
| Image Intensifier | Dunlee | 40 cm diameter | Aurora, IL, USA |
| ImageJ | Open-source (Brown University) | https://imagej.net/Fiji | https://doi.org/10.1038/nmeth.2019 |
| Matlab | The MathWorks, Inc. | R2017a to R2020a | Natick, MA, USA |
| Mimics | Materialise | Version 19.0 to 22.0 | Leuven, Belgium |
| Motion Capture Cameras | Qualisys | Oqus 5+ | Gothenburg, Sweden |
| Pulsed X-ray Generators | EMD Technologies | EPS 45–80 | Saint-Eustache, Quebec, QC, Canada |
| Undistortion Grid | McMaster-Carr | 9255T641 | Steel Perforated Sheet Staggered Holes, 0.048" Thk, 0.125" Hole Dia, 36" X 40" |
| Wrist Implant (In-vitro Study) | Integra LifeSciences | Universal 2 | Plainsboro, NJ, USA |
| Wrist Implant (In-vivo Study) | Integra LifeSciences | Freedom | Plainsboro, NJ, USA |
| WristViz | Open-source (Brown University) | https://github.com/DavidLaidlaw/WristVisualizer/tree/master | Open-source software |
| X-ray Tubes | Varian Medical Systems | Model G-1086 | Palo Alto, CA, USA |
| XMALab | Open-source (Brown University) | https://www.xromm.org/xmalab/ | https://doi.org/10.1242/jeb.145383 |
Referenzen
- Leardini, A., Chiari, L., Croce, U. D., Cappozzo, A. Human movement analysis using stereophotogrammetry: Part 3. Soft tissue artifact assessment and compensation. Gait & Posture. 21 (2), 212-225 (2005).
- Miranda, D. L., Rainbow, M. J., Crisco, J. J., Fleming, B. C. Kinematic differences between optical motion capture and biplanar videoradiography during a jump-cut maneuver. Journal of biomechanics. 46 (3), 567-573 (2013).
- Tashman, S., Anderst, W. In vivo measurement of dynamic joint motion using high speed biplane radiography and CT: application to canine ACL deficiency. Journal of Biomechanical Engineering. 125 (2), 238-245 (2003).
- Moore, D. C., et al. Computed Tomography Image-Based Kinematic Analysis: An Overview. Handbook of Imaging in Biological Mechanics. , 115-126 (2014).
- Fedorov, A., et al. 3D Slicer as an Image Computing Platform for the Quantitative Imaging Network. Magnetic resonance imaging. 30 (9), 1323-1341 (2012).
- Choi, Y. S., et al. Four-dimensional real-time cine images of wrist joint kinematics using dual source CT with minimal time increment scanning. Yonsei medical journal. 54 (4), 1026-1032 (2013).
- Boutin, R. D., et al. Real-Time Magnetic Resonance Imaging (MRI) during Active Wrist Motion-Initial Observations. PLoS ONE. 8 (12), 84004 (2013).
- Miranda, D. L., et al. Static and Dynamic Error of a Biplanar Videoradiography System Using Marker-Based and Markerless Tracking Techniques. Journal of Biomechanical Engineering. 133 (12), 121002-121008 (2011).
- Englander, Z. A., et al. In Vivo Anterior Cruciate Ligament Deformation During a Single-Legged Jump Measured by Magnetic Resonance Imaging and High-Speed Biplanar Radiography , In Vivo Anterior Cruciate Ligament Deformation During a Single-Legged Jump Measured by Magnetic Resonance Imaging and High-Speed Biplanar Radiography. The American Journal of Sports Medicine. 47 (13), 3166-3172 (2019).
- Englander, Z. A., Garrett, W. E., Spritzer, C. E., DeFrate, L. E. In vivo attachment site to attachment site length and strain of the ACL and its bundles during the full gait cycle measured by MRI and high-speed biplanar radiography. Journal of Biomechanics. 98, 109443 (2020).
- Anderst, W. J., Les, C., Tashman, S. In vivo serial joint space measurements during dynamic loading in a canine model of osteoarthritis. Osteoarthritis and cartilage: Osteoarthritis Research Society. 13 (9), 808-816 (2005).
- Bey, M. J., Kline, S. K., Zauel, R., Kolowich, P. A., Lock, T. R. In Vivo Measurement of Glenohumeral Joint Contact Patterns. EURASIP journal on advances in signal processing. 2010, (2010).
- Anderst, W. J., Tashman, S. A method to estimate in vivo dynamic articular surface interaction. Journal of Biomechanics. 36 (9), 1291-1299 (2003).
- Akhbari, B., et al. Accuracy of biplane videoradiography for quantifying dynamic wrist kinematics. Journal of Biomechanics. 92, 120-125 (2019).
- Akhbari, B., et al. Kinematic Accuracy in Tracking Total Wrist Arthroplasty with Biplane Videoradiography using a CT-generated Model. Journal of Biomechanical Engineering. , (2019).
- Akhbari, B., et al. Proximal-distal shift of the center of rotation in a total wrist arthroplasty is more than twice of the healthy wrist. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. 38 (7), 1575-1586 (2020).
- Wang, J., Blackburn, T. J. The AAPM/RSNA Physics Tutorial for Residents. RadioGraphics. 20 (5), 1471-1477 (2000).
- Knörlein, B. J., Baier, D. B., Gatesy, S. M., Laurence-Chasen, J. D., Brainerd, E. L. Validation of XMALab software for marker-based XROMM. The Journal of Experimental Biology. 219 (23), 3701-3711 (2016).
- Brainerd, E. L., et al. X-ray reconstruction of moving morphology (XROMM): precision, accuracy and applications in comparative biomechanics research. Journal of experimental zoology. Part A, Ecological genetics and physiology. 313 (5), 262-279 (2010).
- X-ray Camera Calibration. XROMM / XMALab / wiki Available from: https://bitbucket.org/xromm/xmalab/wiki/X-ray%20Camera%20Calibration (2020)
- Akhbari, B., et al. Biomechanics of the Distal Radioulnar Joint in In vivo Forearm Pronosupination. Journal of Wrist Surgery. , (2020).
- Akhbari, B., et al. Predicting carpal bone kinematics using an expanded digital database of wrist carpal bone anatomy and kinematics. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. , (2019).
- Moore, D. C., et al. Three-dimensional in vivo kinematics of the distal radioulnar joint in malunited distal radius fractures. The Journal of Hand Surgery. 27 (2), 233-242 (2002).
- Knörlein, B. J. Correct for Fluoroscope Distortion (X-ray images). XROMM / XMALab / wiki. , (2020).
- . Autoscoper Available from: https://simtk.org/projects/autoscoper (2019)
- Kennedy, J., Eberhart, R. . Particle swarm optimization. , (1995).
- Nelder, J. A., Mead, R. A Simplex Method for Function Minimization. The Computer Journal. 7, 308 (1965).
- Panjabi, M. A Mathematical Approach for three-dimensional analysis of the mechanics of the spine. J Biomechanics. 4, 203-211 (1971).
- Marai, G. E., et al. Estimating joint contact areas and ligament lengths from bone kinematics and surfaces. IEEE transactions on bio-medical engineering. 51 (5), 790-799 (2004).
- Bland, J. M., Altman, D. G. Measuring agreement in method comparison studies. Statistical methods in medical research. 8 (2), 135-160 (1999).
- . Camera calibration With OpenCV Available from: https://docs.opencv.org/master/index.html (2020)
- Anderst, W. J., Lee, J. Y., Kang, J. D. In Vivo Cervical Facet Joint Capsule Deformation During Flexion-Extension. Spine. 39 (8), 514-520 (2014).
- Anderst, W. J., Vaidya, R., Tashman, S. A technique to measure three-dimensional in vivo rotation of fused and adjacent lumbar vertebrae. The Spine Journal. 8 (6), 991-997 (2008).
- Mozingo, J. D., et al. Validation of imaging-based quantification of glenohumeral joint kinematics using an unmodified clinical biplane fluoroscopy system. Journal of Biomechanics. 71, 306-312 (2018).
- Akbari-Shandiz, M., et al. MRI vs CT-based 2D-3D auto-registration accuracy for quantifying shoulder motion using biplane video-radiography. Journal of Biomechanics. 82, 375-380 (2019).
- Bey, M. J., et al. In vivo shoulder function after surgical repair of a torn rotator cuff: glenohumeral joint mechanics, shoulder strength, clinical outcomes, and their interaction. The American journal of sports medicine. 39 (10), 2117-2129 (2011).
- Bey, M. J., Kline, S. K., Zauel, R., Kolowich, P. A., Lock, T. R. In Vivo Measurement of Glenohumeral Joint Contact Patterns. EURASIP journal on advances in signal processing. 2010, (2010).
- Bey, M. J., Zauel, R., Brock, S. K., Tashman, S. Validation of a New Model-Based Tracking Technique for Measuring Three-Dimensional, In Vivo Glenohumeral Joint Kinematics. Journal of biomechanical engineering. 128 (4), 604-609 (2006).
- Fu, E., et al. Elbow Position Affects Distal Radioulnar Joint Kinematics. The Journal of Hand Surgery. 34 (7), 1261-1268 (2009).
- Martin, D. E., et al. Model-Based Tracking of the Hip: Implications for Novel Analyses of Hip Pathology. The Journal of Arthroplasty. 26 (1), 88-97 (2011).
- Miranda, D. L., et al. Static and Dynamic Error of a Biplanar Videoradiography System Using Marker-Based and Markerless Tracking Techniques. Journal of Biomechanical Engineering. 133 (12), 121002-121008 (2011).
- Stentz-Olesen, K., et al. Validation of static and dynamic radiostereometric analysis of the knee joint using bone models from CT data. Bone & Joint Research. 6 (6), 376-384 (2017).
- Anderst, W., Zauel, R., Bishop, J., Demps, E., Tashman, S. Validation of Three-Dimensional Model-Based Tibio-Femoral Tracking During Running. Medical engineering & physics. 31 (1), 10-16 (2009).
- Kessler, S. E., et al. A Direct Comparison of Biplanar Videoradiography and Optical Motion Capture for Foot and Ankle Kinematics. Frontiers in Bioengineering and Biotechnology. 7, 199 (2019).
- Maharaj, J. N., et al. The Reliability of Foot and Ankle Bone and Joint Kinematics Measured With Biplanar Videoradiography and Manual Scientific Rotoscoping. Frontiers in Bioengineering and Biotechnology. 8, 106 (2020).
- Ito, K., et al. Direct assessment of 3D foot bone kinematics using biplanar X-ray fluoroscopy and an automatic model registration method. Journal of Foot and Ankle Research. 8, 21 (2015).
- Wang, B., et al. Accuracy and feasibility of high-speed dual fluoroscopy and model-based tracking to measure in vivo ankle arthrokinematics. Gait & Posture. 41 (4), 888-893 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten