Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Vidéoradiographie biplanaire pour étudier le poignet et les articulations radio-ulnaires distales
Dans cet article
Résumé
La vidéoradiographie biplanaire (BVR) est une technique d’imagerie avancée permettant de comprendre le mouvement tridimensionnel des os squelettiques et des implants. Combinant des volumes d’images basés sur la densité et des vidéoradiographies du membre supérieur distal, la BVR est utilisée pour étudier le mouvement in vivo du poignet et de l’articulation radio-ulnaire distale, ainsi que les arthroplasties articulaires.
Résumé
Une mesure précise de la cinématique squelettique in vivo est essentielle pour comprendre le fonctionnement normal des articulations, l’influence de la pathologie, la progression de la maladie et les effets des traitements. Les systèmes de mesure qui utilisent des marqueurs de surface de la peau pour déduire le mouvement squelettique ont fourni des informations importantes sur la cinématique normale et pathologique, cependant, une arthrocinématique précise ne peut pas être obtenue à l’aide de ces systèmes, en particulier lors d’activités dynamiques. Au cours des deux dernières décennies, les systèmes de vidéoradiographie biplanaire (BVR) ont permis à de nombreux chercheurs d’étudier directement la cinématique squelettique des articulations pendant les activités de la vie quotidienne. Pour mettre en œuvre des systèmes BVR pour le membre supérieur distal, des vidéoradiographies du radius distal et de la main sont acquises à partir de deux sources de rayons X calibrées pendant qu’un sujet effectue une tâche désignée. Les positions tridimensionnelles (3D) des corps rigides sont calculées à partir des vidéoradiographies via un enregistrement optimal des projections de modèles 3D sur chaque vue BVR. Les modèles 3D sont des volumes d’image basés sur la densité de l’os spécifique dérivés de données de tomodensitométrie acquises indépendamment. Utilisant des processeurs graphiques et des systèmes de calcul haute performance, cette approche de suivi basée sur un modèle s’avère rapide et précise dans l’évaluation de la biomécanique du poignet et de l’articulation radio-ulnaire distale. Dans cette étude, nous avons d’abord résumé les études précédentes qui ont établi la concordance submillimétrique et submillimétrique de la BVR avec un système de capture de mouvement optique in vitro dans l’évaluation de la cinématique du poignet et de l’articulation radio-ulnaire distale. De plus, nous avons utilisé la BVR pour calculer le comportement du centre de rotation de l’articulation du poignet, pour évaluer le modèle d’articulation des composants de l’implant les uns sur les autres et pour évaluer le changement dynamique de la variance ulnaire pendant la pronosupination de l’avant-bras. À l’avenir, les os du carpe pourraient être capturés plus en détail grâce à l’ajout de détecteurs de rayons X à écran plat, d’un plus grand nombre de sources de rayons X (c’est-à-dire la vidéoradiographie multiplanaire) ou d’algorithmes avancés de vision par ordinateur.
Introduction
Une mesure précise de la cinématique squelettique in vivo est essentielle pour comprendre la fonction articulaire saine et remplacée, l’influence de la pathologie, la progression de la maladie et les effets des traitements. La quantification de la cinématique squelettique de manière non invasive à la surface de l’articulation (arthrocinématique) est cruciale pour comprendre les pathologies et les maladies articulaires, telles que l’arthrose, mais elle est techniquement difficile. Auparavant, les techniques qui utilisent des marqueurs de surface cutanée pour déduire le mouvement squelettique ont fourni des informations importantes sur la cinématique saine et pathologique. Cependant, il n’est pas possible d’obtenir une arthrocinématique précise à l’aide de ces techniques, en particulier lors d’activités dynamiques telles que les activités de la vie quotidienne. La précision de ces systèmes optiques est intrinsèquement limitée en raison du mouvement de la peau par rapport aux os sous-jacents, principale source d’erreur dans l’analyse du mouvement humain 1,2.
Les méthodes de pointe actuelles pour quantifier la cinématique squelettique tridimensionnelle (3D) sont le suivi basé sur l’image, à savoir la vidéoradiographie biplan (BVR)3 et la tomodensitométrie en série (TDM) volumes4 et l’imagerie par résonance magnétique (IRM)5. Bien que les technologies habituelles de tomodensitométrie et d’IRM 3D soient très précises et accessibles dans de nombreux hôpitaux à travers le monde, elles sont incapables de mesurer le mouvement dynamique des articulations. Des techniques d’imagerie telles que la tomodensitométrie 4D6 et l’IRM dynamique7 ont été développées ces dernières années pour remédier à cette lacune. Cependant, ces méthodes exposent les patients à une forte dose de rayonnement ou souffrent d’une faible résolution temporelle.
En combinant de nouveaux algorithmes de vision par ordinateur et des systèmes à rayons X traditionnels, la BVR s’est avérée précise pour plusieurs articulations chez les animaux et les humains. résolu à l’aide d’algorithmes de suivi basés sur des marqueurs ou sur des modèles. Les approches basées sur des marqueurs suivent les billes de tantale insérées dans les os ou les tissus mous et sont optimales pour les tests sur les animaux et in vitro. Cependant, ils sont prohibitifs pour la recherche humaine in vivo . Heureusement, les améliorations apportées aux algorithmes de suivi basés sur des modèles offrent une alternative viable. Chez l’homme, les approches de suivi BVR basées sur un modèle consistent à préparer les ensembles d’images volumétriques acquises par TDM ou IRM dans une posture statique et à capturer les mouvements des intérêts dans le champ de vision de deux rayons X. La plupart des applications de suivi basées sur des modèles génèrent ensuite des radiographies reconstruites numériquement (RRC) de l’os ou de l’implant à partir des images statiques de TDM ou d’IRM et les associent à des vidéoradiographies améliorées à l’aide de mesures qui démontrent la similitude entre les RRM et les vidéoradiographies8. Ce processus s’appelle le « suivi » de l’os ou de l’implant.
Les principales variables de sortie du suivi des os ou des implants sont la cinématique des corps rigides, à partir de laquelle la cinématique des articulations, les allongements ligamentaires 9,10, l’espacement des articulations comme substitut de l’épaisseur du cartilage11, le contact articulaire12,13 et d’autres biomarqueurs peuvent être calculés. Récemment, nous avons documenté la précision du suivi BVR basé sur un modèle dans le calcul de la biomécanique du poignet, de l’arthroplastie totale du poignet (TWA) et de l’articulation radio-ulnaire distale (DRUJ)14,15. Dans la section suivante, un protocole détaillé de cette méthode validée pour l’étude du mouvement du poignet squelettique, de l’arthroplastie totale du poignet et de l’articulation radio-ulnaire distale lors de diverses tâches est présenté. Nous segmentons les volumes d’image basés sur la densité des os et des implants à partir des volumes d’images CT, suivons ces volumes d’image partiels dans les vidéoradiographies et déterminons des résultats tels que le centre de rotation, le modèle de contact et la variance ulnaire pour démontrer les forces et les limites de cette méthode.
Protocole
Cette étude a été approuvée par l’Institutional Review Board (IRB) de Lifespan - Rhode Island Hospital, un IRB accrédité par l’AAHRPP. Au total, 16 patients ont fourni un consentement éclairé signé conformément aux directives de l’établissement.
1. Acquisition de données
- Tomodensitométrie (TDM)
- Préparez les échantillons ou les sujets pour le scanner.
REMARQUE : Pour l’évaluation de la précision14,15, 6 avant-bras intacts provenant de quatre spécimens de cadavres intacts (70,5 ± 12,3 ans, 4 droits, 2 femelles) et 1 avant-bras (49 ans, droit, femelle) avec une arthroplastie totale du poignet ont été utilisés. Pour l’étude in vivo 16, 10 volontaires avec des poignets sains (57,0 ± 5,2 ans, huit femmes, neuf dominants droitiers) et 6 patients (74,7 ± 5,6 ans, deux femmes, deux poignets droits, quatre poignets dominants) ayant subi une arthroplastie totale du poignet ont été recrutés. - Pour imager chaque sujet4, acquérir une tomodensitométrie de l’avant-bras distal avec un diamètre de collecte de données de 25 cm et reconstruire avec un champ de vision de 20 cm à l’aide de l’algorithme de convolution Bone Plus à l’aide d’un scanner clinique à 16 coupes. Utilisez des réglages de tube de 80 kVp et 80 mA. Limitez l’exposition de l’avant-bras à environ 7 cm proximal de la surface du radius distal aux phalanges distales.
REMARQUE : Dans cette étude, les dimensions du voxel de l’image étaient de 0,39 × 0,39 mm2 dans le plan transversal de l’avant-bras et de 0,625 mm le long de l’axe long de l’avant-bras. - Transférez les images CT acquises sur l’ordinateur du laboratoire à l’aide d’un protocole approuvé par HIPPA au format DICOM (Digital Imaging and Communications in Medicine).
- Préparez les échantillons ou les sujets pour le scanner.
- Vidéoradiographie biplanaire (BVR)
- Dispositif expérimental (Figure 1). Pour permettre un diamètre de champ de vision capable de capturer le radius distal, le cubitus distal, les os du carpe et les métacarpiens ainsi qu’une perspective permettant de minimiser les occlusions osseuses dans diverses postures du poignet, positionnez les intensificateurs d’image (II) sous les avant-bras et réglez l’angle entre eux à 110° (sources de rayons X dirigées vers le bas vers les II correspondants à 55° par rapport au sol).
- Générez les rayons X en mode continu avec un réglage d’exposition compris entre 65 et 75 kV, en fonction de la visibilité des os/implants, et 80 mA. Ajustez les distances source-image des deux paires X-Ray-II à environ 130 cm et les distances source-main à 90 cm.
REMARQUE : Dans cette étude, les sujets portaient un tablier de plomb de protection contre les rayons X, un collier de protection thyroïdienne et un dosimètre numérique en laboratoire. Tous les chercheurs se tenaient derrière un verre de protection en plomb pendant l’expérience. - Placez un tréteau ou une structure similaire entre les II, et utilisez-le pour placer des objets (par exemple, une poignée de porte, un marteau, un pichet) pour les sujets.
- Générez les rayons X en mode continu avec un réglage d’exposition compris entre 65 et 75 kV, en fonction de la visibilité des os/implants, et 80 mA. Ajustez les distances source-image des deux paires X-Ray-II à environ 130 cm et les distances source-main à 90 cm.
- Dispositif expérimental (Figure 1). Pour permettre un diamètre de champ de vision capable de capturer le radius distal, le cubitus distal, les os du carpe et les métacarpiens ainsi qu’une perspective permettant de minimiser les occlusions osseuses dans diverses postures du poignet, positionnez les intensificateurs d’image (II) sous les avant-bras et réglez l’angle entre eux à 110° (sources de rayons X dirigées vers le bas vers les II correspondants à 55° par rapport au sol).
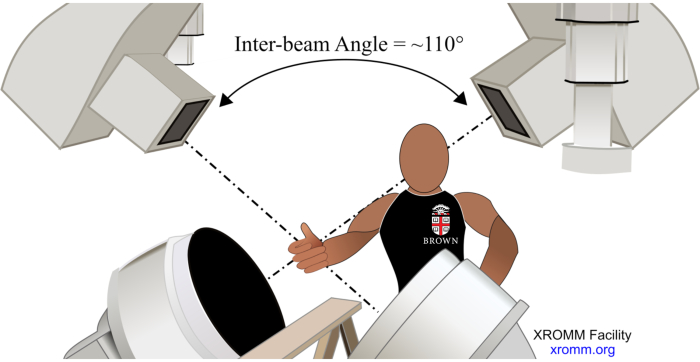
Graphique 1. Dispositif expérimental. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Grille d’absence de distorsion (Figure 2A). Étant donné que les champs électromagnétiques externes affectent le comportement des faisceaux de rayons X (c’est-à-dire les trajets d’électrons), les images radiographiques souffrent de distorsions non linéaires en coussinets et en forme de S17. Pour éliminer ces distorsions, utilisez l’image d’une pièce plate de tôle perforée de précision (c’est-à-dire une grille sans distorsion)18,19. Placez des grilles sans distorsion sur les II et capturez leurs images avant et après les données de vidéoradiographie. Enregistrez ces images en tant qu’images TIFF 8 bits pour les deux sources de rayons X.
- Étalonnage (Figure 2B). L’étalonnage des sources de rayons X est nécessaire pour calculer les coordonnées 3D des objets (os ou implants)20. Utilisez des images d’un objet 3D à la géométrie connue, qui comporte des sphères radio-opaques régulièrement espacées, pour générer une relation entre la position et l’orientation de la source de rayons X dans l’espace 3D et l’espace de l’image (c’est-à-dire les radiographies). Un objet cubique (c’est-à-dire un cube d’étalonnage) fabriqué à partir de feuilles acryliques et d’entretoises avec des sphères d’acier est efficace et précis pour les réglages BVR19.
- Capturez des radiographies du cubed’étalonnage 18 dans différentes orientations 4 fois avant de réaliser l’expérience et 4 fois après l’expérience. Les étalonnages post-expérience servent à confirmer que la configuration expérimentale reste inchangée tout au long de l’expérience. Enregistrez toutes les images en tant qu’images TIFF 8 bits pour les deux sources de rayons X.
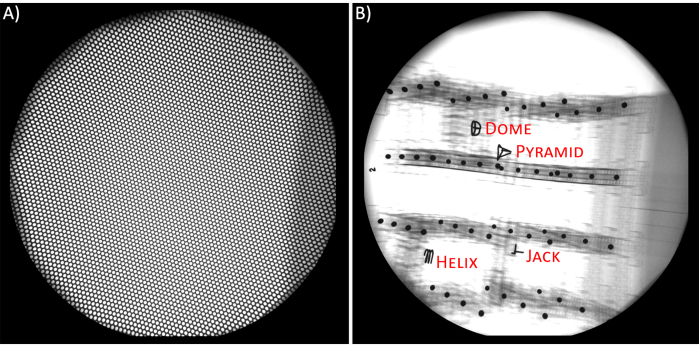
Graphique 2. A) Grille sans distorsion. B) Cube d’étalonnage et ses éléments de référence. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Description des tâches. Définissez diverses tâches pour capturer le mouvement du poignet lors de la flexion-extension, de la déviation radiale-ulnaire et de la circumduction16. Décrire les tâches de mouvement DRUJ comme la rotation de l’avant-bras d’une posture de maintien à une pronation complète ou à une supination complète, définie comme la rotation d’une poignée en T dans le sens des aiguilles d’une montre et dans le sens inverse des aiguilles d’une montre21. Acquérir des vidéoradiographies du poignet dans sa pose neutre (c’est-à-dire une tâche statique neutre). Capturez la pose neutre avec le dos de la main coplanaire avec l’arrière de l’avant-bras.
REMARQUE : Dans la posture de départ pour toutes les tâches, l’épaule était en adduction, le coude était soutenu au niveau de l’articulation et l’avant-bras était sans contrainte et dans sa rotation neutre. Les tâches de flexion-extension et de déviation radiale-ulnaire ont été décrites comme la rotation de la paume par rapport aux côtés palmaire-dorsal et radial-ulnaire de la main, respectivement. La circumduction a été décrite comme le mouvement du poignet qui visait à atteindre une amplitude de mouvement active maximale dans toutes les directions. - Acquisition de vidéoradiographies. Acquérez des vidéoradiographies à une fréquence de 200 Hz avec la vitesse d’obturation de la caméra réglée à 500 μs. Enregistrez deux secondes d’imagerie pour chaque tâche afin d’acquérir 2 répétitions. Stockez les vidéoradiographies acquises à partir de chaque source de rayons X sous forme de vidéo CINE dans un format 8 bits.
REMARQUE : Dans cette étude, les radiographies avaient une taille d’image de 1760×1760 pixels et avaient une résolution d’environ 0,22 mm×0,22 mm par pixel.
REMARQUE : Les sources de rayons X actuelles ont une option pour agrandir le champ de vision (FOV). Cependant, dans cette étude, le mode agrandi offert par le matériel n’a pas été utilisé. D’après la description de la tâche, cette fonctionnalité peut vous permettre d’acquérir des sorties de résolution plus élevée. Si cette fonction est utilisée, les étapes Grille de non-distorsion et Étalonnage doivent être capturées dans ce mode.
- Capture optique de mouvement (OMC) - étude de précision
- Utilisez le protocole d’acquisition de données de capture de mouvement optique (OMC) décrit dans des études précédentes14,15. Fixez directement deux grappes de marqueurs, chacune avec quatre sphères de marqueur rétroréfléchissantes de 9,5 mm de diamètre, au troisième métacarpien et au radius. Capturez le mouvement des marqueurs à l’aide de 8 caméras de capture de mouvement optiques. Synchronisez le démarrage de l’acquisition des données OMC avec le BVR par un déclencheur externe faible actif.
2. Traitement des données
- Tomodensitométrie (TDM)
- Anonymisation DICOM. Anonymisez les DICOM et importez-les dans un logiciel de traitement d’images pour la modélisation 3D (par exemple, Mimics Materialise). Dans le logiciel Mimics Materialise, dans la barre d’outils Fichier , ouvrez la fenêtre Anonymiser . Lorsque la nouvelle fenêtre est ouverte, sélectionnez Anonymiser toutes les images dans l’onglet de gauche, puis appuyez sur le bouton Anonymiser en bas à droite.
- Segmentation (figure 3). Segmenter les os ou les implants d’intérêt en suivant une méthodologie décrite précédemment 16,22,23.
- En bref, dans la barre d’outils Segment , sélectionnez l’option Seuil , puis choisissez les ensembles de seuils prédéfinis pour l’os à l’ouverture de la fenêtre. Une fois le masque seuillé créé, utilisez l’option Masquer scindé et peignez manuellement les régions des os ou les composants de l’implant.
- Lorsque toutes les régions sont colorées, cliquez sur OK et attendez que le logiciel génère automatiquement les masques pour chaque os et le composant de l’implant. Vérifiez tous les masques pour vous assurer qu’ils couvrent complètement les régions prévues. Si ce n’est pas le cas, utilisez l’option Modifier les masques pour modifier les régions manuellement et une par une.
REMARQUE : Pour étudier l’articulation du poignet, les 2eet 3emétacarpiens et les os du radius doivent être segmentés et séparés du reste des os. En option, le capitate et d’autres os peuvent être segmentés pour les étapes de post-traitement.
REMARQUE : Pour étudier le DRUJ, le radius et le cubitus doivent être segmentés et séparés du reste des os.
REMARQUE : Pour les implants, le composant radial et le composant carpien avec ses vis doivent être segmentés.
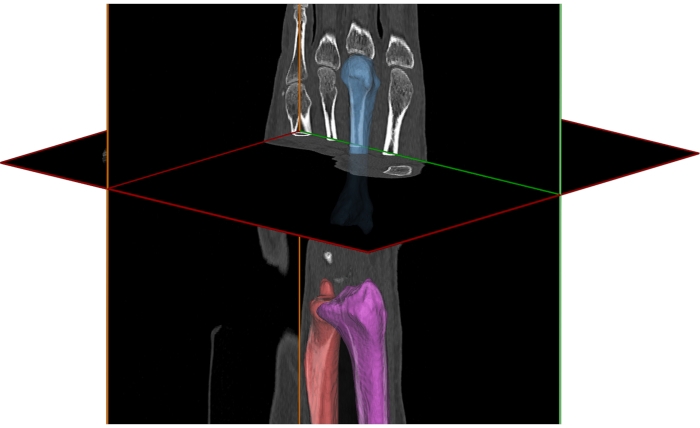
Graphique 3. Image par tomodensitométrie du poignet et modèles reconstruits du radius, du troisième métacarpien et du cubitus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Segmentation - composante radiale uniquement. En raison des artefacts de traînée basés sur la densité de la surface de la composante radiale dans les images CT, le modèle de composante radiale ne peut pas être segmenté à partir des images. Ainsi, utilisez un modèle CAO ou un scanner de surface 3D pour générer le modèle 3D du composant radial explanté. Enregistrez ce modèle dans la segmentation approximative de la composante radiale à l’aide de la méthode par défaut de Mimics Materialise.
REMARQUE : Dans cette étude, nous avons utilisé un scanner de surface d’une résolution de 0,1 mm. L’implant a été placé sur une table et le scanner 3D a été déplacé manuellement autour de l’implant pour capturer toutes les surfaces externes. - Volumes d’images partiels. Le logiciel de suivi nécessite qu’un corps d’intérêt segmenté soit saisi en tant que volume d’image basé sur la densité. Pour créer ce volume partiel, générez un masque complet du volume d’image contenant toutes les valeurs grises en sélectionnant l’option Seuil dans la barre d’outils Segment . Ajustez les valeurs de seuil de la valeur minimale aux valeurs de gris maximales.
- Ensuite, utilisez l’option Opérateurs booléens et soustrayez le volume segmenté de l’os ou de l’implant d’intérêt du masque complet généré. Changez la couleur du masque de ce volume partiel en noir (RVB : 0.0/0.0/0.0) car le logiciel d’enregistrement n’accepte que les volumes d’images en niveaux de gris. Exportez le masque en tant que DICOM en sélectionnant Exporter DICOM... dans le logiciel. La sortie est un volume d’image de l’os isolé ou de l’implant.
- Modèles tridimensionnels. Convertissez les masques segmentés en modèles de surface 3D maillés triangulés en sélectionnant l’option Calculer 3D , avec la résolution optimale. Exportez ces modèles au format VRML, contenant l’emplacement 3D des sommets (points) et leur ordre de connexions. Les modèles sont générés pour la construction du système de coordonnées et une visualisation plus poussée.
- Prétraitement de la vidéoradiographie
- Importation XMALab18 . Pour chaque sujet, créez un nouvel essai dans le logiciel XMALab. Attribuez les chemins d’accès aux dossiers aux images de la grille de non-distorsion et à 8 ensembles d’images d’étalonnage dans la boîte de dialogue Nouvelle version d’essai .
REMARQUE : Si les sources de rayons X sont inversées tout au long de la capture, l’option de retournement doit être cochée dans le logiciel XMALab. - Dé-distorsion. Pour déformer les radiographies, une fois l’essai créé et le logiciel ayant ouvert les images de grille dans l’espace de travail Undistortion, cliquez sur le bouton Compute Undistortion . XMALab détermine et affiche automatiquement les images déformées des grilles. Une fois que les centres de tous les trous ont été détectés par le logiciel, remplacez l’espace de travail dans le menu en haut à gauche par l’espace de travail Étalonnage . Pour plus d’informations et pour obtenir de l’aide sur le débogage des problèmes potentiels, consultez la référence24.
- Calibrage. À ce stade, le logiciel XMALab détermine les matrices d’étalonnage. Dans XMALab, les sources de rayons X sont modélisées comme des caméras à sténopé avec des paramètres intrinsèques et extrinsèques, tels que la distance focale, le centre de la source et les distances source-image, et l’étape d’étalonnage est un processus qui détermine ces métriques18. Pour calibrer, sélectionnez chacun des 4 éléments de référence placés dans le cube de calibration pour tous les ensembles d’images et pour chaque vue. Dans la configuration, ces références étaient Pyramid, Jack, Dome et Helix (Figure 2B). Si le logiciel ne trouve pas automatiquement les points, appuyez sur Compute Calibration. Une fois toutes les images d’étalonnage étalonnées, sélectionnez Optimiser la configuration de l’appareil photo.
- Exportation des matrices d’étalonnage. Après l’optimisation, triez les 8 jeux d’étalonnage (c’est-à-dire les 4 ensembles qui ont été capturés avant l’expérience et les 4 ensembles qui ont été capturés après l’expérience) et sélectionnez l’ensemble d’images avec la plus petite erreur non déformée. Trouvez cette valeur en appuyant sur l’option d’information dans le coin supérieur droit de chaque fenêtre d’étalonnage. Sur l’image avec l’erreur la plus faible, exportez les images en sélectionnant l’option MayaCams 2.0 dans le menu Fichier-Exportation . Pour plus d’informations et pour déboguer d’éventuels problèmes, consultez la référence20.
- Vidéoradiographie sans distorsion. Changez l’espace de travail en Suivi des marqueurs et chargez les vidéoradiographies qui ont été capturées au format CNE en appuyant sur l’icône +. Une fois le processus de chargement terminé, exportez les images d’essai non déformées en sélectionnant Images d’essai non déformées dans le menu Exportation de fichiers .
- Importation XMALab18 . Pour chaque sujet, créez un nouvel essai dans le logiciel XMALab. Attribuez les chemins d’accès aux dossiers aux images de la grille de non-distorsion et à 8 ensembles d’images d’étalonnage dans la boîte de dialogue Nouvelle version d’essai .
- Suivi basé sur un modèle (Figure 4)
- Amélioration des caractéristiques des radiographies/RRM (Figure 4A-B). L’amélioration des caractéristiques de l’image à l’aide de filtres est une étape nécessaire pour obtenir une correspondance précise. Définissez les paramètres des quatre filtres, y compris le contraste (détection de l’intensité), le Sobel (détection des bords), le gaussien (flou/plus lisse) et la netteté (mise en gras des bords) dans le logiciel. Ces filtres peuvent être sélectionnés en cliquant avec le bouton droit de la souris sur les objets Rad Renderer ou DRR Renderer affichés dans le widget en haut à gauche.
REMARQUE : Dans cette étude, les bords des images des os sur les radiographies ont été améliorés à l’aide d’un filtre de Sobel avec un facteur d’échelle de 3 et une valeur de mélange de 0,4. De plus, un filtre de contraste avec un alpha (pour le contraste de l’image) de 2,5 et un bêta (pour la luminosité de l’image) de 0,9 a été utilisé pour améliorer encore la qualité des radiographies. Pour faire correspondre les DRR avec la radiographie, une valeur d’intensité de rayon de 0,35 a été utilisée avec un filtre Sobel avec une valeur de mélange de 0,1 et un facteur d’échelle de 1,7. - Initialisation manuelle. Déplacez la RRC de chaque os et composant à l’aide de son point de pivot pour obtenir la meilleure correspondance possible entre la RRC et les radiographies. Le point de pivot aide les utilisateurs à faire pivoter et à traduire la DRR sélectionnée à partir du widget en bas à gauche. Cette étape est nécessaire car la position de l’os ou de l’implant doit être initialisée au moins une fois, et au moins pour la première image, avant d’effectuer l’optimisation. Appuyez sur W pour déplacer la DRR en mode traduction et appuyez sur E pour faire pivoter la DRR.
REMARQUE : Il n’est généralement pas nécessaire de modifier l’emplacement du point de pivot, mais cela est possible à l’aide de l’option Déplacer le pivot de la barre d’outils. - Optimisation. Appuyez sur C ou cliquez sur le bouton Suivre le courant dans la barre d’outils pour trouver la meilleure correspondance entre la DRR et les radiographies. La procédure par défaut du logiciel utilise la fonction de coût de corrélation croisée normalisée (NCC) et la méthode d’optimisation de l’essaim de particules et les procédures simplex descendantes26,27. Ce processus est également appelé l’étape de correspondance ou de suivi. La procédure d’optimisation peut également être exécutée via la boîte de dialogue de suivi. Pour ce faire, appuyez sur l’option Boîte de dialogue de suivi dans la barre d’outils. Lorsque la nouvelle fenêtre est ouverte, remplacez les options À partir de l’image et À l’image sur 0, puis appuyez sur OK.
REMARQUE : La NCC est une fonction qui mesure la similitude entre les radiographies et les DRR, et une valeur NCC plus proche de 0 a démontré une meilleure correspondance. Cependant, l’obtention de cette valeur dépend entièrement des filtres d’image et des qualités de capture. La valeur de NCC peut être observée dans la « Fenêtre du terminal ». - Processus de jumelage - Premier passage. Une fois que la première image est automatiquement optimisée, ouvrez la boîte de dialogue de suivi, remplacez l’image De l’image par 0 et remplacez l’image de destination par le dernier numéro de l’image (situé en bas à droite). Le processus de correspondance par défaut du logiciel utilise la trame actuelle comme position initiale de la DRR pour le processus de correspondance. Le processus d’optimisation utilise la méthode d’optimisation par essaim de particules (PSO) dans une région de 3 mm et 3° de la position initiale.
- Processus d’appariement - Passage final (figure 4C). Étant donné que le processus de correspondance automatique peut échouer en raison d’une initialisation ou de filtres médiocres, vérifiez manuellement toutes les trames pour vous assurer que l’optimisation a été exécutée correctement. Si la RRC et les radiographies ne sont pas correctement appariées, répétez les étapes décrites aux sections 2.3.2 et 2.3.3.
REMARQUE : Le suivi du poignet a d’abord été effectué avec un modèle qui incluait les deuxième et troisième métacarpiens (MC2-MC3) pour réduire les effets de chevauchement osseux et améliorer la précision de l’appariement. Après avoir localisé la position et la rotation combinées de la RRC dans les radiographies, les emplacements MC2-MC3 appariés et optimisés ont été transformés en volume d’image partiel MC3, et les positions initiales du MC3 ont été amorcées. En utilisant ces positions de tête, le MC3 a été suivi. À l’instar de MC3, le rayon, le cubitus, la composante radiale et la composante carpienne dans les vidéoradiographies pendant toutes les tâches ont également été suivis.
- Amélioration des caractéristiques des radiographies/RRM (Figure 4A-B). L’amélioration des caractéristiques de l’image à l’aide de filtres est une étape nécessaire pour obtenir une correspondance précise. Définissez les paramètres des quatre filtres, y compris le contraste (détection de l’intensité), le Sobel (détection des bords), le gaussien (flou/plus lisse) et la netteté (mise en gras des bords) dans le logiciel. Ces filtres peuvent être sélectionnés en cliquant avec le bouton droit de la souris sur les objets Rad Renderer ou DRR Renderer affichés dans le widget en haut à gauche.
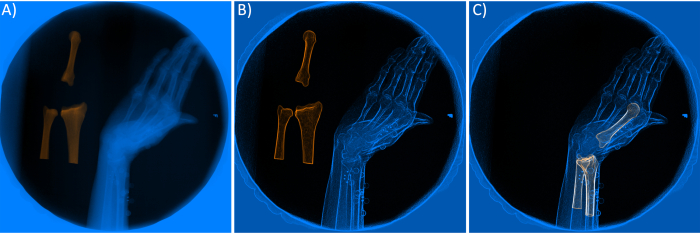
Graphique 4. A) Radiographie capturée d’une source de rayons X avec radiographies reconstruites numériquement (RRC) des os. B) Radiographie améliorée (filtrée) et RRC. C) TRRC appariées après le processus d’optimisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Réduction des données. En fonction de la fréquence d’images et de la qualité des images radiographiques, des approches de filtrage telles que la méthode de la moyenne mobile (avec une portée de 5 images) ont pu être utilisées. Pour ce faire, dans le menu de la barre d’outils, sélectionnez Suivi-Boîte de dialogue avancée, puis cliquez sur le bouton Lisser . Cette approche a été utilisée pour toutes les captures de cette étude.
- Exportation des positions osseuses/implantaires. Une fois que chaque os est adapté aux radiographies et que toutes les images sont optimisées, exportez les matrices de transformation 4x4 des DRR dans le monde de la radiologie en cliquant sur le bouton Enregistrer le suivi et en sélectionnant la sortie du format de matrice. Cette matrice de transformation définit la rotation et la translation du volume d’image partiel de l’os/de l’implant dans le système de coordonnées du monde à rayons X calibré et peut être utilisée pour un traitement supplémentaire et le calcul du mouvement. Le système de coordonnées du volume partiel de l’image doit être transféré au système de coordonnées anatomiques pour une analyse biomécanique significative.
3. Analyse des données
- Systèmes de coordonnées (Figure 5). Pour rendre compte de la cinématique, construisez des systèmes de coordonnées (CS) pour les os et les composants de l’implant en fonction de leurs repères anatomiques et géométriques. Une fois ces CS construits, enregistrez les modèles qui se trouvent dans le nouveau CS dans les modèles de surface qui ont été générés dans la Section 2.1.4 à l’aide de l’algorithme itératif du point le plus proche. Ce recalage un-à-un calcule une nouvelle matrice de transformation 4x4 qui sera utilisée pour les calculs cinématiques.
REMARQUE : Dans cette étude, le CS du radius, du cubitus, du troisième métacarpien, du composant carpien, du composant radial, du chapeau de polyéthylène et du capitate ont été créés à l’aide de méthodes qui ont été décrites précédemment 16,22,23.
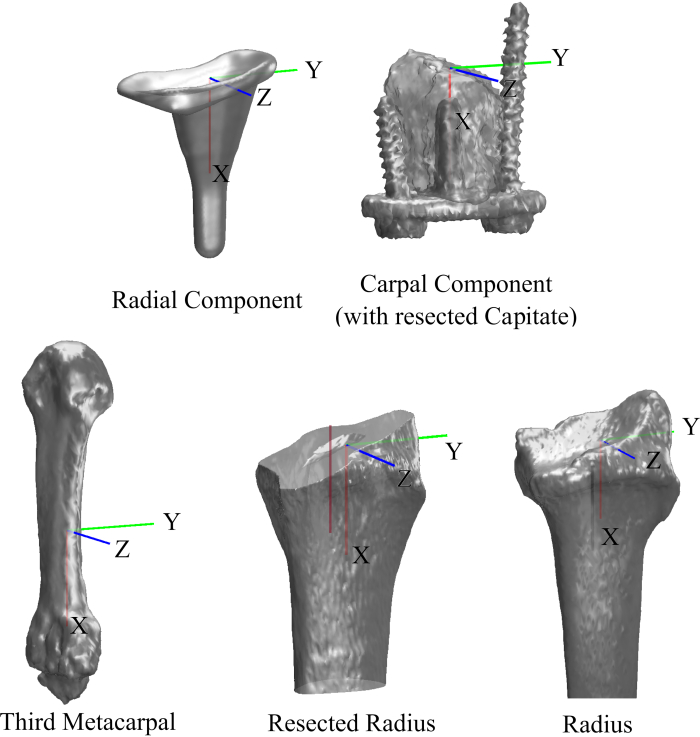
Graphique 5. Systèmes de coordonnées des os et des composants de l’implant. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Cinématique. Dans cette étude, on définit le mouvement du poignet comme le mouvement du MC3 par rapport au rayon, et les données cinématiques ont été rapportées par rapport à la position neutre du poignet.
- Pour l’analyse DRUJ, calculez les paramètres d’axe de mouvement hélicoïdal (HAM)28 pour décrire la cinématique comme le mouvement relatif du rayon CS dans le CS ulnaire, par rapport à la pose neutre.
- Utilisez l’analyse HAM pour déterminer le centre de rotation (COR) du poignet en flexion-extension et en déviation radiale-ulnaire.
- Pour décrire le décalage proximal-distal unidimensionnel du COR, définissez le COR projeté pour les poignets sains comme un point sur l’axe central de l’os capité qui avait la distance la plus courte de l’axe de rotation de la vis16.
- Décomposez la rotation HAM dans le système de coordonnées ulnaires à l’aide de l’axe de vis HAM, et calculez la pronation (positive) et la supination (négative) de l’avant-bras. La pose neutre a été définie lorsque la pronation-supination (PS) était nulle.
- Enfin, définissez la variance ulnaire (UV) comme la distance entre la fovéa ulnaire et l’encoche sigmoïde sur le rayon.
- Modèle d’articulation
- Calculez les modèles de contact entre le capuchon en polyéthylène et le composant radial à l’aide de la cinématique du poignet et des champs de distance spécifiques au composant29.
- Calculez les champs de distance pour chaque modèle de surface 3D sous la forme d’un tableau volumétrique de distances signées par rapport à la surface.
- Calculez les valeurs de proximité des champs de distance sur la surface du capuchon en polyéthylène et de la composante radiale pour chaque posture.
- Pour obtenir la surface de contact entre les composants, ajustez ces valeurs de proximité à la résolution du système d’acquisition (le seuil d’exclusion de distance a été sélectionné à 0,70 mm dans cette étude).
- Analyse statistique
- Utilisez l’analyse de Bland-Altman pour évaluer l’exactitude en démontrant le biais (différences moyennes) et la limite de concordance à 95 % (biais ± 1,96 écart-type) entre le BVR et l’OMC14, 30.
- Utilisez des modèles linéaires généralisés (GLM) pour décrire le comportement du centre de rotation du poignet sain et pour trouver une relation entre la variation des UV et de la PS (UV = p1×PS2 + p2×PS), où p1 et p2 étaient des coefficients optimisés à l’aide de la régression linéaire. Utilisez la métrique d’erreur quadratique moyenne (RMSE) pour évaluer la précision du modèle.
Résultats
Le choix d’un logiciel de recalage d’images 2D-3D pour le suivi basé sur le modèle dépend en partie de l’accès à l’unité de processeur graphique (GPU) et aux systèmes de calcul haute performance (HPC). Ces programmes ont des pipelines différents, et à l’heure actuelle, il n’y a pas de méthodologie commune entre les programmes. Dans cette étude, nous utilisons Autoscoper, un programme open source de recalage d’images 2D-à-3D développé à l’Université Brown
Discussion
La vidéoradiographie biplanaire (BVR) est une méthode basée sur l’image qui peut être utilisée pour mesurer le mouvement de l’os et de l’implant dans le poignet et l’articulation radio-ulnaire distale avec une précision submillimétrique et submillimétrique. Dans les études que nous avons décrites ici, la BVR a été utilisée pour identifier un modèle précis de COR projeté pour un poignet sain ainsi que des modèles de contact TWA. De tels résultats peuvent éclaire...
Déclarations de divulgation
Nous n’avons aucun conflit d’intérêts à déclarer.
Remerciements
Les auteurs tiennent à remercier Joséphine Kalshoven et Lauren Parola d’avoir révisé le protocole. Les auteurs tiennent également à remercier Erika Tavares et Rohit Badida pour leur aide tout au long de l’acquisition des données, ainsi que Kalpit Shah, Arnold-Peter Weiss et Scott Wolfe pour leur aide dans l’interprétation des données. Cette étude a été rendue possible grâce au soutien des National Institutes of Health P30GM122732 (COBRE Bio-engineering Core) et à une subvention de l’American Foundation for Surgery of the Hand (AFSH).
matériels
| Name | Company | Catalog Number | Comments |
| 3D Surface Scanner | Artec 3D | Artec Space SpiderTM | Luxembourg |
| Autoscoper | Brown University | https://simtk.org/projects/autoscoper | https://doi.org/10.1016/j.jbiomech.2019.05.040 |
| CT Scanner | General Electric (GE) | Lightspeed 16 | Milwaukee, WI, USA |
| Geomagic Wrap 3D | 3DSystems | Version 2017 | Rock Hill, SC, USA |
| Graphics Processing Unit (GPU) | Nvidia | GeForce GTX 1080 | CUDA-enabled GPU |
| High-speed Video Cameras | Phantom | Version 10 | Vision Research, Wayne, NJ, USA |
| Image Intensifier | Dunlee | 40 cm diameter | Aurora, IL, USA |
| ImageJ | Open-source (Brown University) | https://imagej.net/Fiji | https://doi.org/10.1038/nmeth.2019 |
| Matlab | The MathWorks, Inc. | R2017a to R2020a | Natick, MA, USA |
| Mimics | Materialise | Version 19.0 to 22.0 | Leuven, Belgium |
| Motion Capture Cameras | Qualisys | Oqus 5+ | Gothenburg, Sweden |
| Pulsed X-ray Generators | EMD Technologies | EPS 45–80 | Saint-Eustache, Quebec, QC, Canada |
| Undistortion Grid | McMaster-Carr | 9255T641 | Steel Perforated Sheet Staggered Holes, 0.048" Thk, 0.125" Hole Dia, 36" X 40" |
| Wrist Implant (In-vitro Study) | Integra LifeSciences | Universal 2 | Plainsboro, NJ, USA |
| Wrist Implant (In-vivo Study) | Integra LifeSciences | Freedom | Plainsboro, NJ, USA |
| WristViz | Open-source (Brown University) | https://github.com/DavidLaidlaw/WristVisualizer/tree/master | Open-source software |
| X-ray Tubes | Varian Medical Systems | Model G-1086 | Palo Alto, CA, USA |
| XMALab | Open-source (Brown University) | https://www.xromm.org/xmalab/ | https://doi.org/10.1242/jeb.145383 |
Références
- Leardini, A., Chiari, L., Croce, U. D., Cappozzo, A. Human movement analysis using stereophotogrammetry: Part 3. Soft tissue artifact assessment and compensation. Gait & Posture. 21 (2), 212-225 (2005).
- Miranda, D. L., Rainbow, M. J., Crisco, J. J., Fleming, B. C. Kinematic differences between optical motion capture and biplanar videoradiography during a jump-cut maneuver. Journal of biomechanics. 46 (3), 567-573 (2013).
- Tashman, S., Anderst, W. In vivo measurement of dynamic joint motion using high speed biplane radiography and CT: application to canine ACL deficiency. Journal of Biomechanical Engineering. 125 (2), 238-245 (2003).
- Moore, D. C., et al. Computed Tomography Image-Based Kinematic Analysis: An Overview. Handbook of Imaging in Biological Mechanics. , 115-126 (2014).
- Fedorov, A., et al. 3D Slicer as an Image Computing Platform for the Quantitative Imaging Network. Magnetic resonance imaging. 30 (9), 1323-1341 (2012).
- Choi, Y. S., et al. Four-dimensional real-time cine images of wrist joint kinematics using dual source CT with minimal time increment scanning. Yonsei medical journal. 54 (4), 1026-1032 (2013).
- Boutin, R. D., et al. Real-Time Magnetic Resonance Imaging (MRI) during Active Wrist Motion-Initial Observations. PLoS ONE. 8 (12), 84004 (2013).
- Miranda, D. L., et al. Static and Dynamic Error of a Biplanar Videoradiography System Using Marker-Based and Markerless Tracking Techniques. Journal of Biomechanical Engineering. 133 (12), 121002-121008 (2011).
- Englander, Z. A., et al. In Vivo Anterior Cruciate Ligament Deformation During a Single-Legged Jump Measured by Magnetic Resonance Imaging and High-Speed Biplanar Radiography , In Vivo Anterior Cruciate Ligament Deformation During a Single-Legged Jump Measured by Magnetic Resonance Imaging and High-Speed Biplanar Radiography. The American Journal of Sports Medicine. 47 (13), 3166-3172 (2019).
- Englander, Z. A., Garrett, W. E., Spritzer, C. E., DeFrate, L. E. In vivo attachment site to attachment site length and strain of the ACL and its bundles during the full gait cycle measured by MRI and high-speed biplanar radiography. Journal of Biomechanics. 98, 109443 (2020).
- Anderst, W. J., Les, C., Tashman, S. In vivo serial joint space measurements during dynamic loading in a canine model of osteoarthritis. Osteoarthritis and cartilage: Osteoarthritis Research Society. 13 (9), 808-816 (2005).
- Bey, M. J., Kline, S. K., Zauel, R., Kolowich, P. A., Lock, T. R. In Vivo Measurement of Glenohumeral Joint Contact Patterns. EURASIP journal on advances in signal processing. 2010, (2010).
- Anderst, W. J., Tashman, S. A method to estimate in vivo dynamic articular surface interaction. Journal of Biomechanics. 36 (9), 1291-1299 (2003).
- Akhbari, B., et al. Accuracy of biplane videoradiography for quantifying dynamic wrist kinematics. Journal of Biomechanics. 92, 120-125 (2019).
- Akhbari, B., et al. Kinematic Accuracy in Tracking Total Wrist Arthroplasty with Biplane Videoradiography using a CT-generated Model. Journal of Biomechanical Engineering. , (2019).
- Akhbari, B., et al. Proximal-distal shift of the center of rotation in a total wrist arthroplasty is more than twice of the healthy wrist. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. 38 (7), 1575-1586 (2020).
- Wang, J., Blackburn, T. J. The AAPM/RSNA Physics Tutorial for Residents. RadioGraphics. 20 (5), 1471-1477 (2000).
- Knörlein, B. J., Baier, D. B., Gatesy, S. M., Laurence-Chasen, J. D., Brainerd, E. L. Validation of XMALab software for marker-based XROMM. The Journal of Experimental Biology. 219 (23), 3701-3711 (2016).
- Brainerd, E. L., et al. X-ray reconstruction of moving morphology (XROMM): precision, accuracy and applications in comparative biomechanics research. Journal of experimental zoology. Part A, Ecological genetics and physiology. 313 (5), 262-279 (2010).
- X-ray Camera Calibration. XROMM / XMALab / wiki Available from: https://bitbucket.org/xromm/xmalab/wiki/X-ray%20Camera%20Calibration (2020)
- Akhbari, B., et al. Biomechanics of the Distal Radioulnar Joint in In vivo Forearm Pronosupination. Journal of Wrist Surgery. , (2020).
- Akhbari, B., et al. Predicting carpal bone kinematics using an expanded digital database of wrist carpal bone anatomy and kinematics. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. , (2019).
- Moore, D. C., et al. Three-dimensional in vivo kinematics of the distal radioulnar joint in malunited distal radius fractures. The Journal of Hand Surgery. 27 (2), 233-242 (2002).
- Knörlein, B. J. Correct for Fluoroscope Distortion (X-ray images). XROMM / XMALab / wiki. , (2020).
- . Autoscoper Available from: https://simtk.org/projects/autoscoper (2019)
- Kennedy, J., Eberhart, R. . Particle swarm optimization. , (1995).
- Nelder, J. A., Mead, R. A Simplex Method for Function Minimization. The Computer Journal. 7, 308 (1965).
- Panjabi, M. A Mathematical Approach for three-dimensional analysis of the mechanics of the spine. J Biomechanics. 4, 203-211 (1971).
- Marai, G. E., et al. Estimating joint contact areas and ligament lengths from bone kinematics and surfaces. IEEE transactions on bio-medical engineering. 51 (5), 790-799 (2004).
- Bland, J. M., Altman, D. G. Measuring agreement in method comparison studies. Statistical methods in medical research. 8 (2), 135-160 (1999).
- . Camera calibration With OpenCV Available from: https://docs.opencv.org/master/index.html (2020)
- Anderst, W. J., Lee, J. Y., Kang, J. D. In Vivo Cervical Facet Joint Capsule Deformation During Flexion-Extension. Spine. 39 (8), 514-520 (2014).
- Anderst, W. J., Vaidya, R., Tashman, S. A technique to measure three-dimensional in vivo rotation of fused and adjacent lumbar vertebrae. The Spine Journal. 8 (6), 991-997 (2008).
- Mozingo, J. D., et al. Validation of imaging-based quantification of glenohumeral joint kinematics using an unmodified clinical biplane fluoroscopy system. Journal of Biomechanics. 71, 306-312 (2018).
- Akbari-Shandiz, M., et al. MRI vs CT-based 2D-3D auto-registration accuracy for quantifying shoulder motion using biplane video-radiography. Journal of Biomechanics. 82, 375-380 (2019).
- Bey, M. J., et al. In vivo shoulder function after surgical repair of a torn rotator cuff: glenohumeral joint mechanics, shoulder strength, clinical outcomes, and their interaction. The American journal of sports medicine. 39 (10), 2117-2129 (2011).
- Bey, M. J., Kline, S. K., Zauel, R., Kolowich, P. A., Lock, T. R. In Vivo Measurement of Glenohumeral Joint Contact Patterns. EURASIP journal on advances in signal processing. 2010, (2010).
- Bey, M. J., Zauel, R., Brock, S. K., Tashman, S. Validation of a New Model-Based Tracking Technique for Measuring Three-Dimensional, In Vivo Glenohumeral Joint Kinematics. Journal of biomechanical engineering. 128 (4), 604-609 (2006).
- Fu, E., et al. Elbow Position Affects Distal Radioulnar Joint Kinematics. The Journal of Hand Surgery. 34 (7), 1261-1268 (2009).
- Martin, D. E., et al. Model-Based Tracking of the Hip: Implications for Novel Analyses of Hip Pathology. The Journal of Arthroplasty. 26 (1), 88-97 (2011).
- Miranda, D. L., et al. Static and Dynamic Error of a Biplanar Videoradiography System Using Marker-Based and Markerless Tracking Techniques. Journal of Biomechanical Engineering. 133 (12), 121002-121008 (2011).
- Stentz-Olesen, K., et al. Validation of static and dynamic radiostereometric analysis of the knee joint using bone models from CT data. Bone & Joint Research. 6 (6), 376-384 (2017).
- Anderst, W., Zauel, R., Bishop, J., Demps, E., Tashman, S. Validation of Three-Dimensional Model-Based Tibio-Femoral Tracking During Running. Medical engineering & physics. 31 (1), 10-16 (2009).
- Kessler, S. E., et al. A Direct Comparison of Biplanar Videoradiography and Optical Motion Capture for Foot and Ankle Kinematics. Frontiers in Bioengineering and Biotechnology. 7, 199 (2019).
- Maharaj, J. N., et al. The Reliability of Foot and Ankle Bone and Joint Kinematics Measured With Biplanar Videoradiography and Manual Scientific Rotoscoping. Frontiers in Bioengineering and Biotechnology. 8, 106 (2020).
- Ito, K., et al. Direct assessment of 3D foot bone kinematics using biplanar X-ray fluoroscopy and an automatic model registration method. Journal of Foot and Ankle Research. 8, 21 (2015).
- Wang, B., et al. Accuracy and feasibility of high-speed dual fluoroscopy and model-based tracking to measure in vivo ankle arthrokinematics. Gait & Posture. 41 (4), 888-893 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon