Method Article
Ein Hochdurchsatz-Assay mit enzymgekoppelter Aktivität zur Untersuchung der Wechselwirkung kleiner Moleküle mit der dNTPase SAMHD1
In diesem Artikel
Zusammenfassung
SAMHD1 ist eine Desoxynukleosidtriphosphat-Triphosphohydrolase, die eine entscheidende Rolle bei der menschlichen Gesundheit und Krankheit spielt. Hier stellen wir einen vielseitigen enzymgekoppelten SAMHD1-Aktivitätsassay vor, der in einem 384-Well-Mikrotiterplattenformat eingesetzt wird und die Evaluierung von kleinen Molekülen und Nukleotidanaloga als SAMHD1-Substrate, Aktivatoren und Inhibitoren ermöglicht.
Zusammenfassung
Steriles Alpha-Motiv und HD-Domänen-enthaltendes Protein 1 (SAMHD1) ist ein zentraler Regulator der intrazellulären Desoxynukleosidtriphosphat (dNTP)-Pools, da dieses Enzym dNTPs in ihre entsprechenden Nukleoside und anorganischen Triphosphate hydrolysieren kann. Aufgrund seiner entscheidenden Rolle im Nukleotidstoffwechsel, seiner Assoziation mit verschiedenen Pathologien und seiner Rolle bei der Therapieresistenz wird derzeit intensiv daran geforscht, sowohl die Regulation als auch die zelluläre Funktion dieses Enzyms besser zu verstehen. Aus diesem Grund ist die Entwicklung einfacher und kostengünstiger Hochdurchsatzmethoden zur Untersuchung der Wechselwirkung kleiner Moleküle mit SAMHD1, wie z. B. allosterische Regulatoren, Substrate oder Inhibitoren, von entscheidender Bedeutung. Zu diesem Zweck ist der enzymgekoppelte Malachitgrün-Assay ein einfacher und robuster kolorimetrischer Assay, der in einem 384-Mikrotiterplatten-Format eingesetzt werden kann, was die indirekte Messung der SAMHD1-Aktivität ermöglicht. Da SAMHD1 die Triphosphatgruppe aus Nukleotidsubstraten freisetzt, können wir eine Pyrophosphatase-Aktivität an diese Reaktion koppeln, wodurch anorganisches Phosphat entsteht, das durch das Malachitgrün-Reagenz durch die Bildung eines Phosphomolybdat-Malachitgrün-Komplexes quantifiziert werden kann. In dieser Arbeit zeigen wir die Anwendung dieser Methodik, um bekannte Inhibitoren von SAMHD1 zu charakterisieren und die Mechanismen zu entschlüsseln, die an der SAMHD1-Katalyse von nicht-kanonischen Substraten und der Regulation durch allosterische Aktivatoren beteiligt sind, am Beispiel von Nukleosid-basierten Krebsmedikamenten. Daher ist der enzymgekoppelte Malachitgrün-Assay ein leistungsfähiges Werkzeug zur Untersuchung von SAMHD1 und könnte darüber hinaus auch bei der Untersuchung mehrerer Enzyme eingesetzt werden, die Phosphatspezies freisetzen.
Einleitung
Steriles Alpha-Motiv und Histidin-Aspartat-Domänen-enthaltendes Protein 1 (SAMHD1) ist ein zentraler Regulator der Nukleotidhomöostase in Säugetierzellen1 mit vielen Rollen bei der menschlichen Gesundheit und Krankheit2. Dieses Enzym ist in der Lage, Desoxynukleosidtriphosphate (dNTPs) in ihre verwandten Desoxynukleosid- und anorganischen Triphosphatmoleküle 3,4 zu hydrolysieren, wobei diese Aktivität allosterisch durch die (d)NTP-Häufigkeit reguliert wird (siehe Referenz5). Jedes SAMHD1-Monomer enthält zwei allosterische Stellen (AS1 und AS2) und eine katalytische Stelle, und die Bildung des aktiven Enzyms erfordert die geordnete Assemblierung eines Homotetramers bei (d)NTP-Bindung. Die Dimerisierung von SAMHD1-Monomeren wird zunächst durch die Bindung eines Guanintriphosphats (GTP oder dGTP) an AS1 ausgelöst, und die anschließende Tetramerisierung wird erreicht, wenn ein zusätzliches dNTP-Molekül an AS2 bindet, was den Substratzugang zum katalytischen Zentrum und die anschließende Hydrolyse ermöglicht.
SAMHD1-Substrate umfassen die vier kanonischen dNTPs 3,4 zusammen mit einigen basen- und zuckermodifizierten Nukleotiden, einschließlich der Triphosphatmetaboliten mehrerer nukleosidbasierter Arzneimittel, die bei der Behandlung von Virusinfektionen und Krebs verwendet werden, von denen einige auch als allosterische Aktivatoren dienen können 6,7,8,9,10,11. In der Konsequenz moduliert SAMHD1 die Wirksamkeit vieler dieser Substanzen in den Krankheitsmodellen 7,8,9,10,11,12,13,14,15 und diktiert darüber hinaus im Falle des Desoxycytidin-Analogons Cytarabin (ara-C), das seit Jahrzehnten die Standardtherapie der akuten myeloischen Leukämie (AML) ist, tatsächlich Wirksamkeit der Behandlung bei dieser Krankheit 7,8,16. SAMHD1 ist daher ein potenzieller Biomarker und therapeutisches Ziel, um die Wirksamkeit von Nukleosid-basierten Therapien zu verbessern17, und dementsprechend haben wir und andere versucht, Strategien zur Inaktivierung von SAMHD1 in Zellen zu identifizieren. Wir schlugen die Verwendung des viralen Proteins X (Vpx) als biologischen Inhibitor vor, um SAMHD1 für den Abbau in Krebszellen zu beeinflussen7, jedoch hat dieser Ansatz eine Reihe von Einschränkungen (diskutiert in Referenz12), und wir berichteten kürzlich auch über einen indirekten Ansatz zur Unterdrückung der SAMHD1-Aktivität durch Hemmung der Ribonukleotidreduktase, den wir in verschiedenen Modellen der AMLdemonstrierten 18. Eine Reihe von Studien hat versucht, kleine Moleküle zu identifizieren, die in der Lage sind, SAMHD1 direkt zu hemmen, und bis heute wurden mehrere solcher Moleküle berichtet, die jedoch nur eine Hemmung in vitro dokumentieren 6,9,19,20,21,22. Folglich unterstreicht der Mangel an kleinen Molekülen, die die SAMHD1-Aktivität in Zellen stark hemmen, in Verbindung mit den komplexen Mechanismen der SAMHD1-Katalyse von Nukleosid-basierten Therapeutika, die Notwendigkeit weiterer Untersuchungen. Daher sind robuste und im Idealfall für den Hochdurchsatz zugängliche Methoden zur Untersuchung der Interaktion kleiner Moleküle mit SAMHD1 ideal, um Substrate, allosterische Regulatoren und Inhibitoren dieses klinisch relevanten Enzyms zu identifizieren.
Es stehen mehrere Methoden zur Verfügung, die die dNTPase-Aktivität von SAMHD1 direkt messen, wie z. B. die Dünnschichtchromatographie (TLC)9,20,23 und die Hochleistungsflüssigkeitschromatographie (HPLC)9,21, aber diese sind für Hochdurchsatzaufbauten nicht ohne weiteres geeignet. Eine Ausnahme ist der von Mauney et al. berichtete Assay, der die Fähigkeit von SAMHD1 ausnutzt, Bis(4-nitrophenyl)phosphat (b4NPP) zu p-Nitrophenol und p-Nitrophenylphosphat zu hydrolysieren, wennMn2+ als aktivierendes Kation verwendet wird, was zu einer kolorimetrischen Änderung führt, die leicht in einer Mikrotiterplatte21 gemessen werden kann. Dieser Assay wurde erfolgreich für die Identifizierung und Charakterisierung von SAMHD1-Inhibitoren eingesetzt, aber es sollte beachtet werden, dass die Hydrolyse in Abwesenheit von (d)NTP-Aktivatoren und in Gegenwart eines wahrscheinlich nicht-physiologischen aktivierenden Kations auftritt, was beides wichtige Vorbehalte sind, die es zu berücksichtigen gilt. Dies macht diesen Assay auch weniger anwendbar für die Untersuchung und Identifizierung allosterischer Regulatoren von SAMHD1.
In diesem Zusammenhang kann ein enzymgekoppelter Ansatz in Kombination mit dem Malachitgrün-Reagenz, wie in diesem Bericht beschrieben, eine vielseitige Methode sein, um die dNTPase-Aktivität von SAMHD1 indirekt zu messen und darüber hinaus den Einfluss verschiedener kleiner Moleküle darauf zu untersuchen. Der Malachitgrün-Assay ist ein robustes und zuverlässiges kolorimetrisches Verfahren zum Nachweis von freiem anorganischem Phosphat (Pi), das auf der Bildung eines Molybdophosphorsäurekomplexes basiert, der zu einer kolorimetrischen Änderung führt, die bei 620 nm24 gemessen wird. Da bei der SAMHD1-Hydrolyse die Triphosphatgruppe aus Nukleotidsubstraten freigesetzt wird, ist es daher notwendig, diese Reaktion vor der Zugabe des Malachitgrün-Reagenzes mit einer (Pyro-)Phosphatase-Aktivität zu koppeln, die freies anorganisches Phosphat erzeugt. Der Malachitgrün-Assay ist empfindlich und kostengünstig und wird häufig zur Identifizierung und Charakterisierung von Inhibitoren und Substraten für Enzyme eingesetzt, die anorganische Phosphatgruppen entweder in ihren Reaktionen oder in Gegenwart eines Kupplungsenzyms freisetzen. Es wurde häufig bei der Charakterisierung der ATPase-Aktivitäten von Helikasen25,26,27 oder bei der Untersuchung der enzymatischen Aktivität CD73 eingesetzt, die den Abbau von AMP zu Adenosin und anorganischem Phosphat vermittelt28. Darüber hinaus wurde es, wenn es gekoppelt ist, bei der Entdeckung von Antibiotika eingesetzt, die auf die UDP-2,3-Diacylglucosamin-Pyrophosphatase LpxH abzielen, ein essentielles Enzym in den meisten gramnegativen Krankheitserregern29. In Bezug auf die Krebsforschung wurde der enzymgekoppelte Ansatz in großem Umfang gegen die NUDIX-Hydrolasen, eine Familie von Nukleotid-metabolisierenden Enzymen, sowohl bei der Charakterisierung von Substraten 30,31,32 als auch bei der Identifizierung und Entwicklung von Medikamenten und chemischen Sonden 33,34,35,36 eingesetzt.
In Bezug auf die dNTPase SAMHD1 wurde dieser Ansatz in mehreren Berichten verwendet. Unter Verwendung der Exopolyphosphatase Ppx1 aus Saccharomyces cerevisiae als Kopplungsenzym wurde dieser Assay verwendet, um mehrere Nukleotidanaloga entweder als Substrate, Aktivatoren oder Inhibitoren von SAMHD1 zu testen, und führte zur Identifizierung des Triphosphatmetaboliten des Antileukämiemedikaments Clofarabin als Aktivator und Substrat6. Darüber hinaus wurde es mit anorganischer Pyrophosphatase aus Escherichia coli als Kupplungsenzym beim Screening einer Bibliothek klinisch zugelassener Verbindungen gegen SAMHD1 eingesetzt, um Inhibitorenzu identifizieren 20. In unserer Forschung haben wir diesen Ansatz verwendet, um zu zeigen, dass ara-CTP, der aktive Metabolit von ara-C, ein SAMHD1-Substrat, aber kein allosterischer Aktivator7 ist, und anschließend diesen Assay verwendet, um zu zeigen, dass mehrere kleine Moleküle, die AML-Modelle auf SAMHD1-abhängige Weise für ara-C sensibilisieren könnten, tatsächlich SAMHD118 nicht direkt hemmen. In diesem Bericht werden wir diese vielseitige Methode detailliert beschreiben und ihre Anwendbarkeit in einem Hochdurchsatz-Aufbau für die Identifizierung von Inhibitoren, Aktivatoren und Substraten von SAMHD1 demonstrieren.
Protokoll
Eine schematische Übersicht der folgenden Methoden ist in Abbildung 1 dargestellt, und eine detaillierte Liste der Materialien und Reagenzien ist in der Materialtabelle verfügbar.
1. Herstellung von Assay-Puffern.
- Vorbereitung von Bestandspuffern.
HINWEIS: Da der Assay empfindlich auf den Nachweis von Phosphaten reagiert, was üblich sein kann, spülen Sie Glaswaren dreimal mit hochreinem oder doppelt destilliertem Wasser, um eine Kontamination zu vermeiden. Alle Puffer können bei Raumtemperatur (RT) gelagert werden.- Bereiten Sie 1 l SAMHD1-Reaktionspuffer-Stammlösung (RB) (25 mM Trisacetat pH 8, 40 mM NaCl, 1 mM MgCl2) vor, indem Sie 4,5 g Trisacetat, 2,3 g NaCl und 0,2 g MgCl2 in etwa 800 ml Wasser auflösen, bevor Sie sich auf pH 8 und das Endvolumen einstellen.
- Bereiten Sie 5 ml 0,1 M TCEP-Stammlösung vor, indem Sie 1 ml 0,5 M TCEP in 4 ml Wasser verdünnen.
- Bereiten Sie 50 ml 11%ige Tween-20-Stammlösung vor, indem Sie 5 ml 100%iges Tween-20 in 44,5 ml Wasser verdünnen.
HINWEIS: Tween-20 ist lichtempfindlich. - Bereiten Sie 50 ml 0,5 M EDTA-Stopplösung vor, indem Sie 9,3 g EDTA in ca. 40 ml Wasser auflösen, bevor Sie sich auf pH 8 und das Endvolumen einstellen.
- Bereiten Sie Malachitgrün (MG) Stammlösung (3,2 mM Malachitgrün inH2SO4) vor, indem Sie langsam 60 ml konzentrierte Schwefelsäure zu 300 ml Wasser in einer Braunglasflasche geben. Die Lösung auf RT abkühlen lassen und 0,44 g Malachitgrün auflösen.
VORSICHT: Die Reaktion von Schwefelsäure mit Wasser ist exotherm, so dass sich die Flasche erhitzen kann, was zu einem Druckaufbau führt. Stellen Sie sicher, dass dieser Druck häufig abgelassen wird.
HINWEIS: Die resultierende orangefarbene Lösung ist lichtempfindlich (daher braune Flasche) und mindestens 1 Jahr bei RT stabil. Niederschlag kann sich im Laufe der Zeit bilden, stellen Sie sicher, dass nur der Überstand verwendet wird. - Bereiten Sie 50 ml 7%ige Ammoniummolybdat-Stammlösung vor, indem Sie 3,75 g Ammoniummolybdat in 50 ml Wasser auflösen.
HINWEIS: Im Laufe der Zeit kann sich Niederschlag bilden, stellen Sie sicher, dass nur der Überstand verwendet wird.
- Herstellung kompletter Assay-Puffer
HINWEIS: Dies sollte am Tag des Experiments erfolgen- Bereiten Sie ein vollständiges SAMHD1 RB vor (25 mM Tris-Acetat pH 8, 40 mM NaCl, 1 mM MgCl2, 0,3 mM TCEP, 0,005 % Tween-20). Verwenden Sie zuvor hergestellte 11 % Tween-20- und 0,1 M TCEP-Stämme, um diese Komponenten in einer Endkonzentration von 0,005 % für Tween-20 und 0,3 mM für TCEP zum SAMHD1 RB-Schaft hinzuzufügen.
- Bereiten Sie die EDTA-Stopplösung vor (25 mM Trisacetat pH 8, 40 mM NaCl, 1 mM MgCl2, 0,3 mM TCEP, 0,005 % Tween-20, 7,9 mM EDTA). Um SAMHD1 RB zu vervollständigen, verwenden Sie 0,5 M EDTA-Stammlösung, um EDTA zu einer Endkonzentration von 7,9 mM hinzuzufügen.
- Bereiten Sie MG-Arbeitslösung (2,5 mM Malachitgrün, 1,4 % Ammoniummolybdat, 0,18 % Tween-20) vor, indem Sie 10 Teile MG-Stammlösung mit 2,5 Teilen 7%igem Ammoniummolybdat und 0,2 Teilen 11%igem Tween-20 mischen.
2. SAMHD1-Inhibitionsassay und Bestimmung der Verbindung IC50
HINWEIS: Die endgültigen Analysebedingungen sind in Tabelle 1 aufgeführt.
- Herstellung von Verbindungen in der Assay-Platte
HINWEIS: Verbindungen mit kleinem Molekulargewicht sind in der Regel in 100% DMSO- und Nukleotidanaloga in Wasser gelöst. Die Stammkonzentration liegt zwischen 10 und 100 mM und wird durch die Wirksamkeit und Löslichkeit der Verbindungen sowie durch die DMSO-Toleranz des Assays beeinflusst. Stellen Sie sicher, dass die endgültige DMSO-Konzentration in der Reaktion 1 % nicht überschreitet, um sicherzustellen, dass die Enzymaktivitäten durch dieses Lösungsmittel nicht beeinträchtigt werden. Es empfiehlt sich, die Toleranz des Assays gegenüber dem Lösungsmittel vor dem Experiment zu testen.- Herstellen von seriell verdünnten Testverbindungen bei 100-facher Endkonzentration im entsprechenden Lösungsmittel (z. B. 100 % DMSO für kleine Moleküle oder Wasser für Nukleotidanaloga) in einer durchsichtigen 96-Well-Platte aus Polypropylen mit rundem Boden entweder mit einer Mehrkanalpipette oder einem automatisierten Liquid-Handling-Gerät.
HINWEIS: Abhängig von der Stabilität der Verbindung können Verdünnungsplatten im Voraus vorbereitet, versiegelt und bei -20 °C gelagert werden. Lassen Sie die Platten auf RT äquilibrieren, bevor Sie mit dem Protokoll fortfahren. - Verdünnen Sie die Verbindungen unter Verwendung von vollständigem SAMHD1 RB auf die 25-fache Endkonzentration (um die endgültige Lösungsmittelkonzentration unter 1 % zu halten) und geben Sie 5 μl in die entsprechenden Vertiefungen einer durchsichtigen 384-Well-Assay-Platte mit flachem Boden. Wiederholen Sie den Vorgang mit reinen Lösungsmittel-Kontrollproben.
- Herstellen von seriell verdünnten Testverbindungen bei 100-facher Endkonzentration im entsprechenden Lösungsmittel (z. B. 100 % DMSO für kleine Moleküle oder Wasser für Nukleotidanaloga) in einer durchsichtigen 96-Well-Platte aus Polypropylen mit rundem Boden entweder mit einer Mehrkanalpipette oder einem automatisierten Liquid-Handling-Gerät.
- Herstellung von Reaktionskomponenten
HINWEIS: Dies sollte am Tag des Tests erfolgen. Rekombinante humane SAMHD1- und E. coli-Pyrophosphatase (PPase)-Aliquots werden langfristig bei -80 °C verdünnt bei 9,1 mg/ml bzw. 23,0 mg/ml in einem Lagerpuffer (20 mM HEPES pH 7,5, 300 mM NaCl, 10% Glycerin, 2 mM TCEP) gelagert. Nach dem Auftauen werden Aliquots kurzzeitig bei -20 °C gelagert.- Bereiten Sie den Enzym-Mastermix (SAMHD1/PPase) vor, indem Sie rekombinantes humanes SAMHD1-Protein und rekombinante PPase in vollständigem SAMHD1 RB auf die 4-fache gewünschte Endkonzentration verdünnen, also 1,4 μM SAMHD1 und 50 U/mL PPase.
- Bereiten Sie Aktivator/Substrat-dGTP vor, indem Sie dGTP-Material (typischerweise 10 oder 100 mM in Wasser) in vollständigem SAMHD1 RB auf die 2-fache Endkonzentration, also 50 μM dGTP, verdünnen.
- Führen Sie den Assay durch
HINWEIS: Alle Assay-Komponenten sollten auf RT äquilibriert sein. Die Zugabe von Flüssigkeiten kann entweder mit einer Mehrkanalpipette oder einem Bulk-Reagenz-Flüssigkeitsspender durchgeführt werden.- Geben Sie 5 μl SAMHD1/PPase-Mastermix auf eine 384-Well-Assay-Platte, die Verbindungsverdünnungen und Lösungsmittelkontrollen enthält. Geben Sie 5 μl vollständiges SAMHD1 RB an keine Enzymkontrollvertiefungen ab. Enzym und Verbindungen 10 Minuten bei RT vorinkubieren.
- Geben Sie 10 μl 2x dGTP-Lösung in alle Vertiefungen, um die Reaktion zu starten.
- Die Reaktion wird 20 Minuten lang bei RT inkubiert.
- Stoppen Sie die Reaktion, indem Sie 20 μl EDTA-Stopplösung in alle Vertiefungen geben.
HINWEIS: Das Experiment kann hier auf Wunsch pausiert werden. - Geben Sie 10 μl MG-Arbeitslösung in alle Vertiefungen.
VORSICHT: MG-Arbeitslösung enthält Schwefelsäure. - Stellen Sie sicher, dass der Well-Inhalt mit einem orbitalen Mikrotiterplattenschüttler gemischt und 1 Minute lang bei 1.000 x g zentrifugiert wird.
- Inkubieren Sie die Platte 20 Minuten lang bei RT.
- Lesen Sie die Absorption bei einer Wellenlänge von 630 nm in einem Mikrotiterplatten-Lesegerät ab.
- Datenvisualisierung und -analyse
- Berechnen Sie den Mittelwert und die Standardabweichung der positiven und negativen Kontrollvertiefungen (positiv, vollständige Reaktion mit Lösungsmittel; negativ, dGTP allein mit Lösungsmittel). Berechnen Sie den Z-Faktor37 als Indikator für die Assay-Qualität.
- Normalisieren Sie jeden Absorptionswert auf die Mittelwerte der Positiv- und Negativkontrollen, wobei die Positivkontrolle auf 100 % SAMHD1-Aktivität und die Negativkontrolle auf 0 % SAMHD1-Aktivität festgelegt wird.
- Zeichnen Sie die SAMHD1-Aktivität (%) als Funktion der Substanzkonzentration auf und passen Sie eine Dosis-Wirkungs-Kurve mit vier Parametern mit variabler Steigung an, die die Bestimmung der Verbindung IC50 ermöglicht.
3. SAMHD1-Aktivator und Substratsieb
HINWEIS: Die endgültigen Analysebedingungen sind in Tabelle 2 aufgeführt. Rekombinante SAMHD1- und PPase-Aliquots werden langfristig bei -80 °C verdünnt bei 9,1 mg/ml bzw. 23,0 mg/ml in einem Lagerpuffer (20 mM HEPES pH 7,5, 300 mM NaCl, 10% Glycerin, 2 mM TCEP) bei -80 °C gelagert. Nach dem Auftauen werden die Aliquots kurzzeitig bei -20 °C gelagert.
- Herstellung von Nukleotidanaloga in der Assayplatte
- Verdünnen Sie Nukleotidanaloga (typischerweise 10 oder 100 mM in Wasser) auf die 4-fache Endkonzentration in vollständigem SAMHD1 RB, in unserem Fall 800 μM Nukleotidanalogon, und übertragen Sie 5 μl in die entsprechenden Vertiefungen einer 384-Well-Assay-Platte.
- Herstellung von Reaktionskomponenten
HINWEIS: Dies sollte am Tag des Tests erfolgen- Bereiten Sie den Enzym-Mastermix (SAMHD1/PPase) vor, indem Sie rekombinantes humanes SAMHD1-Protein und rekombinante E. coli-PPase in vollständigem SAMHD1 RB auf die 2-fache gewünschte Endkonzentration verdünnen, also 0,7 μM SAMHD1 und 25 U/mL PPase.
- Bereiten Sie die PPase allein vor, indem Sie die rekombinante E. coli-PPase in vollständiger SAMHD1-RB auf die 2-fache gewünschte Endkonzentration, also 25 U/ml PPase, verdünnen.
- Bereiten Sie die Aktivatoren GTP (AS1) und dGTPαS (AS1 und AS2) zur Verdünnung (typischerweise 10 oder 100 mM in Wasser) in vollständigem SAMHD1 RB auf die 4-fache Endkonzentration vor, also 50 μM GTP oder dGTPαS.
- Führen Sie den Assay durch
HINWEIS: Alle Assay-Komponenten sollten auf RT äquilibriert sein. Die Zugabe von Flüssigkeiten kann entweder mit einer Mehrkanalpipette oder einem Bulk-Reagenz-Flüssigkeitsspender durchgeführt werden.- Auf eine 384-Well-Assay-Platte mit Nukleotidanaloga geben Sie 5 μl des Aktivators (entweder GTP oder dGTPαS) oder vervollständigen Sie SAMHD1 RB in die entsprechenden Wells.
- Beginnen Sie die Reaktion, indem Sie 10 μl SAMHD1/PPase-Mastermix, PPase allein oder komplettes SAMHD1 RB in die entsprechenden Vertiefungen geben.
- Die Reaktion wird 20 Minuten lang bei RT inkubiert.
- Stoppen Sie die Reaktion, indem Sie 20 μl EDTA-Stopplösung in alle Vertiefungen geben.
HINWEIS: Das Experiment kann hier auf Wunsch pausiert werden. - Geben Sie 10 μl MG-Arbeitslösung in alle Vertiefungen.
VORSICHT: MG-Arbeitslösung enthält Schwefelsäure. - Stellen Sie sicher, dass der Well-Inhalt mit einem orbitalen Mikrotiterplattenschüttler gemischt und 1 Minute lang bei 1.000 x g zentrifugiert wird.
- Inkubieren Sie die Platte 20 Minuten lang bei RT.
- Lesen Sie die Absorption bei einer Wellenlänge von 630 nm in einem Mikrotiterplatten-Lesegerät ab.
- Datenvisualisierung und -analyse
- Berechnen Sie die durchschnittlichen Absorptionswerte für die PPase-reinen Reaktionsvertiefungen (Negativkontrolle oder Hintergrundsignal).
HINWEIS: Als Positivkontrolle eines allosterischen SAMHD1-Aktivators und -Substrats kann dGTP in die Platte eingeschlossen werden. In diesem Fall können Sie diese Bedingung verwenden, um den Z-Faktor als Indikator für die Assay-Qualität zu berechnen. - Subtrahieren Sie den Hintergrundwert von den entsprechenden Vertiefungen in den SAMHD1/PPase-Reaktionen.
- Zeichnen Sie korrigierte Absorptionswerte für jedes Nukleotidanalogon mit Puffer-, GTP- und dGTPαS-Bedingungen auf.
- Berechnen Sie die durchschnittlichen Absorptionswerte für die PPase-reinen Reaktionsvertiefungen (Negativkontrolle oder Hintergrundsignal).
Ergebnisse
Das in Abbildung 1 skizzierte Protokoll beschreibt den grundlegenden Arbeitsablauf für die Verwendung des enzymgekoppelten Malachitgrün-Assays, um die Wechselwirkung kleiner Moleküle mit der dNTPase SAMHD1 zu untersuchen, und kann auf verschiedene Weise angepasst werden, um verschiedene biochemische Fragen zu untersuchen. In den repräsentativen Ergebnissen, die in den folgenden Abschnitten diskutiert werden, veranschaulichen wir Beispiele für die Verwendung dieses Assays, um die hemmenden Eigenschaften kleiner Moleküle gegenüber SAMHD1 zu bestimmen und um zu testen, ob verschiedene Nukleotidanaloga Substrate und/oder Aktivatoren dieses Enzyms sind.
Die in Abbildung 2 dargestellten Ergebnisse veranschaulichen mehrere Kernprinzipien dieses Assays. Das Malachitgrün-Reagenz ermöglicht den kolorimetrischen Nachweis von anorganischem Phosphat durch die Bildung eines Phosphomolybdat-Malachitgrün-Komplexes, und dementsprechend kann dieser Ansatz auf die Untersuchung von enzymatischen Reaktionen angewendet werden, deren Produkt Phosphat ist. Um die Empfindlichkeit dieser Methode zum Nachweis von freiem anorganischem Phosphat zu demonstrieren, zeigt Abbildung 2A die Absorptionswerte, die mit steigenden Konzentrationen von Na3PO4 nach einer 20-minütigen Inkubation mit dem Malachitgrün-Reagenz erhalten wurden. Während das Signal die Sättigung bei 0,25 mMNa3PO4 erreicht, ist der lineare Detektionsbereich von Phosphat von 0,004 bis 0,03 mM sichtbar (Abbildung 2A, rechtes Bild), in Übereinstimmung mit anderen Studien, die einen linearen Phosphatbereich von bis zu 10-20 μM unter Verwendung des Malachitgrün-Assays38 berichteten.
SAMHD1 ist eine dNTPase, die bei der Hydrolyse eines dNTP-Moleküls anorganisches Triphosphat freisetzt, und daher ist ein Kupplungsenzym erforderlich, um freies anorganisches Phosphat für die Detektion durch Malachitgrün zu erzeugen. Anorganische Pyrophosphatase (PPase) aus E. coli hat sich für diesen Zweck als nützlich erwiesen, sowohl im Hinblick auf SAMHD1 7,20, als auch auf andere Nukleotid-metabolisierende Enzyme 30,33,35. Darüber hinaus ist SAMHD1 als Homotetramer eine aktive dNTPase, und dies erfordert eine allosterische Aktivierung durch (d)NTPs, insbesondere ein Guanintriphosphat (GTP oder dGTP) an AS1 und ein beliebiges dNTP an AS2. Anschließend wird die katalytische Stelle für die Substratbindung zugänglich und die enzymatische Reaktion findet statt. Da dGTP die Anforderungen für die Bindung an AS1 und AS2 erfüllt und ein Substrat ist, vereinfacht die Verwendung dieses Nukleotids im Inhibitionsassay den Arbeitsablauf erheblich. Abbildung 2B veranschaulicht die Anforderung der verschiedenen Assay-Komponenten, eine messbare SAMHD1-Aktivität zu erreichen, die durch eine Erhöhung der Absorption bei 630 nm angezeigt wird. Weder SAMHD1 noch PPase allein sind in der Lage, anorganisches Phosphat in Gegenwart von dGTP zu erzeugen, was mit den dokumentierten Aktivitäten dieser Enzyme übereinstimmt. In dem Zustand, in dem alle Assay-Komponenten (SAMHD1, PPase und der dGTP-Aktivator/das Substrat) vorhanden sind, beobachten wir jedoch einen Anstieg des Signals. Der Z-Faktor37 des hier gezeigten Beispiels (ohne Enzyme + dGTP als Negativkontrolle und SAMHD1/PPase + dGTP als Positivkontrolle) betrug 0,74, was auf einen robusten Assay hindeutet.
Eine der möglichen Anwendungen des enzymgekoppelten SAMHD1-Aktivitätsassays ist die Identifizierung von Inhibitoren durch Hochdurchsatz-Screening (HTS). Daher validieren wir in diesem Bericht den Nachweis der SAMHD1-Hemmung in diesem Assay unter Verwendung einer Vielzahl von Verbindungen, die bereits in der Literatur beschrieben sind. Seamon et al. untersuchten die dosisabhängige Hemmung von kanonischen Nukleosiden gegenüber SAMHD1 mit einem ähnlichen Assay wie hier gezeigt und stellten fest, dass Desoxyguanosin (dGuo) das einzige kanonische Nukleosid war, das in der Lage war, SAMHD1 signifikant zu hemmen, mit einem IC50-Wert von 488 μM20. Eine HTS von FDA-zugelassenen Arzneimitteln, die mit dem direkten b4NPP-Assay durchgeführt wurde, ergab mehrere Treffer, die die SAMHD1-Aktivität bei mikromolaren Konzentrationen hemmten, wobei Lolobungin das Molekül war, das die SAMHD1-dNTPase-Aktivität in vitro am stärksten hemmte und einen IC50 von 20,1 μM aufwies, wenn es in Gegenwart von dGTP als Substrat bestimmt wurde21. Darüber hinaus wurden die vier α.β-imido-dNTP-Analoga auch als kompetitive Inhibitoren von SAMHD1 identifiziert, indem der MDCC-PBP-Sensor und SAMHD1 an die Ppx-Aktivität gekoppelt wurden, was zeigte, dass die inhibitorischen Konstanten der dNMPNPP-Analoga im niedrigen mikromolaren / hohen nanomolaren Bereich lagen 6,22. Um zu zeigen, dass der enzymgekoppelte SAMHD1-Aktivitätsassay zur Identifizierung von SAMHD1-Inhibitoren verwendet werden kann, wurden dGuo, Lolomungin und 2'-Desoxythymidin-5'-[(α,β)-Imido]triphosphat (dTMPNPP) zur Validierung der Technik verwendet. Abbildung 3A zeigt die für diese Verbindungen erhaltenen Dosis-Wirkungs-Kurven und zeigt, dass steigende Konzentrationen die SAMHD1-Aktivität wirksam hemmen. Die mittleren IC50-Werte, die für diese Moleküle aus drei unabhängigen Experimenten (± Standardabweichung) erhalten wurden, waren wie folgt: dGuo = 361,9 ± 72,8 μM, Lolomungin 6,78 ± 3,9 μM und dTMPNPP = 2,10 ± 0,9 μM. Als Beispiel für ein negatives Ergebnis wurde auch der Einfluss von Hydroxyharnstoff (HU) auf die SAMHD1-Aktivität bestimmt. HU ist ein Inhibitor der Ribonukleotid-Reduktase, und obwohl es die SAMHD1 ara-CTPase-Aktivität in verschiedenen AML-Modellen einschränkt, wurden die Auswirkungen von HU auf SAMHD1 als indirekt gezeigt und beruhen auf der Störung der allosterischen Regulation von SAMHD118. Die Dosis-Wirkungs-Kurve von HU ist in Abbildung 3B dargestellt, und es wurden keine Veränderungen der SAMHD1-Aktivität mit steigender HU-Dosis beobachtet, was zeigt, dass HU die SAMHD1-Aktivität in vitro nicht hemmt.
Eine weitere Anwendung des enzymgekoppelten SAMHD1-Aktivitätsassays besteht darin, zu untersuchen, ob Nukleotide und ihre Analoga Substrate und/oder allosterische Aktivatoren dieses Enzyms sind, was in Abbildung 4 dargestellt ist. In diesem Experiment wurden kanonische Nukleotide sowie die aktiven Metaboliten mehrerer Anti-Krebs-Nukleosid-Analoga wie Cytarabin (ara-CTP), Clofarabin (Cl-F-ara-ATP) und Gemcitabin (dF-dCTP) als SAMHD1-Substrate und -Aktivatoren getestet. Aufgrund der komplexen allosterischen Regulation von SAMHD1 wird die Reaktion in Gegenwart von GTP als AS1-Aktivator oder dem nicht hydrolysierbaren dGTP-Analogon 2'-Desoxyguanosin-5'-(α-thio)-triphosphat (dGTPαS) durchgeführt, das AS1 und AS2 besetzen kann. Die SAMHD1-Aktivität in Gegenwart des getesteten Nukleotidanalogons und GTP zeigt an, dass das Nukleotid in der Lage ist, an die sekundäre allosterische Stelle und die katalytische Stelle (d. h. AS2-Aktivator und Substrat) zu binden, während die SAMHD1-Aktivität mit dem Nukleotidanalogon und dGTPαS darauf hinweist, dass das Nukleotid nur die katalytische Stelle (d. h. nur ein Substrat) besetzen kann. Wenn das Nukleotid in der Lage ist, sowohl an die allosterische AS1- und AS2-Stelle als auch an die katalytische Stelle zu binden, ist SAMHD1 nur in Gegenwart des Nukleotids aktiv, wie im Fall von dGTP gezeigt. Die Ergebnisse zeigen, dass alle kanonischen dNTPs in der Lage sind, an die AS2-Stelle und an die katalytische Stelle zu binden. Bei Nukleotidanaloga ist Clofarabintriphosphat ein AS2-Aktivator und ein Substrat, während Cytarabintriphosphat nur in der Lage ist, die katalytische Stelle zu besetzen. Auf der anderen Seite wurde keine Aktivität mit Gemcitabintriphosphat beobachtet, was darauf hindeutet, dass Gemcitabintriphosphat unter den getesteten Bedingungen nicht in der Lage ist, als allosterischer Effektor oder Substrat zu wirken. Obwohl dieses Ergebnis mit früheren Vorhersagen übereinstimmt9, zeigten spätere Kristallisations- und kinetische Studien10 , dass Gemcitabintriphosphat in der Lage ist, die katalytische Tasche von SAMHD1 zu binden, und dass es tatsächlich ein Substrat des Enzyms ist. In der letztgenannten Studie10 zeigen die Autoren jedoch, dass die Hydrolyserate im Vergleich zu anderen berichteten Substraten, wie z. B. Cytarabintriphosphat, erheblich niedriger ist, was erklärt, warum wir dies mit diesem Screening-Aufbau nicht beobachten konnten.
Insgesamt validieren diese repräsentativen Ergebnisse die Verwendung des enzymgekoppelten SAMHD1-Aktivitätstests als robuste Technik zur Identifizierung und Charakterisierung von SAMHD1-Inhibitoren, allosterischen Regulatoren und Substraten. Ähnlich wie alle experimentellen Ansätze hat diese Methode jedoch ihre Vorbehalte, so dass orthogonale Assays (z. B. unter Verwendung einer anderen Assay-Technologie) verwendet werden sollten, um die Ergebnisse weiter zu validieren.
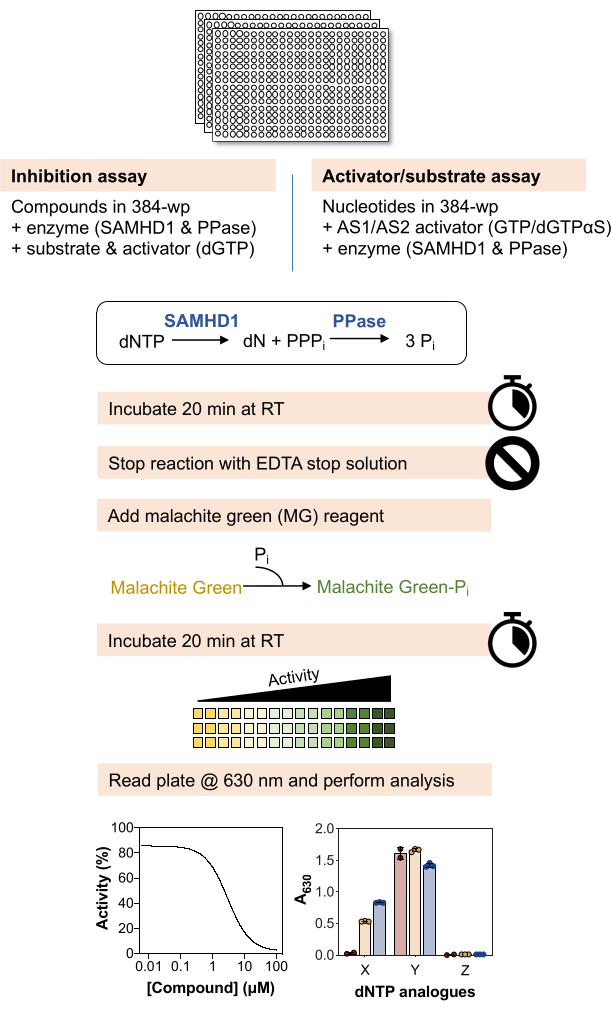
Abbildung 1: Schematische Übersicht über das in diesem Artikel beschriebene Protokoll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
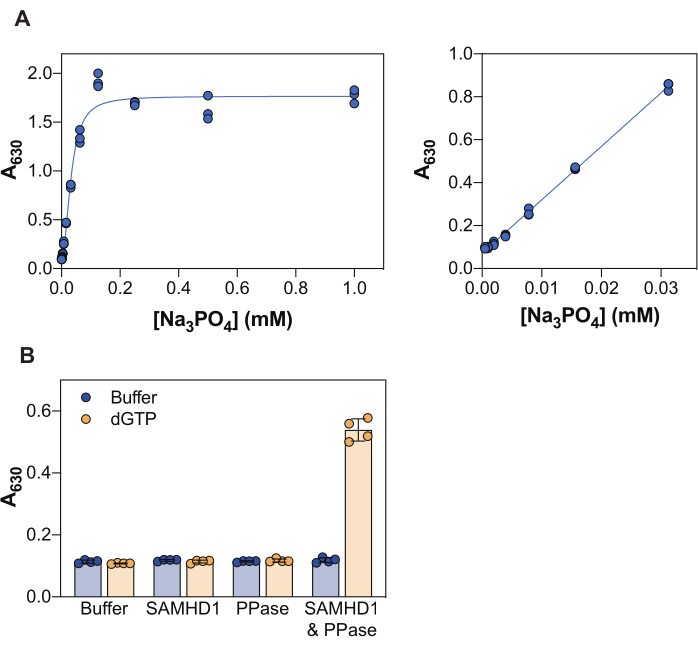
Abbildung 2: Enzymgekoppelter SAMHD1-Aktivitätstest. (A) Na3PO4 Standardkurve im Malachitgrün-Assay. Die serielle Verdünnung vonNa3PO4 (2-fach) wurde von 1 mM bis 0,004 mM in dreifacher Ausführung hergestellt und mit Malachitgrün-Reagenz für 20 min inkubiert. Die Rohabsorptionswerte über den gesamten Bereich der getesteten Konzentrationen sind im linken Feld und der lineare Bereich im rechten Bereich dargestellt. Stellvertretend für zwei unabhängige Experimente gezeigt. (B) Validierung des Assays für enzymgekoppelte Aktivität. SAMHD1 (0,35 μM) und/oder PPase (12,5 U/ml) in Gegenwart oder Abwesenheit von Aktivator/Substrat dGTP (25 μM) wurden für 20 min im Assay mit enzymgekoppelter Aktivität inkubiert. Vierlinge aus einem Repräsentanten von zwei unabhängigen Experimenten, die mit aufgetragenen Rohabsorptionswerten, Balken und Fehlerbalken dargestellt sind, zeigen Mittelwert und SD an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
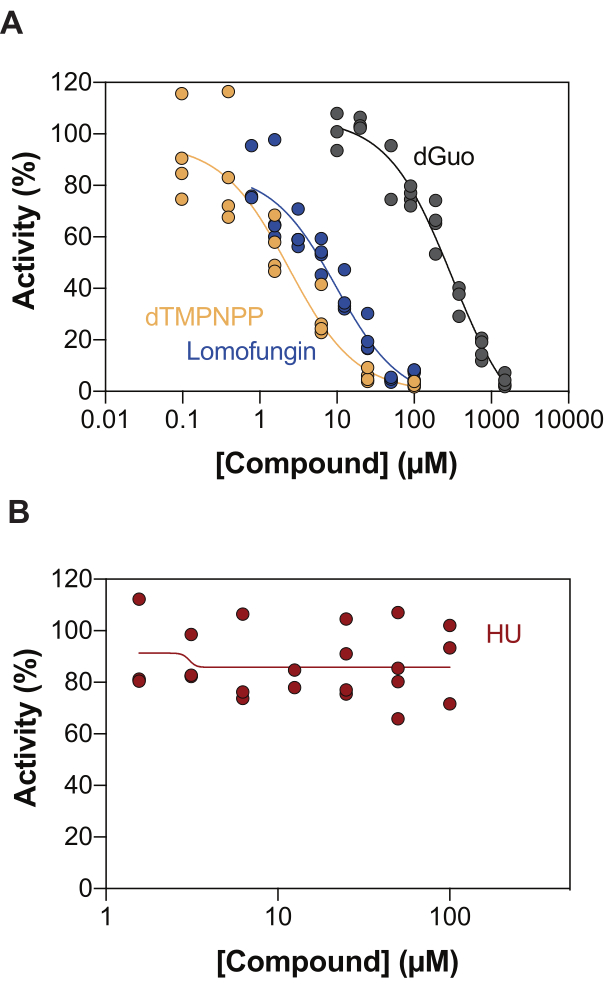
Abbildung 3: Evaluierung von Verbindungen zur Hemmung von SAMHD1 im Assay für enzymgekoppelte Aktivität. Dosis-Wirkungs-Verhältnis von Lopofunget (0,78-100 μM), 2'-Desoxythymidin-5'-[(α,β)-imido]triphosphat (dTMPNPP, 0,01-100 μM) und Desoxyguanosin (dGuo, 10-1.500 μM) (A) oder Hydroxyharnstoff (HU) (0,78-100 μM) (B) im enzymgekoppelten SAMHD1-Aktivitätsassay mit dGTP (25 μM) als Aktivator/Substrat. Prozentuale Aktivität im Verhältnis zu Reaktionskontrollen aus einzelnen Replikaten aufgetragen (DMSO + SAMHD1/PPase + dGTP = 100 % Aktivität, DMSO + dGTP = 0 % Aktivität) mit einem repräsentativen von drei gezeigten Experimenten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
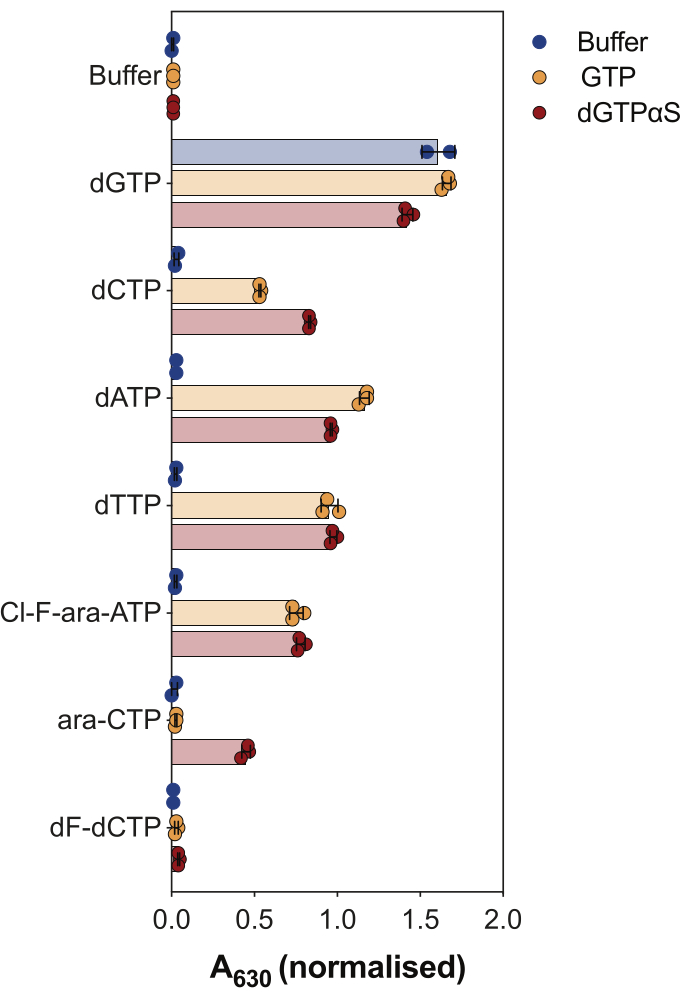
Abbildung 4: Evaluierung von Nukleotidanaloga als allosterische SAMHD1-Aktivatoren und -Substrate im Enzym-gekoppelten Aktivitätsassay. Kanonische Nukleotide und ausgewählte Triphosphat-Metaboliten der Krebsmedikamente Cytarabin (ara-CTP), Clofarabin (Cl-F-ara-ATP) und Gemcitabin (dF-dCTP) wurden bei 200 μM im enzymgekoppelten SAMHD1-Aktivitätsassay in Gegenwart oder Abwesenheit von GTP oder nicht hydrolysierbarem dGTP-Analogon dGTPαS (12,5 μM) getestet. Normierte Absorptionswerte aus einzelnen experimentellen Replikaten werden aufgetragen, Mittelwert und SD werden angezeigt. Repräsentativ für zwei gezeigte unabhängige Experimente, adaptiert aus unserer vorherigen Studie7. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Schritt | Reagenz | Dosiertes Volumen (μL) | Endes Reaktionsvolumen (μl) | Dosierte Konzentration | Faltenverdünnung in Reaktion | Endkonzentration in der Reaktion |
| 1 | Inhibitor | 5 | 20 | 0,4 mM | 4 | 0,1 mM |
| 2 | SAMHD1+PPase-Mischung | 5 | 1,4 μM SAMHD1, 50 U/mL PPase | 4 | 0,35 μM SAMHD1, 12,5 U/ml ppase | |
| 3 | dGTP | 10 | 50 μM | 2 | 25 μM | |
| 4 | Inkubation für 20 Minuten | |||||
| 5 | EDTA-Lösung | 20 | 40 | 7,9 mM | 2 | 3,95 mM |
| 6 | MG-Reagenz | 10 | 50 | 2,5 mM Malachitgrün, 64,4 mM Ammoniummolybdat, 0,18% Tween-20 | 5 | 0,5 mM Malachitgrün, 12,9 mM Ammoniummolybdat, 0,036% Tween-20 |
| 7 | Inkubation für 20 Minuten | |||||
| 8 | Lesen @ 630 nm | |||||
Tabelle 1: Zusammenfassung der Endbedingungen im enzymgekoppelten Assay für das Inhibitoren-Screening.
| Schritt | Reagenz | Dosiertes Volumen (μL) | Endes Reaktionsvolumen (μl) | Dosierte Konzentration | Faltenverdünnung in Reaktion | Endkonzentration in der Reaktion |
| 1 | Allosterischer Regulator | 5 | 20 | 800 μM | 4 | 200 μM |
| 2 | GTP oder dGTPαS | 5 | 50 μM | 4 | 12,5 μM | |
| 3 | SAMHD1 und/oder PPase | 10 | 0,7 μM SAMHD1, 25 U/ml PPase | 2 | 0,35 μM SAMHD1, 12,5 U/mL PPase | |
| 4 | Inkubation für 20 Minuten | |||||
| 5 | EDTA-Lösung | 20 | 40 | 7,9 mM | 2 | 3,95 mM |
| 6 | MG-Reagenz | 10 | 50 | 2,5 mM Malachitgrün, 64,4 mM Ammoniummolybdat, 0,18% Tween-20 | 5 | 0,5 mM Malachitgrün, 12,9 mM Ammoniummolybdat, 0,036% Tween-20 |
| 7 | Inkubation für 20 Minuten | |||||
| 8 | Lesen @ 630 nm | |||||
Tabelle 2: Zusammenfassung der Endbedingungen im enzymgekoppelten Assay für das Screening allosterischer Regulatoren
Diskussion
Der hier beschriebene enzymgekoppelte Aktivitätsassay ist ein kolorimetrischer Hochdurchsatz-Assay, der die indirekte Messung der dNTP-Hydrolyse durch SAMHD1 ermöglicht. Diese Methode nutzt die Fähigkeit der anorganischen PPase aus E. coli, die, wenn sie im Überschuss in der Reaktionsmischung enthalten ist, jedes von SAMHD1 erzeugte anorganische Triphosphat in drei einzelne freie Phosphate umwandelt, die mit dem einfachen und kostengünstigen Malachitgrün-Reagenz quantifiziert werden können. Wir bieten diesen Assay in einem 384-Mikrotiterplattenformat an, das sich ideal für das Screening von Substanzbibliotheken eignet, und demonstrieren die Anwendbarkeit und Vielseitigkeit dieser Technik bei der Identifizierung und Charakterisierung von SAMHD1-Inhibitoren, Aktivatoren und Substraten.
Wie bei allen biochemischen In-vitro-Screening-Assays gibt es eine Reihe kritischer Schritte und wichtiger Überlegungen, von denen viele im frei verfügbaren Assay Guidance Manual39 ausführlich erörtert werden. Die Integrität der gereinigten rekombinanten Enzyme, sowohl SAMHD1 als auch des gekoppelten Enzyms anorganische PPase, ist äußerst wichtig und sollte vor der Etablierung des Assays bestätigt werden. Dementsprechend sollte jede neue Aufreinigung dieser Enzyme einem gewissen Maß an Chargentests unterzogen werden, da Schwankungen von Charge zu Charge zu Inkonsistenzen in den Ergebnissen führen können. Im Idealfall sollte ein orthogonaler Direktassay wie HPLC verwendet werden, der den Nachweis sowohl des Substrats als auch des Reaktionsprodukts ermöglicht, um die dNTP-Triphosphohydrolase-Aktivität des verwendeten gereinigten rekombinanten SAMHD1 zu überprüfen.
Was die Einschränkungen dieses Assays betrifft, so besteht das Prinzip darin, dass er die dNTPase-Aktivität von SAMHD1 auf indirekte Weise misst, wobei die Aktivität der anorganischen PPase ausgenutzt wird, was eine Reihe von Implikationen hat. Es ist wichtig zu bestätigen, dass PPase wenig bis gar keine Aktivität gegenüber Nukleotiden besitzt, die im Assay verwendet werden, und ebenso, dass identifizierte inhibitorische kleine Moleküle keine Aktivität gegenüber PPase besitzen. Daher kann im Hinblick auf das Screening ein Gegenschirm gegen PPase eine wichtige Überlegung sein. Das Vorhandensein von PPase in der Reaktion macht es auch wichtig, einen orthogonalen Assay zu verwenden, um die Ergebnisse zu bestätigen. In Bezug auf direkte Aktivitätsassays wurden bisher eine Reihe von Assays beschrieben, darunter TLC 9,20,23 und HPLC 9,21, die Substraterschöpfung und Produktbildung genau nachweisen. Darüber hinaus könnte der b4NPP-Assay21, der ebenfalls einen hohen Durchsatz aufweist, zum Testen potenzieller Inhibitoren verwendet werden. Es ist jedoch nicht ideal, Substrate oder allosterische Aktivatoren zu testen. Biophysikalische Assays, wie z. B. die dynamische Differenzfluorimetrie (DSF), über die wir bereits mit SAMHD118 berichtet haben, können ebenfalls besonders leistungsfähig sein, um Liganden zu identifizieren und zu charakterisieren. Eine weitere Einschränkung des Assays, insbesondere wie im Aufbau hier zur Identifizierung von Substraten und Aktivatoren gezeigt, ist die Verwendung des nicht-hydrolysierbaren dGTP-Analogons dGTPαS als AS1- und AS2-Aktivator. Während dies die Aktivierung von SAMHD1 ohne beobachtbare Aktivität im Assay ermöglicht, ist dGTPαS ein kompetitiver Inhibitor von SAMHD1, und daher wird die Verwendung hoher Konzentrationen das Enzym inaktivieren. Mit dem Fortschreiten unseres Verständnisses von SAMHD1 könnten zukünftige Studien Moleküle verwenden, die ausschließlich jede Stelle von SAMHD1 besetzen, wodurch dieses potenzielle Problem negiert wird.
Wie wir hier gezeigt haben, ist diese Methode vielseitig einsetzbar und kann zur Beantwortung einer Reihe von biochemischen Fragestellungen eingesetzt werden. Wir haben zwei Variationen dieses Assays beschrieben, eine für die Identifizierung von allosterischen Regulatoren und Substraten von SAMHD1 und eine andere für die Charakterisierung von Inhibitoren, aber es können weitere Anpassungen vorgenommen werden. In Bezug auf potentielle Inhibitoren eignet sich dieser Assay, der auf Mikrotiterplatten basiert, gut für nachgelagerte Studien zum Wirkmechanismus39,40. In ähnlicher Weise kann diese Technik zur weiteren Charakterisierung von Substraten und allosterischen Regulatoren verwendet werden, um kinetische Parameter der Katalyse zu bestimmen, wie wir es für den aktiven Metaboliten von Cytarabin und Clofarabin durchgeführthaben 7. Ein Nachteil ist jedoch, dass es sich bei dem hier berichteten enzymgekoppelten Assay um einen Endpunkt-Assay handelt, so dass ein kontinuierlicher Assay, obwohl er gut für das Screening geeignet ist, für einige mechanistische Studien besser geeignet wäre. Arnold et al. berichteten über einen kontinuierlichen enzymgekoppelten Assay, der den Biosensor MDCC-PBP6 verwendet, der auf der Verwendung des periplasmatischen Phosphatbindungsproteins (PBP) beruht, das mit Cumarinmaleimid (MDCC)-Fluorophor markiert ist und an eine freie Phosphatgruppe binden kann. MDCC-PBP ist sehr empfindlich und ermöglicht die Quantifizierung sehr niedriger Phosphatkonzentrationen, wobei die Reaktionszeit des Sensors im Millisekunden- bis Sekundenbereich liegt.
SAMHD1 spielt eine Reihe wichtiger Funktionen für die menschliche Gesundheit und Krankheit2 , von denen viele mit seiner zentralen Rolle bei der Aufrechterhaltung des intrazellulären dNTP-Spiegels in Verbindung gebracht werdenkönnten 1. Daher wäre die Identifizierung einer hochwertigen chemischen Sonde in Bezug auf die dNTPase-Aktivität von SAMHD1 ein leistungsfähiges Werkzeug zur Definition dieser Verbindungen, und der hier beschriebene enzymgekoppelte Assay könnte leicht zur Identifizierung solcher Sonden eingesetzt werden. Darüber hinaus sind nukleosidbasierte Arzneimittel, von denen viele durch SAMHD1 moduliert werden, eine vielfältige und wichtige Gruppe von therapeutischen41; Chemische Sonden könnten im klinischen Umfeld zu Medikamenten weiterentwickelt werden, die auf SAMHD1 abzielen, mit dem Ziel, die Wirksamkeit dieser Therapien zu erhöhen. Es ist auch wichtig, das volle Ausmaß der Wechselwirkung dieser nukleosidbasierten Verbindungen mit SAMHD1 zu verstehen, eine Frage, die auch mit diesem enzymgekoppelten Assay beantwortet werden kann. Zusammenfassend ist der hier beschriebene enzymgekoppelte SAMHD1-Aktivitätsassay ein kostengünstiger, vielseitiger Hochdurchsatz-Assay, der verwendet werden kann, um unser Verständnis dieses wichtigen Enzyms zu erweitern.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken Thomas Lundbäck und den Mitarbeitern des Labors von Thomas Helleday für Rat und Unterstützung. Ein Teil dieser Arbeit wurde von der Protein Science Facility am Karolinska Institutet/SciLifeLab (http://ki.se/psf) ermöglicht, und wir danken dem National Cancer Institute (NCI), der Division of Cancer Treatment and Diagnosis (DCTD) und dem Developmental Therapeutics Program (DTP) (http://dtp.cancer.gov) für die Bereitstellung eines Wirkstoffs. Die Finanzierung erfolgte durch Zuschüsse, die S.G.R. vom Schwedischen Forschungsrat (2018-02114), der Schwedischen Krebsgesellschaft (19-0056-JIA, 20-0879-PJ), dem Schwedischen Kinderkrebsfonds (PR2019-0014) und dem Karolinska Institutet gewährt wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| 2'-deoxyadenosine-5'-triphosphate (dATP) | Jena bioscience | NU-1001 | Compound tested in SAMHD1 activator/substrate assay |
| 2'-deoxycytidine-5'-triphosphate (dCTP) | Jena bioscience | NU-1002 | Compound tested in SAMHD1 activator/substrate assay |
| 2'-Deoxyguanosine-5'-(α-thio)-triphosphate (dGTPαS) | Jena bioscience | NU-424 | Non-hydrolyzable dGTP analogue for SAMHD1 activator/substrate assay |
| 2'-deoxyguanosine-5'-triphosphate (dGTP) | GE Healthcare | 27-1870-04 | SAMHD1 allosteric activator and substrate used in inhibition assay and activator/substrate assay |
| 2'-Deoxythymidine-5'-[(α,β)-imido]triphosphate (dTMPNPP) | Jena bioscience | NU-907-1 | Compound tested in SAMHD1 inhibition assay |
| 2'-deoxythymidine-5'-triphosphate (dTTP) | Jena bioscience | NU-1004 | Compound tested in SAMHD1 activator/substrate assay |
| 2'Deoxyguanosine mohohydrate (dGuo) | Sigma-Aldrich | D0901 | Compound tested in SAMHD1 inhibition assay |
| 384 well clear flat-bottom microplate | Thermo Fisher Scientific | 262160 | Assay plate |
| 96 well clear U-bottom polypropylene microplate | Thermo Fisher Scientific | 267245 | Compound dilution plate |
| Ammonium heptamolybdate tetrahydrate | Sigma-Aldrich | A1343 | Reagent required for malachite green working reagent |
| ara-Cytidine-5'-triphosphate (ara-CTP) | Jena bioscience | NU-1170 | Compound tested in SAMHD1 activator/substrate assay |
| Clofarabine-5'-triphosphate (Cl-F-ara-ATP) | Jena bioscience | NU-874 | Compound tested in SAMHD1 activator/substrate assay |
| Dimethyl sulphoxide (DMSO) | VWR | 23486.297 | Solvent |
| Ethylenediaminetetraacetic acid disodium salt dihydrate (EDTA) | Sigma-Aldrich | E5134 | EDTA stop solution component |
| Gemcitabine-5'-triphosphate (dF-dCTP) | Jena bioscience | NU-1607 | Compound tested in SAMHD1 activator/substrate assay |
| GraphPad Prism | GraphPad Software | Prism 8 | Data analysis and visualisation |
| Guanosine 5′-triphosphate (GTP) sodium salt hydrate | Sigma-Aldrich | G8877 | Allosteric activator for SAMHD1 activator/substrate assay |
| His-tagged E. coli inorganic pyrophosphatase (PPase) | Generated in house using Protein Science Facility, Karolinska Institutet | - | Recombinant PPase protein, hydrolises inorganic triphosphate and pyrophosphate to orthophosphate so it can form complex with malachite green |
| His-tagged human SAMHD1 | Generated in house using Protein Science Facility, Karolinska Institutet | - | Recombinant SAMHD1 protein, hydrolizes dNTPs into its corresponding nucleoside and inorganic triphosphate |
| Hydroxyurea | Sigma-Aldrich | H8627 | Compound tested in SAMHD1 inhibition assay |
| Lomofungin | National Cancer Institute (NCI)/Division of Cancer Treatment and Diagnosis (DCTD)/Developmental Therapeutics Program (DTP) | NSC106995 | Compound tested in SAMHD1 inhibition assay |
| Magnesium Chloride hexahydrate (MgCl2) | Sigma-Aldrich | M2670 | SAMHD1 reaction buffer component |
| Malachite green Carbinol hydrochloride | Sigma-Aldrich | 213020 | Malachite green stock component |
| Microplate Reader | Hidex | Hidex Sense Microplate reader | Data acquisition, absorption read at 630 nm wavelength |
| Sodium Chloride (NaCl) | Sigma-Aldrich | 31434 | SAMHD1 reaction buffer component |
| Sodium hydroxide (NaOH) | Sigma-Aldrich | 567530 | SAMHD1 reaction buffer component |
| Sodium phosphate (Na3PO4) | Sigma-Aldrich | 342483 | Required for phosphate standard curve |
| Sulphuric acid 95-97% | Sigma-Aldrich | 84720 | Malachite green stock component |
| Tris-Acetate salt | Sigma-Aldrich | T1258 | SAMHD1 reaction buffer component |
| Tris(2-carboxyethyl)phosphine hydrochloride (TCEP) | Sigma-Aldrich | C4706 | SAMHD1 reaction buffer component |
| Tween-20 | Sigma-Aldrich | P1379 | SAMHD1 reaction buffer and malachite green working reagent component |
Referenzen
- Franzolin, E., et al. The deoxynucleotide triphosphohydrolase SAMHD1 is a major regulator of DNA precursor pools in mammalian cells. Proceedings of the National Academy of Sciences of the United States of America. 110 (35), 14272-14277 (2013).
- Coggins, S. A., Mahboubi, B., Schinazi, R. F., Kim, B. SAMHD1 functions and human diseases. Viruses. 12 (4), 382(2020).
- Goldstone, D. C., et al. HIV-1 restriction factor SAMHD1 is a deoxynucleoside triphosphate triphosphohydrolase. Nature. 480 (7377), 379-382 (2011).
- Powell, R. D., Holland, P. J., Hollis, T., Perrino, F. W. Aicardi-Goutieres syndrome gene and HIV-1 restriction factor SAMHD1 is a dGTP-regulated deoxynucleotide triphosphohydrolase. The Journal of Biological Chemistry. 286 (51), 43596-43600 (2011).
- Morris, E. R., Taylor, I. A. The missing link: Allostery and catalysis in the anti-viral protein SAMHD1. Biochemical Society Transactions. 47 (4), 1013-1027 (2019).
- Arnold, L. H., Kunzelmann, S., Webb, M. R., Taylor, I. A. A continuous enzyme-coupled assay for triphosphohydrolase activity of HIV-1 restriction factor SAMHD1. Antimicrobial Agents and Chemotherapy. 59 (1), 186-192 (2015).
- Herold, N., et al. Targeting SAMHD1 with the Vpx protein to improve cytarabine therapy for hematological malignancies. Nature Medicine. 23 (2), 256-263 (2017).
- Schneider, C., et al. SAMHD1 is a biomarker for cytarabine response and a therapeutic target in acute myeloid leukemia. Nature Medicine. 23 (2), 250-255 (2017).
- Hollenbaugh, J. A., et al. Substrates and inhibitors of SAMHD1. PloS One. 12 (1), 0169052(2017).
- Knecht, K. M., et al. The structural basis for cancer drug interactions with the catalytic and allosteric sites of SAMHD1. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10022-10031 (2018).
- Oellerich, T., et al. Selective inactivation of hypomethylating agents by SAMHD1 provides a rationale for therapeutic stratification in AML. Nature Communications. 10 (1), 3475(2019).
- Herold, N., et al. SAMHD1 protects cancer cells from various nucleoside-based antimetabolites. Cell Cycle. 16 (11), 1029-1038 (2017).
- Rothenburger, T., et al. SAMHD1 is a key regulator of the lineage-specific response of acute lymphoblastic leukaemias to nelarabine. Communications Biology. 3 (1), 324(2020).
- Ordonez, P., et al. SAMHD1 enhances nucleoside- analogue efficacy against HIV-1 in myeloid cells. Scientific Reports. 7, 42824(2017).
- Castellví, M., et al. Pharmacological modulation of SAMHD1 activity by CDK4/6 inhibitors improves anticancer therapy. Cancers. 12 (3), 713-719 (2020).
- Rassidakis, G. Z., et al. Low-level expression of SAMHD1 in acute myeloid leukemia (AML) blasts correlates with improved outcome upon consolidation chemotherapy with high-dose cytarabine-based regimens. Blood Cancer Journal. 8 (11), 98(2018).
- Rudd, S. G., Schaller, T., Herold, N. SAMHD1 is a barrier to antimetabolite-based cancer therapies. Molecular & Cellular Oncology. 4 (2), 1287554(2017).
- Rudd, S. G., et al. Ribonucleotide reductase inhibitors suppress SAMHD1 ara-CTPase activity enhancing cytarabine efficacy. EMBO Molecular Medicine. 41, 10419(2020).
- Seamon, K. J., et al. Small molecule inhibition of SAMHD1 dNTPase by tetramer destabilization. Journal of the American Chemical Society. 136 (28), 9822-9825 (2014).
- Seamon, K. J., Stivers, J. T. A high-throughput enzyme-coupled assay for SAMHD1 dNTPase. Journal of Biomolecular Screening. 20 (6), 801-809 (2015).
- Mauney, C. H., Perrino, F. W., Hollis, T. Identification of inhibitors of the dNTP triphosphohydrolase SAMHD1 using a novel and direct high-throughput assay. Biochemistry. 57 (47), 6624-6636 (2018).
- Morris, E. R., et al. Crystal structures of SAMHD1 inhibitor complexes reveal the mechanism of water-mediated dNTP hydrolysis. Nature Communications. 11 (1), 3165(2020).
- Hansen, E. C., Seamon, K. J., Cravens, S. L., Stivers, J. T. GTP activator and dNTP substrates of HIV-1 restriction factor SAMHD1 generate a long-lived activated state. Proceedings of the National Academy of Sciences of the United States of America. 111 (18), 1843-1851 (2014).
- Baykov, A. A., Evtushenko, O. A., Avaeva, S. M. A malachite green procedure for orthophosphate determination and its use in alkaline phosphatase-based enzyme immunoassay. Analytical Biochemistry. 171 (2), 266-270 (1988).
- Hyun, M., Bohr, V. A., Ahn, B. Biochemical characterization of the WRN-1 RecQ helicase of Caenorhabditis elegans. Biochemistry. 47 (28), 7583-7593 (2008).
- Lin, H. -H., Huang, C. -Y. Characterization of flavonol inhibition of DnaB helicase: real-time monitoring, structural modeling, and proposed mechanism. Journal of Biomedicine & Biotechnology. 2012 (4), 735368(2012).
- Yang, M., Wang, G. ATPase activity measurement of DNA replicative helicase from Bacillus stearothermophilus by malachite green method. Analytical Biochemistry. 509, 46-49 (2016).
- Allard, B., Cousineau, I., Spring, K., Stagg, J. Measurement of CD73 enzymatic activity using luminescence-based and colorimetric assays. Methods in Enzymology. 629, 269-289 (2019).
- Lee, M., et al. Structure-activity relationship of sulfonyl piperazine LpxH inhibitors analyzed by an LpxE-coupled malachite green assay. ACS Infectious Diseases. 5 (4), 641-651 (2019).
- Carreras-Puigvert, J., et al. A comprehensive structural, biochemical and biological profiling of the human NUDIX hydrolase family. Nature Communications. 8 (1), 1541(2017).
- Valerie, N. C. K., et al. NUDT15 hydrolyzes 6-thio-deoxyGTP to mediate the anticancer efficacy of 6-thioguanine. Cancer Research. 76 (18), 5501-5511 (2016).
- Carter, M., et al. Human NUDT22 Is a UDP-glucose/galactose hydrolase exhibiting a unique structural fold. Structure. 26 (2), 295-303 (2018).
- Gad, H., et al. MTH1 inhibition eradicates cancer by preventing sanitation of the dNTP pool. Nature. 508 (7495), 215-221 (2014).
- Page, B. D. G., et al. Targeted NUDT5 inhibitors block hormone signaling in breast cancer cells. Nature Communications. 9 (1), 250(2018).
- Zhang, S. M., et al. Development of a chemical probe against NUDT15. Nature Chemical Biology. 16 (10), 1120-1128 (2020).
- Michel, M., et al. In silico druggability assessment of the NUDIX hydrolase protein family as a workflow for target prioritization. Frontiers in Chemistry. 8, 443(2020).
- Zhang, J., Chung, T., Oldenburg, K. A simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67(1999).
- Baykov, A. A., Evtushenko, O. A., Avaeva, S. M. A malachite green procedure for orthophosphate determination and its use in alkaline phosphatase-based enzyme immunoassay. Analytical Biochemistry. 171 (2), 266-270 (1988).
- Markossian, S., et al. Assay guidance manual. Eli Lilly & Company and the National Center for Advancing Translational Sciences. , Bethesda (MD). (2004).
- Holdgate, G. A., Meek, T. D., Grimley, R. L. Mechanistic enzymology in drug discovery: a fresh perspective. Nature Reviews Drug Discovery. 17 (2), 115-132 (2018).
- Tsesmetzis, N., Paulin, C. B. J., Rudd, S. G., Herold, N. Nucleobase and nucleoside analogues: resistance and re-sensitisation at the level of pharmacokinetics, pharmacodynamics and metabolism. Cancers. 10 (7), 240(2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten