Method Article
Anwendung der CRISPR-Interferenz (CRISPRi) zur Gen-Silencing bei pathogenen Leptospira-Spezies
In diesem Artikel
Zusammenfassung
Hier wird die Anwendung der CRISPR-Interferenz (CRISPRi) für spezifisches Gen-Silencing bei Leptospira-Spezies beschrieben. Leptospira-Zellen werden durch Konjugation mit Plasmiden transformiert, die dCas9 (katalytisch "totes" Cas9) und eine Single-Guide-RNA (sgRNA) exprimieren, die für die Basenpaarung zum gewünschten genomischen Ziel verantwortlich ist. Methoden zur Validierung des Gen-Silencing werden vorgestellt.
Zusammenfassung
Leptospirose ist eine weltweit vernachlässigte Zoonose, die für mindestens 1 Million Fälle pro Jahr und fast 60.000 Todesfälle verantwortlich ist. Die Krankheit wird durch pathogene und virulente Bakterien der Gattung Leptospiraverursacht, entweder durch direkten Kontakt mit den Bakterien oder indirekt durch Exposition gegenüber kontaminiertem Wasser oder Boden. Haus- und Wildtiere fungieren als Reservoirwirte der Infektion und scheiden Leptospiren aus kolonisierten Nierentubuli der Niere über Urin in die Umwelt aus. Die Erzeugung mutierter Stämme von Leptospira ist entscheidend, um pathogene Infektionsmechanismen zu bewerten und zu verstehen. CRISPR-Interferenz (CRISPRi) hat sich als einfaches, erschwingliches und spezifisches Werkzeug für das Gen-Silencing in pathogenen Leptospiraerwiesen. Daher werden die methodischen Details zur Gewinnung der Plasmidkonstrukte, die sowohl dCas9 als auch Leit-RNA enthalten, zur Abgabe von Plasmiden an Leptospira durch Konjugation mit dem E. coli-Stamm β2163 und zur transkonjuganten Erholung und Bewertung beschrieben. Darüber hinaus ermöglichen die kürzlich beschriebenen Hornsby-Alt-Nally (HAN) Medien die relativ schnelle Isolierung und Selektion von mutierten Kolonien auf Agarplatten.
Einleitung
Leptospirose ist eine weltweit vernachlässigte Zoonose, die durch pathogene und virulente Arten der Gattung Leptospiraverursacht wird. Beim Menschen ist die Krankheit für mehr als eine Million Fälle und 60.000 Todesfälle pro Jahr weltweitverantwortlich 1,2. Bisher gibt es keinen langfristigen und wirksamen Impfstoff gegen die Krankheit. Die Identifizierung von Virulenzfaktoren und pathogenen Mechanismen ist entscheidend für die Entwicklung besserer therapeutischer und prophylaktischer Strategien. Daher ist die Fähigkeit, genetische Mutationen zu erzeugen und den resultierenden Phänotyp zu bewerten, entscheidend für die funktionelle Genomanalyse3.
Der Aufbau von Mutanten in pathogenem Leptospira galt bisher als von Natur aus ineffizient, mühsam, kostspielig und schwer umzusetzen. Dieses Szenario änderte sich drastisch mit der Anwendung der jüngsten CRISPR-Interferenz (CRISPRi) auf saprophytische4 und pathogene5 Leptospiren.
Gen-Silencing wird durch die Expression von zwei Komponenten erreicht: einer Variante des CRISPR/Cas(clustered regularly interspaced short palindromic repeat/CRISPR associated) Enzym Cas9 aus Streptococcus pyogenes,katalytisch totes Cas9 (dCas9) genannt und einer Single-Guide-RNA (sgRNA), die nach dem gewünschten Target editiert werden kann6,7,8. dCas9-Protein, wenn es an die sgRNA gebunden ist, wird durch Watson- und Crick-Basenpaarung auf spezifische DNA-Ziele gerichtet, was zu einer sterischen Blockade der RNA-Polymerase-Elongation führt, was zu einer Genstummung aufgrund der blockierten Gentranskription führt7 (Abbildung 1).
Dieses Manuskript zielt darauf ab, die Konstruktion des Plasmids für die Exprimation von dCas9 und sgRNA, die Konjugation zwischen Spender E. coli β2163 und Empfänger-Leptospira-Zellen, die transkonjugante Erholung und schließlich die Validierung ausgewählter Mutantenkolonien zu beschreiben.
Protokoll
1. Protospacer-Definition und Plasmid-Konstruktion
HINWEIS: In diesem Abschnitt wird der erste Schritt der Auswahl geeigneter Protospacer für die Konstruktion der sgRNA und die weitere Ligatur in pMaOri.dCas9 beschrieben (Abbildung 1). Diese Protospacer-Sequenz besteht aus einer 20-Nukleotid-Sequenz gegen das gewünschte Ziel.
- Erhalten Sie die Nukleotidsequenz des Gens, das für das Silencing bei GenBank (https://www.ncbi.nlm.nih.gov/genbank) von Interesse ist. Senden Sie es an den CHOPCHOP-Webserver (http://chopchop.cbu.uib.no/), wobei die Parameter für Streptococcus pyogenes Cas9 und protospacer angrenzendes Motiv NGG definiert sind, nachdem Sie das "Fasta Target" ausgewählt haben. Definieren Sie die Parameter zu "CRISPR/Cas9" und PAM (protospacer adjacent motif) NGG.
- Basierend auf den erhaltenen Ergebnissen wählen Sie Protospacer mit der bestmöglichen Punktzahl (grüner Pfeil) aus, die sich so nah wie möglich am 5'-Ende der kodierenden Region befinden und vor allem im Vorlagenstrang (Minus) enthalten sind, da sich die sgRNA für ein vollständiges Gen-Silencing mit dem kodierenden Strang des Gens paaren muss.
HINWEIS: Das NGG-Motiv ist nicht in der endgültigen sgRNA-Sequenz enthalten. - Verwenden Sie den lipL-32-Promotor, um die Einzelleiter-RNA zu exprimieren, die eine variable 20-Nukleotid-Sequenz am 5'-Ende und eine konservierte dCas9-Gerüstsequenz enthält. Verschmelzen Sie die 20-nt-Sequenz, protospacer genannt, mit dem lipL-32-Promotor (an seinem 5'-Ende) und dem sgRNA-Gerüst (3'-Ende) (Abbildung 1B).
HINWEIS: Für einen gut definierten lipL32-Promotor verwenden Sie die Promotorregion, die -334 umfasst, zum TSS (Transcription Start Site, basierend auf Zhukova et al.9). Überprüfen Sie die Ergänzungsdatei für die endgültige sgRNA-Kassette. - Generieren Sie die sgRNA-Kassette mittels sequenzieller PCR 5 oder lassen Sie sie voneinem kommerziellen Anbieter synthetisieren.
- Nachdem Sie die Kassette erhalten haben, ligaieren Sie sie in pMaOri.dCas9-Plasmid an der XmaI-Restriktionsstelle an beiden Enden (cccggg)4.
- Verdauen Sie sowohl die sgRNA-Kassette als auch das pMaOri.dCas9-Plasmid mit dem XmaI-Restriktionsenzym und fahren Sie mit der Ligatur fort (Abbildung 1B).
- Führen Sie die Klonierungsschritte im dT auxotrophen E. coli-Stamm π110durch, aufgrund des pMaOri11 (und damit pMaOri.dCas9) Ursprungs der Replikation, R6K-gamma.
HINWEIS: Ein detailliertes Protokoll für Ligation und Klonauswahl finden Sie in früheren Veröffentlichungen von Fernandes und Nascimento12. sgRNA-gesteuertes dCas9 bindet an den kodierenden Strang des ausgewählten interessierenden Gens und behindert daher die RNA-Polymerase-Elongation (Abbildung 1C), was zu Gen-Silencing führt.
2. Leptospira-Transformation durch Konjugation
HINWEIS: Ein grafisches Schema dieses Schritts ist in Abbildung 2 dargestellt. Um HAN-Medien und HAN-Platten herzustellen, verweisen wir auf Hornsby et al.13 und Fernandes et al.5.
- Züchten Sie pathogene Leptospira-Zellen bei 29 oder 37 °C in HAN-Medien13 unter Agitation, indem Sie eine gesättigte Kultur in frischem HAN bei 1:100 verdünnen; Typischerweise benötigt der L. interrogans serovar Copenhageni Stamm Fiocruz L1-130 4-6 Tage, um die entsprechende Zelldichte zu erreichen.
- Stellen Sie sicher, dass die Kulturen bei 420 nm (2 bis 5 x 10 8 Zellen/ml) einen O.D. von0,2-0,4 erreichen, bevor Sie sie für die Konjugation verwenden.
HINWEIS: Da HAN-Medien mit zunehmender Zelldichte die Farbe ändern (aufgrund von Phenolrot in DMEM-Medien), Zentrifuge (4.000 x g,15 min, Raumtemperatur) 1 ml des Nährmediums, um Leptospiren zu entfernen und Überstand als Rohling für die Messung des O.D. aufzutragen.
- Stellen Sie sicher, dass die Kulturen bei 420 nm (2 bis 5 x 10 8 Zellen/ml) einen O.D. von0,2-0,4 erreichen, bevor Sie sie für die Konjugation verwenden.
- Transformieren Sie den konjugativen E. coli-Stamm β2163, auxotroph für Diaminopimelsäure (DAP), mit dem Plasmid pMaOri.dCas9, das die sgRNA-Kassette enthält. Für E. Coli-Transformation, verwenden Sie entweder Hitzeschockprotokolle oder Elektroporation. Fügen Sie die Transformation mit dem Plasmid pMaOri.dCas9 ohne sgRNA-Kassette als Kontrolle ein.
- Für die Hitzeschock-Transformation die Plasmid-DNA (100 ng) mit chemisch kompetenten E. coli-Zellen mischen und 30 min auf Eis inkubieren. Führen Sie einen Hitzeschock bei 42 °C für 90 s durch und legen Sie ihn für 5 minuten wieder auf Eis. Die Zellen durch Zugabe von 1 ml LB-Medium wiederherstellen, bei 37 °C für 1 h inkubieren und mit der Beschichtung fortfahren.
- Verwenden Sie für die Elektroporation elektrokompetente Zellen, die mit 100 ng Plasmid-DNA gemischt sind. Verwenden Sie die folgenden Parameter für den Puls: 1,8 kV, 100 Ω und 25 μF. Stellen Sie die Zellen wie oben beschrieben wieder her.
- Die transformierten Spender-E. coli-Zellen werden in LB-Agarmedium mit Diaminopimelinsäure (DAP) (0,3 mM) und Spectinomycin (40 μg/ml) zur Auswahl von Plasmiden platten.
- Für die Konjugation wählen Sie eine Kolonie aus jeder Platte einen Tag vor dem Tag der Konjugation (die durch Überwachung des O.D. von Leptospirenkulturen bestimmt wird).
- Wählen Sie eine Kolonie von E. coli β2163 aus den leeren pMaOri.dCas9- und eine aus pMaOri.dCas9sgRNA-Platten aus. Lassen Sie sie über Nacht in 10 ml LB plus DAP und Spectinomycin bei 37 °C wachsen.
- Am nächsten Tag verdünnen Sie die gesättigten Kulturen 1:100 in 10 ml frischem LB plus DAP (hier nicht das Antibiotikum einschließen) bis OD420nm von 0,2-0,4. Normalerweise dauert es 2-3 Stunden, bis E. coli diese Dichten erreicht hat.
- In einer BSL2-Biosicherheitshaube montieren Sie eine Filtervorrichtung, indem Sie einen Membranfilter mit einem Durchmesser von 0,1 μm Porengröße von 25 mm auf der Oberseite der Glasbasis platzieren. Legen Sie einen 15 mL Glastrichter auf die Oberseite und halten Sie beide Teile mit Federklemmen. Schließen Sie das Glas an eine Vakuumpumpe an und fügen Sie die Kulturen zur Filtration in den Trichter ein.
- Fügen Sie 5 ml Leptospira-Kultur in den Trichter hinzu. Fügen Sie ein Volumen von E. coli hinzu, um das 1:1-Verhältnis basierend auf den OD420nm-Werten beider Kulturen zu bilden. Schalten Sie die Vakuumpumpe ein und konzentrieren Sie die Zellen durch Filtration. Nach der Zellkonzentration im Membranfilter vorsichtig abrufen. Stellen Sie sicher, dass das Medium durch die Membran gefiltert wird.
HINWEIS: Die Filtration dauert 5 bis 10 Minuten. - Legen Sie den Filter auf eine handelsübliche EMJH-Platte (siehe Materialtabelle),ergänzt mit DAP (0,3 mM). Stellen Sie sicher, dass die Bakterienseite oben ist. Inkubieren Sie die Platten bei 29 °C für 24 h.
HINWEIS: Wenn HAN oder ergänzte hauseigene EMJH14-Platten verwendet werden, kann sich E. coli vermehren und das beabsichtigte 1:1-Verhältnis überwinden, was wiederum die Konjugationseffizienz verringern kann5. - Nach 24 h die Filter von den Platten zurückholen und jeden einzelnen Filter in ein 50 mL konisches Rohr legen.
- Verwenden Sie 1 ml flüssiges HAN-Medium, um die Zellen durch umfangreiches Pipettieren und Wirbeln von der Filteroberfläche zu lösen.
- Visualisieren Sie die wiederhergestellten gemischten Bakterienlösungen durch Dunkelfeldmikroskopie, um die Zelllebensfähigkeit und -motilität sowie die Leptospira: E. coli-Proportionen zu überprüfen.
HINWEIS: Zu diesem Zeitpunkt können äquivalente Zahlen von E. coli und Leptospira gesehen werden. - Verteilen Sie 100-200 μL dieser Kultur auf HAN-Platten, die 0,4% inaktiviertes Kaninchenserum und 40 μg/ ml Spectinomycin enthalten. Inkubieren Sie Platten bei 37 °C in einer 3% CO2 Atmosphäre.
HINWEIS: Normalerweise bilden L. interrogans serovar Copenhageni Stamm Fiocruz L1-130 Zellen Kolonien in 5-7 Tagen auf Kontrollplatten und in 8-10 Tagen auf Spectinomycinplatten. In diesem Stadium wird E. coli nicht wachsen, da sie für DAP auxotroph sind. - Zur Kontrolle verdünnen Sie Kulturen mit 104 Leptospiren/ml und fügen 100 μL auf Platten ohne Antibiotikum hinzu, um das Leptospiralwachstum zu überwachen.
3. Kolonieselektion und transkonjuganes Wachstum und Validierung
HINWEIS: Kolonien sollten bis zum 10. Tag sichtbar sein. Sie sind jedoch nicht allzu einfach zu visualisieren. Normalerweise sind HAN-Platten zu diesem Zeitpunkt aufgrund der getrockneten Zellen, die verteilt wurden, etwas undurchsichtig und Leptospira-Kolonien können vor dem weißlichen Hintergrund als transparenter Halo erscheinen. Es wird empfohlen, die Platten in verschiedenen Winkeln zu betrachten, um einen unterschiedlichen Lichteinfall zu erzielen, wodurch die Kolonien sichtbarer werden. Bei längeren Inkubationszeiten können Kolonien ein dichteres Aussehen erhalten, und in diesem Fall präsentieren sie sich als milchige Halos vor einem dunklen Hintergrund.
- Fügen Sie 100 μL flüssiges HAN-Medium in jede 1,5-ml-Mikroröhre hinzu, um Mutanten zurückzugewinnen. Nehmen Sie mindestens 3 Kolonien von jedem Teller.
- Mit Hilfe einer Mikropipettenspitze "graben" Sie das Agar, um die Kolonien von den Platten zu holen, da Leptospiralkolonien unter der Oberfläche sein können.
HINWEIS: Es wird erwartet, dass Agar in diesem Stadium mitgenommen wird. Kolonien sollten von Kontrollplatten entnommen werden, die leeres pMaOri.dCas9-Plasmid enthalten, und Platten mit Leptospiren, die Plasmide enthalten, die sowohl dCas9 als auch Einzelleiter-RNA exprimieren, die für das Zielgen entwickelt wurden. - Die gesammelte Kolonie in 100 μL HAN-Medien in einer 1,5 ml Mikroröhrchen abgeben und kräftig homogenisieren. Stellen Sie in diesem Stadium einen maximalen Bruch der Agarintegrität sicher, um Zellen freizusetzen. Wirbel die Aufhängung für 10 s.
- Mit Hilfe einer Mikropipettenspitze "graben" Sie das Agar, um die Kolonien von den Platten zu holen, da Leptospiralkolonien unter der Oberfläche sein können.
- Visualisieren Sie die zurückgewonnenen Zellen durch Dunkelfeldmikroskopie bei einer 200-400-fachen Vergrößerung, indem Sie einen 5 μL-Tropfen auf einen Glasobjektträger geben und die Proben sofort mit einem Deckblatt abdecken.
- Bestätigen Sie das Vorhandensein von lebenden und lebensfähigen Leptospiren, die aus den Kolonien gewonnen wurden.
- Nach der Visualisierung und Bestätigung lebensfähiger Leptospiren werden 100 μL Zellen auf flüssige HAN-Medien mit 40 μg/ml Spectinomycin übertragen.
- Nach dem Wachstum in flüssigen HAN-Medien bewerten Sie die Kulturen auf das Vorhandensein des Plasmids mit Primer pMaOri2 F (ACGCAATGTATCGATACCGAC) und R (ATAGGTGAAGTAGGCCCACCC), die die Region erkennen, die die sgRNA-Kassette flankiert.
- Sammeln Sie 200 μL Kultur, Zentrifuge (4.000 x g,15 min), entsorgen Sie den Überstand und resuspendieren Sie das resultierende Pellet in 20 μL Wasser.
- Verwenden Sie diese Suspension als Vorlage für zusätzliche PCR, ohne dass eine DNA-Extraktionerforderlich ist 12.
HINWEIS: Zellen mit pMaOri.dCas9 geben ein Amplicon von 281 bp ab, verglichen mit denen, die die Plasmide mit der sgRNA-Kassette enthalten, die ein Amplicon von 723 bp ergibt.
- Zur Bestätigung des Gen-Silencings führen Sie einen Immunoblot durch, bei dem Zellextrakte aus Transkonjugantien verwendet werden, die nur pMaOri.dCas9 (Negativkontrolle) und pMaOri.dCas9sgRNA enthalten.
- Tragen Sie das Äquivalent von 5 x 107 Zellen pro Spur des Natriumdodecylsulfat (SDS) Polyacrylamidgels auf.
- Elektrotransferproteine zu einer Membran zur Inkubation mit geeigneten Antikörpern. Neben dem Antikörper gegen das Zielgen zum Stummschalten verwenden Sie einen weiteren zur Belastungskontrolle.
- Bewahren Sie die mutierten Kulturen in HAN-Medien plus Spectinomycin auf, um das Plasmid zu erhalten. Wenn keine Antibiotika auf das Medium aufgetragen werden, kann für mindestens drei Passagen ein vollständiges Gen-Silencing beobachtet werden5.
Ergebnisse
Obwohl der CG-Gehalt in Leptospira spp.-Genomen typischerweise bei etwa 35% liegt; praktisch jedes Gen enthält wahrscheinlich das PAM 5'NGG 3'; dieses Motiv muss im Vorlagenstrang berücksichtigt werden. Nach Eingabe der kodierenden Sequenz eines Gens (von Start bis Stop Codons), basierend auf den CHOPCHOP-Ergebnissen, müssen Protospacer am Minusstrang (-, Template) ausgewählt werden. Es ist wichtig, das NGG-Motiv nicht in den 20-nt-sgRNA-Protospacer aufzunehmen.
Wenn die Konjugation mit einem Spender-Empfänger-Zellanteil von 1:1 durchgeführt wird, sollten Transkonjugantenkolonien nach 24 h auf der Oberfläche von EMJH-Agarplatten plus DAP und 200 μL der wiederhergestellten Bakteriensuspension auf HAN plus Spectinomycin-Agarplatten verteilt werden, transkonjugierende Kolonien sollten nach etwa 8-10 Tagen sichtbar sein. Die Verteilung dieses Volumens führt normalerweise zu 20-40 Kolonien pro Platte (Abbildung 3A). Um die Zelllebensfähigkeit nach der Konjugation zu überprüfen, können Zellen ohne Antibiotikaauswahl auf HAN-Platten verteilt werden. In diesem Fall können Kolonien bereits nach 7 Tagen beobachtet werden. HAN-Platten färben sich in einer 3% CO2-Atmosphäre hellgelb.
Nach Koloniepicking und Wachstum in flüssigen Medien plus Spectinomycin kann die PCR mit ganzen Zellen und pMaOri2-Primern für eine erste Qualitätskontrolle der Transkonjugantien verwendet werden (Abbildung 3B). Leptospiralzellen, die das Kontrollplasmid pMaOri.dCas9 enthalten, sollten zu einem Amplicon von 281 bp führen, während zellen, die das Plasmid zum Stummschalten enthalten, dh sowohl dCas9 als auch sgRNA enthalten, zu einem Amplicon von 723 bp führen sollten. pMaOri2 F- und R-Primer wurden entwickelt, um die XmaI-Restriktionsstelle zu flankieren, die während der sgRNA-Kassettenligatur verwendet wird.
Mit der Bestätigung der Plasmidpräsenz können Zellen aus dem Medium geerntet, zweimal mit PBS gewaschen und dann verwendet werden, um einen Ganzzellextrakt für die Immunblotierung vorzubereiten. Wenn die Stummschaltung stattgefunden hat, sollten die Zielproteine, in diesem Fall entweder LipL32 oder sowohl LigA als auch LigB, nur in den Wildtypzellen und in solchen, die pMaOri.dCas9 enthalten, beobachtet werden; auch bei höheren Expositionszeiten sollten in den Zellen, die pMaOri.dCas9sgRNA enthalten, keine entsprechenden Proteine sichtbar sein (Abbildung 3C).
Wenn Experimente zur Beurteilung der Leptospiralvavirulenz nach dem Gen-Silencing geplant sind, sollten Kulturen, die für die Konjugation verwendet werden, virulente Leptospiramit niedriger Passage sein. Nachdem das Gen-Silencing bestätigt wurde, können mehrere Aliquoten als Backup eingefroren werden. Wenn das stille Gen einen messbaren Phänotyp aufstellt, z.B. basierend auf früheren Arbeiten mit rekombinanten Proteinen, können Kulturen zur Validierung verwendet werden und in diesem Fall können Zellen, die nur pMaOri.dCas9 enthalten, als Negativkontrolle eingeschlossen werden.
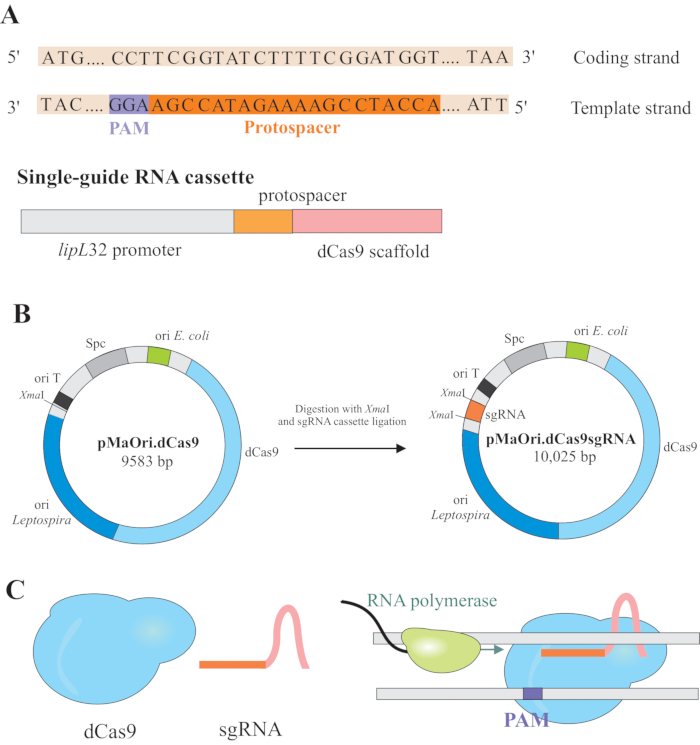
Abbildung 1: Entwicklung von dCas9- und sgRNA-exprimierendem Plasmid. (A) Ein 20-nt langer Protospacer, gefolgt vom S. pyogenes dCas9 PAM 5'-NGG-3', wird innerhalb des Vorlagenstrangs des Zielgens ausgewählt, so dass die nachfolgende sgRNA Watson- und Crick-Basenpaarung zum entsprechenden kodierenden Strang durchführen kann, was zu einem vollständigen Gen-Silencing führt. (B) Die sgRNA-Kassette besteht aus dem lipL32-Promotor, dem 20-nt-Protospacer und dem dCas9-Gerüst. Das pMaOri.dCas9-Plasmid wird als Rückgrat für die sgRNA-Kassettenligatur an der XmaI-Restriktionsstelle verwendet. Das resultierende Plasmid, pMaOri.dCas9sgRNA genannt, wird an Leptospiren abgegeben, und die Expression von dCas9 und sgRNA ist für das Gen-Silencing verantwortlich. (C) sgRNA-gerichtetes dCas9 wirkt als physikalische Barriere für die Dehnung der RNA-Polymerase und behindert daher die Transkription. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
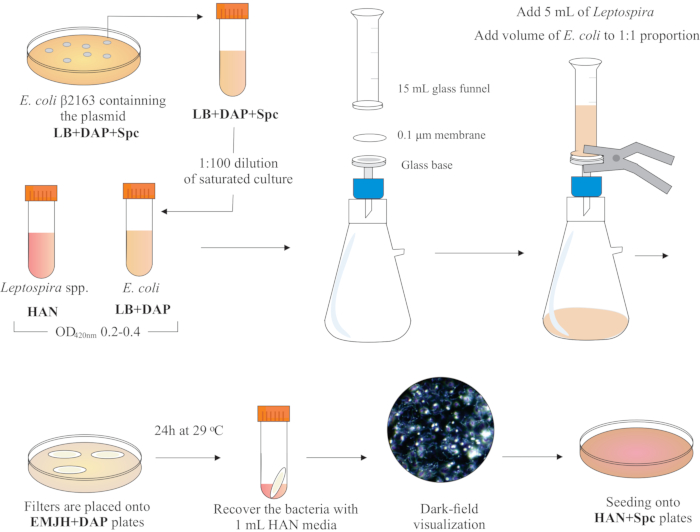
Abbildung 2: Schematische Darstellung des Konjugationsprotokolls. Die gewünschte Leptospira-Art wird in HAN-Medien unter Agitation bis zum O.D. von 0,2-0,4 (Mid-Log-Phase) bei 420 nm gezüchtet. Einen Tag vor der Konjugation wird eine Kolonie rekombinanter Spender E. coli β2163, die das interessierende Plasmid enthält, aus LB + DAP + Spc-Agarplatten ausgewählt, da Zellen über Nacht in flüssigem LB mit der gleichen Ergänzung gezüchtet werden. Am nächsten Tag, gesättigtes E. Coli-Kulturen werden in LB plus DAP verdünnt und bis zu O.D. von 0,2-0,4 bei 420 nm gezüchtet. Sowohl Donor E. coli als auch Empfänger Leptospira werden im Zellverhältnis von 1:1 auf die Oberfläche eines 0,1 μm Filters durch eine Filtrationsvorrichtung unter Unterdruck gemischt. Dann werden Filter auf die mit DAP ergänzten EMJH-Agarplatten gelegt und die Inkubation erfolgt für 24 h bei 29 °C. Die Verwendung von EMJH begrenzt die Proliferation von E. coli, und das beabsichtigte 1:1-Verhältnis wird beibehalten. Bakterien werden aus Filtern durch Pipettieren mit 1 ml HAN-Medien zurückgewonnen, und Suspensionen werden unter Dunkelfeldmikroskopie visualisiert. Schließlich werden 100-200 μL jeder Suspension auf HAN-Agarplatten mit 0,4% Kaninchenserum ausgesät und bei 37 °C in 3% CO2inkubiert. In diesem Stadium wird DAP weggelassen, und infolgedessen wachsen auxotrophe E. coli nicht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
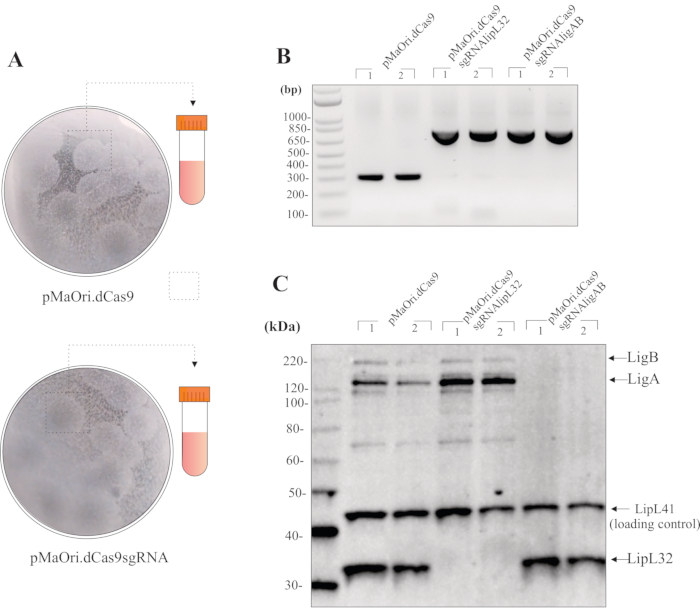
Abbildung 3: Repräsentative Ergebnisse für die Bewertung von Mutanten. (A) Kolonien aus Platten, die Leptospira enthalten, transformiert mit leerem pMaOri.dCas9 (Negativkontrolle für weitere Experimente) und Plasmiden pMaOri.dCas9sgRNA (mit gezieltem Gen, das zum Schweigen gebracht wird) werden gepflückt, in flüssigem HAN kräftig homogenisiert und in flüssigem HAN gezüchtet, das Spectinomycin enthält. Rekombinante Zellen können mittels PCR mit Primern validiert werden, die die XmaI-Stelle in pMaOri.dCas9 flankieren. (B) In diesem Fall führten Zellen, die pMaOri.dCas9 enthielten, nur zu einem Amplicon von 281 bp, während die Zellen, die das Plasmid zum Stummschalten enthielten und sowohl dCas9 als auch sgRNA enthielten, ein Amplicon von 723 bp zeigten. Nach Bestätigung des Vorhandenseins der Plasmide wurde das Gen-Silencing durch Immunoblot-Analyse validiert. (C) Eine Inkubation mit Antikörpern sowohl gegen das Zielprotein als auch gegen ein Ladekontrollprotein wird empfohlen; Im repräsentativen Immunoblot werden Ganzzellextrakte aus Transkonjugantien gezeigt, die pMaOri.dCas9 allein oder mit sgRNA-Kassetten enthalten, die auf lipL32 (pMaOri.dCas9sgRNAlipL32) und sowohl LigA- als auch LigB-Gen (pMaOri.dCas9sgRNAligAB) abzielen. Die Co-Inkubation mit anti-LipL32, anti-LigAB und anti-LipL41 (Non-Target, Loading Control) bestätigt, dass die Expression des LipL32-Proteins in Zellen, die pMaOri.dCas9sgRNAlipL32 und sowohl LigA als auch LigB enthalten, in Zellen, die pMaOri.dCas9sgRNAligAB enthalten, aufgehoben wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei: Single Guide RNA (sgRNA) Kassettensequenz. Die sgRNA-Transkription wird durch den konstitutiven lipL32-Promotor (fette Nukleotide) gesteuert. sgRNA besteht aus 20 Nukleotiden, die sich auf den Protospacer beziehen und für die Basenpaarung mit dem kodierenden Strang des Zielgens verantwortlich sind, und der dCas9-Gerüstsequenz (unterstrichene Nukleotide). Xma I-Restriktionsstellen (cccggg) sind an beiden Enden für die Ligation bei pMaOri.dCas9 Plasmid enthalten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Nach der frühen Sequenzierung der pathogenen15,16,17,18 und saprophytischen19 Leptospira-Arten beleuchtete das Data Mining des Genoms verschiedene Aspekte der leptospiralen Pathogenese. In den meisten Fällen wurde die Proteinfunktion unter Verwendung des rekombinanten Gegenstücks von mutmaßlichen leptospiralen oberflächenexponierten Proteinen und anschließender Spekulation der nativen Proteinfunktion20,21,22,23,24,25,26untersucht.
Die Erzeugung von Mutanten und die Bewertung ihres jeweiligen Phänotyps sind Schlüsselkomponenten der funktionellen Genomanalyse. Erste Versuche, Mutanten in Leptospira spp. zu erzeugen, wurden durch zufällige Transposonmutagenese27,28,29,30erreicht; Nach umfangreicher und mühsamer Analyse zur Ableitung der Identität gestörter Gene wurde jedoch festgestellt, dass nur 15% aller Gene in L. interrogans serovar Manilae gestört waren27. Der gezielte Gen-Knockout wurde weiter durch homologe Rekombination unter Verwendung von Selbstmordplasmiden erreicht, um eine Antibiotikaresistenzkassette zu liefern, die von homologen Armen innerhalb des gewünschten Ziels flankiert wird31,32.
Durch die Anwendung dieser Technologien wurden mehrere Aspekte der leptospiralen Grundlagenbiologie und Virulenz untersucht31,33,34,35,36,37. Die Entwicklung des E. coli-Leptospira konjugativen Shuttle-Vektors, pMaOri 11, ermöglichte die Abgabe von Komponenten für das episomale Gen-Silencing.
Es wurde bereits gezeigt, dass der Cas9-induzierte Doppelstrangbruch für Leptospira spp. tödlich ist und alternativ die katalytisch inaktive Variante des Enzyms, dCas9, verwendet werden kann, um Gen-Silencing sowohl bei saprophytischen als auch bei pathogenen Spezies4,5zu erreichen. Durch die Verwendung des Plasmids pMaOri.dCas9 als Rückgrat für die sgRNA-Kassettenligatur kann aufgrund der Expression von dCas9 und sgRNA ein spezifisches und stabiles Gen-Silencing erzielt werden; dCas9-gebundene sgRNA führt das Protein durch Watson-Crick-Basenpaarung zum gewünschten Ziel.
Für ein vollständiges Gen-Silencing sollte der Protospacer basierend auf dem Vorlagenstrang des gewünschten Gens so konzipiert werden, dass die Basenpaarung der sgRNA mit dem kodierenden Strang erfolgt. Basierend auf einem durchschnittlichen C+G-Gehalt von 35% in Leptospira spp. tritt das PAM 5'-NGG-3' mindestens 3 mal alle 100 bp auf. Daher wird praktisch jedes Gen im Genom von Leptospira mindestens ein PAM enthalten. Wird das Motiv NGG jedoch nicht gefunden, kann das alternative NAG-Motiv bewertet werden.
Frühere Gen-Silencing-Techniken wie Zinkfinger und TALE (Transkriptionsaktivator-ähnliche Effektoren) beruhten auf der Konstruktion eines anderen Proteins für jedes Ziel, was diese Techniken mühsam und kostspielig machte38. Im Falle von CRISPRi ist die variable Komponente die sgRNA, so dass nur die 20 bp am 5'-Ende geändert werden müssen. Vollständiges, stabiles und gezieltes Gen-Silencing wurde nicht nur bei Leptospira spp.4,5, sondern auch bei anderen Bakterien8,39,40,41beobachtet.
Die Entwicklung von HAN media13 begünstigte die Erholung von Mutanten, indem die Inkubationszeit für die Koloniebildung drastisch verkürzt wurde und Leptospira bei 37 oC wachsen kann. Während des Konjugationsschritts wird seine Verwendung jedoch nicht empfohlen, da sich E. coli in diesem Medium stark vermehren und das beabsichtigte 1:1-Verhältnis zwischen Spender- und Empfängerzellen überwinden kann. In diesem Stadium ist EMJH plus DAP die bessere Wahl, da sich E. coli in diesem Medium schlecht replizieren. Es ist erwähnenswert, dass einige Labore intern ergänztes EMJH herstellen, das zusätzliche Komponenten enthalten kann, die auch das Wachstum von E. coli-Zellen unterstützen könnten.
Das hier vorgestellte Konjugationsprotokoll wurde für den L. interrogans serovar Copenhageni Stamm Fiocruz L1-130 optimiert und erwies sich auch bei der Umwandlung eines kürzlich isolierten pathogenen Stammes aus Bodenproben als wirksam5. Erste Versuche mit verschiedenen Serovaren von L. borgpetersenii-Arten deuten auf geringere Konjugationseffizienzen mit dem beschriebenen Protokoll hin. Daher sollten bei der Arbeit mit verschiedenen Spezies/Serovaren von Leptospiradie optimalen Bedingungen für die Konjugation empirisch unter Berücksichtigung der Spender-Empfängerzell-Proportionen, der anfänglichen Zelldichten, der Konjugationsmedien und der Zeit (24 und 48 h) bestimmt werden. Es ist vernünftig anzunehmen, dass sich verschiedene Leptospira-Arten und Serovare mit unterschiedlichen Konjugationsprotokollen unterschiedlich verhalten.
Obwohl saprophytische Leptospira-Kolonien auf Platten relativ einfach zu visualisieren sind, können pathogene Kolonien schwieriger zu beobachten sein. Normalerweise können durch die Verwendung von HAN-Medien, die mit 0,4% Kaninchenserum und Spectinomycin ergänzt werden, transkonjugante Kolonien am Tag 10 beobachtet werden. Unserer Erfahrung nach präsentieren sich Kolonien zunächst als transparenter Halo an der Medienoberfläche. Im Videoprotokoll werden dichtere Kolonien nach 14 Tagen Wachstum gezeigt, da die transparenten schwer zu filmen waren. In diesem Stadium kann das Drehen der Platte, um einen unterschiedlichen Lichteinfall zu erzielen, und das Wechseln zwischen weißem und dunklem Hintergrund helfen, Kolonien zu identifizieren.
Für die Mutantenvalidierung bietet Immunoblotting einen einfachen Ansatz; Da jedoch nicht immer Antikörper gegen Zielproteine zur Verfügung stehen, können alternative Strategien zur Validierung des Gen-Silencing verfolgt werden. Quantitative Reverse-Transkriptase-PCR (qRT-PCR) unter Verwendung von Primern für das Zielgen und einer konstitutiven Kontrolle ist wirksam, um genstummen zu validieren, da sgRNA-gesteuertes dCas9 für die Blockierung der Gentranskription verantwortlich ist. Wenn das Zielgen für ein klar definiertes Proteinband in Proteingelen kodiert, kann SDS-PAGE Silencing und gemäß dem lipL32 Gen Silencing5nachweisen. Wenn LPS-Biosynthesegene zum Schweigen gebracht werden, kann LPS-Färbung eingesetzt werden; im Falle der Stummschaltung von Genen, die für Enzyme mit genau definierten Substraten kodieren, sind kinetische Assays mit chromogenen Substraten gültige Strategien; β-Galactosidase-Schalldämpfen in L. biflexa wurde durch die Verwendung von X-gal- und ONPG-Substraten (Ortho-Nitrophenyl-β-Galactosid) validiert4.
Nach Bestätigung des Gen-Silencing können Experimente zur weiteren Bewertung des Phänotyps konzipiert werden. Bindungsassays können im Falle der Stummschaltung bakterieller Adhäsine durchgeführt werden; Serum-Challenge-Assays bestätigten die Rolle von LigA und LigB beim Serumüberleben, die durch das pathogene Leptospira5gezeigt wurde. Mutanten können auch verwendet werden, um Tiere zu impfen, um die Dämpfung der Virulenz zu beurteilen; In diesem Fall sollten Tiere, die mit der Mutante geimpft wurden, nur mit Solchen verglichen werden, die mit Zellen infiziert sind, die pMaOri.dCas9 enthalten.
Zusammenfassend beschreibt das aktuelle Protokoll die Anwendung von CRISPRi für das Gen-Silencing bei pathogenen Leptospira-Spezies unter Verwendung von HAN-Medien, um die Mutantenerholung innerhalb von 10 Tagen zu erleichtern. Gen-Silencing in Kombination mit funktioneller genomischer Analyse wird unser Verständnis der pathogenen Mechanismen von Leptospiraverbessern und letztendlich zur Entwicklung besserer prophylaktischer Strategien zur Krankheitskontrolle führen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
USDA ist ein Anbieter und Arbeitgeber für Chancengleichheit. Die Erwähnung von Handelsnamen oder Handelsprodukten in dieser Veröffentlichung dient ausschließlich der Bereitstellung spezifischer Informationen und impliziert keine Empfehlung oder Billigung durch das US-Landwirtschaftsministerium. Die brasilianische Agentur FAPESP (Grant 2014/50981-0) unterstützte diese Arbeit finanziell; LGVF wird mit einem Stipendium der FAPESP (2017/06731-8 und 2019/20302-8) gefördert. Die Geldgeber spielten keine Rolle bei der Studiengestaltung, Datenerhebung und -analyse, der Entscheidung zur Veröffentlichung oder der Manuskriptvorbereitung. Die Autoren danken auch Hannah Hill und Alexander Grimes von den USDA Visual Services für das Filmen und Bearbeiten des Videoprotokolls.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.1 µm pore size mixed cellulose esters membrane | Millipore | VCWP02500 | Filtration for bacterial conjugation |
| 2,6-Diaminopimelic acid (DAP) | Sigma | D1377 | Growth of auxotrophic E. coli β2163 |
| Agar Noble | BD & Company | 214230 | Used for preparation of solid EMJH and HAN plates |
| Bacto Agar | BD & Company | 214010 | Used for preparation of solid LB plates |
| Clarity Western ECL substrate | Biorad | 170-5060 | Chemiluminescent substrate |
| dNTP set | Thermo Fisher | 10297-018 | dNTPs for PCR reaction |
| Glass Microanalysis Filter Holder | Millipore | XX1012530 | Filtration for bacterial conjugation |
| Imaging System | Biorad | ChemiDoc MP | Chemiluminescence detection |
| LB broth, Miller | BD & Company | 244620 | Lysogenic liquid medium for E. coli culturing |
| Leptospira Enrichment EMJH | BD & Company | 279510 | Supplementation of EMJH media |
| Leptospira Medium Base EMJH | BD & Company | 279410 | EMJH medium for Leptospira |
| Mini-PROTEAN TGX Gels 12% | Biorad | 4568043 | Used for polyacrylamide gel eletrophoresis |
| Optical density reader | Molecular Devices | SpectraMax M2 | For optical density measurements of bacterial cultures |
| Phosphate Buffered Saline 7.4 | Sigma | 806552 | Saline solution for washing bacterial pellets |
| Spectinomycin | Sigma | S0692 | Selection of pMaOri backbone plasmids |
| Taq DNA Polymerase | Thermo Fisher | EP0402 | Enyme, buffer and MgCl2 for PCR reaction |
| Thermocycler | Applied Biosystem | GeneAmp PCR System 9700 | Used for PCR reaction cycling |
| Thymidine (dT) | Sigma | T9250 | Growth of auxotrophic E. coli π1 |
| XmaI restriction enzyme | New Englan BioLabs | R0180L | Digestion of plasmids and inserts |
Referenzen
- Bharti, A. R., et al. Leptospirosis: A zoonotic disease of global importance. Lancet Infectious Disease. 3 (12), 757-771 (2003).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLoS Neglected Tropical Disease. 9 (9), 0003898(2015).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Fernandes, L. G. V., et al. Gene silencing based on RNA-guided catalytically inactive Cas9 (dCas9): a new tool for genetic engineering in Leptospira. Science Reports. 9 (1), 1839(2019).
- Fernandes, L. G. V., Hornsby, R. L., Nascimento, A. L. T. O., Nally, J. E. Genetic manipulation of pathogenic Leptospira: CRISPR interference (CRISPRi)-mediated gene silencing and rapid mutant recovery at 37 C. Science Reports. 11 (1), 1768(2021).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Choudhary, E., Thakur, P., Pareek, M., Agarwal, N. Gene silencing by CRISPR interference in mycobacteria. Nature Communication. 6, 6267(2015).
- Zhukova, A., et al. Genome-wide transcriptional start site mapping and sRNA identification in the pathogen. Frontiers in Cell and Infectious Microbiology. 7, 10(2017).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPalpha) conjugative machineries, and their cognate Escherichia coli host strains. Research in Microbiology. 156 (2), 245-255 (2005).
- Pappas, C. J., Benaroudj, N., Picardeau, M. A replicative plasmid vector allows efficient complementation of pathogenic Leptospira strains. Applied Environmental Microbiology. 81 (9), 3176-3181 (2015).
- Fernandes, L. G. V., Nascimento, A. L. T. O. Specific gene silencing in Leptospira biflexa by RNA-guided catalytically inactive Cas9 (dCas9). Methods in Molecular Biology. 2134, 109-122 (2020).
- Hornsby, R. L., Alt, D. P., Nally, J. E. Isolation and propagation of leptospires at 37 °C directly from the mammalian host. Science Reports. 10 (1), 9620(2020).
- Haake, D. A. Hamster model of leptospirosis. Current Protocols in Microbiology. , Chapter 12, Unit 12E.12 (2006).
- Nascimento, A. L., et al. Comparative genomics of two Leptospira interrogans serovars reveals novel insights into physiology and pathogenesis. Journal of Bacteriology. 186 (7), 2164-2172 (2004).
- Nascimento, A. L., et al. Genome features of Leptospira interrogans serovar Copenhageni. Brazillian Journal of Medical Biology Research. 37 (4), 459-477 (2004).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Bulach, D. M., et al. Genome reduction in Leptospira borgpetersenii reflects limited transmission potential. Proceedings of the National Academy of Science U. S. A. 103 (39), 14560-14565 (2006).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLoS One. 3 (2), 1607(2008).
- Fernandes, L. G., et al. OmpL1 is an extracellular matrix- and plasminogen-interacting protein of Leptospira spp. Infections and Immunity. 80 (10), 3679-3692 (2012).
- Fernandes, L. G., et al. Leptospira spp.: Novel insights into host-pathogen interactions. Veterinary Immunology and Immunopathology. 176, 50-57 (2016).
- Castiblanco-Valencia, M. M., et al. Leptospiral immunoglobulin-like proteins interact with human complement regulators factor H, FHL-1, FHR-1, and C4BP. Journal of Infectious Diseases. 205 (6), 995-1004 (2012).
- Choy, H. A., et al. The multifunctional LigB adhesin binds homeostatic proteins with potential roles in cutaneous infection by pathogenic Leptospira interrogans. PLoS One. 6 (2), 16879(2011).
- Siqueira, G. H., et al. The recombinant LIC10508 is a plasma fibronectin, plasminogen, fibrinogen and C4BP-binding protein of Leptospira interrogans. Pathogen and Diseases. 74 (2), (2016).
- Teixeira, A. F., et al. Features of two new proteins with OmpA-like domains identified in the genome sequences of Leptospira interrogans. PLoS One. 10 (4), 0122762(2015).
- Kochi, L. T., et al. The interaction of two novel putative proteins of Leptospira interrogans with E-cadherin, plasminogen and complement components with potential role in bacterial infection. Virulence. 10 (1), 734-753 (2019).
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infections and Immunity. 77 (2), 810-816 (2009).
- Bourhy, P., Louvel, H., Saint Girons, I., Picardeau, M. Random insertional mutagenesis of Leptospira interrogans, the agent of leptospirosis, using a mariner transposon. Journal of Bacteriology. 187 (9), 3255-3258 (2005).
- Pětrošová, H., Picardeau, M. Screening of a Leptospira biflexa mutant library to identify genes involved in ethidium bromide tolerance. Applied Environmental Microbiology. 80 (19), 6091-6103 (2014).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods in Molecular Biology. 859, 169-176 (2012).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: Disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infections and Immunity. 76 (12), 5826-5833 (2008).
- Picardeau, M., Brenot, A., Saint Girons, I. First evidence for gene replacement in Leptospira spp. Inactivation of L. biflexa flaB results in non-motile mutants deficient in endoflagella. Molecular Microbiology. 40 (1), 189-199 (2001).
- King, A. M., et al. High-temperature protein G is an essential virulence factor of Leptospira interrogans. Infections and Immunity. 82 (3), 1123-1131 (2014).
- Lambert, A., et al. FlaA proteins in Leptospira interrogans are essential for motility and virulence but are not required for formation of the flagellum sheath. Infections and Immunity. 80 (6), 2019-2025 (2012).
- Murray, G. L., et al. Leptospira interrogans requires heme oxygenase for disease pathogenesis. Microbes and Infections. 11 (2), 311-314 (2009).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathogens. 3 (7), 97(2007).
- Sasaki, Y., et al. Leptospiral flagellar sheath protein FcpA interacts with FlaA2 and FlaB1 in Leptospira biflexa. PLoS One. 13 (4), 0194923(2018).
- Gaj, T., Gersbach, C. A., Barbas, C. F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Cress, B. F., et al. Rapid generation of CRISPR/dCas9-regulated, orthogonally repressible hybrid T7-lac promoters for modular, tuneable control of metabolic pathway fluxes in Escherichia coli. Nucleic Acids Research. 44 (9), 4472-4485 (2016).
- Zhao, C., Shu, X., Sun, B. Construction of a gene knockdown system based on catalytically inactive ("dead") Cas9 (dCas9) in Staphylococcus aureus. Applied Environmental Microbiology. 83 (12), (2017).
- Zhao, Y., et al. CRISPR/dCas9-mediated multiplex gene repression in Streptomyces. Biotechnology Journal. , 1800121(2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten