Method Article
Aplicação de CRISPR Interference (CRISPRi) para Silenciamento genético em espécies patogênicas de Leptospira
Neste Artigo
Resumo
Aqui, é descrita a aplicação da interferência CRISPR (CRISPRi) para silenciamento genético específico em espécies de Leptospira. As células leptospira são transformadas pela conjugação com plasmídeos expressando dCas9 (catalyticamente "morto" Cas9) e um RNA de guia único (sgRNA), responsável pelo emparelhamento base ao alvo genômico desejado. São apresentados métodos para validar o silenciamento genético.
Resumo
A leptospirose é uma zoonose negligenciada global, responsável por pelo menos 1 milhão de casos por ano e quase 60 mil mortes. A doença é causada por bactérias patogênicas e virulentas do gênero Leptospira,seja pelo contato direto com a bactéria ou indiretamente pela exposição à água ou ao solo contaminados. Animais domésticos e silvestres atuam como hospedeiros de infecção, derramando leptospirais de túbulos renais colonizados do rim, via urina, para o meio ambiente. A geração de cepas mutantes de Leptospira é fundamental para avaliar e entender mecanismos patogênicos de infecção. A interferência CRISPR (CRISPRi) provou ser uma ferramenta simples, acessível e específica para o silenciamento genético em Leptospirapatogênico . Portanto, serão descritos os detalhes metodológicos da obtenção dos construtos plasmídeos contendo dCas9 e guia do RNA, a entrega de plasmídeos ao Leptospira por conjugação com a cepa E. coli β2163, e recuperação e avaliação transconjugut. Além disso, a mídia recentemente descrita de Hornsby-Alt-Nally (HAN) permite o isolamento relativamente rápido e a seleção de colônias mutantes em placas de ágar.
Introdução
Leptospirose é uma zoonose mundial negligenciada causada por espécies patogênicas e virulentas do gênero Leptospira. Em humanos, a doença é responsável por mais de um milhão de casos e 60.000 mortes por ano em todo o mundo1,2. Até agora, não há vacina de longo prazo e eficaz para a doença. A identificação de fatores de virulência e mecanismos patogênicos é fundamental para o desenvolvimento de melhores estratégias terapêuticas e profiláticas. Portanto, a capacidade de gerar mutações genéticas e avaliar o fenótipo resultante é fundamental para a análise genômica funcional3.
A construção de mutantes em Leptospira patogênico foi considerada, até agora, inerentemente ineficiente, laboriosa, cara e difícil de implementar. Este cenário mudou drasticamente com a aplicação da recente interferência CRISPR (CRISPRi) para leptospires saprofíticos4 epatogênicos 5.
O silenciamento genético é alcançado pela expressão de dois componentes: uma variante da enzima CRISPR/Cas (clustered regularly interspaced short palindromic repeat/CRISPR comoenciaado) Cas9 de Streptococcus pyogenes, chamado cas9 (dCas9) e um RNA de guia único (sgRNA), que pode ser editado de acordo com o alvo desejado6,7,8. a proteína dCas9, quando vinculada ao sgRNA, é direcionada a alvos específicos de DNA por Watson e Crick, causando um bloqueio estérico ao alongamento da polimerase RNA, resultando em silenciamento genético devido à transcrição genética obstruída7 (Figura 1).
Este manuscrito tem como objetivo descrever a construção do plasmídeo para expressar tanto dCas9 quanto sgRNA, conjugação entre o doador E. coli β2163 e células leptospiras receptoras, recuperação transconjugulante e, finalmente, validação de colônias mutantes selecionadas.
Protocolo
1. Definição protoespacial e construção plasmida
NOTA: Nesta seção, é descrita a primeira etapa de seleção de protoespaços apropriados para a construção do sgRNA e maior ligadura em pMaOri.dCas9 (Figura 1). Esta sequência protoespaço é composta por uma sequência de 20 nucleotídeos contra o alvo desejado.
- Obtenha a sequência nucleotídea do gene de interesse para silenciamento no GenBank (https://www.ncbi.nlm.nih.gov/genbank). Envie-o ao servidor web CHOPCHOP (http://chopchop.cbu.uib.no/), com parâmetros definidos para Streptococcus pyogenes Cas9 e protoespaçor motivo adjacente NGG depois de selecionar o "Alvo Fasta". Defina os parâmetros para "CRISPR/Cas9" e PAM (motivo adjacente protoespaço) NGG.
- Com base nos resultados obtidos, selecione os protoespaciares com a melhor pontuação possível (arqueiro verde), que estão localizados o mais próximo possível da região de codificação de 5'end e, o mais importante, estão contidos no modelo (menos) fio, uma vez que o sgRNA deve emparelhar-se ao fio de codificação do gene para silenciar genes completos.
NOTA: O motivo NGG não está incluído na sequência sgRNA final. - Use o promotor lipL32 para expressar o RNA guia único que contém uma sequência variável de 20 nucleotídeos no final de 5' e uma sequência de andaimes dCas9 conservados. Mescle a sequência de 20 nt, denominada protoespaço, ao promotor lipL32 (no final de 5' ) e ao andaime sgRNA (3'end)(Figura 1B).
NOTA: Para um promotor lipL32 bem definido, utilize a região promotora composta por -334 para o TSS (Transcription Start Site, com base em Zhukova et al.9). Verifique o Arquivo Suplementar para o sgRNA final. - Gere o sgRNA por PCR5 sequencial ou tenha-o sintetizado por um provedor comercial.
- Após a obtenção do, liga-o em pMaOri.dCas9 plasmid no local de restrição de XmaI em ambas as extremidades (cccggg)4.
- Digerir tanto o sgRNA quanto o plasmídeo pMaOri.dCas9 com enzima de restrição de XmaI e proceder à ligadura(Figura 1B).
- Realize as etapas de clonagem na cepa dT auxotrophic E. coli π110, devido à origem pMaOri11 (e por extensão, pMaOri.dCas9) de replicação, R6K-gama.
NOTA: Para um protocolo detalhado para seleção de ligaduras e clones, consulte publicações anteriores de Fernandes e Nascimento12. dCas9 guiado por sgRNA se ligará à cadeia de codificação do gene de interesse selecionado e, portanto, obstruirá o alongamento da polimerase de RNA(Figura 1C),resultando em silenciamento genético.
2. Transformação leptospira por conjugação
NOTA: Um esquema gráfico desta etapa é apresentado na Figura 2. Para fazer mídia HAN e placas HAN, consulte Hornsby et al.13 e Fernandes et al.5.
- Cresça células patogênicas de Leptospira a 29 ou 37 °C na mídia HAN13 sob agitação, diluindo uma cultura saturada em HAN fresco às 1:100; tipicamente, L. interroga serovar Copenhageni tensão Fiocruz L1-130 leva de 4-6 dias para atingir a densidade celular apropriada.
- Certifique-se de que as culturas atinjam uma O.D. de 0,2-0,4 a 420 nm (2 a 5 x 108 células/mL) antes de usar para conjugação.
NOTA: Uma vez que a mídia HAN muda de cor à medida que as densidades celulares aumentam (devido ao vermelho fenol contido na mídia DMEM), centrífuga (4.000 x g, 15 min, temperatura ambiente) 1 mL da mídia cultural para remover leptospires e aplicar supernatante como um branco para medir a O.D.
- Certifique-se de que as culturas atinjam uma O.D. de 0,2-0,4 a 420 nm (2 a 5 x 108 células/mL) antes de usar para conjugação.
- Transforme a cepa conjugal E. coli β2163, auxotrophic para ácido diaminopimelico (DAP), com o plasmídeo pMaOri.dCas9 contendo o sgRNA. Para E. transformação coli, use protocolos de choque térmico ou eletroporação. Inclua a transformação com o pMaOri.dCas9 plasmídeo sem sgRNA como controle.
- Para transformação de choque térmico, misture o DNA plasmídeo (100 ng) com células E. coli quimicamente competentes e incubar no gelo por 30 minutos. Realize o choque térmico a 42 °C por 90 s e coloque-o novamente no gelo por 5 minutos. Recupere as células adicionando 1 mL de mídia LB, incubar a 37 °C por 1 h e seguir para o revestimento.
- Para eletroporação, use células eletrocompetntes misturadas com 100 ng de DNA plasmídeo. Use os seguintes parâmetros para pulso: 1,8 kV, 100 Ω e 25 μF. Recupere as células conforme explicado acima.
- Placa as células E. coli do doador transformado em meio ágar LB suplementadas com ácido diaminopimelico (DAP) (0,3 mM) e espectincina (40 μg/mL) para selecionar para plasmídeos.
- Para a conjugação, selecione uma colônia de cada prato um dia antes do dia da conjugação (que é determinada pelo monitoramento da O.D. das culturas de leptospires).
- Selecione uma colônia de E. coli β2163 do pMaOri.dCas9 vazio, e uma das placas pMaOri.dCas9sgRNA. Permita-lhes crescer durante a noite em 10 mL de LB mais DAP e espectineca a 37 °C.
- No dia seguinte, diluir as culturas saturadas 1:100 em 10 mL de LB fresco mais DAP (não inclua o antibiótico aqui) até OD420nm de 0,2-0,4. Normalmente, leva 2-3 h para E. coli atingir essas densidades.
- Dentro de uma capa biossegurança BSL2, monte um aparelho de filtragem colocando um diâmetro de 25 mm, tamanho de poros de 0,1 μm, filtro de membrana de ésteres de celulose mista na parte superior da base de vidro. Coloque um funil de vidro de 15 mL na parte superior e segure ambas as peças com grampos de mola. Conecte o vidro a uma bomba de vácuo e adicione as culturas ao funil para filtração.
- Adicione 5 mL de cultura Leptospira ao funil. Adicione um volume de E. coli para constituir a proporção de 1:1 com base nos valores de420nm de OD de ambas as culturas. Ligue a bomba de vácuo e concentre as células por filtragem. Após a concentração celular no filtro de membrana, recupere-o cuidadosamente. Certifique-se de que o meio seja filtrado através da membrana.
NOTA: A filtragem leva de 5 a 10 minutos. - Coloque o filtro em uma placa EMJH disponível comercialmente (ver Tabela de Materiais) complementada com DAP (0,3 mM). Certifique-se de que o lado das bactérias está para cima. Incubar as placas a 29 °C por 24 h.
NOTA: Se forem utilizadas placas EMJH14 em casa ou suplementadas internamente, a E. coli pode proliferar e superar a proporção pretendida de 1:1, que por sua vez pode diminuir a eficiência conjugal5. - Após 24 horas, recupere os filtros das placas e coloque cada filtro individual em um tubo cônico de 50 mL.
- Use 1 mL de médio HAN líquido para liberar as células da superfície do filtro por tubulação extensiva e vórtice.
- Visualize as soluções bacterianas mistas recuperadas por microscopia de campo escuro para verificar a viabilidade e a motilidade celular, e as proporções de Leptospira:E. coli.
NOTA: Nesta fase, podem ser vistos números equivalentes de E. coli e Leptospira. - Espalhe 100-200 μL desta cultura em placas HAN contendo 0,4% de soro de coelho inativado e 40 μg/mL especttinomicina. Incubar placas a 37 °C em uma atmosfera de 3% de CO2.
NOTA: Normalmente, L. interroga serovar Copenhageni cepa Fiocruz L1-130 células formam colônias em 5-7 dias em placas de controle e em 8-10 dias em placas de especttinmicina. Nesta fase, e. coli não crescerá, pois são auxotróficos para DAP. - Como controle, diluir culturas a10 4 leptospires/mL e adicionar 100 μL em placas sem antibiótico, para monitorar o crescimento leptospiral.
3. Seleção de colônias e crescimento e validação transconjugult
NOTA: As colônias devem ser aparentes até o dia 10. No entanto, eles não são muito fáceis de visualizar. Normalmente neste momento, as placas HAN são um pouco opacas por causa das células secas que foram espalhadas e as colônias de Leptospira podem aparecer como uma auréola transparente contra o fundo esbranquiçado. Recomenda-se visualizar as placas em diferentes ângulos para alcançar diferentes incidências de luz, tornando as colônias mais aparentes. Em tempos de incubação mais longos, as colônias podem adquirir uma aparência mais densa, e neste caso, apresentam-se como halos leitosos contra um fundo escuro.
- Adicione 100 μL de mídia HAN líquida a cada microtubo de 1,5 mL para recuperar mutantes. Pegue pelo menos 3 colônias de cada prato.
- Com o auxílio de uma ponta de micropipette, "cavar" o ágar para recuperar as colônias das placas, uma vez que colônias leptospirais podem ser subsuperfícies.
NOTA: Espera-se que a Agar seja levada nesta fase. As colônias devem ser retiradas de placas de controle contendo plasmídeos pMaOri.dCas9 vazios, e placas com leptospires contendo plasmídeos expressando dCas9 e RNA guia único, projetado para o gene alvo. - Dispense a colônia coletada em 100 μL de mídia HAN em um microtubo de 1,5 mL e homogeneize vigorosamente. Nesta fase, garanta a quebra máxima da integridade do ágar para liberar células. Vórtice a suspensão por 10 s.
- Com o auxílio de uma ponta de micropipette, "cavar" o ágar para recuperar as colônias das placas, uma vez que colônias leptospirais podem ser subsuperfícies.
- Visualize as células recuperadas por microscopia de campo escuro em uma ampliação de 200-400x adicionando uma gota de 5 μL em um slide de vidro e cubra as amostras imediatamente com uma mancha de cobertura.
- Confirme a presença de leptospires vivos e viáveis recuperados das colônias.
- Após a visualização e confirmação de leptospires viáveis, transfira 100 μL de células para mídia han líquida contendo 40 μg/mL especttinomicina.
- Após o crescimento da mídia han líquida, avalie as culturas para a presença do plasmídeo com primer pMaOri2 F (ACGCAATGTATCGATACCGAC) e R (ATAGGTGAAGTAGGCCCACCC), que reconhecem a região que flanqueia o sgRNA.
- Colete 200 μL de cultura, centrífuga (4.000 x g, 15 min), descarte o supernasce e resuspenque a pelota resultante em 20 μL de água.
- Use esta suspensão como modelo para PCR adicional, sem a necessidade de extração de DNA12.
NOTA: As células com pMaOri.dCas9 renderão um amplicon de 281 bp, em comparação com aqueles que contêm os plasmídeos com o sgRNA que renderá um amplicon de 723 bp.
- Para confirmação do silenciamento genético, realize um imunoblot utilizando extratos celulares de transconjugantes contendo apenas pMaOri.dCas9 (controle negativo) e pMaOri.dCas9sgRNA.
- Aplique o equivalente a 5 x 107 células por pista do gel de poliacrilamida do dodecyl de sódio (SDS).
- Proteínas de eletrotransferência para uma membrana para incubação com anticorpos apropriados. Além do anticorpo contra o gene alvo para silenciar, use outro para carregar o controle.
- Mantenha as culturas mutantes na mídia HAN mais a espectincina para manter o plasmídeo. Se nenhum antibiótico for aplicado na mídia, o silenciamento genético completo pode ser observado por pelo menos três passagens5.
Resultados
Mesmo que o conteúdo CG em genomas leptospira spp. é tipicamente em torno de 35%; praticamente todos os genes são susceptivelmente conter o PAM 5'NGG 3'; este motivo precisa ser considerado na cadeia de modelo. Depois de inserir a sequência de codificação de um gene (do início ao fim dos códons), com base nos resultados do CHOPCHOP, os protoespaciares devem ser selecionados na cadeia menos (-, modelo). É importante não incluir o motivo NGG no protoespaço sgRNA de 20 nts.
Se a conjugação for realizada com uma proporção celular de 1:1:receptor, por 24 h na superfície das placas de ágar EMJH mais DAP, e 200 μL da suspensão bacteriana recuperada são espalhadas em placas de ágar de espectinomicina, as colônias de transconjugantes devem ser visíveis em aproximadamente 8-10 dias. A disseminação desse volume normalmente resulta em 20-40 colônias por placa (Figura 3A). A fim de verificar a viabilidade celular após a conjugação, as células podem ser espalhadas em placas HAN sem seleção de antibióticos. Neste caso, as colônias podem ser observadas logo em 7 dias. As placas HAN ficam amarelas pálidas em uma atmosfera de 3% de CO2.
Após a colheita e crescimento da colônia em mídia líquida mais espectina, o PCR utilizando células inteiras e primers pMaOri2 pode ser usado para uma verificação inicial de qualidade dos transconjugantes(Figura 3B). As células leptospirais contendo o plasmídeo pMaOri.dCas9 de controle devem resultar em um amplicon de 281 bp, enquanto as células que contêm o plasmídeo para silenciamento, ou seja, contendo dCas9 e sgRNA, devem resultar em um amplicon de 723 bp. os primers pMaOri2 F e R foram projetados para flanquear o local de restrição do XmaI, que é o local usado durante a ligadura do sgRNA.
Com a confirmação da presença plasmida, as células podem ser colhidas da mídia, lavadas duas vezes com PBS, e depois usadas para preparar um extrato de células inteiras para imunoblotação. Se o silenciamento ocorreu, as proteínas-alvo, neste caso, tanto lipl32 ou LigA e LigB, devem ser observadas apenas nas células do tipo selvagem e nas que contêm pMaOri.dCas9; mesmo com maiores tempos de exposição, nenhuma proteína correspondente deve ser visível nas células que contêm pMaOri.dCas9sgRNA (Figura 3C).
Se forem planejados experimentos para avaliar a virulência leptospiral após o silenciamento genético, as culturas utilizadas para a conjugação devem ser de baixa passagem virulenta Leptospira. Depois que o silenciamento genético é confirmado, várias alíquotas podem ser congeladas como backup. Se o gene silenciado tiver um fenótipo mensurável, por exemplo, com base em trabalhos anteriores com proteínas recombinantes, culturas podem ser usadas para validação e, neste caso, células que contêm apenas pMaOri.dCas9, podem ser incluídas como um controle negativo.
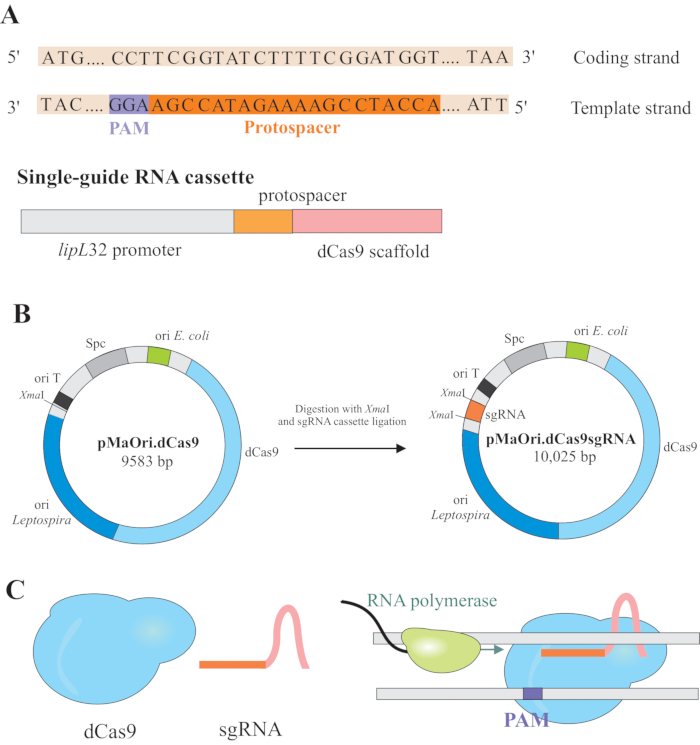
Figura 1: Desenvolvimento de dCas9 e sgRNA expressando plasmid. (A) Um protoespaço de 20 nt de comprimento, seguido pelo S. pyogenes dCas9 PAM 5'-NGG-3', é selecionado dentro do fio de modelo do gene alvo para que o sgRNA subsequente possa realizar o emparelhamento de watson e crick base ao correspondente codificação de fios de codificação, resultando em completo silenciamento genético. (B) O sgRNA é composto pelo promotor lipL32, protoespaço de 20 nt e andaime dCas9. O plasmídeo pMaOri.dCas9 é usado como espinha dorsal para ligadura de sgRNA no local de restrição do XmaI. O plasmídeo resultante, chamado pMaOri.dCas9sgRNA é entregue a leptospires, e a expressão de dCas9 e sgRNA é responsável pelo silenciamento genético. (C) dCas9 dirigido por SGRNA atua como uma barreira física ao alongamento da polimerase de RNA, dificultando, portanto, a transcrição. Clique aqui para ver uma versão maior desta figura.
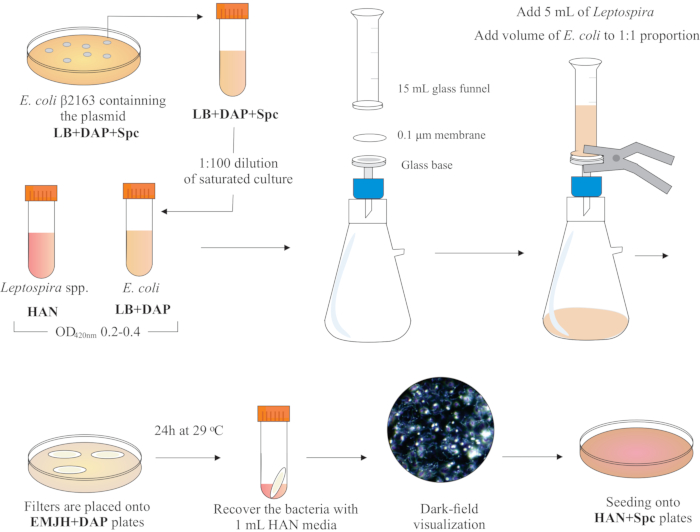
Figura 2: Representação esquemática do protocolo de conjugação. A desejada espécie leptospira é cultivada na mídia HAN, sob agitação, até o O.D. de 0,2-0.4 (fase de registro médio) a 420 nm. Um dia antes da conjugação, uma colônia de doadores recombinantes E. coli β2163 contendo o plasmídeo de interesse é colhida a partir de placas de ágar LB+DAP+Spc, uma vez que as células são cultivadas durante a noite em LB líquido com a mesma suplementação. No dia seguinte, saturado E. as culturas coli são diluídas em LB mais DAP e cultivadas até O.D. de 0,2-0,4 a 420 nm. Tanto o doador E. coli quanto o receptor Leptospira são misturados a proporção celular de 1:1 na superfície de um filtro de 0,1 μm por um aparelho de filtragem sob pressão negativa. Em seguida, os filtros são colocados em cima das placas de ágar EMJH complementadas com DAP, e a incubação prossegue por 24 h a 29 °C. O uso do EMJH limita a proliferação de E. coli, e a proporção pretendida de 1:1 é mantida. As bactérias são recuperadas de filtros por pipetação com mídia HAN de 1 mL, e as suspensões são visualizadas sob microscopia de campo escuro. Finalmente, 100-200 μL de cada suspensão são semeadas em placas de ágar HAN contendo 0,4% de soro de coelho e incubadas a 37 °C em 3% CO2. Nesta fase, o DAP é omitido e, como resultado, o Auxotrophic E. coli não crescerá. Clique aqui para ver uma versão maior desta figura.
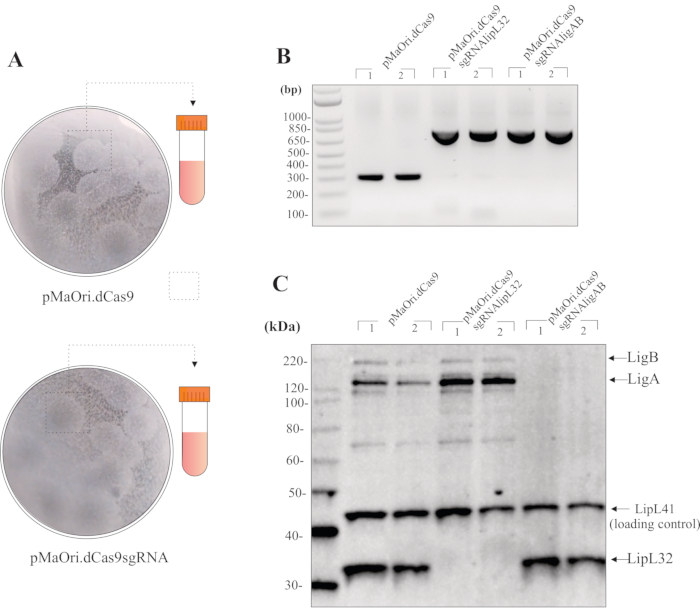
Figura 3: Resultados representativos para avaliação dos mutantes. (A) Colônias de placas contendo Leptospira transformadas com pMaOri.dCas9 vazios (controle negativo para experimentos adicionais) e plasmídeos pMaOri.dCas9sgRNA (com gene silenciado) são colhidos, vigorosamente homogeneizados em HAN líquido e cultivados em HAN líquido contendo espectincina. As células recombinantes podem ser validadas pelo PCR com primers flanqueando o site do XmaI dentro de pMaOri.dCas9. (B) Neste caso, as células contendo pMaOri.dCas9 resultaram apenas em um amplicon de 281 bp, enquanto as células contendo o plasmídeo para silenciamento, contendo dCas9 e sgRNA, apresentaram um amplicon de 723 bp. Após a confirmação da presença dos plasmídeos, o silenciamento genético foi validado pela análise do imunoblot. (C) Recomenda-se a incubação com anticorpos para proteína alvo e uma proteína de controle de carga; no imunobloto representativo, extratos de células inteiras de transconjugantes contendo pMaOri.dCas9 sozinhos ou com sgRNA direcionados aos genes lipL32 (pMaOri.dCas9sgRNAlipL32) e ambos os genes LigA e LigB (pMaOri.dCas9sgRNAligAB). A co-incubação com anti-LipL32, anti-LigAB e anti-LipL41 (controle de carregamento não-alvo) confirma que a expressão da proteína LipL32 é abolida em células que contêm pMaOri.dCas9sgRNAlipL32 e liga e LigB em células contendo pMaOri.dCas9sgRNAABlig. Clique aqui para ver uma versão maior desta figura.
Arquivo suplementar: Sequência de RNA (sgRNA) de guia único. A transcrição do sgRNA é dirigida pelo promotor constitutivo lipL32 (nucleotídeos ousados). o sgRNA é composto por 20 nucleotídeos referentes ao protoespaço, responsável pelo emparelhamento base ao fio de codificação do gene alvo, e sequência de andaimes dCas9 (nucleotídeos sublinhados). Natal I sites de restrição (cccggg) estão incluídos em ambas as extremidades para ligadura em pMaOri.dCas9 plasmid. Clique aqui para baixar este Arquivo.
Discussão
Após o sequenciamento precoce depatogênicos 15,16,17,18 e19espécies de Leptospira, a mineração de dados do genoma lançou luz sobre vários aspectos da patogênese leptospiral. Na maioria dos casos, a função proteica foi explorada utilizando-se a contrapartida recombinante de proteínas leptospirais putativas expostas à superfície e posterior especulação da função proteica nativa20,21,22,23,24,25,26.
A geração de mutantes, e a avaliação de seus respectivos fenótipos, são componentes-chave da análise genômica funcional. As tentativas iniciais de gerar mutantes em Leptospira spp. foram alcançadas por mutagênese transposon aleatória27,28,29,30; no entanto, após análises extensas e trabalhosas para inferir a identidade dos genes rompidos, observou-se que apenas 15% de todos os genes em L. interrogans serovar Manilae foram interrompidos27. O nocaute genético direcionado foi ainda mais alcançado pela recombinação homólogoa utilizando plasmídeos suicidas para entregar um de resistência a antibióticos ladeado por braços homólogos dentro do alvo desejado31,32.
Aplicando essas tecnologias, foram explorados diversos aspectos da biologia básica leptospiral e virulência31,33,34,35,36,37. O desenvolvimento do vetor de transporte conjugal E. coli-Leptospira, pMaOri 11, permitiu a entrega de componentes para silenciamento genético epissomal.
Foi demonstrado anteriormente que a quebra de dois fios induzidos por Cas9 é letal para Leptospira spp. e, como alternativa, a variante catalyticamente inativa da enzima, dCas9, pode ser usada para alcançar o silenciamento genético nas espécies saprofísticas e patogênicas4,5. Usando o plasmid pMaOri.dCas9 como espinha dorsal para ligante de sgRNA, o silenciamento genético específico e estável pode ser obtido devido à expressão de dCas9 e sgRNA; o sgRNA vinculado a dCas9 levará a proteína ao alvo desejado pelo emparelhamento de base Watson-Crick.
Para o silenciamento genético completo, o protoespaço deve ser projetado com base no fio de modelo do gene desejado para que o emparelhamento base do sgRNA ocorra com o fio de codificação. Com base em um teor médio de C+G de 35% em Leptospira spp., o PAM 5'-NGG-3' ocorrerá pelo menos 3 vezes a cada 100 bp. Portanto, praticamente qualquer gene dentro do genoma de Leptospira conterá pelo menos um PAM. No entanto, se o motivo NGG não for encontrado, o motivo alternativo do NAG pode ser avaliado.
Técnicas anteriores de silenciamento genético, como dedos de zinco e TALE (efeitos semelhantes ao ativador de transcrição), contavam com a construção de uma proteína diferente para cada alvo, tornando essas técnicas laboriosas e caras38. No caso do CRISPRi, o componente variável é o sgRNA, tornando necessário apenas alterar os 20 bp no final de 5'. O silenciamento genético completo, estável e direcionado tem sido observado não apenas em Leptospira spp.4,5, mas também em outras bactérias8,39,40,41.
O desenvolvimento da mídia HAN13 favoreceu a recuperação dos mutantes, reduzindo drasticamente o tempo de incubação para a formação de colônias e permitindo que Leptospira crescesse a 37 oC. No entanto, durante a etapa de conjugação, seu uso não é recomendado, uma vez que o E. coli pode proliferar vigorosamente nesta mídia e superar a proporção pretendida de 1:1 entre células doadoras e receptoras. Nesta fase, a EMJH plus DAP é a melhor escolha, já que e. coli se replica mal nesta mídia. Vale ressaltar que alguns laboratórios fazem emjh suplementado internamente, que pode conter componentes adicionais que também podem suportar o crescimento de células E. coli.
O protocolo de conjugação aqui apresentado foi otimizado para l. interrogans serovar Copenhageni cepa Fiocruz L1-130, e também foi comprovado ser eficaz na transformação de uma cepa patogênica recentemente isolada a partir de amostras de solo5. Tentativas iniciais com diferentes serovars de espécies L. borgpetersenii indicam menor eficiência conjugal com o protocolo descrito. Assim, ao trabalhar com diferentes espécies/serovares de Leptospira,as condições ideais para a conjugação devem ser determinadas empiricamente, considerando as proporções de células receptoras, densidades celulares iniciais, mídia de conjugação e tempo (24 e 48 h). É razoável supor que diferentes espécies de Leptospira e serovars se comportarão de forma diferente com diferentes protocolos de conjugação.
Embora as colônias de Leptospira saprofíticas sejam relativamente fáceis de visualizar em placas, colônias patogênicas podem ser mais difíceis de observar. Normalmente, usando mídia HAN suplementada com soro de coelho e espectina de coelho de 0,4% e espectinomicina, colônias transconjugut podem ser observadas no dia 10. Em nossa experiência, colônias inicialmente presentes como um halo transparente na superfície da mídia. No protocolo de vídeo, colônias mais densas, após 14 dias de crescimento, são mostradas já que as transparentes eram difíceis de filmar. Nesta fase, girar a placa para obter diferentes incidência de luz e deslocar entre fundos brancos e escuros pode ajudar a identificar colônias.
Para validação mutante, a imunoblotting oferece uma abordagem simples; no entanto, como os anticorpos nem sempre estão disponíveis contra proteínas-alvo, estratégias alternativas para validar o silenciamento genético podem ser perseguidas. PcR de transcriptase reversa quantitativa (qRT-PCR) usando primers para o gene alvo e um controle constitutivo é eficaz para validar o silenciamento genético, uma vez que o dCas9 guiado pelo SGRNA é responsável pelo bloqueio da transcrição genética. Se o gene alvo codificar uma faixa proteica claramente definida em géis proteicos, o SDS-PAGE pode demonstrar silenciamento, e de acordo com o gene lipL32 silenciando5. Se os genes da biossíntese LPS forem silenciados, a coloração de LPS pode ser empregada; no caso de silenciar genes codificadores para enzimas com substratos bem definidos, ensaios cinéticos com substratos cromogênicos são estratégias válidas; β-galactosidase silenciando em L. biflexa foi validado pelo uso de substratos X-gal e ONPG (ortho-Nitrophenyl-β-galactoside)substratos 4.
Após a confirmação do silenciamento genético, os experimentos podem ser projetados para avaliar ainda mais o fenótipo. Ensaios de vinculação podem ser realizados no caso de silenciamento de adesivos bacterianos; ensaios de desafio sérico confirmaram o papel de LigA e LigB na sobrevivência do soro exibida por Leptospirapatogênico 5. Mutantes também podem ser usados para inocular animais para avaliar atenuação da virulência; neste caso, os animais inoculados com o mutante devem ser comparados com aqueles infectados apenas com células contendo pMaOri.dCas9.
Em conclusão, o protocolo atual descreve a aplicação de CRISPRi para silenciamento genético em espécies patogênicas de Leptospira usando mídia HAN para facilitar a recuperação de mutantes dentro de 10 dias. O silenciamento genético combinado com a análise genômica funcional melhorará nossa compreensão dos mecanismos patogênicos do Leptospira,e, em última instância, levará ao desenvolvimento de melhores estratégias profiláticas para o controle de doenças.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
O USDA é um provedor de igualdade de oportunidades e empregador. A menção de nomes comerciais ou produtos comerciais nesta publicação é exclusivamente com o propósito de fornecer informações específicas, e não implica recomendação ou endosso do Departamento de Agricultura dos EUA. A agência brasileira FAPESP (outorga 2014/50981-0) apoiou financeiramente esse trabalho; A LGVF é financiada com uma bolsa da FAPESP (2017/06731-8 e 2019/20302-8). Os financiadores não tiveram papel na concepção do estudo, coleta e análise de dados, decisão de publicar ou elaboração de manuscritos. Os autores também agradecem Hannah Hill e Alexander Grimes do USDA Visual Services por filmar e editar o protocolo de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.1 µm pore size mixed cellulose esters membrane | Millipore | VCWP02500 | Filtration for bacterial conjugation |
| 2,6-Diaminopimelic acid (DAP) | Sigma | D1377 | Growth of auxotrophic E. coli β2163 |
| Agar Noble | BD & Company | 214230 | Used for preparation of solid EMJH and HAN plates |
| Bacto Agar | BD & Company | 214010 | Used for preparation of solid LB plates |
| Clarity Western ECL substrate | Biorad | 170-5060 | Chemiluminescent substrate |
| dNTP set | Thermo Fisher | 10297-018 | dNTPs for PCR reaction |
| Glass Microanalysis Filter Holder | Millipore | XX1012530 | Filtration for bacterial conjugation |
| Imaging System | Biorad | ChemiDoc MP | Chemiluminescence detection |
| LB broth, Miller | BD & Company | 244620 | Lysogenic liquid medium for E. coli culturing |
| Leptospira Enrichment EMJH | BD & Company | 279510 | Supplementation of EMJH media |
| Leptospira Medium Base EMJH | BD & Company | 279410 | EMJH medium for Leptospira |
| Mini-PROTEAN TGX Gels 12% | Biorad | 4568043 | Used for polyacrylamide gel eletrophoresis |
| Optical density reader | Molecular Devices | SpectraMax M2 | For optical density measurements of bacterial cultures |
| Phosphate Buffered Saline 7.4 | Sigma | 806552 | Saline solution for washing bacterial pellets |
| Spectinomycin | Sigma | S0692 | Selection of pMaOri backbone plasmids |
| Taq DNA Polymerase | Thermo Fisher | EP0402 | Enyme, buffer and MgCl2 for PCR reaction |
| Thermocycler | Applied Biosystem | GeneAmp PCR System 9700 | Used for PCR reaction cycling |
| Thymidine (dT) | Sigma | T9250 | Growth of auxotrophic E. coli π1 |
| XmaI restriction enzyme | New Englan BioLabs | R0180L | Digestion of plasmids and inserts |
Referências
- Bharti, A. R., et al. Leptospirosis: A zoonotic disease of global importance. Lancet Infectious Disease. 3 (12), 757-771 (2003).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLoS Neglected Tropical Disease. 9 (9), 0003898(2015).
- Shapiro, R. S., et al. A CRISPR-Cas9-based gene drive platform for genetic interaction analysis in Candida albicans. Nature Microbiology. 3 (1), 73-82 (2018).
- Fernandes, L. G. V., et al. Gene silencing based on RNA-guided catalytically inactive Cas9 (dCas9): a new tool for genetic engineering in Leptospira. Science Reports. 9 (1), 1839(2019).
- Fernandes, L. G. V., Hornsby, R. L., Nascimento, A. L. T. O., Nally, J. E. Genetic manipulation of pathogenic Leptospira: CRISPR interference (CRISPRi)-mediated gene silencing and rapid mutant recovery at 37 C. Science Reports. 11 (1), 1768(2021).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Choudhary, E., Thakur, P., Pareek, M., Agarwal, N. Gene silencing by CRISPR interference in mycobacteria. Nature Communication. 6, 6267(2015).
- Zhukova, A., et al. Genome-wide transcriptional start site mapping and sRNA identification in the pathogen. Frontiers in Cell and Infectious Microbiology. 7, 10(2017).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPalpha) conjugative machineries, and their cognate Escherichia coli host strains. Research in Microbiology. 156 (2), 245-255 (2005).
- Pappas, C. J., Benaroudj, N., Picardeau, M. A replicative plasmid vector allows efficient complementation of pathogenic Leptospira strains. Applied Environmental Microbiology. 81 (9), 3176-3181 (2015).
- Fernandes, L. G. V., Nascimento, A. L. T. O. Specific gene silencing in Leptospira biflexa by RNA-guided catalytically inactive Cas9 (dCas9). Methods in Molecular Biology. 2134, 109-122 (2020).
- Hornsby, R. L., Alt, D. P., Nally, J. E. Isolation and propagation of leptospires at 37 °C directly from the mammalian host. Science Reports. 10 (1), 9620(2020).
- Haake, D. A. Hamster model of leptospirosis. Current Protocols in Microbiology. , Chapter 12, Unit 12E.12 (2006).
- Nascimento, A. L., et al. Comparative genomics of two Leptospira interrogans serovars reveals novel insights into physiology and pathogenesis. Journal of Bacteriology. 186 (7), 2164-2172 (2004).
- Nascimento, A. L., et al. Genome features of Leptospira interrogans serovar Copenhageni. Brazillian Journal of Medical Biology Research. 37 (4), 459-477 (2004).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Bulach, D. M., et al. Genome reduction in Leptospira borgpetersenii reflects limited transmission potential. Proceedings of the National Academy of Science U. S. A. 103 (39), 14560-14565 (2006).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLoS One. 3 (2), 1607(2008).
- Fernandes, L. G., et al. OmpL1 is an extracellular matrix- and plasminogen-interacting protein of Leptospira spp. Infections and Immunity. 80 (10), 3679-3692 (2012).
- Fernandes, L. G., et al. Leptospira spp.: Novel insights into host-pathogen interactions. Veterinary Immunology and Immunopathology. 176, 50-57 (2016).
- Castiblanco-Valencia, M. M., et al. Leptospiral immunoglobulin-like proteins interact with human complement regulators factor H, FHL-1, FHR-1, and C4BP. Journal of Infectious Diseases. 205 (6), 995-1004 (2012).
- Choy, H. A., et al. The multifunctional LigB adhesin binds homeostatic proteins with potential roles in cutaneous infection by pathogenic Leptospira interrogans. PLoS One. 6 (2), 16879(2011).
- Siqueira, G. H., et al. The recombinant LIC10508 is a plasma fibronectin, plasminogen, fibrinogen and C4BP-binding protein of Leptospira interrogans. Pathogen and Diseases. 74 (2), (2016).
- Teixeira, A. F., et al. Features of two new proteins with OmpA-like domains identified in the genome sequences of Leptospira interrogans. PLoS One. 10 (4), 0122762(2015).
- Kochi, L. T., et al. The interaction of two novel putative proteins of Leptospira interrogans with E-cadherin, plasminogen and complement components with potential role in bacterial infection. Virulence. 10 (1), 734-753 (2019).
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infections and Immunity. 77 (2), 810-816 (2009).
- Bourhy, P., Louvel, H., Saint Girons, I., Picardeau, M. Random insertional mutagenesis of Leptospira interrogans, the agent of leptospirosis, using a mariner transposon. Journal of Bacteriology. 187 (9), 3255-3258 (2005).
- Pětrošová, H., Picardeau, M. Screening of a Leptospira biflexa mutant library to identify genes involved in ethidium bromide tolerance. Applied Environmental Microbiology. 80 (19), 6091-6103 (2014).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods in Molecular Biology. 859, 169-176 (2012).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: Disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infections and Immunity. 76 (12), 5826-5833 (2008).
- Picardeau, M., Brenot, A., Saint Girons, I. First evidence for gene replacement in Leptospira spp. Inactivation of L. biflexa flaB results in non-motile mutants deficient in endoflagella. Molecular Microbiology. 40 (1), 189-199 (2001).
- King, A. M., et al. High-temperature protein G is an essential virulence factor of Leptospira interrogans. Infections and Immunity. 82 (3), 1123-1131 (2014).
- Lambert, A., et al. FlaA proteins in Leptospira interrogans are essential for motility and virulence but are not required for formation of the flagellum sheath. Infections and Immunity. 80 (6), 2019-2025 (2012).
- Murray, G. L., et al. Leptospira interrogans requires heme oxygenase for disease pathogenesis. Microbes and Infections. 11 (2), 311-314 (2009).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathogens. 3 (7), 97(2007).
- Sasaki, Y., et al. Leptospiral flagellar sheath protein FcpA interacts with FlaA2 and FlaB1 in Leptospira biflexa. PLoS One. 13 (4), 0194923(2018).
- Gaj, T., Gersbach, C. A., Barbas, C. F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Cress, B. F., et al. Rapid generation of CRISPR/dCas9-regulated, orthogonally repressible hybrid T7-lac promoters for modular, tuneable control of metabolic pathway fluxes in Escherichia coli. Nucleic Acids Research. 44 (9), 4472-4485 (2016).
- Zhao, C., Shu, X., Sun, B. Construction of a gene knockdown system based on catalytically inactive ("dead") Cas9 (dCas9) in Staphylococcus aureus. Applied Environmental Microbiology. 83 (12), (2017).
- Zhao, Y., et al. CRISPR/dCas9-mediated multiplex gene repression in Streptomyces. Biotechnology Journal. , 1800121(2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados