Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein automatisiertes Kultursystem zur präklinischen Erprobung wirtsgerichteter Therapien für Tuberkulose
In diesem Artikel
Zusammenfassung
Eine schnelle und effiziente Quantifizierung des intrazellulären M . tuberculosis-Wachstums ist entscheidend für verbesserte Therapien gegen Tuberkulose (TB). Dieses Protokoll beschreibt einen brühebasierten kolorimetrischen Detektionsassay unter Verwendung eines automatisierten Flüssigkultursystems zur Quantifizierung des Mtb-Wachstums in Makrophagen, die mit Wirtstherapiekandidaten behandelt werden.
Zusammenfassung
Mycobacterium tuberculosis (Mtb), der Erreger der Tuberkulose (TB), war bis zum Aufkommen von COVID-19 der weltweit bedeutendste Killer für Infektionskrankheiten. Mtb hat sich entwickelt, um in seiner intrazellulären Umgebung zu bestehen, die Abwehr des Wirts zu umgehen und hat Resistenzen gegen viele antituberkulöse Medikamente entwickelt. Ein Ansatz zur Lösung von Resistenzen besteht darin, bestehende zugelassene Medikamente zu identifizieren, die die Immunantwort des Wirts auf Mtb verstärken. Diese Medikamente könnten dann als adjunktive wirtsgerichtete Therapien (HDT) umfunktioniert werden, um die Behandlungszeit zu verkürzen und Antibiotikaresistenzen zu überwinden.
Die Quantifizierung des intrazellulären Mtb-Wachstums in Makrophagen ist ein entscheidender Aspekt bei der Beurteilung potenzieller HDT. Der Goldstandard zur Messung des Mtb-Wachstums ist das Zählen koloniebildender Einheiten (CFU) auf Agarplatten. Dies ist ein langsamer, arbeitsintensiver Assay, der sich nicht für ein schnelles Screening von Medikamenten eignet. In diesem Protokoll wurde ein automatisiertes, brühebasiertes Kultursystem, das häufiger zum Nachweis von Mtb in klinischen Proben verwendet wird, für das präklinische Screening wirtsgerichteter Therapien angepasst. Die Fähigkeit des Flüssigkultur-Assay-Systems zur Untersuchung des intrazellulären Mtb-Wachstums in Makrophagen, die mit HDT behandelt wurden, wurde bewertet. Die HDTs, die auf ihre Fähigkeit getestet wurden, das MTB-Wachstum zu hemmen, waren all-trans-Retinsäure (AtRA), sowohl in Lösung als auch in Poly(lactic-co-glycolic acid) (PLGA) -Mikropartikeln und der Kombination von Interferon-gamma und Linezolid. Zu den Vorteilen dieser automatisierten Flüssigkultur-basierten Technik gegenüber der CFU-Methode gehören die einfache Einrichtung, die weniger arbeitsintensive Vorbereitung und die schnellere Zeit bis zum Ergebnis (5-12 Tage im Vergleich zu 21 Tagen oder mehr für Agarplatten).
Einleitung
Mycobacterium tuberculosis (Mtb), der Erreger der Tuberkulose, war 2019 weltweit der bedeutendste Killer für Infektionskrankheiten1. Um der Abwehr des Wirts zu entgehen, untergräbt Mtb die mykobakterizide Aktivität von angeborenen Immunzellen wie Makrophagen und dendritischen Zellen (DCs), so dass es intrazellulär persistieren und replizierenkann 2. Das Fehlen eines wirksamen Impfstoffs zur Vorbeugung der Lungentuberkulose bei Erwachsenen und das zunehmende Auftreten arzneimittelresistenter Stämme unterstreichen den dringenden Bedarf an neuen Therapien.
Adjunktive wirtsgerichtete Therapien (HDT) könnten die Behandlungszeit verkürzen und helfen, Resistenzen zu überwinden3. Die präklinische Bewertung von HDT-Kandidaten in vitro zur Bestimmung der mykobakteriziden Aktivität in Makrophagen beruht häufig auf der Quantifizierung des Mtb-Wachstums durch koloniebildende Einheiten (CFU) auf festen Agarplatten. Dies ist ein langsamer, arbeitsintensiver Assay, der sich nicht für ein schnelles Screening von Medikamenten eignet. Kommerziell erhältliche automatisierte, brühebasierte mikrobielle Nachweissysteme werden häufiger in klinischen mikrobiologischen Laboratorien für den Nachweis und die Prüfung der Arzneimittelempfindlichkeit von Mtb und anderen mykobakteriellen Spezies in klinischen Proben eingesetzt4. Diese Instrumente messen das Wachstum indirekt auf der Grundlage der bakteriellen Stoffwechselaktivität, die zu physikalischen Veränderungen in den Kulturmedien (Veränderung des CO2- oderO2-Spiegels oder-Drucks) führt, die im Laufe der Zeit überwachtwerden 5. Der Messwert ist Time to Positivity (TPP), von dem zuvor gezeigt wurde, dass sie mit der Mtb-CFU in Sputumproben von TB-Patienten als Reaktion auf Behandlung 6,7 und in Lysaten infizierter muriner Lunge und Milz8 korreliert. Darüber hinaus wurden Flüssigkultur-Detektionssysteme verwendet, um die Wirkung konventioneller pathogengesteuerter Therapien auf das Wachstum von Mykobakterien in axenischen Kulturen und kultivierten Makrophagen zu messen 9,10. Das Instrument wurde auch verwendet, um die angeborene Fähigkeit von dendritischen Zellen und von Alveolarmakrophagen zu untersuchen, das intrazelluläre Wachstum von Mtb11,12 zu kontrollieren. Dieses experimentelle Protokoll zeigt, dass ein Flüssigkultur-Diagnosesystem angepasst werden kann, um ein präklinisches Screening von HDT auf TB in kultivierten Makrophagen durchzuführen. Im Vergleich zur CFU-Aufzählung besteht der Hauptvorteil dieser Technik darin, dass sie den experimentellen Arbeitsaufwand und die Zeit, die zur Quantifizierung des intrazellulären mykobakteriellen Wachstums / Überlebens erforderlich sind, erheblich reduziert. Diese Technik beruht auf dem Zugang zu einem automatisierten Kulturinstrument, mit dem das Überleben intrazellulärer Mykobakterien in Immunzellen beurteilt werden kann, die mit einer breiten Palette pharmakologischer Reagenzien behandelt wurden, die auf zelluläre Funktionen abzielen, um die Immunität des Wirts zu stärken.
Protokoll
Die in diesem Protokoll beschriebenen Experimente wurden mit dem abgeschwächten H37Ra-Stamm von Mtb durchgeführt, der in einem Containment Level 2-Labor gehandhabt werden kann. Alle Manipulationen an lebenden Mykobakterien wurden in einer biologischen Sicherheitswerkbank der Klasse II (BSC) durchgeführt. Experimentelle Verfahren wurden entwickelt, um die Erzeugung von Aerosolen zu minimieren. Eukaryotische Zellkultur (THP-1-Zellen) wurde ebenfalls in einer BSC der Klasse II durchgeführt. Das Labor führte eine Risikobewertung durch und stellte sicher, dass alle Verfahren in Übereinstimmung mit den institutionellen und nationalen biologischen Sicherheitsvorschriften durchgeführt wurden. Die humane monozytäre THP-1-Zelllinie wurde verwendet, um die Methode wie beschrieben durchzuführen (Schritt 1). Die Zellen werden nach Stimulation mit Phorbol-12-Myristat-13-Acetat (PMA) vor der Infektion mit Mykobakterien in Makrophagen differenziert.
1. Zellkultur
- Vermehrung von H37Ra-Samen in die Log-Phase in Middlebrook 7H9 (MB) Brühe, ergänzt mit Albumin-Dextrose-Katalase (ADC) Anreicherung (10%) und 0,05% Polysorbat 80. Lagern Sie H37Ra-Vorräte in 1 ml Aliquots in einem -80 °C Gefrierschrank für bis zu 1 Jahr.
- Eine 1-ml-Durchstechflasche mit Mtb-H37Ra auftauen und etwa 1 Woche vor dem geplanten Experiment in einen T25-Kolben mit einer Filterkappe mit 9 ml MB-ergänzter Brühe geben. Inkubieren Sie bei 37 °C für 5-7 Tage in einem statischen Inkubator.
- THP-1-Zellen in RPMI-1640, ergänzt mit nicht hitzeabgetötetem 10% fetalem Kälberserum (vollständig (c)RPMI) in einem T75-Kolben in einemCO2-befeuchteten Inkubator bei 5% CO2/37 °C und Subkultur zweimal pro Woche, um eine Dichte von weniger als 1 x 106 Zellen/ml aufrechtzuerhalten.
- Differenzieren Sie THP-1-Zellen 3 Tage vor der Infektion in Makrophagen, indem Sie die Zellen mehrmals mit einer serologischen Pipette in T75-Kolben vorsichtig pipettieren, um Klumpen zu dispergieren und in ein 50-ml-konisches Röhrchen zu legen.
- Zentrifugieren Sie die Zellen bei 300 x g für 10 min bei Raumtemperatur, dekantieren Sie den Überstand ab und resuspendieren Sie das Pellet vorsichtig in 2 ml RPMI. Führen Sie die Zellzählung durch, um die Zellen / ml zu schätzen.
- Saatgut 2 ml THP-1-Makrophagen in 12-Well-Gewebekulturplatten bei einer Dichte von 100.000 Zellen/ml in cRPMI mit 100 ng/ml PMA für 72 h. Entfernen Sie PMA-haltiges Medium aus den Zellen und füllen Sie es vor der MTB-Infektion mit frischem cRPMI auf.
- Richten Sie individuelle Platten für jeden gewünschten Zeitpunkt ein.
- Samenzellen mit der gleichen Dichte (100.000 Zellen / ml) in 2-Well-Glaskammerobjektträgern, um die Multiplizität der Infektion (MOI) zu bestimmen.
- In einen 5%CO2 befeuchteten Inkubator für 3 Tage bei 37 °C geben.
2. Quantifizierung der MTB-Aufnahme
- Bestimmung der Mtb-Aufnahme durch Makrophagen (MOI)
- Richten Sie die biologische Sicherheitswerkbank der Klasse II (BSC) am Tag der Infektion ein und arbeiten Sie an zwei Schichten Seidenpapier, um verschüttete Flüssigkeiten aufzufangen. Stellen Sie Abfallentsorgungsbehälter gemäß den örtlichen Vorschriften auf.
- Aus dem T25-Kolben werden 6-8 ml Mykobakterienkultur entnommen und in ein 15-ml-Polypropylenröhrchen überführt.
HINWEIS: Röhrchen mit kleinerem Volumen können für kleinere Experimente verwendet werden. - Zentrifugieren Sie das Röhrchen in einer Tischzentrifuge bei Raumtemperatur für 10 min bei 2890 x g.
- Nehmen Sie das Röhrchen vorsichtig aus der Zentrifuge und bringen Sie es in die biologische Sicherheitswerkbank. Warten Sie 1 Min., damit sich die Bakterien ansiedeln können.
- Gießen Sie den Überstand in den Desinfektionsmittel-Entsorgungsbehälter, verschließen Sie das Röhrchen und resuspendieren Sie die Bakterien im verbleibenden Medium, indem Sie auf die Seite des Röhrchens klopfen. Warten Sie 1 Min., damit sich die Bakterien ansiedeln können.
- Fügen Sie 2 ml vorgewärmtes cRPMI hinzu, mischen Sie vorsichtig und geben Sie es in ein 50-ml-konisches Röhrchen.
- Resuspendieren Sie die Mykobakterien sehr vorsichtig mit einer 25 G Nadel und 5 ml Spritze. Um sie zu resuspendieren, ziehen Sie die Mykobakteriensuspension in die Spritze und werfen Sie sie sehr vorsichtig an der Seitenwand des Röhrchens aus, um die Aerosolproduktion zu minimieren. Wiederholen Sie 6-8 mal.
HINWEIS: Seien Sie äußerste Vorsicht, da es sich um eine hochdichte Kultur von Mykobakterien handelt. Um das Risiko einer Nadelstichverletzung zu vermeiden, verwenden Sie, wenn möglich, stumpfe Nadeln und Luer-Lock-Spritzen. - Entsorgen Sie die Nadel und die Spritze in einem scharfen Behälter im BSC.
- Die Suspension in ein 2-ml-Mikrofugenröhrchen (mit Schraubverschluss) geben und bei Raumtemperatur 3 min bei 100 x g zentrifugieren, um verbleibende Klumpen zu pelletieren. Bringen Sie das Röhrchen in die Sicherheitswerkbank zurück und warten Sie 1 Min., bis sich die Bakterien ansiedeln können.
- Übertragen Sie die oberen 1-1,5 mL des Überstands in ein neues Röhrchen. Entsorgen Sie das Originalrohr im Abfalleimer mit Desinfektionsmittel. Gut mischen und verschiedene Mengen der mykobakteriellen Suspension (z. B. 5, 25, 50, 150 μL) in die 2-Well-Glaskammerobjektträger geben und 3 h in einem CO2-Inkubator bei 37 °C inkubieren.
- Färbung für säurefeste Bakterien (AFB)
HINWEIS: Nach 3 h Inkubation werden die Makrophagen gewaschen und mit Paraformaldehyd fixiert, um Mykobakterien zu inaktivieren. Die Objektträger werden dann mit einem modifizierten Auramine O-Kit (siehe Materialtabelle) gefärbt, um die Phagozytosierung von Mykobakterien pro Zelle zu schätzen. Aufgrund ihrer wachsartigen Zellwand behalten Mykobakterien den Auramin-Farbstoff nach einer Säure-Alkohol-Wäsche. Die Makrophagenkerne werden dann mit Hoechst gegengefärbt. Diese Methode ermöglicht es, die Anzahl der phagozytierten Bakterien pro Zelle zu zählen und wird verwendet, um die Multiplizität der Infektion (MOI) der Makrophagen zu bestimmen.- Entfernen Sie das Medium aus dem Glaskammerobjektträger, nachdem Sie dreimal auf und ab pipettiert haben, um Bakterien zu entfernen, die nicht phagozytiert wurden.
- Einmal mit 2 mL PBS bei Raumtemperatur waschen.
- Lagerbestände von 4% Paraformaldehyd, gelöst in PBS in Aliquoten bei -20 °C für bis zu 6 Monate. Ein Aliquot von 4% Paraformaldehyd unmittelbar vor Gebrauch auftauen. Mit PBS auf 2% Paraformaldehyd verdünnen und 2 ml pro Vertiefung hinzufügen.
- 10 min bei Raumtemperatur inkubieren. Der Glaskammerobjektträger kann in dieser Phase zur Färbung aus der Sicherheitswerkbank entnommen werden.
- Waschen Sie die Rutsche unter einem sanften Leitungswasserstrahl.
- Geben Sie genügend Auramin auf den Objektträger, um die Zellen mit einer Kunststofftransferpipette abzudecken, und inkubieren Sie für 1 min bei Raumtemperatur im Dunkeln (Abdeckung mit Aluminiumfolie).
- Waschen Sie überschüssigen Farbstoff mit Leitungswasser vom Objektträger und fügen Sie den Entfärbungsmittel / Quencher für 1 min im Dunkeln hinzu.
- Den Überschuss mit Leitungswasser abwaschen und 15 min bei Raumtemperatur mit Hoechst 33342 (10 μg/ml in PBS) im Dunkeln inkubieren.
- Den Hoechst-Fleck mit Leitungswasser abwaschen, die Kammern entfernen, überschüssiges Wasser vom Objektträger ablassen, einen Tropfen Antifade und Deckglas hinzufügen und an der Luft trocknen.
- Untersuchen Sie den Objektträger unter dem Fluoreszenzmikroskop mit dem 100-fachen Ölobjektiv. Mykobakterien fluoreszieren unter dem FITC-Filter grün. Die Kerne fluoreszieren blau unter dem DAPI-Filter (Abbildung 1C).
- Bestimmen Sie das MOI, indem Sie die Anzahl der pro Zelle phagozytierten Mykobakterien und den Prozentsatz der infizierten Zellen zählen.
- Berechnen Sie das Volumen der mykobakteriellen Suspension, die benötigt wird, um die erforderliche MOI basierend auf der Oberfläche einer Vertiefung in der Platte zu erreichen. Zum Beispiel beträgt die Oberfläche der in diesem Experiment verwendeten Glaskammerobjektträger 4 cm2. Ein niedriger MOI (ca. 1-2 Bazillen/Zelle) ist wünschenswert für Experimente, die über mehrere Tage (z.B. 5 Tage) durchgeführt werden.
- Infektion von Makrophagen
- Mischen Sie die Mykobakteriensuspension gut und fügen Sie den Zellen die benötigte Menge auf 12-Well-Platten hinzu, sobald das Volumen bestimmt wurde, das erforderlich ist, um den gewünschten MOI zu erreichen.
- Bei 37 °C für 3 h inkubieren, damit Mykobakterien phagozytiert werden können.
- Entfernen Sie extrazelluläre Bakterien, indem Sie die Vertiefungen mehrmals mit warmem RPMI oder HBSS waschen.
- Lysieren Sie die Makrophagen in einer Vertiefung (3-Stunden-Probe), um die prozentuale Zeit bis zur Positivität (TTP) des ursprünglichen Inokulums (3-Stunden-Probe) zu bestimmen, wie in Schritt 3 unten beschrieben.
- Frisches cRPMI und die erforderlichen Medikamentendosen oder Vehikelkontrolle in die verbleibenden Vertiefungen geben, die Platten imCO2-Inkubator bei 37 °C für die erforderliche Zeit inkubieren (abhängig vom Versuchsplan, aber in der Regel in mehreren Abständen zwischen 1 und 8 Tagen).
3. Entnahme von Proben für das Flüssigkultur-Detektionssystem
HINWEIS: Am Tag der Infektion werden extrazelluläre Mykobakterien durch Waschen entfernt, und intrazelluläre Mykobakterien werden durch Lyse einer Vertiefung von Makrophagen (3 h Probe) geerntet, um die anfängliche Menge zu bestimmen, die als Ausgangskontrolle für die Infektion phagozytiert wird. Zu folgenden Zeiten werden sowohl das Medium, das Zelllysat als auch die Waschungen kombiniert, um das gesamte mykobakterielle Wachstum zu messen. Extrazelluläres und intrazelluläres Wachstum können auf Wunsch auch getrennt beurteilt werden.
- Entnahme einer 3-stündigen Probe zur Bestimmung der TTP
- Nach den ersten 3 Stunden der Infektion werden extrazelluläre Mykobakterien aus allen Vertiefungen abgewaschen, wie in Schritt 2.3.3 beschrieben. Fügen Sie 1 ml frische Medien in die 3-Stunden-Kontrollmulde hinzu, um das Lysatvolumen mit denen späterer Zeitpunkte auszugleichen.
HINWEIS: Siehe Schritt 3.2.7, wenn extrazelluläre Mykobakterien von der Analyse ausgeschlossen werden sollen.
- Nach den ersten 3 Stunden der Infektion werden extrazelluläre Mykobakterien aus allen Vertiefungen abgewaschen, wie in Schritt 2.3.3 beschrieben. Fügen Sie 1 ml frische Medien in die 3-Stunden-Kontrollmulde hinzu, um das Lysatvolumen mit denen späterer Zeitpunkte auszugleichen.
- Musterkollektion
- Warme MB-Brühe und Instrumentenkulturflaschen, um sie auf Raumtemperatur zu bringen.
- Überführen Sie das Medium von der 12-Well-Platte in die entsprechenden beschrifteten konischen Rohre.
- 500 μL steriler Lysepuffer (0,1% Triton x-100 in PBS, gefiltert durch einen sterilen 0,2 μm Filter) für 10 min in jede Vertiefung geben.
- Die Zellen vorsichtig mit einem sterilen Schaber aus dem Vertiefung abkratzen und mit dem Medium im entsprechenden konischen Röhrchen verbinden.
- Waschen Sie die Vertiefung mit 0,5 ml sterilem PBS und geben Sie sie in das entsprechende Röhrchen.
- Führen Sie jede Probe 6-8 Mal vorsichtig durch eine Nadel und Spritze (25 G), um die Klumpen aufzubrechen. Verdünnte Proben 1:10 in MB-Brühe; 100 μL Probe + 900 μL MB Medium.
- Zu den erforderlichen Zeiten/Tagen (in der Regel zwischen 1 und 8 Tagen) werden die restlichen Proben mit den Schritten 3.2.1-3.2.6 entnommen.
HINWEIS: Forscher ziehen es möglicherweise vor, extrazelluläre Mykobakterien von ihrer Analyse auszuschließen, in diesem Fall wird in Schritt 3.2.2 oben das Medium aus jeder Vertiefung verworfen und Makrophagen werden mehrmals gewaschen, bevor Lysepuffer hinzugefügt wird.
- Beimpfung und Beladung von Instrumentenkulturflaschen
HINWEIS: Details zum Flüssigkulturgerät und den zugehörigen Verbrauchsmaterialien finden Sie in der Materialtabelle.- Sterilisieren Sie den Gummiverschluss der Instrumentenkulturflasche mit Seidenpapier, das mit 70% Alkohol getränkt ist, und lassen Sie es an der Luft trocknen.
HINWEIS: Dieser Schritt muss im BSC durchgeführt werden. - Bereiten Sie die Flaschen vor, indem Sie genügend Nährstoffergänzungen für alle Proben in ein konisches Röhrchen (0,5 ml / Flasche) geben. Verwenden Sie eine Nadel und eine Spritze, um 0,5 ml Nährstoffergänzung in die Instrumentenkulturflasche zu injizieren.
- 500 μL der verdünnten Probe (1:10) werden in ein 1 ml V-Bodenröhrchen pipettiert.
- Verwenden Sie eine Nadel und eine Spritze, um die 500 μL Probe in die zugewiesene Instrumentenkulturflasche zu injizieren.
- Sterilisieren Sie den Gummiverschluss der Instrumentenkulturflasche mit Seidenpapier, das mit 70% Alkohol getränkt ist, und lassen Sie es an der Luft trocknen. Wischen Sie die Flaschen mit Seidenpapier ab, das mit 70% Alkohol getränkt ist, bevor Sie sie aus dem BSC entfernen.
- Transportieren Sie die Flaschen vorsichtig von der Biosicherheitswerkbank zum Beladungsinstrument.
- Drücken Sie die Ladetaste am automatisierten mikrobiellen Detektionssystem.
- Scannen Sie die Barcodes auf Instrumentenkulturflaschen und legen Sie die Flaschen bis zu 42 Tage lang bei 37 °C in den Inkubator des Detektionssystems. Lesen und notieren Sie die Zeit, die benötigt wird, um eine Positivität vom Instrumentenbildschirm aus zu erreichen.
HINWEIS: Der Barcode ermöglicht es dem Gerät, die Flasche zu identifizieren und Reflexionswerte mit einer bestimmten Flasche zu verknüpfen. - Berechnen Sie die prozentuale Zeit bis zur Positivität (TTP), indem Sie die TTP des anfänglichen intrazellulären Inokulums (Tag 0) mit der von Makrophagen vergleichen, die für die angegebenen Zeiten kultiviert wurden. Eine positive Veränderung des prozentualen TTP bedeutet mykobakterielles Wachstum13.
Zum Beispiel für Tag 3:
Prozentuale Veränderung der Zeit bis zur Positivität =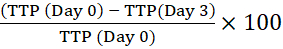
- Sterilisieren Sie den Gummiverschluss der Instrumentenkulturflasche mit Seidenpapier, das mit 70% Alkohol getränkt ist, und lassen Sie es an der Luft trocknen.
Ergebnisse
Das in dieser Studie verwendete automatisierte Flüssigkulturgerät überwacht alle 10 min den CO2 -Gehalt. Eine Farbänderung im Sensor am Boden der Instrumentenflasche wird farbmetrisch gemessen und als Reflexionseinheiten ausgedrückt. Die Gerätesoftware wendet dann Detektionsalgorithmen an, um die Zeit bis zur Positivität (TTP) zu berechnen, d. H. Die Anzahl der Tage von der Inokulation bis zur Markierung der Kulturen als positiv (Abbildung 1A). Eine umgekehrte Beziehung zwi...
Diskussion
Die Autoren haben die in diesem Protokoll beschriebene Flüssigkulturmethode verwendet, um das Mtb-Wachstum in Monozyten-abgeleiteten Makrophagen und Alveolarmakrophagen und THP-1-Zellen zu überwachen, die mit PMA11,16,17 differenziert sind. Diese Technik kann auch für die Verwendung mit nicht adhärenten Zellenmodifiziert werden 12. In jüngerer Zeit wurde das Instrument auch in präklinischen Studien ...
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Arbeit wurde von der Science Foundation Ireland (SFI 08/RFP/BMT1689), dem Health Research Board in Irland [HRA-POR/2012/4 und HRA-POR-2015-1145] und dem Royal City of Dublin Hospital Trust finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| IX51 Fluorescent Microscope | Olympus, Japan | N/A | AFB detection and imaging |
| 2 mL microtube, flat bottom, screw cap, sterile | Sarstedt, North Carolina, USA | 72.694.006 | Mtb infection of macropahges |
| 5 mL syringe, Luer lock | BD Biosciences, San Jose, CA, USA | SZR-150-031K | Mtb infection of macropahges/CFU |
| 50 mL tube, sterile | Sarstedt, North Carolina, USA | 62.547.254 | Mtb infection of macropahges |
| all-trans-Retinoic Acid (ATRA) ≥98% (HPLC) | Sigma Aldrich, Missouri, USA | R2625 | Host directed therapy candidate |
| BacT/ALERT 3D Microbial Detection System | Biomerieux ( Hampshire, UK) | 247001 | Broth-based colormetric detection system |
| BACT/ALERT MP BACT/ALERT MP Nutrient Supplement | Biomerieux ( Hampshire, UK) | 414997 | Broth-based colormetric detection assay |
| BACT/ALERT MP culture bottles | Biomerieux ( Hampshire, UK) | 419744 | Broth-based colormetric detection assay |
| BD BBL Middlebrook ADC Enrichment, 20 mL | BD Biosciences, San Jose, CA, USA | M0553 | Mycobacterium liquid culture |
| BD BBL Middlebrook OADC Enrichment, 20mL | BD Biosciences, San Jose, CA, USA | M0678 | Colony Forming Units |
| Cell scraper, 25 cm | Sarstedt, North Carolina, USA | 83.1830 | Harvest of lmacrophage lysates |
| Corning Syringe Filter, 0.2 µm | Corning Incorporated, Germany | 431219 | Sterilization of lysis buffer |
| Cover glass (borosilicate), 24 x 50 mm, #1.5 thickness | VWR International Limited | 631 - 0147 | |
| Cycloheximide, from microbial | Sigma Aldrich, Missouri, USA | C7698 | Colony Forming Units |
| Dako Fluorescent Mounting Medium | Agilent Technologies Ireland Limited | S3023 | Antifade mounting medium |
| Dulbecco’s Phosphate Buffered Saline | Sigma Aldrich, Missouri, USA | D8537 | Mtb infection of macropahges |
| Fetal Bovine Serum, Gibco | Thermo Fisher, Massachusetts, USA | 10270106 | Macrophage cell culture |
| Glycerol, Difco | BD Biosciences, San Jose, CA, USA | 228220 | Colony Forming Units |
| Hoescht 33342 (bisBenzimide H 33342 trihydrochloride) | Sigma Aldrich, Missouri, USA | B2261 | Nuclear stain |
| IFNγ, recombinant human | R&D Systems Inc, Minnesota, USA | 285-IF | Host directed therapy candidate |
| Labtek 2-well chamber slide, sterile, Nunc | Thermo Fisher, Massachusetts, USA | TKT-210-150R | Mtb infection of macropahges |
| L-Asparagine, anhydrous | Sigma Aldrich, Missouri, USA | A4159 | Colony Forming Units |
| Linezolid | Sigma Aldrich, Missouri, USA | PZ0014 | Antibiotic |
| Microlance Hypodermic Needle 25 G | BD Biosciences, San Jose, CA, USA | 300400 | Mtb infection of macropahges/CFU |
| Middlebrook 7H10 Agar Base | BD Biosciences, San Jose, CA, USA | M0303 | Colony Forming Units |
| Middlebrook 7H9 Broth Base | BD Biosciences, San Jose, CA, USA | M0178 | Mycobacterium liquid culture |
| Modified Auramine O Stain and Decolourizer | Scientific Device Laboratory, IL, USA | 345-250 | AFB stain |
| Paraformaldehyde | Sigma Aldrich, Missouri, USA | 158127 | Mtb infection of macropahges |
| Petri dishes, 92 x 16mm (20/bag) | Sarstedt, North Carolina, USA | 82.1473.001 | Colony Forming Units |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma Aldrich, Missouri, USA | P8139 | Macrophage cell culture |
| Polysorbate 80, Difco | BD Biosciences, San Jose, CA, USA | 231181 | Mycobacterium liquid culture |
| RPMI-1640, Gibco | Thermo Fisher, Massachusetts, USA | 52400025 | Macrophage cell culture |
| Sterile Cell Spreader, L-Shaped | Fisherbrand, Thermo Fisher, MA, USA | RB-44103 | Colony Forming Units |
| T25 TC flask, angled neck, filter cap, sterile, Nunc | Thermo Fisher, Massachusetts, USA | 156367 | Mycobacterium liquid culture |
| THP-1 cell line | ATCC, Virginia, USA | ATCC TIB-202 | Macrophage cell culture |
Referenzen
- WHO. Global Tuberculosis Report. World Health Organization. , (2020).
- Russell, D. G. Mycobacterium tuberculosis and the intimate discourse of a chronic infection. Immunological Reviews. 240 (1), 252-268 (2011).
- Young, C., Walzl, G., Du Plessis, N. Therapeutic host-directed strategies to improve outcome in tuberculosis. Mucosal Immunology. 13 (2), 190-204 (2020).
- Angeby, K. A., et al. Evaluation of the BacT/ALERT 3D system for recovery and drug susceptibility testing of Mycobacterium tuberculosis. Clinical Microbiology and Infection. 9 (11), 1148-1152 (2003).
- Asmar, S., Drancourt, M. Rapid culture-based diagnosis of pulmonary tuberculosis in developed and developing countries. Frontiers in Microbiology. 6, 1184 (2015).
- Diacon, A. H., et al. Time to liquid culture positivity can substitute for colony counting on agar plates in early bactericidal activity studies of antituberculosis agents. Clinical Microbiology and Infection. 18 (7), 711-717 (2012).
- Diacon, A. H., et al. Time to positivity in liquid culture predicts colony forming unit counts of Mycobacterium tuberculosis in sputum specimens. Tuberculosis. 94 (2), 148-151 (2014).
- O'Sullivan, D. M., et al. Evaluation of liquid culture for quantitation of Mycobacterium tuberculosis in murine models. Vaccine. 25 (49), 8203-8205 (2007).
- Heinrichs, M., et al. Mycobacterium tuberculosis Strains H37ra and H37rv have equivalent minimum inhibitory concentrations to most antituberculosis drugs. International Journal of Mycobacteriology. 7 (2), 156-161 (2018).
- Sorrentino, F., et al. Development of an intracellular screen for new compounds able to inhibit mycobacterium tuberculosis growth in human macrophages. Antimicrobial Agents and Chemotherapy. 60 (1), 640-645 (2016).
- O'Leary, S. M., et al. Cigarette smoking impairs human pulmonary immunity to Mycobacterium tuberculosis. American Journal of Respiratory and Critical Care Medicine. 190 (12), 1430-1436 (2014).
- Ryan, R. C., O'Sullivan, M. P., Keane, J. Mycobacterium tuberculosis infection induces non-apoptotic cell death of human dendritic cells. BMC Microbiology. 11, 237 (2011).
- Keane, J., Remold, H. G., Kornfeld, H. Virulent mycobacterium tuberculosis strains evade apoptosis of infected alveolar macrophages. The Journal of Immunology. 164 (4), 2016-2020 (2000).
- Gao, X. F., Yang, Z. W., Li, J. Adjunctive therapy with interferon-gamma for the treatment of pulmonary tuberculosis: a systematic review. International Journal of Infectious Diseases. 15 (9), 594-600 (2011).
- Thorpe, T. C., et al. BacT/Alert: an automated colorimetric microbial detection system. Journal of Clinical Microbiology. 28 (7), 1608-1612 (1990).
- Gleeson, L. E., et al. Cutting edge: Mycobacterium tuberculosis induces aerobic glycolysis in human alveolar macrophages that is required for control of intracellular bacillary replication. The Journal of Immunology. 196 (6), 2444-2449 (2016).
- O'Connor, G., et al. Inhalable poly(lactic-co-glycolic acid) (PLGA) microparticles encapsulating all-trans-Retinoic acid (ATRA) as a host-directed, adjunctive treatment for Mycobacterium tuberculosis infection. European Journal of Pharmaceutics and Biopharmaceutics. 134, 153-165 (2019).
- O'Connor, G., et al. Sharpening nature's tools for efficient tuberculosis control: A review of the potential role and development of host-directed therapies and strategies for targeted respiratory delivery. Advanced Drug Delivery Reviews. 102, 33-54 (2016).
- Rodriguez, D. C., Ocampo, M., Salazar, L. M., Patarroyo, M. A. Quantifying intracellular Mycobacterium tuberculosis: An essential issue for in vitro assays. Microbiologyopen. 7 (2), 00588 (2018).
- Ortalo-Magne, A., et al. Molecular composition of the outermost capsular material of the tubercle bacillus. Microbiology. 141, 1609-1620 (1995).
- Stokes, R. W., et al. The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages. Infection and Immunity. 72 (10), 5676-5686 (2004).
- O'Sullivan, M. P., O'Leary, S., Kelly, D. M., Keane, J. A caspase-independent pathway mediates macrophage cell death in response to Mycobacterium tuberculosis infection. Infection and Immunity. 75 (4), 1984-1993 (2007).
- Cheon, S. H., et al. Bactericidal activity in whole blood as a potential surrogate marker of immunity after vaccination against tuberculosis. Clinical and Diagnostic Laboratory Immunology. 9 (4), 901-907 (2002).
- Nathan, C., Barry, C. E. TB drug development: immunology at the table. Immunological Reviews. 264 (1), 308-318 (2015).
- Bowness, R., et al. The relationship between Mycobacterium tuberculosis MGIT time to positivity and cfu in sputum samples demonstrates changing bacterial phenotypes potentially reflecting the impact of chemotherapy on critical sub-populations. Journal of Antimicrobial Chemotherapy. 70 (2), 448-455 (2015).
- Basu Roy, R., et al. An auto-luminescent fluorescent BCG whole blood assay to enable evaluation of paediatric mycobacterial responses using minimal blood volumes. Frontiers in Pediatrics. 7, 151 (2019).
- Christophe, T., et al. High content screening identifies decaprenyl-phosphoribose 2' epimerase as a target for intracellular antimycobacterial inhibitors. PLoS Pathogens. 5 (10), 1000645 (2009).
- Franzblau, S. G., et al. Comprehensive analysis of methods used for the evaluation of compounds against Mycobacterium tuberculosis. Tuberculosis. 92 (6), 453-488 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten